Method Article
שחזור פעילות החילוץ Msp1 עם רכיבים מטוהרים לחלוטין
In This Article
Summary
כאן, אנו מציגים פרוטוקול מפורט לשיקום פעילות החילוץ Msp1 עם רכיבים מטוהרים לחלוטין בפרוטואוליפוזומים מוגדרים.
Abstract
כמרכז לזרחן חמצוני ורגולציה אפופטוטית, המיטוכונדריה ממלאת תפקיד חיוני בבריאות האדם. תפקוד מיטוכונדריאלי תקין תלוי במערכת בקרת איכות חזקה לשמירה על הומאוסטזיס חלבון (פרוטאוסטזיס). ירידות בפרוטוסטזיס מיטוכונדריאלי נקשרו לסרטן, הזדקנות, ניוון עצבי, ומחלות רבות אחרות. Msp1 הוא ATPase AAA+ המעוגן בקרום המיטוכונדריאלי החיצוני השומר על פרוטאוסטזיס על ידי הסרת חלבונים מעוגנים בזנב. באמצעות רכיבים מטוהרים מחדש לתוך proteoliposomes, הראינו כי Msp1 הוא הכרחי מספיק כדי לחלץ חלבון מעוגן זנב מודל מן bilayer השומנים. המערכת המחודשת הפשוטה שלנו מתגברת על כמה מהמחסומים הטכניים שהפריעו למחקר מפורט של מיצוי חלבון ממברנה. כאן, אנו מספקים שיטות מפורטות ליצירת ליפוזומים, שחזור חלבון ממברנה, ובחאיית החילוץ Msp1.
Introduction
תפקוד תאי תקין תלוי בתהליך הנקרא פרוטאוסטזיס, המבטיח כי חלבונים פונקציונליים נמצאים בריכוז הנכון ובמיקום התאי1. כשלים בפרוטאוסטזיס מובילים לתפקוד אברונים נפגעים והם קשורים למחלות ניווניות רבות2,3,4. חלבוני ממברנה מציגים אתגרים ייחודיים לרשת הפרוטאוסטזיס מכיוון שהם חייבים להיות ממוקדים לממברנה הנכונה תוך הימנעות מצבירה מתחומי הטרנס-מברן ההידרופוביים (TMDs)5. כתוצאה מכך, מכונות מיוחדות התפתחו כדי להגן על TMD הידרופובי מן cytosol ולהקל על מיקוד והכנסת לתוך הממברנה התאית הנכונה6,7,8,9,10,11,12,13,14,15.
המיטוכונדריה הם הרכזת המטבולית של התא והם מעורבים בתהליכים תאיים חיוניים רבים כגון: זרחן חמצוני, ייצור אשכול גופרית ברזל, ורגולציה אפופטוטית16,17. אברונים אנדוזימביוטיים אלה מכילים שתי ממברנות, המכונות הממברנה המיטוכונדריאלית הפנימית (IMM) והממברנה המיטוכונדריאלית החיצונית (OMM). מעל 99% מתוך 1,500 חלבונים מיטוכונדריאליים אנושיים מקודדים בגנום הגרעיני ויש להעבירם על פני ממברנה אחת או שתיים שונות18,19. תפקוד מיטוכונדריאלי תקין ולכן תלוי ברשת פרוטאוסטזיס חזקה כדי לתקן שגיאות כלשהן פילוח חלבון או translocation.
המעבדה שלנו מתמקדת בתת-קבוצה של חלבונים ממברנה מיטוכונדריאלית הנקראים חלבונים מעוגנים בזנב (TA), שיש להם תחום טרנס-מברן יחיד בטרמינל C20,21,22,23,24. חלבוני ת"א מעורבים במספר תהליכים חיוניים, כגון אפופטוזיס, הובלת ארסיה, וטרנסלוקציה של חלבונים25. הטופולוגיה הייחודית של חלבוני ת"א דורשת החדרה לאחר התרגום, המתרחשת ברטיקולום אנדופלסמי (ER) על ידי הכניסה המודרכת של זנב מעוגן (GET) או רטיולמוסית קרום חלבון קומפלקס (EMC) או לתוך OMM על ידי מסלול מאופיין בצורה גרועה20,26,27,28. המאפיינים הביופיזיים של TMD נחוצים ומספיקים כדי להנחות חלבוני ת"א לממברנה הנכונה29. ההכרה במאפיינים ביופיסיים ולא במוטיב רצף מוגדר מגבילה את הנאמנות של מסלולי הפילוח5. לכן, הקצאה שגויה של חלבוני ת"א היא מתח נפוץ עבור רשתות פרוטאוסטזיס. מתח תאי, כגון עיכוב של מסלול GET, גורם לעלייה באילוץ חלבונים ל- OMM ולתפקוד מיטוכונדריאלי אלא אם כן חלבונים אלה מוסרים מייד30,31.
נושא נפוץ בפרוטוסטזיס ממברנה הוא השימוש ב- AAA+(TPase Aהמנוקד לחלבונים Activities תאיים) להסרת חלבונים ישנים, פגומים או לא מאוטצים מהלבייה השומנים1,32,33,34,35,36,37,38 . חלבוני AAA+ הם מנועים מולקולריים היוצרים טבעות הקסמריות ועוברים תנועות תלויות ATP כדי לשפץ מצע, לעתים קרובות על ידי טרנסלוקציה באמצעות נקבובית צירית צרה39,40. למרות מאמץ רב הוקדש לחקר החילוץ של חלבוני ממברנה על ידי AAA + ATPases, reconses הם מורכבים או כרוכים תערובת של שומנים וחומרי ניקוי41,42, אשר מגביל את הכוח הניסיוני לבחון את המנגנון של מיצוי מצע מן bilayer השומנים.
Msp1 הוא AAA + ATPase שמור מאוד המעוגן ב- OMM וב- peroxisomes הממלא תפקיד קריטי בפרוטוסטזיס של הממברנה על ידי הסרת חלבוני ת"א לא מאותרים43,44,45,46,47. Msp1 הוכח לאחרונה גם כדי להקל על לחץ ייבוא חלבון מיטוכונדריאלי על ידי הסרת חלבונים ממברנה לעכב במהלך טרנסלוקציה על פני OMM48. אובדן Msp1 או ההומולוגיה האנושית ATAD1 גורם לפיצול מיטוכונדריאלי, כשלים בזרחן חמצוני, התקפים, פציעה מוגברת בעקבות שבץ, ומוות מוקדם31,49,50,51,52,53,54,55,56.
הראינו כי ניתן לשחזר חלבוני ת"א עם Msp1 ולזהות את החילוץ מ bilayer השומנים57. מערכת פשוטה זו משתמשת בחלבונים מטוהרים לחלוטין המחודשים לליפוזומים מוגדרים המחקים את ה- OMM (איור 1)58,59. רמה זו של שליטה ניסיונית יכולה לענות על שאלות מכניות מפורטות של מיצוי מצע כי הם עקשניים מבחינה ניסיונית עם שחזורים מורכבים יותר מעורבים חלבונים אחרים AAA + . כאן, אנו מספקים פרוטוקולים ניסיוניים המפרטים את השיטות שלנו להכנת ליפוזום, שחזור חלבון ממברנה, ובחאיית החילוץ. תקוותנו היא כי פרטים ניסיוניים אלה יאפשרו מחקר נוסף של תהליך חיוני אך לא מובן היטב של פרוטאוסטזיס ממברנה.
Protocol
1. הכנת ליפוזום
- שלב מלאי כלורופורם של שומנים ביחסים מתאימים כדי לחקות את הממברנה המיטוכונדריאלית החיצונית.
- הכן 25 מ"ג של תערובת שומנים. אנו משתמשים בתערובת שהוקמה בעבר של שומנים המחקים ממברנות מיטוכונדריאליות, מורכב 48:28:10:10:4 יחס טוחנת של ביצת עוף פוספטידיל כולין (PC), ביצת עוף פוספטידיל אתנולמין (PE), כבד בקר פוספטידיל אינוזיטול (PI), סינתטי 1,2-דיאולאויל-sn-גליצריו-3-פוספו-L-serine (DOPS), וסינתטי 1',3'-bis[1,2-דיאולאויל-sn-גליצרי-3-פוספו]-גליצריול (TOCL)58,59. חישובים לדוגמה מוצגים בטבלה 1.
- הביאו את כל מלאי השומנים לטמפרטורת החדר לפני הפתיחה מכיוון שזה יגביל את ה עיבוי. כמו רוב המעבדות אין דרך מדויקת למדוד את הריכוז של השומנים, כל מים נספגים על ידי מלאי כלורופורם ישנה את הריכוז של מלאי השומנים ובכך את היחס של שומנים המשמשים הבדיקה.
- כמו מניות שומנים מגיעים אמפולות זכוכית, להעביר את הכמות הנדרשת של שומנים ל בקבוקון זכוכית באמצעות מזרק 1 מ"ל. הוסף 2 מ ג של dithiothreitol (DTT) למצופה כדי למנוע חמצון שומנים בדם. לעבוד במהירות כמו אידוי של כלורופורם ישנה את הריכוז של השומנים.
- מעבירים את כל השומנים הנותרים ל בקבוקון זכוכית נפרד ומתאימה לספטה PFTE. הוסף 2 מ"ג של DTT למצופה, לעטוף עם parafilm ולאחסן ב -20 °C כדי למנוע חמצון שומנים בדם. נסה להשתמש בליפידים בתוך 3 חודשים של העברה בקבוקונים. כדי למנוע זיהום פוטנציאלי של מלאי כלורופורם על ידי נגר סמן, להעביר את המדבקות מן אמפולות המקוריות בקבוקונים זכוכית במקום תווית עם סמן.
- לאדות כלורופורם תחת זרם עדין מאוד של חנקן תוך ספינינג בקבוקון הזכוכית ברציפות ביד במכסה המנוע אדים, למעשה מתנהג כמו rotovap ידני. יש לסובב את הוויאל במהירות עקבית (20-40 סל"ד) ביד כדי לשמור על השומנים נעים. המטרה היא לאדות את כל הכלורופורם ולקבל ציפוי אחיד של שומנים מעל בקבוקון הזכוכית כולו.
זהירות: כלורופורם הוא neurotoxic, צעד זה צריך להתבצע במכסה המנוע אדים.- חבר פיפטת פסטר טרייה לצינור החנקן. אין לתת לאף אחד מהשומנים להתיז מהחניון או על פיפטת פסטר. כוון את הקצה בתחתית הקרבון כך שהאוויר יקפוץ מהתחתית ודוחף את השומנים למעלה לכיוון מרכז הנקניקיון. קבל ציפוי אחיד על כל הקרבון תוך הימנעות מכל הצטברות בפינות או על ידי הכובע. כל התהליך הזה לוקח בערך 5 דקות.
- כמו התערובת מתעבה לתוך "חרוז" של שומנים, להנחות אותו למרכז של המשחקון על ידי שינוי זווית של המשחקון. ברגע החרוז מתחיל להיות קטן יותר, להפוך את זרם החנקן מעט כדי לפזר את החרוז, להבטיח כי אף אחד השומנים לפוצץ את המשחקון.
- הסר כל כלורופורם שנותר תחת ואקום.
- שים את בקבוקון הזכוכית על ואקום הבית או משאבת ואקום הסרעפת במשך שעה אחת כדי להסיר את רוב כלורופורם שיורית. שואבי אבק אלה הם בדרך כלל לא חזקים מספיק כדי להסיר את כל כלורופורם, אבל הם יכולים לסבול כמויות קטנות של ממס טוב יותר מאשר שואבי אבק סיבובי-לשווא.
- שים בקבוקון על ואקום חזק (<1 mTorr) במשך 12-16 שעות כדי להסיר את כלורופורם שיורית. הקפד להימנע מחבטה של המשחקון במהלך תהליך זה.
- resuspend השומנים ב 1.25 מ"ל של חוצץ ליפוזום (50 mM HEPES KOH pH 7.5, 15% גליצל, 1 mM DTT). כפי שהתחלנו עם 25 מ ג של שומנים, התוצאה היא ריכוז של 20 מ"ג/מ"ל. יש להזרים מחדש את השומנים ללא נתחים נראים לעין. אם השומנים התאגדו בפינת הנקניקיון, זה יכול להיות תהליך ממושך.
- מערבולת את הנקניקיה במרץ עד המדגם הוא חלבי חלק. אנו מוצאים כי אם אידוי כלורופורם נעשה כראוי בלילה הקודם, תהליך זה לוקח בערך 5-10 דקות.
- כדי להבטיח התחדשות מלאה של השומנים, לסובב על גלגל בטמפרטורת החדר במשך 3 שעות ב ~ 80 סל"ד. הסר את הנקינה מהגלגל פעם בשעה במשך דקה אחת של מערבולת כדי להבטיח ערבוב אחיד.
- מעבירים שומנים בזהירות לצינור מיקרוצנטריפוגה נקי של 1.5 מ"ל. בצע 5 מחזורי הפשרת הקפאה באמצעות חנקן נוזלי להקפאה ובלוק חום 30 °C להפשיר. שלב זה מסייע בהמרת שלל רב-לשוני לשלשלדים חד-צדדיים.
- לתפאר את השומנים.
- במהלך מחזורי ההפשרה של ההקפאה, הכינו את המיני-מחול. להרכיב את המיני-מבליט עם תמיכה מסנן 10 מ"מ וקרום פוליקרבונט של גודל הנקבבובית הרצוי (אנו משתמשים 200 ננומטר). גודל המסנן ישפיע על גודל הליפוזום, אשר ישפיע על ריכוז החלבונים הנדרשים לשיקום (שלב 2.3).
- מניחים את המיני-מחול על צלחת חמה ומביאים את טמפרטורת המחול עד 60 מעלות צלזיוס.
- ציירו את השומנים למזרק זכוכית הדוק בגז 1 מ"ל והכניסו בזהירות לקצה אחד של המיני-מחשוף. מניחים את המזרק הריק עם גז בצד השני של המיני-מחול. אפשר השומנים כדי שיווי משקל לטמפרטורת מעמד extruder במשך 5-10 דקות.
- מעבירים את השומנים למזרק החלופי על ידי דחיפת הבוכנה של המזרק המלא. לדחוף את הפתרון מהמזרק החלופי לתוך המזרק המקורי. חזור על תהליך הלוך ושוב 15 פעמים, כך שב-15עובר השומנים מסתיימים במזרק החלופי. נטר את עוצמת הקול בכל מעבר כדי לוודא שאין דליפות.
- הכן aliquots לשימוש יחיד של השומנים, להקפיא פלאש חנקן נוזלי ולאחסן ב -80 °C (70 °F). הליפוזומים יציבים ב -80 מעלות צלזיוס במשך מספר חודשים. השיקום דורש 10 μL של ליפוזומים בכל פעם(שלב 2.3.4),כך שזה נוח להכין 10 μL או 20 aliquots μL.
2 שחזור של חלבון Msp1 ומודל ת"א
- הכן את חיץ השיקום: 50 mM HEPES pH 7.5, 200 mM אשלגן אצטט, 7 mM מגנזיום אצטט, 2 mM DTT, 10% סוכרוז, 0.01% נתרן אזיד, 0.2-0.8% Deoxy Big Chaps (DBC).
- מטב את תנאי השיקום עבור אצווה חדשה של ליפוזומים. הריכוז של DBC ו biobeads הנדרשים עבור שחזור אופטימלי משתנה בהתאם אצווה של ליפוזומים בשימוש. כדי להגביל את ההכנה לשינוייות הכנה, השתמש באותו חלק של DBC עבור כל הניסויים. בעת שינוי של DBC רב, חזור על תהליך המיטוב.
- הגדר סדרה של שחזורים עם ריכוזים שונים של DBC (0.2% - 0.8%) ו biobeads (25 מ"ג - 100 מ"ג) בכל פעם קבוצה חדשה של ליפוזומים מוכן. חשוב לא להוריד את ה-DBC מתחת לריכוז המיצל הקריטי (CMC) של ~0.12%. לאחר התנאים ממוטבים, אנו ממליצים לאסוף את כל הנתונים באמצעות אותם תנאי הכנה ושיקום ליפוזום.
- תבטחו את האפקטיביות של תנאי השיקום השונים באמצעות בדיקות החילוץ המתוארות בשלב 3.
- הכן ביו-פדים בריכוז סופי של 250 מ"ג/מ"ל.
- שוקלים 2.7 גרם של יבלות ביולוגיות מיובשות ומניחים מחדש בצינור צנטריפוגה של 50 מ"ל של 100% מתנול (כ-45 מ"ל) כדי להרטיב את החרוזים. בתחילה הרטיב את הביצים במתנול כדי למנוע מהאוויר להילכד בנקבוביות החרוזים. ברגע במתנול, לשמור על biobeads רטוב כמו כל אוויר לכוד על ידי biobeads ישנה את יכולתם לספוג חומר ניקוי.
- הסר מתנול על ידי שטיפת החרוזים 8x עם כ 45 מ"ל של מים אולטרה-תול (18.2 mΩ), להלן המכונה ddH2O. חרוזים פלטה על ידי ספינינג ב 3,200 x גרם במשך 1 דקה. דקאנט את הנוזל ו resuspend ב ddH2O.
- לאחר הכביסה, resuspend ב 10 מ"ל של ddH2O עם 0.02% נתרן אזיד ולאחסן ב 4 °C (70 °F). Biobeads ניתן לאחסן ב 4 °C (5 °F) במשך כמה חודשים. מלאי זה הוא 250 מ"ג / מ"ל כפי שהוא הניח ~ 0.2 גרם אבד במהלך שלבי לשטוף.
- לחשב את גודל הליפוזום ואת המספר הרצוי של מולקולות של חלבון ת"א ו- Msp1 לכל ליפוזום. זה יקבע את הריכוז של חלבון Msp1 ו- TA הנדרש לשיקום.
- ראשית, לחשב את מספר מולקולות השומנים לכל ליפוזום חד-צדדי (Ntotal) באמצעות המשוואה
 שבה d הוא הקוטר של הליפוזום, h הוא עובי הדו-שכבתית, ו- a הוא אזור קבוצת הראש של השומנים.
שבה d הוא הקוטר של הליפוזום, h הוא עובי הדו-שכבתית, ו- a הוא אזור קבוצת הראש של השומנים.- למדוד את קוטר הליפוזום על ידי DLS. בדוגמה שלנו, ערך של 70 ננומטר עבור קוטר ליפוזום (d) הושג.
- השתמש בערך של 5 ננומטר עבור h ו- 0.71 nm2 עבור, שהוא גודל קבוצת הראש עבור פוספטידילכולין. במצב מסוים זה, Nבסך הכל הוא 37,610.
- לאחר מכן, לחשב את הריכוז הטוחן של שומנים מלומיד M ליפיד באמצעות המשקל המולקולרי הממוצע של השומנים בתערובת. בדוגמה זו, הריכוז של שומנים הוא 20 מ"ג / מ"ל (שלב 1.4) ואת המשקל המולקולרי הממוצע של שומנים הוא 810 גרם / מול (טבלה 1). התוצאה היא ערך של 0.0247 M עבור Mליפיד.
- לאחר מכן, לחשב את הריכוז הטוחן של ליפוזומים, MLiposome, באמצעות המשוואה
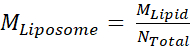 שבה M Lipid הוא ריכוז טוחנות של שומנים מ שלב 2.3.2, ו- Ntotal הוא המספר הכולל של שומנים לליפוזום מחושב בשלב 2.3.1. בדוגמה זו, ריכוז מלאי 20 מ"ג /מ"ל של ליפוזומים הוא כ 660 ננומטר.
שבה M Lipid הוא ריכוז טוחנות של שומנים מ שלב 2.3.2, ו- Ntotal הוא המספר הכולל של שומנים לליפוזום מחושב בשלב 2.3.1. בדוגמה זו, ריכוז מלאי 20 מ"ג /מ"ל של ליפוזומים הוא כ 660 ננומטר. - חשב את כמות חלבון Msp1 ו- TA הנדרש לתגובה של 100 μL לחזרה.
הערה: הריכוז הסופי של שומנים בשחזור הוא 2 מ"ג / מ"ל, שהוא דילול 10x של מלאי ליפוזום. זה נותן ריכוז ליפוזום סופי של 66 ננומטר. הריכוז הסופי של Msp1 הוא 792 ננומטר, אשר נותן בממוצע 12 עותקים סה"כ (2 hexamers פונקציונלי) לכל ליפוזום. הריכוז הסופי של חלבון ת"א הוא 660 ננומטר, אשר נותן בממוצע של 10 עותקים לכל ליפוזום.
- ראשית, לחשב את מספר מולקולות השומנים לכל ליפוזום חד-צדדי (Ntotal) באמצעות המשוואה
- בצינור PCR, יש לערבב יחד Msp1 מטוהר, חלבון ת"א וליפוזומים במאגר שחזור. סדר התוספת הוא חיץ, חלבונים וליפוזומים אחרונים. הנפח הכולל הוא 100 μL. אפשר לתערובת לשבת על קרח במשך 10 דקות. לטהר את חלבון Msp1 ו- TA כפי שתואר בעבר57.
- השתמשו בחלבון ת"א המאופיין היטב בחלבון The-Flag-Sumo-Sec22 כמצע שליטה חיובי בעת הקמת ה-assay לראשונה. למבנה זה יש תג שלו לטיהור קל, תג דגל 3x לזיהוי על ידי כתם מערבי, תחום סומו למסיסות מוגברת, ואת TMD של חלבון ER-TA Sec22 לשיקום והכרה על ידי Msp1.
- ודא כי פתרונות מלאי של Msp1 וחלבון ת"א הם כ 100 מיקרומטר כדי למזער את ההשפעה של N-דודציל β-D-maltoside (DDM) מטיהור החלבון על reconstitution. Msp1 מטוהר הוא 20 mM HEPES pH 7.5, 100 mM נתרן כלורי, 0.1 mM טריס (2-קרבוקסיתיל)פוספין הידרוכלוריד (TCEP), 0.05% DDM ואילו חלבון TA המטוהר הוא 50 mM טריס pH 7.4, 150 mM נתרן כלורי, 10 mM מגנזיום כלוריד, 5 mM β-mercaptoethanol, 10% גליצריול, 0.1% DDM. פתרונות מלאי של כ-100 מיקרומטר ת"א ומונומר Msp1 מבטיחים שרכיבים אלה ירכיבו <-5% מנפח השיקום הסופי, וכתוצאה מכך דילול DDM מתחת ל-CMC.
- מוסיפים את הכמות הרצויה של biobeads לדגימה כדי להסיר חומר ניקוי.
- חותכים את הקצה של קצה פיפטה p200 לקוטר של כ -1/8 אינץ ', כך חרוזים יכולים להתאים דרך הקצה. מערבולת צינור biobeads ביסודיות כדי לקבל תערובת אחידה במהירות להסיר את המכסה ואת pipette את הנפח לפני biobeads להתיישב. העבר את ה-biobeads לצינור פי.סי.אר ריק.
- כאשר השיקום סיים את הדגירה של 10 דקות על קרח, השתמש בקצה פיפטה לא מלוטש כדי להסיר את כל הנוזל מהביורופדים. ואז להעביר את 100 μL שיקום לתוך הצינור עם biobeads. זה חייב להיעשות במהירות, כך biobeads לא ללכוד אוויר, אשר יגרום החרוזים לצוף.
- אפשר לחזרה להסתובב על גלגל במהירות של כ-80 סל"ד למשך 16 שעות ב-4 מעלות צלזיוס.
- הסר חומר מחודש מ biobeads. לעשות סיבוב מהיר picofuge כדי כדורי biobeads, ולאחר מכן להשתמש קצה צינור לא חתוך להעביר את החומר reconstituted לצינור PCR נקי. חזור על תהליך זה 1-2 פעמים עד שלא יישארו ביו-פדים במדגם. שמור על שיקום על קרח.
- יש לנקות מראש את החומר המחודש כדי להסיר חלבונים שלא הצליחו לשחזר את הליפוזומים.
- הכנת חיץ מיצוי: 50 mM HEPES pH 7.5, 200 mM אשלגן אצטט, 7 מ"מ מגנזיום אצטט, 2 mM DTT, 100 ננומטר סידן כלורי.
- ישילב את עמודות הספין של גלוטתיון עם מאגר החילוץ בהתאם להוראות היצרן. זה בדרך כלל כרוך 3 סיבובים של כביסה עם 400 μL של חוצץ ולאחר מכן centrifuging ב 700 x g במשך 2 דקות בטמפרטורת החדר כדי להסיר את המאגר.
- הוסף 5 מיקרומטר של כל מלווה (GST-SGTA ו- GST-Calmodulin) לחומר המחודש. המלווים האלה ייקשרו ל-TMD של חלבונים שלא הצליחו לשחזר את הליפוזומים. טיהור המלווים תואר בעבר6,57.
הערה: מלווים אלה זמינים מסחרית, אך אנו מעדיפים לטהר בבית כדי לשלוט בעלות ובאיכות. שני החלבונים הם 20 mM טריס pH 7.5, 100 mM נתרן כלורי, ו 0.1 mM TCEP, ויש להם ריכוז מלאי סביב 160 מיקרומטר. SGTA מזהה מצעים עם TMD הידרופובי מאוד ואילו Calmodulin קושר TMDs עם הידרופוביה מתונה6,29. יחד, קוקטייל המלווה הזה יכול לזהות מגוון רחב של מצעים. - הוסף 100 μL של חוצץ החילוץ לחומר reconstituted, להביא את הנפח עד 200 μL. הוסף את זה לעמודות ספין גלוטתיון המשווות. שים לב כי עמודות ספין גלוטתיון לספק את ההתאוששות המדגם הגבוה ביותר כאשר עוצמת הקול לפני ניקוי הוא 200 – 400 μL.
- חבר את עמודות הסיבוב וסובב ב- ~ 80 סל"ד ב 4 °C (30 °F) למשך 30 דקות כדי לאפשר למלווים להיקשר לשרף.
- סובבו את העמודים ב-700 x גרם למשך 2 דקות בטמפרטורת החדר. הזרימה דרך היא חומר מנוקה מראש כי הוא מרוקן של חלבונים מצטברים. שמור חומר על קרח ולהמשיך ישירות עם מיצוי assay.
3. אסיית חילוץ
- הכן צינורות לניתוח דף SDS. לכל תגובה יהיו 4 צינורות: קלט (I), FLOW דרך (FT), לשטוף (W) ו- ELUTE (E).
- הוסף 45 μL של ddH2O מדגם צינור הכניסה, 40 μL של ddH2O לצינור FLOW דרך, ו 0 μL צינורות לשטוף ו ELUTE.
הערה: יחס האות לרעש הטוב ביותר בבחיטה זו מתקבל כאשר דגימות ה- WASH ו- ELUTE מרוכזות פי 5 ביחס לדגימות קלט וזרימה. בשל דילול במהלך ההסתייגות, זה דורש לקחת 5 μL של מדגם עבור מדגם קלט, 10 μL של מדגם FLOW דרך המדגם, ו 50 μL של מדגם עבור דגימות לשטוף ו- ELUTE. - הוסף 16.6 μL של 4x SDS עמוד טעינה מאגר לכל צינור. אמצעי האחסון הכולל של כל דגימה הוא 50 μL לפני מאגר טעינת עמוד SDS. אמצעי האחסון הסופי הוא 66.6 μL (50 μL מדגם + 16.6 μL של 4x SDS עמוד טעינה מאגר).
- הוסף 45 μL של ddH2O מדגם צינור הכניסה, 40 μL של ddH2O לצינור FLOW דרך, ו 0 μL צינורות לשטוף ו ELUTE.
- להרכיב את מעש החילוץ.
- הכן את תגובת החילוץ המכילה 60 μL של פרוטאוליפוזומים שנוקו מראש, 5 מיקרומטר של GST-SGTA, 5 מיקרומטר של GST-Calmodulin, ו 2 mM ATP. שלב את כל הריאגנטים למעט ATP, אשר משמש כדי ליזום את התגובה. להביא לנפח סופי של 200 μL עם מאגר החילוץ.
הערה: כמו 60 μL של מדגם משמשים עבור כל מחוק, שחזור אחד יכול לשמש עבור שלוש התקפות מיצוי שונות. בצע פקדים חיוביים ושליליים (+ATP ו- -ATP) בחומר מאותה שחזור. - מיצוי מראש חם assay בלוק חום 30 °C במשך 2 דקות.
- הכן את תגובת החילוץ המכילה 60 μL של פרוטאוליפוזומים שנוקו מראש, 5 מיקרומטר של GST-SGTA, 5 מיקרומטר של GST-Calmodulin, ו 2 mM ATP. שלב את כל הריאגנטים למעט ATP, אשר משמש כדי ליזום את התגובה. להביא לנפח סופי של 200 μL עם מאגר החילוץ.
- ליזום את ההסתה החילוץ על ידי הוספת ATP לריכוז הסופי של 2 mM ושעון התזמון להתחיל.
- תן סיבוב של 5 שניות בפיקופוגה כדי לערבב ATP לתוך התגובה. תגובת דגירה ב 30 °C (50 °F) במשך 30 דקות.
- במהלך הדגירה, לקחת 5 μL של התגובה ולהוסיף צינור קלט. התזמון של זה הוא גמיש.
- במהלך תקופת הדגירה הזו, יש לכלול עמודת ספין גלוטתיון אחת עבור כל דגימה בבחינת החילוץ.
- בצע משיכה כלפי מטה על מלווים כדי לבודד חומר שחולץ.
- לאחר הדגירה של 30 דקות הוא סיים, להוסיף 200 μl של חוצץ החילוץ לצינור כדי להביא נפח כולל ל 400 μL. מוסיפים לשרף גלוטתיון ישוב ומאפשרים לאגד על ההגה ב 4° C במשך 30 דקות.
- סובב את העמודים ב 700 x g במשך 2 דקות בטמפרטורת החדר כדי לאסוף את הזרימה דרך. קח 10 μL עבור צינור זרימה דרך. מדגם זה מכיל מצעים אשר עדיין משולבים bilayer השומנים.
- לשטוף שרף פעמיים עם 400 μL של חוצץ החילוץ, להשליך את הזרימה דרך. על לשטוף השלישי, לשמור על הזרימה דרך ולקחת 50 μL עבור צינור לשטוף.
- הכן 5 מ"ל של מאגר אלוטיון על ידי הוספת גלוטתיון מופחת לריכוז סופי של 5 מ"מ במאגר החילוץ. הכן מאגר זה טרי בכל פעם.
- הוסף 200 μL של מאגר Elution לעמודת הסיבוב. דגירה בטמפרטורת החדר במשך 5 דקות. יש לסובב ב-700 x גרם למשך 2 דקות בטמפרטורת החדר כדי לחמוק. שמור על הזרימה דרך. חזור על התהליך בפעם השנייה כך שנפח ההתחמקות הכולל הוא 400 μL.
- קח 50 μL של מדגם Elution ולהוסיף אותו צינור ELUTE.
- נתח פעילות חילוץ באמצעות SDS-PAGE וכתם מערבי.
הערה: כמו כתם מערבי הוא הליך סטנדרטי למדי, פרוטוקול בסיסי מסופק המדגיש כמה פרטים ייחודיים זה.- טען דגימות לתוך ג'ל פוליאקרילמיד ללא כתמים (4% ערימה, 15% הפרדה) ולהפעיל ב 200 V במשך 50 דקות במאגר Tris-גליצין. אם החלל מאפשר זאת, השתמש בסולם לא מזוהם ומוכתם כדי לאפשר הדמיה של דימוי ג'ל ללא כתמים והעברה לקרום PVDF בהתאמה. הג'ל נטול הכתמים מאפשר הדמיה כמותית של טריפטופן המכיל חלבונים עם הפעלה עם אור אולטרה סגול, ועדיין מאפשר את השימוש בג'ל לכתם מערבי.
- תמונה ג'ל ללא כתמים כדי לאשר כי יש טעינה שווה על פני כל הדגימות. זוהי בקרה חיונית המבטיאה שכל שינוי באות שזוהה על ידי כתם מערבי אינו תוצאה של טעינת חלבון משתנה. צריכות להיות רק להקות גלויות עבור המלווים (GST-calmodulin ו- GST-SGTA) בדגימות קלט ואלוטיון. זכור כי מדגם ELUTE יהיה מרוכז יותר מדגם הקלט.
- להרכיב קלטת כתם מערבי באמצעות קרום PVDF 45 מיקרומטר. מעבירים בזרם קבוע של 300 mA למשך 60 דקות.
- לאחר חסימת הממברנה, להיקשר נוגדן העיקרי עבור 16 שעות ב 4 °C (55 °F) עם רועד עדין ~ 15 סל"ד. כתם למצע עם ארנב אנטי-דגל בדילול 1:1,000.
הערה: הנוגדנים העיקריים והמשניים בהם ייעשה שימוש יהיה ספציפי למצע וייתכן שיהיה צורך למטב את הריכוז לשימוש. - לשטוף קרום ודגורה עם נוגדן משני, נגד ארנב עזים ב 1:10,000 דילול, עם רועד עדין במשך שעה בטמפרטורת החדר.
- לשטוף קרום ותמונה לניתוח באמצעות סוכן זיהוי סופג מערבי.
תוצאות
כדי לפרש כראוי את התוצאות, יש לראות יחד את הג'ל נטול הכתם ואת הכתם המערבי. הג'ל ללא כתמים מבטיח טעינה שווה בכל הדגימות. בעת צפייה בג'ל ללא כתמים, המלווים (GST-calmodulin ו- GST-SGTA) יהיו גלויים בנתיבי קלט (I) ו- ELUTE (E). בדוק שוב כי עוצמת הרצועות האלה אחידה על פני כל דגימות הקלט. כמו כן, ודא כי העוצמה אחידה על פני דגימות ELUTE. ELUTE מרוכז פי 5 מהקלט וההבדל הזה בעוצמה יהיה גלוי בג'לים.
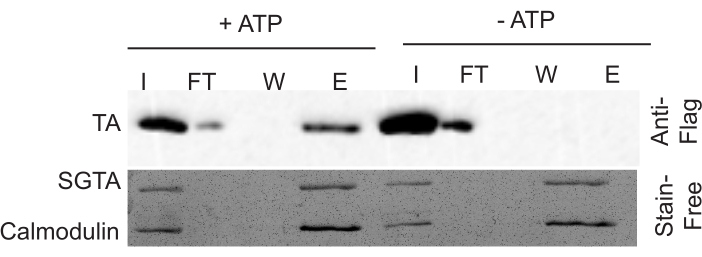
לאחר השימוש בג'ל ללא כתמים כדי לאשר טעינה נכונה, לבחון את כתם המערבי כדי לקבוע את פעילות החילוץ. מדוד פעילות חילוץ על-ידי השוואת כמות המצע בשבר ELUTE (E) ביחס לשבר קלט (I). האות בזרימה דרך (FT) מראה שונות מסוימת, אך דומה בדרך כלל לשבר הקלט. לא אמור להיות אות בשבר 2008 (W). בדרך כלל, יש ~ 10% יעילות החילוץ עבור השליטה החיובית ויעילות החילוץ 1-2% בבקרה השלילית (איור 2). זכור כי שבר ELUTE הוא 5x אינטנסיבי כמו שבר קלט, ולכן זה צריך להילקח בחשבון בעת שיפוט יעילות החילוץ. אם תנאי השיקום אינם ממוטבים, בדרך כלל יש רמות מיצוי דומות הן בדגימות + ATP והן בדגימות ATP (איור 3). תוצאה זו מיוחסת לכישלון של Msp1 כדי לשחזר ביעילות, וכתוצאה מכך proteoliposomes רבים ללא hexamer Msp1 פונקציונלי.
| השומנים | מול % | MW | ממוצע של MW | מיקרומול ב-25 מ"ג | מ"ג ב-25 מ"ג | מלאי כלור (מ"ג/מ"ל) | μL עבור 25 מ"ג |
| מחשב אישי | 48% | 770 | 369.6 | 14.82 | 11.41 | 25 | 456.4 |
| PE | 28% | 746 | 208.88 | 8.64 | 6.45 | 25 | 258.0 |
| PI | 10% | 902 | 90.2 | 3.09 | 2.78 | 10 | 278.5 |
| DOPS | 10% | 810 | 81 | 3.09 | 2.50 | 10 | 250.1 |
| TOCL | 4% | 1502 | 60.08 | 1.23 | 1.85 | 25 | 74.2 |
| קון קור אבג MW | 809.76 | ||||||
| מיקרומול ב-25 מ"ג | 30.87 | ||||||
טבלה 1: חישובים לדוגמה להכנת ליפוזום. המטרות העיקריות של טבלה זו הן לחשב את הריכוז מתוקן משקל מולקולרי ממוצע של תערובת השומנים ואת הנפח של כל מלאי שומנים נדרש כדי להפוך את ליפוזומים. MW וריכוז מלאי מגיעים מתוויות מוצרים. שומנים ושומה % נבחרים על ידי המשתמש.

איור 1: קריקטורה של בדיקת חילוץ ורשימת שלבי מפתח. אנא לחץ כאן כדי להציג גרסה גדולה יותר של דמות זו.
איור 2: נתונים מייצגים המראים בדיקת ישבן מתפקדת כראוי. יעילות החילוץ נקבעת על-ידי השוואת כמות המצע בשבר ELUTE לשבר הקלט. זכור כי הג'ל יש טעינה גבוהה פי 5 של שבר ELUTE ביחס לשבר הקלט. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 3: נתונים מייצגים של תגמולים כושל ובחלוץ. כאן, הפעילות במדגם + ATP דומה לפעילות במדגם ATP. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
Discussion
תפקוד מיטוכונדריאלי תקין תלוי במערכת בקרת איכות חלבון חזקה. בשל מגבלות מובנות בנאמנות של מסלולי פילוח חלבון ת"א, חלבוני ת"א מאושפזים הם מקור מתמיד ללחץ למיטוכונדריה. מרכיב מרכזי ברשת הפרוטאוסטזיס המיטוכונדריאלי הוא Msp1, שהוא ATPase מעוגן AAA+ המודרת שמסיר חלבוני ת"א לא מאוסים מה- OMM. כאן, תיארנו כיצד להכין פרוטאוליפוזומים, שיתוף reconstitute Msp1 וחלבון ת"א מודל, ולבצע מבחן מיצוי. השתמשנו בעבר בבדיקה זו כדי להדגים כי Msp1 מזהה ישירות חלבוני ת"א שלא הוקצו כהלכה ומסוגל לחלץ חלבונים אלה משכבת שומנים ללא חלבוני אביזר או קופקטורים57.
החיסרון של ההסתה הוא שיש שונות מסוימת בתהליך השיקום. כדי לשלוט על השתנות ההכנה-להכנה, אנו תמיד כוללים שליטה חיובית ושלילית על אותו כתם ג'ל / מערבי כדי להבטיח כי בדיקת ה- assay שלנו פועלת כמתוכנן. אנו נמנעים מלעשות השוואות בין שחזורים שנעשו בימים שונים או לצייר השוואות בין כתמים מערביים שונים. ההשוואות היחידות שאנו עושים הן עבור דגימות משוחזרות במקביל ומנוהלות על אותו כתם ג'ל / מערבי. אנחנו גם די שמרנים בפרשנות הנתונים שלנו. למרות שניתן לכמת את יעילות החילוץ באמצעות ImageJ, אנו מתארים בדרך כלל את הניסויים שלנו כבעלי פעילות מלאה, פעילות ביניים או ללא פעילות.
מקור אחד של שונות הוא הכמות הכוללת של חלבון מחדש. בעוד שרוב החלבונים הלא מאוגדים מוסרים על ידי תהליך הפינוי מראש, יעילות ההשבה הפחות מושלמת של Msp1 יכולה להיות בעלת השפעה גדולה על היעילות הנצפית של מיצוי מצע. השפעה זו נובעת מהעובדה כי Msp1 מתפקד כמו homohexamer, אבל מטהר כמו מונומר57. ליפוזומים המכילים כל דבר מלבד 6x עותקים של Msp1 לא יהיו פעילים. לדוגמה, ליפוזום עם 5 עותקים בלבד של Msp1 לא יהיה פעיל. הדרך היחידה ליצור משושים Msp1 יציבים באורך מלא היא לנטרל פעילות ATPase עם אנלוגים ATP שאינם ניתנים ל hydrolyzable (ATPγS) או מוטציה E193Q ווקר B לא פעיל, אף אחד מהם אינם תואמים עם פעילות assay. התגברות על משוכה טכנית זו היא תחום של מחקר פעיל במעבדה שלנו.
תחום נוסף של מחקר פעיל מתמקד בהפיכת ההסתה של החילוץ כמותית יותר. השיטה הנוכחית מסתמכת על משיכות כלפי מטה וכתמים מערביים לזיהוי אותות, שניהם רק חצי כמותיים ומציגים בדיקה כדי לבדוק שונות. שינוי קוולנטי של מצעים שחולצו יבטל את השונות הנובעת מהירידה. כמו כן, שימוש בתוויות רדיואקטיביות או פלואורסצנטיות על מצעים יבטל את הצורך בכתמים מערביים ואת השונות הנלווית.
כוח מרכזי של הבדיקה הוא שהמערכת מוגדרת לחלוטין. חלבוני הממברנה מתבטאים ומטוהרים באופן רקומביננטי וניתן ליצור מוטציות מוגדרות הן ב- Msp1 והן במצע כדי לחקור היבטים ספציפיים של התגובה. תפקידה של סביבת השומנים בפרוטוסטזיס זכה להתעלמות רבה בשל האתגרים הטכניים של לימוד זה באופן מפורט. כי הבדיקה שלנו משתמשת ליפוזומים עם הרכב שומנים מוגדר, זה מאפשר שליטה ניסיונית מלאה של סביבת השומנים. אנו יכולים בקלות לווסת גורמים כגון: נזילות שומנים בדם, עובי bilayer, זהות קבוצת ראש, וגודל ליפוזום. אנו פועלים באופן פעיל כדי להשתמש בבדיקה שלנו כדי לבחון את התפקיד של סביבת השומנים על פעילות Msp1. אנו מקווים כי ההשבה במבחנה ובחיזות המתוארת כאן יכולה לשמש כמערכת מודל פשוטה לחקר התהליך התאי הנפוץ של AAA + ATPase מיצוי בתיווך של חלבוני ממברנה מ bilayer שומנים.
Disclosures
ללא
Acknowledgements
MLW פיתח חלק מפרוטוקול זה במהלך לימודי הפוסט דוקטורט שלו עם ד"ר רוברט קינן באוניברסיטת שיקגו.
עבודה זו ממומנת על ידי מענק NIH 1R35GM137904-01 ל- MLW.
Materials
| Name | Company | Catalog Number | Comments |
| Biobeads | Bio-Rad | 1523920 | |
| Bovine liver phosphatidyl inositol | Avanti | 840042C | PI |
| Chicken egg phosphatidyl choline | Avanti | 840051C | PC |
| Chicken egg phosphatidyl ethanolamine | Avanti | 840021C | PE |
| ECL Select western blotting detection reagent | GE | RPN2235 | |
| Filter supports | Avanti | 610014 | |
| Glass vial | VWR | 60910L-1 | |
| Glutathione spin column | Thermo Fisher | PI16103 | |
| Goat anti-rabbit | Thermo Fisher | NC1050917 | |
| Mini-Extruder | Avanti | 610020 | |
| Polycarbonate membrane | Avanti | 610006 | 200 nM |
| PVDF membrane | Thermo Fisher | 88518 | 45 µM |
| Rabbit anti-FLAG | Sigma-Aldrich | F7245 | |
| Synthetic 1,2-dioleoyl-sn-glycero-3-phospho-L-serine | Avanti | 840035C | DOPS |
| Synthetic 1',3'-bis[1,2-dioleoyl-sn-glycero-3-phospho]-glycerol | Avanti | 710335C | TOCL |
| Syringe, 1 mL | Norm-Ject | 53548-001 | |
| Syringe, 1 mL, gas-tight | Avanti | 610017 |
References
- Song, J., Herrmann, J. M., Becker, T. Quality control of the mitochondrial proteome. Nature Reviews Molecular Cell Biology. 22, 54-70 (2021).
- Phillips, B. P., Miller, E. A. Membrane protein folding and quality control. Current Opinion in Structural Biology. 69, 50-54 (2021).
- Jiang, H. Quality control pathways of tail-anchored proteins. Biochimica et Biophysica Acta - Molecular Cell Research. 1868, 118922 (2020).
- McKenna, M. J., et al. The endoplasmic reticulum P5A-ATPase is a transmembrane helix dislocase. Science. 369, (2020).
- Hegde, R. S., Zavodszky, E. Recognition and Degradation of Mislocalized Proteins in Health and Disease. Cold Spring Harbor Perspectives in Biology. 11, 033902 (2019).
- Shao, S., Hegde, R. S. A calmodulin-dependent translocation pathway for small secretory proteins. Cell. 147, 1576-1588 (2011).
- Samuelson, J. C., et al. YidC mediates membrane protein insertion in bacteria. Nature. 406, 637-641 (2000).
- Anghel, S. A., McGilvray, P. T., Hegde, R. S., Keenan, R. J. Identification of Oxa1 Homologs Operating in the Eukaryotic Endoplasmic Reticulum. Cell Reports. 21, 3708-3716 (2017).
- Aviram, N., et al. The SND proteins constitute an alternative targeting route to the endoplasmic reticulum. Nature. 540, 134-138 (2016).
- Voorhees, R. M., Hegde, R. S. Structure of the Sec61 channel opened by a signal sequence. Science. 351, 88-91 (2016).
- Cichocki, B. A., Krumpe, K., Vitali, D. G., Rapaport, D. Pex19 is involved in importing dually targeted tail-anchored proteins to both mitochondria and peroxisomes. Traffic. 19, 770-785 (2018).
- Mateja, A., et al. Protein targeting. Structure of the Get3 targeting factor in complex with its membrane protein cargo. Science. 347, 1152-1155 (2015).
- Chacinska, A., Koehler, C. M., Milenkovic, D., Lithgow, T., Pfanner, N. Importing mitochondrial proteins: machineries and mechanisms. Cell. 138, 628-644 (2009).
- Chitwood, P. J., Hegde, R. S. An intramembrane chaperone complex facilitates membrane protein biogenesis. Nature. , (2020).
- Chitwood, P. J., Juszkiewicz, S., Guna, A., Shao, S., Hegde, R. S. EMC Is Required to Initiate Accurate Membrane Protein Topogenesis. Cell. 175, 1-30 (2018).
- Bock, F. J., Tait, S. W. G. Mitochondria as multifaceted regulators of cell death. Nature Reviews Molecular Cell Biology. 21, 85-100 (2020).
- Pfanner, N., Warscheid, B., Wiedemann, N. Mitochondrial proteins: from biogenesis to functional networks. Nature Reviews Molecular Cell Biology. 20, (2019).
- Bykov, Y. S., Rapaport, D., Herrmann, J. M., Schuldiner, M. Cytosolic Events in the Biogenesis of Mitochondrial Proteins. Trends in Biochemical Sciences. 45, 650-667 (2020).
- Pfanner, N., Warscheid, B., Wiedemann, N. Mitochondrial proteins: from biogenesis to functional networks. Nature Reviews Molecular Cell Biology. 427, 1135 (2019).
- Borgese, N., Coy-Vergara, J., Colombo, S. F., Schwappach, B. The Ways of Tails: the GET Pathway and more. The Protein Journal. , 1-17 (2019).
- Mateja, A., Keenan, R. J. A structural perspective on tail-anchored protein biogenesis by the GET pathway. Current Opinion in Structural Biology. 51, 195-202 (2018).
- Chio, U. S., Cho, H., Shan, S. Mechanisms of Tail-Anchored Membrane Protein Targeting and Insertion. Annual review of cell and developmental biology. 33, 417-438 (2017).
- Denic, V. A portrait of the GET pathway as a surprisingly complicated young man. Trends in biochemical sciences. , (2012).
- Hegde, R. S., Keenan, R. J. Tail-anchored membrane protein insertion into the endoplasmic reticulum. Nature Reviews Molecular Cell Biology. 12, 787-798 (2011).
- Kalbfleisch, T., Cambon, A., Wattenberg, B. W. A bioinformatics approach to identifying tail-anchored proteins in the human genome. Traffic. 8, 1687-1694 (2007).
- Doan, K. N., et al. The Mitochondrial Import Complex MIM Functions as Main Translocase for α-Helical Outer Membrane Proteins. Cell Reports. 31, (2020).
- McDowell, M. A., et al. Structural Basis of Tail-Anchored Membrane Protein Biogenesis by the GET Insertase Complex. Molecular Cell. 80, (2020).
- Guna, A., Volkmar, N., Christianson, J. C., Hegde, R. S. The ER membrane protein complex is a transmembrane domain insertase. Science. 591, 3099 (2017).
- Rao, M., et al. Multiple selection filters ensure accurate tail-anchored membrane protein targeting. eLife. 5, 21301 (2016).
- Schuldiner, M., et al. The GET complex mediates insertion of tail-anchored proteins into the ER membrane. Cell. 134, 634-645 (2008).
- Chen, Y. -. C., et al. Msp1/ATAD1 maintains mitochondrial function by facilitating the degradation of mislocalized tail-anchored proteins. The EMBO journal. 33, 1548-1564 (2014).
- Wu, X., Rapoport, T. A. Translocation of Proteins through a Distorted Lipid Bilayer. Trends in Cell Biology. , (2021).
- Phillips, B. P., Gomez-Navarro, N., Miller, E. A. Protein quality control in the endoplasmic reticulum. Current Opinion in Cell Biology. 65, 96-102 (2020).
- van de Weijer, M. L., et al. Quality Control of ER Membrane Proteins by the RNF185/Membralin Ubiquitin Ligase Complex. Molecular Cell. 79, (2020).
- Weir, N. R., Kamber, R. A., Martenson, J. S., Denic, V. The AAA protein Msp1 mediates clearance of excess tail-anchored proteins from the peroxisomal membrane. eLife. 6, 28507 (2017).
- Gardner, B. M., et al. The peroxisomal AAA-ATPase Pex1/Pex6 unfolds substrates by processive threading. Nature communications. 9, 135 (2018).
- Puchades, C., et al. Unique Structural Features of the Mitochondrial AAA+ Protease AFG3L2 Reveal the Molecular Basis for Activity in Health and Disease. Molecular Cell. , (2019).
- Castanzo, D. T., LaFrance, B., Martin, A. The AAA+ ATPase Msp1 is a processive protein translocase with robust unfoldase activity. Proceedings of the National Academy of Sciences of the United States of America. 117, 14970-14977 (2020).
- Wang, L., Myasnikov, A., Pan, X., Walter, P. Structure of the AAA protein Msp1 reveals mechanism of mislocalized membrane protein extraction. eLife. 9, (2020).
- Puchades, C., Sandate, C. R., Lander, G. C. The molecular principles governing the activity and functional diversity of AAA+ proteins. Nature Reviews Molecular Cell Biology. , 1-16 (2019).
- Yang, Y., et al. Folding-Degradation Relationship of a Membrane Protein Mediated by the Universally Conserved ATP-Dependent Protease FtsH. Journal of the American Chemical Society. , 10 (2018).
- Baldridge, R. D., Rapoport, T. A. Autoubiquitination of the Hrd1 Ligase Triggers Protein Retrotranslocation in ERAD. Cell. 166, 394-407 (2016).
- Fresenius, H. L., Wohlever, M. L. Sorting out how Msp1 maintains mitochondrial membrane proteostasis. Mitochondrion. 49, 128-134 (2019).
- Wang, L., Walter, P. Msp1/ATAD1 in Protein Quality Control and Regulation of Synaptic Activities. Annual Review of Cell and Developmental Biology. 36, 1-24 (2020).
- Dederer, V., et al. Cooperation of mitochondrial and ER factors in quality control of tail-anchored proteins. eLife. 8, 1126 (2019).
- Matsumoto, S., et al. Msp1 Clears Mistargeted Proteins by Facilitating Their Transfer from Mitochondria to the ER. Molecular Cell. , (2019).
- Li, L., Zheng, J., Wu, X., Jiang, H. Mitochondrial AAA-ATPase Msp1 detects mislocalized tail-anchored proteins through a dual-recognition mechanism. EMBO Reports. 20, (2019).
- Weidberg, H., Amon, A. MitoCPR - a surveillance pathway that protects mitochondria in response to protein import stress. Science. 360, (2018).
- Okreglak, V., Walter, P. The conserved AAA-ATPase Msp1 confers organelle specificity to tail-anchored proteins. Proceedings of the National Academy of Sciences of the United States of America. 111, (2014).
- Piard, J., et al. A homozygous ATAD1 mutation impairs postsynaptic AMPA receptor trafficking and causes a lethal encephalopathy. Brain. , (2018).
- Zhang, J., et al. The AAA+ ATPase Thorase regulates AMPA receptor-dependent synaptic plasticity and behavior. Cell. 145, 284-299 (2011).
- Prendergast, J., et al. Ganglioside regulation of AMPA receptor trafficking. The Journal of Neuroscience. 34, 13246-13258 (2014).
- Umanah, G. K. E., et al. Thorase variants are associated with defects in glutamatergic neurotransmission that can be rescued by Perampanel. Science Translational Medicine. 9, 4985 (2017).
- Pignatelli, M., et al. Synaptic Plasticity onto Dopamine Neurons Shapes Fear Learning. Neuron. 93, 425-440 (2017).
- Zhang, J., et al. The AAA Thorase is neuroprotective against ischemic injury. Journal of Cerebral Blood Flow and Metabolism. , 271678 (2018).
- Umanah, G. K. E., et al. AMPA Receptor Surface Expression Is Regulated by S-Nitrosylation of Thorase and Transnitrosylation of NSF. Cell Reports. 33, 108329 (2020).
- Wohlever, M. L., Mateja, A., McGilvray, P. T., Day, K. J., Keenan, R. J. Msp1 Is a Membrane Protein Dislocase for Tail-Anchored Proteins. Molecular Cell. 67, 194-202 (2017).
- Lovell, J. F., et al. Membrane binding by tBid initiates an ordered series of events culminating in membrane permeabilization by Bax. Cell. 135, 1074-1084 (2008).
- Leshchiner, E. S., Braun, C. R., Bird, G. H., Walensky, L. D. Direct activation of full-length proapoptotic BAK. Proceedings of the National Academy of Sciences of the United States of America. 110, 986-995 (2013).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved