Method Article
Frazione subcellulare delle cellule linfonetiche croniche primarie per monitorare il traffico di proteine nucleari/citoplasmiche
In questo articolo
Riepilogo
Questo protocollo consente l'ottimizzazione e la successiva generazione efficiente di frazioni nucleari e citoplasmiche dalle cellule leucemiche linfocitiche croniche primarie. Questi campioni sono utilizzati per determinare la localizzazione delle proteine e i cambiamenti nel traffico di proteine che si svolgono tra i compartimenti nucleari e citoplasmatici dopo la stimolazione cellulare e il trattamento farmacologico.
Abstract
L'esportazione nucleare di macromolecole è spesso deregolata nelle cellule tumorali. Le proteine soppressori del tumore, come il p53, possono essere rese inattive a causa della localizzazione cellulare aberrante che interrompe il loro meccanismo di azione. La sopravvivenza delle cellule lacofocitiche croniche (LLC), tra le altre cellule tumorali, è favorita dalla deregolazione dell'chiusura da nucleare a citoplasma, almeno in parte attraverso la deregolamentazione del recettore di trasporto XPO1 e l'attivazione Percorsi di segnalazione mediati da PI3K. È essenziale comprendere il ruolo delle singole proteine nel contesto della loro posizione intracellulare per ottenere una comprensione più profonda del ruolo di tali proteine nella patobiologia della malattia. Inoltre, l'identificazione dei processi alla base della stimolazione cellulare e del meccanismo d'azione di specifici inibitori farmacologici, nel contesto del traffico di proteine subcellulari, fornirà una comprensione più completa del meccanismo di azione. Il protocollo qui descritto consente l'ottimizzazione e la successiva generazione efficiente di frazioni nucleari e citoplasmiche dalle cellule leucemiche linfocitiche croniche primarie. Queste frazioni possono essere utilizzate per determinare i cambiamenti nel traffico di proteine tra le frazioni nucleari e citoplasmache durante la stimolazione cellulare e il trattamento farmacologico. I dati possono essere quantificati e presentati in parallelo con immagini immunofluorescenti, fornendo così dati robusti e quantificabili.
Introduzione
Il trasporto di macromolecole tra il nucleo e il citoplasma è stato a lungo stabilito per svolgere un ruolo chiave nella normale funzione cellulare ed è spesso deregolamentato nelle cellule tumorali1,2. Tale deregolamentazione può derivare da sovraespressione/mutazione delle proteine che controllano l'esportazione nucleare. Una di queste proteine Exportin-1 (XPO1), è un recettore di trasporto che esporta >200 proteine contenenti segnale di esportazione nucleare (NES) nel citoplasma del nucleo2. XPO1-cargos includono p53, membri della famiglia FOXO e IB, contribuendo alla loro inattivazione inibendo il loro meccanismo di azione1,2,3. Un'ulteriore dislocalizzazione delle proteine può verificarsi quando i segnali microambientali incidono sulle cellule tumorali, portando all'attivazione di vie di segnalazione intracellulari come il fosfatidil-inositolo-3-kinase (PI3K)/Akt percorso, con conseguente inattivazione dei membri della famiglia FOXO e successiva esportazione dal nucleo4,5. Tale cattiva localizzazione delle proteine soppressori tumorali è stata implicata nella progressione di un certo numero di tumori ematologici e solidi1,2,6.
Lo sviluppo di inibitori di piccole molecole per uso clinico in malignità ematologiche (leucemia mieloide acuta (AML)/CLL), che si legano e inibiscono selettivamente la funzione XPO1, sottolinea l'importanza di sviluppare tecniche appropriate per impatto degli agenti farmacologici sull'chiusura delle proteine tra i compartimenti nucleari e citoplasmamici6,7,8. Le tecniche di imaging sono avanzate in modo significativo consentendo l'identificazione delle proteine nei compartimenti subcellulari sulla stimolazione esterna dei trattamenti farmacologici, tuttavia, l'importanza di tecniche parallele robuste e di supporto è fondamentale per informare un pubblico scientifico della validità di un risultato.
I linfociti a riposo e le cellule cLL-B maligne isolate dai campioni di sangue dei pazienti rappresentano una sfida nella generazione di frazioni nucleari e citoplasmiche a causa dell'elevato rapporto nucleare: citoplasmaico. L'ottimizzazione delle condizioni sperimentali per generare dati sperimentali affidabili e affidabili è ovviamente fondamentale per pianificare futuri programmi sperimentali. Il metodo qui descritto consente la quantificazione delle proteine nelle frazioni nucleari e citoplasmiche e determina come queste proteine possono essere influenzate dalla stimolazione cellulare e/o dal trattamento farmacologico.
Protocollo
L'uso di campioni primari di pazienti affetti da CLL descritti qui è stato approvato dal West of Scotland Research Ethics Service, NHS Greater Glasgow and Clyde (UK) e tutto il lavoro è stato svolto in conformità con le linee guida approvate.
1. Isolamento delle cellule CLL dai campioni di sangue del paziente
-
I campioni di sangue periferici provenienti da pazienti affetti da CLL precedentemente autorizzati vengono ricevuti dalla clinica in tubi di raccolta del sangue EDTA, accompagnati dal numero di cellule bianche (WCC). Purificare i campioni di CLL di sangue periferico secondo il WCC. Per WCC < 40 x 106 celle/mL, procedere al passaggio 1.1.1; per WCC - 40 x 106 celle/mL, procedere al passaggio 1.1.2.
- Versare il contenuto di tutti i tubi sanguigni EDTA in un tubo di centrifuga conica da 50 mL e aggiungere 50 -L di Human B Cell Enrichment Cocktail per 1 mL di sangue. Incubare a temperatura ambiente (RT) per 20 min. Procedere al passaggio 1.1.2.
- Diluire il campione a un rapporto di 1:1 con tampone di lavaggio CLL RT (salina tampone di fosfato (PBS), 0,5% Siero bovino fetale (FBS) e 2 mM EDTA).
- Supporti a gradiente di densità DI RT in un tubo di centrifuga coniliare di dimensioni appropriate per il campione (10 mL in un tubo da 50 mL per 30 mL di campione o 4 mL in un tubo da 15 mL per 10 mL di campione).
- Sovrapporre con attenzione il campione sopra il supporto a gradiente di densità e centrifugare a 400 x g per 30 min a RT.
NOT: Assicurarsi che la centrifuga sia a RT prima che i campioni siano collocati nella centrifuga come un cambiamento di temperatura si tradurrà in un cattivo arricchimento delle cellule mononucleari, e spegnere il freno sulla centrifuga, come frenata improvvisa può interrompere l'interfaccia liquida. - Raccogliere delicatamente lo strato bianco di celle mononucleari che si raccolgono all'interfaccia del supporto a gradiente di densità e del tampone di lavaggio CLL, in un nuovo tubo di centrifuga conica da 50 mL utilizzando una pipetta Pasteur plastica.
- Aggiungere 40 mL di tampone di lavaggio CLL al monostrato isolato per lavare le cellule e centrifugare a 300 x g per 10 min a RT.
- Scartare il supernatante, risospendere il pellet facendo scorrere il fondo del tubo, quindi ripetere la fase di lavaggio descritta al punto 1.5.
- Eliminare il supernatante, risospendere il pellet come descritto al punto 1.6, quindi riutilizzare il pellet in un volume impostato di buffer di lavaggio CLL (fino a 40 mL, a seconda delle dimensioni del pellet cellulare).
- Contare le cellule usando trypan blu e un haemocytometer. Quindi procedere al flusso di citometria per verificare la purezza delle cellule CLL.
NOT: In questa fase le cellule CLL possono essere coltivate ad una concentrazione di 10 x 106 cellule /mL nei media da utilizzare negli esperimenti, e/o crioconservate nel 10% di zolfo dimetilo (DMSO)/FBS per il lavoro futuro a concentrazioni fino a 100 x 106 cellule/fla.
2. Citometria di flusso delle cellule CLL
- Etichettare i tubi in polistirolo rotondo a fondo 12 mm x 75 mm, come descritto nella Tabella 1.
- Nei tubi 2 – 5, mettere una goccia di perline di compensazione e conservare sul ghiaccio. Aggiungere 1 -L dell'anticorpo appropriato (anti-CD5, CD19, CD23 o CD45, come indicato nella tabella 1) ai tubi 2 – 5 e incubare sul ghiaccio per 20 min, protetto dalla luce posizionando la lamina di stagno sopra il secchio di ghiaccio.
NOT: Questi tubi fungono da controlli di compensazione per l'impostazione del modello di citometria di flusso. -
Mettere fino a 1 x 106 celle CLL nei tubi 1, 6 e 7, aggiungere 2 mL di tampone FACS (PBS - 2% FBS) ad ogni tubo e centrifugare a 300 x g per 5 min a RT per lavare le cellule. Eliminare il supernatante e conservare i tubi contenenti pellet cellulari sul ghiaccio.
- Risospendere i pellet cellulari e aggiungere l'appropriata combinazione di anticorpi alle cellule del tubo 7 come indicato nella tabella 1, in un volume finale di 100 l con tampone FACS. Gli anticorpi sono utilizzati ad una concentrazione adeguata secondo le linee guida del produttore.
- Risospendere i pellet cellulari nei tubi 2 e 6 in 100 l di tampone FACS.
- Incubare le cellule sul ghiaccio, insieme alle perline colorate nei tubi 2 – 5, protette dalla luce per 20 min.
- Dopo l'incubazione, aggiungere 2 mL di tampone FACS a tutti i tubi e centrifugare a 300 x g per 5 min a RT per lavare le cellule. Scartare il supernatante e risospendere i pellet perline/cellule facendo scorrere delicatamente i tubi.
- Risospendere i tubi da 1 a 5 su 100 l di TAC tampone e posizionare sul ghiaccio fino a quando non è pronto per l'analisi sul citometro di flusso.
- Diluire la soluzione DAPI a 0,05-0,2 g/mL nel buffer FACS immediatamente prima dell'uso. La concentrazione ottimale può variare, e si raccomanda la titolazione.
- Risospendere i tubi 6 e 7 con 100 l di soluzione DAPI diluita e incubare i tubi sul ghiaccio per un minimo di 5 min per consentire alle cellule di macchiare.
NOT: Non è necessario ulteriore lavaggio, poiché DAPI deve essere presente nel buffer affinché le cellule morte rimangano etichettate. Una volta che DAPI è stato aggiunto, le cellule devono essere analizzate sul citometro di flusso entro 4 h. - Analizzare le celle utilizzando un citometro di flusso.
3. Preparazione delle frazioni subcellulari dalle cellule CLL
NOT: Quando si pianifica l'allestito sperimentale, includere un pozzo di cellule non stimolate/non trattate da cui è possibile generare l'estratto di cellule.
- Eseguire la stimolazione desiderata e/o il trattamento farmacologico della linea cellulare MEC1 CLL o delle cellule CLL primarie isolate utilizzando 10 – 20 x 106 cellule/condizione. Le cellule verranno quindi utilizzate per la frazione subcellulare (passaggi 3.4 e 3.5) o per generare l'estratto di intere cellule (passaggio 3.6).
- Preparazione di soluzioni/tubi: Preparare tutte le soluzioni / buffer appena il giorno della frazione, prima che le cellule vengono raccolte. Conservare le soluzioni sul ghiaccio fino a quando richiesto e utilizzare entro 4 h dalla preparazione.
- Soluzione inibitoriari PBS/phosphatase: Preparare gli inibitori del fosfosi in PBS diluindo gli inibitori del fosfosi 1:20 in 1x PBS (cioè 0,5 mL di inibitori del fosfosi in 9,5 mL di 1x PBS).
NOT: Assicurarsi che gli inibitori del fosfosi non siano precipitati. Se è presente un precipitato, scaldare a 50 gradi centigradi per 10 minuti. - Buffer ipotonico: Preparare 1x buffer ipotonico facendo un 1:10 diluizione di 10x buffer ipotonico in acqua distillata (cioè, 50 L di 10x buffer ipotonico in 450 l di dH2O).
- 10 mM dithiothreitol (DTT): Preparare 10 mM DTT effettuando una diluizione di 1:100 di 1M DTT con acqua distillata (cioè 10L di 1 M DTT in 990 - L di dH2O).
NOT: DTT è altamente labile in modo da preparare questo fresco ogni volta. Evitare ripetuti cicli di congelamento/scongelamento. - Buffer di lisi completa: determinare la quantità di buffer necessaria per ogni esperimento. Ogni campione richiede 50 l di buffer di lisi completa, quindi aggiungere 5 - L di 10 mM DTT (passaggio 3.2.3) a 44,5 l di buffer di lisi, quindi aggiungere 0,5 l di cocktail inibitore della proteasi. Questo importo può essere aumentato a seconda del numero di campioni nell'esperimento.
- Etichettare quattro serie di tubi di microfuge da 1,5 ml per ogni stimolazione e/o trattamento farmacologico per le cellule appena stimolate (passaggio 3.3), le frazioni citoplasmatiche appena generate (passaggio 3,4,3), le frazioni nucleari appena generate (passo 3.5.3) e l'intera cellula lisari (passaggio 3.6.3). Pre-raffreddare questi tubi di microfuge sul ghiaccio fino a quando richiesto.
- Soluzione inibitoriari PBS/phosphatase: Preparare gli inibitori del fosfosi in PBS diluindo gli inibitori del fosfosi 1:20 in 1x PBS (cioè 0,5 mL di inibitori del fosfosi in 9,5 mL di 1x PBS).
- Trasferire le cellule in tubi di microfuge e pellet da 1,5 mL singolarmente centrifugando a 200 x g per 5 min a 4 gradi centigradi. Rimuovere il supernatante e risospendere nuovamente le cellule in 1 mL di inibitori PBS/fosfosi ghiacciati (passaggio 3.2.1). Pellet le cellule per centrifugazione a 200 x g per 5 min a 4 gradi centigradi. Rimuovere il supernatante e tenere i pellet cellulari sul ghiaccio.
- Preparazione delle frazioni citoplasmiche: Risospendere delicatamente i pellet cellulari da utilizzare per la frazione subcellulare in 50 :L di 1x buffer ipotonico (passaggio 3.2.2). Incubare le cellule sul ghiaccio per 15 min per consentire alle cellule di gonfiarsi.
NOT: Il volume del buffer ipotonico utilizzato può essere aumentato empiricamente a seconda del numero di cellulare.- Aggiungere 0,8 - 2,5 gradi (1:20-1:60) di detersivo in ogni campione e vortice sull'impostazione più alta per 10 s.
- Per determinare la concentrazione ottimale di detergente da utilizzare per un tipo di cellula specifico per isolare le frazioni nucleari e citoplasmiche, eseguire inizialmente un gradiente di detersivo. Un intervallo compreso tra 1:20 e 1:60 (vale a dire da 2,5 a 0,8 litri di detersivo in 50 o l di buffer ipotonico) deve essere adeguato.
NOTA: Se il volume del buffer ipotonico nel passaggio 3.4 è regolato, assicurarsi che venga mantenuto il rapporto di detersivo appropriato. - Verificare la lisi delle cellule osservando le cellule utilizzando un microscopio a contrasto di fase prima e dopo l'aggiunta di detersivo. Intere cellule appaiono più grandi con un nucleo denso e scuro. Il citoplasma apparirà come un alone luminoso intorno al nucleo.
NOT: L'appropriata lisi è ulteriormente confermata utilizzando il gonfio occidentale per analizzare specifiche proteine all'interno delle frazioni lismigenerate dal gradiente del detersivo.
- Per determinare la concentrazione ottimale di detergente da utilizzare per un tipo di cellula specifico per isolare le frazioni nucleari e citoplasmiche, eseguire inizialmente un gradiente di detersivo. Un intervallo compreso tra 1:20 e 1:60 (vale a dire da 2,5 a 0,8 litri di detersivo in 50 o l di buffer ipotonico) deve essere adeguato.
- Una volta lievitati, centrificare i campioni a 14.000 x g per 30 s a 4 gradi centigradi.
- Trasferire con attenzione il supernatante in un tubo di microfuge pre-raffreddato e etichettato. Questa frazione citoplasmatica può essere immagazzinata a -80 gradi centigradi fino a quando non è necessaria per ulteriori analisi. Il pellet rimanente contiene la frazione nucleare (passaggio 3.5).
NOT: Evitare ripetuti cicli di congelamento/scongelamento dei campioni.
- Aggiungere 0,8 - 2,5 gradi (1:20-1:60) di detersivo in ogni campione e vortice sull'impostazione più alta per 10 s.
- Preparazione delle frazioni nucleari: Risospendere ogni pellet nucleare in 50 gradi di tampone di lisi completa (passaggio 3.2.4) pipettando su e giù.
NOT: Il volume del buffer di lisi completa può essere regolato empiricamente in base al numero di cella iniziale.- Aggiungere 2,5 litri di detersivo per solubilitare le proteine associate alla membrana nucleare e al vortice sull'impostazione più alta per 10 s. Incubare i campioni sul ghiaccio per 30 min.
- Vortice sull'impostazione più alta per 30 s, quindi centrificare i campioni a 14.000 x g per 20 min a 4 gradi centigradi.
- Trasferire il supernatante in un tubo di microfuge pre-raffreddato e etichettato. Questa frazione nucleare può essere immagazzinata a -80 gradi centigradi fino a quando non è necessaria per ulteriori analisi.
NOT: Evitare ripetuti cicli di congelamento/scongelamento dei campioni.
- Preparazione di lismi a cellule intere (WCL) dalle cellule CLL
NOT: La preparazione dell'estratto di intere cellule può essere effettuata contemporaneamente alla preparazione delle frazioni nucleari (passaggio 3.5).- Risospendere l'intero pellet di estratto di cellule in 100 gradi l di tampone di lisi completa (preparato al passo 3.2.4) pipettando su e giù, quindi aggiungere 5 - L di detersivo per garantire la lisi cellulare completa. Incubare i campioni sul ghiaccio per 30 min.
- Vortice sull'impostazione più alta per 30 s, quindi centrificare i campioni a 14.000 x g per 20 min a 4 gradi centigradi.
- Trasferire il supernatante in un tubo di microfuge pre-raffreddato. Questa intera cellula lisata può essere conservata a -80 gradi centigradi fino a quando non è necessario per ulteriori analisi.
NOT: Evitare ripetuti cicli di congelamento/scongelamento dei campioni.
4. Analisi a valle delle frazioni subcellulari
NOT: In questo protocollo, l'analisi delle frazioni cellulari generate è stata effettuata mediante oscillazioni occidentali utilizzando protocolli standard, caricando numeri cellulari/corsie uguali (equivalenti a 10 dollari di proteine) per le frazioni nucleari e citoplasmiche.
- Quantificazione del traffico di proteine tra frazioni nucleari e citoplasmatiche: Eseguire l'analisi quantitativa delle macchie occidentali attraverso la quantificazione dell'intensità del segnale, o densitometria utilizzando un software di analisi delle macchie occidentali liberamente disponibile.
- Importazione di immagini: Le immagini western blot generate da diversi strumenti in via di sviluppo devono essere importate come file JPG, PNG o TIFF. Si consiglia un file RAW di profondità a 16 bit. Per importare un'immagine, fare clic sull'icona del software e passare il mouse su Importa. Quindi fare clic su Immagini di terze parti. Selezionare il file di immagine e fare clic su Apri.
- Visualizzazione dell'immagine: Nella barra multifunzione Immagine fare clic sul pulsante Scegli nel gruppo Visualizzazione. Si apre la finestra di dialogo Regola visualizzazione per consentire ulteriori regolazioni, se necessario. Implementare ulteriori miglioramenti, tra cui Luminosità o Contrasto utilizzando i cursori regolabili nella scheda LuT immagine.
- Analisi dei dati (deselezione del canale): fare clic sulla barra multifunzione Analisi. Per analizzare un solo canale, deselezionate i canali non analizzati. Fai clic sulla miniatura Non mostrare canale di un canale nelle LUT immagine, lasciando visualizzato solo il canale desiderato. Le immagini importate come file JPG, PNG o TIFF possono richiedere la deselezione di più canali RGB indesiderati.
-
Aggiunta di forme: Per quantificare l'intensità del segnale, fare clic su Aggiungi rettangolo per aggiungere un rettangolo all'immagine. Fare clic al centro di una feature (ad esempio, una banda proteica) per posizionare un rettangolo intorno ad essa. In alternativa, per disegnare manualmente una forma, scegliere Disegna rettangolo. Dopo aver aggiunto tutte le forme desiderate, fare clic su Seleziona per riportare il cursore allo strumento di selezione.
NOT: Aggiungere più forme in ordine logico, poiché i dati vengono ordinati in base a un numero ID generato in sequenza. -
Sottrazione in background: Per sottrarre il disturbo di fondo, fate clic sul primo pulsante nel gruppo Sfondo e selezionate Mediana dal menu a discesa. Impostare lo spessore del bordo su 3 nella finestra di dialogo Sfondo e selezionare i segmenti da utilizzare per il calcolo dello sfondo. Quando scegli i segmenti da utilizzare, seleziona i segmenti che meglio rappresentano lo sfondo dell'immagine.
NOT: Il rumore di fondo può influenzare la quantificazione del segnale, quindi deve essere sottratto per calcolare con precisione il segnale dalle forme di interesse. -
Segnale di taglio e taglio sfondo - OPZIONALE: I file importati nei formati JPG, PNG o TIFF possono presentare la saturazione dei pixel: regioni evidenziate/luminose all'interno di una banda proteica. Trim Signal and Trim Background (Bkgnd) rimuove i pixel saturi dall'analisi. Per visualizzare questi valori, aggiungere Trim Signal e Trim Bkgnd a una tabella facendo clic sul pulsante Colonne a destra della vista tabella.
NOT: La saturazione dei pixel può portare a una quantificazione inaffidabile. I pixel saturi possono essere rimossi solo se meno del 5% dei pixel all'interno di una forma è saturo.
-
Aggiunta di forme: Per quantificare l'intensità del segnale, fare clic su Aggiungi rettangolo per aggiungere un rettangolo all'immagine. Fare clic al centro di una feature (ad esempio, una banda proteica) per posizionare un rettangolo intorno ad essa. In alternativa, per disegnare manualmente una forma, scegliere Disegna rettangolo. Dopo aver aggiunto tutte le forme desiderate, fare clic su Seleziona per riportare il cursore allo strumento di selezione.
- Esporta dati: Fare clic sulla scheda Forme sopra la tabella. Per densitometry, i valori nella colonna Segnale sono obbligatori. Segnale è la somma dei valori di intensità del pixel (Totale) per una forma meno il prodotto del Bkgnd e dell'Area. Fare clic sul pulsante Report. Fare clic su Salva con nome o Avvia foglio di calcolo.
Segnale : Totale – (Bkgnd x Area)
NOT: La scheda Forme fornisce una tabella di valori quantitativi tra cui Segnale, Totale, Area e Bkgrnd. - Quantificare l'espressione proteica: All'interno del foglio di calcolo salvato, calcolare l'espressione normalizzata della proteina di interesse per ogni corsia o variabile dividendo il segnale ottenuto per la proteina di interesse per il segnale per la corrispondente banda di controllo del carico proteico.
NOT: I confronti tra le quantità di una proteina normalizzata di interesse tra le frazioni nucleari e citoplasmatiche non possono essere confrontati direttamente a causa dei diversi controlli di carico utilizzati per distinguere le frazioni nucleari e citoplasmase. Tuttavia, i confronti all'interno di singole frazioni, ad esempio dopo il trattamento farmacologico, sono appropriati. - Esporta immagine per la pubblicazione o la presentazione: Fare clic sulla scheda Immagini che si trova sopra la tabella e quindi fare clic sull'immagine da esportare. Se si utilizza l'immagine per una presentazione di diapositive o altri formati digitali, fare clic sull'icona del software, passare il mouse su Esporta e fare clic su Immagine per Digital Media. Salvare l'immagine come file JPG, PNG o TIFF, in base alle esigenze.
Risultati
Quando si pianificano esperimenti sulle cellule CLL primarie, se i saggi richiedono un gran numero di cellule (>50 x 106 celle), c'è una preferenza per utilizzare cellule CLL appena isolate, piuttosto che cellule crioconservate che richiedono scongelamento, tuttavia questo non è sempre possibile. Questo perché il processo di congelamento / disgelo può provocare la morte fino al 50% delle cellule CLL, anche se questo è dipendente dal campione. L'arricchimento delle cellule CLL con un WCC >40 x 106/mL utilizzando la centricazione della densità come descritto qui (passaggi da 1.3 – 1,5) consente un elevato recupero cellulare con elevata purezza (z 95%) delle cellule CLL primarie. Nel campione mostrato, è stato recuperato il WCC 177 x 106/mL: da un campione di sangue da 30 mL 5 x 109 cellule, che rappresenta una resa cellulare del 94% delle cellule totali. L'analisi di questo campione per citometria di flusso ha rivelato una purezza delle cellule CLL di >95% come indicato dalla doppia espressione superficiale dei marcatori di cella CLL CD19 e CD5 dopo l'analisi su FSC/SSC, singole cellule che erano DAPI negative (cellule vitali) (Figura 1).
L'ottimizzazione della procedura di frazionamento subcellulare è stata effettuata utilizzando una gamma di rapporti detergenti (da 1:20 a 1:60) durante la preparazione della frazione citoplasmatica (passaggio 3.4). Successivamente, sono state preparate le frazioni nucleari e i CL (rispettivamente i passaggi 3.5 e 3.6). Immunoblot sono stati eseguiti sulle frazioni risultanti della linea cellulare CLL MEC1 (Figura 2A) e cellule CLL primarie (Figura 2B). Le macchie sono state sondate per i marcatori di frazione Lamin A/C (nucleare; 74/63 kDa) e la tubulina z-tubulina (citoplasmaico; 55 kDa) per confermare la frazione cellulare di successo. La frazione indica che il livello di detergente ottimale per le cellule MEC1 è una diluizione 1:60 (Figura 2A), rispetto a una diluizione 1:30 ottimale per le cellule CLL primarie (Figura 2B), come indicato da un arricchimento di proteina nucleare e la mancanza di proteine citoplasmiche nelle frazioni e viceversa. I WCL rappresentano la proteina totale e fungono da controllo positivo per gli anticorpi utilizzati per sondare le frazioni subcellulari. È importante scegliere le proteine appropriate come marcatori di frazione: la figura 2C mostra immunoblot delle frazioni nucleari/citoplasmatiche preparate da cellule MEC1 in cui l'RNA polimerasi II (Rpb1 CTD; 250 kDa) e lamin A/C sono stati crollati come marcatori di frazioni nucleari, mentre la tubulina e la tubulina z (50 kDa) sono state usate come marcatori citoplasmatici. È chiaro che la zbulina è arricchita nel citoplasma tuttavia l'espressione è evidente nel nucleo, come mostrato in precedenza9.
Una volta ottimizzate le condizioni sperimentali, è possibile eseguire un esperimento. Negli esempi mostrati, la localizzazione subcellulare di FOXO1 nelle frazioni nucleari e citoplasmiche è stata determinata sulla stimolazione delle cellule con il recettore dell'antigene a cellule B (BCR) in presenza o assenza del doppio inibitore mTORC1/2 A-D8055, nelle cellule MEC1 ( Figura 3A) e celle CLL primarie ( Figura3B)5,10. In entrambi gli esempi, la generazione di frazioni nucleari e citoplasmatiche altamente arricchite è stata ottenuta come indicato dall'espressione quasi esclusiva di Lamin nella frazione nucleare e nella tubulina z-tubulina nelle frazioni citoplasmiche. In entrambi i tipi di cellule, l'espressione di FOXO1 è stata ridotta nel citoplasma dopo il trattamento con l'A-D8055 rispetto a NDC, accompagnata da un aumento dell'espressione FOXO1 nel compartimento nucleare, dimostrando così la traslocazione proteica (Figura 3). Per rimuovere la soggettività dell'interpretazione dei dati, sono stati quantificati singoli immunoblot da cinque campioni primari di LL sottopongo all'interno di frazioni subcellulari (passaggio 4; Figura 4A), utilizzando le rispettive proteine nucleari o citoplasmatiche come controlli di carico interni per ciascun campione e quindi normalizzando ogni frazione al controllo NON stimolato (US) nessun controllo dei farmaci (NDC), come indicato. Il grafico risultante mostra le tendenze del movimento FOXO1 tra le frazioni nucleari e citoplasmiche, con l'A-D8055 che riduce i livelli di espressione FOXO1 nel citoplasma, aumentando contemporaneamente l'espressione nel nucleo. Inoltre, un'elevazione nell'espressione citoplasmica FOXO1 è evidente sul collegamento bCR.
| Tubo | Nome tubo | Celle/Perline | antigene m | Fluoroforo |
| 1 | Innocente | Cellule | Na | Na |
| 2 | Macchia singola | Perline | CD5 (in formato CD5) | Fitc |
| 3 | Macchia singola | Perline | CD19 (informazioni in stato INstato CD1 | PE-Cy7 |
| 4 | Macchia singola | Perline | CD23 (in formato CD23) | Apc |
| 5 | Macchia singola | Perline | CD45 (informazioni in stato indue) | APC Cy7 |
| 6 | Macchia singola | Cellule | attuabilità | Dapi |
| 7 | Macchie CLL | Cellule | CD5, CD19, CD23, CD45 e redditività | FITC, PE-Cy7, APC, APC-Cy7 e DAPI |
Tabella 1: Tabella che mostra il set ideale di tubi campione necessari per la citometria di flusso delle cellule CLL. Ogni esperimento deve includere tutti i controlli appropriati per un'analisi accurata dei risultati ottenuti.

Figura 1: grafico dell'analisi della citometria del flusso rappresentativo dei pazienti arricchiti con CLL. Le cellule mononucleari-CLL arricchite dal sangue periferico di un singolo paziente a base di CoLl sono state gated utilizzando FSC-A contro SSC-A, e i doppietti sono stati poi esclusi utilizzando FSC-A vs FSC-H (A). Le cellule non colorate (tubo 1) e i controlli di compensazione (tubi 2-6) sono stati utilizzati per impostare il citometro di flusso per rilevare le cellule e compensare tra i canali fluorescenti, garantendo così che i segnali di fluorescenza siano stati rilevati correttamente. (B) Un esempio di colorazione negativa (cellule non colorate; tubo 1) nei canali di fluorescenza CD19 e CD5. Le cellule live (DAPI negativo) e le cellule positive CD45 sono state recintate (C) e la percentuale di CD19-CD5 (95,5%) (91,2%) cellule all'interno del DAPI-CD45è stata determinata popolazione (D). Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Ottimizzazione della frazione nucleare/citoplasmaica. Le frazioni citoplasmache e nucleari, e le lismiari intere (WCL), sono state preparate da pellet cellulari (10 a 20 x 106 cellule) della linea cellulare CLL(A) MEC1 o(B)delle cellule CLL primarie arricchite dal sangue periferico dei pazienti Passo 3. L'ottimizzazione della frazione subcellulare è stata effettuata utilizzando una gamma di rapporti detergenti (da 1:20 a 1:60) durante la preparazione della frazione citoplasmatica (come descritto al punto 3.4). I campioni risultanti sono stati immunoblotted e sondati con anticorpi anticorpi anti-Lamin A/C (nucleare) e anti-tubulina (citoplasma) per confermare il successo della frazione cellulare insieme alla WCL. I marcatori di peso molecolare sono mostrati a sinistra della macchia (M). : indica le condizioni ottimali del detersivo per la lisi cellulare. (C) Immunoblot di frazioni nucleari e citoplasmatiche provenienti da cellule MEC1 con condizioni di controllo (NDC) o trattamento farmacologico (8055) in presenza di assenza di stimolazione (rispettivamente il collegamento incrociato di BCR). I blots sono stati sondati con anticorpi anti-Rbp1 CTD (clone 4H8; riconoscendo RNA polymerase II subunit B1), anti-Lamin A/C, anticorpi anti-tubulina o anti-zzbulina (clone GTU-88), per identificare le frazioni subcellulari. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: La frazione subcellulare dimostra l'chiusura di FOXO1 tra il nucleo e il citoplasma in CLL. (A) Le cellule MEC-1 e(B)le cellule CLL primarie sono state pretrattate per 30 min con 100 nM A'D8055 (8055) o lasciate non trattate (NDC) come indicato e quindi BCR è stato ligate per 1 h o lasciato dagli Stati Uniti. Le frazioni nucleari e citoplasmatiche sono state poi preparate e immunoblotte. Dopo la conferma della frazione mediante indagine con anticorpi anticorpi anticorpi anti-lamin A/C (nucleari) e anti-tubulina (citoplasma) anti-lamina, l'effetto sia del trattamento farmacologico che della legatura BCR è stato valutato sull'espressione della proteina FOXO1, utilizzando un anticorpo anti-FOXO1. M indica un marcatore di peso molecolare. Fare clic qui per visualizzare una versione più grande di questa figura.
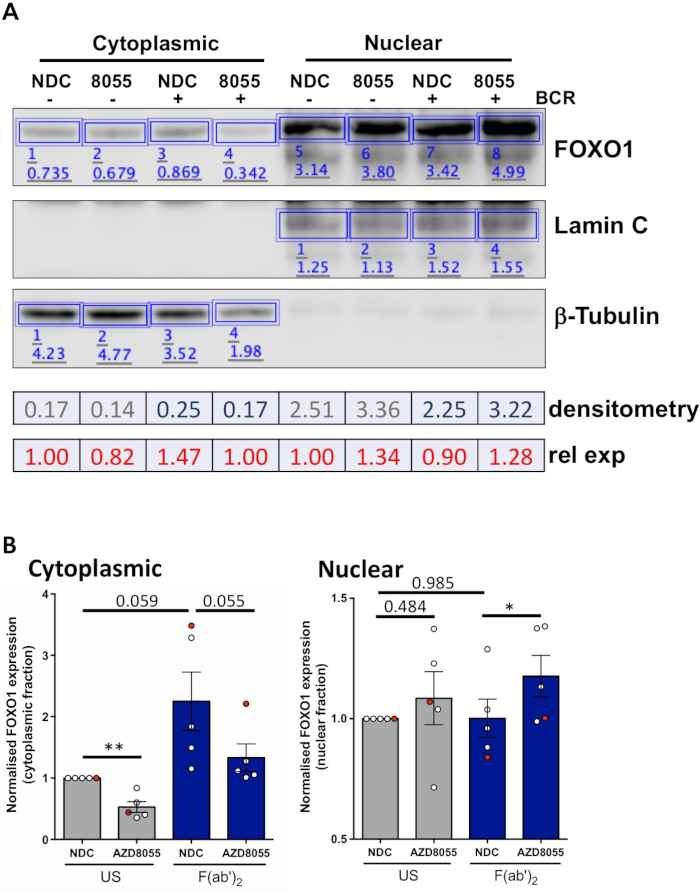
Figura 4: Un esempio elaborato di analisi quantitativa delle macchie occidentali (densitometria). (A) Densitometry è stato eseguito utilizzando il software di analisi western blot disponibile online. In breve, all'interno del nastro Di analisi, i rettangoli sono stati disegnati intorno alle bande proteiche nell'immagine per calcolare l'intensità del segnale. È raffigurata la densitometria di un'immagine commerciale rappresentativa di un campione di pazienti con CLL sottoposto a frazione citoplasmica/nucleare. Le frazioni citoplasmatiche e nucleari si distinguono per l'espressione di marcatori citoplasmatici (z-tubulina) e nucleari (Lamin A/C). L'espressione normalizzata di FOXO1 per una determinata condizione può essere calcolata dividendo il segnale ottenuto per FOXO1 per il segnale corrispondente per z-Tubulin o Lamin A/C, a seconda della frazione analizzata. L'espressione FOXO1 relativa (relativa al controllo del veicolo statunitense), può essere calcolata dividendo l'espressione FOXO1 normalizzata di una determinata condizione dall'espressione FOXO1 normalizzata del controllo veicolo statunitense di una determinata frazione cellulare. (B) Grafico che mostra i livelli di espressione FOXO1 nelle frazioni citoplasmatiche (sinistra) o nucleari (a destra) normalizzate al controllo US-NDC all'interno di ciascuna frazione cellulare. Il punto rosso sul grafico è l'esempio elaborato mostrato. Questi dati mostrano la variazione media della piega nell'espressione FOXO1 rispetto ai valori US-NDC - SEM. P sono stati determinati da studenti a due code abbinati t test. n - 5 campioni di pazienti cLL individuali. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Il protocollo descritto fornisce un metodo rapido ed efficiente per la generazione di frazioni nucleari e citoplasmatiche dalle cellule LL primarie e la successiva quantificazione del traffico di proteine tra le frazioni nucleari e citoplasmiche sulla stimolazione cellulare e il trattamento farmacologico. I dati presentati dimostrano la capacità di rilevare il traffico di proteine specifiche, ad esempio FOXO1, tra le frazioni nucleari e citoplasmiche, dopo il trattamento con un inibitore a doppio mTOR A-D8055 in presenza/assenza di BCR che si incrocia attraverso F( ab')2 stimolazione a frammentazione (Figura 3 e Figura 4 ). L'accoppiamento di questi esperimenti con la quantificazione delle macchie occidentali da singoli campioni di pazienti affetti da CLL, consente analisi obiettive dei dati generati e dimostra la robustezza dell'analisi descritta per quantificare i cambiamenti globali nella localizzazione delle proteine in Cellule CLL isolate dalle coorti dei pazienti (Figura 4). Dai dati è chiaro che una media di cinque campioni di pazienti nelle frazioni citoplasmiche ha raggiunto un significato quasi quasi significativo. Data l'eterogeneità clinica dei pazienti affetti da LLC11,queste analisi vengono normalmente eseguite su coorti di pazienti più grandi e/o focalizzate su sottogruppi prognostici specifici di pazienti per ottenere una comprensione più completa della risposta cellulare della CLL specifici trattamenti farmacologici.
I dati presentati dimostrano l'importanza di scegliere marcatori proteici che risiedono esclusivamente nelle frazioni citoplasmate o nucleari, poiché la purezza della frazione sarà confermata da questi marcatori. Per la conferma della frazione citoplasmica, fu scelta la tubulina e Lamin A/C come marcatore nucleare. Proteine aggiuntive comunemente utilizzate sono GAPDH e tubulina per identificare la frazione citoplasmica o Brg1 (SMARCA4), TFIID e RNA Polymerase II per la purezza della frazione nucleare4,5. Tuttavia, occorre prestare attenzione quando si scelgono proteine altamente arricchite in frazioni specifiche e non presenti in entrambe le frazioni (ad es. tubulina ztubulina) (Figura 2C)9. Infatti, GAPDH e actina, pur generalmente considerate proteine citoplasmiche possono localizzare al nucleo12,13, evidenziando l'importanza di scegliere un marcatore di frazione che non si ripoloche quando la stimolazione o il trattamento è applicato alle cellule. Inoltre, è importante confermare che il marcatore proteico scelto è espresso nella cellula di interesse eseguendo il WCL insieme alle frazioni subcellulari.
Nell'esperimento rappresentativo mostrato, lo stesso numero di cellule CLL è stato utilizzato per ogni condizione (stimolazione / trattamento farmacologico), e successivamente i campioni di frazione sono stati preparati immediatamente. Il caricamento di 10 g di proteine/lane frazionate fornisce materiale sufficiente per il rilevamento delle proteine di interesse. Poiché questi campioni sono stati sottoposti solo a un trattamento e stimolazione farmacologica a breve termine (fino a 4 h), si è ipotizzato che il livello di proteina sarebbe rimasto lo stesso in ogni campione e non è stato eseguito un test proteico. Tuttavia, se i trattamenti cellulari vengono estesi (18- 72 h), il livello di morte cellulare o proliferazione nelle cellule può alterare significativamente la qualità e la quantità di proteine estratte, a seconda della stimolazione farmaco/cellula applicata, alterando così i livelli di proteine nel trattamento /campioni stimolati. In questi casi per trattamenti farmacologici a lungo termine, è consigliabile effettuare la quantificazione delle proteine utilizzando un saggio Bradford o equivalente, prima del gonfiore occidentale per garantire che la stessa quantità di proteine venga eseguita in ogni corsia dell'immunoblot. La presenza di detergenti può interferire con specifici saggi proteici14, questa interferenza può essere ridotta diluindo campioni di proteine della frazione cellulare. Inoltre, utilizzare il buffer di lisi completo come vuoto, utilizzando la stessa diluizione come nei campioni in fase di test.
Per fornire prove a sostegno dei risultati qui descritti, è possibile eseguire esperimenti paralleli utilizzando la microscopia a fluorescenza per analizzare la posizione di FOXO1 all'interno delle cellule CLL per consentire la visualizzazione di questi risultati5. Inoltre, le frazioni subcellulari generate possono essere utilizzate anche per i saggi di attività enzimatica o l'analisi proteomica in ulteriori analisi a valle.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Gli autori desiderano ringraziare la dott.ssa Natasha Malik per aver esaminato criticamente il manoscritto. Questo studio è stato finanziato da una sovvenzione del progetto Bloodwise assegnata ad AMM (18003). Le strutture di analisi FACS sono state finanziate dalla Fondazione Howat. La MWM è stata finanziata da un dottorato di ricerca presso il centro di ricerca Friends of Paul O'Gorman Leukaemia, JC è stato finanziato dal Centro di ricerca Friends of Paul O'Gorman Leukaemia e JH è stato finanziato da una sovvenzione per il progetto Bloodwise (18003).
Materiali
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge Tubes | Griener Bio one | 616201 | |
| 3 mL Pasteur Pipettes | Griener Bio one | 612398 | |
| 12 mm x 75 mm FACS Tubes | Elkay | 2052-004 | |
| 15 mL Tube | Griener Bio one | 188271 | |
| 50 mL Tube | Griener Bio one | 227261 | |
| anti-CD5 FITC antibody | BD Biosciences | 555352 | phenotypic surface marker |
| anti-CD19 PE Cy7 antibody | BD Biosciences | 557835 | phenotypic surface marker |
| anti-CD23 APC antibody | BD Biosciences | 558690 | phenotypic surface marker |
| anti-CD45 APC Cy7 antibody | BD Biosciences | 557833 | phenotypic surface marker |
| anti-β-Tubulin antibody | Cell Signaling | 2146 | cytoplasmic marker |
| anti-γ-Tubulin Mouse antibody (clone GTU-88) | Sigma-Aldrich | T5326 | |
| anti-FoxO1 (C29H4) Rabbit antibody | Cell Signaling | 2880 | |
| anti-Lamin A/C antibody | Cell Signaling | 2032 | nuclear marker |
| anti-mouse IgG, HRP-linked Antibody | Cell Signaling | 7076 | Secondary antibody |
| anti-rabbit IgG, HRP-linked Antibody | Cell Signaling | 7074 | Secondary antibody |
| anti-Rpb1 CTD antibody (clone 4H8) | Cell Signaling | 2629 | nuclear marker |
| BDFACS Canto II | BD Biosciences | By Request | Flow Cytometer |
| DAPI Solution | BD Biosciences | 564907 | live/dead discriminator |
| DMSO | Sigma | D2650 | |
| EDTA | Sigma | EDS | |
| Fetal Bovine Serum | Thermofisher | 10500064 | |
| GraphPad Prism 6 | GraphPad Software | Software | |
| Histopaque1077 density gradient media | Sigma | H8889 | |
| HyperPAGE 10 - 190 kDa protein marker | Bioline | BIO-33066 | Molecular weight marker |
| Image Studio Lite (version 5.2.5) | LI-COR | www.licor.com | Software |
| Labnet VX100 | Fisher Scientific | Vortex | |
| Nucelar Extract Kit | Active Motif | 40010 | |
| OneComp eBeads | Thermofisher | 01-111-42 | |
| Trypan Blue Solution | Thermofisher | 15250061 | |
| PageRuler Plus Prestained Protein Ladder | Thermo Fisher Scientific | 26619 | Molecular weight marker |
| PBS Tablets | Fisher Scientific | BR0014G | |
| RosetteSep Human B Cell Enrichment Cocktail | Stem Cell Technologies | 15064 | |
| Sigma 3-16P | SciQuip | Centrifuge | |
| Sigma 1-15PK | SciQuip | Centrifuge | |
Riferimenti
- Kau, T. R., Way, J. C., Silver, P. A. Nuclear transport and cancer: from mechanism to intervention. Nature Reviews Cancer. 4, 106-117 (2004).
- Turner, J. G., Dawson, J., Sullivan, D. M. Nuclear export of proteins and drug resistance in cancer. Biochemical Pharmacology. 83, 1021-1032 (2012).
- Nakamura, N., et al. Forkhead transcription factors are critical effectors of cell death and cell cycle arrest downstream of PTEN. Molecular and Cellular Biology. 20, 8969-8982 (2000).
- Calnan, D. R., Brunet, A. The FoxO code. Oncogene. 27, 2276-2288 (2008).
- Cosimo, E., et al. AKT/mTORC2 inhibition activates FOXO1 function in CLL cells reducing B cell receptor-mediated survival. Clinical Cancer Research. 25, 1574-1587 (2019).
- Mahipal, A., Malafa, M. Importins and exportins as therapeutic targets in cancer. Pharmacology & Therapeutics. 164, 135-143 (2016).
- Hing, Z. A., et al. Next-generation XPO1 inhibitor shows improved efficacy and in vivo tolerability in hematological malignancies. Leukemia. 30, 2364-2372 (2016).
- Lapalombella, R., et al. Selective inhibitors of nuclear export show that CRM1/XPO1 is a target in chronic lymphocytic leukemia. Blood. 120, 4621-4634 (2012).
- Hořejší, B., et al. Nuclear γ-tubulin associates with nucleoli and interacts with tumor suppressor protein C53. Journal of Cellular Physiology. 227, 367-382 (2012).
- McCaig, A. M., Cosimo, E., Leach, M. T., Michie, A. M. Dasatinib inhibits B cell receptor signalling in chronic lymphocytic leukaemia but novel combination approaches are required to overcome additional pro-survival microenvironmental signals. British Journal of Haematology. 153, 199-211 (2011).
- Fischer, K., Hallek, M. Optimizing frontline therapy of CLL based on clinical and biological factors. Hematology. American Society of Hematology. Education Program. 2017, 338-345 (2017).
- Butera, G., et al. Regulation of autophagy by nuclear GAPDH and its aggregates in cancer and neurodegenerative disorders. International Journal of Molecular Sciences. 20, 2062(2019).
- Virtanen, J. A., Vartiainen, M. K. Diverse functions for different forms of nuclear actin. Current Opinion in Cell Biology. 46, 33-38 (2017).
- Friedenauer, S., Berlet, H. H. Sensitivity and variability of the Bradford protein assay in the presence of detergents. Analytical Biochemistry. 178, 263-268 (1989).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon