Method Article
Un metodo automatizzato per eseguire l'analisi in vitro micronucleoutilizzando la citometria del flusso di imaging multispettrale
In questo articolo
Riepilogo
L'analisi in vitro micronucleo è un metodo consolidato per valutare la genotossicità e la citotossicità, ma segnare l'analisi utilizzando la microscopia manuale è laborioso e soffre di soggettività e variabilità inter-marcatore. Questo documento descrive il protocollo sviluppato per eseguire una versione completamente automatizzata dell'analisi utilizzando la citometria del flusso di imaging multispettrale.
Abstract
L'analisi in vitro micronucleo (MN) è spesso usata per valutare la citotossicità e la genotossicità, ma segnare l'analisi tramite microscopia manuale è laborioso e introduce incertezza nei risultati a causa della variabilità tra i marcatori. Per ovviare a questo problema, sono state introdotte la microscopia automatica a scansione delle diapositive e i metodi di citometria di flusso convenzionali nel tentativo di rimuovere la distorsione del marcatore e migliorare la produttività. Tuttavia, questi metodi hanno le loro limitazioni intrinseche come l'incapacità di visualizzare il citoplasma della cellula e la mancanza di verifica MN visiva o archiviazione dei dati immagine con citometria di flusso. La citometria MIFC (Multispectral Imaging Flow) ha il potenziale per superare queste limitazioni. MIFC combina le immagini fluorescenti ad alta risoluzione della microscopia con la robustezza statistica e la velocità della citometria di flusso convenzionale. Inoltre, tutte le immagini raccolte possono essere memorizzate in file specifici della dose. Questo documento descrive il protocollo sviluppato per eseguire una versione completamente automatizzata del test MN su MIFC. Le cellule linfoblastoidi umane TK6 sono state ingrandite utilizzando una soluzione ipotonica (75 mM KCl), fissata con il 4% di formalina e il contenuto nucleare è stato macchiato con Hoechst 33342. Tutti i campioni sono stati eseguiti in sospensione sul MIFC, consentendo l'acquisizione di immagini ad alta risoluzione di tutti gli eventi chiave necessari per il saggio (ad esempio cellule binucleate con e senza MN, nonché cellule mononucleate e polucleate). Le immagini sono state automaticamente identificate, categorizzate ed enumerate nel software di analisi dei dati MIFC, consentendo il punteggio automatizzato sia della citotossicità che della genotossicità. I risultati dimostrano che l'utilizzo di MIFC per eseguire il test in vitro MN consente di rilevare aumenti statisticamente significativi della frequenza di MN a diversi livelli di citotossicità rispetto ai controlli dei solventi a seguito dell'esposizione delle cellule TK6 Mitomicina C e Colchicina, e che non si osservano aumenti significativi della frequenza di MN in seguito all'esposizione a Mannitol.
Introduzione
L'analisi in vitro micronucleoleus (MN) è un test comunemente usato per valutare la citotossicità e la genotossicità come strumento di screening in diversi campi di studio come lo sviluppo chimico e farmaceutico, nonché il biomonitoraggio umano tra gli individui esposti a vari fattori ambientali, professionali o di stile di vita1,2,3. MN è costituito da frammenti di cromosomi o cromosomi interi generati durante la divisione cellulare che non sono incorporati in uno dei due nuclei principali della figlia. Dopo la telofase, questo materiale cromosomico si forma in un corpo individuale arrotondato all'interno del citoplasma separato da uno dei nuclei principali2. Pertanto, MN sono rappresentativi del danno al DNA e sono stati utilizzati per molti anni come endpoint nei test di genotossicità4. Il metodo più appropriato per misurare la MN è il saggio citochinesi-blocco micronucleo (CBMN). Utilizzando il saggio CBMN, la frequenza di MN nelle cellule binucleate (BNC) può essere valutata incorporando la CitochalasinA B (Cyt-B) nel campione. Cyt-B consente la divisione nucleare ma previene la divisione cellulare e quindi, limita il punteggio di MN ai BNC che si sono divisi solo una volta5.
I protocolli che utilizzano microscopia e citometria di flusso sono stati sviluppati e convalidati e sono regolarmente utilizzati per eseguire l'analisi in vitro MN6,7,8,9,10, 11,12,13,14. La microscopia trae vantaggio dall'essere in grado di confermare visivamente che le MN sono legittime, ma richiede molto tempo e è soggetta a variabilità tra i marcatori15. Per risolvere questo problema, sono stati sviluppati metodi automatizzati di microscopia per scansionare diapositive e catturare immagini di nuclei e MN16,17,18,19, ma il citoplasma non può essere visualizzato, rendendolo difficile determinare se un MN è effettivamente associato a una cella specifica. Inoltre, questi metodi hanno difficoltà nell'identificare le cellule polinucleate (POLY) (comprese le cellule tri e quadranucle) che sono necessarie per il calcolo della citotossicità quando si utilizza Cyt-B9. Metodi di citometria di flusso sviluppati per eseguire il saggio MN impiegano fluorescenza così come intensità di dispersione in avanti e laterale per identificare le popolazioni dei nuclei e MN che sono stati liberati dalla cellula dopo lisi20,21 ,22. Questo permette di acquisire i dati da diverse migliaia di celle in pochi minuti e permette l'analisi automatizzata23; tuttavia, l'incapacità di visualizzare le cellule rende impossibile confermare che gli eventi segnati sono autentici. Inoltre, il lising della membrana cellulare inibisce l'uso di Cyt-B, nonché la creazione di una sospensione che contiene altri detriti come aggregati cromosomici o corpi apoptotici e non c'è modo di differenziarli da MN24.
Alla luce di queste limitazioni, la citometria MIFC (Multispectral Imaging Flow) è un sistema ideale per eseguire il saggio MN in quanto combina le immagini fluorescenti ad alta risoluzione della microscopia con la robustezza statistica e la velocità della citometria di flusso convenzionale. In MIFC, tutte le cellule vengono introdotte in un sistema fluidico e quindi idrodinamicamente focalizzate al centro di una cuvette di celle di flusso. L'illuminazione ortogonale di tutte le cellule avviene attraverso l'uso di un diodo a emissione luminosa (LED) a campo luminoso (BF), un laser a dispersione laterale e (almeno) un laser fluorescente. I fotoni fluorescenti vengono catturati da uno dei tre obiettivi obiettivo ad alta apertura 20x, 40x o 60x e quindi passano attraverso un elemento di decomposizione spettrale. I fotoni vengono quindi concentrati su una fotocamera a dispositivo accoppiato a carica (CCD) per ottenere immagini ad alta risoluzione di tutte le celle che passano attraverso la cella di flusso. Per evitare sfocature o striature, il CCD opera in modalità DiC (Time Delay Integration) che tiene traccia degli oggetti trasferendo il contenuto dei pixel da una riga all'altra del CCD in sincronia con la velocità della cella in flusso. Le informazioni sui pixel vengono quindi raccolte dall'ultima riga di pixel. L'imaging TDI combinato con la decomposizione spettrale consente di acquisire simultaneamente fino a 12 immagini (2 BF, 10 fluorescenti) da tutte le cellule che passano attraverso la cella di flusso. Tutte le immagini acquisite vengono memorizzate in file di dati specifici del campione, consentendo l'analisi da eseguire in qualsiasi momento utilizzando il software di analisi dei dati MIFC. Infine, i file di dati mantengono il collegamento tra immagini cellulari e punti su tutti i grafici bivariati. Ciò significa che qualsiasi punto su una trama bivariata tradizionale può essere evidenziato e le corrispondenti immagini BF e fluorescenti verranno visualizzate25.
Recentemente, sono stati sviluppati metodi basati su MIFC per eseguire il test MN sia per la biodosimetria di radiazione triage26,27,28,29,30,31 e genetica tossicologia32,33 test. Questo lavoro ha dimostrato che le immagini cellulari dei nuclei principali, MN e il citoplasma possono essere immagine con una maggiore produttività rispetto ad altri metodi26. Tutti i tipi di cellule necessari per l'analisi, incluse le celle MONO, i BNC (con e senza MN) e le celle POLY, possono essere identificati automaticamente nel software di analisi dei dati MIFC e l'implementazione dei criteri di punteggio sviluppati da Fenech et al. l'uso di vari algoritmi matematici6,34. I risultati della biodosimetria hanno mostrato che le curve di calibrazione della risposta alla dose erano simili in grandezza a quelle ottenute da altri metodi automatizzati nella letteratura quando si quantifica il tasso di MN per BNC29. Inoltre, recenti lavori in tossicologia hanno dimostrato che le immagini di cellule MONO, BNC (con e senza MN) e cellule POLY possono essere automaticamente catturate, identificate, classificate ed enumerate utilizzando MIFC. Il protocollo e l'analisi dei dati hanno permesso il calcolo della citotossicità e genotossicità dopo aver esposto le cellule TK6 a diversi clastogeni e aneugeni32.
Il protocollo presentato in questo documento descrive un metodo per eseguire il saggio in vitro MN utilizzando MIFC. La tecnica di elaborazione del campione utilizzata in questo lavoro richiede meno di 2 h per elaborare un singolo campione ed è relativamente facile da eseguire rispetto ad altri metodi. L'analisi dei dati nel software di analisi MIFC è complicata, ma la creazione del modello di analisi può essere eseguita in poche ore seguendo i passaggi descritti in questo documento. Inoltre, una volta che il modello è stato creato, può essere applicato automaticamente a tutti i dati raccolti senza ulteriori lavori. Il protocollo descrive tutti i passaggi necessari per esporre le cellule TK6 a clastogeni e aneugeni, descrive come coltura, elaborare e macchiare le cellule e dimostra come acquisire immagini ad alta risoluzione utilizzando MIFC. Inoltre, questo documento illustra le attuali best practice per l'analisi dei dati nel software MIFC per identificare e assegnare automaticamente un punteggio alle cellule MONO, ai BNC e POLY allo scopo di calcolare sia la citotossicità che la genotossicità.
Protocollo
1. Preparazione del mezzo di coltura e coltura delle cellule TK6
NOTA: Alcune sostanze chimiche utilizzate in questo protocollo sono tossiche. Inalare, deglutire o contattare la pelle con Cytochalasin B può essere fatale. Indossare attrezzature protettive personali appropriate, tra cui un cappotto da laboratorio e due paia di guanti di nitrile. Lavare accuratamente le mani dopo il maneggiamento. La formalina/formaldeide è tossica se inalata o inghiottita; è irritante per gli occhi, il sistema respiratorio e la pelle; e può causare sensibilizzazione per inalazione o contatto cutaneo. C'è il rischio di gravi danni agli occhi. È un potenziale cancerogeno.
- Preparare 565 mL di 1x RPMI supporto di coltura. Aggiungere 5 mL di aminoacidi non essenziali MEM (100x), 5 mL di pyruvate di sodio (100 mM), 5 mL di penicillina-streptomycin-glutamine (100x) e 50 mL di siero bovino fetale (FBS) a una bottiglia da 500 mL di 1xMI140. Preparare il mezzo in un armadio di biosicurezza e conservare a 2-8 gradi centigradi. Riscaldare il mezzo a 37 gradi centigradi prima di aggiungerlo alle celle TK6 (vedere Tabella dei materiali).
- Scongelare 1 mL di cellule TK6 (conservate a -80 gradi centigradi in DMSO) in 10 mL di media. Centrifugare le cellule a 200 x g per 8 min e aspirare il super-natante. Trasferire le cellule a 50 mL di supporti e incubare a 37 gradi centigradi, 5% DI CO2. Il tempo di raddoppio delle cellule TK6 varia da 12 a 18 h e saranno necessari alcuni (3 o 4) passaggi affinché le cellule raggiungano il loro tasso massimo di proliferazione (vedi Tabella deimateriali).
- Coltura 100 mL di cellule ad una concentrazione di 7-8 x 105 cellule / mL.
2. Preparazione di clastogeni e/o aneugeni e cytochalasin B
- Preparare le concentrazioni di scorte appropriate di clastogeni e aneugeni desiderati. Ad esempio, per Mitomycin C, sciogliere una bottiglia piena da 2 mg in 10 mL di acqua sterile per ottenere una concentrazione finale di scorte di 200 g/mL. La mitomicina C può essere conservata a 4 gradi centigradi per tre mesi (cfr. tabella dei materiali).
- Il giorno dell'esperimento, preparare le diluizioni delle sostanze chimiche desiderate che sono 10 o 100 volte superiori alle concentrazioni di esposizione desiderate se si diluisce rispettivamente in acqua sterile o DMSO.
- Per Mitomycin C, preparare diluizioni da 3 mL in acqua sterile di 0,5, 1,0, 2,0, 3,0, 4,0 e 5,0 g/mL. Per la colchiina, preparare diluizioni da 3 mL in acqua sterile di 0,05, 0,1, 0,2, 0,3, 0,4 e 0,5 g/mL. Infine, per Mannitol, preparare diluizioni da 3 mL in acqua sterile di 5, 10, 20, 30, 40 e 50 mg/mL.
- Preparare una concentrazione di scorte di Cytochalasin B di 200 g/mL di Cytochalasin B sciogliendo una bottiglia da 5 mg in 25 mL di DMSO. La ciclica B può essere conservata a -20 gradi centigradi per diversi mesi.
3. Esposizione delle cellule a clastogeni e/o aneugeni
- Aggiungere 1 mL di sostanze chimiche desiderate (ad esempio Mitomycin C) a 9 mL di cellule a 7-8x105 celle /mL in un pallone T25. Per i campioni di controllo, aggiungere 1 mL di acqua sterile. Collocare i flaconi in un'incubatrice di CO2 a 37 gradi, 5% per 3 h.
NOTA: Se le sostanze chimiche vengono diluite in DMSO, aggiungere solo 100 l l della sostanza chimica a ciascun pallone e aggiungere 100 L di DMSO ai controlli. Ogni pallone deve contenere 9.900 mL di celle. - Dopo 3 h, rimuovere i flaconi dall'incubatrice e trasferire le cellule a tubi di polipropilene da 15 mL. Centrifuga a 200 x g per 8 min, aspirare le cellule supernatali e di trasferimento a nuovi flaconi T25 contenenti un totale di 10 mL di mezzo di coltura fresca. Aggiungete 150 l della concentrazione di stock (200 g/mL) di Cytochalasin B ad ogni flacone per ottenere una concentrazione finale di 3 g/mL.
- Riportare i flaconi all'incubatore di CO2 del 5% per un tempo di recupero pari a 1,5-2,0 tempi di raddoppio, come raccomandato dalle linee guida dell'OCSE9. Per le cellule TK6 utilizzate in questo lavoro, il tempo di recupero era di 24 h.
NOTA: Il tempo di raddoppio delle cellule TK6 utilizzate qui è stato di 15 h ed è stato utilizzato un tempo di recupero di 24 h (1,6 tempi di raddoppio). I tempi di recupero inferiori a 1,5 volte il raddoppio ridurranno la proliferazione di campioni esposti a dosi più elevate che influiscono sul numero di BNC. Al contrario, i tempi di recupero superiori a 2,0 produrranno un numero sproporzionato di cellule polurtate nei campioni di controllo, che inclinano i calcoli della citotossicità.
4. Preparazione di buffer per la fissazione e l'etichettatura del contenuto del DNA (vedere Tabella dei materiali)
- Preparare 75 mM di cloruro di potassio (KCl) aggiungendo 2,79 g a 500 mL di acqua ultrapura. Mescolare la soluzione per 5 min utilizzando un agitatore magnetico e filtro sterile attraverso un filtro da 200 m. La soluzione KCl da 75 mM può essere conservata a 4 gradi centigradi per diversi mesi.
- Preparare una quantità sufficiente di 4% formalina per l'esperimento, prevedendo che un totale di 2,1 mL deve essere aggiunto ad ogni campione. Ad esempio, per preparare 10 mL di formalina del 4%, aggiungere 4 mL di 10% di scorte formaline a 6 mL di 1x soluzione Saline fosfato di Dulbecco senza Ca2 o Mg2 (PBS). Questa formalina del 4% può essere conservata a temperatura ambiente per diverse settimane.
- Preparare 510 mL di buffer di lavaggio (2% FBS in 1X PBS) aggiungendo 10 mL di FBS a una bottiglia da 500 mL di 1x PBS.
- Preparare 10 mL di una concentrazione di 100 g/mL di Hoechst 33342 aggiungendo 100 l della concentrazione di stock (1 mg/mL) a 9.900 gradi l di 1X PBS. La soluzione Hoechst 33342 può essere conservata a 4 gradi centigradi per diversi mesi.
5. Elaborazione del campione: gonfiore ipotonico, fissazione, conteggio cellulare ed etichettatura del contenuto di DNA
- Alla fine del periodo di recupero, rimuovere tutti i flaconi dall'incubatrice e trasferire tutti i campioni a tubi di polipropilene da 15 mL. Centrifuga tutti i campioni a 200 x g per 8 min.
- Aspirati il superatante, risospendere le cellule e aggiungere 5 mL di 75 mM KCl. Mescolare delicatamente per inversione tre volte e incubare a 4 gradi centigradi per 7 min.
- Aggiungere 2 mL di 4% di formalina ad ogni campione, mescolare delicatamente per inversione tre volte e incubare a 4 gradi centigradi per 10 min. Questo passaggio funge da "fissazione morbida".
- Centrifugare tutti i campioni a 200 x g per 8 min. Aspirate the supernatant and resuspend in 100 : L of 4% formalin for 20 min. Questo passaggio funge da "fissazione rigida".
- Aggiungere 5 mL di tampone di lavaggio e centrifugare a 200 x g per 8 min. Aspirare il supernatante e risospendere in 100 .
- Trasferire tutti i campioni su tubi di microcentrifuga da 1,5 ml.
- Eseguire un conteggio delle celle su ogni campione per determinare il numero di celle per campione. I campioni saranno altamente concentrati, quindi sarà probabilmente necessaria una diluizione di 1:100 in 1x PBS (10 : L di campione in 990 l di PBS) per ottenere un conteggio accurato.
NOTA: A questo punto è meglio eseguire il numero di cellule utilizzando un emocitometro. L'aggiunta di KCl conferisce al citoplasma un aspetto traslucido, rendendo difficile per i contatori cellulari automatizzati riconoscerli. Inoltre, i contatori automatici hanno difficoltà a segnare le cellule polincleate a causa delle loro dimensioni. - Se non si eseguono i campioni sul MIFC immediatamente, possono essere conservati a 4 gradi centigradi per diversi giorni. Quando si è pronti per l'esecuzione dei campioni, aggiungere ad ogni campione 5 o luna di 100 g/mL per 1x106 celle/mL. Aggiungete anche 10 luna di 500 g/mL di RNase per 100 l' di campione per una concentrazione finale di 50 g/mL. Incubare i campioni a 37 , 5% CO2 per 30 min.
- Microcentrifuga tutti i campioni a 200 x g per 8 min e utilizzare una pipetta per rimuovere il super-natante lasciando 30 dollari l. Utilizzare una pipetta per risospendere tutti i campioni prima di essere in esecuzione sul MIFC assicurando che non ci siano bolle nel tubo. Non vortice.
6. Avvio e calibrazione del MIFC
- Assicurarsi che la boroda, il reagente di calibrazione del sistema, il debubbler, il detergente e i contenitori sterilizzatori siano pieni e che il serbatoio dei rifiuti sia vuoto. Accendere il sistema e fare doppio clic sull'icona del software MIFC. Fare clic sul pulsante Avvio e assicurarsi che la casella di controllo Avvia tutte le tarature e i test sia selezionata. Questo scaricherà il sistema, caricherà la diaposizione e i reagenti di calibrazione del sistema e calibra il sistema (vedere Tabella dei materiali).
7. Esecuzione di campioni sul MIFC
NOTA: in questa sezione si presuppone l'uso di una telecamera MIFC a 2. Se si utilizza una fotocamera 1 MIFC, si prega di consultare Supplemento 1 - Protocollo completo, sezione 7 per la creazione di grafici durante l'acquisizione
- Avviare il software di acquisizione dati MIFC (vedere Tabella dei materiali). Figura 1 Mostra le impostazioni dello strumento. Accendere il laser da 405 nm e impostare la potenza del laser su 10 mW (A). Disattivare tutti gli altri laser (incluso SSC) e impostare il BF sui canali 1 e 9 (B). Verificare che il dispositivo di scorrimento dell'ingrandimento sia impostato su 60x (C), che sia selezionata la modalità ad alta sensibilità (D) e che nella raccolta immagini vengano visualizzati solo i canali 1, 7 e 9.
- Fare clic sull'icona Grafico a dispersione. Selezionare Popolazione Tutto e selezionare Area M01 sull'asse X e Rapporto aspetto M01 sull'asse Y. Fare clic sull'icona Regione quadrata e disegnare un'area intorno alle singole celle. Assegnare all'area il nome Celle singole. Fare clic con il pulsante destro del mouse sul grafico e selezionare Regioni. Evidenziare l'area Celle singole e modificare le coordinate x in 100 e 900 e modificare le coordinate y in 0,75 e 1 (Figura 1I).
- Fare clic sull'icona Grafico a dispersione. Selezionare Celle singole come popolazione padre, selezionare Gradient RMS M01 sull'asse X e Gradient RMS M07 sull'asse Y. Fare clic sull'icona Regione quadrata e disegnare un'area intorno alla maggior parte delle celle. Assegnare all'area il nome Celle attive. Fare clic con il pulsante destro del mouse sul grafico e selezionare Regioni. Evidenziare l'area Celle con lo stato attivo e modificare le coordinate x in 55 e 75 e modificare le coordinate y in 9.5 e 20 ( Figura1J).
- Fare clic sull'icona Istogramma. Selezionare la popolazione Di celle focalizzate e selezionare Intensità M07 come funzione. Fare clic sull'icona Regione lineare e disegnare una regione sul picco principale dell'istogramma. Denominare questa regione DNA-positivo. Fare clic con il pulsante destro del mouse sul grafico e selezionare Regioni. Evidenziare la regione di DNA positivo e modificare le coordinate in 2 x 105 e 2 x 106. L'intervallo potrebbe dover essere regolato a seconda del picco di intensità dell'istogramma (Figura 1K).
- Impostare i parametri di acquisizione (Figura 1E). Specificare il nome del file e la cartella di destinazione, modificare il numero di eventi in 20.000 e selezionare la popolazione DNA-positiva.
- Fare clic su Carica (Figura 1F) e inserire l'esempio di controllo nel MIFC. Fare clic sul pulsante Acquisisci per raccogliere i dati (Figura 1G). Una volta completata l'acquisizione, fare clic sul pulsante Indietro per restituire l'esempio (Figura 1H). Rimuovere il tubo campione dallo strumento. Ripetere questo processo per tutti i campioni rimanenti nell'esperimento.
8. Apertura di un file di dati in IDEAS
- Avviare il pacchetto software di analisi MIFC (vedere la Tabella dei materiali). Fare clic su Avvia analisi per avviare l'Apertura guidata file. Selezionare un file di dati individuando il file di immagine raw desiderato (.rif). Fare clic sul pulsante Apri e quindi su Avanti.
- Poiché si tratta di un singolo saggio a colori, la compensazione non è necessaria, quindi fare clic su Avanti per ignorare la fase di compensazione. In questa fase non è disponibile alcun modello di analisi da applicare, quindi fare di nuovo clic su Avanti. Se il modello di analisi è stato scaricato dal materiale supplementare, selezionarlo ora. Questi modelli funzionano solo con una telecamera 2 MIFC con BF impostato su canali 1 e 9 e immagini nucleari nel canale 7 durante l'acquisizione.
- Per impostazione predefinita, i nomi dei file .cif e .daf vengono generati automaticamente in modo che corrispondano a .rif. Non è consigliabile modificare i nomi dei file .cif e .daf. Fare clic su Avanti. Impostare le proprietà di visualizzazione dell'immagine selezionando i file 01 e 07. Fare clic su Avanti. Non esiste una procedura guidata per questa applicazione, quindi fare clic su Fine. È molto importante salvare il file di analisi dei dati (.daf) e il modello di analisi (.ast) spesso durante le sezioni da 9 a 14 per evitare perdite di stato.
9. Creazione di maschere e funzioni per identificare i BNC
- Fare clic sull'icona Proprietà Raccolta immagini (icona blu/bianca). Nella scheda Proprietà di visualizzazione, fare clic su Imposta intervallo su pixel dati, quindi modificare il colore in giallo. Fare clic su OK. Le immagini Hoechst sono ora più facili da visualizzare sullo sfondo nero.
- Creare il grafico di celle non apoptotiche.
- Fare clic sulla scheda Analisi, quindi fare clic su Maschere. Fare clic su Nuovo, quindi su Funzione. In Funzione scegliere Soglia, in Maschera scegliere M07 e impostare Percentuale intensità su 50. Fare clic su OK, quindi di nuovo su OK. Fare clic su Chiudi.
- Fare clic sulla scheda Analisi , fare clic su Funzionalità, quindi fare clic su Nuovo. Per Il tipo di funzione, selezionare Area. In Maschera selezionare Soglia(M07,Ch07,50). Fate clic su Imposta nome predefinito (Set default Name) e quindi su OK. Fare clic su Chiudi per avviare il calcolo dei valori della funzione.
- Fare clic sull'icona Stampa punti. Selezionare la popolazione Tutto. Per la feature dell'asse X, scegliete la feature Contrasto_M01_Ch01 e per la feature dell'asse Y, scegliete Soglia_area(M07,Ch07,50). Fare clic su OK.
- Fare clic sul pulsante Area quadrata e disegnare un'area intorno alla maggior parte delle celle. Chiamare questa regione Non-apoptotic. Fare clic con il pulsante destro del mouse sul grafico e scegliere Regioni. Evidenziare la regione non apoptotica. Impostare le coordinate x su 0 e 15 e impostare le coordinate y su 50 e 300. Fare clic su Chiudi.
- Creare la maschera BNC (passaggi 9.3.1-9.3.5) per identificare le cellule che contengono solo due nuclei.
- Cercare un BNC nella galleria di immagini e fare clic su di esso. Questo per visualizzare la creazione della maschera nel canale Hoechst.
- Fare clic sulla scheda Analisi, quindi fare clic su Maschere. Fare clic su Nuovo, quindi su Funzione. In Funzione scegliere LevelSet, in Maschera scegliere M07, selezionare il pulsante di opzione Maschera di livello intermedio e impostare Scala dettagli contorno su 3.00. Fare clic su OK, quindi di nuovo su OK.
- Fare clic su Nuovo, quindi su Funzione. In Funzionescegliere Dilatae in Maschera scegliere LevelSet(M07,Ch07,Middle,3). Impostare l'immagine da visualizzare su Ch07e impostare Numero di pixel su 2. Fare clic su OK, quindi di nuovo su OK.
- Fare clic su Nuovo, quindi su Funzione. In Funzione scegliere Spartiacquee in Maschera scegliere Dilato(LevelSet(M07,Ch07,Middle,3)2). Impostare l'immagine da visualizzare su Ch07e spessore linea su 1. Fare clic su OK, quindi di nuovo su OK.
- Fare clic su Nuovo, quindi su Funzione. In Funzione scegliere Intervallo, in Maschera scegliere Bacino idrografico(Dilate(LevelSet(M07,Ch07,Middle,3)2)). Impostare l'immagine da visualizzare su Ch07. Impostare i valori di area minimo e massimo rispettivamente su 115 e 5000. Impostare i valori delle proporzioni minime e massime rispettivamente su 0,4 e 1. Fare clic su OK. Nel campo Nome modificare il testo in cui leggere BNC, quindi fare clic su OK.
- Creare le feature e i grafici per ottenere la popolazione BNC finale
- Funzione BNC di conteggio spot: fare clic sulla scheda Analisi , quindi su Feature, quindi su Nuovo. Per Tipo di feature, selezionare Conteggio macchie. In Maschera ( Maschera), selezionate la maschera BNC finale creata in 9.3.5. Impostare Connessione su Quattro e modificare il nome in Conteggio macchie BNC. Fare clic su OK, quindi su Chiudi per calcolare i valori della funzione.
- Istogramma del conte spot BNC. Fare clic sull'icona Istogramma. Selezionare Non apoptotico come popolazione padre. Per la funzione Asse X, scegliete la funzione Conteggio macchie BNC. Fare clic su OK. Fare clic sull'icona Regione lineare. Disegnare un'area nel raccoglitore 2. Chiamare questa regione 2N.
NOTA: Fare riferimento alla sezione 9 del Supplemento 1 - Protocollo completo per creare le maschere, le caratteristiche e i grafici rimanenti per identificare la popolazione finale di BNC
10. Creazione di maschere e funzionalità per identificare MN all'interno della popolazione BNC
- Creare la maschera MN. Cercare un BNC che contiene un MN nella galleria di immagini e fare clic su di esso. Questo per visualizzare la creazione della maschera MN nel canale Hoechst. Fare clic sulla scheda Analisi, quindi fare clic su Maschere.
- Crea maschera di identificazione spot 1:
- Fare clic su Nuovo, quindi su Funzione. In Funzione scegliere Spot e verificare che il pulsante di opzione Luminoso sia selezionato. In Maschera scegliere M07, impostare Rapporto sfondo spot-cella su 2,00. Impostare Raggio minimo su 2 e Raggio massimo su 6. Fare clic su OK, quindi di nuovo su OK.
- Fare clic su Nuovo, quindi su Funzione. In Funzione scegliere Intervalloe in Maschera scegliere LevelSet(M07,Ch07,Middle,3). Impostare l'immagine da visualizzare su Ch07. Impostare rispettivamente Area minima e Massima su 80 e 5000. Impostare il rapporto di aspetto minimo e massimo su 0 e 1, rispettivamente. Fare clic su OK, quindi di nuovo su OK.
- Fare clic su Nuovo, quindi su Funzione. In Funzione scegliere Dilata, in Maschera scegliere Intervallo(Setdi livello(M07,Ch07,Medio,3),80-5000,0-1). Impostare l'immagine da visualizzare su Ch07. Impostare Il numero di pixel su 2. Fare clic su OK, quindi di nuovo su OK.
- Fare clic su Nuovo. Fare doppio clic sulla maschera Spot(M07, Ch07, Bright, 2, 6, 2) per aggiungerla alla definizione della maschera. Fare clic sull'operatore And e quindi sull'operatore Not. Fare doppio clic sulla maschera Dilate(Range(LevelSet(M07, Ch07, Middle, 3), 80-5000, 0-1), 2) per aggiungerla alla definizione della maschera. Fare clic su OK.
- Fare clic su Nuovo, quindi su Funzione. In Intervallo di scelta funzione e sotto maschera scegliere la maschera creata in 10.1.1.4:
- Selezionare Spot(M07, Ch07, Luminoso, 2, 6, 2) E Non dilatarne (Intervallo (Insieme di livelli (M07, Ch07, Medio, 3), 80-5000, 0-1), 2).
- Impostare l'immagine su Ch07. Impostare l'Area minima e massima su 10 e 80, rispettivamente. Impostare il rapporto di aspetto minimo e massimo su 0,4 e 1, rispettivamente. Fare clic su OK, quindi di nuovo su OK. La maschera di identificazione spot 1 è completa.
NOTA: Fare riferimento alla sezione 10 del Supplemento 1 - Protocollo completo per creare le maschere, le caratteristiche e i grafici per identificare la popolazione MN finale
- Crea maschera di identificazione spot 1:
11. Creare maschere, feature e trame per identificare le popolazioni mononucleate e poliincleate
- Create la maschera POLY. Fare clic su Analisi, quindi su Maschere, quindi su Nuovo e quindi su Funzione. In Funzione scegliere Intervallo, in Maschera scegliere Bacino idrografico(Dilate(LevelSet(M07, Ch07, Middle, 3), 2)). Impostare l'immagine da visualizzare su Ch07. Impostare i valori Area minima e Massima su 135 e 5000, rispettivamente. Impostare i valori di Rapporto aspetto minimo e massimo rispettivamente su 0,4 e 1. Fare clic su OK. Nel campo Nome modificare il testo in POLY, quindi fare clic su OK, quindi su Chiudi. La maschera di celle Polynucleated è completa.
- Create le maschere dei componenti POLY.
- Maschera componente POLY 1: fare clic sulla scheda Analisi, quindi su Maschere, quindi su Nuovo, quindi su Funzione. In Funzione selezionare Componentee in Maschera selezionare la maschera POLY. Per Caratteristica di classificazione selezionare Areae per Ordinamento fare clic sul pulsante di opzione Decrescente. Impostare Classifica su 1. Fare clic su OK, quindi di nuovo su OK.
- Poly Component Masks 2, 3 e 4: Ripetere tutti i passaggi in 11.2.1 ad eccezione di Rank su 2, 3 e 4 per creare le singole maschere componente.
- Conteggio macchie con maschera POLY.
- Fare clic sulla scheda Analisi , quindi su Funzionalità, quindi su Nuovo. Per Tipo di funzione, selezionare Conteggio macchie. Per Maschera, scegliete la maschera POLY e impostate Connessione a 4. Fare clic su Imposta nome predefinito e quindi su OK, quindi su Chiudi per calcolare i valori della funzione.
- Fare clic sull'icona Istogramma. Selezionare la popolazione non apoptotica. Per la feature dell'asse X, scegliete la feature Conteggio macchie_POLY_4.
- regione di conteggio spot MONO. Fare clic sull'icona Regione lineare. Disegnare una regione nel raccoglitore 1 sull'istogramma creato in 11.3.2. Chiamare questa regione 1N.
- Regione di conteggio spot TRI. Fare clic sull'icona Regione lineare. Disegnare una regione nel raccoglitore 3 sull'istogramma creato in 11.3.2. Chiamare questa regione 3N.
- QUAD MONO spot count regione. Fare clic sull'icona Regione lineare. Disegnare una regione nel raccoglitore 4 sull'istogramma creato in 11.3.2. Chiamare questa regione 4N.
- Identificare la popolazione MONO.
- Creare la funzione di proporzioni MONO. Fare clic sulla scheda Analisi , quindi su Funzionalità, quindi su Nuovo. In Tipo di feature, selezionare la funzione Proporzioni e in Maschera selezionare Componente(1, Area, POLI, Decrescente). Fare clic su Imposta nome predefinito, quindi su OK.
- Create la funzione di circolarità MONO. Con la finestra Gestione feature ancora aperta, fare clic su Nuovo. In Tipo di funzione, selezionare la funzione Circolarità e in Maschera selezionare Componente(1, Area, POLI, Decrescente). Fare clic su Imposta nome predefinito, quindi su OK, quindi su Chiudi per calcolare i valori della funzione.
- Per il grafico a punti delle celle MONO circolari, fare clic sull'icona Grafico punto. Selezionare 1N come popolazione padre. Per la Feature asse X scegliere Circularity_Component(1, Area, POLY, Descending) e per La Feature asse Y scegliere Aspect Ratio_Component(1, Area, POLY, Descending). Fare clic su OK. Fare clic sul pulsante Area quadrata e disegnare un'area intorno alla popolazione di celle verso la parte superiore destra del grafico. Denominare questa regione Circular_1N. Fare clic con il pulsante destro del mouse sul grafico e scegliere Regioni. Evidenziare l'area Circular_1N. Impostare le coordinate X su 20 e 55 e le coordinate Y su 0,85 e 1,0. Fare clic su Chiudi.
- Create la feature Poli/Area_M07. Fare clic sulla scheda Analisi , quindi su Funzionalità, quindi su Nuovo. In Tipo di feature, selezionare la funzione Area e in Maschera selezionare Componente(1, Area, POLI, Decrescente). Fare clic su Imposta nome predefinito, quindi su OK.
- Con la finestra Gestione funzionalità ancora aperta, fare clic su Nuovo, quindi in Tipo di feature fare clic sul pulsante di opzione Combinato. Dall'elenco delle feature, evidenziare Area_Component(1, Area, POLI, Discendente) e fare clic sulla freccia giù per aggiungerla alla definizione della feature. Fare clic sul simbolo di divisione (/). Selezionate la funzione Area_M07 e fate clic sulla freccia giù per aggiungerla alla definizione della feature. Fate clic su Imposta nome predefinito (Set default Name) e quindi su OK. Fare clic su Chiudi per avviare il calcolo dei valori della funzione.
- Per il grafico a punti di popolazione MONO finale, fare clic sull'icona del grafico a punti. Selezionate Circular_1N come popolazione padre. Per La feature asse X, scegliete Rapporto aspetto_M07 e per Feature asse Y, scegliete Area_Componente(1, Area, Poliero, Decrescente) / Area_M07. Fare clic su OK. Fare clic sul pulsante Area quadrata e disegnare un'area intorno alla maggior parte delle celle. Nome in questa regione Mononucleated. Fare clic con il pulsante destro del mouse sul grafico e scegliere Regioni. Evidenziare la regione mononucleated. Impostare le coordinate X su 0,85 e 1,0 e impostare le coordinate Y su 0,55 e 1,0. Fare clic su Chiudi.
NOTA: Fare riferimento alla sezione 11 del Supplemento 1 - Protocollo completo per creare le maschere, le caratteristiche e i grafici per identificare le popolazioni trinucleate e polucleate finali.
12. Creare una vista personalizzata per esaminare le maschere BNC e MN
- Fare clic sul pulsante Proprietà Raccolta immagini, quindi fare clic sulla scheda Visualizza. In Tipo di nome Ch01/Ch07. Fare clic su Aggiungi immagine. In Immagine scegliere Ch01 e impostare Percentuale su 100. Fare di nuovo clic su Aggiungi immagine, in Immagine scegliere Ch07 e impostare Percentuale su 100.
- Fare clic su Nuovo e in Tipo di nome Maschere BNC e MN
- Fare clic su Aggiungi colonna. In Tipo immagine scegliere Ch01 e in Maschera scegliere Nessuno
- Fare clic su Aggiungi colonna. In Tipo immagine scegliere Ch07 e in Maschera scegliere Nessuno
- Fare clic su Aggiungi colonna. In Tipo immagine scegliere Ch07 e in Maschera scegliere BNC
- Fare clic su Aggiungi colonna. In Tipo immagine scegliere Ch07 e in Maschera scegliere Maschera MN
- Fare clic su Aggiungi colonna. In Tipo immagine fare clic sul pulsante di opzione Composito. L'immagine composita Ch01/Ch07 deve essere aggiunta automaticamente alla vista. Fare clic su OK per chiudere la finestra Proprietà Raccolta immagini.
13. Creare una vista personalizzata per esaminare la maschera POLY
- Fare riferimento alla sezione 13 del Supplemento 1 - Protocollo completo per creare una visualizzazione personalizzata per esaminare la maschera POLY
14. Creare una tabella di statistiche per enumerare gli eventi chiave
- Fare clic sulla scheda Report, quindi fare clic su Definisci rapporto statistiche. Nella nuova finestra fare clic su Aggiungi colonne.
- Aggiungere la statistica del conteggio BNC. In Statistiche selezionare Conteggio e in Popolazione selezionata scegliere la popolazione BNC. Fare clic su Aggiungi statistiche per aggiungere la statistica all'elenco.
- Ripetere il passaggio 14.2 per creare colonne separate per le popolazioni MN BNCs, MONO, TRI e POLY . Fare clic su Chiudi, quindi su OK.
- Il modello di analisi dei dati è completo (Figura 2). Salvare il modello (File, Salva come modello). L'elenco completo delle maschere è disponibile in Supplemento 2 - Elenco maschere.
15. File di esperimento di processo batch utilizzando il modello di analisi dei dati
- Nel menu Strumenti fare clic su Batch Data Files, quindi fare clic su Aggiungi batch nella nuova finestra.
- Nella nuova finestra fare clic su Aggiungi file per selezionare i file dell'esperimento (.rif) da aggiungere al batch. Nell'opzione Selezionare un modello o un file di analisi dei dati (.ast, .daf) fare clic sull'icona della cartella aperta per individuare e aprire il modello di analisi dei dati (file ast) salvato nel passaggio 14.4.
- Fare clic sul pulsante Anteprima rapporto statistiche per visualizzare in anteprima la tabella delle statistiche. Nessun valore verrà visualizzato qui poiché non sono ancora stati calcolati. Tuttavia, questo passaggio serve come un controllo per assicurarsi che sia stato selezionato il modello di analisi corretto prima di eseguire il batch.
- Fare clic su OK per chiudere la finestra corrente. Quindi fare clic su Invia batch per avviare l'elaborazione batch di tutti i file.
- Al termine dell'elaborazione batch, nella cartella che contiene tutti i file rif sarà disponibile un file con estensione txt. Utilizzare queste statistiche per calcolare la genotossicità e la citotossicità.
16. Calcolo dei parametri di genotossicità e citotossicità
- Calcolo della genotossicità: per calcolare la genotossicità, utilizzare la tabella delle statistiche creata in 15,5. Dividere il numero di celle nella popolazione MN BNC per il numero di celle nella popolazione di BNM quindi moltiplicare per 100:

- Calcolo della citotossicità: determinare il numero totale di cellule POLY sommando il numero di celle TRI e QUAD.
- Calcolare l'indice di proliferazione del blocco di citocinesi (CBPI) utilizzando il numero di celle nei campi MONO, BNC e POLY come segue:
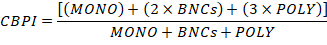
- Infine, calcolare la citotossicità di ogni coltura utilizzando i valori CBPI delle colture di controllo (C) e della coltura esposta chimicamente (T) come segue:

- Calcolare l'indice di proliferazione del blocco di citocinesi (CBPI) utilizzando il numero di celle nei campi MONO, BNC e POLY come segue:
Risultati
Il metodo di analisi descritto in questo documento consente l'identificazione e il punteggio automatici delle BNC, con e senza MN, per calcolare la genotossicità. Inoltre, le cellule MONO e POLY vengono identificate e valutate automaticamente per calcolare la citotossicità. I criteri di punteggio pubblicati6,34 che devono essere aderiti quando si segnano questi eventi vengono implementati nel software di analisi dei dati MIFC. I risultati qui presentati indicano che aumenti statisticamente significativi della frequenza di MN con crescente citotossicità possono essere rilevati dopo l'esposizione delle cellule TK6 del linfoblastoide umano a sostanze chimiche che inducono MN ben note (Mitomycin C e Colchicina). Risultati analoghi per le sostanze chimiche aggiuntive testate sono stati dimostrati in una pubblicazione separata32. Inoltre, i risultati dell'uso di Mannitol mostrano che le sostanze chimiche che non inducono MN possono anche essere identificate correttamente utilizzando il metodo MIFC qui descritto. I parametri descritti nel protocollo per creare tutte le maschere, le funzioni e i limiti delle regioni dovranno probabilmente essere regolati se vengono utilizzati diversi tipi di cellule (ad esempio celle di Cesster cinese) per eseguire l'analisi.
La figura 3 mostra quattro pannelli selezionati per identificare i BNC (Figura3A-3D). Di seguito è mostrato un istogramma che consente la selezione di celle con due nuclei (Figura 3A) e grafici bivariati che consentono la selezione di BNC con circolarità simile (Figura 3B), aree e intensità simili (Figura 3C ) e BnMc che hanno nuclei ben separati e non sovrapposti (Figura 3D) come da parte del punteggio critieria6,34. Figura 3 E mostra le immagini BF e Hoechst, nonché le maschere BNC e MN che indicano che i BNC con MN singolo o multiplo possono essere identificati ed enumerati. Ciò consente di calcolare la genotossicità determinando il tasso di Micronucleoned BNC nella popolazione finale del BNC. Figura 4 mostra l'applicazione della funzione Spot Count utilizzando la maschera POLY per identificare le celle MONO, TRI e QUAD. Il numero di celle TRI e QUAD può quindi essere sommato per ottenere il numero finale di celle POLY (Tabella1). Ciò consente di calcolare la citotossicità utilizzando la formula mostrata nel protocollo. Pertanto, ogni punto di dose nell'esperimento può essere valutato da entrambi i parametri di genotossicità e citotossicità.
Figura 5 Mostra i valori di genotossicità e citotossicità per l'aneugen Colchicine, il clastogen Mitomycin C e per un controllo negativo, Mannitol. Per la Colchiina (Figura 5A) le dosi da 0,02 a 0,05 g/mL hanno prodotto aumenti statisticamente significativi della frequenza DiN, che vanno rispettivamente dall'1,28% al 2,44% rispetto al controllo del solvente (Tabella 1). Nel caso di Mitomycin C (Figura 5B) le due dosi massime di 0,4 e 0,5 g/mL hanno prodotto frequenze MN statisticamente significative rispetto ai controlli del solvente. Queste frequenze di MN erano dello 0,93% a 0,4 g/mL e dell'1,02% a 0,5 g/mL (tabella 2). Infine, per Mannitol (Figura 5C), nessuna dose testata inducono una citotossicità superiore al 30%, né produceva aumenti significativi della frequenza di MN rispetto ai controlli dei solventi, come previsto (Tabella 3).

Figura 1 : impostazioni dello strumento MIFC. Schermata delle impostazioni MIFC come descritto nella sezione 7 del protocollo. (A) Impostare la potenza laser da 405 nm a 10 mW. (B) Impostazione dei canali BF 1 e 9. (C) Selezione dell'obiettivo di ingrandimento 60x. (D) Selezionare la velocità di flusso più lenta che genera immagini con la risoluzione più alta. (E) Specificare il numero di eventi da raccogliere a 20.000. (F) Fare clic sul pulsante Carica per avviare il processo di caricamento del campione. (G) Fare clic sul pulsante Acquisisci per iniziare ad acquisire immagini. (H) Fare clic sul pulsante Indietro per restituire qualsiasi campione inutilizzato. (I) Grafico a dispersione di BF Aspect Ratio rispetto all'area BF per la selezione di singole celle. (J) Scatterplot di Hoechst Gradient RMS contro BF Gradient RMS per la selezione delle celle a fuoco. (K) Istogramma dell'intensità di Hoechst per la selezione delle cellule positive del DNA. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2 : Strategia di gating software di analisi. Uno screenshot della strategia di gating descritta nella sezione 9 del protocollo. Le regioni sono mostrate in ordine sequenziale per l'identificazione delle cellule federate (scatola rossa), dei micronuclei (scatola gialla) e delle cellule monoe polucleate (scatola blu). Fare clic qui per visualizzare una versione più grande di questa figura.
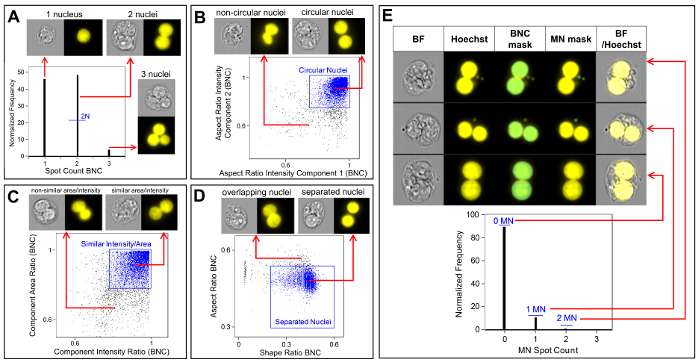
Figura 3 : Identificazione e punteggio dei BNC con e senza MN. (A) Selezione di cellule che hanno due nuclei distinti. (B) Identificazione delle cellule federate (BNC) che hanno due nuclei altamente circolari attraverso l'uso della funzione Intensità proporzioni. (C) Selezione dei BNC con nuclei con aree e intensità simili. Ciò si ottiene calcolando il rapporto tra l'area di entrambi i nuclei e il rapporto tra il rapporto di aspetto di entrambi i nuclei. (D) Uso delle funzioni Proporzioni forma e Rapporto di aspetto per identificare i BNC con due nuclei ben separati. (E) La funzione Spot Count che utilizza la maschera micronucleo (MN) dimostra che i BNC con MN singolo o multiplo possono essere identificati ed enumerati. Fare clic qui per visualizzare una versione più grande di questa figura.
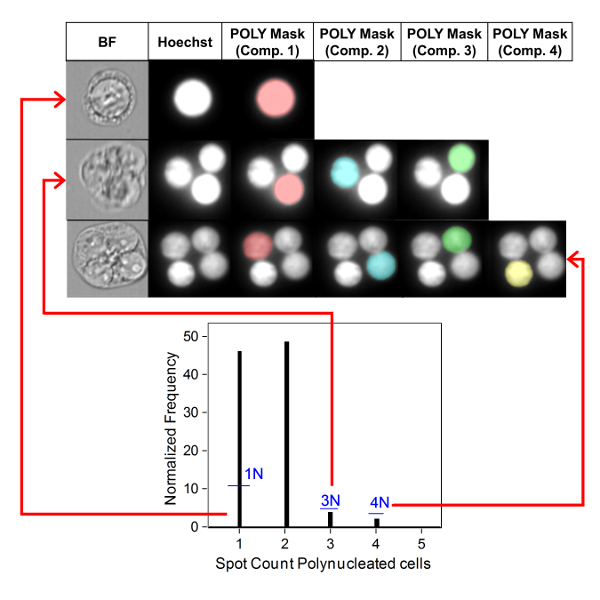
Figura 4 : Identificazione e punteggio delle celle MONO e POLY. Uso della funzione di conteggio spot per identificare ed enumerare le celle mono, tri e quadraricane. La maschera componente 1 consente l'identificazione delle celle mononucleate (immagine superiore). Le maschere componenti da 1 a 3 consentono l'identificazione delle celle trinucleate (immagine media). Le maschere componenti da 1 a 4 consentono l'identificazione delle celle quadracle (immagine inferiore). Questa cifra è stata modificata da Rodrigues 201832. Fare clic qui per visualizzare una versione più grande di questa figura.
Figura 5 : Quantificazione della citotossicità. Citotossicità quantificata utilizzando l'indice di proliferazione del blocco di citocinesi (cerchi neri) e genotossicità quantificata utilizzando la percentuale di MN (barre chiare) in seguito a un'esposizione di 3 h e 24 h di recupero per (A) Colchicina, (B) Mitomycin C e ( C) Mannitol. Gli aumenti statisticamente significativi della frequenza di MN rispetto ai controlli sono indicati da stelle (test chi quadrato;s< 0,05,s< 0,01 USD,p < 0,001 USD< 0,001). Tutte le quantità sono la media di due repliche ad ogni punto di dose. Questa cifra è stata modificata da Rodrigues 201832. Fare clic qui per visualizzare una versione più grande di questa figura.
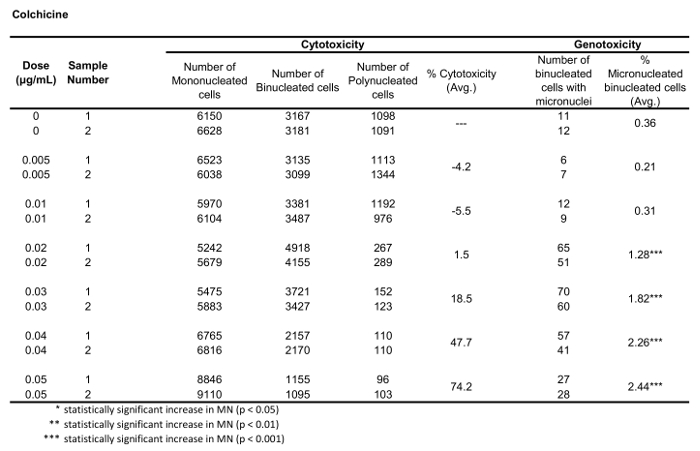
Tabella 1: I parametri necessari per calcolare la citotossicità (il numero di cellule mono, biepolle e polincleate) e la genotossicità (il numero e la percentuale di cellule binucleate micronucleate) per la colichina. Tutte le quantità calcolate sono la media di due repliche ad ogni punto di dose.
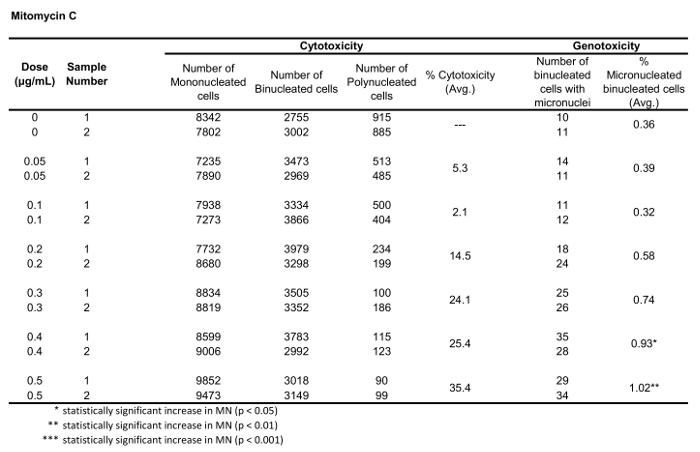
Tabella 2: Tegli parametri necessari per calcolare la citotossicità (il numero di cellule mono-, bi- e polinucleate) e la genotossicità (il numero e la percentuale di cellule binucleated micronucleate) per Mitomycin C. Tutte le quantità calcolate sono la media di due repliche ad ogni punto di dose.
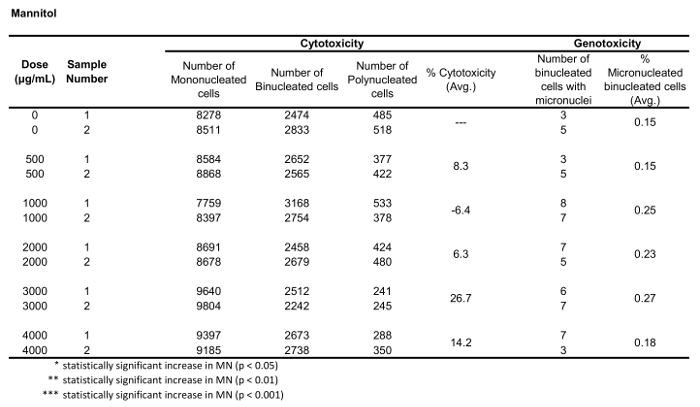
Tabella 3: I parametri necessari per calcolare la citotossicità (il numero di cellule mono, bi e polnucleate) e la genotossicità (il numero e la percentuale di cellule binucleate micronucleate) per Mannitol. Tutte le quantità calcolate sono la media di due repliche ad ogni punto di dose.
Supplemento 1: Protocollo completo. Fare clic qui per scaricare questo file.
Supplemento 2: Elenco maschere. Fare clic qui per scaricare questo file.
Discussione
In una recente pubblicazione Verma e altri hanno sottolineato l'importanza di sviluppare un sistema che combini il vantaggio ad alta produttività della citometria di flusso con i vantaggi di archiviazione dei dati e delle immagini dell'analisi delle immagini35. Il MIFC in vitro MN assay descritto in questo articolo soddisfa questa citazione e ha il potenziale per superare molte delle sfide di cui sopra nei metodi di microscopia e citometria di flusso. Il protocollo qui descritto dimostra che sia la citotossicità che la genotossicità possono essere valutate utilizzando MIFC. La preparazione dei campioni, la colorazione cellulare e la raccolta dei dati sono semplici, ma ci sono alcuni passaggi critici nel protocollo che devono sempre essere implementati. L'aggiunta di cloruro di potassio (KCl) alle cellule è fondamentale per gonfiare le cellule, generando la separazione tra i nuclei principali. Ciò garantisce che l'algoritmo di mascheramento possa identificare tutti i singoli nuclei nei BNM e nelle cellule POLY (cellule POLY) che è necessario per la loro enumerazione. Inoltre, KCL fornisce la separazione tra nuclei e MN, che è essenziale per mascheramento e quantificazione MN accurati. Inoltre, l'uso della formalina dopo l'aggiunta di KCl impedisce alle cellule di lising durante la centrifugazione. L'aggiunta di Cytochalasin B fa sì che le cellule TK6 che hanno subito più di una divisione nucleare siano piuttosto grandi. Di conseguenza, il citoplasma diventa fragile e può lisi se la centrifugazione viene eseguita immediatamente dopo l'aggiunta di KCl. Inoltre, è molto importante introdurre Hoechst nel campione in base al numero di cellule nel campione e non in base a una concentrazione finale. Ad esempio, una concentrazione finale di 10 g/mL di Hoechst macchia uniformemente un campione di 1 x 106 cellule, ma potrebbe non macchiare adeguatamente un campione contenente 5 x 106 cellule e può provocare molte cellule con nuclei macchiati devasamente, rendendo difficile l'analisi. È inoltre importante notare che Hoechst può essere sostituito con un altro tintire di DNA come DAPI se il MIFC è dotato del laser di eccitazione 405 nm o DRAQ5 se il MIFC è dotato del 488 nm e/o 642nm excitation laser(s). Se si modifica la macchia nucleare, è fondamentale titrate la macchia al fine di trovare la concentrazione appropriata per la potenza laser richiesta / desiderata.
Quando si raccolgono dati nel MIFC è importante determinare i limiti di area ottimali per le funzionalità RMS di gradiente. I confini presentati in questo protocollo possono richiedere un aggiustamento a causa di alcune lievi variazioni tra gli strumenti MIFC. L'applicazione di questa funzionalità durante la raccolta dei dati è essenziale per garantire l'acquisizione di immagini altamente focalizzate. Se i file di dati contengono molte immagini sfocate o non sfocate, è probabile che gli algoritmi di mascheramento nel software di analisi evidenzieranno erroneamente gli artefatti di colorazione nelle aree sfocate, portando a un numero elevato di falsi artefatti positivi che vengono segnati come MN. Anche se le tecniche di elaborazione delle immagini qui descritte possono essere difficili, una volta che un modello di analisi è stato sviluppato nel software MIFC, l'elaborazione batch consente di analizzare automaticamente i file di dati, eliminando l'intervento dell'utente e quindi il marcatore pregiudizio. Inoltre, se una linea cellulare diversa dalle celle TK6 viene utilizzata per eseguire l'esecuzione dell'esecuzione, sarà necessario modificare le maschere e i limiti delle aree in quanto le proprietà morfologiche (ad esempio, le dimensioni) delle celle saranno diverse da quelle delle cellule TK6.
I risultati qui presentati (Figura 5) mostrano aumenti statisticamente significativi nell'induzione di MN quando si espongono le cellule TK6 a varie dosi di Mitomycin C e Colchicine. Sono stati osservati aumenti statisticamente significativi della frequenza di MN rispetto ai controlli dei solventi per diverse dosi in entrambe le sostanze chimiche. Inoltre, nessuna dose di Mannitol ha indotto una citotossicità superiore al 30%, né un aumento statisticamente significativo della frequenza di MN rispetto ai controlli dei solventi, come previsto. Il protocollo descritto in questo documento utilizzando MIFC per eseguire il test in vitro MN fornisce i risultati attesi da sostanze chimiche di controllo positive e negative. È molto importante eseguire una serie di esperimenti utilizzando sia i controlli dei solventi che le sostanze chimiche di controllo negativo per sviluppare i valori di base sia della frequenza di MN che dell'indice di proliferazione del blocco di Citocinesi (CBPI). Per la genotossicità, aumenti statisticamente significativi della frequenza MN sono determinati attraverso il confronto con le frequenze MN di base che devono essere ben note per il tipo di cellula utilizzato. Inoltre, tutti i calcoli della citotossicità si basano sulla CBPI dei campioni di controllo e, pertanto, i tassi di base delle cellule MONO, BNC e POLY devono essere ben quantificati nei controlli.
Diverse limitazioni e vantaggi dell'utilizzo di MIFC nel contesto del MN assay sono stati descritti nel lavoro precedente29,32. Le principali limitazioni riguardano frequenze MN più basse rispetto alla microscopia, che probabilmente deriva sia dalla mancanza di flessibilità nell'implementazione dei criteri di punteggio nel software di analisi, sia dalla limitata profondità di campo del MIFC. Le maschere ben sagomate possono essere create per identificare con precisione i nuclei principali, ma MN che stanno toccando (o molto vicino a) i nuclei principali potrebbero essere catturati all'interno della maschera BNC. Inoltre, MN molto piccolo che può essere ottenuto piuttosto facilmente utilizzando la microscopia sono probabilmente erroneamente mancati quando si utilizza MIFC a causa del limite inferiore sul parametro di area della maschera MN per evitare di segnare piccoli artefatti. Oltre alle difficoltà presenti nell'analisi dei dati basata su immagini, grazie al suo design, MIFC ottiene immagini di proiezione bidimensionale di oggetti cellulari tridimensionali. Questo probabilmente fa sì che alcuni MN per essere catturato ad una diversa profondità di messa a fuoco che i due MN principale, facendoli apparire molto fiodo e non-scorable utilizzando mascheramento. Inoltre, una piccola frazione di MN potrebbe risiedere dietro uno dei due nuclei principali, rendendoli impossibili da visualizzare e segnare. Pertanto, considerando queste difficoltà, è necessario prestare attenzione quando si interpretano aumenti significativi della frequenza di MN a basse dosi.
Nonostante queste carenze, il metodo MIFC descritto qui offre diversi vantaggi rispetto ad altre tecniche. Fenech et al. ha proposto criteri e linee guida che dovrebbero essere presi in considerazione quando si sviluppano sistemi e metodologie automatizzate per i test MN36. Questi includono, ma non sono limitati a, la visualizzazione diretta dei principali nuclei e del citoplasma, la determinazione della frequenza di MN da varie dosi della sostanza chimica o dell'agente in fase di test e la capacità di quantificare la morfologia e determinare la posizione di tutti i nuclei e MN per assicurarsi che siano all'interno del citoplasma. Questo documento mostra che il metodo MIFC sviluppato per eseguire il saggio in vitro MN soddisfa (o possiede il potenziale per soddisfare) questi criteri. In particolare, le immagini dei nuclei e MN possono essere catturate dai laser fluorescenti, mentre le immagini citoplasmiche possono essere ottenute utilizzando il LED BF. Le immagini di cellule con morfologia nucleare normale possono essere differenziate automaticamente da quelle cellule con morfologia irregolare utilizzando una combinazione di maschere avanzate e caratteristiche. I risultati presentati per Colchicine e Mitomycin C (Figura 5) mostrano che sia la genotossicità che la citotossicità possono essere valutate a varie dosi rispetto ai controlli dei solventi e che si osservano frequenze di MN statisticamente significative dove Previsto. Inoltre, la linea 487 della Guida al test dell'OCSE raccomanda di segnare 2.000 BNC per concentrazione di test per valutare la presenza di MN per determinare la genotossicità insieme ad almeno 500 cellule per concentrazione di prova per determinare la citotossicità9; questo può richiedere più di 1 h utilizzando la microscopia manuale. Il protocollo e i risultati in questo documento mostrano che una media di circa 6.000 BNC, 16.000 cellule MONO e 800 cellule POLY sono state catturate e valutate per concentrazione di test in circa 20 min. Il rapido tasso di acquisizione dei dati e l'elevato numero di cellule candidate segnate in così poco tempo evidenziano un altro importante vantaggio dell'impiego di MIFC per eseguire il test in vitro MN.
Mentre i risultati presentati in questo documento sono incoraggianti, sono rappresentativi di un metodo di prova precoce del concetto. Questo lavoro dovrebbe essere seguito da un'indagine più approfondita di un insieme chimico più ampio e diversificato che copre più classi e meccanismi di genotossicità e citotossicità come quelli suggeriti da Kirkland et al.37 Condurre tali studi sono tuttavia, questi studi su larga scala forniranno preziose informazioni sulla capacità del metodo di identificare in modo affidabile agenti debolmente mutotossici. La metodologia qui presentata non è ancora stata miniaturizzata in un formato di microwell, il che consentirebbe uno screening più rapido ed efficiente su una gamma di dosi più ampia. Come tale, nella sua forma attuale, il saggio in vitro MN basato su MIFC qui presentato può essere più adatto per studi di follow-up ad alta intensità di lavoro o la ricerca su buone pratiche di laboratorio. Tuttavia, il metodo continuerà ad essere ottimizzato e convalidato, e possiede il potenziale per consentire una maggiore flessibilità nel rilevamento di eventi chimici specifici legati alla morfologia, come l'esposizione all'aneugeno che aumenta la proporzione di cellule con nuclei non circolari che sono ancora commisti biliabili38. Infine, il metodo MIFC offre l'opportunità di introdurre ulteriori biomarcatori nel test MN (ad esempio colorazione del cinetochore) per fornire una visione più completa del meccanismo di induzione di MN.
Divulgazioni
L'autore è impiegato da Luminex Corporation, il creatore del cytometro di flusso di imaging multispettrale ImageStream che è stato utilizzato in questo lavoro.
Riconoscimenti
L'autrice ringrazia Christine Probst (Luminex Corporation) per i suoi sforzi nello sviluppo di forme precedenti del modello di analisi dei dati, nonché la dott.ssa Haley Pugsley (Luminex Corporation) e la dott.ssa Phil Morrissey (Luminex Corporation) per la revisione e la manoscritto.
Materiali
| Name | Company | Catalog Number | Comments |
| 15 mL centrifuge tube | Falcon | 352096 | |
| Cleanser - Coulter Clenz | Beckman Coulter | 8546931 | Fill container with 200 mL of Cleanser. https://www.beckmancoulter.com/wsrportal/page/itemDetails?itemNumber=8546931#2/10//0/25/1/0/asc/2/8546931///0/1//0/ |
| Colchicine | MilliporeSigma | 64-86-8 | |
| Corning bottle-top vacuum filter | MilliporeSigma | CLS430769 | 0.22 um filter, 500 mL bottle |
| Cytochalasin B | MilliporeSigma | 14930-96-2 | 5 mg bottle |
| Debubbler - 70% Isopropanol | EMD Millipore | 1.3704 | Fill container with 200 mL of Debubbler. http://www.emdmillipore.com/US/en/product/2-Propanol-70%25-%28V%2FV%29-0.1-%C2%B5m-filtred,MDA_CHEM-137040?ReferrerURL=https%3A%2F%2Fwww.google.com%2F |
| Dimethyl Sulfoxide (DMSO) | MilliporeSigma | 67-68-5 | |
| Dulbecco's Phosphate Buffered Saline 1X | EMD Millipore | BSS-1006-B | PBS Ca++MG++ Free |
| Fetal Bovine Serum | HyClone | SH30071.03 | |
| Formaldehyde, 10%, methanol free, Ultra Pure | Polysciences, Inc. | 04018 | This is what is used for the 4% and 1% Formalin. CAUTION: Formalin/Formaldehyde toxic by inhalation and if swallowed. Irritating to the eyes, respiratory systems and skin. May cause sensitization by inhalation or skin contact. Risk of serious damage to eyes. Potential cancer hazard. http://www.polysciences.com/default/catalog-products/life-sciences/histology-microscopy/fixatives/formaldehydes/formaldehyde-10-methanol-free-pure/ |
| Hoechst 33342 | Thermo Fisher | H3570 | 10 mg/mL solution |
| Mannitol | MilliporeSigma | 69-65-8 | |
| MEM Non-Essential Amino Acids 100X | HyClone | SH30238.01 | |
| MIFC - ImageStreamX Mark II | EMD Millipore | 100220 | A 2 camera ImageStreamX Mark II eqiped with the 405nm, 488nm, and 642nm lasers was used. http://www.emdmillipore.com/US/en/life-science-research/cell-analysis/amnis-imaging-flow-cytometers/imagestreamx-Mark-ii-imaging-flow-cytometer/VaSb.qB.QokAAAFLzRop.zHe,nav?cid=BI-XX-BDS-P-GOOG-FLOW-B325-0006 |
| MIFC analysis software - IDEAS | EMD Millipore | 100220 | The companion software to the MIFC (ImageStreamX MKII) |
| MIFC software - INSPIRE | EMD Millipore | 100220 | This is the software that runs the MIFC (ImageStreamX MKII) |
| Mitomycin C | MilliporeSigma | 50-07-7 | |
| NEAA Mixture 100X | Lonza BioWhittaker | 13-114E | |
| Penicllin/Streptomycin/Glutamine solution 100X | Gibco | 15070063 | |
| Potassium Chloride (KCl) | MilliporeSigma | P9541 | |
| Rinse - Ultrapure water or deionized water | NA | NA | You can use any ultrapure water or deionized water. Fill container with 900 mL of Rinse. |
| RNase | MilliporeSigma | 9001-99-4 | |
| RPMI-1640 Medium 1X | HyClone | SH30027.01 | |
| Sheath - PBS | EMD Millipore | BSS-1006-B | This is the same as Dulbecco's Phosphate Buffered Saline 1X Ca++MG++ free. Fill container with 900mL of Sheath. |
| Sterile water | HyClone | SH30529.01 | |
| Sterilizer - 0.4-0.7% Hypochlorite | VWR | JT9416-1 | This is assentually 10% Clorox bleach that can be made by deluting Clorox bleach with water. Fill container with 200 mL of Sterilzer. |
| System Calibration Reagent - SpeedBead | EMD Millipore | 400041 | Each tube holds ~10 mL. https://www.emdmillipore.com/US/en/life-science-research/cell-analysis/amnis-imaging-flow-cytometers/support-training/XDqb.qB.wQMAAAFLBDUp.zHu,nav |
| T25 flask | Falcon | 353109 | |
| T75 flask | Falcon | 353136 | |
| TK6 cells | MilliporeSigma | 95111735 |
Riferimenti
- Bonassi, S., et al. An increased micronucleus frequency in peripheral blood lymphocytes predicts the risk of cancer in humans. Carcinogenesis. 28 (3), 625-631 (2007).
- Fenech, M. The in vitro micronucleus technique. Mutation Research - Fundamental and Molecular Mechanisms of Mutagenesis. 455 (1-2), 81-95 (2000).
- Fenech, M. The Lymphocyte Cytokinesis-Block Micronucleus Cytome Assay and its Application in Radiation Biodosimetry. Health Physics. 98 (2), 234-243 (2010).
- Hintzsche, H., et al. Fate of micronuclei and micronucleated cells. Mutation Research - Reviews in Mutation Research. 771, 85-98 (2017).
- Fenech, M. The advantages and disadvantages of the cytokinesis-block micronucleus method. Mutation Research. 392 (1-2), 11-18 (1997).
- Fenech, M. Cytokinesis-block micronucleus cytome assay. Nature Protocols. 2 (5), 1084-1104 (2007).
- Fenech, M., Holland, N., Chang, W. P., Zeiger, E., Bonassi, S. The HUman MicroNucleus Project - An international collaborative study on the use of the micronucleus technique for measuring DNA damage in humans. Mutation Research - Fundamental and Molecular Mechanisms of Mutagenesis. 428 (1-2), 271-283 (1999).
- Kirsch-Volders, M., et al. Report from the in vitro micronucleus assay working group. Mutation Research - Genetic Toxicology and Environmental Mutagenesis. 540 (2), 153-163 (2003).
- OECD Library. Test No. 487: In vitro Mammalian Cell Micronucleus Test. OECD Guidelines for the Testing of Chemicals, Section 4. , (2016).
- Aardema, M. J., et al. SFTG international collaborative study on in vitro micronucleus test. III. Using CHO cells. Mutation Research - Genetic Toxicology and Environmental Mutagenesis. 607 (1), 61-87 (2006).
- Clare, M. G., et al. SFTG international collaborative study on in vitro micronucleus test. II. Using human lymphocytes. Mutation Research - Genetic Toxicology and Environmental Mutagenesis. 607 (1), 37-60 (2006).
- Lorge, E., et al. SFTG international collaborative study on in vitro micronucleus test. I. General conditions and overall conclusions of the study. Mutation Research - Genetic Toxicology and Environmental Mutagenesis. 607 (1), 13-36 (2006).
- Oliver, J., et al. SFTG international collaborative study on in vitro micronucleus test. V. Using L5178Y cells. Mutation Research - Genetic Toxicology and Environmental Mutagenesis. 607 (1), 125-152 (2006).
- Wakata, A., et al. SFTG international collaborative study on in vitro micronucleus test. IV. Using CHL cells. Mutation Research - Genetic Toxicology and Environmental Mutagenesis. 607 (1), 88-124 (2006).
- Fenech, M., et al. Intra- and inter-laboratory variation in the scoring of micronuclei and nucleoplasmic bridges in binucleated human lymphocytes: Results of an international slide-scoring exercise by the HUMN project. Mutation Research - Genetic Toxicology and Environmental Mutagenesis. 534 (1-2), 45-64 (2003).
- Decordier, I., et al. Automated image analysis of cytokinesis-blocked micronuclei: an adapted protocol and a validated scoring procedure for biomonitoring. Mutagenesis. 24 (1), 85-93 (2009).
- Decordier, I., et al. Automated image analysis of micronuclei by IMSTAR for biomonitoring. Mutagenesis. 26 (1), 163-168 (2011).
- Schunck, C., Johannes, T., Varga, D., Lorch, T., Plesch, A. New developments in automated cytogenetic imaging: unattended scoring of dicentric chromosomes, micronuclei, single cell gel electrophoresis, and fluorescence signals. Cytogenetic and Genome Research. 104 (1-4), 383-389 (2004).
- Rossnerova, A., Spatova, M., Schunck, C., Sram, R. J. Automated scoring of lymphocyte micronuclei by the MetaSystems Metafer image cytometry system and its application in studies of human mutagen sensitivity and biodosimetry of genotoxin exposure. Mutagenesis. 26 (1), 169-175 (2011).
- Nüsse, M., Marx, K. Flow cytometric analysis of micronuclei in cell cultures and human lymphocytes: Advantages and disadvantages. Mutation Research - Genetic Toxicology and Environmental Mutagenesis. 392 (1-2), 109-115 (1997).
- Avlasevich, S. L., Bryce, S. M., Cairns, S. E., Dertinger, S. D. In vitro micronucleus scoring by flow cytometry: Differential staining of micronuclei versus apoptotic and necrotic chromatin enhances assay reliability. Environmental and Molecular Mutagenesis. 47 (1), 56-66 (2006).
- Bryce, S. M., Bemis, J. C., Avlasevich, S. L., Dertinger, S. D. In vitro micronucleus assay scored by flow cytometry provides a comprehensive evaluation of cytogenetic damage and cytotoxicity. Mutation Research/Genetic Toxicology and Environmental Mutagenesis. 630 (1-2), 78-91 (2007).
- Bryce, S. M., et al. Flow cytometric 96-well microplate-based in vitro micronucleus assay with human TK6 cells: Protocol optimization and transferability assessment. Environmental and Molecular Mutagenesis. 54 (3), 180-194 (2013).
- Fenech, M. Commentary on the SFTG international collaborative study on the in vitro micronucleus test: to Cyt-B or not to Cyt-B. Mutation Research. 607 (1), 9-12 (2006).
- Basiji, D. A. . Methods in Molecular Biology. 1389, 13-21 (2016).
- Rodrigues, M. A., Beaton-Green, L. A., Kutzner, B. C., Wilkins, R. C. Automated analysis of the cytokinesis-block micronucleus assay for radiation biodosimetry using imaging flow cytometry. Radiation and Environmental Biophysics. 53 (2), 273-282 (2014).
- Rodrigues, M. A., Beaton-Green, L. A., Kutzner, B. C., Wilkins, R. C. Multi-parameter dose estimations in radiation biodosimetry using the automated cytokinesis-block micronucleus assay with imaging flow cytometry. Cytometry Part A. 85 (10), 883-893 (2014).
- Rodrigues, M. A., Beaton-Green, L. A., Wilkins, R. C. Validation of the cytokinesis-block micronucleus assay using imaging flow cytometry for high throughput radiation biodosimetry. Health Physics. 110 (1), 29-36 (2016).
- Rodrigues, M. A., Probst, C. E., Beaton-Green, L. A., Wilkins, R. C. Optimized automated data analysis for the cytokinesis-block micronucleus assay using imaging flow cytometry for high throughput radiation biodosimetry. Cytometry Part A. 89 (7), 653-662 (2016).
- Rodrigues, M. A., Probst, C. E., Beaton-Green, L. A., Wilkins, R. C. The effect of an optimized imaging flow cytometry analysis template on sample throughput in the reduced culture cytokinesis-block micronucleus assay. Radiation Protection Dosimetry. 172 (1-3), 223-229 (2016).
- Wang, Q., et al. Automated Triage Radiation Biodosimetry: Integrating Imaging Flow Cytometry with High-Throughput Robotics to Perform the Cytokinesis-Block Micronucleus Assay. Radiation Research. 191 (4), 342-351 (2019).
- Rodrigues, M. A. Automation Of The In vitro Micronucleus Assay Using The ImageStream® Imaging Flow Cytometer. Cytometry Part A. 93, 706-726 (2018).
- Rodrigues, M. A., Beaton-Green, L. A., Wilkins, R. C., Fenech, M. F. The potential for complete automated scoring of the cytokinesis block micronucleus cytome assay using imaging flow cytometry. Mutation Research/Genetic Toxicology and Environmental Mutagenesis. 836, 53-64 (2018).
- Fenech, M., et al. HUMN project: Detailed description of the scoring criteria for the cytokinesis-block micronucleus assay using isolated human lymphocyte cultures. Mutation Research - Genetic Toxicology and Environmental Mutagenesis. 534 (1-2), 65-75 (2003).
- Verma, J. R., et al. Evaluation of the automated MicroFlow® and Metafer™ platforms for high-throughput micronucleus scoring and dose response analysis in human lymphoblastoid TK6 cells. Archives of Toxicology. 91 (7), 2689-2698 (2017).
- Fenech, M., et al. HUMN project initiative and review of validation, quality control and prospects for further development of automated micronucleus assays using image cytometry systems. International Journal of Hygiene and Environmental Health. 216 (5), 541-552 (2013).
- Kirkland, D., et al. Updated recommended lists of genotoxic and non-genotoxic chemicals for assessment of the performance of new or improved genotoxicity tests. Mutation Research/Genetic Toxicology and Environmental Mutagenesis. 795, 7-30 (2016).
- Verma, J. R., et al. Investigating FlowSight® imaging flow cytometry as a platform to assess chemically induced micronuclei using human lymphoblastoid cells in vitro. Mutagenesis. 33 (4), 283-289 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon