Method Article
Une méthode automatisée pour effectuer l'essai micronutrique in vitro à l'aide de la cytométrie du flux d'imagerie multispectrale
Dans cet article
Résumé
L'analyse micronutrique in vitro est une méthode bien établie pour évaluer la génotoxicité et la cytotoxicité, mais la notation de l'essai à l'aide de la microscopie manuelle est laborieuse et souffre de subjectivité et de variabilité inter-scorer. Cet article décrit le protocole développé pour effectuer une version entièrement automatisée de l'essai utilisant la cytométrie multispectrale de flux d'imagerie.
Résumé
L'analyse du micronoyau in vitro (MN) est souvent utilisée pour évaluer la cytotoxicité et la génotoxicité, mais la notation de l'essai par microscopie manuelle est laborieuse et introduit l'incertitude dans les résultats en raison de la variabilité entre les marqueurs. Pour remédier à cela, la microscopie automatique de balayage de diapositive ainsi que les méthodes conventionnelles de cytométrie de flux ont été introduites dans une tentative d'éliminer le biais de marqueur et d'améliorer le débit. Cependant, ces méthodes ont leurs propres limitations inhérentes telles que l'incapacité de visualiser le cytoplasme de la cellule et l'absence de vérification visuelle MN ou de stockage de données d'image avec cytométrie de flux. La cytométrie multispectrale de flux d'imagerie (MIFC) a le potentiel de surmonter ces limitations. MIFC combine l'imagerie fluorescente à haute résolution de la microscopie avec la robustesse statistique et la vitesse de la cytométrie de débit classique. En outre, toutes les images recueillies peuvent être stockées dans des fichiers spécifiques à la dose. Cet article décrit le protocole développé pour effectuer une version entièrement automatisée de l'analyse MN sur MIFC. Les lymphoblastoïdes humains TK6 cellules ont été agrandies à l'aide d'une solution hypotonique (75 mM KCl), fixé avec 4% de formaline et la teneur nucléaire a été tachée avec Hoechst 33342. Tous les échantillons ont été exécutés en suspension sur le MIFC, permettant l'acquisition d'images haute résolution de tous les événements clés requis pour l'essai (par exemple, les cellules binuclées avec et sans MN ainsi que les cellules mononucléées et polynucléées). Les images ont été automatiquement identifiées, classées et énumérées dans le logiciel d'analyse des données du MIFC, ce qui a permis une notation automatisée de la cytotoxicité et de la génotoxicité. Les résultats démontrent que l'utilisation du MIFC pour effectuer l'analyse In vitro mN permet de détecter des augmentations statistiquement significatives de la fréquence mN à plusieurs niveaux différents de cytotoxicité par rapport aux contrôles des solvants à la suite de l'exposition des cellules TK6 à Mitomycine C et Colchicine, et qu'aucune augmentation significative de la fréquence de MN n'est observée après exposition au Mannitol.
Introduction
L'analyse du micronucyaux in vitro (MN) est un test couramment utilisé pour évaluer la cytotoxicité et la génotoxicité comme outil de dépistage dans plusieurs domaines d'étude tels que le développement chimique et pharmaceutique ainsi que la biosurveillance humaine chez les personnes exposées à divers facteurs environnementaux, professionnels ou de style de vie1,2,3. MN se composent de fragments de chromosomes ou de chromosomes entiers générés au cours de la division cellulaire qui ne sont pas incorporés dans l'un des deux principaux noyaux filles. Après la télophase, ce matériau chromosomique se forme en un corps individuel et arrondi à l'intérieur du cytoplasme qui est séparé de l'un ou l'autre des noyaux principaux2. Par conséquent, MN sont représentatifs des dommages à l'ADN et ont été utilisés pendant de nombreuses années comme un point de terminaison dans les tests de génotoxicité4. La méthode la plus appropriée pour mesurer mN est l'analyse du micronoyau de bloc de cytokinesis (CBMN). À l'aide de l'analyse CBMN, la fréquence du MN dans les cellules binuclées (BNC) peut être notée en incorporant la cytochalacine B (Cyt-B) dans l'échantillon. Cyt-B permet la division nucléaire, mais empêche la division cellulaire et, par conséquent, limite la notation de MN aux BNC qui n'ont divisé qu'une seule fois5.
Les protocoles utilisant à la fois la microscopie et la cytométrie de flux ont été développés et validés et sont couramment utilisés pour effectuer l'essai in vitro MN6,7,8,9,10, 11,12,13,14. La microscopie bénéficie de la façon de confirmer visuellement que les MN sont légitimes, mais qu'elles prennent beaucoup de temps et qu'elles sont sujettes à une variabilité entre les marqueurs15. Pour remédier à cela, des méthodes automatisées de microscopie ont été développées pour numériser et capturer des images de noyaux et MN16,17,18,19, mais le cytoplasme ne peut pas être visualisé, ce qui le rend difficile de déterminer si un MN est réellement associé à une cellule spécifique. En outre, ces méthodes ont des difficultés à identifier les cellules polynucléées (POLY) (y compris les cellules tri et quadranuclées) qui sont nécessaires pour le calcul de la cytotoxicité lors de l'utilisation de Cyt-B9. Les méthodes de cytométrie de flux développées pour effectuer l'essai de MN emploient la fluorescence aussi bien que les intensités de diffusion vers l'avant et latérales pour identifier des populations des noyaux et du MN qui ont été libérées de la cellule suivant la lyse20,21 ,22. Cela permet d'acquérir des données à partir de plusieurs milliers de cellules en quelques minutes et permet une analyse automatisée23; cependant, l'incapacité de visualiser les cellules rend impossible de confirmer que les événements marqués sont authentiques. En outre, lysing la membrane cellulaire inhibe l'utilisation de Cyt-B ainsi que la création d'une suspension qui contient d'autres débris tels que les agrégats chromosomiques ou des corps apoptotiques et il n'y a aucun moyen de différencier ces de MN24.
À la lumière de ces limitations, la cytométrie multispectrale de flux d'imagerie (MIFC) est un système idéal pour effectuer l'essai MN puisqu'il combine l'imagerie fluorescente à haute résolution de la microscopie avec la robustesse statistique et la vitesse de la cytométrie conventionnelle de flux. Dans MIFC, toutes les cellules sont introduites dans un système fluidique et sont ensuite hydrodynamiquement focalisés dans le centre d'une cuvette de cellules d'écoulement. L'éclairage orthogonal de toutes les cellules est accompli par l'utilisation d'une diode électroluminescente (DEL) brightfield (BF), d'un laser de diffusion latérale et (au moins) d'un laser fluorescent. Les photons fluorescents sont capturés par l'un des trois objectifs objectifs d'ouverture numérique (20x, 40x ou 60x) et passent ensuite par un élément de décomposition spectrale. Les photons sont ensuite concentrés sur un appareil couplé à la charge (CCD) pour obtenir des images haute résolution de toutes les cellules qui passent à travers la cellule de flux. Pour éviter le flou ou les stries, le CCD fonctionne en mode d'intégration de retard de temps (TDI) qui suit les objets en transférant le contenu pixel d'une ligne à l'autre en synchronie avec la vitesse de la cellule en flux. Les informations Pixel sont ensuite collectées à partir de la dernière rangée de pixels. L'imagerie TDI combinée à la décomposition spectrale permet de capturer simultanément jusqu'à 12 images (2 BF, 10 fluorescents) à partir de toutes les cellules passant par la cellule d'écoulement. Toutes les images capturées sont stockées dans des fichiers de données spécifiques à l'échantillon, ce qui permet d'effectuer une analyse à tout moment à l'aide du logiciel d'analyse de données MIFC. Enfin, les fichiers de données conservent le lien entre les images cellulaires et les points sur toutes les parcelles bivariées. Cela signifie que n'importe quel point sur une parcelle bivariée traditionnelle peut être mis en évidence et son BF correspondant et l'imagerie fluorescente sera affiché25.
Récemment, des méthodes miFC ont été développées pour effectuer l'analyse MN pour la biodosimétrie de rayonnement de triage26,27,28,29,30,31 et génétique toxicologie32,33 tests. Ce travail a démontré que les images cellulaires des noyaux principaux, MN et le cytoplasme peuvent être photographiés avec un débit plus élevé que d'autres méthodes26. Tous les types de cellules nécessaires à l'analyse, y compris les cellules MONO, les BNC (avec et sans MN) et les cellules POLY, peuvent être automatiquement identifiés dans le logiciel d'analyse des données MIFC, et la mise en œuvre des critères de notation développés par Fenech et al. est réalisée grâce à l'utilisation de divers algorithmes mathématiques6,34. Les résultats de la biodosimétrie ont montré que les courbes d'étalonnage de la réponse à la dose étaient de même que celles obtenues à partir d'autres méthodes automatisées dans la littérature lors de la quantification du taux de MN par BNC29. En outre, des travaux récents en toxicologie ont démontré que les images des cellules MONO, des BNC (avec et sans MN) et des cellules POLY peuvent être automatiquement capturées, identifiées, classifiées et énumérées à l'aide du MIFC. Le protocole et l'analyse des données ont permis le calcul de la cytotoxicité et de la génotoxicité après avoir exposé les cellules TK6 à plusieurs clastogènes et aneugènes32.
Le protocole présenté dans cet article décrit une méthode pour effectuer l'examen in vitro MN à l'aide de MIFC. La technique de traitement de l'échantillon utilisée dans ce travail nécessite moins de 2 h pour traiter un seul échantillon et est relativement facile à exécuter par rapport à d'autres méthodes. L'analyse des données dans le logiciel d'analyse MIFC est compliquée, mais la création du modèle d'analyse peut être réalisée en quelques heures suivant les étapes décrites dans le présent document. En outre, une fois le modèle créé, il peut être automatiquement appliqué à toutes les données collectées sans aucun autre travail. Le protocole décrit toutes les étapes nécessaires pour exposer les cellules TK6 aux clastogènes et aux aneugènes, décrit comment la culture, le traitement et la tache des cellules, et démontre comment acquérir des images à haute résolution à l'aide du MIFC. En outre, cet article illustre les meilleures pratiques actuelles pour analyser les données dans le logiciel MIFC pour identifier et marquer automatiquement les cellules MONO, BNCs, et les cellules poly aux fins de calculer la cytotoxicité et la génotoxicité.
Protocole
1. Préparation du milieu de culture et de la culture des cellules TK6
REMARQUE : Certains produits chimiques utilisés dans ce protocole sont toxiques. L'inhalation, la déglutition ou le contact de la peau avec cytochalasinE B peuvent être mortels. Portez l'équipement de protection individuelle approprié, y compris un manteau de laboratoire et deux paires de gants nitriles. Lavez-vous soigneusement les mains après la manipulation. La formaline/formaldéhyde est toxique s'il est inhalé ou avalé; est irritant pour les yeux, le système respiratoire et la peau; et peut provoquer une sensibilisation par inhalation ou contact cutané. Il y a un risque de dommages graves aux yeux. C'est un cancérogène potentiel.
- Préparer 565 ml de 1x rpMI culture moyenne. Ajouter 5 ml d'acides aminés non essentiels MEM (100x), 5 mL de pyruvate de sodium (100 ml), 5 mL de pénicilline-streptomycine-glutamine (100x), et 50 mL de sérum bovin fœtal (FBS) à une bouteille de 500 ml de 1x RPMI 1640 moyen. Préparer le milieu dans une armoire de biosécurité et le conserver à 2-8 oC. Chauffer le milieu à 37 oC avant de l'ajouter aux cellules TK6 (voir tableau des matériaux).
- Décongeler 1 ml de cellules TK6 (stockées à -80 oC en DMSO) dans 10 ml de milieu. Centrifuger les cellules à 200 x g pendant 8 min et aspirer le supernatant. Transférer les cellules à 50 ml de support et incuberà 37 oC, 5 % DE CO 2. Le temps de doublement des cellules TK6 varie de 12 à 18 h et quelques (3 ou 4) passages seront nécessaires pour que les cellules atteignent leur taux de prolifération maximum (voir tableau des matériaux).
- Culture 100 ml de cellules à une concentration de 7-8 x 105 cellules/ml.
2. Préparation des clastogènes et/ou des aneugènes et de la cytochachiasine B
- Préparer les concentrations de stocks appropriées de clastogènes et d'aneugènes désirés. Par exemple, pour la mitomycine C, dissoudre une bouteille complète de 2 mg dans 10 ml d'eau stérile pour atteindre une concentration finale de 200 g/mL. La mitomycine C peut être stockée à 4 oC pendant trois mois (voir Tableau des matériaux).
- Le jour de l'expérience, préparer des dilutions des produits chimiques désirés qui sont 10 fois plus élevés ou 100 fois plus élevés que les concentrations d'exposition souhaitées si vous diluez dans de l'eau stérile ou du DMSO, respectivement.
- Pour la mitomycine C, préparer des dilutions de 3 ml dans de l'eau stérile de 0,5, 1,0, 2,0, 3,0, 4,0 et 5,0 g/mL. Pour la colchicine, préparer des dilutions de 3 ml dans de l'eau stérile de 0,05, 0,1, 0,2, 0,3, 0,4 et 0,5 g/mL. Enfin, pour Mannitol, préparer des dilutions de 3 ml dans de l'eau stérile de 5, 10, 20, 30, 40 et 50 mg/ll.
- Préparer une concentration de 200 g/mL de Cytochalasin B en dissolvant une bouteille de 5 mg en 25 ml de DMSO. La cytochalacine B peut être stockée à -20 oC pendant plusieurs mois.
3. Exposition des cellules aux clastogènes et/ou aux aneugènes
- Ajouter 1 ml de produit chimique désiré (p. ex. Mitomycine C) à 9 ml de cellules à 7-8x105 cellules/mL dans un flacon T25. Pour les échantillons témoins, ajouter 1 ml d'eau stérile. Placer les flacons dans un incubateur de CO2 de 37 oC et 5 % pendant 3 h.
REMARQUE : Si les produits chimiques sont dilués dans le DMSO, ajoutez seulement 100 L de ce produit chimique à chaque flacon et ajoutez 100 L de DMSO aux contrôles. Chaque flacon doit contenir 9.900 ml de cellules. - Après 3 h, retirer les flacons de l'incubateur et transférer les cellules dans des tubes de polypropylène de 15 ml. Centrifugeuse à 200 x g pendant 8 min, aspirez le supernatant et transférez les cellules vers de nouveaux flacons T25 contenant un total de 10 ml de milieu de culture frais. Ajouter 150 oL de la concentration de stock (200 g/mL) de Cytochalasin B à chaque flacon pour atteindre une concentration finale de 3 g/mL.
- Remettre les flacons à l'incubateur de CO2 de 37 oC et 5 % pour un temps de récupération égal à 1,5 à 2,0 fois, comme le recommandent les lignes directrices de l'OCDE9. Pour les cellules TK6 utilisées dans ce travail, le temps de récupération était de 24 h.
REMARQUE : Le temps de doublement des cellules TK6 utilisées ici était de 15 h et un temps de récupération de 24 h (1,6 temps de doublement) a été utilisé. Les temps de récupération inférieurs à 1,5 fois doublerréront la prolifération des échantillons exposés à des doses plus élevées ayant une incidence sur le nombre de BNC. Inversement, les temps de récupération de plus de 2,0 produiront un nombre disproportionné de cellules polynucléées dans les échantillons témoins, calcul de cytotoxicité.
4. Préparation de tampons pour la fixation et l'étiquetage du contenu de l'ADN (voir Tableau des matériaux)
- Préparer 75 mM de chlorure de potassium (KCl) en ajoutant 2,79 g à 500 ml d'eau ultrapure. Remuer la solution pendant 5 min à l'aide d'un agitateur magnétique et d'un filtre stérile à l'aide d'un filtre de 200 m. La solution de 75 mM KCl peut être stockée à 4 oC pendant plusieurs mois.
- Préparer une quantité suffisante de 4% de formaline pour l'expérience, en anticipant qu'un total de 2,1 ml doit être ajouté à chaque échantillon. Par exemple, pour préparer 10 ml de formaline de 4 %, ajouter 4 ml de stock de formaline à 10 % à 6 ml de la solution Saline tamponnée par phosphate de 1x Dulbecco sans Ca2 ou Mg2MD (PBS). Cette formaline de 4% peut être stockée à température ambiante pendant plusieurs semaines.
- Préparer 510 ml de tampon de lavage (2 % de FBS en 1X PBS) en ajoutant 10 ml de FBS à une bouteille de 500 ml de 1x PBS.
- Préparer 10 ml d'une concentration de Hoechst 33342 de 100 ml en ajoutant 100 l de la concentration de stock (1 mg/mL) à 9 900 oL de 1X PBS. La solution Hoechst 33342 peut être stockée à 4 oC pendant plusieurs mois.
5. Traitement d'échantillon : gonflement hypotonique, fixation, comptage cellulaire et contenu d'étiquetage d'ADN
- À la fin de la période de récupération, retirer tous les flacons de l'incubateur et transférer tous les échantillons dans des tubes de polypropylène de 15 ml. Centrifugetous les échantillons à 200 x g pendant 8 min.
- Aspirer le supernatant, resuspendre les cellules et ajouter 5 ml de 75 ml de KCl. Mélanger délicatement par inversion trois fois et incuber à 4 oC pendant 7 min.
- Ajouter 2 ml de formaline de 4 % à chaque échantillon, mélanger délicatement par inversion trois fois et incuber à 4 oC pendant 10 min. Cette étape agit comme une « fixation douce ».
- Centrifuger tous les échantillons à 200 x g pendant 8 min. Aspirer le supernatant et resuspendre dans 100 L de 4% de formaline pendant 20 min. Cette étape agit comme une « fixation difficile ».
- Ajouter 5 ml de tampon de lavage et centrifugeuse à 200 x g pendant 8 min. Aspirer le supernatant et resuspendre dans 100 l l de tampon de lavage.
- Transférer tous les échantillons dans des tubes microcentrifuges de 1,5 ml.
- Effectuer un compte cellulaire sur chaque échantillon pour déterminer le nombre de cellules par échantillon. Les échantillons seront très concentrés, de sorte qu'une dilution de 1:100 dans 1x PBS (10 l d'échantillon dans 990 L de PBS) sera probablement nécessaire pour obtenir un décompte précis.
REMARQUE : À ce stade, il est préférable d'effectuer des comptes cellulaires à l'aide d'un hémocytomètre. L'ajout de KCl donne au cytoplasme un aspect translucide, ce qui rend difficile pour les compteurs cellulaires automatisés de les reconnaître. En outre, les compteurs automatisés ont de la difficulté à marquer les cellules polynucléées en raison de leur taille. - S'ils ne sont pas analysés immédiatement sur le MIFC, ils peuvent être conservés à 4 oC pendant plusieurs jours. Lorsque vous êtes prêt à exécuter des échantillons, ajouter 5 'L's de 100 'g/mL par 1x106 cellules/mL à chaque échantillon. Ajoutez également 10 oL de 500 g/mL de RNase par 100 l d'échantillon pour une concentration finale de 50 g/mL. Incuber les échantillons à 37 oC, 5 % de CO2 pendant 30 min.
- Micro centrifuge tous les échantillons à 200 x g pendant 8 min et utiliser une pipette pour enlever le supernatant laissant 30 l. Utilisez une pipette pour resuspendre tous les échantillons avant de s'exécuter sur le MIFC en s'assurant qu'il n'y a pas de bulles dans le tube. Ne pas vortex.
6. Démarrage et étalonnage du MIFC
- Assurez-vous que les contenants de gaine, de réamorçant du système, de débulleur, nettoyant et de stérilisateur sont pleins et que le réservoir de déchets est vide. Alimentez le système et double-cliquez sur l'icône logicielle MIFC. Cliquez sur le bouton Startup et assurez-vous que tous les étalonnages et tests Démarrer sont cochés. Ceci va rincer le système, charger la gaine et les réactifs d'étalonnage du système, et calibrer le système (voir tableau des matériaux).
7. Exécuter des échantillons sur le MIFC
REMARQUE: Cette section suppose l'utilisation d'un 2 caméra MIFC. Si vous utilisez un MIFC 1 caméra, veuillez consulter le Supplément 1 - Protocole complet, section 7 pour la création de parcelles lors de l'acquisition
- Lancer le logiciel d'acquisition de données MIFC (voir Tableau des matériaux). La figure 1 montre les paramètres de l'instrument. Allumez le laser de 405 nm et fixez la puissance du laser à 10 mW (A). Désactiver tous les autres lasers (y compris SSC) et définir le BF aux canaux 1 et 9 (B). Confirmez que le curseur de grossissement est réglé à 60x (C), mode haute sensibilité est sélectionné (D), et que seuls les canaux 1, 7 et 9 sont montrant dans la galerie d'images.
- Cliquez sur l'icône Scatterplot. Sélectionnez la population all et sélectionnez la zone M01 sur l'axe X et le ratio aspect m01 sur l'axe Y. Cliquez sur l'icône De la Région Carrée et dessinez une région autour des cellules individuelles. Nommez cette région Cellules uniques. Cliquez à droite sur l'intrigue et sélectionnez Régions. Mettez en évidence la région des cellules individuelles et modifiez les x-coordonnées à 100 et 900 et modifiez les coordonnées y à 0,75 et 1 (Figure 1I).
- Cliquez sur l'icône Scatterplot. Sélectionnez Cellules uniques comme population mère, sélectionnez Gradient RMS M01 sur l'axe X et Gradient RMS M07 sur l'axe Y. Cliquez sur l'icône De la Région Carrée et dessinez une région autour de la majorité des cellules. Nommez cette région Cellules ciblées. Cliquez à droite sur l'intrigue et sélectionnez Régions. Mettre en évidence la région des cellules focales et modifier les coordonnées x à 55 et 75 et changer les coordonnées y à 9,5 et 20 (figure 1J).
- Cliquez sur l'icône Histogram. Sélectionnez la population de cellules focales et sélectionnez Intensité M07 comme fonctionnalité. Cliquez sur l'icône Région linéaire et dessinez une région à travers le pic principal de l'histogramme. Nommez cette région POSITIVE à l'ADN. Cliquez à droite sur l'intrigue et sélectionnez Régions. Mettez en évidence la région positive à l'ADN et changez les coordonnées en 2 x 105 et 2 x 106. La plage peut devoir être ajustée en fonction du pic d'intensité de l'histogramme (figure 1K).
- Définir les paramètres d'acquisition (Figure 1E). Spécifiez le nom du fichier et le dossier de destination, modifiez le nombre d'événements à 20 000 et sélectionnez la population positive à l'ADN.
- Cliquez sur Charge (Figure 1F) et placez l'échantillon de contrôle dans le MIFC. Cliquez sur le bouton Acquérir pour collecter les données (Figure 1G). Une fois l'acquisition terminée, cliquez sur le bouton Retour pour retourner l'échantillon (Figure 1H). Retirez le tube de l'échantillon de l'instrument. Répétez ce processus pour tous les échantillons restants de l'expérience.
8. Ouverture d'un fichier de données dans IDEAS
- Lancement du logiciel d'analyse MIFC (voir tableau des matériaux). Cliquez sur L'analyse de démarrage pour démarrer l'assistant de fichier ouvert. Sélectionnez un fichier de données en naviguant vers le fichier d'image brute souhaité (.rif). Cliquez sur le bouton Ouvrez et cliquez sur Suivant.
- Puisqu'il s'agit d'un seul jeu de couleur, la compensation n'est pas nécessaire alors cliquez ensuite pour contourner l'étape de compensation. À ce stade, il n'y a pas de modèle d'analyse à appliquer, alors cliquez à nouveau sur Suivant. Si le modèle d'analyse a été téléchargé à partir du matériel supplémentaire, sélectionnez-le maintenant. Ces modèles ne fonctionnent qu'avec un MIFC 2 caméras avec BF réglé sur les canaux 1 et 9 et l'imagerie nucléaire dans le canal 7 lors de l'acquisition.
- Par défaut, les noms de fichiers .cif et .daf sont automatiquement générés pour correspondre au .rif. Il n'est pas recommandé de changer les noms de la .cif et .daf. Cliquez ensuite. Définir les propriétés d'affichage d'image en sélectionnant les 01 et 07. Cliquez ensuite. Il n'y a pas d'assistant pour cette application, alors cliquez sur Finish. Il est très important d'enregistrer le fichier d'analyse de données (.daf) et le modèle d'analyse (.ast) souvent pendant les sections 9 à 14 pour éviter la perte de progrès.
9. Création de masques et de fonctionnalités pour identifier les BNC
- Cliquez sur l'icône Propriétés de la galerie d'images (icône bleue/blanche). Dans l'onglet Propriétés d'affichage, cliquez sur Définir la plage de données Pixel, puis changez la couleur en jaune. Cliquez OK. Les images Hoechst sont maintenant plus faciles à voir sur le fond noir.
- Créez la parcelle de cellules non apoptotiques.
- Cliquez sur l'onglet Analyse, puis cliquez sur Masques. Cliquez sur Nouveau puis cliquez sur Fonction. Sous Fonction choisissez Seuil, sous Masque choisissez M07 et définir le pourcentage d'intensité à 50. Cliquez OK puis OK à nouveau. Cliquez sur Fermer.
- Cliquez sur l'onglet Analyse, cliquez sur Caractéristiques, puis cliquez sur Nouveau. Pour la zonede sélection du type de fonctionnalité . Pour Masque sélectionnez le Seuil (M07,Ch07,50). Cliquez sur Définir le nom par défaut et cliquez sur OK. Cliquez sur Fermer pour commencer à calculer les valeurs de fonctionnalité.
- Cliquez sur l'icône Dot Plot. Sélectionnez la population All. Pour la fonction X-axe, choisissez la fonction Contraste M01-Ch01 et pour la fonction Axe Y, choisissez Zone-Seuil (M07,Ch07,50). Cliquez OK.
- Cliquez sur le bouton Région carrée et dessinez une région autour de la majorité des cellules. Appelez cette région Non-apoptotic. Cliquez à droite sur l'intrigue et cliquez sur Régions. Mettez en valeur la région non apoptotique. Définir les coordonnées x à 0 et 15 et définir les coordonnées y à 50 et 300. Cliquez sur Fermer.
- Créez le masque BNC (étapes 9.3.1-9.3.5) pour identifier les cellules qui ne contiennent que deux noyaux.
- Parcourez un BNC dans la galerie d'images et cliquez dessus. Il s'agit de visualiser la création du masque dans le canal Hoechst.
- Cliquez sur l'onglet Analyse, puis cliquez sur Masques. Cliquez sur Nouveau puis cliquez sur Fonction. Sous Fonction choisissez LevelSet, sous Masque choisissez M07, sélectionnez le bouton de radio De niveau moyen de masque, et fixez l'échelle de détail de contour à 3.00. Cliquez OK puis OK à nouveau.
- Cliquez sur Nouveau puis cliquez sur Fonction. Sous fonction, choisissez Dilate, et sous Masque choisir LevelSet(M07,Ch07,Middle,3). Définir l'image pour afficher à Ch07, et définir le nombre de pixels à 2. Cliquez OK puis OK à nouveau.
- Cliquez sur Nouveau puis cliquez sur Fonction. Sous fonction choisissez Watershed, et sous Masque choisir Dilate (LevelSet(M07,Ch07,Middle,3)2). Définir l'image pour afficher à Ch07, et définir l'épaisseur de la ligne à 1. Cliquez OK puis OK à nouveau.
- Cliquez sur Nouveau puis cliquez sur Fonction. Sous Fonction choisissez Range, sous Masque choisissez Watershed(Dilate(LevelSet(M07,Ch07,Middle,3)2)). Définir l'image pour afficher à Ch07. Fixez les valeurs minimales et maximales de la zone à 115 et 5000, respectivement. Définir les valeurs de ratio d'aspect minimum et maximum à 0,4 et 1, respectivement. Cliquez sur OK. Dans le champ Nom changer le texte pour lire BNC puis cliquez sur OK.
- Créer les caractéristiques et les parcelles pour obtenir la population finale bnC
- Spot Count BNC fonctionnalité: Cliquez sur l'onglet Analyse, puis Caractéristiques, puis Nouveau. Pour le type de fonctionnalité sélectionnez Spot Count. Pour Masque, sélectionnez le masque FINAL BNC créé en 9.3.5. Définir la connectivité à quatre et changer le nom de Spot Count BNC. Cliquez OK puis Fermez pour calculer les valeurs de fonctionnalité.
- Spot Count BNC histogramme. Cliquez sur l'icône Histogram. Sélectionnez Non-apoptotic comme la population parente. Pour la fonction X-axis, choisissez la fonction Spot Count BNC. Cliquez OK. Cliquez sur l'icône Région linéaire. Dessiner une région à travers le bac 2. Appelez cette région 2N.
REMARQUE : Reportez-vous à la section 9 du Supplément 1 - Protocole complet pour créer les masques, les caractéristiques et les parcelles restants afin d'identifier la population finale du BNC
10. Création de masques et de fonctionnalités pour identifier les MN au sein de la population BNC
- Créez le masque MN. Parcourez pour un BNC qui contient un MN dans la galerie d'images et cliquez dessus. Il s'agit de visualiser la création du masque MN dans le canal Hoechst. Cliquez sur l'onglet Analyse, puis cliquez sur Masques.
- Créer le masque d'identification spot 1 :
- Cliquez sur Nouveau puis cliquez sur Fonction. Under Function choisir Spot et s'assurer que le bouton radio Bright est sélectionné. Sous Masque choisissez M07, définir le rapport spot to Cell Background à 2.00. Définir le rayon minimum à 2 et le rayon maximum à 6. Cliquez OK puis OK à nouveau.
- Cliquez sur Nouveau puis cliquez sur Fonction. Sous fonction choisissez gamme, et sous masque choisir LevelSet(M07,Ch07,Middle,3). Définir l'image pour afficher à Ch07. Définir la zone minimale et maximale à 80 et 5000, respectivement. Définir le ratio d'aspect minimum et maximum à 0 et 1, respectivement. Cliquez OK puis OK à nouveau.
- Cliquez sur Nouveau puis cliquez sur Fonction. Sous Fonction choisissez Dilate, sous Masque choisissez Range(LevelSet(M07,Ch07,Middle,3),80-5000,0-1). Définir l'image pour afficher à Ch07. Définir le nombre de pixels à 2. Cliquez OK puis OK à nouveau.
- Cliquez sur Nouveau. Double cliquez sur le masque Spot(M07, Ch07, Bright, 2, 6, 2) pour l'ajouter à la définition du masque. Cliquez sur l'opérateur Et, puis l'opérateur Not. Double cliquez sur le masque Dilate(Range(LevelSet(M07, Ch07, Middle, 3), 80-5000, 0-1), 2) pour l'ajouter à la définition du masque. Cliquez OK.
- Cliquez sur Nouveau, puis Fonction. Sous Fonction choisissez Range et sous masque choisissez le masque créé en 10.1.1.4:
- Sélectionnez Spot(M07, Ch07, Bright, 2, 6, 2) Et non Dilate(Range(LevelSet(M07, Ch07, Middle, 3), 80-5000, 0-1), 2).
- Définir l'image à afficher sur Ch07. Définir la zone minimale et maximale à 10 et 80, respectivement. Définir le ratio d'aspect minimum et maximum à 0,4 et 1, respectivement. Cliquez OK puis OK à nouveau. Le masque d'identification spot 1 est complet.
REMARQUE : Se référer à la section 10 du Supplément 1 - Protocole complet pour créer les masques, les caractéristiques et les parcelles afin d'identifier la population finale du MN
- Créer le masque d'identification spot 1 :
11. Créer des masques, des caractéristiques et des parcelles pour identifier les populations mononuclites et polynuclées
- Créez le masque POLY. Cliquez sur Analyse, puis Masques, puis Nouvelle puis Fonction. Sous Fonction choisissez Range, sous Masque choisissez Watershed(Dilate(LevelSet(M07, Ch07, Middle, 3), 2)). Définir l'image pour afficher à Ch07. Définir les valeurs de la zone minimale et maximale à 135 et 5000, respectivement. Définir les valeurs du ratio d'aspect minimum et maximum à 0,4 et 1, respectivement. Cliquez OK. Dans le champ Nom, modifiez le texte pour lire POLY puis cliquez sur OK puis Fermez. Le masque cellulaire polynucléest est complet.
- Créez les masques polycomposants.
- Poly Component Mask 1: Cliquez sur l'onglet Analyse, puis Masques, puis Nouveau, puis Fonction. Sous Fonction sélectionnez Composant, et sous Masque sélectionnez le masque POLY. Pour la zonede sélection des fonctionnalités de classement et pour l'ordre de tri, cliquez sur le bouton De radio Descendant. Définir le rang à 1. Cliquez OK puis OK à nouveau.
- Masques de composants POLY 2, 3 et 4 : Répétez toutes les étapes en 11.2.1 sauf définir Rank à 2, 3 et 4 pour créer les masques de composants individuels.
- Compte spot à l'aide du masque POLY.
- Cliquez sur l'onglet Analyse, puis Caractéristiques, puis Nouveau. Pour le type de fonctionnalité, sélectionnez Spot Count. Pour Masque, choisissez le masque POLY et fixez la Connectivité à 4. Cliquez sur Définir le nom par défaut et cliquez sur OK puis Fermer pour calculer les valeurs de fonctionnalité.
- Cliquez sur l'icône Histogram. Sélectionnez la population non-apoptotique. Pour la fonction X-axis, choisissez la fonction Spot Count-POLY-4.
- Région de comptage des taches MONO. Cliquez sur l'icône Région linéaire. Dessiner une région à travers le bac 1 sur l'histogramme créé en 11.3.2. Appelez cette région 1N.
- Région de comptage des taches TRI. Cliquez sur l'icône Région linéaire. Dessiner une région à travers le bac 3 sur l'histogramme créé en 11.3.2. Appelez cette région 3N.
- Quad MONO région de comptage des taches. Cliquez sur l'icône Région linéaire. Dessiner une région à travers le bac 4 sur l'histogramme créé en 11.3.2. Appelez cette région 4N.
- Identifier la population mono.
- Créez la fonction mono Aspect Ratio Feature. Cliquez sur l'onglet Analyse, puis Caractéristiques, puis Nouveau. Sous le typede fonctionnalité , sélectionnez la fonction Ratio Aspect et sous Masque sélectionnez Composant(1, Zone, POLY, Descendant). Cliquez sur Définir le nom par défaut, puis cliquez sur OK.
- Créez la fonction de circularité MONO. Avec la fenêtre Feature Manager toujours ouverte, cliquez sur Nouveau. Sous le typede fonctionnalité , sélectionnez la fonction Circularity et sous Masque sélectionnez Composant(1, Zone, POLY, Descendant). Cliquez sur Le nom par défaut, puis cliquez sur OK, puis cliquez sur Fermer pour calculer les valeurs de fonctionnalité.
- Pour la parcelle circulaire de points de cellules MONO, cliquez sur l'icône Dot Plot. Sélectionnez 1N comme population parente. Pour la fonction X Axis, choisissez Circularity-Component(1, Area, POLY, Descending) et pour la fonction Axe Y, choisissez Aspect Ratio-Composant(1, Zone, POLY, Descendant). Cliquez OK. Cliquez sur le bouton Région carrée et dessinez une région autour de la population cellulaire vers la partie supérieure droite de la parcelle. Nommez cette région Circulaire 1N. Cliquez à droite sur l'intrigue et cliquez sur Régions. Mettez en évidence la région Circulaire 1N. Modifier les coordonnées X à 20 et 55 et changer les coordonnées Y à 0,85 et 1,0. Cliquez sur Fermer.
- Créez la fonction Zone POLY/Area-M07. Cliquez sur l'onglet Analyse, puis Caractéristiques, puis Nouveau. Sous Le typede fonctionnalité , sélectionnez la fonction Zone et sous Masque sélectionnez Composant(1, Zone, POLY, Descendant). Cliquez sur Définir le nom par défaut, puis cliquez sur OK.
- Avec la fenêtre Feature Manager toujours ouverte, cliquez sur Nouveau puis sous Type de fonctionnalité cliquez sur le bouton radio combiné. À partir de la liste des entités, mettez en évidence la composante zone (1, zone, POLY, descente) et cliquez sur la flèche vers le bas pour l'ajouter à la définition de fonctionnalité. Cliquez sur le symbole de division (/). Sélectionnez la fonction Zone M07 et cliquez sur la flèche vers le bas pour l'ajouter à la définition de la fonctionnalité. Cliquez sur Définir le nom par défaut et cliquez sur OK. Cliquez sur Fermer pour commencer à calculer les valeurs de fonctionnalité.
- Pour la dernière parcelle de point de population MONO, cliquez sur l'icône de l'intrigue point. Sélectionnez Circular 1N comme population parente. Pour la fonction X Axis, choisissez Aspect Ratio-M07 et pour la fonction Axe Y, choisissez Area-Component(1, Area, POLY, Descending) / Area-M07. Cliquez OK. Cliquez sur le bouton Région carrée et dessinez une région autour de la majorité des cellules. Nommez cette région Mononucleated. Cliquez à droite sur l'intrigue et cliquez sur Régions. Mettez en valeur la région mononuclée. Modifier les coordonnées X à 0,85 et 1.0 et changer les coordonnées Y à 0,55 et 1,0. Cliquez sur Fermer.
REMARQUE : Reportez-vous à l'article 11 du Supplément 1 - Protocole complet pour créer les masques, les caractéristiques et les parcelles afin d'identifier les populations trinucléaires et polynuclées finales.
12. Créer une vue personnalisée pour examiner les masques BNC et MN
- Cliquez sur le bouton Propriétés de la galerie d'images, puis cliquez sur l'onglet Afficher. Cliquez sur l'onglet Composites puis cliquez sur Nouveau. Sous nom type Ch01/Ch07. Cliquez sur Ajouter l'image. Sous Image choisissez Ch01 et fixez le Pourcentage à 100. Cliquez à nouveau sur Ajouter l'image, sous Image choisissez Ch07 et fixez le pourcentage à 100.
- Cliquez sur Les masques BNC et MN de type Nouveau et dessous
- Cliquez sur Ajouter colonne. Sous Type d'image choisissez Ch01 et sous Masque choisissez Aucun
- Cliquez sur Ajouter colonne. Sous Type d'image choisissez Ch07 et sous Masque choisissez Aucun
- Cliquez sur Ajouter colonne. Sous Type d'image choisissez Ch07 et sous Masque choisissez BNC
- Cliquez sur Ajouter colonne. Sous Type d'image choisissez Ch07 et sous Masque choisissez masque MN
- Cliquez sur Ajouter colonne. Sous Image Type cliquez sur le bouton de radio Composite. L'image composite Ch01/Ch07 doit être automatiquement ajoutée à la vue. Cliquez sur OK pour fermer la fenêtre Image Gallery Properties.
13. Créer une vue personnalisée pour examiner le masque POLY
- Se référer à l'article 13 du Supplément 1 - Protocole complet pour créer une vue personnalisée pour examiner le masque POLY
14. Créer un tableau statistique pour énumérer les événements clés
- Cliquez sur l'onglet Rapports puis cliquez sur Définir le rapport statistique. Dans la nouvelle fenêtre, cliquez sur Ajouter des colonnes.
- Ajoutez la statistique du nombre de BNC. Sous Statistiques, sélectionnez Compter et sous Population sélectionnée choisir la population bnCs. Cliquez sur Ajouter des statistiques pour ajouter la statistique à la liste.
- Répétez l'étape 14.2 pour créer des colonnes séparées pour les populations MN BNCs, MONO, TRI et POLY. Cliquez sur Fermer puis cliquez sur OK.
- Le modèle d'analyse de données est complet (Figure 2). Enregistrer le modèle (Fichier, Enregistrer comme template). La liste complète des masques peut être trouvée dans Le Supplément 2 - Liste des Masques.
15. Fichiers d'expérience de processus de lots à l'aide du modèle d'analyse de données
- Sous le menu Outils, cliquez sur Fichiers de données par lots, puis cliquez sur Ajouter Batch dans la nouvelle fenêtre.
- Dans la nouvelle fenêtre, cliquez sur Ajouter des fichiers pour sélectionner les fichiers d'expérience (.rif) à ajouter au lot. Sous l'option Sélectionnez un modèle ou un fichier d'analyse de données (.ast, .daf), cliquez sur l'icône de dossier ouvert pour naviguer et ouvrir le modèle d'analyse de données (fichier.ast) qui a été enregistré à l'étape 14.4.
- Cliquez sur le bouton Rapport statistique préliminaire pour prévisualiser le tableau des statistiques. Aucune valeur ne sera affichée ici puisqu'elles n'ont pas encore été calculées. Toutefois, cette étape sert de vérification pour s'assurer que le modèle d'analyse approprié a été sélectionné avant l'exécution du lot.
- Cliquez ok pour fermer la fenêtre actuelle. Cliquez ensuite sur Soumettre des lots pour commencer le traitement par lots de tous les fichiers.
- Lorsque le traitement par lots est terminé, un fichier .txt sera disponible dans le dossier qui contient tous les fichiers .rif. Utilisez ces statistiques pour calculer la génotoxicité et la cytotoxicité.
16. Calcul des paramètres de génotoxicité et de cytotoxicité
- Calcul de la génotoxicité : Pour calculer la génotoxicité, utilisez le tableau statistique créé en 15,5. Diviser le nombre de cellules dans la population de BNC MN par le nombre de cellules dans la population de BNCs puis multiplier par 100 :

- Calcul de la cytotoxicité : Déterminer le nombre total de cellules POLY en résumant le nombre de cellules TRI et QUAD.
- Calculez l'indice de prolifération de cytokinesis-bloc (CBPI) en utilisant le nombre de cellules dans le MONO, les BNC et le POLY comme suit :
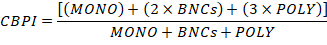
- Enfin, calculez la cytotoxicité de chaque culture en utilisant les valeurs CBPI de la culture témoin (C) et de la culture chimiquement exposée (T) comme suit :

- Calculez l'indice de prolifération de cytokinesis-bloc (CBPI) en utilisant le nombre de cellules dans le MONO, les BNC et le POLY comme suit :
Résultats
La méthode d'analyse décrite dans le présent document permet l'identification et la notation automatiques des BNC, avec et sans MN, pour calculer la génotoxicité. En outre, les cellules MONO et POLY sont également automatiquement identifiées et notées pour calculer la cytotoxicité. Les critères de notation publiés6,34 qui doivent être respectés lors de la notation de ces événements sont implémentés dans le logiciel d'analyse de données MIFC. Les résultats présentés ici indiquent que des augmentations statistiquement significatives de la fréquence de MN avec la cytotoxicité croissante peuvent être détectées après exposition des cellules tK6 lymphoblastoïdes humaines aux produits chimiques induisants bien connus de MN (Mitomycin C et Colchicine). Des résultats similaires pour d'autres produits chimiques testés ont été démontrés dans une publication distincte32. En outre, les résultats de l'utilisation de Mannitol montrent que les produits chimiques induisant non-MN peuvent également être correctement identifiés en utilisant la méthode MIFC décrite ici. Les paramètres décrits dans le protocole pour créer tous les masques, les caractéristiques et les limites de la région devront probablement être ajustés si différents types de cellules (par exemple les cellules de hamster chinois) sont utilisés pour effectuer l'attaque.
La figure 3 montre quatre panneaux sélectionnés pour identifier les BNC (figure3A-3D). Voici un histogramme qui permet de sélectionner des cellules avec deux noyaux (figure 3A) et des parcelles bivariées qui permettent la sélection de BNC s'agit d'une circularité similaire (figure 3B), de zones et d'intensités similaires (figure 3C ) et les BNC qui ont des noyaux bien séparés et non superposés (figure 3D) selon la notation critieria6,34. Figure 3 E montre les images BF et Hoechst ainsi que les masques BNC et MN indiquant que les BNC avec un seul ou plusieurs MN peuvent être identifiés et énumérés. Cela permet de calculer la génotoxicité en déterminant le taux de BNC micronucléées dans la population finale de BNC. La figure 4 montre l'application de la fonction Spot Count à l'aide du masque POLY pour identifier les cellules MONO, TRI et QUAD. Le nombre de cellules TRI et QUAD peut alors être résumé pour obtenir le nombre final de cellules POLY (tableau 1). Cela permet de calculer la cytotoxicité en utilisant la formule indiquée dans le protocole. Par conséquent, chaque point de dose de l'expérience peut être évalué par des paramètres de génotoxicité et de cytotoxicité.
La figure 5 montre les valeurs de génotoxicité et de cytotoxicité pour l'aneugène Colchicine, le clastogène Mitomycin C et pour un contrôle négatif, Mannitol. Pour la colchicine (figure 5A) les doses de 0,02 à 0,05 g/mL ont produit des augmentations statistiquement significatives de la fréquence du MN, allant respectivement de 1,28 % à 2,44 % par rapport au contrôle des solvants (tableau 1). Dans le cas de la mitomycine C (figure 5B) les deux doses supérieures de 0,4 et 0,5 g/mL ont produit des fréquences MN statistiquement significatives par rapport aux contrôles des solvants. Ces fréquences MN étaient de 0,93 % à 0,4 g/mL et de 1,02 % à 0,5 g/mL (tableau2). Enfin, pour Mannitol (figure 5C), aucune doses testées n'induit une cytotoxicité de plus de 30 %, et elles n'ont pas non plus produit d'augmentation significative de la fréquence du MN par rapport aux contrôles des solvants, comme prévu (tableau 3).

Figure 1 : Réglages d'instruments MIFC. Une capture d'écran des paramètres MIFC tel que décrit dans la section 7 du protocole. (A) Réglage de la puissance laser de 405 nm à 10 mW. (B) Réglage des canaux BF 1 et 9. (C) Sélection de l'objectif de grossissement 60x. (D) Sélection de la vitesse de débit la plus lente qui génère des images avec la plus haute résolution. (E) Préciser le nombre d'événements à collecter à 20 000. (F) Cliquer sur le bouton Charge pour commencer le processus de charge de l'échantillon. (G) Cliquer sur le bouton Acquérir pour commencer à acquérir des images. (H) Cliquer sur le bouton Retour pour retourner tout échantillon inutilisé. (I) Scatterplot de BF Aspect Ratio versus BF Area pour la sélection des cellules individuelles. (J) Scatterplot de Hoechst Gradient RMS versus BF Gradient RMS pour la sélection des cellules focalisées. (K) Histogramme de l'intensité Hoechst pour la sélection des cellules positives d'ADN. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 2 : Stratégie de gating logiciel logiciel d'analyse. Une capture d'écran de la stratégie de gating décrite à la section 9 du protocole. Les régions sont indiquées dans l'ordre séquentiel pour l'identification des cellules binuclées (boîte rouge), des micronucléées (boîte jaune), et des cellules mono- et polynucléées (boîte bleue). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
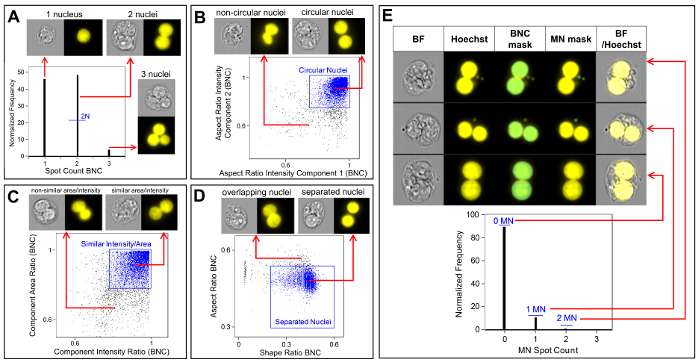
Figure 3 : Identification et notation des BNC avec et sans MN. (A) Sélection de cellules qui ont deux noyaux distincts. (B) Identification des cellules binucleated (BNC) qui ont deux noyaux très circulaires grâce à l'utilisation de la fonction d'intensité de ratio d'aspect. (C) Sélection de BNC s'il y a des noyaux avec des zones et des intensités similaires. Ceci est accompli en calculant le rapport de la zone des deux noyaux et le rapport du rapport d'aspect des deux noyaux. (D) Utilisation des caractéristiques du ratio de forme et du ratio d'aspect pour identifier les BNC qui ont deux noyaux bien séparés. (E) La fonction Spot Count utilisant le masque micronunomique (MN) démontrant que les BNC avec un seul ou plusieurs MN peuvent être identifiés et énumérés. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
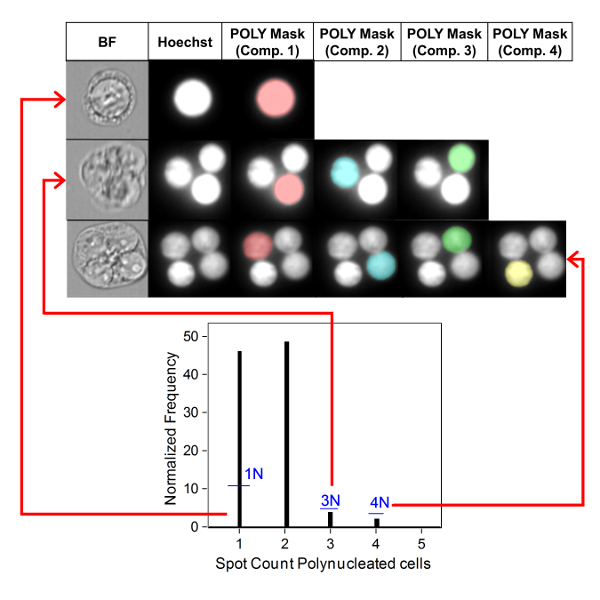
Figure 4 : Identification et notation des cellules MONO et POLY. Utilisation de la fonction de comptage des taches pour identifier et énumérer les cellules mono-, tri et quadranuclées. Le masque de composants 1 permet l'identification des cellules mononucléées (image du haut). Les masques de composants 1 à 3 permettent d'identifier les cellules trinulées (image du milieu). Les masques de composants 1 à 4 permettent d'identifier les cellules quadranuclées (image du bas). Ce chiffre a été modifié à partir de Rodrigues 201832. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
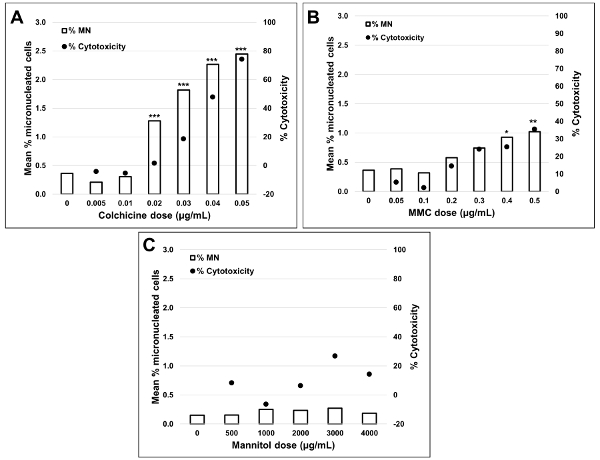
Figure 5 : Quantification de la cytotoxicité. Cytotoxicité quantifiée à l'aide de l'indice de prolifération des blocs de cytokinesis (cercles noirs) et de génotoxicité quantifiée à l'aide du pourcentage de MN (barres claires) à la suite d'une exposition de 3 h et d'une récupération de 24 h pour (A) Colchicine, (B) Mitomycine C et ( C) Mannitol. Les augmentations statistiquement significatives de la fréquence mN par rapport aux témoins sont indiquées par les étoiles (test au carré de chi;p 'lt; 0.05,'p 'lt; 0.01,'p 'lt; 0.001). Toutes les quantités sont la moyenne de deux répliques à chaque point de dose. Ce chiffre a été modifié à partir de Rodrigues 201832. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
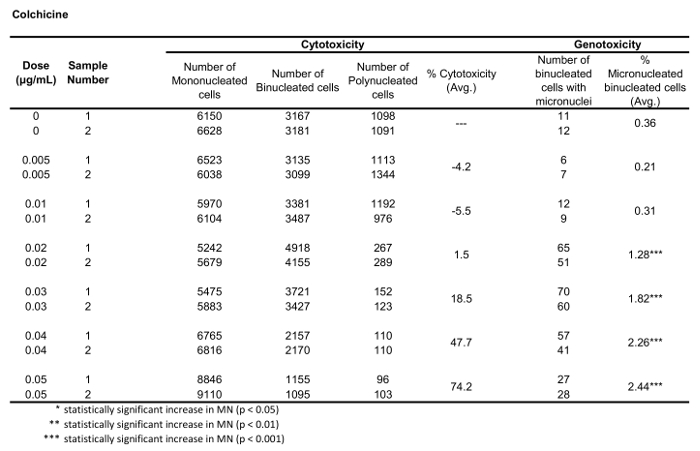
Tableau 1 : Les paramètres requis pour calculer la cytotoxicité (le nombre de cellules mono-, bi et polynucléées) et la génotoxicité (nombre et pourcentage de cellules binucléées micronucléées) pour la colchicine. Toutes les quantités calculées sont la moyenne de deux répliques à chaque point de dose.
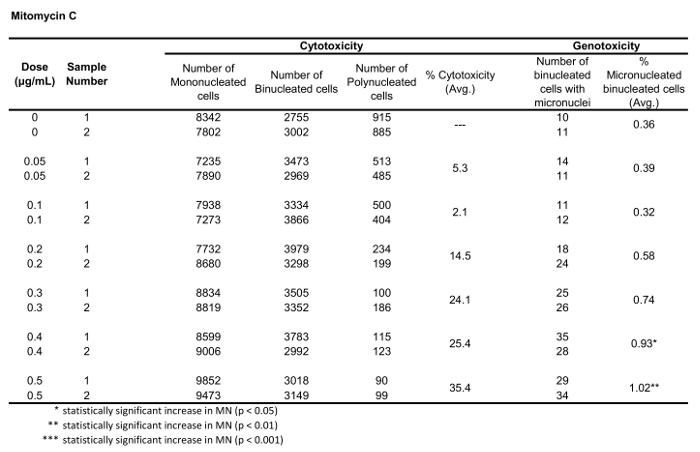
Tableau 2 : T il paramètres nécessaires pour calculer lacytotoxicité (le nombre de cellules mono-, bi et polynucléées) et la génotoxicité (le nombre et le pourcentage de cellules binuclées micronucléées) pour la mitomycine C. Toutes les quantités calculées sont la moyenne de deux répliques à chaque point de dose.
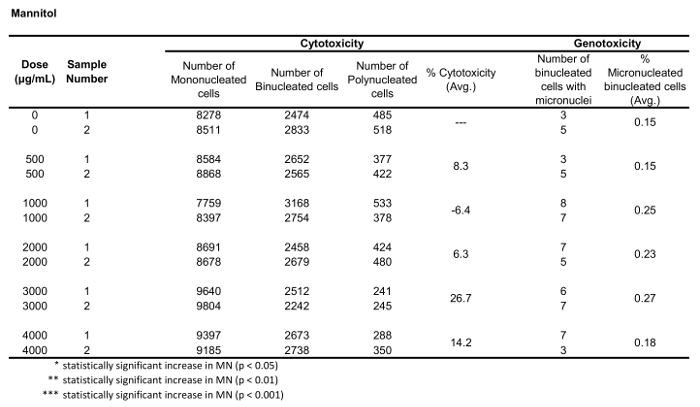
Tableau 3 : Les paramètres requis pour calculer la cytotoxicité (nombre de cellules mono-, bi et polynucléées) et la génotoxicité (nombre et pourcentage de cellules binucléées micronucléées) pour Mannitol. Toutes les quantités calculées sont la moyenne de deux répliques à chaque point de dose.
Supplément 1: Protocole complet. Veuillez cliquer ici pour télécharger ce fichier.
Supplément 2: Liste des masques. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Dans une publication récente, Verma et coll. ont souligné l'importance de développer un système qui combine l'avantage à haut débit de la cytométrie des flux avec les avantages de l'analyse d'images35. L'analyse IN vitro MIFC MN décrite dans cet article satisfait à cette citation et a le potentiel de surmonter de nombreux défis susmentionnés dans les méthodes de microscopie et de cytométrie de flux. Le protocole décrit ici démontre que la cytotoxicité et la génotoxicité peuvent être évaluées à l'aide de MIFC. La préparation des échantillons, la coloration cellulaire et la collecte de données sont simples, mais il y a des étapes critiques dans le protocole qui devraient toujours être mises en œuvre. L'ajout de chlorure de potassium (KCl) aux cellules est essentiel pour gonfler les cellules, générant une séparation entre les noyaux principaux. Cela garantit que l'algorithme de masquage peut identifier tous les noyaux individuels dans les BNC et les cellules POLY (cellules POLY) qui est nécessaire pour leur énumération. En outre, KCL fournit la séparation entre les noyaux et MN, qui est essentiel pour le masquage mN précis et la quantitation. En outre, l'utilisation de formaline après l'ajout de KCl empêche les cellules de lysing pendant la centrifugation. L'ajout de Cytochalasin B provoque des cellules TK6 qui ont subi plus d'une division nucléaire d'être assez grande. En conséquence, le cytoplasme devient fragile et peut lyse si la centrifugation est effectuée immédiatement après l'ajout de KCl. En outre, il est très important d'introduire Hoechst à l'échantillon en fonction du nombre de cellules dans l'échantillon et non en fonction d'une concentration finale. Par exemple, une concentration finale de 10 g/mL de Hoechst tachera uniformément un échantillon de 1 x 106 cellules, mais peut ne pas tacher adéquatement un échantillon contenant 5 x 106 cellules et peut entraîner de nombreuses cellules avec des noyaux faiblement tachés, ce qui rend l'analyse difficile. Il est également important de noter que Hoechst peut être remplacé par un autre colorant d'ADN tel que DAPI si le MIFC est équipé du laser d'excitation 405 nm ou DRAQ5 si le MIFC est équipé du laser d'excitation de 488 nm et/ou 642nm d'excitation(s). Si vous modifiez la tache nucléaire, il est essentiel de titrer la tache afin de trouver la concentration appropriée pour la puissance laser requise/désirée.
Lors de la collecte de données sur le MIFC, il est important de déterminer les limites optimales de la région pour les fonctionnalités Gradient RMS. Les limites présentées dans ce protocole peuvent nécessiter un ajustement en raison de légères variations entre les instruments MIFC. L'application de cette fonctionnalité lors de la collecte de données est essentielle pour s'assurer que l'imagerie hautement ciblée est capturée. Si les fichiers de données contiennent de nombreuses images floues ou non focalisées, il est probable que les algorithmes de masquage dans le logiciel d'analyse mettront mal en évidence les artefacts de coloration dans les zones floues, conduisant à un grand nombre d'artefacts faux positifs étant marqués comme MN. Bien que les techniques de traitement d'image décrites ici puissent être difficiles, une fois qu'un modèle d'analyse a été développé dans le logiciel MIFC, le traitement par lots permet d'analyser automatiquement les fichiers de données, éliminant ainsi l'intervention de l'utilisateur et, par conséquent, le marqueur parti-pris. En outre, si une lignée cellulaire autre que les cellules TK6 est utilisée pour effectuer l'évaluation, il sera nécessaire de modifier les masques et les limites de la région que les propriétés morphologiques (par exemple, la taille) des cellules diffèrent de celles des cellules TK6.
Les résultats présentés ici (figure 5) montrent des augmentations statistiquement significatives de l'induction de MN lors de l'exposition des cellules TK6 à diverses doses de Mitomycine C et de Colchicine. Des augmentations statistiquement significatives de la fréquence du MN par rapport aux contrôles de solvants ont été observées pour plusieurs doses dans les deux produits chimiques. En outre, aucune dose de Mannitol n'a induit une cytotoxicité au-dessus de 30%, ni une augmentation statistiquement significative de la fréquence de MN par rapport aux contrôles de solvant, comme prévu. Le protocole décrit dans ce document utilisant MIFC pour effectuer l'analyse in vitro MN donne des résultats attendus à partir de produits chimiques de contrôle positifs et négatifs. Il est très important d'effectuer un certain nombre d'expériences à l'aide de la fois des contrôles solvants et des produits chimiques de contrôle négatifs pour développer des valeurs de base de la fréquence de MN ainsi que l'indice de prolifération des blocs de cytokinesis (CBPI). Pour la génotoxicité, des augmentations statistiquement significatives de la fréquence mN sont déterminées par comparaison avec les fréquences MN de base qui doivent être bien connues pour le type de cellule utilisé. En outre, tous les calculs de cytotoxicité sont basés sur le CBPI des échantillons témoins et, par conséquent, les taux de référence des cellules MONO, BNC et POLY doivent être bien quantifiés dans les contrôles.
Plusieurs limitations et avantages de l'utilisation du MIFC dans le contexte de l'analyse MN ont été décrits dans les travaux précédents29,32. Les principales limitations concernent des fréquences MN inférieures par rapport à la microscopie, qui résulte probablement à la fois du manque de flexibilité lors de la mise en œuvre des critères de notation dans le logiciel d'analyse ainsi que de la profondeur de champ limitée du MIFC. Des masques bien profilés peuvent être créés pour identifier avec précision les noyaux principaux, mais MN qui touchent (ou très près) les noyaux principaux pourraient être capturés dans le masque BNC. En outre, très petit MN qui peut être assez facilement marqué à l'aide de microscopie sont probablement mal manqué lors de l'utilisation de MIFC en raison de la limite inférieure sur le paramètre de zone du masque MN pour éviter de marquer de petits artefacts. En plus des difficultés présentes dans l'analyse des données basées sur l'image, en raison de sa conception, MIFC obtient des images de projection en deux dimensions d'objets cellulaires tridimensionnels. Cela provoque probablement certains MN d'être capturé à une profondeur différente de mise au point que les deux MN principal, ce qui les rend très sombre et non décorable en utilisant le masquage. En outre, une petite fraction de MN pourrait résider derrière l'un des deux noyaux principaux, ce qui les rend impossibles à visualiser et à marquer. Par conséquent, compte tenu de ces difficultés, il faut faire preuve de prudence lorsqu'on interprète des augmentations importantes de la fréquence des MN à faibles doses.
Malgré ces lacunes, la méthode MIFC décrite ici offre plusieurs avantages par rapport à d'autres techniques. Fenech et coll. ont proposé des critères et des lignes directrices qui devraient être pris en considération lors de l'élaboration de systèmes et de méthodologies automatisés pour les analyses MN36. Il s'agit notamment, sans s'y limiter, de la visualisation directe des noyaux principaux et du cytoplasme, de la détermination de la fréquence du MN à partir de diverses doses du produit chimique ou de l'agent testé et de la capacité de quantifier la morphologie et de déterminer la position de tous les noyaux et MN pour s'assurer qu'ils sont dans le cytoplasme. Cet article montre que la méthode MIFC développée pour effectuer l'examen in vitro MN satisfait (ou possède le potentiel de satisfaire) ces critères. Plus précisément, les images des noyaux et mN peuvent être capturées par les lasers fluorescents tandis que l'imagerie cytoplasmique peut être obtenue en utilisant la LED BF. L'imagerie des cellules avec la morphologie nucléaire normale peut être automatiquement différenciée de ces cellules avec la morphologie irrégulière utilisant une combinaison des masques et des dispositifs avancés. Les résultats présentés pour la colchicine et la mitomycine C (figure 5) montrent que la génotoxicité et la cytotoxicité peuvent être évaluées à diverses doses par rapport aux contrôles des solvants et que des fréquences MN statistiquement significatives sont observées Attendu. En outre, la ligne directrice 487 de l'OCDE recommande de marquer 2 000 BNC par concentration d'essai afin d'évaluer la présencede MN afin de déterminer la génotoxicité ainsi qu'au moins 500 cellules par concentration d'essai pour déterminer la cytotoxicité 9; cela peut prendre plus de 1 h à l'aide de la microscopie manuelle. Le protocole et les résultats de cet article montrent qu'une moyenne d'environ 6 000 BNC, 16 000 cellules MONO et 800 cellules POLY ont été capturées et notées par concentration d'essai dans environ 20 min. Le taux rapide d'acquisition de données et le nombre élevé de cellules candidates notées en si peu de temps soulignent un autre avantage important de l'utilisation du MIFC pour effectuer l'analyse in vitro du MN.
Bien que les résultats présentés dans le présent document soient encourageants, ils sont représentatifs d'une méthode de preuve de concept précoce. Ce travail devrait être suivi d'une étude plus approfondie d'un ensemble chimique plus vaste et plus diversifié qui couvre de multiples classes et mécanismes de génotoxicité et de cytotoxicité, comme ceux suggérés par Kirkland et al.37 La réalisation de telles études est temps et laborieux, et tombent en dehors de la portée de ce document cependant, ces études à plus grande échelle fourniront un aperçu précieux dans la capacité de la méthode à identifier de manière fiable les agents faiblement génotoxiques. La méthodologie présentée ici n'a pas encore été miniaturisée à un format de micropuits, ce qui permettrait un dépistage plus rapide et plus efficace dans une plus grande gamme de doses. En tant que tel, dans sa forme actuelle, l'essais IN vitro MN mi-IN, présenté ici, pourrait être le mieux adapté aux études de suivi à forte intensité de main-d'œuvre ou à la recherche sur les bonnes pratiques de laboratoire. Cependant, la méthode continuera d'être optimisée et validée, et possède le potentiel de permettre une plus grande flexibilité dans la détection d'événements chimiques spécifiques liés à la morphologie, tels que l'exposition à l'aneugène qui augmente la proportion de cellules noyaux non circulaires qui sont encore scorables38. Enfin, la méthode MIFC offre l'occasion d'introduire des biomarqueurs supplémentaires dans l'analyse MN (p. ex. coloration kinetochore) afin de fournir une vue plus complète du mécanisme d'induction du MN.
Déclarations de divulgation
L'auteur est employé par Luminex Corporation, le fabricant du cytomètre de flux d'imagerie multispectrale ImageStream qui a été utilisé dans ce travail.
Remerciements
L'auteure remercie Christine Probst (Luminex Corporation) pour les efforts qu'elle a déployés dans l'élaboration de formes antérieures du modèle d'analyse des données, ainsi que la Dre Haley Pugsley (Luminex Corporation) et le Dr Phil Morrissey (Luminex Corporation) pour l'examen et l'édition de la manuscrit.
matériels
| Name | Company | Catalog Number | Comments |
| 15 mL centrifuge tube | Falcon | 352096 | |
| Cleanser - Coulter Clenz | Beckman Coulter | 8546931 | Fill container with 200 mL of Cleanser. https://www.beckmancoulter.com/wsrportal/page/itemDetails?itemNumber=8546931#2/10//0/25/1/0/asc/2/8546931///0/1//0/ |
| Colchicine | MilliporeSigma | 64-86-8 | |
| Corning bottle-top vacuum filter | MilliporeSigma | CLS430769 | 0.22 um filter, 500 mL bottle |
| Cytochalasin B | MilliporeSigma | 14930-96-2 | 5 mg bottle |
| Debubbler - 70% Isopropanol | EMD Millipore | 1.3704 | Fill container with 200 mL of Debubbler. http://www.emdmillipore.com/US/en/product/2-Propanol-70%25-%28V%2FV%29-0.1-%C2%B5m-filtred,MDA_CHEM-137040?ReferrerURL=https%3A%2F%2Fwww.google.com%2F |
| Dimethyl Sulfoxide (DMSO) | MilliporeSigma | 67-68-5 | |
| Dulbecco's Phosphate Buffered Saline 1X | EMD Millipore | BSS-1006-B | PBS Ca++MG++ Free |
| Fetal Bovine Serum | HyClone | SH30071.03 | |
| Formaldehyde, 10%, methanol free, Ultra Pure | Polysciences, Inc. | 04018 | This is what is used for the 4% and 1% Formalin. CAUTION: Formalin/Formaldehyde toxic by inhalation and if swallowed. Irritating to the eyes, respiratory systems and skin. May cause sensitization by inhalation or skin contact. Risk of serious damage to eyes. Potential cancer hazard. http://www.polysciences.com/default/catalog-products/life-sciences/histology-microscopy/fixatives/formaldehydes/formaldehyde-10-methanol-free-pure/ |
| Hoechst 33342 | Thermo Fisher | H3570 | 10 mg/mL solution |
| Mannitol | MilliporeSigma | 69-65-8 | |
| MEM Non-Essential Amino Acids 100X | HyClone | SH30238.01 | |
| MIFC - ImageStreamX Mark II | EMD Millipore | 100220 | A 2 camera ImageStreamX Mark II eqiped with the 405nm, 488nm, and 642nm lasers was used. http://www.emdmillipore.com/US/en/life-science-research/cell-analysis/amnis-imaging-flow-cytometers/imagestreamx-Mark-ii-imaging-flow-cytometer/VaSb.qB.QokAAAFLzRop.zHe,nav?cid=BI-XX-BDS-P-GOOG-FLOW-B325-0006 |
| MIFC analysis software - IDEAS | EMD Millipore | 100220 | The companion software to the MIFC (ImageStreamX MKII) |
| MIFC software - INSPIRE | EMD Millipore | 100220 | This is the software that runs the MIFC (ImageStreamX MKII) |
| Mitomycin C | MilliporeSigma | 50-07-7 | |
| NEAA Mixture 100X | Lonza BioWhittaker | 13-114E | |
| Penicllin/Streptomycin/Glutamine solution 100X | Gibco | 15070063 | |
| Potassium Chloride (KCl) | MilliporeSigma | P9541 | |
| Rinse - Ultrapure water or deionized water | NA | NA | You can use any ultrapure water or deionized water. Fill container with 900 mL of Rinse. |
| RNase | MilliporeSigma | 9001-99-4 | |
| RPMI-1640 Medium 1X | HyClone | SH30027.01 | |
| Sheath - PBS | EMD Millipore | BSS-1006-B | This is the same as Dulbecco's Phosphate Buffered Saline 1X Ca++MG++ free. Fill container with 900mL of Sheath. |
| Sterile water | HyClone | SH30529.01 | |
| Sterilizer - 0.4-0.7% Hypochlorite | VWR | JT9416-1 | This is assentually 10% Clorox bleach that can be made by deluting Clorox bleach with water. Fill container with 200 mL of Sterilzer. |
| System Calibration Reagent - SpeedBead | EMD Millipore | 400041 | Each tube holds ~10 mL. https://www.emdmillipore.com/US/en/life-science-research/cell-analysis/amnis-imaging-flow-cytometers/support-training/XDqb.qB.wQMAAAFLBDUp.zHu,nav |
| T25 flask | Falcon | 353109 | |
| T75 flask | Falcon | 353136 | |
| TK6 cells | MilliporeSigma | 95111735 |
Références
- Bonassi, S., et al. An increased micronucleus frequency in peripheral blood lymphocytes predicts the risk of cancer in humans. Carcinogenesis. 28 (3), 625-631 (2007).
- Fenech, M. The in vitro micronucleus technique. Mutation Research - Fundamental and Molecular Mechanisms of Mutagenesis. 455 (1-2), 81-95 (2000).
- Fenech, M. The Lymphocyte Cytokinesis-Block Micronucleus Cytome Assay and its Application in Radiation Biodosimetry. Health Physics. 98 (2), 234-243 (2010).
- Hintzsche, H., et al. Fate of micronuclei and micronucleated cells. Mutation Research - Reviews in Mutation Research. 771, 85-98 (2017).
- Fenech, M. The advantages and disadvantages of the cytokinesis-block micronucleus method. Mutation Research. 392 (1-2), 11-18 (1997).
- Fenech, M. Cytokinesis-block micronucleus cytome assay. Nature Protocols. 2 (5), 1084-1104 (2007).
- Fenech, M., Holland, N., Chang, W. P., Zeiger, E., Bonassi, S. The HUman MicroNucleus Project - An international collaborative study on the use of the micronucleus technique for measuring DNA damage in humans. Mutation Research - Fundamental and Molecular Mechanisms of Mutagenesis. 428 (1-2), 271-283 (1999).
- Kirsch-Volders, M., et al. Report from the in vitro micronucleus assay working group. Mutation Research - Genetic Toxicology and Environmental Mutagenesis. 540 (2), 153-163 (2003).
- OECD Library. Test No. 487: In vitro Mammalian Cell Micronucleus Test. OECD Guidelines for the Testing of Chemicals, Section 4. , (2016).
- Aardema, M. J., et al. SFTG international collaborative study on in vitro micronucleus test. III. Using CHO cells. Mutation Research - Genetic Toxicology and Environmental Mutagenesis. 607 (1), 61-87 (2006).
- Clare, M. G., et al. SFTG international collaborative study on in vitro micronucleus test. II. Using human lymphocytes. Mutation Research - Genetic Toxicology and Environmental Mutagenesis. 607 (1), 37-60 (2006).
- Lorge, E., et al. SFTG international collaborative study on in vitro micronucleus test. I. General conditions and overall conclusions of the study. Mutation Research - Genetic Toxicology and Environmental Mutagenesis. 607 (1), 13-36 (2006).
- Oliver, J., et al. SFTG international collaborative study on in vitro micronucleus test. V. Using L5178Y cells. Mutation Research - Genetic Toxicology and Environmental Mutagenesis. 607 (1), 125-152 (2006).
- Wakata, A., et al. SFTG international collaborative study on in vitro micronucleus test. IV. Using CHL cells. Mutation Research - Genetic Toxicology and Environmental Mutagenesis. 607 (1), 88-124 (2006).
- Fenech, M., et al. Intra- and inter-laboratory variation in the scoring of micronuclei and nucleoplasmic bridges in binucleated human lymphocytes: Results of an international slide-scoring exercise by the HUMN project. Mutation Research - Genetic Toxicology and Environmental Mutagenesis. 534 (1-2), 45-64 (2003).
- Decordier, I., et al. Automated image analysis of cytokinesis-blocked micronuclei: an adapted protocol and a validated scoring procedure for biomonitoring. Mutagenesis. 24 (1), 85-93 (2009).
- Decordier, I., et al. Automated image analysis of micronuclei by IMSTAR for biomonitoring. Mutagenesis. 26 (1), 163-168 (2011).
- Schunck, C., Johannes, T., Varga, D., Lorch, T., Plesch, A. New developments in automated cytogenetic imaging: unattended scoring of dicentric chromosomes, micronuclei, single cell gel electrophoresis, and fluorescence signals. Cytogenetic and Genome Research. 104 (1-4), 383-389 (2004).
- Rossnerova, A., Spatova, M., Schunck, C., Sram, R. J. Automated scoring of lymphocyte micronuclei by the MetaSystems Metafer image cytometry system and its application in studies of human mutagen sensitivity and biodosimetry of genotoxin exposure. Mutagenesis. 26 (1), 169-175 (2011).
- Nüsse, M., Marx, K. Flow cytometric analysis of micronuclei in cell cultures and human lymphocytes: Advantages and disadvantages. Mutation Research - Genetic Toxicology and Environmental Mutagenesis. 392 (1-2), 109-115 (1997).
- Avlasevich, S. L., Bryce, S. M., Cairns, S. E., Dertinger, S. D. In vitro micronucleus scoring by flow cytometry: Differential staining of micronuclei versus apoptotic and necrotic chromatin enhances assay reliability. Environmental and Molecular Mutagenesis. 47 (1), 56-66 (2006).
- Bryce, S. M., Bemis, J. C., Avlasevich, S. L., Dertinger, S. D. In vitro micronucleus assay scored by flow cytometry provides a comprehensive evaluation of cytogenetic damage and cytotoxicity. Mutation Research/Genetic Toxicology and Environmental Mutagenesis. 630 (1-2), 78-91 (2007).
- Bryce, S. M., et al. Flow cytometric 96-well microplate-based in vitro micronucleus assay with human TK6 cells: Protocol optimization and transferability assessment. Environmental and Molecular Mutagenesis. 54 (3), 180-194 (2013).
- Fenech, M. Commentary on the SFTG international collaborative study on the in vitro micronucleus test: to Cyt-B or not to Cyt-B. Mutation Research. 607 (1), 9-12 (2006).
- Basiji, D. A. . Methods in Molecular Biology. 1389, 13-21 (2016).
- Rodrigues, M. A., Beaton-Green, L. A., Kutzner, B. C., Wilkins, R. C. Automated analysis of the cytokinesis-block micronucleus assay for radiation biodosimetry using imaging flow cytometry. Radiation and Environmental Biophysics. 53 (2), 273-282 (2014).
- Rodrigues, M. A., Beaton-Green, L. A., Kutzner, B. C., Wilkins, R. C. Multi-parameter dose estimations in radiation biodosimetry using the automated cytokinesis-block micronucleus assay with imaging flow cytometry. Cytometry Part A. 85 (10), 883-893 (2014).
- Rodrigues, M. A., Beaton-Green, L. A., Wilkins, R. C. Validation of the cytokinesis-block micronucleus assay using imaging flow cytometry for high throughput radiation biodosimetry. Health Physics. 110 (1), 29-36 (2016).
- Rodrigues, M. A., Probst, C. E., Beaton-Green, L. A., Wilkins, R. C. Optimized automated data analysis for the cytokinesis-block micronucleus assay using imaging flow cytometry for high throughput radiation biodosimetry. Cytometry Part A. 89 (7), 653-662 (2016).
- Rodrigues, M. A., Probst, C. E., Beaton-Green, L. A., Wilkins, R. C. The effect of an optimized imaging flow cytometry analysis template on sample throughput in the reduced culture cytokinesis-block micronucleus assay. Radiation Protection Dosimetry. 172 (1-3), 223-229 (2016).
- Wang, Q., et al. Automated Triage Radiation Biodosimetry: Integrating Imaging Flow Cytometry with High-Throughput Robotics to Perform the Cytokinesis-Block Micronucleus Assay. Radiation Research. 191 (4), 342-351 (2019).
- Rodrigues, M. A. Automation Of The In vitro Micronucleus Assay Using The ImageStream® Imaging Flow Cytometer. Cytometry Part A. 93, 706-726 (2018).
- Rodrigues, M. A., Beaton-Green, L. A., Wilkins, R. C., Fenech, M. F. The potential for complete automated scoring of the cytokinesis block micronucleus cytome assay using imaging flow cytometry. Mutation Research/Genetic Toxicology and Environmental Mutagenesis. 836, 53-64 (2018).
- Fenech, M., et al. HUMN project: Detailed description of the scoring criteria for the cytokinesis-block micronucleus assay using isolated human lymphocyte cultures. Mutation Research - Genetic Toxicology and Environmental Mutagenesis. 534 (1-2), 65-75 (2003).
- Verma, J. R., et al. Evaluation of the automated MicroFlow® and Metafer™ platforms for high-throughput micronucleus scoring and dose response analysis in human lymphoblastoid TK6 cells. Archives of Toxicology. 91 (7), 2689-2698 (2017).
- Fenech, M., et al. HUMN project initiative and review of validation, quality control and prospects for further development of automated micronucleus assays using image cytometry systems. International Journal of Hygiene and Environmental Health. 216 (5), 541-552 (2013).
- Kirkland, D., et al. Updated recommended lists of genotoxic and non-genotoxic chemicals for assessment of the performance of new or improved genotoxicity tests. Mutation Research/Genetic Toxicology and Environmental Mutagenesis. 795, 7-30 (2016).
- Verma, J. R., et al. Investigating FlowSight® imaging flow cytometry as a platform to assess chemically induced micronuclei using human lymphoblastoid cells in vitro. Mutagenesis. 33 (4), 283-289 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon