Method Article
Alta velocità effettiva In Vitro valutazione della latenza inversione agenti sulla trascrizione di HIV e Splicing
In questo articolo
Riepilogo
Un protocollo di throughput elevato per la valutazione funzionale di riattivazione efficiente HIV e clearance del provirus latenti è descritto e applicato dalla prova dell'impatto di interventi sulla trascrizione di HIV e di splicing. Risultati rappresentativi dell'effetto di latenza inversione agenti sulla trascrizione LTR-driven e splicing sono forniti.
Abstract
L'HIV rimane incurabile a causa dell'esistenza di un serbatoio di cellule che nutre forma stabile e latente del virus, che rimane invisibile al sistema immunitario e non è mirato dall'attuale terapia antiretrovirale (cART). Trascrizione e splicing sono stati indicati per rafforzare la latenza di HIV-1 nelle cellule T CD4 + di riposo. Inversione di latenza mediante l'uso di agenti di inversione di latenza (LRA) nell'approccio "shock and kill" è stata studiata estesamente nel tentativo di eliminare questo serbatoio ma finora non ha dimostrato alcun successo negli studi clinici a causa della mancanza di sviluppo di piccoli adeguato molecole che possono perturbare in modo efficiente questo serbatoio. Il protocollo qui presentato fornisce un metodo per attendibilmente ed efficientemente valutare gli agenti di inversione di latenza (LRA) sulla trascrizione di HIV e giunzione. Questo approccio si basa sull'uso di un reporter di doppio colore LTR-driven che consente di misurare contemporaneamente l'effetto di un LRA sulla trascrizione e splicing tramite flusso cytometry. Il protocollo descritto qui è adeguato per le celle aderenti, come pure le cellule in sospensione. È utile per il testing di un gran numero di farmaci in un sistema di alta velocità di trasmissione. Il metodo è tecnicamente semplice da implementare ed economicamente vantaggiosa. Inoltre, l'uso di citometria a flusso permette la valutazione di attuabilità delle cellule e così droga tossicità allo stesso tempo.
Introduzione
Nonostante la terapia antiretrovirale a lungo termine efficace HIV persiste in uno stato latente come un provirus integrato in memoria di cellule T CD4 +1. La struttura della cromatina del HIV-1 5' promotore di ripetizione terminale lunga (litro) e modificazioni epigenetiche come metilazione dell'istone e deacetilazione di methyltransferases del DNA (DNMT) e istone deacetilasi (HDAC) sono importanti meccanismi che conducono alla repressione trascrizionale e così post-integrazione latenza2,3. Una grande varietà di agenti inversione di latenza (LRA) è stata studiata per la loro efficacia per indurre la produzione di virus in vitro e in vivo da latente infettati riposo T CD4 + cellule4,5,6,7 ,8. Tra gli enti locali e regionali testato, HDACi (inibitori HDAC) e scommessa bromodomain inibitori (BETis) inducono decondensazione della cromatina e rilascio della trascrizione positivo allungamento fattore b (P-TEFb) rispettivamente, che porta a successive alleviare il trascrizionale repressione a 5' LTR e attivazione di HIV espressione9,10,11,12,13. Tuttavia, la grandezza della riattivazione raggiunta da questi enti era limitata, come è stato osservato solo un modesto aumento associato unspliced HIV mRNA (RNA noi), indicativo della trascrizione virale, ex vivo14,15. D'importanza, questi enti anche non è riuscito a indurre una riduzione della frequenza delle cellule latente infettate.
L'espressione di HIV può essere ulteriormente limitata da splicing inefficiente16 , nonché i difetti in export nucleare di moltiplicare impiombato HIV RNA (RNA MS)17. Così, sono necessari identificazione nuove classi di enti locali e regionali che sono più potenti e possono interessare diversi aspetti di post-integrazione produzione del virus. Inoltre, lo sviluppo di nuovi saggi che aiutano a definire i composti ottimali per invertire in modo efficiente la latenza è richiesto.
Qui, un protocollo è presentato, che utilizza un approccio ad alta produttività per la valutazione funzionale dell'impatto degli interventi su trascrizionale LTR di HIV e splicing. In breve, un nuovo dual LTR-driven colore reporter sistema pLTR.gp140/EGFP. RevΔ38/DsRed (Figura 1) viene utilizzato per valutare la riattivazione di HIV tramite flusso cytometry. In questo reporter fluorescente, l'espressione del mRNA di unspliced HIV (4 kb) conduce all'espressione migliorata della proteina fluorescente verde (EGFP), mentre l'espressione di mRNA splicing (2Kb) porterebbe all'espressione della proteina fluorescente di Discosoma sp. rosso (DsRed). In breve, abbiamo usato una proteina di fusione di Env-EGFP fluorescente, gp140unc. EGFP, dove la sequenza codificante di EGFP è stata posizionata nel telaio con una forma non-spaccata e troncata della busta (Env). Le modifiche sono state introdotte per l'ablazione del sito di clivaggio impedendo la dissociazione di Env in gp120 e gp41-EGFP e troncare la proteina gp160 prima il dominio transmembrana creando un analogo solubile di Env, che facilita la piegatura corretta e espressione di EGFP. Al momento l'espressione all'interno di una cella, Rev si localizza nel nucleo dove media l'esportazione nucleare-citoplasmico di 4KB env mRNA tramite l'interazione con l'elemento sensibile rev (RRE). Il troncamento di Env non compromette la RRE, che si trova tra gp120, gp41 e A7 3' luogo della giuntura. In questo sistema, splicing a HIV-1 splice donatore 4 (SD4) e della giuntura acceptor 7 (SA7) risultati nella produzione di un 2 kb mRNA che codifica per una proteina non funzionale di Rev troncata all'amminoacido 38 fusa alla proteina fluorescente di DsRed, RevΔ38-DsRed. Brevemente, DsRed è stato inserito in essone 2nd di Rev all'amminoacido 38 da sovrapposizione estensione18. Per facilitare l'esportazione nucleare di unspliced mRNA, un vettore di espressione mammifera codifica Rev (pCMV-RevNL4.3) è stato co-trasfettato con la costruzione del reporter fluorescenti (Figura 2). Questo costrutto unico reporter qui descritto è utile nella valutazione di alto-rendimento di trascrizione di HIV e intestature, senza la necessità di utilizzare vettori virali.
Protocollo
Nota: Procedure per la clonazione, trasformazione e sequenziamento sono discussi altrove18,19. I protocolli qui iniziano dalla trasfezione di vettori di espressione mammifera (Figura 3).
1. transfezione delle cellule HEK293T con doppio colore Reporter costruire
- Coltivare le cellule HEK293T nel mezzo dell'Aquila di Dulbecco modificato (DMEM) completati con 10% (v/v) siero bovino fetale (FBS), penicillina (100 U/mL) e streptomicina (100 µ g/mL) in un 5% CO2 incubatore a 37 ° C. Dopo lo scongelamento, gli esperimenti di cellule HEK293T passaggio 2 - 3 volte prima di utilizzarli nella transfezione. Questo darebbe il tempo di cellule per recuperare dalla procedura di scongelamento.
Attenzione: Contaminazione del micoplasma di colture cellulari rimane un problema serio. Buona pratica di laboratorio e test di routine di colture cellulari sono essenziali per ridurre il rischio di contaminazione del micoplasma ed evitare diffusione20. - Dividere le celle 1:2 un giorno prima della semina e trasferirli nel terreno DMEM fresco. Coltivare le cellule per 24 h in un 5% CO2 incubatore a 37 ° C.
- Il giorno prima di transfezione, rimuovere il supporto dal pallone dall'aspirazione e lavare le cellule con tamponato fosfato salino (DPBS) soluzione di Dulbecco priva di calcio (Ca2 +) e magnesio (Mg2 +) per rimuovere tracce di siero.
- Pipettare 5 mL della soluzione di tripsina-EDTA (0.05% - 10 mM in PBS) nel recipiente di cultura e posizionarlo a 37 ° C in un incubatore di 5% CO2 per 2-5 min.
- Quando le cellule sono disconnessi, aggiungere 5 mL di DMEM supplementato con 10% (v/v) FBS ulteriormente inibire l'attività della tripsina che può danneggiare le cellule.
- Risospendere delicatamente pipettando la sospensione cellulare su e giù per rompere i grumi.
- Diluire le celle 01:10 in trypan blu macchia (0,4%).
- Contare le cellule vive che non occupano trypan blue utilizzando un microscopio (obiettivo 10x) e un emocitometro.
- Calcolare il numero di cellule vitali per millilitro prendendo la media del conteggio delle cellule e moltiplicandolo per 10.000 e per il fattore di diluizione (01:10) dal trypan blu macchia.
- Piastra 2 x 104 cellule in 100 μL di medium DMEM supplementato con 10% FBS senza antibiotici in una piastra a 96 pozzetti a fondo piatto per 24 h.
- Per tutti i pozzetti, diluire 400 ng di pLTR.gp140/EGFP. RevD38/DsRed, 20 ng di pCMV-RevNL4.3 e 100 ng di pCMV-Tat101AD8-bandiera DNA in 50 μL di mezzo libero del siero. Mescolare delicatamente. Per gli esperimenti senza Tat, utilizzare un vuoto abbinati Tat vettore pcDNA3.1 (-).
Nota: Si consiglia vivamente l'uso di DNA privo di endotossina. Per questo, preparare il DNA privo di endotossina utilizzando un kit di purificazione dell'acido nucleico midi o maxiprep. Determinare la purezza di DNA misurando il rapporto di 260/280 OD, che dovrebbe essere compreso tra 1,7 e 1,9. - Mix il reagente del lipido delicatamente prima dell'uso, diluire 0.65 μL in 50 μL di mezzo libero del siero. Mescolare delicatamente e incubare per 5 min a temperatura ambiente.
- Combinare il DNA diluito con il reagente del lipido diluito.
- Mescolare delicatamente e incubare per 20 minuti a temperatura ambiente. La soluzione potrebbe apparire nuvolosa.
Nota: Passare al punto 1.15. entro 30 min. - Pipettare 100 μL per pozzetto dei complessi lipidici reagente-DNA drop-wise in cima le cellule.
- Mescolare delicatamente a dondolo avanti e indietro la piastra.
-
Incubare la piastra per 5 h in una 5% CO2 incubatrice umidificata a 37 ° C.
- Ogni miscela di reazione è sufficiente per la transfezione un singolo pozzo (96-well). Regolare la quantità e volume dei componenti secondo il numero di droga e combinazione che sarebbe stato testato e conti per pipettaggio variazioni. Tutte le condizioni sono solitamente testate in triplici copie.
- Transfect ulteriori pozzi con 100 ng di CMV-driven EGFP e DsRed-Express DNA plasmidico fini di compensazione.
2. trattamento di HEK293T Transfected le cellule con latenza agenti di retromarcia
Nota: Prima di ogni analisi, è possibile determinare lo stato fisiologico di ogni LRA misurando la vitalità delle cellule dopo l'esposizione ad alta e bassa della dose della droga con analisi delle cellule di proliferazione.
- Diluire ciascuna LRA alla concentrazione di lavoro appropriato con il mezzo di crescita (ad esempio, 1 μM per JQ1(+)).
Attenzione: Ricostituire i farmaci liofilizzati con il solvente appropriato a una concentrazione di 10 mM. Conservare Stock in tutti gli enti locali e regionali a-80 ° C in un'uso singola aliquota (5 µ l) per evitare ripetuti cicli di congelamento-scongelamento. - Attentamente aspirare mezzo di transfezione con un multicanale e sostituire con 100 μL contenente appropriato LRA (tabella 1). Aggiungere il terreno molto delicatamente lungo la parete del pozzo per evitare di staccare le cellule transfected HEK293T, che sono conosciute per staccare facilmente dalla superficie della placca di cultura.
- Incubare la piastra per 48 h in una 5% CO2 incubatrice umidificata a 37 ° C.
3. colorazione delle cellule transfettate con tintura di attuabilità risolvibile per analisi di citometria a flusso
Attenzione: Rimozione detriti e cellule morte è indispensabile per eliminare i falsi positivi e di ottenere risultati di altissima qualità.
- Staccare le cellule nei mezzi usando 100 μL di tampone fosfato salino (PBS) per pozzetto e pipettando delicatamente alti e Bassi (~ 5 volte) con un multicanale, evitando la bava dei media. Se necessario, staccare le cellule dalla piastra utilizzando 35 µ l per pozzetto di soluzione di tripsina-EDTA (0.05% - 10 mM in PBS) e incubare 5 min a 37 ° C, prima neutralizzazione con mezzo contenente siero.
- Trasferire le cellule a una piastra a 96 pozzetti fondo V.
- Girare le cellule per 5 min a 500 x g a 4 ° C, quindi aspirare accuratamente il medio/PBS senza toccare le cellule.
- Lavare le cellule almeno 1 volta con 200 µ l di PBS gratis proteina/del siero.
- Centrifugare a 500 g per 5 min a 4 ° C, quindi eliminare il supernatante inclinando la piastra e rimuovere il tampone di lavaggio senza toccare le cellule.
- Preparare una soluzione di lavoro di attuabilità risolvibile colorante diluendo l'attuabilità tintura 1: 1000 in proteina/siero libero PBS. Preparare 50 µ l della macchia diluita per pozzetto.
Nota: Attuabilità coloranti sono disponibili in una gamma di colori adatti per l'utilizzo con laser blu, rossi e violetto. Preparare una soluzione stock di attuabilità risolvibile tintura di risospensione un flaconcino di liofilizzato tintura (componente A) con 50 μL di DMSO anidro (componente B). Conservare a-20 ° C in un uso singola aliquota (1 μL), al riparo dalla luce. - Aggiungere 50 μL di colorante diluito vitalità ad ogni pozzetto e mescolare le cellule pipettando su e giù con un multicanale.
- Macchia per 10-15 min a 4 ° C, al riparo dalla luce.
- Lavare 1 - 2 volte con 150 µ l di tampone di lavaggio (PBS con 1% bovino dell'albumina di siero e 2 mM EDTA).
- Centrifugare a 500 g per 5 min a 4 ° C. Scartare il surnatante.
- Fissare le cellule con 100 μL di preparati 1% formaldeide nel tampone di lavaggio per 10-15 min a 4 ° C al buio.
Attenzione: La formaldeide è molto tossica. Preparare la soluzione di 1% di formaldeide in una cappa per evitare l'inalazione indossando guanti e occhiali di sicurezza per la protezione. - Lavare le cellule 1 - 2 volte con 100 μL di tampone di lavaggio.
- Centrifugare a 500 g per 5 min a 4 ° C. Scartare il surnatante.
- Risospendere il pellet cellulare in 70 μL di tampone di lavaggio.
Nota: Il protocollo può essere messo in pausa qui. Fissa le cellule potrebbero essere conservate a 4 ° C al buio per l'analisi il giorno successivo il citofluorimetro.
4. EGFP e DsRed misure di citometria a flusso e analisi dei dati
Nota: Analizzare la trascrizione di HIV (% EGFP) e splicing (% DsRed) su un citometro a flusso. Filtrare il campione prima della corsa con un cellulare-setaccio di 70 μm o maglia di nylon 100 μm per evitare di intasare l'ugello.
- Avviare il citometro e il computer almeno 10 minuti prima dell'uso per garantire laser scaldare.
- Prima di eseguire i campioni e iniziare l'acquisizione dei dati, è necessario riempire il serbatoio di guaina con soluzione fisiologica 0,9% e assicurarsi che il serbatoio di scarico viene aggiunta una tavoletta di ipoclorito di sodio.
- Controllare la cella di flusso per le bolle d'aria.
- Verificare che i filtri e i rivelatori siano appropriati per l'esperimento.
Nota: Per EGFP, l'utilizzo del laser blu (488 nm) e 530/30 passa-banda, mentre DsRed Express viene rilevato in modo ottimale utilizzando il laser giallo (561 nm) e 610/20 passa-banda. Un laser blu (488 nm) e passa-banda 610/20 può essere utilizzato anche per rilevare DsRed espressione. - Controllare il citometro prestazioni e sensibilità attraverso rivelatori eseguendo perle di calibrazione.
- Regolare il forward (FSC-A) e tensioni (SSC-A) dispersione laterale con senza macchia campione affinché la popolazione principale è sullo schermo e chiaramente percepibile.
Nota: FSC (luce diffusa diretta) è una misura della luce diffrazione in un angolo piatto, che dipende dal volume della cella (cella superficie o dimensione), mentre SSC (lato-sparsi luce) è una misura di diffrazione della luce in un angolo retto, che è proporzionale alla granularità cellulare e complessità interna. - Eseguire la compensazione manuale o automatica utilizzando i campioni di singolo-macchiato garantendo minimo spillover di EGFP + popolazione nella DsRed rivelatore e viceversa.
Attenzione: Ai fini della compensazione, è necessario utilizzare un campione di cellule che esprimono ciascuno della proteina fluorescente colore singolo così come le cellule singolarmente colorate con il colorante di attuabilità risolvibile. Non utilizzare perline di compensazione FITC e PE per compensazioni di proteine fluorescenti EGFP e DsRed a causa di differenti caratteristiche spettrali. Controlli di compensazione dovrebbero essere abbastanza brillanti da risolvere popolazione positiva e negativa. - Creare trame e impostare le porte usando la fluorescenza meno uno (FMO) controlli.
- Acquisire e registrare un minimo di 10.000 eventi di cellule vitali per campione. Eseguire gli esempi a media o bassa velocità per evitare doppietti passando per l'intercetta di laser. Utilizzare impulsi geometria gating come SSC-A contro SSC-H per eliminare doppietti. Verificare la stabilità della corsa tracciando tempo contro SSC-A per vedere come anche il flusso è durante l'esecuzione.
- Alla fine della corsa, pulire la fluidica di citometria a flusso con detergenti concentrati poi acqua per 5 minuti ciascuno.
- Arresto del sistema, eliminare i rifiuti e ri-riempire il serbatoio di guaina dopo l'acquisizione.
- Analizzare i dati utilizzando un software di analisi dati di citometria a flusso. Escludere i detriti cellulari e ciuffo (doppiette) basato su dispersione avanti e laterali, quindi eliminare le cellule morte usando la macchia di tintura di redditività (negativo della popolazione = cellule vive). Identificare le cellule che esprimono EGFP e DsRed (Figura 4), così come la percentuale di prodotto impiombato DsRed /(DsRed + EGFP) (Figura 5).
- Determinare l'effetto del LRA sulla trascrizione di HIV e splicing confrontando le cellule non trattate e le cellule esposte a individuo o una combinazione di enti locali e regionali (Figura 5).
Risultati
Risultati rappresentativi sono illustrati nella Figura 5 per l'espressione di HIV-1 unspliced (EGFP) e impiombato (DsRed) prodotti dopo il trattamento con l'inibitore bromodomain JQ1. Sia JQ1(+) e Tat ha aumentato significativamente la percentuale di cellule che esprimono EGFP (2.18 e 4.13 FC sopra DMSO rispettivamente; n = 3) indicativo di unspliced trascrizioni. Inoltre, JQ1(+) aumentato significativamente la percentuale di cellule esprimenti DsRed (46,6 FC sopra DMSO) così come la percentuale di prodotto impiombato (7,37 FC sopra DMSO) a un livello analogo come Tat (59,6 e 5,83 FC sopra DMSO, rispettivamente) che conferma la capacità di JQ1(+) per attivare la trascrizione di HIV e splicing. D'altra parte, il trattamento con il controllo di stereoisomero JQ1(-) abolisce JQ1(+) effetto sulla trascrizione di HIV e splicing confermando il successo del protocollo.
In una recente pubblicazione, abbiamo dimostrato da analisi di RNA un accumulo di HIV impiombato trascrizioni seguendo JQ1(+) trattamento21. Infatti, i nostri dati hanno rivelato i cambiamenti coerenti nei livelli di unspliced e impiombate trascrizioni che sono stati rispecchiati dall'espressione della proteina EGFP e DsRed in questo modello dopo il trattamento con JQ1(+). Questi risultati indicano che EGFP e DsRed che esprimono le cellule riflettono la capacità dell'inibitore bromodomain JQ1(+) di indurre la trascrizione di HIV e splicing.
In sintesi, abbiamo validato l'uso della pLTR.gp140/EGFP. RevΔ38/DsRed reporter in un set-up ad alta produttività per la valutazione dell'effetto di enti locali e regionali sulla trascrizione di HIV-1 e splicing. Sub-ottimale quantità di LRA o aumento della tossicità potrebbe portare a risultati di scud. Ottimizzare la quantità di farmaco utilizzato, preservando la vitalità cellulare può migliorare l'accuratezza dei risultati.
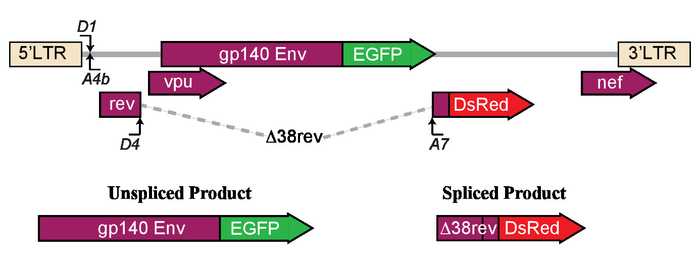
Figura 1: schematico del modello utilizzato per determinare gli effetti della latenza inversione agenti sulla trascrizione LTR-driven e splicing. L'esprime di costrutto LTR a che o proteina di unspliced busta (Env) fuso enhanced proteina fluorescente verde (EGFP) o impiombato proteine Δ38rev con DsRed. Questa figura è stata ristampata da Khoury et al.21. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: costruisce design. PLTR.gp140/EGFP (A). RevΔ38/DsRed d'impionbatura reporter permette l'espressione di EGFP (gp140-EGFP) e non-funzionali della proteina Rev in busta troncata all'amminoacido 38 fusa alla proteina fluorescente DsRed (RevΔ38-DsRed). (B) pCMV-RevNL4.3 permette l'espressione della proteina di Rev. (C) pCMV-Tat101AD8-Flag consente l'espressione della proteina di Tat101(AD8) con una bandierina del C-terminale. I plasmidi di espressione sono costruiti usando PCR sovrapposizioni e raccolta di limitazione. Tutte le mappe vettoriali sono state create utilizzando SnapGene software, versione 4.1.9. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: analisi progettazione per rapida valutazione della latenza di HIV inversione agenti. Il metodo corrente per la valutazione dell'effetto di enti locali e regionali sulla trascrizione di HIV-1 e splicing include i) semina delle cellule HEK293T un giorno prima del ii) trasfezione con vettori di espressioni seguite da trattamento iii) LRA. IV) le cellule vengono analizzate dal post-trattamento 48 h di citometria a flusso. Usando questo protocollo, 32 (in triplice copia) a 96 condizioni potrebbero essere testati per piastra. Clicca qui per visualizzare una versione più grande di questa figura.
Tabella 1: esempio rappresentativo del set-up piatto e i controlli necessari. 1 a 3 colonne corrispondono ai controlli negativo (-) con nessuna droga e solvente solo (DMSO 1: 5000) e controlli positivo (+) quali Tat (100 ng), PMA/PHA = forbolo miristato acetato/phytohaemagglutinin (10 nM PMA, PHA 10 μg/mL), JQ1 (+ /-) (1 μM), VOR = vorinostat ( 0,5 μM) e PAN = panobinostat (30 nM). Enti locali e regionali sconosciuti sono testati in triplice copia sopra una gamma di concentrazioni (15.625 a 1000 nM). Ai fini della compensazione, cellule che esprimono ciascuno della proteina fluorescente colore singolo (EGFP + e DsRed +) così come le cellule colorate con la tintura di attuabilità e cellule non macchiate sono inclusi in ogni seduta. Per favore clicca qui per scaricare questo file.

Figura 4: Gating strategia utilizzata per l'analisi di citometria a flusso. Il primo passo nella strategia di gating è basato sulla dispersione in avanti e laterale, che permette la distinzione delle cellule di interesse basato sulle proprietà size e granularità di queste cellule. È consigliabile che questa gating essere generoso come possibile includere tutti gli eventi, mentre la rimozione di detriti cellulari e cellule morte, che si trovano nell'angolo sinistro inferiore della trama densità. Quindi rimuovere doppietti da dataset utilizzando la geometria di impulso gating, SSC-A contro SSC-H e ulteriore stretta su cellule vive usando la tintura di attuabilità. Infine, un canale di scarico (viola) ed EGFP o DsRed vengono utilizzati per definire le celle di interesse. L'uso della fluorescenza meno uno (FMO) controlli sono fondamentali nella definizione della popolazione di interesse e affrontare le questioni di ricaduta da un altro fluorocromo nel canale di interesse. Dopo l'acquisizione, i dati sono analizzati utilizzando software di analisi dati di citometria a flusso. Questa figura è stata ristampata nella forma adattata Khoury et al.21. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5: risultati rappresentativi dell'effetto degli inibitori bromodomain sulla trascrizione di HIV e splicing. (A) esempi di due parametri dual colore fluorescenza EGFP contro DsRed densità trame dalle cellule untransfected (-Tat), le cellule transfected con 100 ng di pCMV-Tat101AD8-bandiera (+ Tat) e cellule trattati con DMSO, JQ1 (+) o JQ1 (-). (B) la media percentuale (%) delle cellule che esprimono EGFP, DsRed o prodotto impiombato (DsRed / DsRed + EGFP) da 3 esperimenti indipendenti è indicato. Confronti di ciascuna condizione di DMSO sono stati fatti usando il 2-way ANOVA test. Sono mostrati solo i confronti statisticamente significativi. p < 0,05; p < 0.01; p < 0,001. Le linee nere rappresentano il mean±SEM. DMSO (1: 5000), JQ1 (+) e JQ1(-) (1 μM). Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Data la difficoltà nel misurare la riattivazione del virus ex vivo, una vasta gamma di in vitro sono stati sviluppati modelli nel tempo al fine di studiare la latenza di HIV tra cui latente infettato linee a cellula T (J-Lats, ACH2, U1), modelli primari dell'infezione latente di riposo ( Modelli o ' Doherty, Lewin, Greene e Spina) o pre-attivato le cellule T CD4 + (Serafini, Marini, Planelles, Siliciano, modelli di Karn) con singolo turno o replica competente reporter virus22. Per modellare le condizioni fisiologiche della latenza di HIV in cellule T CD4 + di riposo, sistemi dual reporter fluorescente è seguito come il reporter RGH (Red-Green-HIV) sviluppato dal gruppo di Ivan Sadowski con un marcatore di LTR-driven Gag-eGFP e un mCherry CMV-driven in luogo di Nef23. Un simile virus reporter di doppio colore, Duo-Fluo, è stato sviluppato dal laboratorio di E. Verdin con un'espressione di EGFP LTR-driven e un marcatore fluorescente mCherry guidato da un promotore di EF1α24. In questo sistema, la EGFP è stato inserito all'estremità 3' del provirus al posto di Nef. L'eliminazione della busta in questi due sistemi impedisce più Arrotonda di infezioni. Inoltre, la combinazione delle due proteine fluorescenti permette la rilevazione di produttivamente (eGFP + mCherry +) e latente (eGFP - mCherry +) cellule infette. Tuttavia, parecchie imperfezioni dei virus reporter doppio colore erano notevoli in cellule T CD4 +, come l'infezione diretta delle cellule latente infettate dipende in larga misura lo stato di attivazione cellulare delle cellule di T23,24. In realtà, molto basso tasso di infezione è stata osservata mediante questi virus due reporter in cellule T CD4 + di riposo: 0,1% per RGH23 e 0,2% per DuoFluo25. Inoltre, come il CMV promotore è stato indicato per essere espressa a livelli inferiori in cellule non attivato26,27,28, un CMV inattivi o EF1α promotore porterebbe alla sottovalutazione di latente infetti popolazioni.
Abbiamo generato e caratterizzato un nuovo vettore reporter HIV per studiare l'istituzione di latenza e riattivazione del virus in un dosaggio elevato throughput. Diversi vantaggi al nostro metodo di screening includono i) costo efficacia con la capacità di valutare la trascrizione e splicing simultaneamente, ii) la possibilità di testare un gran numero di farmaci o combinazioni in un singolo esperimento, iii) rapida valutazione della effetto di enti locali e regionali mediante citometria a flusso (tempo lettura tipica 30-45 min per piastra ben 96 indipendentemente dal numero di parametri fluorescenti), iv) ad alta dinamica, v) adatto per automatizzato di transfezione elevato throughput e citometria a flusso mediante bracci robotici e piattaforma HTS, vi) veloce indurimento dei dati utilizzando un modello di area di lavoro di citometria di flusso, vii) ottimale per sospensione e cellule aderenti, viii) semplicità del modello e adattabilità alle misure di luminescenza e piastra lettore (dual luciferasi Firefly e Renilla) e ix) scalabilità (96 e 384 pozzetti formati di patè).
La propensione di un LRA o una combinazione di enti locali e regionali per attivare trascrizionale LTR di HIV e splicing così espressione di proteine fluorescenti EGFP e DsRed potrebbe essere esaminata mediante citometria a flusso utilizzando la nostra reporter d'impionbatura di HIV. Tuttavia, prima di testare la sinergia del romanzo enti locali e regionali, si consiglia di determinare le condizioni fisiologiche di ogni LRA misurando la vitalità delle cellule dopo l'esposizione ad una gamma di concentrazioni del farmaco singolo. Pertanto, compreso un marcatore delle cellule vivi e morti è essenziale per eliminare i falsi positivi e di ottenere risultati di altissima qualità. I controlli di compensazione o FMO è un altro aspetto importante dell'analisi di citometria a flusso. Si consiglia di utilizzare campioni singolarmente macchiati con garanzia minimo spillover di EGFP + popolazione nella DsRed e viceversa. Inoltre, è essenziale per conto di autofluorescenza delle cellule includendo cellule non colorate. Infine, un controllo regolare del citometro prestazioni e sensibilità attraverso i rivelatori è fondamentale soprattutto quando si misura campioni per uno studio che attraversa un lungo periodo di tempo.
Anche se non discusso nella sezione protocollo, ci sono diverse limitazioni del nostro sistema di reporter d'impionbatura. Per una discussione più approfondita, vedere la nostra precedente pubblicazione (Khoury et al., 2018). Esportazione della parzialmente o unspliced varianti di HIV mRNA è facilitata dalla proteina virale Rev e la sua associazione con l'elemento reattivo Rev (RRE) in questi mRNA specie29,30,31,32. La sovraespressione di Rev nel nostro sistema ci ha permesso di eludere il blocco principale di ritenzione nucleare di moltiplica impiombato e unspliced mRNAs osservati nel latente infettate T cellule17 e messa a fuoco sulla comprensione di come gli enti locali e regionali inducono la trascrizione e splicing così la riattivazione del virus. Inoltre, dato che il nostro sistema richiede la transfezione di 3 plasmidi, abbiamo scelto di HEK293T cellule perché l'efficienza di trasfezione alta di queste cellule. Causa la diversa disponibilità temporale e spaziale di fattori cellulari in cellule di T rispetto ad una linea cellulare di cancro, è importante convalidare i risultati del reporter d'impionbatura acquisito in cellule HEK293T in linee a cellula T e le cellule primarie, che potrebbe fornire grandi problemi tecnici a causa della loro limitata transfectability. Pertanto, l'uso di reagenti di consegna alternativi del DNA per la transfezione quali DMRIE-C, che ha dimostrato di essere particolarmente efficace per la transfezione di cellule in sospensione (ad es., Jurkat), o metodi di nucleofection come nucleoporatore Nucleofactor o Neon sistema di transfezione che hanno dimostrato per facilitare la consegna efficiente e affidabile di single o plasmidi più difficili a transfect cellule compreso le cellule di T di CD4 + primarie. In alternativa, sarebbe interessante testare questo sistema reporter nel contesto del full-length virus in un modello di cellula primaria di latenza utilizzando i metodi di transfezione di cui sopra. In realtà, le differenze nella disponibilità di trascrizione di host, allungamento e splicing fattori in una cellula di T rCD4 + possono influenzare la capacità di un LRA per riattivare provirus latenti. Infine, il nostro modello non riguarda se il LRA può colpire la trascrizione e la giunzione delle cellule degli astanti, e se può indurre le competenti virus replica. Tuttavia, avevamo il sospetto che misurando un aumento associato HIV U.S. e MS RNA, questo porterebbe alla produzione di virus competente replica nel surnatante della cultura. Questo potrebbe essere confermato conducendo il gold standard quantitativi virus escrescenza assay (QVOA)33.
A causa della natura complessa della latenza e per garantire la riattivazione del virus efficiente, una strategia combinatoria multiforme può essere richiesto34. Trattamento potenziale deve stimolare le vie multiple tra cui trascrizione e splicing, che precedentemente è stato indicato per svolgere i ruoli essenziali nella creazione di latenza16,35,36, 37. our LTR-driven reporter d'impionbatura consentirebbe un screening su vasta scala delle droghe che possono essere destinate in modo ottimale sia passi, trascrizione e splicing, aumentando la probabilità di trovare molecole che possono sinergizzare in modo efficiente, che porterebbe ad un più basso livelli di dose e quindi la tossicità di ogni farmaco.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Questo lavoro è stato supportato dal progetto grant APP1129320 e programma grant APP1052979 da NHMRC dell'Australia. Ringraziamo Dr. Adam Wheatley, Dr. Marina Alexander, Dr. Jenny L. Anderson e Michelle Y. Lee per la fornitura di costrutti essenziali e consigli per il corretto completamento di questo lavoro. Ringraziamo anche il personale della struttura di flusso di DMI per i loro consigli e la generosa assistenza nel mantenere il citometro a flusso utilizzato in questo studio.
Materiali
| Name | Company | Catalog Number | Comments |
| Cell culture | |||
| HEK293T cells (Human Embryonic Kidney cells) | ATCC | CRL-3216 | Replicates vectors carrying the SV40 region of replication. |
| Dulbecco's Modified Eagle's Medium (DMEM 1x + GlutaMAX-I) | Gibco | 10569-010 | + 4.5 g/L D-Glucose + 110 mg/L Sodium Pyruvate |
| Fetal Bovine serum | Gibco | 10099-141 | Origin Australia |
| Penicillin-Streptomycin | Sigma | P4458 | |
| Dulbecco's phosphate buffered saline (DPBS), no calcium, no magnesium | Gibco | 14190-136 | |
| Trypan blue Stain, 0.4% | Gibco | 15250 | |
| Trypsin-EDTA (0.05%), phenol red | Gibco | 25300054 | |
| Lipofectamine 2000 | Invitrogen | 11668-019 | Lipid transfection reagent |
| Opti-MEM I (1x) reduced serum medium | Gibco | 31985-070 | Serum free medium |
| NucleoBond Xtra Maxi | Marcherey-Nagel | 740414.50 | |
| pEGFP-N1 plasmid | Clontech (TaKaRa) | 6085-1 | Expression of EGFP in mammalian cells, CMVIE promoter. |
| pDsRed-Express-N1 | Clontech (TaKaRa) | 632429 | Expression of DsRed-Express in mammalian cells, CMVIE promoter. |
| pLTR.gp140/EGFP.RevD38/DsRed | Addgene | 115775 | |
| pCMV-RevNL4.3 | Addgene | 115776 | |
| pCMV-Tat101AD8-Flag | Addgene | 115777 | |
| Dimethyl sulfoxide (DMSO) | Millipore | 67-68-5 | |
| JQ1(+) | Cayman Chemical | 11187 | Stock at 10 mM in DMSO; working concentration 1 μM |
| JQ1(-) | Cayman Chemical | 11232 | Stock at 10 mM in DMSO; working concentration 1 μM |
| Phorbol Myristate Acetate (PMA) | Sigma-Aldrich | 16561-29-8 | Stock at 100 μg/mL in DMSO; working concentration 10 nM |
| Phytohaemagglutinin (PHA) | Remel | HA15/R30852701 | Stock at 1 μg/μL in PBS; working concentration 10 μg/mL |
| Vorinostat (VOR) | Cayman Chemical | 10009929 | Stock at 10 mM in DMSO; working concentration 0.5 μM |
| Panobinostat (PAN) | TRC | P180500 | Stock at 10 mM in DMSO; working concentration 30 nM |
| CellTiter 96 AQueous One Solution Cell Proliferation Assay | Promega | 63581 | |
| Venor GeM Classic | Minerva Biolabs | 11-1100 | Mycoplasma Detection Kit, PCR-based |
| Name | Company | Catalog Number | Comments |
| Flow cytometry reagents | |||
| LSR Fortessa | BD Biosciences | Flow cytometer (4 lasers-blue, red, violet and yellow) | |
| LSR II | BD Biosciences | Flow cytometer (3 lasers-blue, red and violet) | |
| LIVE/DEAD Fixable Near-IR Dead Cell Stain Kit | Life Technologies | L34976 | Viability dye: for 633 or 635 nm excitation, 400 assays. Component A and B are both provided in the kit. |
| Bovine Serum Albumin | Sigma | A2153 | |
| EDTA 0.5M pH8 | Gibco | 15575-038 | |
| Formaldehyde Solution 37/10 (37%) | Chem-Supply | FA010 | |
| BD FACS Diva CS&T Research Beads | BD Biosciences | 655050 | Calibration beads |
| Sphero Rainbow Calibration Particles (8 peaks) | BD Biosciences | 559123 | 3.0 - 3.4 mm |
| Sheath solution | Chem-Supply | SA046 | 90 g NaCl in 10 L water |
| HAZ-Tabs | Guest Medical | H8801 | Chlorine release tablets for disinfection |
| Decon 90 | Decon Laboratories Limited | N/A | Concentrated cleaning agents of flow cytometer. Working solution Decon 90 5%. |
| Sodium Hypochlorite (12-13% Solution) | Labco | SODHYPO-5L | Concentrated cleaning agents of flow cytometer. Working solution bleach 1%. |
| 7x | MPBio | IM76670 | Concentrated cleaning agents of flow cytometer. Working solution 7x 1%. |
| Name | Company | Catalog Number | Comments |
| Materials | |||
| Tissue culture flasks (75 cm2, canted neck, cap vented) | Corning | 430641U | |
| Tissue culture plates (96 well flat bottom with lid) | Costar | 3599 | |
| Tissue culture plates (96 well V-bottom without lid) | Costar | 3896 | |
| Centrifuge tubes (10 mL) | SARSTEDT | 62.9924.284 | 100x16 mm |
| Centrifuge tubes (50 mL) | CellStar | 227261 | 30x115 mm |
| Microcentrifuge tubes (1.5 mL) | Corning Axygen | MCT-150-C | |
| Serological Pipette (25 mL), sterile | Corning | CLS4489-200EA | |
| Serological Pipette (10 mL), sterile | Corning | CLS4488-200EA | |
| Serological Pipette (5 mL), sterile | Corning | CLS4487-200EA | |
| Reagent reservoirs (50 mL), sterile | Corning | CLS4470-200EA | |
| 5 mL Round-Bottom polystyrene test tube, with cell-strainer cap | Corning | 352235 | 12 x 75 mm style, 70 mm |
| Nylon Mesh | SEFAR | 03-100/32 | 100 mm |
| Titertube Micro test tubes, bulk | BIO-RAD | 2239391 | microfacs tubes |
| 5 mL Round-Bottom polystyrene test tube, without cap | Corning | 352008 | 12x75 mm style |
| Snap Caps for 12x75 mm Test Tubes | Corning | 352032 | |
| Counting chamber, Neubauer improved double net ruling, bright-line (Haemocytometer, LO-Laboroptik) | ProSciTech | SVZ4NIOU | 3x3 large squares of 1 mm2; Depth 0.100 mm; volume 0.1 mL; area minimum 0.0025 mm2 |
| Coverslips (Menzel-Gläser) | Grale Scientific | HCS2026 | 20 x 26 mm |
| Microscope | Nikon TMS | 310528 | |
| Centrifuge 5810R refrigerated | Eppendorf | 5811000487 | with rotor A-4-81 including adapters for 15/50 mL conical tubes |
| FLUOstar Omega microplate reader | BMG Labtech | N/A | Plate reader for cell proliferation assay. Filter 490 nm. |
| Name | Company | Catalog Number | Comments |
| Softwares | |||
| FACS Diva | BD Biosciences | Flow cytometer data acquisition and analysis program, version 8.0.1 | |
| FlowJo | FlowJo | FlowJo 10.4.2 | Flow cytometer data analysis program, FlowJo Engine v3.05481 |
| Omega | BMG Labtech | FLUOstar multi-user reader control, version 5.11 | |
| Omega - Data Analysis | BMG Labtech | MARS | FLUOstar data analysis, version 3.20R2 |
| Microsoft Excel | Microsoft | Excel:mac 2011 | version 14.0.0 |
| Prism | GraphPad | Prism 7 | version 7.0c |
Riferimenti
- Siliciano, J. D., et al. Long-term follow-up studies confirm the stability of the latent reservoir for HIV-1 in resting CD4+ T cells. Nature Medicine. 9 (6), 727-728 (2003).
- Khoury, G., et al. Ch. 8. HIV vaccine and cure - The Path Towards Finding an Effective Cure and Vaccine. 1075, Springer Nature. Adv Exp Med Biol (2018).
- Van Lint, C., Bouchat, S., Marcello, A. HIV-1 transcription and latency: an update. Retrovirology. 10, 67(2013).
- Archin, N. M., et al. HIV-1 Expression Within Resting CD4+ T Cells After Multiple Doses of Vorinostat. Journal of Infectious Diseases. 210, 728-735 (2014).
- Elliott, J. H., et al. Activation of HIV Transcription with Short-Course Vorinostat in HIV-Infected Patients on Suppressive Antiretroviral Therapy. PLoS Pathogens. 10, (2014).
- Leth, S., et al. Combined effect of Vacc-4x, recombinant human granulocyte macrophage colony-stimulating factor vaccination, and romidepsin on the HIV-1 reservoir (REDUC): a single-arm, phase 1B/2A trial. The Lancet HIV. 3, e463-e472 (2016).
- Rasmussen, T. A., et al. Panobinostat, a histone deacetylase inhibitor, for latent-virus reactivation in HIV-infected patients on suppressive antiretroviral therapy: a phase 1/2, single group, clinical trial. The Lancet HIV. 1, e13-e21 (2014).
- Søgaard, O. S., et al. The Depsipeptide Romidepsin Reverses HIV-1 Latency In Vivo. PLoS Pathogens. 11, (2015).
- Bartholomeeusen, K., Xiang, Y., Fujinaga, K., Peterlin, B. M. Bromodomain and extra-terminal (BET) bromodomain inhibition activate transcription via transient release of Positive Transcription Elongation Factor b (P-TEFb) from 7SK small nuclear ribonucleoprotein. Journal of Biological Chemistry. 287, 36609-36616 (2012).
- Boehm, D., et al. BET bromodomain-targeting compounds reactivate HIV from latency via a Tat-independent mechanism. Cell Cycle. 12, 452-462 (2013).
- Contreras, X., et al. Suberoylanilide hydroxamic acid reactivates HIV from latently infected cells. Journal of Biological Chemistry. 284 (11), 6782-6789 (2009).
- Rasmussen, T. A., et al. Comparison of HDAC inhibitors in clinical development: effect on HIV production in latently infected cells and T-cell activation. Human Vaccines & Immunotherapeutics. 9 (5), 993-1001 (2013).
- Wei, D. G., et al. Histone deacetylase inhibitor romidepsin induces HIV expression in CD4 T cells from patients on suppressive antiretroviral therapy at concentrations achieved by clinical dosing. PLoS Pathogens. 10 (4), e1004071(2014).
- Blazkova, J., et al. Effect of histone deacetylase inhibitors on HIV production in latently infected, resting CD4(+) T cells from infected individuals receiving effective antiretroviral therapy. Journal of Infectious Diseases. 206 (5), 765-769 (2012).
- Bullen, C. K., Laird, G. M., Durand, C. M., Siliciano, J. D., Siliciano, R. F. New ex vivo approaches distinguish effective and ineffective single agents for reversing HIV-1 latency in vivo. Nature Medicine. 20 (4), 425-429 (2014).
- Yukl, S. A., et al. HIV latency in isolated patient CD4+T cells may be due to blocks in HIV transcriptional elongation, completion, and splicing. Science Translational Medicine. 10, (2018).
- Lassen, K. G., Ramyar, K. X., Bailey, J. R., Zhou, Y., Siliciano, R. F. Nuclear retention of multiply spliced HIV-1 RNA in resting CD4+T cells. PLoS Pathogens. 2, 0650-0661 (2006).
- Alexander, M. R., Wheatley, A. K., Center, R. J., Purcell, D. F. J. Efficient transcription through an intron requires the binding of an Sm-type U1 snRNP with intact stem loop II to the splice donor. Nucleic Acids Research. 38, 3041-3053 (2010).
- Anderson, J. L., Johnson, A. T., Howard, J. L., Purcell, D. F. J. Both Linear and Discontinuous Ribosome Scanning Are Used for Translation Initiation from Bicistronic Human Immunodeficiency Virus Type 1 env mRNAs. Journal of Virology. 81, 4664-4676 (2007).
- Nikfarjam, L., Farzaneh, P. Prevention and detection of Mycoplasma contamination in cell culture. Cell J. 13 (4), 203-212 (2012).
- Khoury, G., et al. HIV latency reversing agents act through Tat post translational modifications. Retrovirology. 15 (1), 36(2018).
- Hakre, S., Chavez, L., Shirakawa, K., Verdin, E. HIV latency: experimental systems and molecular models. FEMS Microbiology Reviews. 36 (3), 706-716 (2012).
- Dahabieh, M. S., et al. Direct non-productive HIV-1 infection in a T-cell line is driven by cellular activation state and NFkappaB. Retrovirology. 11, 17(2014).
- Calvanese, V., Chavez, L., Laurent, T., Ding, S., Verdin, E. Dual-color HIV reporters trace a population of latently infected cells and enable their purification. Virology. 446 (1-2), 283-292 (2013).
- Chavez, L., Calvanese, V., Verdin, E. HIV Latency Is Established Directly and Early in Both Resting and Activated Primary CD4 T Cells. PLoS Pathogens. 11 (6), e1004955(2015).
- Hunninghake, G. W., Monick, M. M., Liu, B., Stinski, M. F. The promoter-regulatory region of the major immediate-early gene of human cytomegalovirus responds to T-lymphocyte stimulation and contains functional cyclic AMP-response elements. Journal of Virology. 63 (7), 3026-3033 (1989).
- Reeves, M., Sinclair, J. Aspects of human cytomegalovirus latency and reactivation. Current Topics in Microbiology and Immunology. 325, 297-313 (2008).
- Sambucetti, L. C., Cherrington, J. M., Wilkinson, G. W., Mocarski, E. S. NF-kappa B activation of the cytomegalovirus enhancer is mediated by a viral transactivator and by T cell stimulation. EMBO Journal. 8 (13), 4251-4258 (1989).
- Kula, A., et al. Characterization of the HIV-1 RNA associated proteome identifies Matrin 3 as a nuclear cofactor of Rev function. Retrovirology. 8, 60(2011).
- Kula, A., Marcello, A. Dynamic Post-Transcriptional Regulation of HIV-1 Gene Expression. Biology (Basel). 1 (2), 116-133 (2012).
- Yedavalli, V. S., Jeang, K. T. Rev-ing up post-transcriptional HIV-1 RNA expression. RNA Biology. 8 (2), 195-199 (2011).
- Zolotukhin, A. S., et al. PSF acts through the human immunodeficiency virus type 1 mRNA instability elements to regulate virus expression. Molecular and Cellular Biology. 23 (18), 6618-6630 (2003).
- Laird, G. M., Rosenbloom, D. I., Lai, J., Siliciano, R. F., Siliciano, J. D. Measuring the Frequency of Latent HIV-1 in Resting CD4(+) T Cells Using a Limiting Dilution Coculture Assay. Methods in Molecular Biology. 1354, 239-253 (2016).
- Cary, D. C., Peterlin, B. M. Targeting the latent reservoir to achieve functional HIV cure. F1000Res. 5, (2016).
- Han, Y., et al. Resting CD4+ T cells from human immunodeficiency virus type 1 (HIV-1)-infected individuals carry integrated HIV-1 genomes within actively transcribed host genes. Journal of Virology. 78 (12), 6122-6133 (2004).
- Laird, G. M., et al. Ex vivo analysis identifies effective HIV-1 latency - reversing drug combinations. Journal of Clinical Investigation. 125, 1901-1912 (2015).
- Lenasi, T., Contreras, X., Peterlin, B. M. Transcriptional interference antagonizes proviral gene expression to promote HIV latency. Cell Host Microbe. 4 (2), 123-133 (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon