Method Article
Hoher Durchsatz In Vitro Bewertung der Latenz Umkehrung Agenten auf HIV Transkription und Spleißen
In diesem Artikel
Zusammenfassung
Ein hoher Durchsatz-Protokoll für funktionale Bewertung von HIV effiziente Reaktivierung und Clearance von latenten Proviruses ist beschrieben und durch die Auswirkungen von Interventionen auf die Transkription von HIV-Tests und Spleißen angewendet. Repräsentative Ergebnisse des Effekts der Latenz Umkehrung Agenten auf LTR-driven Transkription und Spleißen stehen zur Verfügung.
Zusammenfassung
HIV bleibt aufgrund der Existenz eines Reservoirs von Zellen, die stabil und latente Form des Virus, birgt für das Immunsystem unsichtbar bleibt und nicht durch die aktuelle antiretrovirale Therapie (cART) unheilbar. Transkription und Spleißen wurden gezeigt, HIV-1 Latenzzeiten in ruhenden CD4 + T-Zellen zu verstärken. Umkehrung der Latenz durch den Einsatz von Latenz Umkehr Agenten (Gebietskörperschaften) in der "Shock and Kill" Ansatz wurde ausgiebig untersucht, in dem Versuch, dieses Reservoir zu bereinigen, aber keinen Erfolg in klinischen Studien wegen mangelnder Entwicklung von angemessenen kleinen bisher nicht nachgewiesen Moleküle, die effizient dieses Reservoir durcheinanderbringen können. Die hier vorgestellten Protokoll stellt eine Methode zur zuverlässig und effizient Bewertung Latenz Umkehr Agents (Gebietskörperschaften) auf HIV Transkription und Spleißen. Dieser Ansatz basiert auf der Verwendung von ein LTR-gesteuerte dual Color-Reporter, der gleichzeitig die Wirkung von einem LRA auf Transkription und Spleißen messen kann durch Durchflusszytometrie. Das hier beschriebene Protokoll ist ausreichend für adhärente Zellen als auch die Zellen in der Suspension. Es eignet sich für eine große Anzahl von Drogen in einem hohen Durchsatz-System testen. Die Methode ist technisch einfach zu implementieren und kostengünstig. Darüber hinaus ist die Verwendung der Durchflusszytometrie ermöglicht die Beurteilung der Zellviabilität und somit Drogen Toxizität zur gleichen Zeit.
Einleitung
Trotz wirksame langfristige antiretrovirale Therapie besteht HIV in einem latenten Zustand als eine integrierte Provirus im Speicher CD4 + T-Zellen-1. Das Chromatinstruktur des HIV-1-5 "lange terminal Repeat (LTR) Promoter und epigenetische Modifikationen wie Histon-Methylierung und Deacetylation durch DNA-Methyltransferasen (DNMT) und Histon Deacetylases (HDAC) sind wichtige Mechanismen, die transcriptional Repression und damit nach Integration Latenz2,3. Eine Vielzahl von Latenz Umkehr Agenten (Gebietskörperschaften) wurde untersucht, auf ihre Wirksamkeit induzieren Virus Produktion in-vitro- und in-vivo von latent infizierten ruhenden CD4 + T Zellen4,5,6,7 ,8. Unter den lokalen und regionalen Gebietskörperschaften getestet, HDACi (HDAC-Inhibitoren) und BET Bromodomain-Hemmer (BETis) induzieren Chromatin Enttauung und Freisetzung von positiven Transkription Dehnung Faktor b (P-TEFb) bzw., was zu nachfolgenden entlasten die transkriptionelle Repression auf die 5' LTR und Aktivierung von HIV Ausdruck9,10,11,12,13. Das Ausmaß der Reaktivierung durch diese Gebietskörperschaften erreicht war jedoch begrenzt, da nur ein mäßiger Anstieg der Zelle-assoziierten unspliced HIV mRNA (uns RNA), bezeichnend für virale Transkription, ex Vivo14,15beobachtet wurde. Wichtig ist, versäumt diese lokalen und regionalen Gebietskörperschaften auch, eine Verringerung der Häufigkeit von latent infizierten Zellen induzieren.
HIV-Ausdruck kann weiter durch ineffiziente Spleißen16 sowie Mängel im nuklearen Export mehrfach gespleißte HIV-RNA (MS-RNA)17beschränkt sein. Daher sind zur Identifizierung neue Klassen von lokalen und regionalen Gebietskörperschaften, die sind stärker und können Einfluss auf verschiedene Aspekte der Virus Produktion Post-Integration erforderlich. Darüber hinaus ist die Entwicklung von neuartigen Assays, die helfen, definieren die optimale Verbindungen effizient Latenz umzukehren erforderlich.
Hier ist ein Protokoll präsentiert, was nutzt einen Hochdurchsatz-Ansatz für funktionale Bewertung der Auswirkungen von Interventionen auf die HIV-LTR-driven Transkription und Spleißen. Kurz gesagt, eine neue LTR-gesteuerte Dual color Reporter System pLTR.gp140/EGFP. RevΔ38/DsRed (Abbildung 1) wird zur Durchflusszytometrie HIV Reaktivierung zu beurteilen. In dieser fluoreszierende Reporter führt die Expression des unspliced mRNA HIV (4 kb), verbesserte grün fluoreszierendes Protein (EGFP) Ausdruck, während der Begriff der gespleißten mRNA (2 kb) Discosoma SP. rote (DsRed) fluoreszierendes Protein-Expression führen würde. Kurz gesagt, haben wir eine fluoreszierende Env-EGFP Schmelzverfahren Protein, gp140unc verwendet. EGFP, wo die kodierende Sequenz des EGFP in Frame mit einer UN-gespalten und verkürzten Form des Umschlags (Env) platziert wurde. Änderungen wurden eingeführt, um die Spaltstelle verhindern die Dissoziation der Env in gp120 und gp41-EGFP abtragen und zum Abschneiden der gp160-Protein vor der transmembrane Domäne erstellen eine lösliche Env analog, das erleichtert die korrekte Faltung und Ausdruck von EGFP. Auf den Ausdruck innerhalb einer Zelle, Rev lokalisiert in den Zellkern, wo es den nuklearen zytoplasmatischen Export von 4 kb Env vermittelt, mRNA über die Interaktion mit der Rev-responsive Element (RRE). Abschneiden der Env beeinträchtigt nicht die RRE liegt zwischen gp120 und gp41 und der A7 3' Splice Site. In diesem System Spleißen bei HIV-1 splice Geber 4 (SD4) und Spleiß-Akzeptor 7 (SA7) Ergebnisse bei der Herstellung von 2 kb mRNA Kodierung ein nichtfunktionaler Rev-Protein bei Aminosäure 38 abgeschnitten mit DsRed fluoreszierendes Protein, RevΔ38-DsRed verschmolzen. Kurz gesagt, war das Exon 2Nd von Rev an Aminosäure 38 durch Überlappung Erweiterung18DsRed eingefügt. Um den nuklearen Export von unspliced mRNA zu erleichtern, wurde ein Säugetier Expressionsvektor Codierung Rev (pCMV-RevNL4.3) gemeinsam mit dem fluoreszierenden Reporter Konstrukt (Abbildung 2) transfiziert. Dieses einzigartige Reporter Konstrukt hier beschriebenen eignet sich im Hochdurchsatz-Bewertung der HIV-Übertragung und Spleißen, ohne die Notwendigkeit, virale Vektoren verwenden.
Protokoll
Hinweis: Verfahren zum Klonen, Transformation und Sequenzierung sind an anderer Stelle erläutert18,19. Die Protokolle hier beginnt die Transfektion von Säugetieren Expressionsvektoren (Abbildung 3).
1. die Transfektion von HEK293T Zellen mit Dual Color Reporter zu konstruieren
- Pflegen Sie HEK293T Zellen in Dulbeccos modifizierten Eagle Medium (DMEM) ergänzt mit 10 % (V/V) fetalen bovine Serum (FBS), Penicillin (100 U/mL) und Streptomycin (100 μg/mL) in einem 5 % CO2 Inkubator bei 37 ° C. Nach dem Auftauen Durchgang HEK293T Zellen 2-3 Mal vor der Verwendung in Transfektion experimentiert. Dies würde die Zellen Zeit wieder aus dem tauenden Verfahren geben.
Vorsicht: Mykoplasmen Kontamination von Zellkulturen bleibt ein ernstes Problem. Gute Laborpraxis und Routineuntersuchung von Zellkulturen sind wesentlich verringern das Risiko einer Kontamination der Mykoplasmen und Verbreitung20vermeiden. - Teilen Sie die Zellen, 1:2 einen Tag vor der Aussaat und in frische DMEM Medium überführen. Pflegen Sie die Zellen für 24 h in einem 5 % CO2 Inkubator bei 37 ° C.
- Am Vortag Transfektion, entfernen Sie das Medium aus der Flasche durch Absaugen und waschen Sie die Zellen mit Dulbeccos Phosphat gepuffert (DPBS) Kochsalzlösung frei von Kalzium (Ca2 +) und Magnesium (Mg2 +) um Spuren von Serum zu entfernen.
- 5 mL Trypsin-EDTA-Lösung (0,05 % - 10 mM mit PBS-Puffer) in das Kulturgefäß und legen Sie sie bei 37 ° C in einem 5 % CO2 Inkubator für 2 bis 5 min.
- Wenn die Zellen getrennt sind, fügen Sie 5 mL DMEM mit 10 % (V/V) FBS ergänzt, weiter die Trypsin-Aktivität zu hemmen, die Zellen schädigen können.
- Aufschwemmen Sie sanft durch Pipettieren der Zellsuspension rauf und runter, bis die Klumpen zu brechen.
- Verdünnen Sie die Zellen 01:10 in Trypan blau Fleck (0,4 %).
- Zählen Sie die lebenden Zellen, die keinen Trypan blau mit einem Mikroskop (10 X-Objektiv) und eine Zählkammern belegen.
- Berechnen Sie die Anzahl der lebensfähigen Zellen pro Milliliter zu, indem man den Durchschnitt der die Zellzahl und mit 10.000 multipliziert und durch den Verdünnungsfaktor (01:10) aus der Trypan blau zu färben.
- Platte 2 x 104 Zellen in 100 μL DMEM Medium ergänzt mit 10 % FBS ohne Antibiotika in einer 96-Well-Wohnung-Bodenplatte für 24 h.
- Für jedes gut verdünnen 400 ng von pLTR.gp140/EGFP. RevD38/DsRed, 20 ng pCMV-RevNL4.3 und 100 ng pCMV-Tat101AD8-Flagge DNA in 50 μL Serum freie Medium. Mischen Sie vorsichtig. Verwenden Sie für Experimente ohne Tat eine abgestimmte leer Tat-Vektor-pcDNA3.1 (-).
Hinweis: Die Verwendung von Endotoxin-freie DNA wird dringend empfohlen. Dafür bereiten Sie Endotoxin-freie DNA mit Hilfe einer Nukleinsäure Reinigung Kit MIDI- oder Maxiprep. Bestimmen Sie die DNA Reinheit durch die Messung der 260/280 AD-Verhältnis, das zwischen 1,7 und 1,9 sein sollte. - Mischen Sie der Lipid-Reagenz gemischt, dann verdünnen Sie 0,65 μL in 50 μL Serum freie Medium. Vorsichtig mischen und 5 min bei Raumtemperatur inkubieren.
- Kombinieren Sie die verdünnte DNA mit dem verdünnten Lipid-Reagenz.
- Vorsichtig mischen und 20 min bei Raumtemperatur inkubieren. Die Lösung erscheinen trüb.
Hinweis: Fahren Sie mit Schritt 1.15. innerhalb von 30 Minuten. - 100 μL pro Well der Lipid-Reagenz-DNA-komplexe tropfenweise auf die Zellen zu verzichten.
- Mischen Sie leicht von der Platte hin und her schaukeln.
-
Inkubieren Sie die Platte für 5 h in einem 5 % CO2 befeuchteten Inkubator bei 37 ° c
- Jede Reaktion Mischung reicht für einen einzigen Brunnen (96-Well) Transfection. Passen Sie die Höhe und Volumen der Komponenten entsprechend der Anzahl der Droge und Kombination, die getestet werden würde und Konten für Pipettier Variationen. Alle Bedingungen werden in der Regel in Triplicates getestet.
- Transfizieren weitere Brunnen mit 100 ng von CMV-driven EGFP und DsRed-Express DNA Plasmid zwecks Entschädigung.
2. Behandlung von transfizierten HEK293T Zellen mit Latenz Umkehrung Agenten
Hinweis: Bestimmen Sie vor jedem Test den physiologischen Zustand der jede LRA durch Messung der Lebensfähigkeit der Zellen nach der Exposition auf hohe und niedrige Dosis des Medikaments mit Cell Proliferation Assay.
- Jede LRA auf die entsprechenden Arbeiten Konzentration mit Wachstumsmedium verdünnen (z. B., 1 μM für JQ1(+)).
Vorsicht: Bereiten Sie die lyophilisierten Medikamente mit dem entsprechenden Lösungsmittel zu einer Konzentration von 10 mM. Aufbewahren Sie alle Lager Gebietskörperschaften bei-80 ° C in einer einmaligen Gebrauch aliquoten (5 μl), um wiederholte Einfrieren Auftauen Zyklen zu vermeiden. - Vorsichtig aspirieren Sie Transfektion Medium mit einem Mehrkanal- und ersetzen Sie mit 100 μl Medium mit entsprechenden LRA (Tabelle 1). Fügen Sie Medium sehr sanft entlang der Mauer des Brunnens zu vermeiden, trennen die transfizierten HEK293T Zellen, die bekannt sind, leicht zu trennen von der Kultur Plattenoberfläche.
- Inkubieren Sie die Platte für 48 h in einem 5 % CO2 befeuchteten Inkubator bei 37 ° c
3. Färbung von transfizierten Zellen mit fixierbar Lebensfähigkeit Farbstoff für Flow Cytometry Analysis
Vorsicht: Entfernen abgestorbene Zellen und Schutt ist erforderlich, um Fehlalarme zu beseitigen und Ergebnisse von höchster Qualität zu erhalten.
- Lösen Sie die Zellen in den Medien mit 100 μL der Phosphat-gepufferte Kochsalzlösung (PBS) pro Bohrloch und sanft Pipettieren wechselvollen (~ 5 Mal) mit einer Mehrkanal Aufschäumen der Medien zu vermeiden. Falls erforderlich, lösen Sie die Zellen von der Platte mit 35 µL pro Bohrloch Trypsin-EDTA-Lösung (0,05 % - 10 mM mit PBS-Puffer) und Inkubation 5 min bei 37 ° C, vor Neutralisierung mit Serum-haltigem Medium.
- Übertragen Sie die Zellen auf eine 96-Well-V-Boden-Platte.
- Drehen Sie die Zellen für 5 min bei 500 X g bei 4 ° C, dann aspirieren Sie vorsichtig Medium/PBS ohne die Zellen zu berühren.
- Waschen Sie die Zellen mindestens 1 Mal mit 200 μl Protein/Serum frei PBS.
- 500 X g für 5 min bei 4 ° C zentrifugiert, dann verwerfen des Überstands durch Kippen der Plattenrandes und der Waschpuffer ohne die Zellen zu entfernen.
- Bereiten Sie eine funktionierende Lösung des Farbstoffes fixierbar Lebensfähigkeit durch die Lebensfähigkeit Farbstoff 1: 1000 in Protein/Serum frei PBS verdünnen. Bereiten Sie 50 µL des verdünnten Flecks pro Bohrloch.
Hinweis: Lebensfähigkeit Farbstoffe sind in einer Palette von Farben geeignet für den Einsatz mit blauen, roten und violetten Lasern zur Verfügung. Bereiten Sie eine Stammlösung fixierbar Lebensfähigkeit Farbstoff durch resuspending ein Fläschchen des lyophilisierten Farbstoffs (Komponente A) mit 50 μL wasserfreiem DMSO (Komponente B). Shop bei-20 ° C in einen einzigen Verwendungszweck aliquoten (1 μl), vor Licht geschützt. - 50 μL der verdünnten Lebensfähigkeit Farbstoff in jede Vertiefung und mischen Sie die Zellen von oben und unten mit einer Mehrkanal pipettieren.
- Fleck für 10-15 min bei 4 ° C, vor Licht geschützt.
- 1-bis 2-Mal mit 150 μl Waschpuffer (PBS mit 1 % bovine Serum Albumin und 2 mM EDTA) waschen.
- Zentrifuge bei 500 X g für 5 min bei 4 ° C. Verwerfen Sie den überstand.
- Befestigen Sie die Zellen mit 100 μL der frisch zubereiteten 1 % Formaldehyd in Waschpuffer für 10-15 min bei 4 ° C im Dunkeln.
Vorsicht: Formaldehyd ist sehr giftig. Bereiten Sie 1 % Formaldehyd-Lösung in einer Dampfhaube, Einatmen beim Tragen von Handschuhen und Schutzbrille zum Schutz zu vermeiden. - Waschen Sie die Zellen 1-2 Mal mit 100 μl Waschpuffer.
- Zentrifuge bei 500 X g für 5 min bei 4 ° C. Verwerfen Sie den überstand.
- Die Zelle Pellet in 70 μl Waschpuffer aufzuwirbeln.
Hinweis: Das Protokoll kann hier angehalten werden. Fixe Zellen könnten bei 4 ° C im Dunkeln für die Analyse am nächsten Tag auf das Durchflusszytometer gespeichert werden.
4. EGFP und DsRed Messungen durch Durchflusszytometrie und Datenanalyse
Hinweis: HIV-Übertragung (% EGFP) und Spleißen (% DsRed) auf einem Durchflusszytometer analysieren. Filtern Sie die Probe vor dem Lauf mit einem 70 μm Zelle-Sieb oder 100 μm-Nylon-Mesh zu vermeiden, die Düse verstopfen.
- Starten Sie die Cytometer und Computer mindestens 10 min vor mithilfe von Lasern aufwärmen zu gewährleisten.
- Füllen Sie vor Beispiele ausführen und Beginn der Datenerfassung den Mantel Tank mit 0,9 % Kochsalzlösung und sicherstellen Sie, dass der Fäkalientank Natriumhypochlorit Tablet hinzugefügt wird.
- Überprüfen Sie die Messzelle für Luftblasen.
- Stellen Sie sicher, dass die Melder und Filter für das Experiment geeignet sind.
Hinweis: Für EGFP, Nutzung der blauen Laser (488 nm) und 530/30 Bandpass, während DsRed Express optimal erkannt wird mit dem gelben Laser (561 nm) und 610/20 Bandpass. Ein blauer Laser (488 nm) und 610/20 Bandpass kann auch verwendet werden, um DsRed Ausdruck zu erkennen. - Überprüfen Sie die Cytometer Leistung und Empfindlichkeit über Detektoren durch Kalibrierung Perlen ausführen.
- Passen Sie den vorwärts (FSC-A) und Side Scatter (SSC-A) Spannungen mit ungefärbten Probe, so dass die Bevölkerung auf dem Bildschirm ist und klar erkennbar.
Hinweis: FSC (Forward verteilt Licht) ist eine Messung des Lichts Beugung in einem flachen Winkel, die abhängig von dem Volumen der Zelle (Zelle Oberfläche oder Größe), beim SSC (Seite-Streulicht) ist eine Messung der Lichtbeugung im rechten Winkel, die proportional zur Zelle Granularität und interne Komplexität. - Führen Sie manuelle oder automatische Kompensation mithilfe der Einzel-gefärbten Proben gewährleistet minimale Spillover EGFP + Bevölkerung in die DsRed Detektor und umgekehrt aus.
Vorsicht: Verwenden Sie für Entschädigung Zwecke eine Probe von Zellen ausdrückt jede einzelne Farbe fluoreszierendes Protein und die Zellen einzeln mit dem fixierbar Lebensfähigkeit Farbstoff gefärbt. Verwenden Sie FITC und PE Entschädigung Perlen nicht für EGFP und DsRed fluoreszierende Proteine Entschädigungen aufgrund der unterschiedlichen spektralen Eigenschaften. Vergütung Kontrollen sollte hell genug, um positive und negative Bevölkerung zu lösen. - Erstellen Sie Grundstücke und legen Sie die Tore mit Fluoreszenz minus 1 (FMO) Kontrollen.
- Erwerben Sie und notieren Sie ein Minimum von 10.000 lebensfähigen Zelle Ereignisse pro Probe. Ausführen von Beispielen mit mittlerer oder niedriger Geschwindigkeit, Dubletten, die durch den Laser abfangen zu vermeiden. Verwenden Sie Puls Geometrie gating wie SSC-A vs. SSC-H, Dubletten zu beseitigen. Überprüfen Sie die Stabilität des Laufs durch Plotten Zeit gegen SSC-A um zu sehen, wie auch die Strömung ist während des Laufs.
- Am Ende des Laufs Flow Cytometry Fluidik mit konzentrierten Reinigungsmitteln zu reinigen, dann Wasser für 5 Minuten.
- Herunterfahren des Systems die Abfälle entsorgen und neu auftanken Scheide nach Erwerb.
- Analysieren Sie die Daten mit Hilfe einer Flow Cytometry Datenanalyse-Software. Zellenrückstand und Klumpen (Dubletten) basierend auf vorwärts- und seitliche Streuung ausschließen, dann beseitigen abgestorbene Zellen mit der Lebensfähigkeit Farbstoff Fleck (negative Bevölkerung = lebende Zellen). Identifizieren Sie die Zellen mit dem Ausdruck EGFP und DsRed (Abbildung 4), sowie der Prozentsatz der gespleißten Produkt DsRed /(DsRed + EGFP) (Abbildung 5).
- Bestimmen Sie die Wirkung der LRA auf HIV Transkription und Spleißen durch Vergleich von unbehandelten Zellen und den Zellen, einzelnen oder einer Kombination aus lokalen und regionalen Gebietskörperschaften (Abbildung 5).
Ergebnisse
Repräsentative Ergebnisse sind in Abbildung 5 gezeigt, die Expression von HIV-1 unspliced (EGFP) und gespleißt (DsRed) Produkte, die nach der Behandlung mit Bromodomain-Hemmer JQ1. JQ1(+) und Tat deutlich stieg der Anteil der Zellen mit dem Ausdruck EGFP (2,18 und 4.13 FC über DMSO bzw.; n = 3) bezeichnend für unspliced Transkripte. Darüber hinaus JQ1(+) deutlich stieg der Anteil der Zellen mit dem Ausdruck DsRed (46,6 FC über DMSO) sowie der Anteil der gespleißten Produkt (7,37 FC über DMSO) auf einem ähnlichen Niveau wie Tat (59,6 und 5,83 FC über DMSO, beziehungsweise) bestätigt die Fähigkeit der JQ1(+) HIV-Übertragung und Spleißen aktivieren. Auf der anderen Seite schafft Behandlung mit dem Stereoisomer Regler JQ1(-) JQ1(+) Auswirkungen auf die Transkription von HIV und Spleißen bestätigen den Erfolg des Protokolls.
In einer kürzlich erschienenen Publikation haben wir gezeigt, durch RNA-Analyse, eine Ansammlung von HIV Transkripte nach JQ1(+) Behandlung21gespleißt. In der Tat ergaben unsere Daten konsistente Änderungen in der Höhe von unspliced und gespleißt Transkripte, die durch die EGFP und DsRed Proteinexpression in diesem Modell nach der Behandlung mit JQ1(+) gespiegelt wurden. Diese Ergebnisse deuten darauf hin, dass EGFP und DsRed Ausdruck Zellen die Fähigkeit des Bromodomain-Inhibitors JQ1(+), HIV-Transkription und Spleißen induzieren widerspiegeln.
Zusammenfassend lässt sich sagen validiert wir den Einsatz von der pLTR.gp140/EGFP. RevΔ38/DsRed Reporter in einer Hochdurchsatz-Set-up für die Beurteilung der Wirkung von lokalen und regionalen Gebietskörperschaften auf HIV-1 Transkription und Spleißen. Sub-optimale Höhe der LRA oder erhöhte Toxizität könnte zu Scud Ergebnissen führen. Optimieren die Menge an Wirkstoff, die Beibehaltung der Zellviabilität verwendet kann die Genauigkeit der Ergebnisse verbessern.
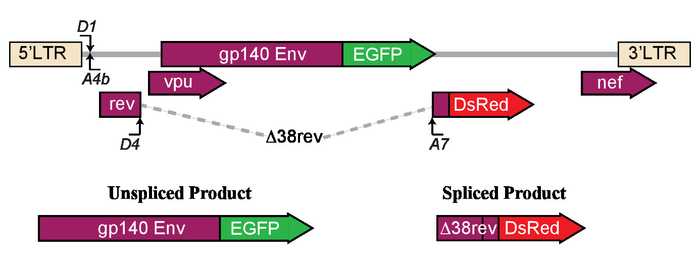
Abbildung 1: Schematische des Modells zur Ermittlung der Auswirkungen der Latenz Umkehrung Agenten auf LTR-driven Transkription und Spleißen. Der LTR Konstrukt bringt zum Ausdruck, die, denen entweder unspliced Hüllprotein (Env) an fusioniert, verstärkt grünes fluoreszierendes Protein (EGFP) oder gespleißt Δ38rev Protein mit DsRed. Diese Zahl wurde von Khoury Et Al.21nachgedruckt. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 2: Design konstruiert. (A) pLTR.gp140/EGFP. RevΔ38/DsRed Spleißen Reporter ermöglicht der Ausdruck des Umschlags verschmolzen EGFP (gp140-EGFP) und nicht-funktionalen Rev-Protein an Aminosäure 38 verschmolzen zu DsRed fluoreszierenden Proteins (RevΔ38-DsRed) abgeschnitten. (B) pCMV-RevNL4.3 erlaubt die Expression von Rev-Protein. (C) pCMV-Tat101AD8-Flagge erlaubt die Expression von Tat101(AD8) Protein mit einer C-terminalen Flag-Tag. Der Ausdruck Plasmide werden mittels PCR Überschneidungen und Beschränkungsauswahl konstruiert. Alle Vektorkarten wurden mit SnapGene -Software, Version 4.1.9 erstellt. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 3: Assay-Design für schnelle Einschätzung der HIV Latenz Umkehrung Agenten. Die aktuelle Methode für die Bewertung der Wirkung von lokalen und regionalen Gebietskörperschaften auf HIV-1 Transkription und Spleißen enthält i) Aussaat von HEK293T Zellen einen Tag vor Ii) Transfektion mit Ausdrücke Vektoren, gefolgt von der LRA Iii) Behandlung. (IV) Zellen werden von Flow Cytometry 48 h nach der Behandlung analysiert. Unter Verwendung dieses Protokolls, konnten pro Platte 32 (in dreifacher Ausfertigung) bis 96 Bedingungen getestet werden. Bitte klicken Sie hier für eine größere Version dieser Figur.
Tabelle 1: repräsentatives Beispiel für die Platte auf- und Kontrollen erforderlich. Spalten 1 bis 3 entsprechen Negative (-) Kontrollen mit kein Medikament und Lösungsmittel nur (DMSO 1: 5.000) sowie Positive (+) Steuerelemente wie Tat (100 ng), PMA/PHA = Phorbol Myristate Acetat/Phytohaemagglutinin (10 nM PMA, 10 μg/mL PHA), JQ1 (+/-) (1 μM), VOR = Vorinostat ( 0,5 μM), und PAN = Panobinostat (30 nM). Unbekannte lokalen und regionalen Gebietskörperschaften sind in dreifacher Ausfertigung über einen Bereich von Konzentrationen (15,625 bis 1000 nM) getestet. Zwecken der Entschädigung Zellen ausdrückt jede einfarbige fluoreszierenden Proteins (EGFP + und DsRed +) sowie Zellen mit der Lebensfähigkeit Farbstoff befleckt und ungefärbte Zellen sind in jedem Lauf enthalten. Bitte klicken Sie hier, um diese Datei herunterladen.

Abbildung 4: Strategie in der Flow-Zytometrie-Analyse verwendet Gating. Der erste Schritt in der gating-Strategie basiert auf der vorderen und seitlichen streuen, wodurch die Unterscheidung der Zellen von Interesse, die anhand der Größe und Granularität Eigenschaften dieser Zellen. Es wird empfohlen, dass diese gating werden so großzügig wie möglich, jedenfalls beim Entfernen Zelltrümmer und abgestorbenen Zellen, die in der unteren linken Ecke der Dichte Handlung zu finden sind. Dann entfernen Sie Dubletten aus dem Dataset mit der Puls-Geometrie gating, SSC-A vs. SSC-H und weitere schmale auf die lebenden Zellen mit der Lebensfähigkeit Farbstoff. Zu guter Letzt ein Dump Kanal (violett) und EGFP oder DsRed dienen zum definieren, Zellen von Interesse. Die Verwendung von Fluoreszenz minus 1 (FMO) Steuerelemente sind entscheidend bei der Festlegung der Bevölkerung von Interesse und die Fragen der Spillover aus einem anderen Fluorochrom im Kanal von Interesse. Nach dem Erwerb sind Daten mit Flow Cytometry Datenanalyse-Software analysiert. Diese Zahl hat in adaptierter Form Khoury Et Al.21abgedruckt. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 5: repräsentative Ergebnisse der Wirkung von Bromodomain-Inhibitoren auf HIV Transkription und Spleißen. (A) Beispiele für zwei-Parameter Dual color Fluoreszenz EGFP versus DsRed Dichte aus untransfected Zellen Grundstücke (-Tat), transfizierten Zellen mit 100 ng pCMV-Tat101AD8-Flagge (+ Tat) und Zellen mit DMSO, JQ1 behandelt (+) oder JQ1 (-). (B) die mittlere Prozentsatz (%) der Zellen, die mit dem Ausdruck EGFP, DsRed oder gespleißt Produkt (DsRed / DsRed + EGFP) aus 3 unabhängigen Experimenten gezeigt. Jede Bedingung, DMSO wurden Vergleiche mit dem 2-Wege-ANOVA-Test. Statistisch signifikante Vergleiche werden nur angezeigt. p < 0,05; p < 0,01; p < 0,001. Die schwarzen Linien stellen die Mean±SEM. DMSO (1: 5.000), JQ1 (+) und JQ1(-) (1 μM). Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Angesichts der Schwierigkeiten bei der Messung der Virus-Reaktivierung ex Vivo, infiziert eine Vielzahl von in-vitro-Modelle im Laufe der Zeit entwickelt wurden, um HIV-Latenz einschließlich latent studieren T-Zell-Linien (J-Lats, ACH2, U1), primäre Modelle der latenten Infektion ruhen ( O' Doherty, Lewin, Greene und Spina-Modelle) oder pre-activated CD4 + T-Zellen (Sahu, Marini, Planelles, Siliciano, Karn Modelle) mit einzelnen Runde oder Replikation zuständigen Reporter Viren22. Um die physiologischen Bedingungen der Latenz von HIV in ruhenden CD4 + T-Zellen zu modellieren, folgte dual fluoreszierende Reporter Systeme wie der RGH (rot-grün-HIV) Reporter von Ivan Sadowskis Gruppe mit einer CMV-driven mCherry im Ort und eine LTR-driven Gag-eGFP Marker entwickelt der Nef-23. Eine ähnliche dual Farbe Reporter Virus, Duo-Fluo, wurde von E. Verdin Labor mit LTR-driven EGFP Ausdruck und eine mCherry fluoreszierenden Marker angetrieben durch eine EF1α-Promotor-24entwickelt. In diesem System wurde die EGFP am 3' Ende des Provirus anstelle von Nef eingefügt. Die Löschung in den Umschlag in beide Systeme verhindert, dass mehrere Runden von Infektionen. Darüber hinaus ermöglicht die Kombination der beiden fluoreszierende Proteine die Erkennung von produktiv (eGFP + mCherry +) und latent infizierten (eGFP - mCherry +) Zellen. Jedoch waren einige Mängel der dual Farbe Reporter Viren in CD4 + T-Zellen bemerkbar, da die direkte Infektion latent infizierten Zellen weitgehend abhängig von der zellulären Aktivierungsstatus der T-Zellen23,24 ist. In der Tat sehr niedrige Rate von Infektion war mit dieser zwei Reporter Viren in ruhenden CD4 + T-Zellen beobachtet: 0,1 % für RGH23 und 0,2 % für die DuoFluo25. Darüber hinaus als die CMV Promoter hat gezeigt, dass auf den unteren Ebenen nicht aktivierte Zellen26,27,28, eine inaktive CMV ausgedrückt werden oder EF1α Veranstalter würde zu einer Unterschätzung der latent infizierten führen Populationen.
Wir haben generiert und zeichnet sich einen neuen HIV-Reporter-Vektor um die Einrichtung von Latenz und Virus-Reaktivierung in einem hohen Durchsatz-Assay zu studieren. Unsere Screening-Methode hat mehrere Vorteile umfassen i) Preis-/Leistungsverhältnis mit der Fähigkeit, Transkription und Spleißen gleichzeitig, (Ii) die Möglichkeit, eine große Anzahl von Drogen oder Kombinationen in einem einzigen Experiment, Iii) schnelle Beurteilung der testen bewerten die Wirkung der lokalen und regionalen Gebietskörperschaften durch Durchflusszytometrie (30-45 min typische Ablesezeit für 96-well-Platte unabhängig von der Anzahl der fluoreszierenden Parameter), iv) hoher Dynamikbereich, V) geeignet für hohen Durchsatz Transfektion und Durchflusszytometrie mit Roboterarmen und HTS-Plattform automatisiert (VI) schnell aushärten der Daten mithilfe einer Flow Cytometry Workspace-Vorlage, Vii) optimal für Fahrwerk und adhärente Zellen, Viii) Einfachheit des Modells und der Anpassungsfähigkeit an Leuchtkraft und Platte Leser-Messungen (dual Luciferase Firefly und Renilla) und Ix) Skalierbarkeit (96 und 384-Well-Pastete-Formate).
Die Neigung der ein LRA oder eine Kombination aus lokalen und regionalen Gebietskörperschaften, HIV-LTR-driven Transkription und Spleißen somit EGFP und DsRed fluoreszierende Proteine Ausdruck aktivieren könnte durch Durchflusszytometrie mit unserer HIV Spleißen Reporter geprüft werden. Jedoch bevor Sie testen die Synergie des Romans lokalen und regionalen Gebietskörperschaften, empfiehlt es die physiologischen Bedingungen der einzelnen LRA durch Messung die Lebensfähigkeit der Zellen nach der Exposition zu einer Reihe von Konzentrationen der einzelnen Droge zu bestimmen. Daher ist es wichtig, um Fehlalarme zu beseitigen und Ergebnisse von höchster Qualität zu erhalten, einschließlich eine Markierung von lebenden und toten Zellen. Ein weiterer wichtiger Aspekt der Flow-Zytometrie-Analyse ist die Entschädigung Steuerelemente oder FMO. Es wird dringend empfohlen, einzeln gefärbten Proben gewährleistet minimale Spillover EGFP + Bevölkerung in die DsRed und umgekehrt zu verwenden. Darüber hinaus muss auf Konto Autofluoreszenz der Zellen durch die Einbeziehung ungefärbten Zellen. Schließlich ist eine regelmäßige Kontrolle der Cytometer Leistung und Empfindlichkeit über die Detektoren entscheidend vor allem bei der Messung Proben für eine Studie über einen langen Zeitraum hinweg.
Obwohl nicht im Protokoll Abschnitt erläutert, gibt es mehrere Einschränkungen unseres Spleißen Reporter-Systems. Eine tiefer gehende Diskussion entnehmen Sie bitte unserer früheren Publikation (Khoury Et Al., 2018). Export von den teilweise oder unspliced Varianten von HIV mRNA wird durch das virale Protein Rev und seine Verbindung mit Rev Responsive Element (RRE) in diese mRNA Arten29,30,31,32erleichtert. Die Überexpression von Rev in unserem System erlaubt uns, den großen Block der nuklearen Eigentumsvorbehalt multiplizieren zu umgehen gespleißt und unspliced mRNAs beobachtet in latent infizierten T Zellen17 und konzentrieren sich auf das Verständnis, wie die lokalen und regionalen Gebietskörperschaften induzieren Transkription und Spleißen so Virus-Reaktivierung. Darüber hinaus angesichts der Tatsache, dass unser System die Transfektion von 3 Plasmide erfordert, wir entschieden uns für HEK293T Zellen, weil die hohen Transfection Leistungsfähigkeit dieser Zellen. Aufgrund der verschiedenen zeitlichen und räumlichen Verfügbarkeit von zellulären Faktoren in T-Zellen im Vergleich zu einem Krebs-Zell-Linie, ist es wichtig, überprüfen Sie die Ergebnisse der Spleißen Reporter erwarb HEK293T Zellen in T-Zell-Linien und Primärzellen, die unter Umständen große aufweisen technische Herausforderungen aufgrund ihrer begrenzten Transfectability. Daher ist die Verwendung von alternativen DNA Lieferung Reagenzien für die Transfektion wie DMRIE-C, das gezeigt worden ist, besonders effektiv für die Transfektion von Aussetzung Zellen (z. B. Jurkat) oder Nucleofection Methoden wie Amaxa Nucleofector oder Neon Transfektion System, das nachweislich auf um effiziente und zuverlässige Lieferung von einzelnen oder mehreren Plasmide schwierig zu transfizieren Zellen einschließlich der primären CD4 + T-Zellen zu erleichtern. Alternativ wäre es interessant, diese Reporter-System im Rahmen des Full-length-Virus in einer Primärzelle Modell der Latenz, die mit den vorgenannten Transfektion Methoden zu testen. In der Tat beeinträchtigen die Unterschiede in der Verfügbarkeit von Host-Transkription, Dehnung und Spleißen Faktoren in einer rCD4 + T-Zelle die Fähigkeit der eine LRA, die latente Provirus zu reaktivieren. Schließlich befasst sich unser Modell nicht die LRA Transkription und Spleißen der Bystander Zellen beeinflussen kann und ob es Replikation zuständigen Virus hervorrufen kann. Allerdings würden wir vermuten, dass durch die Messung einer Zunahme Zelle verbunden U.S. HIV und MS RNA, das zur Produktion von Replikation zuständigen Virus in der Kultur überstand führen würde. Dies konnte durch die Durchführung der Goldstandard quantitativen Virus Auswuchs Assay (QVOA)33bestätigt werden.
Aufgrund der komplexen Natur der Latenz und effizienten Viren Reaktivierung sicherzustellen kann eine vielseitige kombinatorische Strategie erforderlich34sein. Mögliche Behandlung muss mehrere Wege einschließlich Transkription und Spleißen, stimulieren die zuvor nachweislich die wesentliche Rolle bei der Festlegung der Latenz16,35,36, 37. unser LTR-driven Spleißen Reporter würde eine Hochdurchsatz-Screening von Medikamenten, die Schritte, Transkription und Spleißen optimal ausrichten können ermöglichen, erhöhen die Chance der Feststellung Moleküle, die effizient, synergize kann zu einem niedrigeren führen würde Dosierungen und somit Toxizität jedes Medikament.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Diese Arbeit wurde von Projekt Grant APP1129320 und Programm APP1052979 Stipendium der NHMRC Australia unterstützt. Wir danken Dr. Adam Wheatley, Dr. Marina Alexander, Dr. Jenny L. Anderson und Michelle Y. Lee für die wesentlichen Konstrukte und Beratung für den erfolgreichen Abschluss dieser Arbeit. Wir danken auch das DMI-Flow-Anlage Personal für ihre Ratschläge und großzügige Unterstützung bei der Aufrechterhaltung der in dieser Studie verwendeten Durchflusszytometer.
Materialien
| Name | Company | Catalog Number | Comments |
| Cell culture | |||
| HEK293T cells (Human Embryonic Kidney cells) | ATCC | CRL-3216 | Replicates vectors carrying the SV40 region of replication. |
| Dulbecco's Modified Eagle's Medium (DMEM 1x + GlutaMAX-I) | Gibco | 10569-010 | + 4.5 g/L D-Glucose + 110 mg/L Sodium Pyruvate |
| Fetal Bovine serum | Gibco | 10099-141 | Origin Australia |
| Penicillin-Streptomycin | Sigma | P4458 | |
| Dulbecco's phosphate buffered saline (DPBS), no calcium, no magnesium | Gibco | 14190-136 | |
| Trypan blue Stain, 0.4% | Gibco | 15250 | |
| Trypsin-EDTA (0.05%), phenol red | Gibco | 25300054 | |
| Lipofectamine 2000 | Invitrogen | 11668-019 | Lipid transfection reagent |
| Opti-MEM I (1x) reduced serum medium | Gibco | 31985-070 | Serum free medium |
| NucleoBond Xtra Maxi | Marcherey-Nagel | 740414.50 | |
| pEGFP-N1 plasmid | Clontech (TaKaRa) | 6085-1 | Expression of EGFP in mammalian cells, CMVIE promoter. |
| pDsRed-Express-N1 | Clontech (TaKaRa) | 632429 | Expression of DsRed-Express in mammalian cells, CMVIE promoter. |
| pLTR.gp140/EGFP.RevD38/DsRed | Addgene | 115775 | |
| pCMV-RevNL4.3 | Addgene | 115776 | |
| pCMV-Tat101AD8-Flag | Addgene | 115777 | |
| Dimethyl sulfoxide (DMSO) | Millipore | 67-68-5 | |
| JQ1(+) | Cayman Chemical | 11187 | Stock at 10 mM in DMSO; working concentration 1 μM |
| JQ1(-) | Cayman Chemical | 11232 | Stock at 10 mM in DMSO; working concentration 1 μM |
| Phorbol Myristate Acetate (PMA) | Sigma-Aldrich | 16561-29-8 | Stock at 100 μg/mL in DMSO; working concentration 10 nM |
| Phytohaemagglutinin (PHA) | Remel | HA15/R30852701 | Stock at 1 μg/μL in PBS; working concentration 10 μg/mL |
| Vorinostat (VOR) | Cayman Chemical | 10009929 | Stock at 10 mM in DMSO; working concentration 0.5 μM |
| Panobinostat (PAN) | TRC | P180500 | Stock at 10 mM in DMSO; working concentration 30 nM |
| CellTiter 96 AQueous One Solution Cell Proliferation Assay | Promega | 63581 | |
| Venor GeM Classic | Minerva Biolabs | 11-1100 | Mycoplasma Detection Kit, PCR-based |
| Name | Company | Catalog Number | Comments |
| Flow cytometry reagents | |||
| LSR Fortessa | BD Biosciences | Flow cytometer (4 lasers-blue, red, violet and yellow) | |
| LSR II | BD Biosciences | Flow cytometer (3 lasers-blue, red and violet) | |
| LIVE/DEAD Fixable Near-IR Dead Cell Stain Kit | Life Technologies | L34976 | Viability dye: for 633 or 635 nm excitation, 400 assays. Component A and B are both provided in the kit. |
| Bovine Serum Albumin | Sigma | A2153 | |
| EDTA 0.5M pH8 | Gibco | 15575-038 | |
| Formaldehyde Solution 37/10 (37%) | Chem-Supply | FA010 | |
| BD FACS Diva CS&T Research Beads | BD Biosciences | 655050 | Calibration beads |
| Sphero Rainbow Calibration Particles (8 peaks) | BD Biosciences | 559123 | 3.0 - 3.4 mm |
| Sheath solution | Chem-Supply | SA046 | 90 g NaCl in 10 L water |
| HAZ-Tabs | Guest Medical | H8801 | Chlorine release tablets for disinfection |
| Decon 90 | Decon Laboratories Limited | N/A | Concentrated cleaning agents of flow cytometer. Working solution Decon 90 5%. |
| Sodium Hypochlorite (12-13% Solution) | Labco | SODHYPO-5L | Concentrated cleaning agents of flow cytometer. Working solution bleach 1%. |
| 7x | MPBio | IM76670 | Concentrated cleaning agents of flow cytometer. Working solution 7x 1%. |
| Name | Company | Catalog Number | Comments |
| Materials | |||
| Tissue culture flasks (75 cm2, canted neck, cap vented) | Corning | 430641U | |
| Tissue culture plates (96 well flat bottom with lid) | Costar | 3599 | |
| Tissue culture plates (96 well V-bottom without lid) | Costar | 3896 | |
| Centrifuge tubes (10 mL) | SARSTEDT | 62.9924.284 | 100x16 mm |
| Centrifuge tubes (50 mL) | CellStar | 227261 | 30x115 mm |
| Microcentrifuge tubes (1.5 mL) | Corning Axygen | MCT-150-C | |
| Serological Pipette (25 mL), sterile | Corning | CLS4489-200EA | |
| Serological Pipette (10 mL), sterile | Corning | CLS4488-200EA | |
| Serological Pipette (5 mL), sterile | Corning | CLS4487-200EA | |
| Reagent reservoirs (50 mL), sterile | Corning | CLS4470-200EA | |
| 5 mL Round-Bottom polystyrene test tube, with cell-strainer cap | Corning | 352235 | 12 x 75 mm style, 70 mm |
| Nylon Mesh | SEFAR | 03-100/32 | 100 mm |
| Titertube Micro test tubes, bulk | BIO-RAD | 2239391 | microfacs tubes |
| 5 mL Round-Bottom polystyrene test tube, without cap | Corning | 352008 | 12x75 mm style |
| Snap Caps for 12x75 mm Test Tubes | Corning | 352032 | |
| Counting chamber, Neubauer improved double net ruling, bright-line (Haemocytometer, LO-Laboroptik) | ProSciTech | SVZ4NIOU | 3x3 large squares of 1 mm2; Depth 0.100 mm; volume 0.1 mL; area minimum 0.0025 mm2 |
| Coverslips (Menzel-Gläser) | Grale Scientific | HCS2026 | 20 x 26 mm |
| Microscope | Nikon TMS | 310528 | |
| Centrifuge 5810R refrigerated | Eppendorf | 5811000487 | with rotor A-4-81 including adapters for 15/50 mL conical tubes |
| FLUOstar Omega microplate reader | BMG Labtech | N/A | Plate reader for cell proliferation assay. Filter 490 nm. |
| Name | Company | Catalog Number | Comments |
| Softwares | |||
| FACS Diva | BD Biosciences | Flow cytometer data acquisition and analysis program, version 8.0.1 | |
| FlowJo | FlowJo | FlowJo 10.4.2 | Flow cytometer data analysis program, FlowJo Engine v3.05481 |
| Omega | BMG Labtech | FLUOstar multi-user reader control, version 5.11 | |
| Omega - Data Analysis | BMG Labtech | MARS | FLUOstar data analysis, version 3.20R2 |
| Microsoft Excel | Microsoft | Excel:mac 2011 | version 14.0.0 |
| Prism | GraphPad | Prism 7 | version 7.0c |
Referenzen
- Siliciano, J. D., et al. Long-term follow-up studies confirm the stability of the latent reservoir for HIV-1 in resting CD4+ T cells. Nature Medicine. 9 (6), 727-728 (2003).
- Khoury, G., et al. Ch. 8. HIV vaccine and cure - The Path Towards Finding an Effective Cure and Vaccine. 1075, Springer Nature. Adv Exp Med Biol (2018).
- Van Lint, C., Bouchat, S., Marcello, A. HIV-1 transcription and latency: an update. Retrovirology. 10, 67(2013).
- Archin, N. M., et al. HIV-1 Expression Within Resting CD4+ T Cells After Multiple Doses of Vorinostat. Journal of Infectious Diseases. 210, 728-735 (2014).
- Elliott, J. H., et al. Activation of HIV Transcription with Short-Course Vorinostat in HIV-Infected Patients on Suppressive Antiretroviral Therapy. PLoS Pathogens. 10, (2014).
- Leth, S., et al. Combined effect of Vacc-4x, recombinant human granulocyte macrophage colony-stimulating factor vaccination, and romidepsin on the HIV-1 reservoir (REDUC): a single-arm, phase 1B/2A trial. The Lancet HIV. 3, e463-e472 (2016).
- Rasmussen, T. A., et al. Panobinostat, a histone deacetylase inhibitor, for latent-virus reactivation in HIV-infected patients on suppressive antiretroviral therapy: a phase 1/2, single group, clinical trial. The Lancet HIV. 1, e13-e21 (2014).
- Søgaard, O. S., et al. The Depsipeptide Romidepsin Reverses HIV-1 Latency In Vivo. PLoS Pathogens. 11, (2015).
- Bartholomeeusen, K., Xiang, Y., Fujinaga, K., Peterlin, B. M. Bromodomain and extra-terminal (BET) bromodomain inhibition activate transcription via transient release of Positive Transcription Elongation Factor b (P-TEFb) from 7SK small nuclear ribonucleoprotein. Journal of Biological Chemistry. 287, 36609-36616 (2012).
- Boehm, D., et al. BET bromodomain-targeting compounds reactivate HIV from latency via a Tat-independent mechanism. Cell Cycle. 12, 452-462 (2013).
- Contreras, X., et al. Suberoylanilide hydroxamic acid reactivates HIV from latently infected cells. Journal of Biological Chemistry. 284 (11), 6782-6789 (2009).
- Rasmussen, T. A., et al. Comparison of HDAC inhibitors in clinical development: effect on HIV production in latently infected cells and T-cell activation. Human Vaccines & Immunotherapeutics. 9 (5), 993-1001 (2013).
- Wei, D. G., et al. Histone deacetylase inhibitor romidepsin induces HIV expression in CD4 T cells from patients on suppressive antiretroviral therapy at concentrations achieved by clinical dosing. PLoS Pathogens. 10 (4), e1004071(2014).
- Blazkova, J., et al. Effect of histone deacetylase inhibitors on HIV production in latently infected, resting CD4(+) T cells from infected individuals receiving effective antiretroviral therapy. Journal of Infectious Diseases. 206 (5), 765-769 (2012).
- Bullen, C. K., Laird, G. M., Durand, C. M., Siliciano, J. D., Siliciano, R. F. New ex vivo approaches distinguish effective and ineffective single agents for reversing HIV-1 latency in vivo. Nature Medicine. 20 (4), 425-429 (2014).
- Yukl, S. A., et al. HIV latency in isolated patient CD4+T cells may be due to blocks in HIV transcriptional elongation, completion, and splicing. Science Translational Medicine. 10, (2018).
- Lassen, K. G., Ramyar, K. X., Bailey, J. R., Zhou, Y., Siliciano, R. F. Nuclear retention of multiply spliced HIV-1 RNA in resting CD4+T cells. PLoS Pathogens. 2, 0650-0661 (2006).
- Alexander, M. R., Wheatley, A. K., Center, R. J., Purcell, D. F. J. Efficient transcription through an intron requires the binding of an Sm-type U1 snRNP with intact stem loop II to the splice donor. Nucleic Acids Research. 38, 3041-3053 (2010).
- Anderson, J. L., Johnson, A. T., Howard, J. L., Purcell, D. F. J. Both Linear and Discontinuous Ribosome Scanning Are Used for Translation Initiation from Bicistronic Human Immunodeficiency Virus Type 1 env mRNAs. Journal of Virology. 81, 4664-4676 (2007).
- Nikfarjam, L., Farzaneh, P. Prevention and detection of Mycoplasma contamination in cell culture. Cell J. 13 (4), 203-212 (2012).
- Khoury, G., et al. HIV latency reversing agents act through Tat post translational modifications. Retrovirology. 15 (1), 36(2018).
- Hakre, S., Chavez, L., Shirakawa, K., Verdin, E. HIV latency: experimental systems and molecular models. FEMS Microbiology Reviews. 36 (3), 706-716 (2012).
- Dahabieh, M. S., et al. Direct non-productive HIV-1 infection in a T-cell line is driven by cellular activation state and NFkappaB. Retrovirology. 11, 17(2014).
- Calvanese, V., Chavez, L., Laurent, T., Ding, S., Verdin, E. Dual-color HIV reporters trace a population of latently infected cells and enable their purification. Virology. 446 (1-2), 283-292 (2013).
- Chavez, L., Calvanese, V., Verdin, E. HIV Latency Is Established Directly and Early in Both Resting and Activated Primary CD4 T Cells. PLoS Pathogens. 11 (6), e1004955(2015).
- Hunninghake, G. W., Monick, M. M., Liu, B., Stinski, M. F. The promoter-regulatory region of the major immediate-early gene of human cytomegalovirus responds to T-lymphocyte stimulation and contains functional cyclic AMP-response elements. Journal of Virology. 63 (7), 3026-3033 (1989).
- Reeves, M., Sinclair, J. Aspects of human cytomegalovirus latency and reactivation. Current Topics in Microbiology and Immunology. 325, 297-313 (2008).
- Sambucetti, L. C., Cherrington, J. M., Wilkinson, G. W., Mocarski, E. S. NF-kappa B activation of the cytomegalovirus enhancer is mediated by a viral transactivator and by T cell stimulation. EMBO Journal. 8 (13), 4251-4258 (1989).
- Kula, A., et al. Characterization of the HIV-1 RNA associated proteome identifies Matrin 3 as a nuclear cofactor of Rev function. Retrovirology. 8, 60(2011).
- Kula, A., Marcello, A. Dynamic Post-Transcriptional Regulation of HIV-1 Gene Expression. Biology (Basel). 1 (2), 116-133 (2012).
- Yedavalli, V. S., Jeang, K. T. Rev-ing up post-transcriptional HIV-1 RNA expression. RNA Biology. 8 (2), 195-199 (2011).
- Zolotukhin, A. S., et al. PSF acts through the human immunodeficiency virus type 1 mRNA instability elements to regulate virus expression. Molecular and Cellular Biology. 23 (18), 6618-6630 (2003).
- Laird, G. M., Rosenbloom, D. I., Lai, J., Siliciano, R. F., Siliciano, J. D. Measuring the Frequency of Latent HIV-1 in Resting CD4(+) T Cells Using a Limiting Dilution Coculture Assay. Methods in Molecular Biology. 1354, 239-253 (2016).
- Cary, D. C., Peterlin, B. M. Targeting the latent reservoir to achieve functional HIV cure. F1000Res. 5, (2016).
- Han, Y., et al. Resting CD4+ T cells from human immunodeficiency virus type 1 (HIV-1)-infected individuals carry integrated HIV-1 genomes within actively transcribed host genes. Journal of Virology. 78 (12), 6122-6133 (2004).
- Laird, G. M., et al. Ex vivo analysis identifies effective HIV-1 latency - reversing drug combinations. Journal of Clinical Investigation. 125, 1901-1912 (2015).
- Lenasi, T., Contreras, X., Peterlin, B. M. Transcriptional interference antagonizes proviral gene expression to promote HIV latency. Cell Host Microbe. 4 (2), 123-133 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten