Method Article
Una piattaforma di High-throughput per lo Screening della Salmonella spp /Shigella spp.
In questo articolo
Riepilogo
Salmonella spp /Shigella spp sono agenti patogeni comuni attribuiti alla diarrea. Qui, descriviamo una piattaforma di alto-rendimento per lo screening della Salmonella spp /Shigella spp usando la PCR in tempo reale combinati con cultura guidato.
Abstract
Trasmissione feco-orale di gastroenterite acuta si verifica di tanto in tanto, soprattutto quando le persone che hanno gestito il cibo e l'acqua sono infetti da Salmonella spp./Shigella spp. Il metodo gold standard per la rilevazione della Salmonella spp./Shigella spp è coltura diretta ma questo è laborioso e che richiede tempo. Qui, descriviamo una piattaforma di alto-rendimento per Salmonella spp./Shigella spp., screening, usando reazione a catena della polimerasi in tempo reale (PCR) combinata con cultura guidata. Ci sono due fasi principali: PCR in tempo reale e la cultura guidata. Per la prima fase (Real-Time PCR), spieghiamo ogni passaggio del metodo: esempio di collezione, pre-arricchimento, estrazione del DNA e PCR in tempo reale. Se il risultato in tempo reale di PCR è positivo, allora viene eseguita la seconda fase (guidata cultura): coltura selettiva, identificazione biochimica e sierologica caratterizzazione. Illustriamo anche risultati rappresentativi generati da esso. Il protocollo descritto qui sarebbe una valida piattaforma per lo screening rapido, specifico, sensibile ed alta-velocità di trasmissione di Salmonella spp /Shigella spp.
Introduzione
La diarrea è ancora un problema di salute comune con un'alta incidenza globalmente tasso1,2. Anche se la mortalità è relativamente bassa, alcuni pazienti mostrano vari sintomi per settimane (come sgabelli allentati e acquosi, l'urgenza di andare in bagno), che rendono l'impatto socio-economico molto alto3,4. Più seriamente, alcuni pazienti possono anche sviluppare sindrome dell'intestino irritabile se lasciato non trattato5. Ci sono vari tipi di batteri, virus e parassiti che possono causare diarrea6. Salmonella spp /Shigella spp sono tra i più comuni batteri per la trasmissione di gastroenterite acuta7,8,9,10,11. Di conseguenza, molte contee hanno emesso leggi o regolamenti per regolare Salmonella spp /Shigella spp screening fra la gente che dovrebbe gestire cibo e acqua. Ad esempio, il governo cinese hanno emesso leggi obbligatorie Salmonella spp /Shigella spp screening una volta all'anno.
Il metodo gold standard per Salmonella spp /Shigella spp. rilevamento è coltura dei batteri. Attraverso la coltura dei batteri e successiva identificazione biochimica e sierologica caratterizzazione, possiamo identificare la specie di batteri, che potrebbero facilitare la gestione del focolaio di malattia e profilatura antimicrobica per facilitare il trattamento dei pazienti 12. potrebbe anche aiutare a rintracciare la fonte di infezione durante la Salmonella spp /Shigella spp. scoppio13. Tuttavia, questo metodo è laborioso (che richiedono funzionamento manuale) e che richiede tempo (prendendo diversi giorni), soprattutto per il collaudo di un gran numero di esempi7. Inoltre, vitali ma non coltivabili (VBNC) Salmonella spp /Shigella spp.,possono esistere in alcuni campioni di feci14. In considerazione di questi inconvenienti, molti laboratori hanno cercato di sviluppare nuove tecniche per la rilevazione della Salmonella spp /Shigella spp.15,16,17,18 , 19 , 20 , 21 , 22 , 23 , 24 , 25. tutti questi metodi utilizzano il test nucleare acido amplificazione (NAAT), tra i quali la reazione a catena della polimerasi (PCR) è il più comune. Uno dei principali limiti di questi metodi NAAT basata è che i batteri morti, anche batteriche detriti contenenti DNA genomic incompleta, potrebbero mostrare risultati positivi26, che in gran parte potrebbe influenzare la diagnosi accurata della malattia. Blanco et al hanno mostrato che quel saggio molecolare è altamente sensibile, non solo praticabile Salmonella nelle culture, ma anche di parziali genomi e batteri morti o impraticabili dal passato infezioni o contaminazioni26. Di conseguenza, la nuova tecnologia dovrebbe essere sviluppata.
Qui, abbiamo descritto un nuovo metodo che combina il NAAT basato su metodo e coltura. Come illustrato nella Figura 1, questo nuovo metodo si applica in tempo reale PCR prima di screening e quindi vengono inviati campioni positivi per l'identificazione e la coltura dei batteri.
Protocollo
Il protocollo segue le linee guida del comitato etico di ricerca umana di Zhuhai International Travel Healthcare Center. Si prega di utilizzare standard di funzionamento sterile durante l'esperimento.
1. preparazione e composizione di terreni di coltura
- Preparare il brodo nutritivo: Sciogliere 1% peptone, 0,3% di Estratto di carne di manzo, 0,5% cloruro di sodio, 0,1% di glucosio in H2O, regolare il pH a 7,5 e autoclave a 121 ° C per 15 min.
- Preparare il supporto di Selenite cistina: sciogliere 0,5% peptone, lattosio 0,4%, 1% Na2HCO3, selenito di 0,4% sodio idrogeno, 0.001% L-cistina in H2O, regolare il pH a 7.0 e portare a ebollizione per 5 min.
- Preparare il xilosio, lisina, desossicolato piastra di agar (XLD): Estratto di lievito di sciogliere 0,3%, 0,5% L-lisina, xilosio 0.375%, 0,75% lattosio, 0,75% saccarosio, 0,5% cloruro di sodio, 0,008% di rosso fenolo, 0,68% sodio tiosolfato, 0,08% citrato ferrico di ammonio, sodio 0,25% desossicolato, agar 1,5% in H2O e regolare il pH a 7,4. Bollire per 5 minuti, quindi versarlo in piastre da 90 mm.
- Preparare la piastra di agar cromogenico Salmonella : sciogliere 1,5% agar, 0,7% peptone e lievito estratto, il reagente selettiva 1,29% in H2O. Bollire per 5 minuti, quindi versarlo in piastre da 90 mm.
- Preparare la piastra di agar nutriente: sciogliere 1% peptone, 0,3% di Estratto di carne di manzo, cloruro di sodio 0,5%, 1,5% agar in H2O e regolare il pH a 7,3. Quindi autoclave a 121 ° C per 15 min e versarlo in piastre da 90 mm.
- Preparare la piastra di MacConkey agar (MAC): sciogliere 2% peptone, 1% lattosio, 0,5% cloruro di sodio, 0.5% sale di Bile di bue, 0,0025% Neutral Red, 1,5% agar, viola di cristallo di 0,0001% in H2O e regolare il pH a 7,2. Quindi autoclave a 115 ° C per 20 min e versarlo in piastre da 90 mm.
2. Real-Time PCR
- Raccolta del campione
- Inserire un tampone anale ano del paziente 3-5 cm di profondità e ruotarlo di 360° intorno.
- Mettere il tampone anale in una provetta sterile. Marco campione ID.
- Inviare il campione al laboratorio appena possibile.
Nota: I campioni potrebbero essere conservati a 4 ° C per non più di 24 h.
- Pre-arricchimento
- Aggiungere 3 mL di brodo nutriente in ogni campione nel tubo di raccolta.
- Incubare a 36 ° C per 6 h in un'incubatrice.
- Esempio di miscelazione (opzionale)
- Raccogliere 100 µ l di ogni cultura pre-arricchimento e mescolare 8-10 campioni di 1 campione in una provetta da 1,5 mL, se ci sono più di 10 campioni.
- Contrassegnare correttamente.
- Estrazione del DNA
- Centrifugare la coltura pre-arricchimento a 800 x g per 2 min consentire grandi particelle di stabilirsi, e trasferimento del surnatante in una nuova provetta e centrifugare a 12.000 x g per 5 min. eliminare il surnatante tramite aspirazione.
- Aggiungere 100 µ l di soluzione di estrazione del DNA (0,01 M, pH 8.0 Tris-EDTA, 0,01% Nonidet P 40 (NP40)) per il pellet. Vortexare vigorosamente per 1 min.
- Bollire a 100 ° C per 5 minuti in un bagno asciutto.
- Centrifugare a 12.000 x g per 5 min, raccogliere il surnatante utilizzando un tubo nuovo, che sarà il modello per le successiva analisi di PCR in tempo reale.
- PCR in tempo reale
- La miscela di reazione come di installazione seguire: per ogni campione, aggiungere 12,5 µ l della miscela di reazione, 0,4 µM di ogni primer, 0,2 µM di ogni sonda (sequenze in tabella 1)27, 5 µ l del modello come preparata al punto 2.4.4: 2x e utilizzare ddH2O per aggiungere fino a 25 µ l totale volume.
- Impostare il programma in bicicletta come segue: 95 ° C per 3 min, seguiti da 40 cicli di 95 ° C per 15 s, 55 ° C per 30 s, 72 ° C per 34 s. Raccogli segnali fluorescenti da 6-carboxy-fluoresceina (FAM) e Hexachlorofluorescein (HEX) canali nella fase di allungamento (72 ° C) automaticamente dalla fluorescente macchina PCR in tempo reale.
- Eseguire PCR in tempo reale su una macchina fluorescente di PCR in tempo reale, secondo le istruzioni dello strumento.
- Andare direttamente al passaggio 4 ed emettere rapporti negativi in caso di risultati negativi sui canali FAM/HEX, che significa che i campioni sono negativi per Salmonella spp /Shigella spp.
- Passare al punto 3.1 o 3.2 se positivi risultati si verificano sui canali HEX e/o FAM, che significa che il campione può essere positivo per Salmonella spp e/o Shigella spp., rispettivamente.
Nota: Se miscelazione del campione è stata eseguita nel passaggio 2.3, Real-Time PCR su campioni individuali che compone quella positiva dovrebbe essere eseguita per escludere il campione reale positivo.
3. guidata cultura
-
Campione di Salmonella spp. PCR positivo
- Coltura selettiva in mezzo
- Aggiungere 100 µ l della cultura pre-arricchimento in 5 mL di mezzo di Selenite cistina in una provetta. Incubare a 36 ° C per 18-24 h in un'incubatrice.
- Cultura sulla piastra di separazione
- Raccogliere un ciclo della coltura con un micro-ciclo e stendere su una piastra XLD o Salmonella chromogenic agar. Incubare a 36 ° C per 18-24 h in un'incubatrice.
- Identificazione biochimica
- Seleziona colonia sospetta su XLD piastra (Colonia Rosa con/senza cuore scuro; scuro Colonia Colonia giallo con/senza cuore scuro) o Salmonella chromogenic agar (Colonia viola o prunosus, dolci e rotonde) (Figura 2).
- Colonia di soggetto sospetto per identificazione biochimica sul sistema automatizzato di identificazione microbica, secondo le istruzioni dello strumento.
- Caratterizzazione sierologica
- O caratterizzazione di antigene
- Aggiungere una goccia di siero polivalente dell'antigene O su un vetrino pulito.
- Raccogliere un ciclo della Colonia con un micro-ciclo e macinare nei sieri.
- Andare al passaggio 3.1.4.1.4 se sembra che scorre sabbia, che significa che la Colonia è reattiva al sera (Figura 3). In caso contrario, andare al passaggio 3.1.4.1.5.
- Utilizzare sieri monovalenti di antigene O per ripetere i passaggi da 3.1.4.1.1 a 3.1.4.1.3 fino a quando l'antigene specifico O è caratterizzato.
- Utilizzare sieri Vi per ripetere i passaggi da 3.1.4.1.1 a 3.1.4.1.3. Raccogliere quelle colonie reattive di sieri di Vi in un tubo e far bollire a 100 ° C per 5 min in un bagno secco. Centrifugare a 12.000 x g per 5 min, raccogliere il pellet e ripetere i passaggi da 3.1.4.1.1 a 3.1.4.1.4 fino a specifici O antigen è caratterizzato.
- Caratterizzazione dell'antigene H
- Aggiungere una goccia di siero polivalente dell'antigene H su un vetrino pulito.
- Raccogliere un ciclo della Colonia con un micro-ciclo e macinare nei sieri.
- Andare al passaggio 3.1.4.2.4 se sembra che scorre sabbia, che significa che la Colonia è reattiva al sera.
- Utilizzare sieri monovalenti antigene di H per ripetere i passaggi 3.1.4.2.1 a 3.1.4.2.3 specifico antigene di H è caratterizzata.
Nota: A volte induzione del siero può essere necessario per caratterizzare l'antigene di fase H secondo. In questo caso, è necessario eseguire i passaggi facoltativi seguenti. - (Opzionale) Aggiungere una goccia di sieri specifici dell'antigene di H sulla piastra di agar nutriente. Attendere che tutti i sieri vengono assorbiti.
- (Opzionale) Raccogliere un ciclo della Colonia con un micro-loop e la diffusione sulla piastra dove sono assorbiti i sieri specifici di antigene H. Incubare a 36 ° C per 18-24 h in un'incubatrice.
- (Opzionale) Utilizzare sieri monovalenti antigene di H per ripetere i passaggi 3.1.4.2.1 a 3.1.4.2.3 seconda fase antigene H è caratterizzata.
- Andare al passaggio 4.
- O caratterizzazione di antigene
- Coltura selettiva in mezzo
-
Campione di Shigella spp. PCR positivo
- Cultura sulla piastra di separazione
- Raccogliere un ciclo della cultura pre-arricchimento con un micro-ciclo e stendere su una piastra XLD o MAC. Incubare a 36 ° C per 18-24 h in un'incubatrice.
- Identificazione biochimica
- Raccogliere la colonia sospetta su XLD piastra (liscio, tondo, Colonia trasparente e rossa) o MAC (liscio, tondo, trasparente e incolore Colonia con 2-3 mm di diametro; Shigella sonnei può essere più grande e girare alla luce rosa come l'allungamento del tempo di incubazione) (Figura 4).
- Colonia di soggetto sospetto per identificazione biochimica sul sistema automatizzato di identificazione microbica, secondo le istruzioni dello strumento.
- Caratterizzazione sierologica
- Aggiungere una goccia di siero polivalente Shigella spp. su un vetrino pulito.
- Raccogliere un ciclo della Colonia con un micro-ciclo e macinare nei sieri.
- Andare al punto 3.2.3.4 se sembra che scorre sabbia, che significa che la Colonia è reattiva al sera.
- Utilizzare sieri monovalenti di Shigella spp. per ripetere i passaggi 3.2.3.1 per 3.2.3.3 specifico Shigella spp è caratterizzato.
- Andare al passaggio 4.
- Cultura sulla piastra di separazione
4. report
- Emettere rapporti positivi o negativi in base ai risultati di cui sopra.
Risultati
Il protocollo è stato applicato per lo screening della Salmonella spp /Shigella spp nelle feci anale campioni da chi dovrebbe gestire cibo e acqua.
In fase PCR in tempo reale, come mostrato in Figura 5A, c'era un'amplificazione successo nel canale di HEX, che significava che il misto del campione era positivo per Salmonella spp. Quindi un ulteriore PCR in tempo reale è stato condotto su campioni singoli che compone quella positiva. Come illustrato nella Figura 5B, campione 2 è stata positiva. Di conseguenza, esempio 2 è stato scelto per la cultura guidata di Salmonella spp. Nella Figura 5C, c'era un'amplificazione successo nel canale di FAM, che significava che il misto del campione era positivo per Shigella spp. Poi è stato condotto un ulteriore PCR in tempo reale e campione 10 è stato trovato per essere positivo(Figura 5). Di conseguenza, campione 10 è stato scelto per la cultura guidata di Shigella spp.
Nella cultura guidata di Salmonella spp., c'erano colonie rosa e viola colonie su XLD piastra e Salmonella chromogenic agar, separatamente, come mostrato nella Figura 2. Quindi queste colonie sono stati sottoposti ad identificazione biochimica il sistema automatizzato di identificazione microbica. Risultati hanno mostrato che si trattava di Salmonella spp., con specie sconosciuta. Di conseguenza, Caratterizzazione sierologica è stata eseguita (Figura 3) e che era reattivo a O4, O12, Hb, H1, 2. Secondo lo schema di Kauffmann-White-Le minori, esempio 2 infine è stato segnalato come positivi per Salmonella paratyphi B. Mentre per la cultura guidata di Shigella spp., c'erano colonie rosa rossi e incolore su XLD piastra e MAC, separatamente, come mostrato nella Figura 4. Quindi queste colonie sono stati anche sottoposti ad identificazione biochimica il sistema automatizzato di identificazione microbica. Risultati hanno mostrato che si trattava di Shigella sonnei. Per confermare la sua sierotipo specifico, Caratterizzazione sierologica è stata eseguita (Figura 3) e che era reattivo sonnei fase II. Di conseguenza, campione 10 Infine è stato segnalato come positivo per fase di Shigella sonnei II.

Figura 1 : Lo schema del protocollo. Due passaggi principali sono stati mostrati e separati da dash linea. Clicca qui per visualizzare una versione più grande di questa figura.
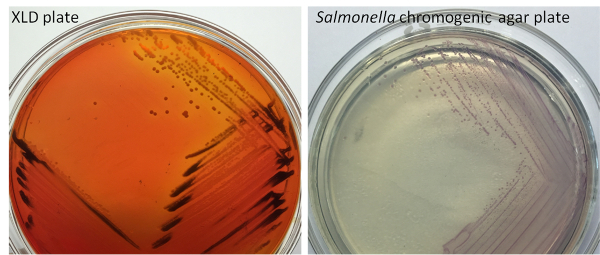
Figura 2 : Risultati rappresentativi della coltura della Salmonella spp su piastra di XLD e Salmonella agar cromogenico. Sul piatto XLD, c'erano colonie rosa con/senza cuore oscuro, mentre sulla piastra di agar cromogenico di Salmonella , c'erano colonie viola. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3 : Risultato rappresentativo Caratterizzazione sierologica. Se la colonia era reattiva al sera, sembrava che scorre sabbia (a sinistra). In caso contrario, era torbida (destra). Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4 : Risultati rappresentativi della coltura di Shigella spp su piastra di XLD e MAC. Sul piatto XLD, c'erano colonie rosse, mentre sulla piastra di MAC, c'erano colonie trasparente e incolore. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5 : Risultati rappresentativi di PCR in tempo reale. (A), HEX canale per mix campioni. Canale (B), HEX per singoli campioni. (C) FAM canale per mix campioni. (D) FAM canale per singoli campioni. PC: controllo positivo. NC: controllo negativo. Clicca qui per visualizzare una versione più grande di questa figura.
| Agente patogeno | Nome | Sequence(5'-3') |
| Salmonella | Sal-F Sal-R Sal-sonda | gctcatattaattccggcatttac caggtcaatagccagaaagg HEX-ataagtaatccaatccgaaatgcctgcgt-Eclipse |
| Shigella | Shi-F Shi-R Shi-sonda | ccgggataaagtcagaactc cagtggagagctgaagtttc FAM-aggccaggtagacttctatctcatccac-Eclipse |
Tabella 1: primer e sonde utilizzate. Il nome e le sequenze del primer e sonde erano forniti.
Discussione
Da Salmonella spp /Shigella spp sono spesso associati con l'intossicazione alimentare e trasmissione fecale-orale di gastroenterite acuta28,29 e il metodo di routine è laborioso o richiede molto tempo 7, descriviamo una piattaforma ad alta produttività per la Salmonella spp / proiezione diShigella spp., usando la PCR in tempo reale combinati con cultura guidata.
Ci sono diversi passaggi che hanno bisogno di considerazione per massimizzare la capacità di questa piattaforma. Il primo è il passaggio di pre-arricchimento dei campioni in brodo nutriente (punto 2.2). Anche se nessun passaggio di pre-arricchimento è necessario per campioni di feci raccolti da quei pazienti con sintomi evidenti, quali diarrea, ecc., un passo di pre-arricchimento generale 6 h è ancora necessaria per campioni da tampone anale, quando raccolti durante pre-l'occupazione esame fisico per le persone che vuoi gestire cibo e acqua, come quelle persone erano principalmente gli adulti sani o almeno asintomatici. Se il protocollo è stato applicato solo per la rilevazione di Salmonella spp., pre-arricchimento poteva essere condotte nel mezzo di Selenite cistina al fine di aumentare la sensibilità. Il secondo passaggio fondamentale è l'identificazione delle colonie sospette (passo 3.1.3.1 e 3.2.2.1). Ci sono molti interferenti sfondo flora in campioni di feci che possono mascherare il rilevamento e l'isolamento di destinazione patogeni30. Pertanto, la caratteristica delle colonie sospette come definito nel passaggio 3.1.3.1 e 3.2.2.1 dovrebbe essere tenuta presente durante l'esperimento. Il terzo passaggio fondamentale è la caratterizzazione sierologica di Salmonella spp. Poiché una maggioranza di Salmonella spp. contengono una seconda fase per l'antigene H31, induzione del siero deve essere eseguita. Tuttavia, l'induzione del siero non è sempre successo, e diversi cicli di induzione possono essere necessaria per determinare la corretta fase seconda H.
Il protocollo è altamente specifico e sensibile come verificato dal nostro precedente studio27. L'elevata specificità del protocollo è dimostrato dalla sua capacità, in quali Salmonella spp /Shigella spp potrebbero essere distinti da altri agenti patogeni correlati27. Il limite inferiore di rilevamento del protocollo è 104 CFU/mL e 103 CFU/mL per Salmonella spp e Shigella spp., rispettivamente27, che sono paragonabili alle precedenti relazioni7,32 , 33 , 34. come abbiamo detto sopra, la sensibilità di rilevazione di Salmonella spp. potrebbe essere ulteriormente aumentata di pre-arricchimento Selenite cistina se il protocollo è stato applicato solo per la rilevazione di Salmonella spp. Inoltre, il protocollo potrebbe aumentare il tasso positivo di due pieghe e diminuire il carico di lavoro/mediana turnaround tempo significativamente27.
Simile ad altri dosaggi NAAT, uno dei principali limiti del protocollo, rispetto al metodo di coltura di batteri classico, è che il protocollo potrebbe identificare solo Salmonella spp /Shigella spp., mentre altri batteri comuni che causano diarrea sono omesso di7. Al contrario, durante la coltura di batteri classico, quei batteri potrebbero essere identificati in parallelo se esistessero. Un'altra limitazione del protocollo è che alcune delle Salmonella spp /Shigella spp., infatti, non potrebbe essere identificata dalla PCR in tempo reale a causa di variazioni di sequenza27. Tuttavia, se i risultati negativi di PCR in tempo reale vengono visualizzati per quei campioni da pazienti con sintomi clinici evidenti, tecnici di laboratorio dovrebbero prestare attenzione e possono condurre altri esperimenti per confermare i risultati. Durante un'epidemia di grandi dimensioni, possiamo utilizzare un singolo campione invece campioni riuniti per il primo round dello screening di PCR.
In conclusione, il protocollo fornito qui potrebbe servire come una valida piattaforma per lo screening della Salmonella spp /Shigella spp.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Questo lavoro è stato supportato da scienza e tecnologia programma di Zhuhai, Cina (concessione numero 20171009E030064), la scienza e tecnologia programma del Guangdong, Cina (concessione numero 2015A020211004) e scienza e tecnologia programma dell'amministrazione generale di supervisione della qualità, ispezione e quarantena della Repubblica popolare cinese (concessione numero 2016IK302, 2017IK224).
Materiali
| Name | Company | Catalog Number | Comments |
| Tris | Sigma | 10708976001 | |
| EDTA | Sigma | 798681 | |
| NP40 | Sigma | 11332473001 | |
| ddH2O | Takara | 9012 | |
| PrimeSTAR HS (Premix) | Takara | R040Q | |
| Nutrient Broth | LandBridge | CM106 | |
| Nutrient agar | LandBridge | CM107 | |
| Selenite Cystine medium | LandBridge | CM225 | |
| XLD | LandBridge | CM219 | |
| MAC | LandBridge | CM908 | |
| Salmonella chromogenic agar | CHROMagar | SA130 | |
| Salmonella diagnostic serum | Tianrun | SAL60 | |
| Shigella diagnostic serum | Tianrun | SHI54 | |
| anal swab (collecting tube plus) | Huachenyang | ||
| slide | Mingsheng | 7102 | |
| micro-loop | Weierkang | W511 | |
| incubator | Jinghong | DNP-9082 | |
| autoclave | AUL | SS-325 | |
| dry bath | Jinghong | KB-20 | |
| automated microbial identification system | bioMérieux | VITEK2 | other equivalent system could be used |
| fluorescent real-time PCR machine | ThermoFisher | ABI7500 | other equivalent machine could be used |
Riferimenti
- Roy, S. L., Scallan, E., Beach, M. J. The rate of acute gastrointestinal illness in developed countries. Journal of Water and Health. 4, Suppl 2 31-69 (2006).
- Wilking, H., et al. Acute gastrointestinal illness in adults in Germany: a population-based telephone survey. Epidemiology and Infection. 141 (11), 2365-2375 (2013).
- Friesema, I. H. M., Lugnér, A. K., van Duynhoven, Y. T. H. P. Costs of gastroenteritis in the Netherlands, with special attention for severe cases. European Journal of Clinical Microbiology & Infectious Diseases. 31 (8), 1895-1900 (2012).
- Henson, S. J., et al. Estimation of the costs of acute gastrointestinal illness in British Columbia, Canada. International Journal of Food Microbiology. 127 (1-2), 43-52 (2008).
- Okhuysen, P. C., Jiang, Z. D., Carlin, L., Forbes, C., DuPont, H. L. Post-diarrhea chronic intestinal symptoms and irritable bowel syndrome in North American travelers to Mexico. The American Journal of Gastroenterology. 99 (9), 1774-1778 (2004).
- Wongboot, W., Okada, K., Chantaroj, S., Kamjumphol, W., Hamada, S. Simultaneous detection and quantification of 19 diarrhea-related pathogens with a quantitative real-time PCR panel assay. Journal of Microbiological Methods. 151, 76-82 (2018).
- Van Lint, P., De Witte, E., Ursi, J. P., Van Herendael, B., Van Schaeren, J. A screening algorithm for diagnosing bacterial gastroenteritis by real-time PCR in combination with guided culture. Diagnostic Microbiology and Infectious Disease. 85 (2), 255-259 (2016).
- Liu, J., et al. Use of quantitative molecular diagnostic methods to identify causes of diarrhoea in children: a reanalysis of the GEMS case-control study. Lancet. 388 (10051), 1291-1301 (2016).
- Wang, S. M., et al. Surveillance of shigellosis by real-time PCR suggests underestimation of shigellosis prevalence by culture-based methods in a population of rural China. Journal of Infection. 61 (6), 471-475 (2010).
- Wikswo, M. E., Hall, A. J. Outbreaks of acute gastroenteritis transmitted by person-to-person contact--United States, 2009-2010. MMWR Surveillance Summaries. 61 (9), 1-12 (2012).
- Shen, H., et al. The 12 Gastrointestinal Pathogens Spectrum of Acute Infectious Diarrhea in a Sentinel Hospital, Shenzhen, China. Frontiers in Microbiology. 7, 1926(2016).
- Tariq, A., et al. Molecular profiling of antimicrobial resistance and integron association of multidrug-resistant clinical isolates of Shigella species from Faisalabad, Pakistan. Canadian Journal of Microbiology. 58 (9), 1047-1054 (2012).
- Ferrari, R. G., Panzenhagen, P. H. N., Conte-Junior, C. A. Phenotypic and Genotypic Eligible Methods for Salmonella Typhimurium Source Tracking. Frontiers in Microbiology. 8, 2587(2017).
- Oliver, J. D. The viable but nonculturable state in bacteria. The Journal of Microbiology. 43, Spec No 93-100 (2005).
- Rintala, A., Munukka, E., Weintraub, A., Ullberg, M., Eerola, E. Evaluation of a multiplex real-time PCR kit Amplidiag(R) Bacterial GE in the detection of bacterial pathogens from stool samples. Journal of Microbiological Methods. 128, 61-65 (2016).
- Wohlwend, N., Tiermann, S., Risch, L., Risch, M., Bodmer, T. Evaluation of a Multiplex Real-Time PCR Assay for Detecting Major Bacterial Enteric Pathogens in Fecal Specimens: Intestinal Inflammation and Bacterial Load Are Correlated in Campylobacter Infections. Journal of Clinical Microbiology. 54 (9), 2262-2266 (2016).
- Van Lint, P., et al. Evaluation of a real-time multiplex PCR for the simultaneous detection of Campylobacter jejuni, Salmonella spp., Shigella spp./EIEC, and Yersinia enterocolitica in fecal samples. Eur Journal of Clinical Microbiology Infect Dis. 34 (3), 535-542 (2015).
- Kamkamidze, G., et al. Rapid Identification Of The Etiological Factors Causing Diarrheal Diseases. Georgian Medical News. (258), 89-92 (2016).
- Li, Y. Establishment and Application of a Visual DNA Microarray for the Detection of Food-borne Pathogens. Analytical Sciences. 32 (2), 215-218 (2016).
- Zhuang, L., et al. Detection of Salmonella spp. by a loop-mediated isothermal amplification (LAMP) method targeting bcfD gene. Letters in Applied Microbiology. 59 (6), 658-664 (2014).
- Shi, X. L., et al. Rapid simultaneous detection of Salmonella and Shigella using modified molecular beacons and real-time PCR. Zhonghua Liu Xing Bing Xue Za Zhi. 27 (12), 1053-1056 (2006).
- Mo, Q. H., et al. Preparation of a 96-microwell plate DNA diagnostic chip for detection of foodborne bacteria and its application in an incident of food poisoning. Nan Fang Yi Ke Da Xue Xue Bao. 30 (3), 417-421 (2010).
- Wang, H. B., et al. Probe-free and sensitive detection of diarrhea-causing pathogens using RT-PCR combined high resolution melting analysis. Biologicals. 44 (5), 360-366 (2016).
- Sun, H., et al. Rapid simultaneous screening of seven clinically important enteric pathogens using a magnetic bead based DNA microarray. World Journal of Microbiology and Biotechnology. 27 (1), 163-169 (2011).
- Qi, W., et al. Multiplex PCR assay for rapid detection of five important pathogenic vibrios. Chinese Journal of health laboratory technology. (24), 3497-3500 (2014).
- Blanco, G., Diaz de Tuesta, J. A. Culture- and molecular-based detection of swine-adapted Salmonella shed by avian scavengers. Science of the Total Environment. 634, 1513-1518 (2018).
- Tang, X. J., Yang, Z., Chen, X. B., Tian, W. F., Tu, C. N., Wang, H. B. Verification and large scale clinical evaluation of a national standard protocol for Salmonella.spp./Shigella.spp. screening using real-time PCR combined with guided culture. Journal of Microbiological Methods. 145, 14-19 (2018).
- Dekker, D. M., et al. Drinking water from dug wells in rural ghana--salmonella contamination, environmental factors, and genotypes. International Journal of Environmental Research and Public Health. 12 (4), 3535-3546 (2015).
- Gargano, J. W., et al. Mortality from selected diseases that can be transmitted by water - United States, 2003-2009. Journal of Water and Health. 15 (3), 438-450 (2017).
- Kumar, R., Surendran, P. K., Thampuran, N. Evaluation of culture, ELISA and PCR assays for the detection of Salmonella in seafood. Letters in Applied Microbiology. 46 (2), 221-226 (2008).
- Herrera-Leon, S., et al. Blind comparison of traditional serotyping with three multiplex PCRs for the identification of Salmonella serotypes. Research in Microbiology. 158 (2), 122-127 (2007).
- Cunningham, S. A., et al. Three-hour molecular detection of Campylobacter, Salmonella, Yersinia, and Shigella species in feces with accuracy as high as that of culture. Journal of Clinical Microbiology. 48 (8), 2929-2933 (2010).
- Eriksson, E., Aspan, A. Comparison of culture, ELISA and PCR techniques for salmonella detection in faecal samples for cattle, pig and poultry. BMC Veterinary Research. 3, 21(2007).
- Dutta, S., et al. Sensitivity and performance characteristics of a direct PCR with stool samples in comparison to conventional techniques for diagnosis of Shigella and enteroinvasive Escherichia coli infection in children with acute diarrhoea in Calcutta, India. Journal of Medical Microbiology. 50 (8), 667-674 (2001).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon