Method Article
Una plataforma de alto rendimiento para la detección de Salmonella spp. /Shigella spp.
En este artículo
Resumen
Salmonella sppShigella spp., son patógenos comunes atribuidas a la diarrea. Aquí, describimos una plataforma de alto rendimiento para la detección de Salmonella spp. /Shigella spp., utilizando PCR en tiempo real combinado con cultura guiada.
Resumen
Transmisión fecal-oral de gastroenteritis aguda ocurre de vez en cuando, especialmente cuando las personas que manejan alimentos y agua están infectadas por Salmonella spp./Shigella spp. El método estándar de oro para la detección de Salmonella spp./Shigella spp es cultura directa pero esto es desperdiciador de tiempo y mano de obra intensiva. Aquí, describimos una plataforma de alto rendimiento para Salmonella spp./Shigella spp de detección, usando la reacción en cadena de la polimerasa en tiempo real (PCR) combinada con cultura guiada. Hay dos etapas principales: la cultura guiada y PCR en tiempo real. Para la primera etapa (PCR en tiempo real), se explica cada paso del método: la muestra recogida, previo enriquecimiento, extracción de ADN y PCR en tiempo real. Si el resultado PCR en tiempo real es positivo, entonces se realiza la segunda etapa (cultura guiada): cultivo selectivo, identificación bioquímica y serológica caracterización. También ilustramos resultados representativos generados de él. El protocolo descrito aquí sería una plataforma valiosa para la detección rápida, específica, sensible y de alto rendimiento de Salmonella spp. /Shigella spp.
Introducción
La diarrea es aún un problema de salud común con una incidencia alta tasa global1,2. Aunque la mortalidad es relativamente baja, algunos pacientes muestran varios síntomas durante semanas (por ejemplo, las heces blandas y acuosas, una urgencia para ir al baño), que hacen que el impacto socioeconómico muy alto3,4. Más grave aún, algunos pacientes incluso pueden desarrollar síndrome de intestino irritable si se deja sin tratar5. Hay varias clases de bacterias, virus y parásitos que pueden causar diarrea6. Salmonella sppShigella spp., están entre las bacterias más comunes para la transmisión de la gastroenteritis aguda7,8,9,10,11. Por lo tanto, muchos condados han emitido leyes o reglamentos para regular spp Salmonella /Shigella spp. cribado entre las personas que se manejan alimentos y agua. Por ejemplo, el gobierno chino ha emitido leyes para obligatorio Salmonella spp. /Shigella spp. de detección una vez al año.
El método estándar de oro para Salmonella spp. / detección deShigella spp es la cultura de las bacterias. A través de bacterias cultura y sucesiva identificación bioquímica y serológica caracterización, podemos identificar las especies de bacterias, lo que podrían facilitar la gestión del brote de la enfermedad y perfiles de antimicrobianos para el tratamiento de pacientes 12. también podría ayudar a rastrear el origen de la infección durante la Salmonella spp. /Shigella spp. brote13. Sin embargo, este método es intensiva (que requieren operación manual) y lento (tomando varios días), especialmente para la prueba de un gran número de muestras7. Por otra parte, viable pero no cultivable (VBNC) Salmonella spp. /Shigella spppueden existir en algunas muestras de heces14. En vista de estos inconvenientes, muchos laboratorios han tratado de desarrollar nuevas técnicas para la detección de Salmonella spp. /Shigella spp15,16,17,18 , 19 , 20 , 21 , 22 , 23 , 24 , 25. todos estos métodos utilizan la prueba de amplificación de ácido nuclear (NAAT), entre los que la reacción en cadena de polimerasa (PCR) es la más común. Una limitación importante de estos métodos NAAT base es que las bacterias muertas, bacteriana incluso ruina que contiene ADN genómico incompleta, podrían demostrar resultados positivos26, que podrían influir en gran medida el diagnóstico preciso de la enfermedad. Blanco et al demostró que análisis molecular es altamente sensible, no sólo viable Salmonella en culturas, pero también a genomas parciales y bacterias muertas o inviables de infecciones pasadas o contaminación26. Por lo tanto, se deben desarrollar nuevas tecnologías.
Aquí, describimos un nuevo método que combina el NAAT basado en método y cultivo. Como se muestra en la figura 1, este nuevo método aplica a PCR en tiempo real detección primero y luego se envían muestras positivas para la identificación y cultivo de bacterias.
Protocolo
El protocolo sigue los lineamientos establecidos por el Comité de ética de investigación de Zhuhai International Travel Healthcare Center. Utilice estándar operación estéril durante el experimento.
1. preparación y composición de los medios de cultivo
- Preparar el caldo nutritivo: Disolver la peptona 1%, extracto de carne de res de 0.3%, cloruro de sodio 0,5%, glucosa 0.1% en H2O, ajustar el pH a 7,5 y autoclave en 121 ° C durante 15 minutos.
- Preparar medio de selenito cistina: disolver la peptona 0.5%, 1% Na2HCO3, 0.4% de lactosa, selenito de hidrógeno de sodio 0.4%, 0,001% L-cistina en H2O, ajustar el pH a 7.0 y hervir durante 5 minutos.
- Preparar la xilosa, lisina, desoxicolato placa de agar (XLD): Extracto de disolver levadura de 0,3%, 0,5% L-lisina xilosa de 0.375%, 0.75% de lactosa, 0.75% de sacarosa, cloruro de sodio 0,5%, 0.008% de rojo de fenol, 0,68% tiosulfato de sodio, citrato férrico de amonio 0.08%, sodio 0.25% desoxicolato, agar 1,5% en H2O y ajustar el pH a 7,4. Luego hervir durante 5 minutos y verter en placas de 90 mm.
- Preparar la placa de agar cromogénico de Salmonella : disolver 1.5% de agar, peptona y levadura Extracto de 0,7%, reactivo selectivo de 1.29% en H2O. Luego hervir durante 5 minutos y verter en placas de 90 mm.
- Preparar la placa de agar nutritivo: disolver 0.3% Extracto de carne, cloruro de sodio 0,5%, peptona 1%, agar 1,5% en H2O y ajustar el pH a 7.3. Entonces el autoclave en 121 ° C durante 15 minutos y verter en placas de 90 mm.
- Preparar la placa de MacConkey agar (MAC): disolver la peptona 2%, 1% de lactosa, cloruro de sodio 0.5% 0.5% sal de bilis de buey, 0,0025% rojo neutro, agar 1,5%, un 0,0001% de cristal violeta en H2O y ajustar el pH a 7.2. Entonces el autoclave en 115 ° C por 20 min y verter en placas de 90 mm.
2. en tiempo real PCR
- Recogida de muestras
- Inserte un hisopo anal en el ano del paciente 3-5 cm de profundidad y girar 360° alrededor.
- Poner el hisopo anal en un tubo estéril para recogida. Marca muestra ID.
- Enviar la muestra al laboratorio tan pronto como sea posible.
Nota: Las muestras podrían almacenarse a 4 ° C por no más de 24 h.
- Enriquecimiento de la
- Añadir 3 mL de caldo nutriente en cada muestra en el tubo de la colección.
- Incubar a 36 ° C por 6 horas en una incubadora.
- Muestra de mezcla (opcional)
- Recoger 100 μl de cada cultivo de enriquecimiento previo y mezclar 8-10 muestras de 1 pieza en un tubo de 1,5 mL si hay más de 10 muestras.
- Marque correctamente.
- Extracción de ADN
- Centrifugue la cultura pre-enriquecimiento a 800 x g durante 2 min permitir que las partículas grandes a establecerse, y traslado el sobrenadante a un nuevo tubo y centrifugar a 12.000 x g durante 5 minutos eliminar el sobrenadante por aspiración.
- Añada 100 μl de solución de extracción de ADN (0.01 M pH 8,0 Tris-EDTA, 0.01% Nonidet P 40 (NP40)) a la pelotilla. Vortex vigorosamente por 1 minuto.
- Hervir a 100 ° C por 5 min en un baño seco.
- Centrifugar a 12.000 x g durante 5 min recoger el sobrenadante utilizando un tubo nuevo, que será la plantilla para el posterior análisis PCR en tiempo real.
- PCR en tiempo real
- Configuración de la mezcla de reacción como sigue: para cada muestra, añadir 12,5 μl de 2 x la mezcla de reacción, 0,4 μm de cada cebador, 0.2 μm de cada sonda (secuencias en la tabla 1)27, 5 μl de la plantilla como preparado en el paso 2.4.4 y utilizar ddH2O para agregar total hasta 25 μl volumen.
- Configurar el programa de ciclismo como sigue: 95 ° C por 3 min, seguido de 40 ciclos de 95 ° C por 15 s, 55 ° C por 30 s, 72 ° C durante 34 s. recogemos señales fluorescentes de canales Hexachlorofluorescein (HEX) en la etapa de elongación (72 ° C) y 6-carboxi-fluoresceína (FAM) automáticamente por la máquina PCR en tiempo real fluorescente.
- Realizar PCR en tiempo real en una máquina PCR en tiempo real fluorescente, según instrucciones del instrumento.
- Ir directamente al paso 4 y emitir informes negativos si resultados negativos se producen en los canales de FAM/hexagonal, lo que significa que las muestras son negativas para Salmonella spp. /Shigella spp.
- Vaya al paso 3.1 o 3.2 si positivos resultados se producen en los canales hexagonales o FAM, que significa que la muestra puede ser positiva para Salmonella spp y Shigella spp., respectivamente.
Nota: Si la mezcla de la muestra se ha realizado en el paso 2.3, entonces PCR en tiempo real en muestras individuales que constituyen un positivo debe realizarse para descartar la muestra real positiva.
3. Guía de la cultura
-
Muestras positivas de Salmonella spp. polimerización en cadena
- Cultivo selectivo en medio
- Añadir 100 μl del cultivo de enriquecimiento previo en 5 mL de medio de selenito cistina en un tubo de ensayo. Incubar a 36 ° C por 18 – 24 h en una incubadora.
- Separar el cultivo en placa
- Recoger un loop de la cultura con un micro-loop y extender sobre una placa XLD o Salmonella agar cromogénico. Incubar a 36 ° C durante 18-24 h en una incubadora.
- Identificación bioquímica
- Seleccione Colonia sospechosa en placa XLD (Colonia rosa con/sin corazón oscuro; oscuro Colonia Colonia amarillo con o sin corazón oscuro) o Salmonella agar cromogénico (Colonia púrpura o prunosus, liso y redondo) (figura 2).
- Colonia sospechosa de tema para identificación bioquímica en el sistema automatizado de identificación microbiana, según las instrucciones del instrumento.
- Caracterización serológica
- O caracterización del antígeno
- Añada una gota de los sueros polivalentes del antígeno O en un portaobjetos limpio.
- Recoger un loop de la Colonia con un micro-loop y la rutina en el suero.
- Ir a paso 3.1.4.1.4 Si parece que fluye la arena, lo que significa que la Colonia es reactiva a los sueros (figura 3). De lo contrario, vaya al paso 3.1.4.1.5.
- Utilice sueros monovalentes de antígeno O repetir 3.1.4.1.1 a 3.1.4.1.3 hasta que caracteriza el antígeno específico de la O.
- Utilice sueros Vi repetir 3.1.4.1.1 a 3.1.4.1.3. Recoger las colonias reactivas de Vi sera en un tubo y hierve a 100 ° C por 5 min en un baño seco. Centrifugar a 12.000 x g durante 5 minutos recoge los pellets y repita los pasos 3.1.4.1.1 a 3.1.4.1.4 hasta antígeno específico O se caracteriza.
- Caracterización del antígeno de H
- Añada una gota de los sueros polivalentes del antígeno de H en un portaobjetos limpio.
- Recoger un loop de la Colonia con un micro-loop y la rutina en el suero.
- Ir a paso 3.1.4.2.4 Si parece que arena, que significa que la Colonia es reactiva a los sueros.
- Utilice sueros monovalentes de antígeno H repetir 3.1.4.2.1 a 3.1.4.2.3 hasta antígeno específico de la H se caracteriza.
Nota: A veces la inducción del suero puede necesitarse para caracterizar el segundo antígeno de fase H. Si es así, deben realizarse los siguientes pasos opcionales. - (Opcional) Añada una gota de concreto sera de antígeno H en placa de agar nutritivo. Espere hasta que todos los sueros son absorbidos.
- (Opcional) Recoger un loop de la Colonia con un micro-loop y propagación en la placa donde se absorben específicos sueros antígeno de H. Incubar a 36 ° C durante 18-24 h en una incubadora.
- (Opcional) Utilice sueros monovalentes de antígeno H repetir 3.1.4.2.1 a 3.1.4.2.3 hasta segunda fase antígeno H se caracteriza.
- Vaya al paso 4.
- O caracterización del antígeno
- Cultivo selectivo en medio
-
Muestra positiva de Shigella spp. polimerización en cadena
- Separar el cultivo en placa
- Recoger un loop de la cultura pre-enriquecimiento con un micro-loop y extender sobre una placa XLD o MAC. Incubar a 36 ° C por 18 – 24 h en una incubadora.
- Identificación bioquímica
- Recoger la Colonia sospechosa sobre placa XLD (lisa, redonda, transparente y rojo Colonia) o MAC (liso, redondo, transparente e incoloro Colonia con 2-3 mm de diámetro; Shigella sonnei puede ser más grande y vuelven a la luz de color rosa como el alargamiento del tiempo de incubación) (figura 4).
- Colonia sospechosa de tema para identificación bioquímica en el sistema automatizado de identificación microbiana, según las instrucciones del instrumento.
- Caracterización serológica
- Añada una gota de los sueros polivalentes de Shigella spp. en un portaobjetos limpio.
- Recoger un loop de la Colonia con un micro-loop y la rutina en el suero.
- Ir a paso 3.2.3.4 Si parece que arena, que significa que la Colonia es reactiva a los sueros.
- Utilice sueros monovalentes de Shigella spp. repetir pasos 3.2.3.1 a 3.2.3.3 hasta específicos Shigella spp se caracteriza.
- Vaya al paso 4.
- Separar el cultivo en placa
4. Informe
- Emitir informes positivos o negativos según los resultados anteriores.
Resultados
Se aplicó el protocolo para la detección de Salmonella spp. /Shigella spp en heces anal muestras de personas que se manejan alimentos y agua.
En el paso de la PCR en tiempo real, como se muestra en la figura 5A, hubo una amplificación exitosa en canal HEX, que significó que la muestra mezclada era positiva para Salmonella spp. Luego se realizó una PCR en tiempo real más en muestras individuales que constituyen un positivo. Como se muestra en la figura 5B, muestra 2 fue positivo. Por lo tanto, muestra 2 fue elegida por la cultura guiada de Salmonella spp. En la figura 5C, hubo una amplificación exitosa en el canal de la FAM, que significa que la muestra mezclada era positiva para Shigella spp. Luego se realizó una PCR en tiempo real más y muestra 10 fue encontrada para ser positiva(figura 5). Por lo tanto, muestra 10 fue elegida por la cultura guiada de Shigella spp.
En la cultura guiada de Salmonella spp., había colonias de color rosa y púrpura colonias en placa XLD y en placa de agar cromogénico de Salmonella , por separado, como se muestra en la figura 2. Entonces estas colonias fueron sometidas a identificación bioquímica en el sistema automatizado de identificación microbiana. Los resultados mostraron que fue Salmonella spp., con especies desconocidas. Por lo tanto, la caracterización serológica fue realizada (figura 3) y fue reactiva a O4, O12, Hb, H1, 2. Siguiendo el esquema de Kauffmann-White-Le menor muestra 2 informó finalmente positivo para Salmonella paratyphi B. Mientras que para la cultura guiada de Shigella spp., hubo colonias rojo e incoloras rosa en XLD placa y placa de MAC, por separado, como se muestra en la figura 4. Entonces estas colonias también fueron sometidas a identificación bioquímica en el sistema automatizado de identificación microbiana. Los resultados mostraron que fue Shigella sonnei. Para confirmar su serotipo específico, caracterización serológica fue realizada (figura 3) y fue reactiva a sonnei fase II. Por lo tanto, muestra 10 finalmente fue reportado como positivo para fase de Shigella sonnei II.

Figura 1 : El esquema del Protocolo de. Dos pasos importantes fueron demostrados y separados por la línea del salpicadero. Haga clic aquí para ver una versión más grande de esta figura.
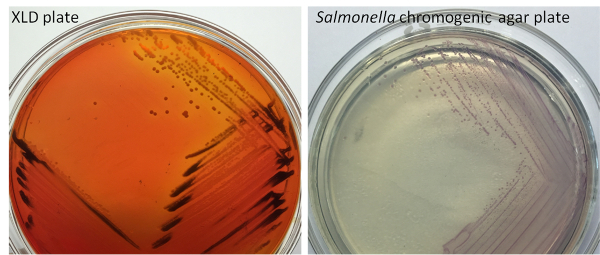
Figura 2 : Resultados representativos de la cultura de Salmonella spp. en XLD y Salmonella cromogénico agar placa. En placa XLD, había colonias rosadas con/sin corazón oscuro, mientras que en la placa de agar cromogénico de Salmonella , había colonias de color púrpura. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3 : Resultado de la caracterización serológica representante. Si la Colonia era reactiva a los sueros, parecía fluir arena (izquierda). De lo contrario, era turbia (derecha). Haga clic aquí para ver una versión más grande de esta figura.

Figura 4 : Resultados de los cultivos representativos de Shigella spp en XLD y MAC placa. En placa XLD, había colonias rojo, mientras que en la placa de MAC, había colonias transparentes e incoloras. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5 : Representante resultados PCR en tiempo real. Canal (A) hexagonal para las muestras de la mezcla. (B) hexagonal canal para muestras individuales. (C) FAM canal para las muestras de la mezcla. (D) FAM canal para muestras individuales. PC: control positivo. NC: control negativo. Haga clic aquí para ver una versión más grande de esta figura.
| Patógeno | Nombre | Sequence(5'-3') |
| Salmonella | Sal-F Sal-R Sonda de sal | gctcatattaattccggcatttac caggtcaatagccagaaagg HEX-ataagtaatccaatccgaaatgcctgcgt-Eclipse |
| Shigella | Shi-F Shi-R Shi-probe | ccgggataaagtcagaactc cagtggagagctgaagtttc FAM-aggccaggtagacttctatctcatccac-Eclipse |
Tabla 1: cebadores y sondas. Proporcionan el nombre y secuencias de los cebadores y sondas.
Discusión
Desde Salmonella sppShigella spp. a menudo se asocian a la intoxicación alimentaria y transmisión fecal-oral de gastroenteritis aguda28,29 y el método de rutina es desperdiciador de tiempo o mano de obra intensiva 7, se describe una plataforma de alto rendimiento para la Salmonella spp. / detección deShigella spp., utilizando PCR en tiempo real combinado con cultura guiada.
Hay varios pasos que requieren consideración para maximizar la capacidad de esta plataforma. El primero es el paso previo de enriquecimiento de las muestras en caldo nutriente (criterio 2.2). Aunque no paso de enriquecimiento previo es necesario para las muestras de heces recogidas de los pacientes con síntomas evidentes, tales como diarrea, etc., un paso de pre enriquecimiento general 6 h todavía es necesaria para las muestras de hisopo anal, cuando durante la previa al empleo examinación física para las personas que se manejan alimentos y agua, como esas personas eran principalmente adultos sanos o menos asintomáticos. Si sólo se aplicó el protocolo para la detección de Salmonella spp., luego pre-enriquecimiento podría llevar a cabo en medio de selenito cistina para aumentar la sensibilidad. El segundo paso crítico es la identificación de colonias sospechosas (paso 3.1.3.1 y 3.2.2.1). Hay muchos flora de fondo que interfieren en las muestras de heces que pueden enmascarar la detección y aislamiento de patógenos de destino30. Por lo tanto, la característica de las colonias sospechosas como paso definido en 3.1.3.1 y 3.2.2.1 debe tenerse en cuenta durante el experimento. El tercer paso crítico es la caracterización serológica de Salmonella spp. Una mayoría de Salmonella spp., contiene una segunda fase para el antígeno de H31, inducción de suero debe realizarse. Sin embargo, la inducción del suero no es siempre acertada, y varias rondas de inducción pueden ser necesaria para determinar la correcta segunda fase H.
El protocolo es altamente específico y sensible como verificado por nuestro anterior estudio27. La alta especificidad del protocolo es demostrada por su capacidad, en que Salmonella spp. /Shigella spp podría ser distinguido de otros patógenos relacionados con27. El límite inferior de detección del protocolo es 104 UFC/mL y 103 UFC/mL de Salmonella spp y Shigella spp., respectivamente27, que son comparables con los anteriores informes7,32 , 33 , 34. como dijimos anteriormente, la sensibilidad de la detección de Salmonella spp. podría ser incrementada por el enriquecimiento previo de selenito cistina si sólo se aplicó el protocolo para la detección de Salmonella spp. Por otra parte, el protocolo podría aumentar la tasa de positiva por dos pliegues y disminuir el carga de trabajo mediano cambio significativamente tiempo27.
Similar a otros ensayos de NAAT, una limitación importante del Protocolo, en comparación con el método de cultivo de las bacterias clásicas, es que el protocolo sólo podría identificar Salmonella spp. /Shigella spp., mientras que otras bacterias comunes causantes de diarrea son omite el7. En contraste, durante la cultura clásica de las bacterias, las bacterias podían ser identificadas en paralelo si existieron. Otra limitación del protocolo es que algunos de Salmonella spp. /Shigella spp., no pudieron ser identificados por PCR en tiempo real debido a las variaciones de secuencia27. Sin embargo, si resultados negativos de PCR en tiempo real aparecen para las muestras de pacientes con síntomas clínicos evidentes, técnicos de laboratorio deben prestar atención y realización otros experimentos para confirmar los resultados. Durante un brote grande, podemos utilizar una sola muestra en lugar de muestras agrupadas para la primera ronda de la detección de PCR.
En conclusión, el protocolo que aquí podría servir como una plataforma valiosa para la detección de Salmonella spp. /Shigella spp.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por la ciencia y tecnología programa de Zhuhai, China (concesión número 20171009E030064), la ciencia y tecnología programa de Guangdong, China (concesión número 2015A020211004) y ciencia y programa de Administración General de tecnología de supervisión de calidad, inspección y cuarentena de la República Popular de China (número de concesión 2016IK302, 2017IK224).
Materiales
| Name | Company | Catalog Number | Comments |
| Tris | Sigma | 10708976001 | |
| EDTA | Sigma | 798681 | |
| NP40 | Sigma | 11332473001 | |
| ddH2O | Takara | 9012 | |
| PrimeSTAR HS (Premix) | Takara | R040Q | |
| Nutrient Broth | LandBridge | CM106 | |
| Nutrient agar | LandBridge | CM107 | |
| Selenite Cystine medium | LandBridge | CM225 | |
| XLD | LandBridge | CM219 | |
| MAC | LandBridge | CM908 | |
| Salmonella chromogenic agar | CHROMagar | SA130 | |
| Salmonella diagnostic serum | Tianrun | SAL60 | |
| Shigella diagnostic serum | Tianrun | SHI54 | |
| anal swab (collecting tube plus) | Huachenyang | ||
| slide | Mingsheng | 7102 | |
| micro-loop | Weierkang | W511 | |
| incubator | Jinghong | DNP-9082 | |
| autoclave | AUL | SS-325 | |
| dry bath | Jinghong | KB-20 | |
| automated microbial identification system | bioMérieux | VITEK2 | other equivalent system could be used |
| fluorescent real-time PCR machine | ThermoFisher | ABI7500 | other equivalent machine could be used |
Referencias
- Roy, S. L., Scallan, E., Beach, M. J. The rate of acute gastrointestinal illness in developed countries. Journal of Water and Health. 4, Suppl 2 31-69 (2006).
- Wilking, H., et al. Acute gastrointestinal illness in adults in Germany: a population-based telephone survey. Epidemiology and Infection. 141 (11), 2365-2375 (2013).
- Friesema, I. H. M., Lugnér, A. K., van Duynhoven, Y. T. H. P. Costs of gastroenteritis in the Netherlands, with special attention for severe cases. European Journal of Clinical Microbiology & Infectious Diseases. 31 (8), 1895-1900 (2012).
- Henson, S. J., et al. Estimation of the costs of acute gastrointestinal illness in British Columbia, Canada. International Journal of Food Microbiology. 127 (1-2), 43-52 (2008).
- Okhuysen, P. C., Jiang, Z. D., Carlin, L., Forbes, C., DuPont, H. L. Post-diarrhea chronic intestinal symptoms and irritable bowel syndrome in North American travelers to Mexico. The American Journal of Gastroenterology. 99 (9), 1774-1778 (2004).
- Wongboot, W., Okada, K., Chantaroj, S., Kamjumphol, W., Hamada, S. Simultaneous detection and quantification of 19 diarrhea-related pathogens with a quantitative real-time PCR panel assay. Journal of Microbiological Methods. 151, 76-82 (2018).
- Van Lint, P., De Witte, E., Ursi, J. P., Van Herendael, B., Van Schaeren, J. A screening algorithm for diagnosing bacterial gastroenteritis by real-time PCR in combination with guided culture. Diagnostic Microbiology and Infectious Disease. 85 (2), 255-259 (2016).
- Liu, J., et al. Use of quantitative molecular diagnostic methods to identify causes of diarrhoea in children: a reanalysis of the GEMS case-control study. Lancet. 388 (10051), 1291-1301 (2016).
- Wang, S. M., et al. Surveillance of shigellosis by real-time PCR suggests underestimation of shigellosis prevalence by culture-based methods in a population of rural China. Journal of Infection. 61 (6), 471-475 (2010).
- Wikswo, M. E., Hall, A. J. Outbreaks of acute gastroenteritis transmitted by person-to-person contact--United States, 2009-2010. MMWR Surveillance Summaries. 61 (9), 1-12 (2012).
- Shen, H., et al. The 12 Gastrointestinal Pathogens Spectrum of Acute Infectious Diarrhea in a Sentinel Hospital, Shenzhen, China. Frontiers in Microbiology. 7, 1926(2016).
- Tariq, A., et al. Molecular profiling of antimicrobial resistance and integron association of multidrug-resistant clinical isolates of Shigella species from Faisalabad, Pakistan. Canadian Journal of Microbiology. 58 (9), 1047-1054 (2012).
- Ferrari, R. G., Panzenhagen, P. H. N., Conte-Junior, C. A. Phenotypic and Genotypic Eligible Methods for Salmonella Typhimurium Source Tracking. Frontiers in Microbiology. 8, 2587(2017).
- Oliver, J. D. The viable but nonculturable state in bacteria. The Journal of Microbiology. 43, Spec No 93-100 (2005).
- Rintala, A., Munukka, E., Weintraub, A., Ullberg, M., Eerola, E. Evaluation of a multiplex real-time PCR kit Amplidiag(R) Bacterial GE in the detection of bacterial pathogens from stool samples. Journal of Microbiological Methods. 128, 61-65 (2016).
- Wohlwend, N., Tiermann, S., Risch, L., Risch, M., Bodmer, T. Evaluation of a Multiplex Real-Time PCR Assay for Detecting Major Bacterial Enteric Pathogens in Fecal Specimens: Intestinal Inflammation and Bacterial Load Are Correlated in Campylobacter Infections. Journal of Clinical Microbiology. 54 (9), 2262-2266 (2016).
- Van Lint, P., et al. Evaluation of a real-time multiplex PCR for the simultaneous detection of Campylobacter jejuni, Salmonella spp., Shigella spp./EIEC, and Yersinia enterocolitica in fecal samples. Eur Journal of Clinical Microbiology Infect Dis. 34 (3), 535-542 (2015).
- Kamkamidze, G., et al. Rapid Identification Of The Etiological Factors Causing Diarrheal Diseases. Georgian Medical News. (258), 89-92 (2016).
- Li, Y. Establishment and Application of a Visual DNA Microarray for the Detection of Food-borne Pathogens. Analytical Sciences. 32 (2), 215-218 (2016).
- Zhuang, L., et al. Detection of Salmonella spp. by a loop-mediated isothermal amplification (LAMP) method targeting bcfD gene. Letters in Applied Microbiology. 59 (6), 658-664 (2014).
- Shi, X. L., et al. Rapid simultaneous detection of Salmonella and Shigella using modified molecular beacons and real-time PCR. Zhonghua Liu Xing Bing Xue Za Zhi. 27 (12), 1053-1056 (2006).
- Mo, Q. H., et al. Preparation of a 96-microwell plate DNA diagnostic chip for detection of foodborne bacteria and its application in an incident of food poisoning. Nan Fang Yi Ke Da Xue Xue Bao. 30 (3), 417-421 (2010).
- Wang, H. B., et al. Probe-free and sensitive detection of diarrhea-causing pathogens using RT-PCR combined high resolution melting analysis. Biologicals. 44 (5), 360-366 (2016).
- Sun, H., et al. Rapid simultaneous screening of seven clinically important enteric pathogens using a magnetic bead based DNA microarray. World Journal of Microbiology and Biotechnology. 27 (1), 163-169 (2011).
- Qi, W., et al. Multiplex PCR assay for rapid detection of five important pathogenic vibrios. Chinese Journal of health laboratory technology. (24), 3497-3500 (2014).
- Blanco, G., Diaz de Tuesta, J. A. Culture- and molecular-based detection of swine-adapted Salmonella shed by avian scavengers. Science of the Total Environment. 634, 1513-1518 (2018).
- Tang, X. J., Yang, Z., Chen, X. B., Tian, W. F., Tu, C. N., Wang, H. B. Verification and large scale clinical evaluation of a national standard protocol for Salmonella.spp./Shigella.spp. screening using real-time PCR combined with guided culture. Journal of Microbiological Methods. 145, 14-19 (2018).
- Dekker, D. M., et al. Drinking water from dug wells in rural ghana--salmonella contamination, environmental factors, and genotypes. International Journal of Environmental Research and Public Health. 12 (4), 3535-3546 (2015).
- Gargano, J. W., et al. Mortality from selected diseases that can be transmitted by water - United States, 2003-2009. Journal of Water and Health. 15 (3), 438-450 (2017).
- Kumar, R., Surendran, P. K., Thampuran, N. Evaluation of culture, ELISA and PCR assays for the detection of Salmonella in seafood. Letters in Applied Microbiology. 46 (2), 221-226 (2008).
- Herrera-Leon, S., et al. Blind comparison of traditional serotyping with three multiplex PCRs for the identification of Salmonella serotypes. Research in Microbiology. 158 (2), 122-127 (2007).
- Cunningham, S. A., et al. Three-hour molecular detection of Campylobacter, Salmonella, Yersinia, and Shigella species in feces with accuracy as high as that of culture. Journal of Clinical Microbiology. 48 (8), 2929-2933 (2010).
- Eriksson, E., Aspan, A. Comparison of culture, ELISA and PCR techniques for salmonella detection in faecal samples for cattle, pig and poultry. BMC Veterinary Research. 3, 21(2007).
- Dutta, S., et al. Sensitivity and performance characteristics of a direct PCR with stool samples in comparison to conventional techniques for diagnosis of Shigella and enteroinvasive Escherichia coli infection in children with acute diarrhoea in Calcutta, India. Journal of Medical Microbiology. 50 (8), 667-674 (2001).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados