Method Article
Misura sensibile di Mitophagy di flusso Cytometry utilizzando il pH-dipendente Reporter fluorescente mt-Keima
In questo articolo
Riepilogo
Mitophagy, la degradazione selettiva dei mitocondri, è stato implicato nell'omeostasi mitocondriale ed è liberalizzato in varie malattie umane. Tuttavia, convenienti metodi sperimentali per misurare l'attività di mitophagy sono molto limitati. Qui, forniamo un test sensibile per misurare l'attività di mitophagy mediante citometria a flusso.
Abstract
Mitophagy è un processo di rimozione selettiva dei mitocondri danneggiati o non necessari utilizzando macchinari di autofagia. Stretti legami sono state trovate fra mitophagy difettoso e varie malattie umane, tra cui le malattie neurodegenerative, cancro e malattie metaboliche. Inoltre, recenti studi hanno dimostrato che mitophagy è coinvolto nei processi cellulari normali, quali la differenziazione e lo sviluppo. Tuttavia, il ruolo preciso di e meccanismi molecolari alla base di mitophagy richiedono ulteriore studio. Pertanto, è fondamentale per sviluppare un metodo robusto e conveniente per misurare i cambiamenti nell'attività di mitophagy. Qui, descriviamo un protocollo dettagliato per valutare quantitativamente mitophagy attività tramite citofluorimetria usando la proteina fluorescente mitocondrio-mirati Keima (mt-Keima). Questa analisi di citometria a flusso è in grado di analizzare più rapidamente e con sensibilità rispetto ai metodi convenzionali di microscopia o immunoblotting basato mitophagy attività. Questo protocollo può essere applicato per analizzare l'attività di mitophagy in vari tipi cellulari.
Introduzione
I mitocondri sono organelli che sono essenziali per la fisiologia e la proliferazione delle cellule. I mitocondri sono responsabili della generazione più dell'80% del rifornimento di ATP tramite fosforilazione ossidativa, e forniscono anche vari intermedi metabolici per biosintesi e metabolismo1,2. Oltre ai loro ruoli nella fornitura di energia e metabolismo, i mitocondri giocano un ruolo centrale in molti altri processi importanti, tra cui la generazione di specie (ROS) reattive dell'ossigeno, nella regolazione della morte cellulare e intracellulare Ca2 + dynamics3 . Le alterazioni nella funzione mitocondriale sono state associate con malattie umane, compreso il cancro, diabete mellito e vari neurodegenerative malattie4,5. Aumentata la disfunzione mitocondriale e mutazioni del DNA mitocondriale sono anche pensate di contribuire ai processi di invecchiamento normale6,7,8. Di conseguenza, il controllo di qualità è una questione cruciale in mitocondri. I mitocondri sono organelli altamente dinamici che possono continuamente cambiare la loro forma tra reti allungate e forma breve, frammentato. Dinamiche di mitocondri svolge un ruolo importante nel mantenimento della funzione dei mitocondri, come pure la loro degradazione attraverso mitophagy9.
Mitophagy è un meccanismo che coinvolge la degradazione selettiva dei mitocondri interi utilizzando le macchine di autofagia. Mitophagy è il meccanismo di principio sottostante fatturato mitocondriale e la rimozione dei mitocondri danneggiati o disfunzionale. In questo processo, i mitocondri sono in primo luogo circondati da una membrana, causando la formazione di autofagosomi, che poi si fondono con i lisosomi per degradazione idrolitica9. Precedenti studi genetici in Drosophila hanno suggerito che due geni legati al morbo di Parkinson, indotta da PTEN putativa chinasi 1 (PINK1) (PARK6) e parkina (PARK2), funzionare nella stessa via10,11. Sottosequenza studi hanno dimostrato che la via di PINK1-Parkin è responsabile per l'eliminazione dei mitocondri danneggiati e difetti in questo risultato di percorso in mitophagy disfunzionali e può contribuire a malattie umane12,13. Difetti nei processi di mitophagy recentemente sono stati trovati in varie malattie umane, compreso il cancro, malattie cardiache, malattie del fegato e neurodegenerative malattia 14. Inoltre, recenti studi hanno inoltre dimostrato che mitophagy è fondamentale per molti processi fisiologici, quali la risposta immunitaria15,16,17,18, sviluppo e differenziazione ,19, suggerendo che mitophagy può svolgere un ruolo più attivo nel controllare le funzioni delle cellule.
Nonostante la recente conferma che mitophagy svolge un ruolo importante nel controllo di qualità sia nei mitocondri e malattia umana, i meccanismi molecolari sottostanti mitophagy rimangono mal capito. Anche se meccanismi mitophagy-relativi sono stati studiati sistematicamente in lievito, studi volti a esplorare meccanismi correlati mitophagy in cellule di mammifero sono concentrati principalmente sulla via di PINK1-Parkin. Precedenti studi hanno stabilito che la via di PINK1-Parkin è principalmente responsabile per la rimozione selettiva dei mitocondri danneggiati via mitophagy12,20,21. Tuttavia, studi recenti hanno riferito che in alcuni modelli, può essere attivato anche in assenza del funzionale PINK122,23,24mitophagy. Questi risultati indicano che oltre la via di PINK1-Parkin, lì un percorso aggiuntivo non identificato che può attivare mitophagy.
L'assenza di un metodo pratico per valutare l'attività mitophagy è un ostacolo importante a esplorare le vie che regolano mitophagy e il suo ruolo in eventi patofisiologici. La microscopia elettronica è uno strumento potente per visualizzare direttamente i mitocondri autofagosoma-inghiottito. Tuttavia, la microscopia elettronica ha limitazioni nella quantificazione mitophagy attività. Anche se le strategie che utilizzano microtubule-associated protein-1 luce catena 3 (LC3)-coniugati sonde fluorescenti, come GFP-LC3, sono attualmente i più diffusi approcci25, la natura transitoria del segnale basato su LC3 e suo alto tasso di falsi positivi segnali limitano la sua sensibilità per la rilevazione mitophagy in cellule26. Una combinazione di western blotting per misurare il livello di proteina mitocondriale, la quantificazione di DNA mitocondriale e analisi di microscopia di fluorescenza per colocalize mitocondri con autofagosomi o lisosomi sarebbe un buon approccio per la valutazione mitophagy. Tuttavia, limiti di quantificazione e una mancanza di convenienza delle metodologie esistenti richiedono nuovi approcci. L'introduzione di una nuova proteina fluorescente pH-dipendente, l'obiettivo mitocondriale Keima (mt-Keima), ha notevolmente migliorato la capacità di rilevare mitophagy27. Fondendo la sequenza mitocondriale-targeting di citocromo c ossidasi secondaria VIII (COX VIII) in Keima, mt-Keima è diretto alla matrice mitocondriale. Il grande cambiamento nel picco di eccitazione di Keima da 440 nm a 586 nm (corrispondente a pH 7 e pH 4, rispettivamente) possono essere utilizzati per valutare mitophagy con buona sensibile in sia in vitro che in vivo esperimenti28,29 . Ancora più importante, la resistenza di mt-Keima a degradazione lisosomiale causa l'integrazione del segnale del mt-Keima nei lisosomi, consentendo la facile misurazione quantitativa dell'attività mitophagy. La variazione di fluorescenza che si verifica in mt-Keima flusso cytometry28,29o possa essere analizzata usando sia la microscopia confocal. Tuttavia, un metodo basato su citometria a flusso per misurare l'attività di mitophagy utilizzando mt-Keima non è stato fornito in dettaglio fino ad oggi.
Qui, descriviamo un protocollo dettagliato per una tecnica di base di cytometry di flusso per misurare l'attività di mitophagy in cellule utilizzando mt-Keima. Anche se vi abbiamo mostrato qui che l'analisi di citometria a flusso rilevato con successo mitophagy CCCP-indotta in cellule HeLa esprimendo Parkin, questa tecnica può essere applicata a una vasta gamma di tipi di cellule.
Protocollo
1. generazione di cellule HeLa esprimendo mito-Keima (mt-Keima)
-
Preparazione di mt-Keima lentivirus
- Ricoprire una piastra di coltura 100 mm con l'aggiunta di 2 mL di 0,001% poly-L-lisina/tampone fosfato salino (PBS) e lasciare riposare per 5 min a temperatura ambiente.
- Rimuovere la soluzione di poli-L-lisina usando una pipetta di vetro collegata ad un vuoto e lavare la piastra di coltura con l'aggiunta di 2 mL di 1X PBS.
- Piastra a 1,5 x 106 HEK293T cellule sulla cultura rivestito piatto con 10 mL di DMEM contenente 10% FBS e 1% di penicillina/streptomicina e cultura le cellule a 37 ° C in un incubatore di coltura del tessuto di CO2 per un giorno.
- Transfect 2 µ g di mt-Keima lentivirali plasmide29 DNA insieme imballaggio DNAs (psPAX 2 µ g e pVSVG 0,25 µ g) utilizzando un reagente di transfezione per 8 h secondo le istruzioni del produttore.
- Rimuovere il supporto di transfezione e aggiungere 8 mL di terreno fresco.
- Raccogliere i supporti contenenti particelle virali 48 ore più tardi. Rimuovere i detriti cellulari da parte dei media raccolti mediante centrifugazione a 500 x g per 5 min e filtrare con un filtro per siringa da 0,45 µm.
Nota: Per uso a lungo termine, rendere le aliquote di particelle lentivirali e conservare i campioni a-80 ° C. Evitare di sottoporre i campioni virali per cicli per infezione virale efficiente di gelo-disgelo.
-
Infettare le cellule HeLa-Parkin con mt-Keima lentivirus
- Cellule HeLa4 piastra 5x10 esprimendo Parkin (HeLa-Parkin) in una piastra di coltura di 60 mm con 4 mL di DMEM contenente 10% FBS e 1% di penicillina/streptomicina a 37 ° C un giorno prima della raccolta i lentivirus mt-Keima.
Nota: A causa dell'assenza di espressione endogena di Parkin in HeLa cellule30, le cellule HeLa-Parkin usate qui per più chiaramente Visualizza CCCP-indotto mitophagy. Questa procedura può essere applicata anche ad altre linee cellulari immortalizzate o primario. - Rimuovere il mezzo di crescita il giorno successivo e aggiungere 1 mL di mt-Keima lentivirali media. Aggiungere un ulteriore 2 mL di terreno di coltura contenente 3 µ l di soluzione madre polybrene (8 mg/mL in acqua distillata). Incubare per un giorno in un incubatore a CO2 coltura del tessuto.
- Rimuovere la miscela lentivirali mt-Keima il giorno successivo e lavare le cellule due volte con PBS 1X. Aggiungere 4 mL di terreno di coltura e trattare le cellule con 2 con puromicina µ g/mL per due giorni.
- Dopo due giorni di selezione con puromicina, rimuovere il mezzo di crescita e lavare le cellule due volte con PBS 1x 1. Aggiungere mezzo di sviluppo e cultura le cellule in un incubatore a CO2 fino all'utilizzo.
Nota: Questo metodo può essere applicato per generare cellule che esprimono stabilmente mt-Keima, nonché sulle altre linee cellulari e cellule primarie.
- Cellule HeLa4 piastra 5x10 esprimendo Parkin (HeLa-Parkin) in una piastra di coltura di 60 mm con 4 mL di DMEM contenente 10% FBS e 1% di penicillina/streptomicina a 37 ° C un giorno prima della raccolta i lentivirus mt-Keima.
2. indurre mitophagy utilizzando CCCP trattamento e preparazione dei campioni di FACS
- Piastra 5 x 104 HeLa-Parkin cellule esprimendo mt-Keima in una cultura di 60 mm piatto con 4 mL di DMEM contenente 10% FBS e 1% di penicillina/streptomicina e cultura li per un giorno a 37 ° C in un incubatore a CO2 coltura del tessuto.
- Rimuovere il mezzo di crescita il giorno successivo e aggiungere 4 mL di fresco DMEM contenente 10 µM Carbonil cianuro m-chlorophenylhydrazone (CCCP). Incubare le cellule HeLa-Parkin-mt-Keima in incubatore a CO2 tessuto per 6 h.
- Rimuovere il mezzo di crescita 6 ore più tardi e lavare le cellule con 2 mL di PBS 1X. Staccare le cellule trattandole con soluzione di tripsina/EDTA e inattivare la tripsina aggiungendo terreno di coltura contenente 10% FBS. Trasferire le cellule in una provetta conica da 15 mL.
- Centrifugare le cellule a 500 x g per 5 min.
- Rimuovere il mezzo di crescita dall'aspirazione con cautela e risospendere le cellule con 1 mL di PBS 1X freddo.
- Trasferire le cellule nella provetta FACS appropriata e posizionarlo sul ghiaccio.
Nota: Preparare le cellule HeLa non infette come controllo negativo.
Nota: Le cellule sono vitali sul ghiaccio per diverse ore, e segnali di fluorescenza mt-Keima anche rimangano stabili.
3. analisi FACS di mitophagy
Nota: In questo studio, le cellule sono state analizzate con un citometro a flusso dotato di un laser 405 nm e 561 nm. Le cellule erano eccitate con un laser violetto (405 nm) con emissione rilevato a 610 nm ± 10 con un rilevatore di BV605 e con un laser di colore giallo-verde (561 nm) con emissione rilevato a 610 nm ± 10 da un rivelatore di PE-CF594 contemporaneamente (Figura 1).
-
Gating di cellule vive
- Accendere il citofluorimetro e il computer e accedere al software di analisi.
- Generare un nuovo esperimento o duplicare un esperimento esistente. Per eccitare le cellule con il violetto (405 nm) e giallo-verde (561 nm) laser e rilevare emissioni a 610 rilevatore di nm ± 10, selezionare BV605 e PE-CF594 oltre a forward scatter (FSC) e side scatter (SSC) nella finestra di parametro come richiesto fluorofori.
Nota: Tutti i fluorophores deve essere aggiunto in un modo lineare. - Vortex ciascun campione cellulare brevemente per disperdere cella aggrega.
- Collocare la provetta contenente un campione di cellule di controllo HeLa-Parkin che non è infetto con mt-Keima lentivirus sulla porta di iniezione del campione e premere il pulsante "eseguire" il citometro.
- Nella trama puntino del FSC vs SSC, regolare la tensione FSC e SSC per posizionare le cellule al centro della trama del puntino.
- Disegnare un cancello P1 utilizzando l'icona del poligono intorno cellule vive ed eliminare le cellule morte e detriti cellulari (Figura 2A).
-
Regolare le tensioni per laser violetto e verde
- Collocare la provetta contenente cellule HeLa-Parkin infettate con mt-Keima lentivirus sull'iniezione del campione di porta e premere il pulsante "eseguire" il citometro.
- Nella trama del puntino di BV605 (405 nm) contro SSC, regolare la tensione di BV605 in modo che le cellule HeLa-Parkin esprimendo mt-Keima vengano tenuti chiaramente distinte dalle cellule di controllo che non esprimono mt-Keima (Figura 2B).
- Nella trama del puntino di PE-CF594 (561 nm) contro SSC, regolare la tensione PE-CF594 in modo che le cellule HeLa-Parkin esprimendo mt-Keima vengano tenuti chiaramente distinte dalle cellule di controllo (Figura 2B).
-
Valutare la percentuale di cellule in mitophagy
- Acquisire il BV605 contro PE-CF594 trama di dot utilizzando una scala lineare. Disegnare un cancello utilizzando l'icona del poligono intorno le cellule che esprimono mt-Keima ed eliminare le cellule non esprimono mt-Keima (Figura 3A). La popolazione di fluorescenza-positiva delle cellule mt-Keima è riferita come il cancello "mt-Keima".
- Creare un altro puntino trama di BV605 versus PE-CF594 mostrando solo "mt-Keima"-gated cellule. In questa trama di dot, disegnare un cancello intorno le cellule non trattate di mt-Keima-positivo. L'attività basale mitophagy di controllo HeLa cells è basso, e il rapporto di emissione a PE-CF594/BV605 è minore di 1. Così, questa porta è denominata il cancello "basso".
- Disegnare un altro cancello che contengono la regione sopra del cancello "basso". Questo cancello si riferisce a come "alto" cancello perché contiene cellule con attività di alta mitophagy, vale a dire le cellule con un alto rapporto di emissione a PE-CF594/BV605 (Figura 3B).
- Per calcolare la percentuale di cellule nel cancello "alta", selezionare il menu "Visualizza gerarchia di popolazione". Il valore "% del padre" (% padre) rappresenta la percentuale di cellule nel cancello "alta" tra la popolazione di mt-Keima-positivo.
- Impostare almeno 10.000 eventi per registrare nel "mt-Keima" arresto cancello ed eseguire ogni esempio.
Risultati
Nella Figura 4è riportato un esempio dell'analisi di citometria a flusso di mitophagy CCCP-indotta in cellule HeLa-Parkin. Utilizzando il metodo di analisi di citometria a flusso sopra descritto, possiamo rilevare un aumento drammatico nelle cellule mitophagic nel cancello "alta". La percentuale di cellule nel cancello "alta" è stata aumentata di più di 10 volte rispetto alle cellule non trattate di controllo (Figura 4A). Questo aumento nell'attività di mitophagy è stata completamente abolito dal co-trattamento con methyladenine A (BFA) (Figura 4A e 4B).

Figura 1 . Rappresentazione schematica del metodo utilizzato per misurare mitophagy con citometria a flusso. Lo schema illustra i passaggi principali necessari per quantificare le cellule che subiscono mitophagy tramite flusso cytometry. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2 . Parametrizzazione di citometro a flusso. (A) Gating delle cellule viventi. Una regione per le cellule viventi è stata determinata su FSC contro plot SSC per escludere le cellule morte e detriti cellulari. (B) regolare il laser 405 nm e 531-nm utilizzando controllo HeLa cellule e HeLa cellule esprime mt-Keima. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3 . Calcolare la proporzione di cellule mitophagic da citometria a flusso. (A) cellule HeLa di Gating esprimendo mt-Keima. (B) mitophagic cellule di Gating e calcolare la proporzione utilizzando lo strumento di statistiche. Clicca qui per visualizzare una versione più grande di questa figura.
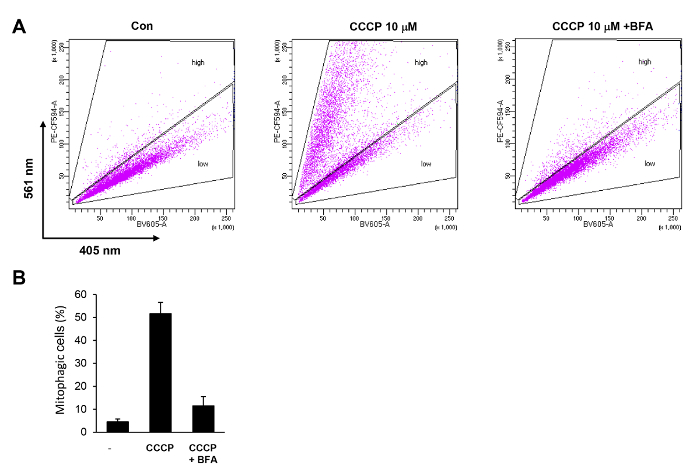
Figura 4 . Quantificazione di mitophagy CCCP-indotta in cellule HeLa tramite flusso cytometry. (A) cellule HeLa-Parkin esprimendo mt-Keima sono state trattate con 10 µM CCCP per 6 h con o senza 100 nM methyladenine A (BFA). Le cellule sono state analizzate da FACS usando rivelatori BV605 e PE-CF594. (B) cellule con un alto rapporto di PE-CF594/BV605 sono state selezionate ("alto"), e loro percentuale è stata calcolata. I risultati di tre esperimenti ripetuti vengono tracciati con deviazioni standard. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Qui, presentiamo un metodo veloce e sensibile per mediante citometria a flusso per misurare l'attività cellulare mitophagy in cellule che esprimono mt-Keima. Le cellule che subiscono un elevato livello di mitophagy presentano un rapporto aumentato di PE-CF594 (561 nm) / BV605 (405 nm) eccitazione. Quindi, mitophagy attività può essere espresso come la percentuale di cellule che esibiscono un alto rapporto di 561/405. Abbiamo calcolato la percentuale delle cellule della regione "alto" cancello su un terreno di puntino di PE-CF594 (561 nm) contro BV605 (405 nm), e i risultati hanno mostrato che il trattamento con CCCP ha indotto un aumento in mitophagy in cellule HeLa-Parkin. Precedentemente abbiamo dimostrato che la sovraespressione di lentivirus-mediata di mt-Keima non sé influisce la funzione mitocondriale o fisiologia cellulare31. Anche se abbiamo mostrato nel nostro manoscritto che analisi di citometria a flusso possono rilevare correttamente mitophagy CCCP-indotta in HeLa cells esprimendo Parkin, questa tecnica può essere applicata a qualsiasi tipo di cellula dopo l'introduzione di mt-Keima. Precedentemente abbiamo indicato che mitophagy indotta da ipossia potrebbe essere rilevato in cellule HeLa senza di espressione ectopica Parkin29. Inoltre, gli studi precedenti, compresi i nostri e quelli di altri gruppi, ha mostrato che l'analisi di fluorescenza di mt-Keima può rilevare mitophagy in cellule primarie quali i fibroblasti embrionali del mouse (MEFs) e immortalato cellule come le cellule HeLa, HEK293 e SH-SY5Y 28 , 29 , 32.
Confrontato con altri dosaggi basati su immunoblotting o microscopia mitophagy, analisi di citometria a flusso presentano vantaggi in quanto essi consentono analisi rapide e riproducibile di un gran numero di cellule31. Quindi, considerando che nelle analisi di base di microscopia, bias ricercatore può falsare i risultati, questa tendenza non è un problema quando si utilizza l'analisi di citometria a flusso.
Meno di 1 x 106 celle sono necessari per l'analisi di citometria a flusso. Inoltre, poiché l'analisi di un campione accetta solo uno o due minuti, fino a decine di campioni possa essere analizzato all'interno di una o due ore. Inoltre, le analisi di citometria a flusso possono rilevare aumenti in cellule mitophagic con buona sensibilità. Nella nostra analisi di citometria a flusso, abbiamo rilevato un aumento di 10 volte in cellule mitophagic dopo 6 h di trattamento con CCCP.
Il passaggio fondamentale per l'analisi mitophagy mediante citometria a flusso è impostato correttamente le porte di "alte" e "basse" per distinguere cellule che subiscono un elevato livello di mitophagy. Perché i livelli basali mitophagy possono essere diversi a seconda del tipo di cellula, può essere difficile in alcune linee cellulari impostare il cancello "alto" e "basso". In tal caso, è utile includere una linea cellulare di controllo con attività mitophagy basale basso come cellule HeLa. Inoltre, per le cellule trattate con un inibitore lisosomiale come clorochina, methyladenine A possa essere incluse come un controllo di basso mitophagy per impostare il corretto gating. Sebbene lentivirus sono comunemente utilizzati per l'espressione ectopica di proteine interessanti, alcune linee cellulari possono essere soggette all'infezione del virus. Se l'infezione dei lentivirus stessa provoca cambiamenti nella crescita delle cellule o funzione di mitocondri, dovrebbe essere considerato un altro metodo per mt-Keima espressione. Il livello di espressione di mt-Keima può anche variare a seconda del tipo di cella. Se il segnale di fluorescenza di mt-Keima è troppo debole o troppo eterogenea, solo le celle con un segnale di fluorescenza forte mt-Keima possono essere raccolti usando un Classificatore celle o mt-Keima cellule trattate nuovamente con una maggiore concentrazione di antibiotici con puromicina per diversi giorni . Il segnale di mt-Keima delle cellule indipendenti sono stabili fino a quando le cellule sono vitali, ma si consiglia di analizzare i campioni mediante citometria a flusso più rapidamente possibile.
La nostra analisi di citometria a flusso sono un metodo robusto e conveniente per l'analisi di mitophagy, ma ci sono alcune limitazioni. In primo luogo, perché le cellule devono essere separate come un livello di singola cellula per citometria a flusso, questo metodo è difficile da applicare per l'analisi mitophagy di tessuti o organi senza alterare la fisiologia. Inoltre, mt-Keima fluorescenza perde le sue caratteristiche di pH-dipendente dopo la fissazione, e così, tutti i campioni devono essere analizzati come cellule vive. Questo provoca la limitazione di conservazione dei campioni citati sopra e un'altra limitazione quando si applica una macchia di immunofluorescenza supplementari. Questa tecnica può misurare quantitativamente mitophagy attività, ma è in grado di monitorare i cambiamenti nella morfologia mitocondriale o dinamiche, che sono conosciuti per svolgere un ruolo importante nel processo mitophagy9. I ricercatori dovrebbero tenere a mente la complessità e indeterminati aspetti di mitophagy. Pertanto, al fine di evitare il possibile errore di valutazione, il processo di mitophagy dovrebbe essere ulteriormente confermato dai risultati combinati di altri metodi di analisi di mitophagy comuni, tra cui la microscopia elettronica, turnover delle proteine mitocondriali, una riduzione DNA mitocondriale e microscopia a fluorescenza misura la colocalizzazione dei mitocondri con i lisosomi o autofagosomi come descritto in precedenza33,34,35. Qualsiasi aumento osservato di mitophagy possa anche essere ulteriormente esaminato tramite microscopia confocale delle cellule che esprimono mt-Keima. Nelle cellule mitophagic, mt-Keima è espresso in un reticolo punctata luminoso con un alto rapporto di 586/440 che possa essere osservato in microscopia confocale28,29.
Data la sua capacità di rapida e sensibile rilevare mitophagy, questa analisi di citometria a flusso fornisce un'approssimazione di primo-punto che sarebbe utile per una varietà di studi. Semplicemente infettando le cellule con la mt-Keima lentivirus, cambiamenti di attività mitophagy possono essere facilmente misura in qualsiasi tipo di cella desiderata. In combinazione con la funzione di ordinatore delle cellule, cellule con uno specifico livello di attività mitophagy possono essere specificamente isolate e utilizzate per studiare i ruoli di e meccanismi di regolazione alla base mitophagy.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Questo lavoro è stato supportato da una sovvenzione dal National Research Foundation of Korea (2016R1D1A1B03931949) (di J. U.) e dalla National Research foundation di Corea (NRF) sovvenzione finanziati dalla government(MSIT) di Corea (n. 2016R1A2B2008887, n. 2016R1A5A2007009) (a J.Y. .)
Materiali
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| poly-L-lysine | Sigma-Aldrich | P2636 | |
| FBS | GIBCO | 16000-044 | |
| penicillin/streptomycin | wellgene | LS202-02 | |
| PBS | Hyclone | SH30013.02 | |
| HEK293T | ATCC | CRL-3216 | |
| DMEM | GIBCO | 12800-082 | |
| OPTI-MEM | GIBCO | 31985-070 | |
| Turbofect | Thermos scientific | R0531 | |
| 0.45 μm syringe filter | sartorius | 16555 | |
| HeLa | ATCC | CCL-2 | |
| polybrene | Sigma-Aldrich | H9268 | 8 mg/ml |
| puromycin | Sigma-Aldrich | P8833 | 2 mg/ml |
| Carbonyl cyanide m-chlorophenyl hydrazine (CCCP) | Sigma-Aldrich | C2759 | 10 mM |
| trypsin-EDTA | wellgene | LS015-01 | |
| EQUIPMENTS | |||
| BD LSRFortessa | BD Bioscience | LSRFortessa | |
| FACSDIVA | BD Bioscience | FACSDIVA (v8.0.1) |
Riferimenti
- McBride, H. M., Neuspiel, M., Wasiak, S. Mitochondria: More than just a powerhouse. Current Biology. 16 (14), R551-R560 (2006).
- Zorov, D. B., Krasnikov, B. F., Kuzminova, A. E., Vysokikh, M., Zorova, L. D. Mitochondria revisited. Alternative functions of mitochondria. Bioscience Reports. Bioscience Reports. 17 (6), 507-520 (1997).
- Taylor, R. W., Turnbull, D. M. Mitochondrial DNA mutations in human disease. Nature Reviews Genetics. 6 (5), 389-402 (2005).
- Galluzzi, L., Kepp, O., Trojel-Hansen, C., Kroemer, G. Mitochondrial control of cellular life, stress, and death. Circulation Research. 111 (9), 1198-1207 (2012).
- Kang, D., Hamasaki, N. Alterations of mitochondrial DNA in common diseases and disease states: aging, neurodegeneration, heart failure, diabetes, and cancer. Current Medicinal Chemistry. 12 (4), 429-441 (2005).
- Sun, N., Youle, R. J., Finkel, T. The mitochondrial basis of aging. Molecular Cell. 61 (5), 654-666 (2016).
- Wallace, D. C., Brown, M. D., Melov, S., Graham, B., Lott, M. Mitochondrial biology, degenerative diseases and aging. BioFactors. 7 (3), 187-190 (1998).
- Yun, J., Finkel, T. Mitohormesis. Cell Metabolism. 19 (5), 757-766 (2014).
- Ashrafi, G., Schwarz, T. L. The pathways of mitophagy for quality control and clearance of mitochondria. Cell Death and Differentiation. 20 (1), 31-42 (2013).
- Clark, I. E., et al. Drosophila pink1 is required for mitochondrial function and interacts genetically with parkin. Nature. 441 (7097), 1162-1166 (2006).
- Park, J., et al. Mitochondrial dysfunction in Drosophila PINK1 mutants is complemented by parkin. Nature. 441 (7097), 1157-1161 (2006).
- Narendra, D., Tanaka, A., Suen, D. F., Youle, R. J. Parkin is recruited selectively to impaired mitochondria and promotes their autophagy. The Journal of Cell Biology. 183 (5), 795-803 (2008).
- Zhu, J., Wang, K. Z., Chu, C. T. After the banquet: Mitochondrial biogenesis, mitophagy, and cell survival. Autophagy. 9 (11), 1663-1676 (2013).
- Um, J. H., Yun, J. Emerging role of mitophagy in human diseases and physiology. BMB Reports. 50 (6), 299-307 (2017).
- Al Rawi, S., et al. Postfertilization autophagy of sperm organelles prevents paternal mitochondrial DNA transmission. Science. 334 (6059), 1144-1147 (2011).
- Kim, M. J., et al. SESN2/sestrin2 suppresses sepsis by inducing mitophagy and inhibiting NLRP3 activation in macrophages. Autophagy. 12 (8), 1272-1291 (2016).
- Kim, M. J., Yoon, J. H., Ryu, J. H. Mitophagy: A balance regulator of NLRP3 inflammasome activation. BMB Reports. 49 (10), 529-535 (2016).
- Sandoval, H., et al. Essential role for Nix in autophagic maturation of erythroid cells. Nature. 454 (7201), 232-235 (2008).
- Sato, M., Sato, K. Degradation of paternal mitochondria by fertilization-triggered autophagy in C. elegans embryos. Science. 334 (6059), 1141-1144 (2011).
- Geisler, S., et al. PINK1/Parkin-mediated mitophagy is dependent on VDAC1 and p62/SQSTM1. Nature Cell Biology. 12 (2), 119-131 (2010).
- Matsuda, N., et al. PINK1 stabilized by mitochondrial depolarization recruits Parkin to damaged mitochondria and activates latent Parkin for mitophagy. The Journal of Cell Biology. 189 (2), 211-221 (2010).
- Allen, G. F., Toth, R., James, J., Ganley, I. G. Loss of iron triggers PINK1/Parkin-independent mitophagy. EMBO Reports. 14 (12), 1127-1135 (2013).
- Chu, C. T., et al. Cardiolipin externalization to the outer mitochondrial membrane acts as an elimination signal for mitophagy in neuronal cells. Nature Cell Biology. 15 (10), 1197-1205 (2013).
- Kubli, D. A., et al. PINK1 Is dispensable for mitochondrial recruitment of Parkin and activation of mitophagy in cardiac myocytes. PloS One. 10 (6), e0130707(2015).
- Dolman, N. J., Chambers, K. M., Mandavilli, B., Batchelor, R. H., Janes, M. S. Tools and techniques to measure mitophagy using fluorescence microscopy. Autophagy. 9 (11), 1653-1662 (2013).
- Kuma, A., Matsui, M., Mizushima, N. LC3, an autophagosome marker, can be incorporated into protein aggregates independent of autophagy: caution in the interpretation of LC3 localization. Autophagy. 3 (4), 323-328 (2007).
- Katayama, H., Kogure, T., Mizushima, N., Yoshimori, T., Miyawaki, A. A sensitive and quantitative technique for detecting autophagic events based on lysosomal delivery. Chemistry & Biology. 18 (8), 1042-1052 (2011).
- Lee, I. H., Yun, J., Finkel, T. The emerging links between sirtuins and autophagy. Methods in Molecular Biology. 1077, 259-271 (2013).
- Sun, N., et al. Measuring In vivo Mitophagy. Molecular Cell. 60 (4), 685-696 (2015).
- Denison, S. R., et al. Alterations in the common fragile site gene Parkin in ovarian and other cancers. Oncogene. 22 (51), 8370-8378 (2003).
- Adan, A., Alizada, G., Kiraz, Y., Baran, Y., Nalbant, A. Flow cytometry: Basic principles and applications. Critical Reviews in Biotechnology. 37 (2), 163-176 (2017).
- Bingol, B., et al. The mitochondrial deubiquitinase USP30 opposes parkin-mediated mitophagy. Nature. 510 (7505), 370-375 (2014).
- Burbulla, L. F., Kruger, R. The use of primary human fibroblasts for monitoring mitochondrial phenotypes in the field of Parkinson's disease. Journal of Visualized Experiments. 68, e4228(2012).
- Di Sante, G., Casimiro, M. C., Pestell, T. G., Pestell, R. G. Time-lapse video microscopy for assessment of EYFP-Parkin aggregation as a marker for cellular mitophagy. Journal of Visualized Experiments. 111, e53657(2016).
- Fang, E. F., et al. In vitro and in vivo detection of mitophagy in human cells, C. Elegans, and mice. Journal of Visualized Experiments. 129, e56301(2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon