Method Article
Fosfopeptide arricchimento accoppiata alla spettrometria di massa privo di etichetta quantitativa per indagare il Phosphoproteome nel carcinoma della prostata
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo protocollo descrive una procedura per estrarre e arricchire fosfopeptidi da linee cellulari di carcinoma della prostata o tessuti per un'analisi della phosphoproteome via basati sulla spettrometria di massa proteomica.
Abstract
Fosfoproteomica coinvolge lo studio su larga scala di proteine fosforilate. Fosforilazione della proteina è un passo fondamentale in molte vie di trasduzione del segnale ed è strettamente regolata da chinasi e fosfatasi. Di conseguenza, che caratterizzano il phosphoproteome può fornire intuizioni di identificare nuovi target e biomarcatori per la terapia oncologica. Spettrometria di massa fornisce un modo globalmente individuare e quantificare le migliaia di eventi di fosforilazione unico. Tuttavia, fosfopeptidi sono molto meno abbondanti che non-fosfopeptidi, rendendo l'analisi biochimica più impegnativo. Per ovviare a questa limitazione, sono necessari metodi di arricchire fosfopeptidi prima l'analisi di spettrometria di massa. Descriviamo una procedura per estrarre e digerire le proteine dal tessuto per produrre peptidi, seguite da un arricchimento per phosphotyrosine (pY) e peptidi di fosfoserina/treonina (pST) utilizzando un anticorpo-based e/o biossido di titanio (TiO2)-base Metodo di arricchimento. Dopo la preparazione del campione e la spettrometria di massa, abbiamo successivamente identificare e quantificare fosfopeptidi usando spettrometria della cromatografia-massa liquida e software di analisi.
Introduzione
Circa 165.000 nuovi casi e circa 29.000 morti si verificherà nel 2018 a causa di carcinoma della prostata, che rappresenta il tumore più comune e la seconda causa di morte per cancro negli uomini negli Stati Uniti1. Stadi precoci del carcinoma della prostata sono trattabili con resezione o radioterapia della malattia organo-limitata, dove il tasso di ricorrenza di dieci anni è compresa tra 20% e 40% per i pazienti che subiscono la prostatectomia e tra 30% e 50% per i pazienti che ricevono la radiazione terapia2. Perché il cancro alla prostata si basa sulla segnalazione per la crescita dell'androgeno, terapie di castrazione chirurgica e chimiche vengono impiegate anche per pazienti ad alto rischio. Tuttavia, ricaduta si verifica quando il cancro non risponde alla terapia di deprivazione androgenica come evidenziato dalla recidiva biochimica, dove l'antigene prostatico specifico nel siero aumenta ancora. A questo punto nella progressione, metastasi spesso vengono rilevate come bene. Questa fase avanzata, chiamata carcinoma della prostata metastatico resistente alla castrazione, rappresenta la forma letale della malattia dove la prognosi è un tempo di sopravvivenza mediano di meno di due anni3. Poche opzioni di trattamento sono disponibili in ritardo-fase malattia, compreso la seconda generazione antiandrogeni quali enzalutamide e abiraterone, come pure la chemioterapia a base di taxano come docetaxel. Malgrado i trattamenti disponibili, la malattia spesso progredisce. Pertanto, la scoperta e lo sviluppo di modalità di trattamento novello sono necessarie per migliorare la cura dei pazienti di carcinoma della prostata con la malattia avanzata.
Spettrometria di massa (MS)-approcci basati forniscono un'analisi globale del proteoma attraverso l'individuazione di centinaia di migliaia di peptide analiti4. In particolare, scoperta proteomica, acquisizione di dati-dipendente (DDA), noto anche come può produrre l'identificazione e la quantificazione di migliaia di peptidi4,5. Proteomica di rilevamento basato su MS può essere ulteriormente delineato in proteomica verticistica, dove proteine intatte sono caratterizzati, e proteomica ascendente (anche conosciuto come fucile da caccia), dove i peptidi sono analizzati per la caratterizzazione di proteine5. Così, in proteomica di fucile da caccia, un passo di proteolisi avviene nella preparazione del campione precedente l'analisi MS fendere le proteine nei peptidi. Alla fine, viene eseguita una ricerca di database per mappare i peptidi delle proteine per l'identificazione. Privo di etichetta così come parecchie isotopo-etichettatura [ad es., isotopo stabile etichettatura da amminoacidi nella coltura delle cellule (SILAC)] metodi possono essere utilizzati per confrontare quantitativamente peptidi tra campioni6,7. Mentre le tecniche d'etichettatura degli isotopi sono il gold standard, privo di etichetta metodi hanno dimostrato simile quantificazione esattezze8,9 e sono comparabili compromessi tra sensibilità e specificità10. Privo di etichetta quantificazione fornisce una maggiore copertura e permette confronti tra molti più campioni, mentre metodi basati su etichetta sono limitati dal costo e multiplexing capacità6,7,8.
Inoltre, il fucile MS può essere utilizzato anche per interrogare modificazioni post-traduzionali (PTM) come fosforilazione11. A causa della natura inferiore stechiometrica di fosfopeptidi rispetto al totale peptidi, sono impiegati diversi metodi per arricchire per fosfopeptidi, tra cui immunoprecipitazione anticorpo-basato di peptidi phosphotyrosine (pY), biossido di titanio (TiO2 ) e immobilizzato metallo affinità cromatografia (IMAC)5,12. Perché la fosforilazione della proteina è un passaggio chiave nella cella molte vie, di segnalazione fucile fosfoproteomica permette ai ricercatori di studiare i cambiamenti in diversi tipi di cancro, compresi seno13, prostata14, renale15, di segnalazione delle cellule e alle ovaie,16,17 per comprendere meglio la biologia del tumore e per identificare potenziali nuovi target per la terapia.
Questo metodo di fosfoproteomica fucile privo di etichetta è stato costruito e raffinato basato sul precedente lavoro di Graeber gruppo18,19,20. Questo protocollo inizia descrivendo l'estrazione e la digestione delle proteine e fosfoproteine dal tessuto in peptidi. Abbiamo poi dettaglio l'arricchimento dei peptidi di pY utilizzando anticorpi specifici phosphotyrosine e TiO2. Inoltre descriviamo l'arricchimento dei peptidi di fosfoserina/treonina (pST) utilizzando scambio cationico forte (SCX) seguita da TiO2. Questo protocollo si conclude con la presentazione dei campioni ad un impianto di MS e l'uso del software di analisi di MS per identificare e quantificare fosfopeptidi e loro fosfoproteine corrispondente. L'applicazione del presente protocollo può estendersi oltre il carcinoma della prostata in altri campi di fuori di oncologia e cancri.
Protocollo
Gli esperimenti usando tumori xenotrapiantati sono stati approvati dal comitato di uso e Rutgers University istituzionale Animal Care come set indietro sotto le linee guida del National Institutes of Health.
1. estrazione di proteine
- Preparare il tampone di lisi (tabella 1). (Il volume dipende dal numero di campioni per essere raccolti). Per campioni di cellule in vitro , passare al punto 1.2. Per il tessuto del tumore, passare al punto 1.3.
-
Raccolta delle cellule
- Raccogliere le cellule in una provetta conica da 50 mL e li spin a 700 x g per 5 min a 4 ° C. Scartare il surnatante e tenere la pallina sul ghiaccio. Ripetere questo passaggio per tutti i piatti raccogliere le cellule in una pallina. (In genere, sono necessari circa 5 piatti 15 cm quasi confluenti di cellule per 5 mg di proteine, ma questo può dipendere la linea cellulare e deve essere determinata empiricamente da ciascun investigatore.)
- Lavare la pallina con 30 mL di soluzione tampone fosfato (PBS) e refrigerati e spin a 700 x g per 5 min a 4 ° C prima di aspirare il PBS. Aggiungere 1,5 mL di tampone di lisi per 5 mg di proteina utilizzata per il pellet cellulare. Dispensare su e giù per un paio di volte. Passare al punto 1.4.
-
Raccolta di tessuti
- Pesare il tumore e aggiungere 2 mL di tampone di lisi ghiacciata per ogni 100 mg di tessuto in una provetta di cultura. (In genere, 50-150 mg di peso bagnato del tessuto è necessario.)
- Omogeneizzare il lisato utilizzando un omogeneizzatore portatile o da banco (impulsi x 2 per 15 s.) Pulire l'omogeneizzatore prima il primo campione e tra i campioni utilizzando candeggina al 10%, 70% di etanolo e acqua deionizzata in successione.
- Per ridurre e alchilata, riscaldare i campioni omogeneizzati a 95 ° C per 5 min. Quindi raffreddarli in ghiaccio per 15 minuti. Su ghiaccio, sottoporre ad ultrasuoni il lisato x 3 (cioè, impulso per 30 s con 60 s pause tra gli impulsi). Il campione non dovrebbe essere a questo punto clumpy o viscosi. Calore il lisato a 95 ° C per 5 min21.
- Centrifuga tubo lisato nella sonicazione stessa utilizzando un rotore swing a 3.500 x g a 15 ° C per 15 min. raccogliere il surnatante e scartare il pellet.
- Determinare la concentrazione di proteina mediante l'esecuzione di un saggio di Bradford22. Se necessario, diluire il lisato a 5 mg/mL con un tampone di lisi. Conservare a-20 ° C.
Nota: L'esperimento può essere messo in pausa qui. Congelare i campioni a-80 ° C e continuare in un secondo momento.
2. lisata digestione
- Diluire il campione 12x utilizzando 100 mM Tris (pH = 8,5) per ridurre la quantità di guanidinium. Diluire i campioni tutti allo stesso volume per ridurre al minimo gli effetti della digestione disuguale. Salvare 12,5 µ g del non digerito lisato per confermarla su un gel di Coomassie macchiati23.
- 5 mg di proteina, aggiungere 10 µ g di lisil endopeptidasi (Lys-C) e incubare a temperatura ambiente per 5-6 h. Adjust pH a 8.0 di aggiunta di 1 M untitrated Tris (pH ~ 11).
- Preparare 1 mg/mL di L-1-tosylamido-2-phenylethyl clorometil chetone (TPCK)-trattata tripsina in HCl di 1 mM (con 20 mM CaCl2). Aggiungere la tripsina in un rapporto di tripsina: proteine di 1: 100 e incubare a 37 ° C per 3 h.
- Aggiungere la stessa quantità di tripsina fresca come al punto 2.3. Incubare a 37 ° C durante la notte.
- Salvare 12,5 µ g del digerito lisato per confermare la digestione completa su un gel di Coomassie macchiati23.
3. estrazione in fase inversa
- Registrare il volume lisato. Filtrare il campione utilizzando un filtro cutoff di kDa 15 mL 10. Centrifugare il campione a 3.500 x g utilizzando il rotore swing (o 3.500 x g in un rotore ad angolo fisso) a 15 ° C fino a quando il volume di retentato è inferiore a 250 µ l (tempo di percorrenza circa 45-60 min). Raccogliere il flusso continuo e scartare il retentato.
Nota: L'esperimento può essere messo in pausa qui. Congelare i campioni a-80 ° C e continuare in un secondo momento. - Per acidificare il campione, aggiungere circa 20 µ l di 5% acido trifluoroacetico (TFA) per mL di lisato. Mescolare bene e misurare il pH del campione utilizzando strisce di pH. Regolare il pH a 2.5 utilizzando 5% TFA.
- Collegare l'estremità più corta di una colonna di C-18 per un collettore ad aspirazione. Impostare il vuoto fra 17 e 34 kPa (o secondo le istruzioni del produttore). Utilizzando pipette in vetro , bagnate la colonna con 3 mL di acetonitrile 100% (ACN). Non lasciare asciugare la colonna.
- Utilizzando pipette in vetro , equilibrare la colonna con 6 mL di 0,1% TFA applicato come 2 x 3 mL. Caricare il campione acidificato nella colonna. Non aggiungere più di 3 mL alla volta. Regolare il vuoto per circa 1 o 2 gocce al secondo di destinazione.
- Utilizzando pipette in vetro , lavare la colonna con 9 mL di 0,1% TFA applicato come 3 x 3 mL. Eluire la colonna con 2 mL di 40% ACN, 0,1% TFA. Raccogliere le due frazioni di 2 mL in provette di coltura di vetro . Scartare la colonna.
- Coprire le provette di eluato con parafilm e perforare i fori di 3-5 sulla copertina utilizzando un ago di 20G. Congelare l'eluato su ghiaccio secco per almeno 30 min fino a quando non è completamente solido.
- Lyophilize le frazioni durante la notte. Il giorno seguente, assicurarsi che i campioni siano completamente asciutti prima di arrestare i liofilizzatori. Conservare i capillari in una provetta conica 50 mL con salviette delicate a-80 ° C.
Nota: L'esperimento può essere messo in pausa qui.
4. immunoprecipitazione e l'arricchimento di pY peptidi24
- Risospendere la polvere liofilizzata con 0,5 mL di tampone di associazione ghiacciata immunoprecipitazione (IP) in ogni frazione. Piscina le frazioni trasferendo il volume di risospensione di 0,5 mL dalla seconda frazione la prima frazione e salvare la punta della pipetta. Vigorosamente vortice (invece di pipettaggio su e giù) per assicurarsi che il campione è completamente sciolto prima di trasferirlo a un stoccaggio PROVETTE CRIOGENIA di 3,6 mL tappo a vite.
- Come descritto al punto 4.1., sciacquare i tubi di liofilizzazione con un altro 0,5 mL di buffer di binding IP (tabella 1) in ogni provetta. Trasferire la soluzione nella provetta tappo a vite di 3,6 mL utilizzando la stessa punta della pipetta per ridurre al minimo eventuali perdite di campione. Ripetere il risciacquo 1 x altro, rendendo il volume finale di risospensione 2 mL (per 5 mg di proteina). Misurare il pH del campione per assicurarsi che è circa 7,4. Se è troppo acido, in modo iterativo aggiungere 10 µ l di 1 M Tris (untitrated, pH ~ 11). Se è troppo di base, in modo iterativo aggiungere 10 µ l di acido cloridrico diluito (01:25 o 1: 100).
-
Pre-lavare le perle di pY (per 5 mg di a partire lisato)
- 25 µ g di anticorpo 4 10 e 12,5 µ g di 27B10.4 anticorpo sono necessari per campione. Dopo aver utilizzato un p200 pipetta con una punta di taglio per trasferire gli anticorpi in microcentrifuga separata, lavare gli anticorpi con 450 µ l di tampone di binding IP ghiacciata x 2. Centrifugare a loro a 100 x g per 1 min a 4 ° C e aspirato fuori il supernatante.
- Risospendere le perline per una concentrazione stock di 0,5 mg/mL utilizzando IP associazione buffer. (Do non vortice le perline). Dopo Aliquotare il liquame necessario (50 µ l di anticorpo 4 10 fanghi e 25 liquami di anticorpo 27B10.4 µ l per campione) in una singola provetta, girare giù i tubi di centrifuga stock a 200 x g per 1 min a 4 ° C. Lavare le pareti laterali con surnatante prima di restituire le perle alla conservazione in frigorifero.
- Aggiungere alla soluzione di campione di sedimento nel tappo a vite provette per criogenia prelavati pY perline. Incubare per loro a 4 ° C in un rotatore fine sopra una notte.
- Inserire le provette per criogenia tappo a vite in una provetta da centrifuga 50ml foderata con un panno delicato. Rotazione verso il basso le perline a 100 x g per 1 min. Risparmia il surnatante, che verrà utilizzato per arricchire per peptidi pST. (L'arricchimento per pST comincia al punto 7 e può essere eseguita in parallelo con l'elaborazione di peptide pY).
- Risospendere le perline con 300 µ l di tampone di associazione di IP. Trasferirli in una provetta da 2 mL microcentrifuga e loro si sono fermati a 100 x g per 1 min a 4 ° C.
- Sciacquare l'incubazione tubo 3x con 200 µ l di tampone di associazione di IP. Trasferire il contenuto al tubo del Microcentrifuge stesso ogni volta. Poi li spin giù.
- Lavare le perle nella microcentrifuga tube 3x con 500 µ l di tampone di associazione di IP e li spin giù a 100 x g per 1 min. Poi lavare le perle 4 x con 450 µ l di 25mm NH4HCO3, pH 7.5 e spin loro giù a 100 x g per 1 min. uso un fresco 25 millimetri NH4HCO3 soluzione da polvere ogni volta.
- Centrifugare le perline a 1.500 x g per 1 min. uso una punta di gel-caricamento per rimuovere il surnatante completamente immergendo la punta della punta della gel-caricamento leggermente di sotto di superficie le palline.
- Aggiungere 4 volte il volume della perla dello 0,1% TFA ai talloni (vale a dire, aggiungere 300 µ l di 0,1% TFA per 75 µ g di liquami perlina pY). Mescolare bene e incubare la miscela in un thermomixer a 1.000 giri/min per 15 min a 37 ° C.
- Trasferire la risospensione in un filtro di spin di 0,2 µm. Rapidamente giù per il tubo di eluizione e trasferire il volume residuo allo stesso filtro spin usando una pipetta P10. Spin-down il filtro spin a 850 x g per 1 min. trasferire l'eluizione a un tubo del microcentrifuge basso legame . Vuoto concentrare l'eluato sino ad essiccazione durante la notte a 40 ° C e con un tempo di calore di 300 min.
Nota: L'esperimento può essere messo in pausa qui. Congelare i campioni a-80 ° C e continuare in un secondo momento.
5. biossido di titanio arricchimento25 di pY peptidi
- Risospendere i secchi giù fosfopeptidi in 200 µ l di 50% ACN, 0,1% TFA. Vortex e centrifugare a 10.000 x g per 30 s. ripetizione di questo 1x per risospendere il loro bene.
- Preparando il TiO2 perle contenute in consigli che hanno una capacità di 200 campioni di µ l.
- Picchiettare delicatamente sul lato punta piccolo della punta per spostare il materiale a tal fine. Lavare l'estremità con l'aggiunta di 200 µ l di 100% ACN, seguita da invertendo la punta e sfogliando l'estremità più piccola per spostare il liquido verso il tappo.
- Utilizzando una lama di rasoio, tagliare la punta piccola della punta e metterlo sopra un basso legame con le proteine tubo. (Evitare l'uso di provette di polistirene come TiO2 si attaccano ai lati del tubo). Rimuovere il tappo e inserire una micropipetta per immergersi fuori i restanti ACN. Ripetere il lavaggio con 200 µ l di 100% ACN. Le perle di TiO2 si trovano ora nel tubo basso legame proteico per i seguenti passaggi.
- Presupposto di TiO2 con 500 µ l di 100% ACN 2 x. Pipetta per mescolare le perline con il solvente. Centrifugare a loro a 100 x g per 1 min.
- Condizione di TiO2 con 500 µ l di tampone di fosfato di sodio 0,2 M (pH ~ 7) x 2. Lavare le perle con 300 µ l di equilibrazione tampone x 3. Perché TiO2 è molto denso, le perline si depositerà rapidamente.
- Aggiungere 400 µ l di 50% ACN, 0,1% TFA nel tubo basso legame proteico, seguita da aggiunta 84 µ l di acido lattico. Trasferire il sedimento fosfopeptidi nel tubo basso legame proteico e li Incubare per 1 h a temperatura ambiente utilizzando un rotatore di over-fine.
- Centrifugare le perline a 100 x g per 1 min a pellet li. Lavarli con 300 µ l di tampone di equilibrazione (tabella 1), 2 x e li spin giù a 100 x g per 1 min.
- Sciacquare le perline con 300 µ l di risciacquo tampone x 2. Trasferirli a un filtro di spin di 0,2 µm. Spin loro a 1.500 x g per 1 min.
- Trasferire l'unità filtro in una provetta di legame proteico basso pulito 1,5 mL. Eluire il contenuto 2 x con 200 µ l di 0,9% NH3 in H2O. misura del pH con strisce di pH, che deve essere compreso tra 10 e 11. Vuoto concentrare l'eluato sino ad essiccazione durante la notte per far evaporare l'ammoniaca.
6. desalificazione pY peptidi per MS Analyses
- Ricostituire il fosfopeptidi con 15 µ l di 0,1% TFA di Vortex e loro centrifugazione a 10.000 x g per 30 s per risospendere li. Ripetere questo 1x per risospendere il loro bene. Non pipettare su e giù.
- Pulire il campione utilizzando una punta di C-18 con una capacità di legame di 5 µ g e seguire il protocollo del produttore.
- Asciugare completamente il volume di eluizione di concentrazione sotto vuoto. Questo richiede 1-2 h. Risospendere fosfopeptidi secchi in 12,5 µ l della soluzione di spettrometria di massa (Vedi tabella 1) (o come raccomandato dal MS del ricercatore proteomica core facility). Vortice e brevemente rallentare la soluzione a 10.000 x g per 30 s. Ripeti questo 2x risospendere il loro bene. I campioni sono pronti per la presentazione di un impianto di spettrometria di massa (punto 11).
Nota: La seguente procedura riportata di seguito è relative all'arricchimento di peptide pST solo.
7. invertire la fase di estrazione dei peptidi di pST
- Misurare la concentrazione di peptide del surnatante acquisito dal passaggio 4.6 eseguendo un'analisi di peptidi. Una quantità sufficiente per spettrometria di massa di pST è di 2,5 mg.
- Regolare il pH a 3.5 con 5% TFA.
- Collegare l'estremità più corta di una colonna di C-18 per un collettore ad aspirazione. Impostare il vuoto fra 17 e 34 kPa (o secondo le istruzioni del produttore). Bagnate la colonna con 3 mL di 100% ACN. Non lasciare asciugare la colonna.
- Equilibrare la colonna con 6 mL di 0,1% TFA applicato come 2 x 3 mL. Caricare il campione acidificato nella colonna. Non aggiungere più di 3 mL alla volta. Regolare il vuoto per circa 1-2 gocce al secondo di destinazione.
- Lavare la colonna con 9 mL di 0,1% TFA applicato come 3 x 3 mL. Eluire la colonna con 2 mL di 40% ACN, 0,1% TFA. Raccogliere le due frazioni di 2 mL in provette di coltura di vetro. Scartare la colonna.
- Coprire le provette di eluato con parafilm e perforare i fori di 3-5 sulla copertina utilizzando un ago di 20G. Congelare l'eluato su ghiaccio secco per almeno 30 min fino a quando non è completamente solido.
- Lyophilize le frazioni selezionate durante la notte. Il giorno seguente, assicurarsi che i campioni siano completamente asciutti prima di arrestare i liofilizzatori. Conservare i capillari in una provetta conica 50 mL con salviette delicate a-80 ° C.
Nota: L'esperimento può essere messo in pausa qui.
8. forte scambio cationico (SCX) dei peptidi di pST
- Risospendere i peptidi liofilizzati in 2 mL di tampone A (tabella 1). Piscina le frazioni per ciascun campione. (La soluzione sarà nuvolosa).
- Preparare il collettore del vuoto. Collegare una colonna SCX ad una siringa da 3 mL con lo stantuffo rimosso. Impostare il vuoto fra 17 e 34 kPa (o secondo le istruzioni del produttore).
- Confezionare la colonna SCX con 4 mL di ACN, seguita da 4 mL di Buffer A.
- Caricare i 2 mL del campione dal punto 8.1 e raccogliere l'eluato immediatamente. Caricare 3 mL di tampone A:B (80.9:19.1) e raccogliere l'eluato. La piscina di loro eluati di ciascun campione e aliquota in provette di 2 mL basso legame proteico.
- Vuoto si concentrano tutti i campioni fino a circa il 30% del volume rimane. (Questo passaggio richiede circa 2-4 h.) Piscina le aliquote in 1 basso legame con le proteine provetta per ciascun campione.
- Collegare l'estremità più corta di una colonna di C-18 per un collettore ad aspirazione. Impostare il vuoto fra 17 e 34 kPa (o secondo le istruzioni del produttore). Bagnate la colonna con 3 mL di 100% ACN 2 x. Non lasciare che la colonna asciutta.
- Equilibrare la colonna con 3 mL di 0,1% TFA 2 x. Caricare il campione in colonna. Non aggiungere più di 3 mL alla volta. Regolare il vuoto per circa 1-2 gocce al secondo di destinazione.
- Lavare la colonna con 3 mL di 0,1% TFA 2 x. Eluire la colonna con 4 mL di 50% ACN, 0,1% TFA.
9. biossido di titanio arricchimento dei peptidi di pST
-
Preparando il TiO2 perle contenute in consigli che hanno una capacità di 200 campioni di µ l
- Picchiettare delicatamente sul lato piccola punta della punta per spostare le perline a tal fine. Rimuovere il tappo e versare le perle in una provetta conica da 15 mL in polipropilene.
- Lavare l'estremità con l'aggiunta di 200 µ l di 100% ACN, invertendo la punta un paio di volte e sfogliando l'estremità più piccola per spostare il liquido verso il tappo. Utilizzando una lama di rasoio, tagliare la punta piccola della punta e metterlo sopra il tubo conico da 15 mL in polipropilene. Rimuovere il tappo e inserire una micropipetta per immergersi fuori i restanti ACN. Ripetere il lavaggio con 200 µ l di 100% ACN. Il TiO2 perle si trovano ora nel tubo conico da 15 mL per le fasi successive.
- Presupposto di TiO2 con 500 µ l di 100% ACN 2 x. Pipetta per mescolare le perline con il solvente. Centrifugare a loro a 100 x g per 1 min.
- Condizione di TiO2 con 500 µ l di tampone di fosfato di sodio 0,2 M (pH ~ 7) due volte. Lavare le perle con 300 µ l di equilibrazione tampone x 3.
- Trasferimento fosfopeptidi eluiti nella provetta conica da 15 mL in polipropilene. 560 µ l di acido lattico e incubare per 1 h a temperatura ambiente utilizzando un rotatore di over-fine.
- Centrifugare la miscela a 100 x g per 1 minuto agglomerare le perline. Lavarli con 300 µ l di tampone di equilibrazione (tabella 1), 3 x. Spin loro giù a 100 x g per 1 min.
- Sciacquare le perline con 300 µ l di risciacquo tampone x 2. Trasferirli a un filtro di spin di 0,2 µm. Spin loro giù a 1.500 x g per 1 min.
- Trasferire l'unità filtro in una provetta di legame proteico basso pulito 1,5 mL. Eluire il contenuto 2 x con 200 µ l di 0,9% NH3 in H2O. Lasciate che la soluzione di sedersi sul fosfopeptidi per 2 min prima eluizione li. Misurare il pH, che deve essere compreso tra 10 e 11.
- Vuoto concentrare l'eluato sino ad essiccazione durante la notte per far evaporare l'ammoniaca.
10. pST peptidi di desalificazione per analisi MS
- Picchiettare delicatamente sul lato punta piccolo della punta per spostare il materiale a tal fine. Lavare l'estremità con l'aggiunta di 200 µ l di 100% ACN, seguita da invertendo la punta e sfogliando l'estremità più piccola per spostare il liquido verso il tappo.
- Utilizzando una lama di rasoio, tagliare la punta piccola della punta e metterlo sopra una provetta conica da 15 mL in polipropilene. Rimuovere il tappo e inserire una micropipetta per immergersi fuori i restanti ACN. Ripetere il lavaggio con 200 µ l di 100% ACN. Le perle di TiO2 si trovano ora nel tubo conico da 15 mL in polipropilene per le fasi successive.
- Pulire il campione utilizzando una punta di C-18 con una capacità di legame di 100 µ g. (seguire le istruzioni del produttore).
- Asciugare completamente il volume di eluizione di concentrazione sotto vuoto. Questo richiede 1-2 h.
- Risospendere fosfopeptidi secchi in 12,5 µ l della soluzione di spettrometria di massa (o come raccomandato da impianto di nucleo del ricercatore MS proteomica). Vortex e centrifugare li a 10.000 x g per 30 s. Ripetere 2x per risospendere il loro bene. (Non pipettare su e giù.)
11. spettrometria totale analisi
- Inviare i campioni alla struttura di nucleo di proteomica MS per eseguire liquida cromatografia-tandem MS (LC-MS/MS) utilizzando le impostazioni consigliate. Impostazioni di esempio sono i seguenti (Vedi tabella 2 per il sommario):
- Caricare 5 µ l di campioni su una colonna di trappola (2 cm lunghezza x 75 micron di diametro) e lavarli con 0,1% TFA per 5 min con una portata di 5 µ l/min.
- Portare la trappola in linea con una colonna analitica nano (20 cm x 75 µm) con una portata di 300 nL/min.
- I gradienti lineari segmentati (una percentuale di acido formico di 0,16%, 80% ACN in acido formico 0,2%) sono diversi tra campioni pY e pST:
- Per gli esempi di pY, eluire con una pendenza del 4-15% in 5 min, 15-50% in 40 min e 50-90% in 5 min.
- Per esempi di pST, eluire con una pendenza del 4-15% in 30 min, 15-25% in 40 min, 25-50% in 44 min e 50-90% in 11 min.
- Acquisire i dati di MS in modalità di acquisizione dati-dipendente con una serie ciclica di una scansione completa con una risoluzione di 120.000 seguita da MS/MS (HCD, energia di collisione relativa del 27%) degli 20 ioni più intensi e una durata di esclusione dinamica di 20 s.
- Dopo il MS eseguire completamento, è possibile importare i file raw di MS in un programma di software di analisi di MS per identificare e quantificare fosfopeptidi. (MaxQuant software8,26,27 è stato utilizzato in questo esperimento. Se non diversamente specificato nella tabella 3, sono state utilizzate le impostazioni predefinite.)
Risultati
Questo protocollo viene descritto in dettaglio un metodo per l'estrazione di proteine e digestione seguita da phosphopeptide arricchimento e la successiva analisi MS (Figura 1). Le composizioni di tutti i buffer e le soluzioni che vengono utilizzate in questo protocollo sono elencate nella tabella 1. L'uso sequenza di Lys-C e la tripsina fornisce una digestione efficiente. Un gel Coomassie macchiati di pre-digeriti lisato conferma la presenza di proteine, mentre la macchiatura del post-digerito lisato conferma la digestione completa (Figura 2A). Per una digestione completa, nessuna band dovrebbe apparire sopra 15 kDa, tranne il 30 kDa e 23,3 kDa bande per Lys-C e tripsina, rispettivamente. L'aggiunta di Lys-C riduce anche il numero di divisioni perse (Figura 2B). Poiché i peptidi pY rappresentano solo il 2% del phosphoproteome28, immunoprecipitazione dei peptidi pY utilizzando un anticorpo specifico pY è il primo passo di arricchimento del peptide pY. Il surnatante risultante diventa l'input per l'arricchimento del peptide pST. L'immunoprecipitazione di pY separa efficacemente pY peptidi da peptidi pST dove mediamente sono 85% dei fosfopeptidi identificato dalla preparazione pY pY (Figura 3A) e oltre il 99% dei fosfopeptidi identificato dalla preparazione pST sono pST (Figura 3B). Biossido di titanio viene utilizzato per arricchire per fosfopeptidi in entrambe le preparazioni. La percentuale prevista di peptidi nella preparazione MS-ready che vengono fosforilate è tra 30-50% (Figura 4A). La variabilità della percentuale di arricchimento phosphopeptide può essere maggiore nella preparazione pY come risultato ci siano molti meno pY peptidi di peptidi pST. La maggior parte dei fosfopeptidi rilevato in termini di specie phosphopeptide, dispone di un gruppo di phosphoryl singola o doppia (Figura 4B).
Dopo l'esecuzione di spettrometria di massa, i file raw di MS vengono caricati in un software di analisi di MS. Le impostazioni dei parametri utilizzate nell'esperimento sono riportate nella tabella 3 , ma varieranno da software a software e possono variare da versione a versione. I parametri che non sono elencati sono stati lasciati come impostazione predefinita, tra cui un FDR taglio dell'1% per peptide-spettro corrispondente (PSM) con un punteggio minimo di Andromeda di 40 per l'identificazione di peptidi modificati27. Impostazione di un taglio di probabilità di localizzazione di maggiore di 0,75 filtri fuori circa 5% dei peptidi pY e 15% e il 34% dei pS e pT peptidi, rispettivamente (Figura 5A). Dopo l'applicazione di questi filtri, il numero di identificazioni di phosphopeptide alla fine dell'analisi MS previsto è di circa 300 circa 7.500 pS peptidi e pY peptidi (per 5 mg di proteina partenza) e 640 pT peptidi (per 2,5 mg del peptide partenza ammontare) dai preparativi rispettivi arricchimento (figura 5B). Il numero delle ripetizioni e la variabilità dell'intensità del segnale phosphopeptide determina un'adeguata alimentazione per confronti statistici. In quattro esperimenti separati con gruppi che contengono duplicati biologici o triplici copie, sono stati calcolati i coefficienti delle percentuali di variazione (% CV) per fosfopeptidi rilevati. Distribuzioni di variabilità inferiore (ad es., pST gruppi 1-5 in Figura 5) indicano che la raccolta del campione, la preparazione e funzionamenti di spettrometria di massa erano coerenti. D'altra parte, distribuzioni di maggiore variabilità (ad es., pST gruppo 6 in Figura 5) indica i dati più rumorosi che richiederebbero più grande piega-modifiche per rilevare differenze significative nelle analisi differenziale a valle.
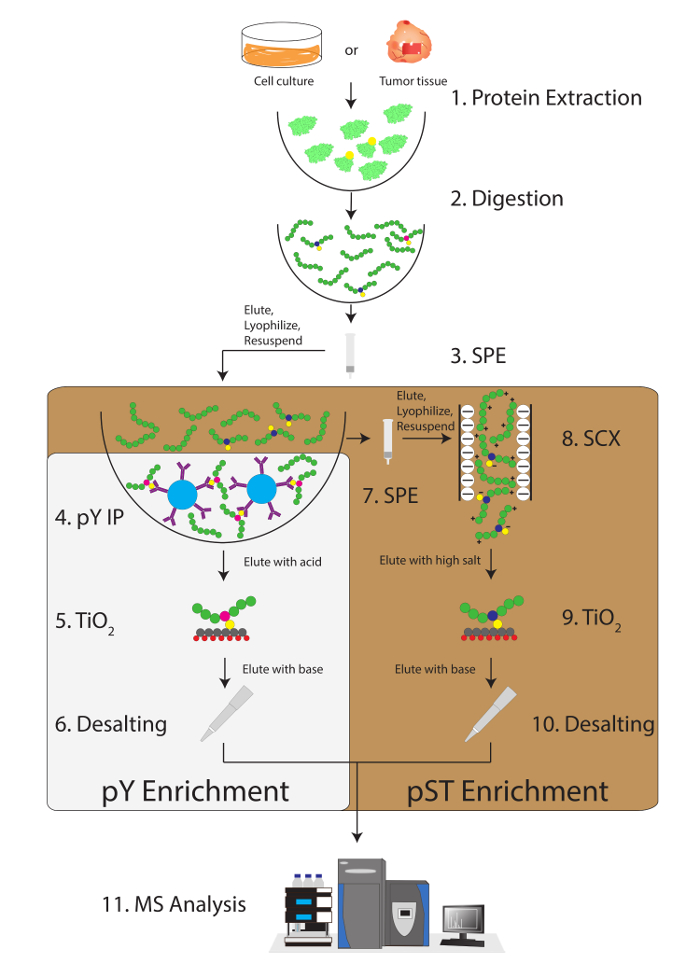
Figura 1: diagramma di flusso di lavoro. Proteine da campioni vengono estratti e digerite. Peptidi sono estratte da estrazione in fase solida (SPE), e peptidi phosphotyrosine (pY) sono immunoprecipitated. In parallelo, i peptidi di fosfoserina/treonina (pST) sono arricchiti dal surnatante nel passaggio immunoprecipitazione pY. Scambio cationico forte (SCX) viene eseguito sul surnatante per rimuovere peptidi molto caricate per ridurre la soppressione dello ione12. Entrambe le preparazioni subiscono phosphopeptide arricchimento tramite biossido di titanio (TiO2). Dopo la pulitura di campione, liquida cromatografia-spettrometria di massa (LC-MS/MS) viene eseguita per misurare l'abbondanza phosphopeptide. I dati raw viene quindi caricati in un software di analisi di MS per identificare fosfopeptidi. Clicca qui per visualizzare una versione più grande di questa figura.
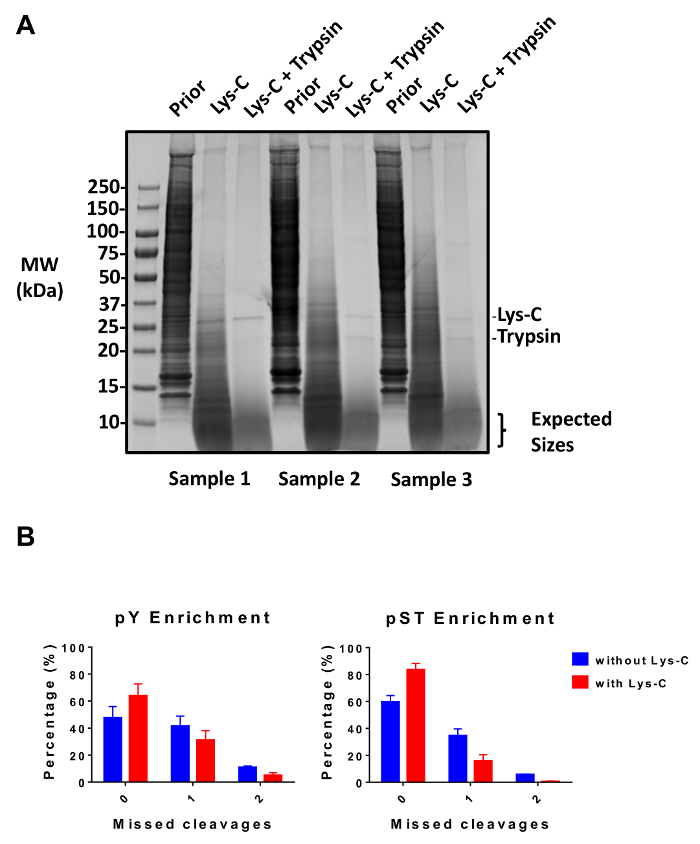
Figura 2: valutazione della digestione. (A) tre campioni con 12,5 µ g di lisato digestione pre-digestione, post-Lys-C e digestione della post-tripsina sono mostrati. Un test di gel-macchia di Coomassie Mostra una digestione pulita dopo l'uso sequenza di Lys-C e la tripsina. I marcatore di peso molecolare (MW) sono in kilodaltons (kDa). (B) A riduzione di fenditure perse è osservata dopo Lys-C è stato aggiunto al protocollo. La percentuale di fosfopeptidi senza fenditure perse aumentata dal 48% al 64% e dal 60% all'84% in media per preparazioni di arricchimento di pY e pST, rispettivamente. I grafici riassumono i dati ottenuti da due esperimenti eseguiti senza Lys-C e cinque esperimenti compiuti con Lys-C. Le barre di errore sono deviazioni standard che rappresenta pY 38 e 38 campioni pST da 2 esperimenti separati (senza Lys-C) e pY 62 60 campioni di pST da 5 esperimenti separati (con Lys-C). Clicca qui per visualizzare una versione più grande di questa figura.
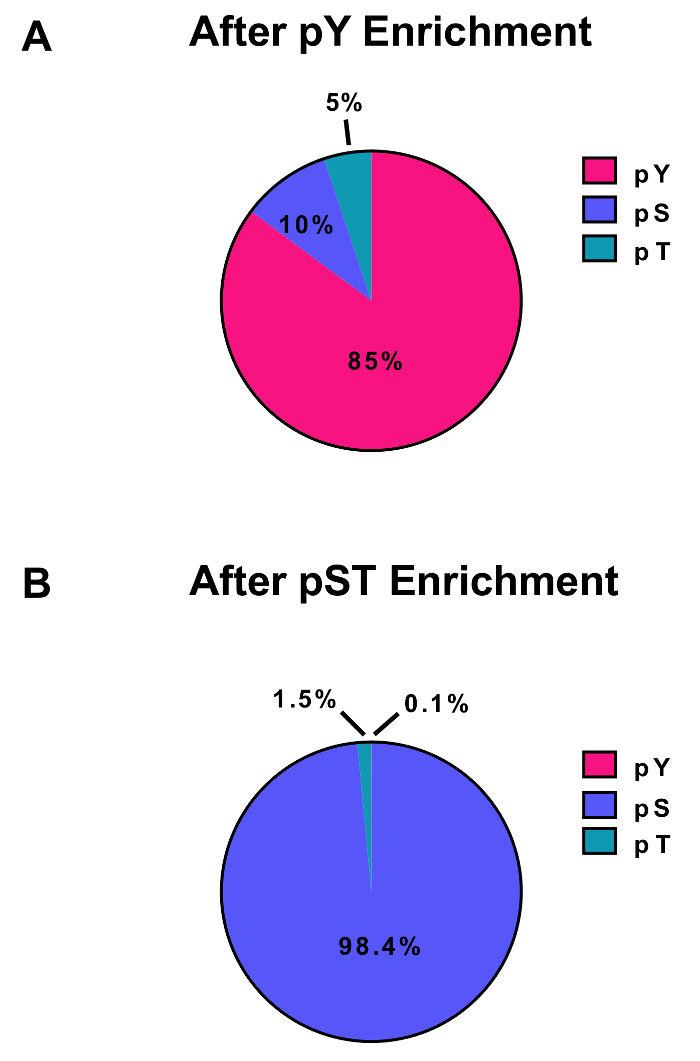
Figura 3: nell'arricchimento delle fosfopeptidi pY e pST. Questi pannelli mostrano le percentuali di pSTY fosfopeptidi da (A) il pY o (B) le preparazioni di arricchimento di pST. L'arricchimento di pY pY immunoprecipitazione e biossido di titanio ha provocato 85% fosfopeptidi essendo per pY peptidi, mentre solo lo 0,1% di fosfopeptidi nell'arricchimento pST sono pY. Questi valori sono stati disegnati dall'esaminare il Phospho (STY)Sites.txt file di rappresentante un esperimento dopo il filtraggio contaminanti, sequenze d'inversione e fosfopeptidi con probabilità di localizzazione meno di 0,75. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: Phosphopeptide arricchimento con biossido di titanio. (A) la percentuale di fosfopeptidi rilevati (rispetto al totale peptidi) da campioni in quattro esperimenti separati è indicata. (B), questo pannello mostra la composizione media del mono - doppio- e peptidi fosforilati in multi in quattro esperimenti separati. Le barre di errore in pannello A sono deviazioni standard. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5: previsto identificazioni phosphoresidue. (A), questo pannello mostra le probabilità di localizzazione di fosforilazione di ID da pY arricchimento (a sinistra) e l'arricchimento di pST (a destra). La percentuale media di ID che soddisfano il cutoff di probabilità > 0.75 è 93%, 75% e 52% per pY, pS e pT, rispettivamente. (B) la media numero di ID con un > 0.75 localizzazione probabilità è 300 per pY, 7.500 per pS e 640 in pT. (C), questo pannello mostra trame di violino del coefficiente percentuale di variazione (% CV) di fosfopeptidi. Una valutazione di % CV è stata eseguita solo se un valore di intensità del segnale è stato rilevato in ogni replica biologico o gruppo triplicata. Dati è stata presa da quattro esperimenti separati. Le barre di errore nei pannelli A e B sono deviazioni standard da 34 pY e 34 campioni di pST da 4 esperimenti separati. Clicca qui per visualizzare una versione più grande di questa figura.
| Buffer | Volume | Composizione | |
| Buffer di lisi del cloruro di guanidinio 6m | 50 mL | Cloruro di guanidinio 6m, 100 mM tris pH 8.5, 10 mM tris (2-carboxyethyl) fosfina, chloroacetamide 40 mM, 2 mM sodio ortovanadato, pirofosfato di sodio 2,5 mM, 1mm β-glicerofosfato, 500 mg n-octil-glicoside, ultra-acqua pura per volume | |
| pirofosfato di sodio 100 mM | 50 mL | 2,23 g sodio pirofosfato decaidrato, acqua ultra pura a volume | |
| Β-glicerofosfato di 1M | 50 mL | 15,31 g β-glicerofosfato, acqua ultra pura a volume | |
| 5% acido trifluoroacetico | 20 mL | Aggiungere 1 mL di acido trifluoroacetico 100% in 19 mL di acqua ultra-pura | |
| 0,1 acido trifluoroacetico % | 250 mL | Aggiungere 5 mL 5% di acido trifluoroacetico a 245 mL di acqua ultra-pura | |
| tampone di eluizione di pY | 250 mL | 0,1% trifluoroacetico acido, 40% acetonitrile, acqua ultra pura a volume | |
| tampone di eluizione di pST | 250 mL | 0,1% trifluoroacetico acido, 50% acetonitrile, acqua ultra pura a volume | |
| Buffer di associazione di IP | 200 mL | 50 mM tris-HCl pH 7.4, 50 mM di cloruro di sodio, acqua ultra pura a volume | |
| bicarbonato d'ammonio di 25 mM, pH 7.5 | 10 mL | Sciogliere 19,7 mg in 10 mL di acqua sterile ultra-puro, pH a 7,5 con soluzione di acido cloridrico 1 N (~ 10-15 µ l/10 ml di soluzione), rendere fresca | |
| 1M di tampone fosfato, pH 7 | 1.000 mL | 423 mL 1 M sodio fosfato monobasico, 577 mL 1 M sodio idrogeno fosfato | |
| Buffer di equilibrazione | 14 mL | 6,3 mL acetonitrile, 280 Acido trifluoroacetico 5% µ l, 1740 µ l acido lattico, acqua ultra pura 5,68 mL | |
| Buffer di risciacquo | 20 mL | 9 mL acetonitrile, 400 µ l 5% trifluoroacetico acido, 10,6 mL di acqua ultra-pura | |
| Soluzione di spettrometria di massa | 10 mL | 500 µ l acetonitrile, acido trifluoroacetico di 200 µ l 5%, acqua ultra pura 9,3 mL | |
| Tampone A | 250 mL | Diidrogenofosfato di 5 mM (pH 2,65), 30% acetonitrile, 5 mM di cloruro di potassio, acqua ultra-pura a volume | |
| Tampone B | 250 mL | Diidrogenofosfato di 5 mM (pH 2,65), 30% acetonitrile, 350 mM di cloruro di potassio, acqua ultra pura a volume | |
| 0,9% idrossido di ammonio | 10 mL | 300 μL 29,42% idrossido di ammonio, 9,7 mL di acqua ultra-pura | |
Tabella 1: buffer e soluzioni. Questa tabella mostra le composizioni del buffer e soluzioni utilizzate nel presente protocollo.
| Impostazioni di LC-MS/MS | ||
| Parametro | pY impostazione | pST impostazione |
| Caricamento (µ l) del campione | 5 | |
| Portata di carico (µ l/min) | 5 | |
| Gradiente della portata (nL/min) | 300 | |
| Sfumatura lineare (percentuale 0,16% acido formico, 80% ACN in 0,2% acido formico) | 4 - 15% per 5 min | 4 - 15% per 30 min |
| 15 - 50% per 40 min | 15 - 25% per 40 min | |
| 50 - 90% per 5 min | 25 - 50% per 44 min | |
| 50 - 90% per 11 min | ||
| Risoluzione di scansione completa | 120.000 | |
| Numero di ioni più intensi selezionati | 20 | |
| Energia di collisione relativa (%) (HCD) | 27 | |
| Esclusione di dinamica (s) | 20 | |
Tabella 2: impostazioni di LC-MS. Questo è un esempio di impostazioni di LC-MS in un esperimento di fosfoproteomica tipico fucile da caccia. I campioni sono stati caricati su una colonna di trappola. La trappola è stata portata in linea con una colonna analitica. Queste impostazioni sono state ottimizzate per l'utilizzo il sistema LC-MS elencato nella tabella materiali e reagenti. Queste impostazioni avrebbero bisogno di essere regolato per altri sistemi di LC-MS.
| Impostazioni dei parametri di MaxQuant | ||
| Impostazione | Azione | |
| Parametri specifici del gruppo | ||
| Tipo | Tipo | Selezionare Standard |
| Molteplicità | Impostare su 1 | |
| Modalità di digestione | Enzima | Selezionare tripsina/P |
| Max. fenditure perse | Impostato su 2 | |
| Modifiche | Modifiche variabile | Aggiungere fosfo (STY) |
| Privo di etichetta quantificazione | Privo di etichetta quantificazione | Selezionare IFQ |
| Conteggio di rapporto minimo IFQ | Impostare su 1 | |
| LFQ veloce | Spuntare le | |
| Varie | Ri-quantificare | Spuntare le |
| Parametri globali | ||
| Sequenze | File FASTA | Fasta selezionare file scaricato da UniProt |
| Modifiche fisse | Aggiungere Carbamidomethyl (C) | |
| Identificazione di adv. | Una partita tra le esecuzioni | Spuntare le |
| Finestra di tempo partita | Situato a 5 min | |
| Finestra di tempo di allineamento | Impostare a 20 min | |
| Abbinare le caratteristiche di non identificati | Spuntare le | |
| Quantificazione della proteina | Conteggio di rapporto min. | Impostare su 1 |
| Percorsi delle cartelle | Modificare di conseguenza | |
Tabella 3: le impostazioni di software di analisi MS. In MaxQuant, i parametri globali e gruppo-specifici in questa tabella sono stati selezionati o regolati. Tutti gli altri parametri è rimasto a default. Questi esperimenti sono stati condotti utilizzando la versione 1.5.3.30. I parametri possono variare da versione a versione e da software a software.
Discussione
Prima di utilizzare questo protocollo per arricchire per fosfopeptidi, un attento esame del disegno sperimentale è fondamentale. Utilizzando replicati biologici è un più costo-efficace utilizzo delle risorse di spettrometria di massa rispetto tecnici replicati. Il numero di repliche che sono necessarie dipenderà in parte la variabilità dei dati. Un recente studio ha dimostrato che, mentre aumentando il numero delle ripetizioni oltre le tre solo marginalmente aumenta il numero di identificazioni, il numero di identificazioni significative tra gruppi aumenta con più replica10.
A causa della bassa abbondanza di fosfoproteine nella cella, quantità sufficiente di proteina partenza sono necessari per ottenere un phosphoproteome globale da campioni di carcinoma della prostata in modalità discovery. In questi esperimenti, è stato utilizzato 5 mg di proteina. Circa cinque piatti 15 cm quasi confluenti di cellule forniscono proteine a sufficienza come input in questo protocollo, anche se si tratta di cella linea-dipendente. Per quanto riguarda il tessuto del tumore, del rendimento atteso di proteina è circa 6-8% del peso del tessuto. Nella cornice in vitro , un campione di controllo positivo da considerare è l'aggiunta di vanadato di 1 mM per 30 min prima della raccolta delle cellule. Vanadato, un inibitore di fosfatasi della proteina competitiva phosphotyrosyl, conserverà la fosforilazione della tirosina, aumentando così il numero di identificazioni di pY peptide29.
Digestione pulita è un passo fondamentale per massimizzare phosphopeptide identificazione. Oltre alla prova di macchia di Coomassie, la percentuale di mancata fenditure nei dati può essere utilizzata per valutare l'efficienza di digestione (Figura 2). Software di controllo di qualità è disponibile che analizza fenditure perse ed altre metriche per valutare MS dati qualità30. Mentre la tripsina è che le proteasi più comuni, alternative sono disponibili5 per colmare le lacune copertura nel proteoma dove peptidi triptici ottimale non possono essere generato31. Le impostazioni del software di analisi di MS sarebbero quindi bisogno di essere modificati di conseguenza per regolare le modifiche di proteasi.
Il protocollo si avvale di immunoprecipitazione (per arricchimento pY) così come biossido di titanio (TiO2) arricchire per fosfopeptidi. Approcci alternativi per arricchire per peptidi includono cromatografia di affinità immobilizzata di metallo (IMAC), altri ossidi metallici per cromatografia di affinità di ossido di metallo (MOAC) come idrossido di alluminio e la cattura di affinità dello ione del metallo a base di polimeri (PolyMAC) 5,12. Studi precedenti hanno dimostrato che diverse arricchimento dei metodi arricchisca per le diverse popolazioni di fosfopeptidi32. Per esempio, IMAC arricchisce più peptidi multi-fosforilati mentre MOAC preferibilmente arricchisce per peptidi fosforilati in mono33. I Risultati di rappresentante del presente protocollo riflettono questa osservazione (Figura 4B). Una recente pubblicazione ha dimostrato che la combinazione di IMAC e MOAC utilizzando un materiale ibrido potenzialmente potrebbe fornire una maggiore copertura di phosphopeptide specie34. Così, questo protocollo potrebbe essere modificato per utilizzare altri metodi di arricchimento in parallelo per consentire analisi fosfoproteomica ancora più complete.
La suite di software26 MaxQuant viene utilizzata per analizzare i dati di MS in questo protocollo, ma applicazioni commerciali35 sono anche disponibili per fosfopeptide identificazione e quantificazione. Per fosfopeptide identificazione, viene applicata una probabilità di localizzazione cutoff. Questo filtro viene eseguito per selezionare per fosfopeptidi con un'elevata probabilità (cioè, maggiore di 0,75) in phosphoresidue identificazione10,28. In altre parole, la probabilità sommata tutti i altri residui che potrebbero potenzialmente contenere il phospho-gruppo è inferiore a 0,25. Questa frequenza di taglio poteva essere sollevata per aumentare il rigore della selezione phosphopeptide. Per quanto riguarda il numero di identificazioni, il numero atteso di peptidi pY è nell'ordine delle centinaia, mentre il numero previsto di peptidi pST è in migliaia ad alte. Questi valori riflettono precedentemente osservati phosphoproteome distribuzione dove circa il 2%, 12% e 86% della phosphosites sono pY, pT e pS, rispettivamente28.
Se i passaggi di arricchimento pY e pST vengono eseguiti in parallelo, la procedura di preparazione del campione nel protocollo può essere completata in sei giorni. In abbinamento con il potente strumento di MS, protocolli di arricchimento phosphopeptide come questo forniscono un approccio globale per gli scienziati raccogliere i dati per analizzare il phosphoproteome nei loro campi rispettivi ricerca.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Ringraziamo i membri del laboratorio Drake per fornire consulenza e ingresso sul manoscritto. Ringraziamo anche i membri del biologico massa spettrometria Facility di Robert Wood Johnson Medical School e Rutgers, The State University of New Jersey, per fornire consulenza e l'esecuzione di spettrometria di massa sui nostri campioni. Larry C. Cheng è supportato dal National Institute of General Medical Sciences del National Institutes of Health, sotto numero premio T32 GM008339. Thomas G. Graeber è supportato dal NIH/NCI (SPORE in prostata cancro P50 CA092131; P01 CA168585) e un American Cancer Society Research Scholar Award (RSG-12-257-01-TBE). Justin M. Drake è supportato dal dipartimento della difesa prostata cancro ricerca programma W81XWH-15-1-0236, prostata Cancer Foundation Young Investigator Award, il New Jersey Health Foundation e un premio di precisione medicina iniziativa pilota dalla Rutgers Cancer Institute del New Jersey.
Materiali
| Name | Company | Catalog Number | Comments |
| Ultra-Low Temperature Freezer | Panasonic | MDF-U76V | |
| Freezer -20 °C | VWR | scpmf-2020 | |
| Swing rotor bucket | ThermoFisher Scientific | 75004377 | |
| Vacuum manifold | Restek | 26080 | |
| Lyophilizer | Labconco | 7420020 | |
| CentriVap Benchtop Vacuum Concentrator | Labconco | 7810010 | |
| End-over-end rotator | ThermoFisher Scientific | 415110Q | |
| Razor blade | Fisher Scientific | 620177 | |
| Amicon Ultra-15 Centrifugal Filter Units | Millipore Sigma | UFC901024 | |
| Glass culture tubes | Fisher Scientific | 14-961-26 | |
| Parafilm | Fisher Scientific | 13-374-12 | |
| 20G needle | BD | B305175 | |
| Kimwipes | Fisher Scientific | 06-666A | |
| Screw cap cryotube | ThermoFisher Scientific | 379189 | |
| Nunc 15 mL conical tubes | ThermoFisher Scientific | 12-565-268 | |
| Gel loading tips | Fisher Scientific | 02-707-181 | |
| Millipore 0.2 µm spin filter | Millipore Sigma | UFC30GVNB | |
| Low protein-binding Eppendorf tubes | Eppendorf | 22431081 | |
| anti-Phosphotyrosine, Agarose, Clone: 4G10 | Millipore Sigma | 16101 | |
| 27B10.4 antibody | Cytoskeleton | APY03-beads | |
| Peptide assay kit | Thermo Scientific | 23275 | Step 7 |
| TopTip | PolyLC Inc | TT200TIO.96 | Steps 5 and 9 |
| SCX columns (PolySULFOETHYL A) | PolyLC Inc | SPESE1203 | |
| 3 mL syringe | BD | 309657 | |
| Trifluoracetic Acid (TFA) | Fisher Scientific | PI-28904 | |
| Acetonitrile (ACN) | Fisher Scientific | A21-1 | |
| Lactic acid | Sigma-Aldrich | 69785-250ML | |
| Ammonium Hydroxide | Fisher Scientific | A669S-500 | |
| Potassium Phosphate Monobasic | Fisher Scientific | BP362-500 | |
| Potassium Chloride | Fisher Scientific | BP366-500 | |
| Calcium Chloride Dihydrate | Fisher Scientific | BP510-500 | |
| Tris Base | Fisher Scientific | BP152-5 | |
| Trypsin, TPCK Treated | Worthington Biochemicals | LS003740 | |
| Lysyl Endopeptidase | Wako Pure Chemical Industries, Ltd. | 125-05061 | |
| MonoTip | PolyLC Inc | TT200TIO.96 | Step 10 |
| ZipTip | MilliporeSigma | ZTC18S096 | Step 6 |
| nanoEase, MZ peptide BEH C18, 130A, 1.7 μm, 75 μm x 20 cm | Waters | 186008794 | Step 11: analytical column |
| Acclaim PepMap 100 C18 LC Columns | ThermoFisher Scientific | 164535 | Step 11: trap column |
| Ultimate 3000 RLSCnano System | Dionex | ULTIM3000RSLCNANO | Step 11 |
| Q Exactive HF | ThermoFisher Scientific | IQLAAEGAAPFALGMBFZ | Step 11 |
| MilliQ water | deionized water used to prepare all solutions and bufferes | ||
| Sonic Dismembrator | Fisher Scientific | FB-120 | sonicator |
| Polytron System PT | Kinematica AG | PT 10-35 GT | homogenizer |
Riferimenti
- Siegel, R. L., Miller, K. D., Jemal, A. Cancer statistics, 2018. CA: A Cancer Journal for Clinicians. 68 (1), 7-30 (2018).
- Paller, C. J., Antonarakis, E. S. Management of biochemically recurrent prostate cancer after local therapy: evolving standards of care and new directions. Clinical Advances in Hematology & Oncology. 11 (1), 14-23 (2013).
- Lowrance, W. T., Roth, B. J., Kirkby, E., Murad, M. H., Cookson, M. S. Castration-resistant prostate cancer: AUA guideline amendment 2015. The Journal of Urology. 195 (5), 1444-1452 (2016).
- Domon, B., Aebersold, R. Options and considerations when selecting a quantitative proteomics strategy. Nature Biotechnology. 28 (7), 710-721 (2010).
- Zhang, Y. Y., Fonslow, B. R., Shan, B., Baek, M. C., Yates, J. R. Protein analysis by shotgun/bottom-up proteomics. Chemical Reviews. 113 (4), 2343-2394 (2013).
- Bantscheff, M., Schirle, M., Sweetman, G., Rick, J., Kuster, B. Quantitative mass spectrometry in proteomics: a critical review. Analytical and Bioanalytical Chemistry. 389 (4), 1017-1031 (2007).
- Bantscheff, M., Lemeer, S., Savitski, M. M., Kuster, B. Quantitative mass spectrometry in proteomics: critical review update from 2007 to the present. Analytical and Bioanalytical Chemistry. 404 (4), 939-965 (2012).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513-2526 (2014).
- Rubbi, L., et al. Global phosphoproteomics reveals crosstalk between Bcr-Abl and negative feedback mechanisms controlling Src signaling. Science Signaling. 4 (166), ra18 (2011).
- Hogrebe, A., et al. Benchmarking common quantification strategies for large-scale phosphoproteomics. Nature Communications. 9 (1), 1045 (2018).
- Rush, J., et al. Immunoaffinity profiling of tyrosine phosphorylation in cancer cells. Nature Biotechnology. 23 (1), 94-101 (2005).
- Fila, J., Honys, D. Enrichment techniques employed in phosphoproteomics. Amino Acids. 43 (3), 1025-1047 (2012).
- Mertins, P., et al. Proteogenomics connects somatic mutations to signalling in breast cancer. Nature. 534 (7605), 55-62 (2016).
- Drake, J. M., et al. Phosphoproteome integration reveals patient-specific networks in prostate cancer. Cell. 166 (4), 1041-1054 (2016).
- Lue, H. W., et al. Metabolic reprogramming ensures cancer cell survival despite oncogenic signaling blockade. Genes & Development. 31 (20), 2067-2084 (2017).
- Francavilla, C., et al. Phosphoproteomics of primary cells reveals druggable kinase signatures in ovarian cancer. Cell Reports. 18 (13), 3242-3256 (2017).
- Zhang, H., et al. Integrated proteogenomic characterization of human high-grade serous ovarian cancer. Cell. 166 (3), 755-765 (2016).
- Skaggs, B. J., et al. Phosphorylation of the ATP-binding loop directs oncogenicity of drug-resistant BCR-ABL mutants. Proceedings of the National Academy of Sciences of the United States of America. 103 (51), 19466-19471 (2006).
- Zimman, A., et al. Activation of aortic endothelial cells by oxidized phospholipids: a phosphoproteomic analysis. Journal of Proteome Research. 9 (6), 2812-2824 (2010).
- Zimman, A., Berliner, J. A., Graeber, T. G. Phosphoproteomic analysis of aortic endothelial cells activated by oxidized phospholipids. Methods in Molecular Biology. , 53-69 (2013).
- Humphrey, S. J., Azimifar, S. B., Mann, M. High-throughput phosphoproteomics reveals in vivo insulin signaling dynamics. Nature Biotechnology. 33 (9), 990-995 (2015).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72, 248-254 (1976).
- Meyer, T. S., Lamberts, B. L. Use of coomassie brilliant blue R250 for the electrophoresis of microgram quantities of parotid saliva proteins on acrylamide-gel strips. Biochimica el Biophysica Acta. 107 (1), 144-145 (1965).
- Bergstrom Lind, S., et al. Immunoaffinity enrichments followed by mass spectrometric detection for studying global protein tyrosine phosphorylation. Journal of Proteome Research. 7 (7), 2897-2910 (2008).
- Pinkse, M. W., Uitto, P. M., Hilhorst, M. J., Ooms, B., Heck, A. J. Selective isolation at the femtomole level of phosphopeptides from proteolytic digests using 2D-NanoLC-ESI-MS/MS and titanium oxide precolumns. Analytical Chemistry. 76 (14), 3935-3943 (2004).
- Cox, J., Mann, M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nature Biotechnology. 26 (12), 1367-1372 (2008).
- Cox, J., et al. Andromeda: a peptide search engine integrated into the MaxQuant environment. Journal of Proteome Research. 10 (4), 1794-1805 (2011).
- Olsen, J. V., et al. Global, in vivo, and site-specific phosphorylation dynamics in signaling networks. Cell. 127 (3), 635-648 (2006).
- Huyer, G., et al. Mechanism of inhibition of protein-tyrosine phosphatases by vanadate and pervanadate. The Journal of Biological Chemistry. 272 (2), 843-851 (1997).
- Bielow, C., Mastrobuoni, G., Kempa, S. Proteomics quality control: quality control software for MaxQuant results. Journal of Proteome Research. 15 (3), 777-787 (2016).
- Swaney, D. L., Wenger, C. D., Coon, J. J. Value of using multiple proteases for large-scale mass spectrometry-based proteomics. Journal of Proteome Research. 9 (3), 1323-1329 (2010).
- Bodenmiller, B., Mueller, L. N., Mueller, M., Domon, B., Aebersold, R. Reproducible isolation of distinct, overlapping segments of the phosphoproteome. Nature Methods. 4 (3), 231-237 (2007).
- Leitner, A., Sturm, M., Lindner, W. Tools for analyzing the phosphoproteome and other phosphorylated biomolecules: a review. Analytica Chimica Acta. 703 (1), 19-30 (2011).
- Yang, D. S., et al. Design and synthesis of an immobilized metal affinity chromatography and metal oxide affinity chromatography hybrid material for improved phosphopeptide enrichment. Journal of Chromatography A. 1505, 56-62 (2017).
- Al Shweiki, M. R., et al. Assessment of label-free quantification in discovery proteomics and impact of technological factors and natural variability of protein abundance. Journal of Proteome Research. 16 (4), 1410-1424 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon