Method Article
Phosphopeptide Bereicherung gepaart mit quantitativen markierungsfreie Massenspektrometrie zu untersuchen, die Phosphoproteome bei Prostatakrebs
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt ein Verfahren zum Extrahieren und Phosphopeptides von Prostatakrebs-Zelllinien oder Gewebe für eine Analyse der Phosphoproteome per Massenspektrometrie-basierte Proteomics zu bereichern.
Zusammenfassung
Phosphoproteomics umfasst die groß angelegte Untersuchung der phosphorylierten Proteine. Protein-Phosphorylierung ist ein entscheidender Schritt in vielen Signal Transduction Bahnen und ist streng reguliert durch Kinasen und Phosphatasen. Daher können Charakterisierung der Phosphoproteome Einblicke in neue Targets und Biomarker für die onkologische Therapie zu identifizieren. Massenspektrometrie bietet eine Möglichkeit, weltweit zu erkennen und zu quantifizieren, Tausende von einzigartigen Phosphorylierung Veranstaltungen. Phosphopeptides sind jedoch viel weniger reichlich vorhanden als Phosphopeptides, so dass biochemische Analyse schwieriger. Um diese Einschränkung zu umgehen, müssen Methoden zur Phosphopeptides vor der Massenspektrometrie-Analyse zu bereichern. Wir beschreiben eine Prozedur, um zu extrahieren und zu verdauen Proteine aus Gewebe, Peptide, gefolgt von eine Bereicherung für die Phosphotyrosine (pY) und Phosphoserine/Threonin (pST) Peptide mit einer Antikörper-basierten und/oder Titandioxid (TiO2) Ausbeute-Basis Anreicherung-Methode. Nach der Probenvorbereitung und Massenspektrometrie wir anschließend identifizieren und quantifizieren Phosphopeptides mit flüssigen Gaschromatographie-Massenspektrometrie und Analysesoftware.
Einleitung
Einen geschätzten 165.000 Neuerkrankungen und etwa 29.000 Todesfälle treten im Jahr 2018 wegen Prostatakrebs, repräsentieren die am häufigsten auftretende Krebsart und die zweithäufigste Ursache für krebsbedingte Todesursache bei Männern in den Vereinigten Staaten1. Frühe Stadien von Prostatakrebs sind behandelbar mit Resektion oder Strahlung Therapie der Orgel beschränkt Krankheit, wo die Zehn-Jahres-Rezidivrate ist zwischen 20 % und 40 % für Patienten, die Prostatektomie unterzogen und zwischen 30 % und 50 % bei Patienten, die Strahlung zu erhalten Therapie2. Da Prostatakrebs auf Androgen-Signalisierung für Wachstum beruht, sind chirurgische und chemische Kastration Therapien auch für Hochrisiko-Patienten eingesetzt. Rückfall tritt jedoch wenn der Krebs nicht mehr auf Androgen-Deprivation Therapie reagiert, wie biochemisches Rezidiv belegt, wo die Prostata-spezifisches Antigen im Serum aufersteht. An dieser Stelle werden in der Reihenfolge Metastasen oft auch erkannt. Diese fortgeschrittenen Stadium, genannt metastasiertem Kastration-resistenten Prostatakrebs stellt die tödliche Form der Krankheit, wo die Prognose eine mediane Überlebenszeit von weniger als zwei Jahren3ist. Nur wenige Behandlungsmöglichkeiten gibt es im späten Stadium der Erkrankung, einschließlich der zweiten Generation Antiandrogene wie enzalutamid sowie Abiraterone und Taxan-basierte Chemotherapie wie Docetaxel. Trotz verfügbaren Behandlungen schreitet die Krankheit oft. Daher sind die Erforschung und Entwicklung von neuartigen Behandlungsmethoden notwendig, um die Versorgung von Prostatakrebs-Patienten mit fortgeschrittener Erkrankung zu verbessern.
Massenspektrometrie (MS)-basierte Ansätze bieten eine Gesamtanalyse des Proteoms durch die Erkennung von Hunderten bis Tausenden von Peptid Analyten4. Insbesondere können die Identifikation und Quantifizierung von Tausenden von Peptiden4,5Entdeckung Proteomics, auch bekannt als datenabhängiges Erwerb (DDA), erzielt werden. MS-basierte Erkennung Proteomics können weiter in Top-Down-Proteomics, wo intakte Proteine auszeichnen, und unten nach oben (auch bekannt als Schrotflinte) Proteomics, wo Peptide analysiert werden, um Proteine5charakterisieren abgegrenzt werden. So findet Schrotflinte Proteomics, ein Proteolyse Schritt in der Probenvorbereitung vor der MS-Analyse, Proteine in Peptide zu Spalten. Am Ende wird eine Suche in der Datenbank durchgeführt, um die Peptide zurück an die Proteine zur Identifikation Karte. Markierungsfreie sowie mehrere Isotop-Kennzeichnung [z.B., stabile Isotope, die Kennzeichnung von Aminosäuren in der Zellkultur (SILAC)] Methoden lässt sich quantitativ Peptide zwischen Proben6,7vergleichen. Während Isotop Kennzeichnung Techniken der Goldstandard, markierungsfreie Methoden zeigten ähnliche Quantifizierung Genauigkeiten8,9 und vergleichbare Kompromisse zwischen Empfindlichkeit und Spezifität10. Markierungsfreie Quantifizierung bietet eine größere Abdeckung und erlaubt Vergleiche zwischen viele weitere Proben, während Label-basierte Methoden durch Kosten und multiplexing Kapazitäten6,7,8begrenzt sind.
Darüber hinaus einsetzbar Schrotflinte MS auch Post-translationalen Modifikationen (PTMs) wie z. B. Phosphorylierung11zu befragen. Aufgrund der niedrigeren stöchiometrischen Phosphopeptides im Vergleich zum gesamten Peptide werden verschiedene Methoden eingesetzt, um für Phosphopeptides, einschließlich Antikörper-basierten Immunopräzipitation Phosphotyrosine (pY) Peptide, Titandioxid (TiO2 bereichern ), und Metall Affinität Chromatographie (IMAC)5,12immobilisiert. Weil Protein Phosphorylierung ein wichtiger Schritt in vielen Zelle Signalwege, Schrotflinte Phosphoproteomics Zelle Änderungen bei verschiedenen Krebsarten ist, einschließlich Brust13, Prostata14, renale15untersuchen Forscher ermöglicht, und Eierstockkrebs,16,17 , Krebsbiologie besser zu verstehen und mögliche neue Ziele für die Therapie zu identifizieren.
Diese markierungsfreie Schrotflinte Phosphoproteomic Methode wurde gebaut und raffinierte basierend auf Vorarbeiten durch die Graeber Gruppe18,19,20. Dieses Protokoll beginnt mit der Beschreibung der Gewinnung und die Verdauung von Proteinen und Phosphoproteine aus Gewebe in Peptide. Wir dann ausführlich die Anreicherung von pY Peptide mit spezifischen Phosphotyrosine Antikörper und TiO2. Wir beschreiben auch die Anreicherung der Phosphoserine/Threonin (pST) Peptide mit starken KATIONENAUSTAUSCH (SCX) gefolgt von TiO2. Dieses Protokoll schließt mit der Vorlage von Mustern in eine MS-Einrichtung und die Verwendung von MS-Analyse-Software zu identifizieren und zu quantifizieren, Phosphopeptides und ihre entsprechenden Phosphoproteine. Die Anwendung dieses Protokolls kann Prostatakrebs in anderen Krebsarten und Felder außerhalb der Onkologie hinausgehen.
Protokoll
Experimente mit Xenograft Tumoren wurden von der Rutgers University institutionelle Animal Care und Nutzung als Set her nach den Richtlinien des National Institutes of Health genehmigt.
(1) Proteingewinnung
- Bereiten Sie Lyse Puffer (Tabelle 1). (Das Volumen hängt die Anzahl der Samples, geerntet zu werden.) Fahren Sie für in-vitro- Zellproben mit Schritt 1.2. Fahren Sie für Tumorgewebe mit Schritt 1.3.
-
Ernte der Zellen
- Sammeln Sie die Zellen in einem 50 mL konische Röhrchen und drehen sie bei 700 X g für 5 min bei 4 ° C. Verwerfen Sie den überstand und halten Sie das Pellet auf Eis. Wiederholen Sie diesen Schritt für alle Gerichte, die Zellen in einem Pellet zu sammeln. (In der Regel ca. 5 fast konfluierende 15-cm-Gerichte der Zellen für 5 mg Protein benötigt werden, aber dies kann abhängig von der Zell-Linie und muss von jedem Ermittler empirisch bestimmt werden.)
- Waschen Sie das Pellet mit 30 mL gekühlten Phosphat-gepufferte Kochsalzlösung (PBS) und Spin bei 700 X g für 5 min bei 4 ° C vor dem Absaugen der PBS. Hinzugeben Sie 1,5 mL Lyse Puffer pro 5 mg Protein verwendet, um die Zelle Pellet. Pipettieren nach oben und unten ein paar Mal. Fahren Sie mit Schritt 1.4.
-
Ernte von Gewebe
- Wiegen des Tumors und 2 mL eiskaltes Lyse Puffer für jede 100 mg des Gewebes im Reagenzglas Kultur. (In der Regel wird 50 bis 150 mg Gewebe Frischgewicht benötigt.)
- Homogenisieren der lysate mit einem Handgerät oder Benchtop Homogenisator (Puls 2 X für 15 s.) Reinigen Sie den Homogenisator vor der ersten Probe und zwischen den Proben mit 10 % Bleichmittel, 70 % Ethanol und deionisiertes Wasser in Folge.
- Zu reduzieren und Alkylat, homogenisierten Proben bei 95 ° C für 5 min erwärmen. Anschließend kühlen sie für 15 min auf Eis. Auf dem Eis, beschallen die lysate 3 X (d.h., Pulse für 30 s mit 60 s Pause zwischen den Pulsen). Die Probe sollte an dieser Stelle nicht zähflüssig oder klumpig sein. Hitze der lysate bei 95 ° C für 5 min21.
- Zentrifuge lysate in der gleichen Beschallung Tube mit einem Swing-Eimer-Rotor bei 3.500 x g bei 15 ° C für 15 min. überstand zu sammeln und entsorgen das Pellet.
- Bestimmen Sie die Proteinkonzentration durch Ausführen einer Bradford-Test22. Bei Bedarf verdünnen Sie lysate bis 5 mg/mL mit einer Lyse-Puffer. Bei-20 ° c Lagern
Hinweis: Das Experiment kann hier angehalten werden. Die Proben bei-80 ° C eingefroren und zu einem späteren Zeitpunkt fortsetzen.
(2) lysate Verdauung
- Die Probe verdünnen 12-fachen mit 100 mM Tris (pH = 8,5), die Guanidinium zu reduzieren. Alle Proben auf derselben Festplatte zur Minimierung der Auswirkungen der ungleichen Verdauung zu verdünnen. Speichern Sie 12,5 µg von den unverdauten lysate auf ein Coomassie gefärbt Gel23bestätigen.
- Für 5 mg Protein, fügen Sie 10 µg Lysyl Endopeptidase (Lys-C) und inkubieren Sie es bei Raumtemperatur für ca. 5-6 h. Adjust pH bis 8,0 durch untitrated Hinzufügen von 1 M Tris (pH ~ 11).
- Bereiten Sie 1 mg/mL L-1-Tosylamido-2-phenyl Chloromethyl Keton (TPCK)-Trypsin in 1 mM HCl (mit 20 mM CaCl2) behandelt. Fügen Sie die Trypsin bei einem Trypsin: Protein-Verhältnis von 1: 100 und bei 37 ° C für 3 h inkubieren.
- Fügen Sie die gleiche Menge an frischer Trypsin wie in Schritt 2.3. Inkubieren Sie es über Nacht bei 37 ° C.
- Speichern Sie 12,5 µg der verdauten lysate, die komplette Verdauung auf ein Coomassie gefärbt Gel23zu bestätigen.
3. Phase Extraktion umkehren
- Das lysate Volumen aufnehmen. Filtern Sie die Probe mithilfe eines 15 mL 10 kDa cutoff-Filters. Zentrifugieren Sie die Probe bei 3500 x g mit der Schaukel-Eimer-Rotor (oder 3.500 x g in einem festen Winkel Rotor) bei 15 ° C, bis das Retentat Volumen weniger als 250 µL beträgt (Dies dauert ca. 45-60 min). Die Flow-through zu sammeln Sie und entsorgen Sie das Retentat.
Hinweis: Das Experiment kann hier angehalten werden. Die Proben bei-80 ° C eingefroren und zu einem späteren Zeitpunkt fortsetzen. - Um die Probe Ansäuern, fügen Sie ca. 20 µL 5 % Trifluoroacetic Säure (TFA) pro mL lysate. Mischen Sie gut und Messen Sie den pH-Wert der Probe mittels pH-Streifen. Justieren pH bis 2,5 mit 5 % TFA.
- Verbinden Sie das kürzere Ende einer C-18 Spalte vielfältige Vakuum. Legen Sie das Vakuum zwischen 17 und 34 kPa (oder entsprechend den Anweisungen des Herstellers). Verwendung von glaspipetten , nasse Spalte mit 3 mL 100 % Acetonitril (ACN). Lassen Sie sich nicht die Spalte trocken.
- Verwendung von glaspipetten , equilibrate der Spalte mit 6 mL 0,1 % TFA als 2 x 3 mL angewendet. Laden Sie die gesäuerte Probe in der Spalte. Fügen Sie zu einem Zeitpunkt nicht mehr als 3 mL. Passen Sie das Vakuum auf etwa 1 bis 2 Tropfen pro Sekunde.
- Verwendung von glaspipetten , waschen Sie die Spalte mit 9 mL 0,1 % TFA als 3 x 3 mL angewendet. Die Spalte mit 2 mL 40 % eluieren ACN, 0,1 % TFA. Sammeln Sie zwei 2 mL Fraktionen in Glasröhren Kultur . Entsorgen Sie die Spalte.
- Das Eluat Rohre mit Parafilm abdecken und 3-5 Lochen auf dem Cover mit einer 20G-Nadel. Frieren Sie das Eluat auf Trockeneis für mindestens 30 min so lange ein, bis es vollständig fest ist.
- Die Fraktionen über Nacht lyophilize. Am nächsten Tag sicherstellen Sie, dass die Proben vor dem Anhalten der Gefriertrockner vollständig trocken sind. Speichern Sie die Rohre in einem 50 mL konische Röhrchen mit zarten Tücher bei-80 ° C.
Hinweis: Das Experiment kann hier angehalten werden.
4. Immunopräzipitation und Anreicherung von pY Peptide24
- Aufschwemmen des lyophilisierten Pulvers mit 0,5 mL eiskaltes Immunopräzipitation (IP) Bindung Puffer in jeder Bruchteil. Bündeln Sie die Bruchteile von 0,5 mL Wiederfreisetzung Volumen aus der zweiten Fraktion auf der ersten Fraktion übertragen und speichern Sie die PIPETTENSPITZE. Energisch Wirbel (statt pipettieren rauf und runter) um sicherzustellen, dass die Probe wird vor der Übertragung auf eine 3,6 mL Schraubverschluss Cryotube vollständig aufgelöst.
- Wie in Schritt 4.1., spülen Sie die Lyophilisation Rohre mit einem anderen 0,5 mL IP-Bindung Puffer (Tabelle 1) in jedem Röhrchen. Übertragen Sie die Lösung mit dem 3,6 mL Schraubverschluss Rohr unter Verwendung der gleichen PIPETTENSPITZE Probenverlust zu minimieren. Wiederholen Sie die Spülung 1 x mehr, so dass die endgültige Wiederfreisetzung Volumen 2 mL (für 5 mg Protein). Messen Sie den pH-Wert der Probe um sicherzustellen, dass es ungefähr 7.4 ist. Wenn es zu sauer wird, fügen Sie iterativ 10 µL 1 M Tris (untitrated, pH ~ 11). Wenn es zu einfach ist, fügen Sie iterativ 10 µL verdünnter HCl (01:25 oder 1: 100).
-
Vorspülen Sie die pY-Perlen (für 5 mg beginnend lysate)
- je Probe sind 25 µg 4 10 Antikörper und 12,5 µg 27B10.4 Antikörper erforderlich. Nach der Verwendung einer p200 pipette mit einer abgeschnittenen Spitze zu übertragen die Antikörper in separaten Mikrozentrifugenröhrchen, waschen Sie die Antikörper mit 450 µL eiskalte IP-Bindung Puffer 2 X. Zentrifugieren sie 100 X g für 1 min bei 4 ° C und absaugen, überstand.
- Aufzuwirbeln Sie die Perlen auf ein Lager Konzentration von 0,5 mg/mL mit IP-Bindung Puffer. (Haben Sie keine Wirbel die Perlen.) Nach Aliquotierung der notwendigen Gülle (50 µL 4 10 Antikörper Gülle und 25 µL 27B10.4 Antikörper Gülle pro Probe) in ein einzelnes Rohr spin-down Lager Zentrifuge Schläuche am 200 X g für 1 min bei 4 ° C. Waschen Sie die Seitenwände mit überstand vor der Rückkehr der Perlen nach Lagerung im Kühlschrank.
- Die resuspendierte Beispiellösung in der Schraubkappe Cryoröhrchen vorgewaschen pY Perlen hinzufügen. Brüten sie über Nacht bei 4 ° C auf eine durchgängige über Rotator.
- Legen Sie die Schraubkappe Cryoröhrchen in ein 50 mL Zentrifugenröhrchen mit einem feinen Tuch ausgekleidet. Spin-down der Perlen bei 100 X g für 1 min. Speichern der Überstand, die verwendet werden, zu bereichern für PST-Peptide. (Die Bereicherung für pST beginnt bei Schritt 7 und parallel zu den pY-Peptid-Verarbeitung durchgeführt werden kann).
- Die Perlen mit 300 µL IP-Bindung Puffer aufzuwirbeln. Übertragen Sie sie auf einem 2 mL Microcentrifuge Schlauch und drehen Sie sie nach unten bei 100 X g für 1 min bei 4 ° C.
- Spülen Sie die Inkubation Rohr 3 X mit 200 µL des IP-Bindung Puffer. Übertragen Sie den Inhalt mit dem gleichen Microcentrifuge Schlauch jedes Mal. Dann drehen sie sich nach unten.
- Die Perlen in den Microcentrifuge Schlauch 3 X mit 500 µL IP-Bindung Puffer und drehen sie sich bei 100 X g für 1 min waschen. Dann waschen Sie die Perlen 4 X mit 450 µL 25 mM NH4HCO3, pH 7,5 und Spin sie 100 X g für 1 min. verwenden Sie ein frisches 25 mM NH4HCO3 -Lösung von jedesmal Pulver.
- Zentrifugieren Sie den Perlen 1.500 x g für 1 min. Einsatz einen Gel-laden-Tipp den überstand vollständig zu entfernen, indem man die Spitze der Gel-Loading Spitze leicht unter den Perlen-Oberfläche.
- Fügen Sie 4 x die Wulst Volumen von 0,1 % TFA, die Perlen (d. h., fügen Sie 300 µL von 0,1 % TFA für 75 µg pY Wulst Gülle). Gut mischen und die Mischung in einem Thermomixer bei 1.000 u/min für 15 min bei 37 ° c inkubieren
- Übertragen Sie die Wiederfreisetzung auf einen 0,2 µm Spin Filter. Schnell spin-down der Elution Röhre und übertragen das Restvolumen an den gleichen Spin Filter mit einem P10 pipettieren. Spin-down der Spin-Filter bei 850 X g für 1 min. die Elution auf eine geringe Proteinbindung Microcentrifuge Schlauch übertragen. Über Nacht bei 40 ° C und mit einer Wärme-Zeit von 300 min konzentrieren Vakuum das Eluat zu Trockenheit.
Hinweis: Das Experiment kann hier angehalten werden. Die Proben bei-80 ° C eingefroren und zu einem späteren Zeitpunkt fortsetzen.
(5) Titandioxid Anreicherung25 PJ Peptide
- Aufschwemmen der getrockneten unten Phosphopeptides in 200 µL 50 % ACN, 0,1 % TFA. Wirbel und Zentrifugieren sie bei 10.000 x g 30 S. Wiederholung dieser 1 X, um sie gut aufzuwirbeln.
- Vorbereitung der TiO2 Perlen in Tipps, die eine für 200 µL Proben Kapazität enthalten.
- Klopfen Sie leicht auf der kleinen Tipp-Seite der Spitze, das Material zu diesem Zweck zu bewegen. Spülen Sie die Spitze durch Zugabe von 200 µL 100 % ACN, gefolgt von invertieren die Spitze und streichen das schmale Ende um die Flüssigkeit auf die Kappe zu verschieben.
- Mit einer Rasierklinge, schneiden Sie die kleinen Spitze der Spitze und legen Sie es über eine geringe Proteinbindung Rohr. (Verwenden Sie Polystyrol Rohre wie die TiO2 an den Seiten des Rohres wird.) Entfernen Sie die Kappe und einfügen Sie einer Mikropipette um die verbleibenden ACN zu stürzen. Wiederholen Sie das Waschen mit 200 µL 100 % ACN. Die TiO2-Perlen befinden sich jetzt in das geringe Proteinbindung Rohr für die folgenden Schritte.
- Voraussetzung TiO2 mit 500 µL 100 % ACN 2 X. Pipettieren sie die Perlen mit dem Lösungsmittel zu vermischen. Zentrifugieren sie 100 X g für 1 min.
- TiO2 mit 500 µL 0,2 M-Natrium-Phosphat-Puffer (pH ~ 7) Zustand 2 X. Die Perlen mit 300 µL Gleichgewichtherstellung Puffer 3 X waschen. Da TiO2 sehr dicht ist, werden die Perlen schnell einleben.
- Fügen Sie 400 µL 50 % ACN, 0,1 % TFA in das geringe Proteinbindung Rohr, gefolgt von Hinzufügen von 84 µL der Milchsäure. Übertragen Sie die resuspendierte Phosphopeptides in das geringe Proteinbindung Rohr und inkubieren sie für 1 h bei Raumtemperatur mit einer durchgängigen über Rotator.
- Zentrifugieren Sie die Perlen bei 100 X g für 1 min zu ihnen Pellets. Waschen Sie sie mit 300 µL Gleichgewichtherstellung Puffer (Tabelle 1) 2 X und drehen Sie sie nach unten bei 100 X g für 1 min.
- Spülen Sie die Perlen mit 300 µL des ausspülens Puffer 2 X. Übertragen Sie sie auf einen 0,2 µm Spin Filter. Drehen sie mit 1.500 x g für 1 min.
- Übertragen Sie die Filtereinheit auf eine saubere 1,5 mL geringe Proteinbindung Rohr. Eluieren Inhalt 2 X mit 200 µL von 0,9 % NH3 H2O. messen den pH-Wert mit pH-Streifen, die zwischen 10 und 11 werden sollte. Vakuum zu konzentrieren das Eluat zu Trockenheit über Nacht das Ammoniak verdampft.
(6) Entsalzung pY Peptide für MS Analyses
- Wiederherstellen der Phosphopeptides mit 15 µL von 0,1 % TFA durch aufschütteln und Zentrifugieren sie bei 10.000 x g für 30 s, um sie erneut. Wiederholen Sie diese 1 X, um sie gut aufzuwirbeln. Nicht nach oben und unten pipette.
- Reinigen Sie die Probe mit einer C-18 Spitze mit einem Bindungskapazität von 5 µg und folgen Sie Protokoll des Herstellers.
- Trocknen Sie vollständig die Elution Lautstärke durch Vakuum Konzentration. Dieser Vorgang dauert ca. 1-2 h Aufschwemmen der getrockneten Phosphopeptides in 12,5 µL Massenspektrometrie-Lösung (siehe Tabelle 1) (oder wie empfohlen von der Forscher MS Proteomics core Facility). Wirbel und kurz die Lösung spin-down bei 10.000 x g 30 S. Wiederholung dieser 2 X zu ihnen gut aufzuwirbeln. Die Proben sind bereit für die Einreichung einer Massenspektrometrie-Anlage (Schritt 11).
Hinweis: Die folgenden Schritte unten beziehen sich auf PST-Peptid Anreicherung nur.
7. Phase Extraktion von PST-Peptide umkehren
- Messen Sie die Peptid-Konzentration des Überstands gewonnenen Schritt 4.6 durch ein Peptid-Test durchführen. Eine ausreichende Menge für die PST-Massenspektrometrie beträgt 2,5 mg.
- Justieren pH bis 3.5 mit 5 % TFA.
- Verbinden Sie das kürzere Ende einer C-18 Spalte vielfältige Vakuum. Legen Sie das Vakuum zwischen 17 und 34 kPa (oder entsprechend den Anweisungen des Herstellers). Nasse Spalte mit 3 mL 100 % ACN. Lassen Sie sich nicht die Spalte trocken.
- Equilibrate der Spalte mit 6 mL 0,1 % TFA als 2 x 3 mL angewendet. Laden Sie die gesäuerte Probe in der Spalte. Fügen Sie zu einem Zeitpunkt nicht mehr als 3 mL. Passen Sie das Vakuum auf etwa 1 bis 2 Tropfen pro Sekunde.
- Waschen Sie die Spalte mit 9 mL 0,1 % TFA als 3 x 3 mL angewendet. Die Spalte mit 2 mL 40 % eluieren ACN, 0,1 % TFA. Sammeln Sie zwei 2 mL Fraktionen in Glasröhren Kultur. Entsorgen Sie die Spalte.
- Das Eluat Rohre mit Parafilm abdecken und 3-5 Lochen auf dem Cover mit einer 20G-Nadel. Frieren Sie das Eluat auf Trockeneis für mindestens 30 min so lange ein, bis es vollständig fest ist.
- Lyophilize der gewählten Fraktionen über Nacht. Am nächsten Tag sicherstellen Sie, dass die Proben vor dem Anhalten der Gefriertrockner vollständig trocken sind. Speichern Sie die Rohre in einem 50 mL konische Röhrchen mit zarten Tücher bei-80 ° C.
Hinweis: Das Experiment kann hier angehalten werden.
8. starke KATIONENAUSTAUSCH (SCX) PST-Peptide
- Aufschwemmen Sie lyophilisierter Peptide in 2 mL Puffer A (Tabelle 1). Bündeln Sie die Brüche für jede Probe. (Die Lösung trüb sein.)
- Bereiten Sie das Vakuum Krümmer. Verbinden Sie eine SCX-Spalte mit einer 3 mL Spritze mit den Kolben entfernt. Legen Sie das Vakuum zwischen 17 und 34 kPa (oder entsprechend den Anweisungen des Herstellers).
- Zustand der SCX-Spalte mit 4 mL von ACN, gefolgt von 4 mL Puffer A.
- 2 mL der Probe aus Schritt 8.1 Laden und das Eluat sofort zu sammeln. 3 mL umlenkern (80.9:19.1) Puffer zu laden und das Eluat zu sammeln. Bündeln sie die Eluate von jeder Probe und aliquoten in 2 mL geringe Proteinbindung Röhren.
- Vakuum konzentrieren sich alle Proben bis ca. 30 % des Volumens bleibt. (Dieser Schritt dauert ca. 2-4 h.) Bündeln Sie die Aliquote in 1 geringe Proteinbindung Röhre für jede Probe.
- Verbinden Sie das kürzere Ende einer C-18 Spalte vielfältige Vakuum. Legen Sie das Vakuum zwischen 17 und 34 kPa (oder entsprechend den Anweisungen des Herstellers). Nasse Spalte mit 3 mL 100 % ACN 2 X. Tun nicht die Spalte trocken lassen.
- Equilibrate die Spalte mit 3 mL 0,1 % TFA 2 X. Laden Sie das Beispiel in der Spalte. Fügen Sie zu einem Zeitpunkt nicht mehr als 3 mL. Passen Sie das Vakuum auf etwa 1 bis 2 Tropfen pro Sekunde.
- Waschen Sie die Spalte mit 3 mL 0,1 % TFA 2 X. Die Spalte mit 4 mL 50 % eluieren ACN, 0,1 % TFA.
9. Titandioxid-Anreicherung von PST-Peptide
-
Vorbereitung der TiO2 Perlen in Tipps, die eine für 200 µL Proben Kapazität enthalten
- Klopfen Sie leicht kleiner Tipp an die Spitze, die Perlen zu diesem Zweck zu bewegen. Entfernen Sie die Kappe und Gießen Sie die Perlen in einem Polypropylen 15 mL konische Rohr.
- Spülen Sie die Spitze durch Zugabe von 200 µL 100 % ACN, invertieren die Spitze ein paar Mal und streichen das schmale Ende um die Flüssigkeit auf die Kappe zu verschieben. Mit einer Rasierklinge, schneiden Sie die kleinen Spitze der Spitze und legen Sie es über das Polypropylen 15 mL konische Rohr. Entfernen Sie die Kappe und einfügen Sie einer Mikropipette um die verbleibenden ACN zu stürzen. Wiederholen Sie das Waschen mit 200 µL 100 % ACN. Die TiO2 Perlen befinden sich jetzt in der 15 mL konische Rohr für die folgenden Schritte.
- Voraussetzung TiO2 mit 500 µL 100 % ACN 2 X. Pipettieren sie die Perlen mit dem Lösungsmittel zu vermischen. Zentrifugieren sie 100 X g für 1 min.
- TiO2 mit 500 µL 0,2 M-Natrium-Phosphat-Puffer (pH ~ 7) zweimal Zustand. Die Perlen mit 300 µL Gleichgewichtherstellung Puffer 3 X waschen.
- Übertragen der eluierten Phosphopeptides in das Polypropylen 15 mL konische Röhrchen. Hinzufügen von 560 µL von Milchsäure und 1 h bei Raumtemperatur mit einer durchgängigen über Rotator inkubieren.
- Zentrifugieren Sie die Mischung bei 100 X g für 1 min um die Perlen zu Pellets. Waschen Sie sie mit 300 µL Gleichgewichtherstellung Puffer (Tabelle 1) 3 X. Mit 100 X g für 1 min nach unten drehen.
- Spülen Sie die Perlen mit 300 µL des ausspülens Puffer 2 X. Übertragen Sie sie auf einen 0,2 µm Spin Filter. Mit 1.500 x g für 1 min nach unten drehen.
- Übertragen Sie die Filtereinheit auf eine saubere 1,5 mL geringe Proteinbindung Rohr. Eluieren Inhalt 2 X mit 200 µL von 0,9 % NH3 H2O. Lassen Sie die Lösung auf die Phosphopeptides für 2 min sitzen, bevor sie eluierenden. Messen Sie den pH-Wert, die zwischen 10 und 11 werden sollte.
- Vakuum zu konzentrieren das Eluat zu Trockenheit über Nacht das Ammoniak verdampft.
10. Entsalzung PST-Peptide für MS-Analysen
- Klopfen Sie leicht auf der kleinen Tipp-Seite der Spitze, das Material zu diesem Zweck zu bewegen. Spülen Sie die Spitze durch Zugabe von 200 µL 100 % ACN, gefolgt von invertieren die Spitze und streichen das schmale Ende um die Flüssigkeit auf die Kappe zu verschieben.
- Schneiden Sie die kleinen Spitze der Spitze mit einer Rasierklinge, und legen Sie es über ein Polypropylen 15 mL konische Rohr. Entfernen Sie die Kappe und einfügen Sie einer Mikropipette um die verbleibenden ACN zu stürzen. Wiederholen Sie das Waschen mit 200 µL 100 % ACN. Die TiO2-Perlen befinden sich nun im Polypropylen 15 mL konische Röhrchen für die folgenden Schritte.
- Reinigen Sie die Probe mit einer C-18 Spitze mit einem Bindungskapazität von 100 µg. (folgen Sie den Anweisungen des Herstellers.)
- Trocknen Sie vollständig die Elution Lautstärke durch Vakuum Konzentration. Dieser Vorgang dauert ca. 1-2 h.
- Aufschwemmen der getrockneten Phosphopeptides in 12,5 µL Massenspektrometrie-Lösung (oder wie von der Forscher MS Proteomics zentrale Einrichtung empfohlen). Wirbel und Zentrifugieren sie bei 10.000 x g für 30 s. 2 X zu ihnen gut Aufschwemmen wiederholen. (Nicht pipette rauf und runter.)
11. die Massenspektrometrie Analyse
- Senden Sie die Proben an die MS Proteomics zentrale Einrichtung, flüssige Chromatographie-Tandem-MS (LC-MS/MS) mit den empfohlenen Einstellungen durchzuführen. Beispieleinstellungen sind wie folgt (siehe Tabelle 2 für die Zusammenfassung):
- Laden 5 µL der Proben auf einer Trap-Säule (2 cm lang x 75 µm Durchmesser) und waschen Sie sie mit 0,1 % TFA für 5 min mit einem Volumenstrom von 5 µL/min.
- Bringen Sie die Falle im Einklang mit einer Nano-Trennsäule (20 cm x 75 µm) mit einer Durchflussmenge von 300 nL/min.
- Die segmentierte lineare Verläufe (ein Prozentsatz von 0,16 % Ameisensäure, 80 % ACN in 0,2 % Ameisensäure) pY und pST Proben unterschiedlich sind:
- Eluieren Sie für die Proben pY mit einer Steigung von 4-15 % in 5 min, 15-50 % in 40 min und 50-90 % in 5 Minuten.
- Eluieren Sie für PST-Proben mit einer Steigung von 4-15 % in 30 min, 15-25 % in 40 min, 25-50 % in 44 min und 50-90 % in 11 min..
- MS Daten in datenabhängiges Akquisitionsmodus mit einer zyklischen Reihe von einen vollständigen Scan mit einer Auflösung von 120.000 gefolgt von MS/MS (HCD, relative Aufprallenergie von 27 %) von den 20 am intensivsten Ionen und eine dynamische Dauer 20 s.
- Importieren Sie nach der Fertigstellung laufen MS MS-raw-Dateien in ein MS-Analyse-Software-Programm zu identifizieren und quantifizieren Phosphopeptides. (MaxQuant Software8,26,27 wurde in diesem Experiment verwendet. Es sei denn, in Tabelle 3angegeben, wurden die Standardeinstellungen verwendet.)
Ergebnisse
Dieses Protokoll beschreibt ausführlich eine Methode zur Proteingewinnung und Verdauung, gefolgt von Phosphopeptide Bereicherung und anschließende Analyse in MS (Abbildung 1). Die Kompositionen der Puffer und Lösungen, die in diesem Protokoll verwendet werden sind in Tabelle 1aufgeführt. Die fortlaufende Nutzung der Lys-C und Trypsin bietet eine effiziente Verdauung. Eine Coomassie gefärbt Gel des bereits verdauten lysate bestätigt das Vorhandensein von Proteinen, während Färbung des Post-verdaute lysate die komplette Verdauung (Abbildung 2A bestätigt). Für eine vollständige Verdauung sollte keine Bands bzw. über 15 kDa, außer die 30 kDa und 23,3 kDa-Bands für Lys-C und Trypsin, angezeigt. Die Zugabe von Lys-C reduziert auch die Anzahl der verpassten Spaltungen (Abb. 2 b). Da pY Peptide nur 2 % der Phosphoproteome28darstellen, ist Immunopräzipitation der pY Peptide mit einer pY-spezifischen Antikörper der erste Schritt der pY Peptid Bereicherung. Der daraus resultierende Überstand wird die Eingabe für PST-Peptid Bereicherung. PY-Immunopräzipitation trennt effektiv pY Peptide aus PST-Peptide, wo im Durchschnitt 85 % der Phosphopeptides von der VJ-Vorbereitung identifiziert pY (Abbildung 3A) und mehr als 99 % der Phosphopeptides aus der PST-Datei-Erstellung identifiziert werden pST (Abb. 3 b). Titandioxid wird verwendet, um für Phosphopeptides in beiden Präparaten zu bereichern. Der erwartete Anteil von Peptiden in der MS-Ready-Vorbereitung, die phosphoryliert sind ist zwischen 30-50 % (Abb. 4A). Die Variabilität der Phosphopeptide Bereicherung Anteil kann in pY Vorbereitung durch es gibt viele weniger pY-Peptide als PST-Peptide größer sein. In Bezug auf die Phosphopeptide Arten haben die meisten von den Phosphopeptides erkannt eine einfache oder doppelte Phosphoryl-Gruppe (Abbildung 4 b).
Nach der Durchführung Massenspektrometrie, werden die MS-raw-Dateien in eine MS-Analyse-Software geladen. Die Parameter-Einstellungen, die im Experiment sind in Tabelle 3 aufgeführten aber variiert von Software zu Software und von Version zu Version variieren. Die Parameter, die nicht aufgeführt sind als Standard, einschließlich einer FDR cutoff von 1 % für passende Peptid-Spektrum (PSM) mit einer Mindestpunktzahl von Andromeda von 40 für die Identifizierung der modifizierte Peptide27blieben. Eine Lokalisierung Wahrscheinlichkeit Cutoff von größer als 0,75 filtert ca. 5 % der pY Peptide und 15 % und 34 % der pS und pT Peptide, bzw. einstellen (Abb. 5A). Nach diesen Filter anwenden, ist die erwartete Anzahl von Phosphopeptide Identifikationen am Ende der MS-Analyse etwa 300 pY Peptide (für 5 mg Start Protein) und ca. 7.500 pS Peptide und 640 pT Peptide (für 2,5 mg des Peptids ab Betrag) von den jeweiligen Bereicherung Vorbereitungen (Abb. 5 b). Die Anzahl der Wiederholungen und die Variabilität der Phosphopeptide Signalintensität bestimmt ausreichende Stromversorgung für statistische Vergleiche. In vier separaten Experimente mit Gruppen, die entweder biologische Duplikate oder Triplicates wurden die prozentuale Koeffizienten der Variation (% CV) für erkannten Phosphopeptides berechnet. Verteilungen von geringer Variabilität (z.B. PST-Gruppen 1-5 in Abbildung 5) zeigen, dass die Entnahme von Proben, die Vorbereitung und die Massenspektrometrie läuft beständig waren. Auf der anderen Seite Verteilungen von höheren Variabilität (z.B. PST-Gruppe 6 in Abb. 5) zeigt lautere Daten, die größeren Falten-Änderungen erhebliche Unterschiede in den nachgelagerten differenzierte Analysen erkennen erfordern würde.
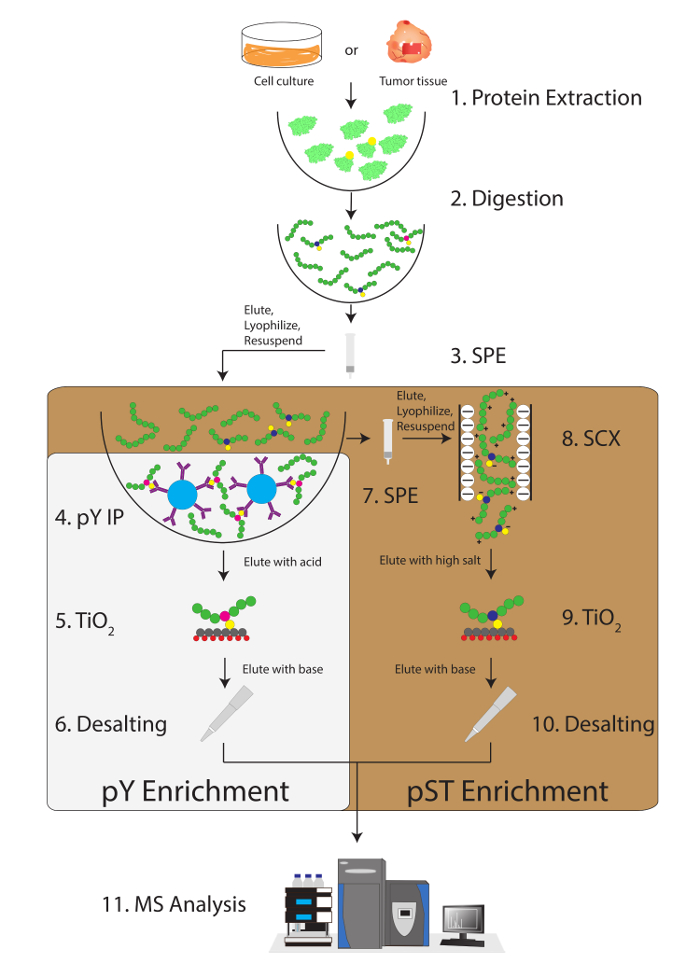
Abbildung 1: Workflow-Diagramm. Proteine aus Proben extrahiert und verdaut. Peptide werden durch Festphasenextraktion (SPE) extrahiert und Phosphotyrosine (pY) Peptide sind Immunoprecipitated. Parallel werden die Phosphoserine/Threonin (pST) Peptide aus der Überstand in pY Immunopräzipitation Schritt bereichert. Starke KATIONENAUSTAUSCH (SCX) erfolgt auf der Überstand, stark geladenen Peptide zur Reduzierung der Ionen-Unterdrückung12zu entfernen. Beide Präparate werden Phosphopeptide Bereicherung über Titandioxid (TiO2) unterzogen. Nach Bereinigung der Probe wird flüssige Chromatographie-Tandem-Massenspektrometrie (LC-MS/MS) durchgeführt, um die Phosphopeptide Fülle zu messen. Die raw-Daten werden dann in eine MS-Analyse-Software um Phosphopeptides zu identifizieren geladen. Bitte klicken Sie hier für eine größere Version dieser Figur.
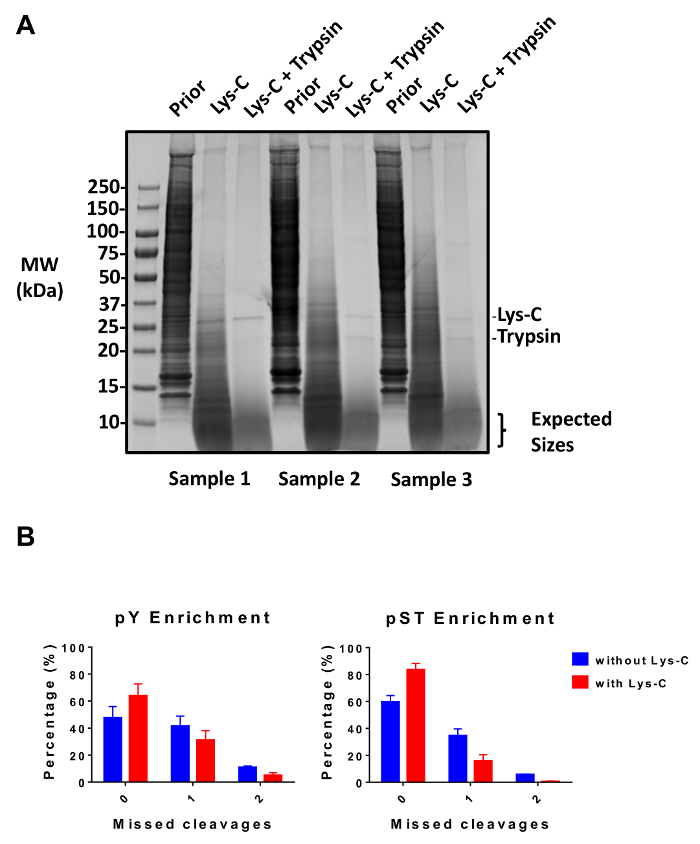
Abbildung 2: Bewertung der Verdauung. (A) drei Proben mit 12,5 µg lysate Pre-Verdauung, post-Lys-C Verdauung und Post-Trypsin-Verdauung werden angezeigt. Ein Coomassie Gel-Fleck Test zeigt eine saubere Verdauung sequentielle Nachgebrauch von Lys-C und Trypsin. Das Molekulargewicht (MW) Größe Marker sind in Kilodaltons (kDa). (B) A Verringerung der verpassten Spaltungen wird beobachtet, nachdem Lys-C das Protokoll hinzugefügt wurde. Der Anteil der Phosphopeptides ohne verpasste Spaltungen von 48 % auf 64 % und von 60 % auf 84 % im Durchschnitt für pY und pST Bereicherung Vorbereitungen, bzw. erhöht. Die Diagramme zusammenfassen, die Daten aus zwei Experimente ohne Lys-C und fünf Experimente mit Lys-C. Die Fehlerbalken sind Standardabweichungen repräsentieren 38 pY und 38 pST aus 2 separaten Experimenten (ohne Lys-C) und 62-pY und 60 PST-Proben aus 5 separaten Experimente (mit Lys-C). Bitte klicken Sie hier für eine größere Version dieser Figur.
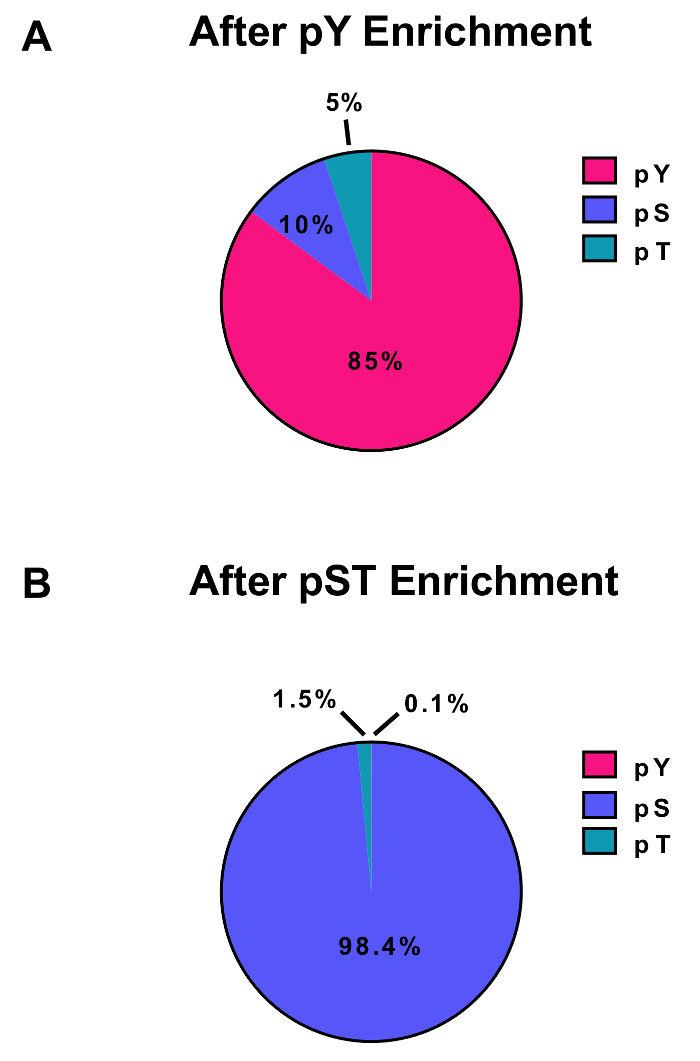
Abbildung 3: Anreicherung von pY und PST-Phosphopeptides. Diese Tafeln zeigen die Prozentsätze der pSTY Phosphopeptides von entweder (A) die pY oder (B) die pST Bereicherung Vorbereitungen. Die pY-Anreicherung von pY Immunopräzipitation und Titandioxid führte zu 85 % Phosphopeptides wird für pY Peptide, während nur 0,1 % der Phosphopeptides in der PST-Anreicherung pY sind. Diese Werte wurden gezeichnet von der Prüfung der Phospho (STY)Sites.txt-Datei eines repräsentativen Experiments nach herausfiltern von Verunreinigungen, umgekehrte Sequenzen und Phosphopeptides mit weniger als 0,75 Lokalisierung Wahrscheinlichkeiten. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 4: Phosphopeptide Anreicherung mit Titandioxid. (A) ist der Anteil der gefundenen Phosphopeptides (bezogen auf insgesamt Peptide) von Proben in vier separaten Experimente gezeigt. (B) dieses Panel zeigt die durchschnittliche Zusammensetzung des Mono-, Doppel- und Multi-phosphoryliert Peptide in vier separaten Experimente. Die Fehlerbalken in zentrale A sind Standardabweichungen. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 5: Phosphoresidue Identifikationen erwartet. (A) dieses Panel zeigt die Phosphorylierung Lokalisierung Wahrscheinlichkeiten von IDs von pY Anreicherung (links) und PST-Anreicherung (rechts). Der mittlere Anteil der IDs, die die Wahrscheinlichkeit Cutoff > 0,75 zu erfüllen ist 93 %, 75 % und 52 % für pY, pS und pT, beziehungsweise. (B) die mittlere Anzahl der IDs mit einem > 0,75 Lokalisierung Wahrscheinlichkeit beträgt 300 für pY, 7.500 für pS und 640 für pT. (C) dieses Panel zeigt Violine Grundstücke der prozentuale Variationskoeffizient (% CV) von den Phosphopeptides. Eine Auswertung der % CV wurde nur durchgeführt, wenn ein Signal Intensitätswert in jedem biologischen replizieren oder dreifacher Gruppe erkannt wurde. Daten stammen von vier separate Experimente. Die Fehlerbalken in Platten A und B sind Standardabweichungen vom 34 pY und 34 PST-Proben aus 4 separaten Experimente. Bitte klicken Sie hier für eine größere Version dieser Figur.
| Puffer | Volumen | Zusammensetzung | |
| 6 M Guanidinium Chlorid Lyse Puffer | 50 mL | 6 M Guanidinium Chlorid, 100 mM Tris pH 8,5, 10 mM Tris (2-Carboxyethyl) Phosphin, Chloracetamid 40 mM, 2 mM Natrium Orthovanadate, 2,5 mM Natrium Pyrophosphat, 1 mM β-glycerophosphat, 500 mg n-Octyl-Glykosid, Ultrareines Wasser Volumen | |
| 100 mM Natrium Pyrophosphat | 50 mL | 2,23 g Natrium Pyrophosphat Decahydrate, Reinstwasser, Volumen | |
| 1M β-glycerophosphat | 50 mL | 15,31 g β-glycerophosphat, Reinstwasser, Volumen | |
| 5 % Trifluoroacetic Säure | 20 mL | Fügen Sie 1 mL 100 % Trifluoroacetic Säure in 19 mL Reinstwasser | |
| 0,1 % Trifluoroacetic Säure | 250 mL | 245 mL Reinstwasser 5 mL 5 % Trifluoroacetic Säure hinzufügen | |
| pY Elution buffer | 250 mL | 0,1 % Trifluoroacetic Säure, 40 % Acetonitril, Reinstwasser, Volumen | |
| PST-Elution buffer | 250 mL | 0,1 % Trifluoroacetic Säure, 50 % Acetonitril, Reinstwasser, Volumen | |
| IP-Bindung Puffer | 200 mL | 50 mM Tris pH 7.4, 50 mM Natrium-Chlorid, Reinstwasser, Volumen | |
| 25 mM Ammonium Bicarbonat, pH 7,5 | 10 mL | 19,7 mg in 10 mL sterile Ultrareines Wasser, pH-Wert auf 7,5 mit 1 N Salzsäure auflösen (~ 10-15 µL/10 ml Lösung), frisch | |
| 1M Phosphatpuffer pH 7 | 1.000 mL | 423 mL 1 M Dihydrogen Natriumphosphat, 577 mL 1 M Natriumphosphat Wasserstoff | |
| Gleichgewichtherstellung Puffer | 14 mL | 6,3 mL Acetonitril, 280 µL 5 % Trifluoroacetic Säure, 1740 µL Milchsäure, 5,68 mL Reinstwasser | |
| Puffer spülen | 20 mL | 9 mL Acetonitril, 400 µL 5 % Trifluoroacetic Säure, 10,6 mL Reinstwasser | |
| Massenspektrometrie-Lösung | 10 mL | 500 µL Acetonitril, 200 µL 5 % Trifluoroacetic Säure, 9,3 mL Reinstwasser | |
| Puffer A | 250 mL | 5 mM Monopotassium Phosphat (pH-Wert 2,65), 30 % Acetonitril, 5 mM Kaliumchlorid, Ultra-reines Wasser, Volumen | |
| Puffer B | 250 mL | 5 mM Monopotassium Phosphat (pH-Wert 2,65), 30 % Acetonitril, 350 mM Kaliumchlorid, Reinstwasser, Volumen | |
| 0,9 % Ammonium Hydroxid | 10 mL | 300 μL 29.42 % Ammonium Hydroxid, 9,7 mL Reinstwasser | |
Tabelle 1: Puffer und Lösungen. Diese Tabelle zeigt die Kompositionen der Puffer und Lösungen, die in diesem Protokoll verwendeten.
| LC-MS/MS-Einstellungen | ||
| Parameter | pY Einstellung | PST-Datei Einstellung |
| Probieren Sie laden (µL) | 5 | |
| Laden von Durchfluss (µL/min) | 5 | |
| Gradient Durchflussmenge (nL/min) | 300 | |
| Linearer Farbverlauf (Prozentsatz 0,16 % Ameisensäure, 80 % ACN in 0,2 % Ameisensäure) | 4 - 15 % für 5 min | 4 - 15 % für 30 min. |
| 15 - 50 % für 40 min. | 15 - 25 % für 40 min. | |
| 50 - 90 % für 5 min | 25 - 50 % für 44 min. | |
| 50 - 90 % für 11 min | ||
| Vollständiger Scan-Auflösung | 120.000 | |
| Anzahl der intensivsten Ionen ausgewählt | 20 | |
| Relative Aufprallenergie (%) (HCD) | 27 | |
| Dynamische Ausgrenzung (s) | 20 | |
Tabelle 2: LC-MS Einstellungen. Dies ist ein Beispiel für LC-MS-Einstellungen in einem typischen Schrotflinte Phosphoproteomic Experiment. Die Proben wurden auf einer Trap-Säule verladen. Die Falle wurde im Einklang mit einer Trennsäule gebracht. Diese Einstellungen wurden optimiert für die Verwendung der LC-MS-System in der Tabelle der Werkstoffe und Reagenzienaufgeführt. Diese Einstellungen für andere LC-MS Systeme berichtigt werden müssten.
| MaxQuant Parameter-Einstellungen | ||
| Einstellung | Aktion | |
| Gruppe-spezifische Parameter | ||
| Typ | Typ | Wählen Sie Standard |
| Multiplizität | Auf 1 gesetzt | |
| Verdauung-Modus | Enzym | Wählen Sie Trypsin/P |
| Max. verpasste Spaltungen | Legen Sie auf 2 | |
| Änderungen | Variable Änderungen | Hinzufügen von Phospho (Stall) |
| Markierungsfreie Quantifizierung | Markierungsfreie Quantifizierung | Wählen Sie LFQ |
| LFQ min. Verhältnis Graf | Auf 1 gesetzt | |
| Schnelle LFQ | Abhaken | |
| Diverses | Re zu quantifizieren | Abhaken |
| Globale Parameter | ||
| Sequenzen | FASTA-Dateien | Wählen Sie Fasta-Datei von UniProt heruntergeladen |
| Festen Modifikationen | Carbamidomethyl (C) hinzufügen | |
| ADV. Identifikation | Übereinstimmung zwischen den Läufen | Abhaken |
| Spiel-Zeit-Fenster | Legen Sie auf 5 min | |
| Ausrichtung-Zeitfenster | Legen Sie auf 20 min | |
| Unbekannte Funktionen entsprechen | Abhaken | |
| Protein Quantifizierung | Min. Verhältnis Graf | Auf 1 gesetzt |
| Ordnerspeicherorte | Ändern Sie entsprechend | |
Tabelle 3: MS-Analyse-Software-Einstellungen. In MaxQuant waren die gruppenspezifischen und globale Parameter in dieser Tabelle ausgewählt oder angepasst. Alle anderen Parameter blieben auf Standard. Diese Experimente wurden mit Version 1.5.3.30. Die Parameter können von Version zu Version und von Software zu Software variieren.
Diskussion
Vor Verwendung dieses Protokolls für Phosphopeptides zu bereichern, ist eine sorgfältige Prüfung der experimentellen Design entscheidend. Unter Verwendung biologischer Replikate ist ein kostengünstiger Ressourceneinsatz Massenspektrometrie als technische repliziert. Die Anzahl der Wiederholungen, die notwendig sind hängt teilweise von der Variabilität der Daten. Eine kürzlich durchgeführte Studie zeigte, dass, während die Anzahl Identifikationen die Zahl der Wiederholungen über drei nur marginal steigt, die Anzahl der signifikanten Identifikationen zwischen Gruppen steigt mit mehr10repliziert.
Aufgrund der niedrigeren Fülle von Phosphoproteine in der Zelle sind ausreichend Protein Startbeträge notwendig, um eine globale Phosphoproteome von Prostata-Krebs Proben in den Suchmodus zu erhalten. In diesen Experimenten wurde 5 mg Protein verwendet. Ungefähr fünf fast konfluierende 15 cm Gerichte von Zellen bieten genügend Protein als Eingabe in dieses Protokoll, obwohl dies Zelle Zeile abhängig sein wird. Für Tumorgewebe ist die erwartete Rendite des Proteins etwa 6-8 % der Gewebe Gewicht. In der in-Vitro -Einstellung ist eine Positivkontrolle Probe zu prüfen, die Zugabe von 1 mM Vanadat für 30 min vor der Ernte der Zellen. Bismutvanadat, eine wettbewerbsfähige Protein Phosphotyrosyl Phosphatase Inhibitor, bewahrt die Tyrosin-Phosphorylierung, wodurch sich die Anzahl der pY Peptid Identifikationen29.
Saubere Verdauung ist ein wichtiger Schritt um Phosphopeptide Identifikation zu maximieren. Neben dem Coomassie Fleck Test kann Prozent verpasste Spaltungen in den Daten zur Verdauung Effizienz (Abbildung 2) zu bewerten. Qualitätskontrolle Software steht zur Verfügung, die analysiert verpasste Spaltungen und andere Metriken zur Bewertung MS Daten Qualität30. Während Trypsin ist die am weitesten verbreiteten, Alternativen Proteasen zur Verfügung stehen5 bis Adresse Deckungslücken in der Proteomforschung wo optimale tryptic Peptide werden können nicht generiert31. Die Einstellungen der MS-Analyse-Software müsste dann entsprechend geändert werden, um auf Veränderungen der Proteasen einzustellen.
Das Protokoll beschäftigt Immunopräzipitation (für pY Bereicherung) sowie Titandioxid (TiO2), für Phosphopeptides zu bereichern. Alternative Ansätze zur Bereicherung für Peptide enthalten immobilisierten Metall Affinitätschromatographie (IMAC), andere Metalloxide für Metall-Oxid-Affinitätschromatographie (Verwaltungscenter) wie z. B. Aluminiumhydroxid und Polymer-basierten Metallion Affinität Capture (PolyMAC) 5,12. Frühere Studien haben gezeigt, dass verschiedene Anreicherungsverfahren für verschiedene Populationen von Phosphopeptides32bereichern. Zum Beispiel bereichert IMAC mehr Multi-phosphoryliert Peptide während Verwaltungscenter vorzugsweise für Mono-phosphoryliert Peptide33bereichert. Die Ergebnisse der Vertreter dieses Protokolls spiegeln diese Beobachtung (Abbildung 4 b). Eine kürzlich erschienenen Publikation gezeigt, dass die Kombination von IMAC und Verwaltungscenter mit einem Hybridmaterial potenziell größere Abdeckung des Phosphopeptide Arten34bieten könnte. So könnte dieses Protokoll geändert werden, um anderen Anreicherungsverfahren parallel für noch umfassendere Phosphoproteomic Analysen ermöglichen zu nutzen.
Die MaxQuant-26 -Software-Suite wird verwendet, um die MS-Datenanalyse in dieses Protokoll, aber kommerzielle Anwendungen35 gibt es auch für Phosphopeptide-Identifizierung und Quantifizierung. Phosphopeptide Identifikation ist eine Lokalisierung Wahrscheinlichkeit cutoff beantragt. Dieser Filter wird durchgeführt, um für Phosphopeptides mit einem hohen Vertrauen (d.h.größer als 0,75) Phosphoresidue Kennung10,28auswählen. Mit anderen Worten ist die summierte Wahrscheinlichkeit von anderen Rückständen, die potenziell die Phospho-Gruppe enthalten könnte weniger als 0,25. Dieser Cutoff kann erhöht werden, um die strenge der Phosphopeptide Auswahl zu erhöhen. In Bezug auf die Anzahl der Identifikationen ist die erwartete Anzahl von pY Peptiden in die Hunderte, während die erwartete Anzahl von PST-Peptiden in den hohen Tausenden ist. Diese Werte reflektieren bisher beobachteten Phosphoproteome Verteilung, wo etwa 2 %, 12 % und 86 % der der Phosphosites pY, pT und pS, bzw.28sind.
Wenn die pY und pST Bereicherung Schritte parallel ausgeführt werden, können die Probe Vorbereitungsschritte im Protokoll in sechs Tagen abgeschlossen werden. Durch die Kombination mit dem leistungsstarken Tool von MS, bieten Phosphopeptide Bereicherung Protokolle wie dies ein globales Konzept für Wissenschaftler, Daten zu analysieren, die Phosphoproteome in ihren jeweiligen Gebieten zu sammeln.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Wir danken Mitglieder des Drake Lab für die Beratung und Eingang auf dem Manuskript. Wir danken auch die Mitgliedern des biologischen Massenspektrometrie Anlage von Robert Wood Johnson Medical School und Rutgers, The State University of New Jersey, für die Beratung und Durchführung von Massenspektrometrie auf unseren Proben. Larry C. Cheng wird durch das National Institute of General Medical Sciences von den National Institutes of Health unter prämiennummer T32 GM008339 unterstützt. Thomas G. Graeber wird unterstützt durch das NCI/NIH (SPORE in Prostata-Krebs-P50-CA092131; P01 CA168585) und ein American Cancer Society Research Scholar Award (RSG-12-257-01-TBE). Justin M. Drake wird von der Abteilung der Verteidigung Prostata Krebs Forschung Programm W81XWH-15-1-0236, Prostata Krebs Foundation Young Investigator Award der New Jersey Health Foundation und einen Precision Medicine Initiative Pilot Award von der Rutgers unterstützt. Cancer Institute von New Jersey.
Materialien
| Name | Company | Catalog Number | Comments |
| Ultra-Low Temperature Freezer | Panasonic | MDF-U76V | |
| Freezer -20 °C | VWR | scpmf-2020 | |
| Swing rotor bucket | ThermoFisher Scientific | 75004377 | |
| Vacuum manifold | Restek | 26080 | |
| Lyophilizer | Labconco | 7420020 | |
| CentriVap Benchtop Vacuum Concentrator | Labconco | 7810010 | |
| End-over-end rotator | ThermoFisher Scientific | 415110Q | |
| Razor blade | Fisher Scientific | 620177 | |
| Amicon Ultra-15 Centrifugal Filter Units | Millipore Sigma | UFC901024 | |
| Glass culture tubes | Fisher Scientific | 14-961-26 | |
| Parafilm | Fisher Scientific | 13-374-12 | |
| 20G needle | BD | B305175 | |
| Kimwipes | Fisher Scientific | 06-666A | |
| Screw cap cryotube | ThermoFisher Scientific | 379189 | |
| Nunc 15 mL conical tubes | ThermoFisher Scientific | 12-565-268 | |
| Gel loading tips | Fisher Scientific | 02-707-181 | |
| Millipore 0.2 µm spin filter | Millipore Sigma | UFC30GVNB | |
| Low protein-binding Eppendorf tubes | Eppendorf | 22431081 | |
| anti-Phosphotyrosine, Agarose, Clone: 4G10 | Millipore Sigma | 16101 | |
| 27B10.4 antibody | Cytoskeleton | APY03-beads | |
| Peptide assay kit | Thermo Scientific | 23275 | Step 7 |
| TopTip | PolyLC Inc | TT200TIO.96 | Steps 5 and 9 |
| SCX columns (PolySULFOETHYL A) | PolyLC Inc | SPESE1203 | |
| 3 mL syringe | BD | 309657 | |
| Trifluoracetic Acid (TFA) | Fisher Scientific | PI-28904 | |
| Acetonitrile (ACN) | Fisher Scientific | A21-1 | |
| Lactic acid | Sigma-Aldrich | 69785-250ML | |
| Ammonium Hydroxide | Fisher Scientific | A669S-500 | |
| Potassium Phosphate Monobasic | Fisher Scientific | BP362-500 | |
| Potassium Chloride | Fisher Scientific | BP366-500 | |
| Calcium Chloride Dihydrate | Fisher Scientific | BP510-500 | |
| Tris Base | Fisher Scientific | BP152-5 | |
| Trypsin, TPCK Treated | Worthington Biochemicals | LS003740 | |
| Lysyl Endopeptidase | Wako Pure Chemical Industries, Ltd. | 125-05061 | |
| MonoTip | PolyLC Inc | TT200TIO.96 | Step 10 |
| ZipTip | MilliporeSigma | ZTC18S096 | Step 6 |
| nanoEase, MZ peptide BEH C18, 130A, 1.7 μm, 75 μm x 20 cm | Waters | 186008794 | Step 11: analytical column |
| Acclaim PepMap 100 C18 LC Columns | ThermoFisher Scientific | 164535 | Step 11: trap column |
| Ultimate 3000 RLSCnano System | Dionex | ULTIM3000RSLCNANO | Step 11 |
| Q Exactive HF | ThermoFisher Scientific | IQLAAEGAAPFALGMBFZ | Step 11 |
| MilliQ water | deionized water used to prepare all solutions and bufferes | ||
| Sonic Dismembrator | Fisher Scientific | FB-120 | sonicator |
| Polytron System PT | Kinematica AG | PT 10-35 GT | homogenizer |
Referenzen
- Siegel, R. L., Miller, K. D., Jemal, A. Cancer statistics, 2018. CA: A Cancer Journal for Clinicians. 68 (1), 7-30 (2018).
- Paller, C. J., Antonarakis, E. S. Management of biochemically recurrent prostate cancer after local therapy: evolving standards of care and new directions. Clinical Advances in Hematology & Oncology. 11 (1), 14-23 (2013).
- Lowrance, W. T., Roth, B. J., Kirkby, E., Murad, M. H., Cookson, M. S. Castration-resistant prostate cancer: AUA guideline amendment 2015. The Journal of Urology. 195 (5), 1444-1452 (2016).
- Domon, B., Aebersold, R. Options and considerations when selecting a quantitative proteomics strategy. Nature Biotechnology. 28 (7), 710-721 (2010).
- Zhang, Y. Y., Fonslow, B. R., Shan, B., Baek, M. C., Yates, J. R. Protein analysis by shotgun/bottom-up proteomics. Chemical Reviews. 113 (4), 2343-2394 (2013).
- Bantscheff, M., Schirle, M., Sweetman, G., Rick, J., Kuster, B. Quantitative mass spectrometry in proteomics: a critical review. Analytical and Bioanalytical Chemistry. 389 (4), 1017-1031 (2007).
- Bantscheff, M., Lemeer, S., Savitski, M. M., Kuster, B. Quantitative mass spectrometry in proteomics: critical review update from 2007 to the present. Analytical and Bioanalytical Chemistry. 404 (4), 939-965 (2012).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513-2526 (2014).
- Rubbi, L., et al. Global phosphoproteomics reveals crosstalk between Bcr-Abl and negative feedback mechanisms controlling Src signaling. Science Signaling. 4 (166), ra18 (2011).
- Hogrebe, A., et al. Benchmarking common quantification strategies for large-scale phosphoproteomics. Nature Communications. 9 (1), 1045 (2018).
- Rush, J., et al. Immunoaffinity profiling of tyrosine phosphorylation in cancer cells. Nature Biotechnology. 23 (1), 94-101 (2005).
- Fila, J., Honys, D. Enrichment techniques employed in phosphoproteomics. Amino Acids. 43 (3), 1025-1047 (2012).
- Mertins, P., et al. Proteogenomics connects somatic mutations to signalling in breast cancer. Nature. 534 (7605), 55-62 (2016).
- Drake, J. M., et al. Phosphoproteome integration reveals patient-specific networks in prostate cancer. Cell. 166 (4), 1041-1054 (2016).
- Lue, H. W., et al. Metabolic reprogramming ensures cancer cell survival despite oncogenic signaling blockade. Genes & Development. 31 (20), 2067-2084 (2017).
- Francavilla, C., et al. Phosphoproteomics of primary cells reveals druggable kinase signatures in ovarian cancer. Cell Reports. 18 (13), 3242-3256 (2017).
- Zhang, H., et al. Integrated proteogenomic characterization of human high-grade serous ovarian cancer. Cell. 166 (3), 755-765 (2016).
- Skaggs, B. J., et al. Phosphorylation of the ATP-binding loop directs oncogenicity of drug-resistant BCR-ABL mutants. Proceedings of the National Academy of Sciences of the United States of America. 103 (51), 19466-19471 (2006).
- Zimman, A., et al. Activation of aortic endothelial cells by oxidized phospholipids: a phosphoproteomic analysis. Journal of Proteome Research. 9 (6), 2812-2824 (2010).
- Zimman, A., Berliner, J. A., Graeber, T. G. Phosphoproteomic analysis of aortic endothelial cells activated by oxidized phospholipids. Methods in Molecular Biology. , 53-69 (2013).
- Humphrey, S. J., Azimifar, S. B., Mann, M. High-throughput phosphoproteomics reveals in vivo insulin signaling dynamics. Nature Biotechnology. 33 (9), 990-995 (2015).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72, 248-254 (1976).
- Meyer, T. S., Lamberts, B. L. Use of coomassie brilliant blue R250 for the electrophoresis of microgram quantities of parotid saliva proteins on acrylamide-gel strips. Biochimica el Biophysica Acta. 107 (1), 144-145 (1965).
- Bergstrom Lind, S., et al. Immunoaffinity enrichments followed by mass spectrometric detection for studying global protein tyrosine phosphorylation. Journal of Proteome Research. 7 (7), 2897-2910 (2008).
- Pinkse, M. W., Uitto, P. M., Hilhorst, M. J., Ooms, B., Heck, A. J. Selective isolation at the femtomole level of phosphopeptides from proteolytic digests using 2D-NanoLC-ESI-MS/MS and titanium oxide precolumns. Analytical Chemistry. 76 (14), 3935-3943 (2004).
- Cox, J., Mann, M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nature Biotechnology. 26 (12), 1367-1372 (2008).
- Cox, J., et al. Andromeda: a peptide search engine integrated into the MaxQuant environment. Journal of Proteome Research. 10 (4), 1794-1805 (2011).
- Olsen, J. V., et al. Global, in vivo, and site-specific phosphorylation dynamics in signaling networks. Cell. 127 (3), 635-648 (2006).
- Huyer, G., et al. Mechanism of inhibition of protein-tyrosine phosphatases by vanadate and pervanadate. The Journal of Biological Chemistry. 272 (2), 843-851 (1997).
- Bielow, C., Mastrobuoni, G., Kempa, S. Proteomics quality control: quality control software for MaxQuant results. Journal of Proteome Research. 15 (3), 777-787 (2016).
- Swaney, D. L., Wenger, C. D., Coon, J. J. Value of using multiple proteases for large-scale mass spectrometry-based proteomics. Journal of Proteome Research. 9 (3), 1323-1329 (2010).
- Bodenmiller, B., Mueller, L. N., Mueller, M., Domon, B., Aebersold, R. Reproducible isolation of distinct, overlapping segments of the phosphoproteome. Nature Methods. 4 (3), 231-237 (2007).
- Leitner, A., Sturm, M., Lindner, W. Tools for analyzing the phosphoproteome and other phosphorylated biomolecules: a review. Analytica Chimica Acta. 703 (1), 19-30 (2011).
- Yang, D. S., et al. Design and synthesis of an immobilized metal affinity chromatography and metal oxide affinity chromatography hybrid material for improved phosphopeptide enrichment. Journal of Chromatography A. 1505, 56-62 (2017).
- Al Shweiki, M. R., et al. Assessment of label-free quantification in discovery proteomics and impact of technological factors and natural variability of protein abundance. Journal of Proteome Research. 16 (4), 1410-1424 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten