Method Article
Rilevazione e quantificazione del Plasmodium falciparum in acquoso globuli rossi da attenuato spettroscopia infrarossa totale riflessione e l'analisi multivariata dei dati
In questo articolo
Riepilogo
Qui, presentiamo un protocollo per la rilevazione e la quantificazione del Plasmodium falciparum in acquosi globuli rossi infetti utilizzando un spettrometro a infrarossi riflessione totale attenuata e analisi multivariata dei dati.
Abstract
Dimostriamo un metodo di quantificazione e individuazione di parassiti in acquosa di globuli rossi (RBCs) utilizzando un semplice benchtop attenuato totale riflessione Fourier Transform spettrometro a infrarossi (ATR-FTIR) in combinazione con l'analisi multivariata dei dati ( MVDA). 3 7 p. falciparum sono stati coltivati a parassiti fase 10% parassitemia anello e utilizzato a picco i globuli rossi del donatore fresco isolato per creare una diluizione serie tra 0 – 1%. 10 µ l di ciascun campione sono stati collocati al centro della finestra di diamante ATR per acquisire lo spettro. I dati di esempio è stati trattati per migliorare il rapporto segnale-rumore e rimuovere il contributo di acqua, e quindi la derivata seconda è stata applicata per risolvere caratteristiche spettrali. I dati sono stati quindi analizzati utilizzando due tipi di MVDA: primo Principal Component Analysis (PCA) per determinare eventuali valori anomali e quindi regressione di minimi quadrati parziali (PLS-R) per compilare il modello di quantificazione.
Introduzione
La malaria è tra le più devastanti malattie del nostro tempo; oltre la metà della popolazione vive a rischio nelle regioni endemiche ed esso sproporzionatamente opprime il povero1,2,3,4. Gran parte del problema è i portatori asintomatici e primi pazienti di stadio che fungono da serbatoi per zanzare vettori5, causando picchi di infezione durante stagioni umide e permettendogli di persistere nelle comunità. La malaria è causata da parassiti Plasmodium cinque, il più mortale di cui è p. falciparum che causa la forma più grave della malattia2.
Attualmente, le tecniche per la diagnosi di malaria sono meno che perfetto. Microscopia ottica, il gold standard attuale, è in grado di rilevare solo 62-88 parassiti / µ l a seconda del metodo utilizzato6. Inoltre, a causa della intensità e alta abilità richiesto, in molte regioni microscopia mal diagnostica > 50% dei casi, soprattutto quelli con bassa parassitemia livelli2, che possono essere direttamente attribuiti alla mancanza di risorse nella zona e risultati in l'uso improprio di farmaci anti-malarici. Gli altri 2 metodi diagnostici principali sono test diagnostici rapidi (RDT), che utilizzano gli anticorpi per la rilevazione e l'analisi di reazione a catena della polimerasi (PCR), che discriminano e quantificare i parassiti dal DNA. Attualmente, RDTs sono solo in grado di rilevare p. falciparum e p. vivax di almeno 100 parassiti / µ l di sangue con conseguente forme più rare della malattia non viene trattata. 7 , 8 al contrario, analisi di PCR discriminare e quantificare le diverse specie di Plasmodium ad una sensibilità di 0,0004-5 parassiti / µ l di sangue. Tuttavia, esso richiede costosi reagenti, equipaggiamento e abilità tecnica e pertanto non è adatto per l'applicazione del campo.
Una tecnica altamente sensibile, affidabile e conveniente è essenziale per migliorare i tempi di diagnosi e così migliorando i risultati pazienti e rendendo possibile l'eliminazione della malattia. Attenuato totale riflessione spettroscopia trasformata di Fourier (ATR-FTIR) offre una possibile soluzione a questo problema. Il lavoro precedente ha indicato che era possibile rilevare e quantificare da p. falciparum in metanolo fissata pellicole di anima raggiungendo i limiti di rilevazione di < 1 parassita / µ l di sangue (< 0,0002% parassitemia)9, che è paragonabile ai metodi PCR. Studi recenti hanno dimostrato che è possibile rilevare e quantificare i parassiti in campioni acquosi e quindi eliminare il punto di fissazione. Tuttavia, fattori come il vapore acqueo, rumore spettrale e trattamento dei dati devono essere presi in considerazione per un risultato ottimale10.
Questo protocollo si propone di mostrare nuovi utenti come acquisire spettri ATR-FTIR e preparare un modello di regressione per il rilevamento di p. falciparum da acquose di campioni di globuli rossi (RBC)10.
Protocollo
Consultare appropriato Material Safety Data Sheets (MSDS) e cercare la formazione adeguata di livello 2 di biosicurezza (BSL-2). Tutti i passaggi di coltura devono essere fatto in un cabinet di BSL-2 usando una tecnica asettica, il che significa che c'è un rischio di esposizione a radiazioni UV nocive da operazioni di decontaminazione, punture accidentali ed esposizione potenziale biologico e infezione se la cultura di parassita entra tutte le lesioni. Inoltre, è un potenziale rischio sangue stock da banche del sangue è solo proiettato per alcune malattie e il potenziale per la diffusione di malattie a carico del sangue. Cercare aiuto medico immediato nell'avvenimento della lesione.
1. preparazione e misurazione dei 3 7 serie di diluizioni del parassita Plasmodium falciparum
Nota: Plasmodium SP. cultura è altamente sensibile. Usare i reagenti freschi/scaduto e nutrire i parassiti regolarmente modificando il supporto. Nutrire le colture sotto 5% parassitemia ogni secondo giorno; nutrire le colture tra 6-10% parassitemia una o due volte al giorno; e nutrire le colture tra 11 – 20% fino a 4 volte al giorno. Parassiti che stanno cominciando a morire di fame si perdono la loro forma e inizia a contrarsi. In tal caso, mangimi e diluire immediatamente cambiando i media e aggiungendo brodo di RBCs. raccogliere il sangue del donatore in provette contenenti eparina come anticoagulante e misura entro le prime 6 ore.
- Cultura un totale di 30 mL di 3 7 ceppo parassiti Plasmodium falciparum secondo il protocollo standard fino a quando la parassitemia raggiunge 10% anelli.
-
Sincronizzazione dei parassiti
- 11 h dopo shizogeny nel ciclo di vita del parassita, Risospendere la cultura e il trasferimento in una provetta conica 50 mL utilizzando una pipetta automatica.
- Centrifugare la coltura a 300-400 x g e a condizioni di laboratorio standard (25° C e 100 kPa) per 5 min.
- Eliminare il surnatante tramite elaborazione dello stesso con una pipetta o usando una pipetta di Pasteur attaccata ad un vuoto senza disturbare il pellet. Gettare il terreno dei rifiuti in una soluzione di candeggina al 10%.
- Lentamente aggiungere 12 – 15 mL di soluzione al 4% sorbitolo al pellet utilizzando una pipetta automatica e mescolare la cultura tappatura ed invertendo il tubo fino a quando la cultura è omogenea.
- Incubare la coltura a 37 ° C per 15 min.
- Centrifugare la coltura a 300-400 x g e le condizioni standard di laboratorio per 5 min.
- Rimuovere il surnatante come descritto al punto 1.2.3.
- Aggiungere 10 – 15 mL di soluzione fisiologica 0,9% al pellet e miscelare la soluzione di tappatura e invertendo il tubo fino a quando la cultura è omogenea.
- Ripetere i passaggi 1.2.6–1.2.8 altre due volte per lavare tutti i resti di sorbitolo.
-
Preparazione della parassitemia diluizione serie
- Lavare Stock in RBCs come in passaggi 1.2.6–1.2.9.
- Centrifugare la coltura in condizioni standard di laboratorio, Stock in RBCs a 300-400 x g per 5 min ed eliminare il surnatante come descritto al punto 1.2.3.
- Etichetta per microcentrifuga come 0%, 0.010%, 0,025%, 0,075%, 0,100%, 0.250%, 0.750% e 1.000% e aggiungere 0 µ l, 1 µ l, 2,5 µ l, 7,5 µ l, 10 µ l, 25 µ l, 75 µ l e 100 µ l di cultura rispettivamente usando una pipetta di 0,2-20 µ l.
- Aggiungere 100 µ l, µ l 99, 97,5 µ l, µ l 92,5, 80 µ l, 75 µ l, 0 e 25 µ l di Stock in RBCs al tubi etichettati 0%, 0.010%, 0,025%, 0,075%, 0,100%, 0.250%, 0.750% e 1.000%, rispettivamente.
- Centrifugare il sangue del donatore freschi raccolto in provette dell'anticoagulante a 1.200 x g e condizioni standard di laboratorio per 10 min.
- Rimuovere il plasma di elaborazione dello stesso con una pipetta e scartando come descritto al punto 1.2.3.
- Aggiungere 900 µ l di RBCs isolato in ciascuna provetta per microcentrifuga e miscelare accuratamente capovolgendo 10x.
-
Acquisizione spettrale
- Utilizzando il programma di acquisizione spettrale accompagnamento, fare clic su "Strumento set-up" e impostare i parametri di raccolta di dati seguenti: 128 scansioni per sfondo; 32 scansioni per il campione; e risoluzione a 8/cm sopra la gamma di frequenza di campionamento massima dello strumento.
- Impostare la temperatura dello spettrometro FTIR-ATR per la raccomandazione del produttore o durante la notte.
- Pulire il cristallo di strofinare delicatamente con un movimento circolare utilizzando lanugine gratis salviette inumidito con acqua ultrapura. Quindi asciugare con un altro panno libero wipe.
- Prendere una misura di sfondo dell'aria facendo clic su "Sfondo misura". Ogni 20 min, pulire il cristallo come descritto al punto 1.4.3 e ripetere la misurazione.
- Aprire la visualizzazione live facendo clic su "Anteprima". Osservare una linea di base piana, orizzontale che indica che il cristallo sia pulito.
- Pipettare 10 µ l di acqua deionizzata direttamente sul mezzo del cristallo e fare clic su "Esempio di misura". Ripetere questa misurazione di acqua dopo ogni campione quinto.
- Asciugare il cristallo usando un panno libero wipe e un delicato movimento circolare.
- Pipettare 10 µ l di campione e fare clic su "Esempio di misura".
- Pulire il cristallo tra i campioni come descritto al punto 1.4.3.
- Ripetere fino a 3 replicati vengono acquisiti, assicurandosi di randomizzare l'ordine di entrambi i campioni e le repliche.
2. multivariata analisi (MVDA)
Nota: Trattamento dei dati deve essere fatto sopra l'intero set di dati al fine di evitare il rumore e l'aggiunta di cime di origine non biologica. Al contrario, analizzare sopra le regioni biologicamente rilevanti: 2980-2800/cm e 1750-850/cm.
-
Trattamento dei dati
Nota: Due software, ad esempio, Matlab (d'ora in poi denominati software 1) e il riordinatore X (d'ora in poi denominati software 2) sono utilizzati come esempi per MVDA. Software 1 ha la capacità di eseguire MVDA senza l'aggiunta di un'interfaccia utente grafica (GUI). Tuttavia, è consigliabile acquistare una GUI (Tabella materiali). Le seguenti istruzioni quando riferendosi al software 1 presupporrà che è in congiunzione con il toolbox di GUI disponibile in commercio, e lo script di trattamento di dati di esempio è stato allegato.- Aprire il software di analisi di dati multivariati appropriato, fare clic su "Importa dati" e selezionare il tipo di file per l'analisi.
- Nel software 1, ingresso "Analisi" nella finestra di comando per aprire l'interfaccia grafica. Fare clic destro la X box per trovare "Importa dati".
- Nel software 2, fare clic sulla scheda "File" per trovare "Importazione".
- Importare gli spettri del campione, acqua e linea di base come insiemi di dati nello spazio di lavoro selezionando tutti gli spettri in ogni set separatamente e facendo clic su "Apri" e dare ogni set un nome breve, ad esempio, 'wat' per set di dati di spettri di acqua.
- Selezionare nuova tabella o matrice facendo clic su "nuovo Matrix/Vector" e generare un vettore di n × 1, dove n è il numero di campioni. La parassitemia di ogni campione di input e dare il vettore il nome 'Parassitemia'.
- Nel software 1, fare clic su "Nuova variabile" nella finestra di comando.
- Nel software 2, fare clic sull'icona "Nuova matrice".
- Tracciare i dati facendo clic su "Stampa dati icona". Ispezionare gli spettri per gli effetti del vapore acqueo facendo clic su "Zoom" e zoomando su 1800-1400/cm; più chiaramente osservato come breve, tagliente, stretti picchi lungo le pendici del ammide io e ammide Band II.
- In casi di estrema vapor d'acqua, aprire la scheda Modifica/pre-process dati e selezionare "Smoothing". Ridurre il rumore e/o contributi di forte vapore acqueo lisciando gli spettri del campione e acqua utilizzando fino a 25 punti di lisciatura o utilizzare un metodo di correzione di vapore acqueo.
- Corretta rispetto al basale non orizzontali utilizzando l'algoritmo di correzione della linea di base, se del caso, la stessa scheda di dati di modifica/pre-process nel passaggio 2.1.4.
- Media acqua spettri e copiare le righe in una matrice di n × m equivalente al set di dati campione e ridurlo al 70% intensità moltiplicandolo per 0,7.
- Nel software 1, farlo nella finestra di comando inserendo il seguente script "AverageWater=mean(WaterDataset)". Quindi copiare e incollare le righe per corrispondere al set di dati di esempio. Per ridurre l'intensità, di input "AverageWater70 = AverageWater * 0.70".
- Sottrarre acqua medio spettri da ogni spettro campione.
- Nel software 1, farlo nella finestra di comando inserendo il seguente script "WaterCorrectedData = SampleDataset-AverageWater70".
- Aprire la scheda Modifica/pre-process dati per applicare una seconda funzione derivative e quindi normalizzare i dati selezionando la funzione di singola variabile normale (SNV) e significa centro dati.
- Nel software 1, è possibile farlo in una sola volta. In primo luogo selezionare "Derivato" e ingresso 25 punti di lisciatura, ordine polinomiale di 3 e derivati ordine sul campione impostato utilizzando una funzione Savitzky Golay e 25 punti di lisciatura. Quindi selezionare "SNV" e "Media Center". Fare clic su "OK/applicare".
- Aprire la scheda di dati di modifica/pre-process e nella colonna "variabili", selezionare 2.980 – 2.800/cm e 1.750 – 850/cm assicurandosi che siano selezionate solo le caselle.
- Aprire il software di analisi di dati multivariati appropriato, fare clic su "Importa dati" e selezionare il tipo di file per l'analisi.
-
Analisi dei dati
- Analisi delle componenti principali (PCA)
- Fare clic su "analisi | Decomposizione"e seleziona PCA.
- Nel software 1, cliccare su "Build Model".
- Nel software 2, il PCA metodi di input. Immettere il numero ottimo di componenti principali (PC) - in questo lavoro, 7 - e un massimo di 100 iterazioni e seleziona Convalida incrociata per il metodo di convalida. Fare clic su "Esegui".
- Osservare il limite di confidenza del 95%, l'anello tratteggiata, la trama di punteggi tra PC1 e PC2.
- Contrassegnare gli spettri del campione con i punteggi che si verificano di fuori del limite come potenziali outlier utilizzando lo "strumento di spettri seleziona".
- Se gli spettri contrassegnati hanno alti valori di2 Hotellings T e alta Q residui escludono da ulteriori analisi.
- Fare clic su "analisi | Decomposizione"e seleziona PCA.
- Regressione di minimi quadrati parziali (PLS-R)
- Fare clic su "analisi | Regressione"e selezionare"PLS-R".
- Nel software 1, fare clic destro Y blocco e selezionare il vettore "parassitemia | Costruire il modello".
- Nel software 2, i PLS-R metodi di input. Selezionare il vettore "Parassitemia" come il "riferimento Y" e il set di dati del campione come "insieme di dati X" e seleziona Convalida incrociata come il metodo di convalida. Fare clic su "Esegui".
- Analizzare il modello di regressione. R2 valori sopra 0,80 ed errore quadratico medio dei valori di cross-validazione (RMSECV) che sono meno di 0.1% parassitemia sono modelli accettabili.
- Il vettore di regressione di analizzare e individuare le bande biologiche.
- Fare clic su "analisi | Regressione"e selezionare"PLS-R".
- Analisi delle componenti principali (PCA)
Risultati
Trama di minimi quadrati parziali (PLS-R) e un vettore di regressione associato, Figura 1a e 1b rispettivamente, mostrano che il segnale da parassiti sono abbastanza distinta da globuli rossi che servono per formare una regressione lineare modello da utilizzare per la previsione dei parassiti in insiemi di dati futuri.
Un robusto modello lineare PLS-R è stato generato con un valore di R al quadrato di 0,87 e una media di radice quadrata errore di validazione incrociata (RMSECV) 0,13% parassitemia, Figura 1a. I livelli aumentanti di parassitemia sono principalmente associati con bande di acidi nucleici, soprattutto 1042, 1083 e 1226/cm, che sono assegnate alla ν(C=O) dal ribosio di RNA di gruppo, νs(PO4 -) e νun(PO4 -) rispettivamente e formano la maggior parte negativa del vettore regressione. Infatti, i globuli rossi privi di un nucleo e così acidi nucleici sono forti indicatori per i parassiti. Inoltre, la band di 1226/cm indica che la struttura ha una forma B-DNA, che mette in evidenza l'importanza della misurazione nell' acquoso stato12. Al contrario, parassiti consumano RBC componenti, principalmente dell'emoglobina, e quindi una proteina più alta rapporto del lipido è previsto in RBCs non infetti come indicato dalla bande del lipido a 2913, 2879 e 1397 cm-1 e il 1631 e il 1555 cm-1 dall'ammide io e ammide II modalità12,13.
Le bande distinte mostrano chiaramente che i segnali sono vere bande biologiche piuttosto che differenziazione basata sulle bande spurie di origine non biologica come vapore acqueo, che presentano come bande strette e intense. In particolare, la presenza di bande di DNA, tabella 1, può essere direttamente attribuita al parassita11,12,13. In generale, il vettore di regressione può essere interpretato in modo simile ai carichi nell'APC. Bande dovrebbero correlano bene con bande e turni degli spettri originali.
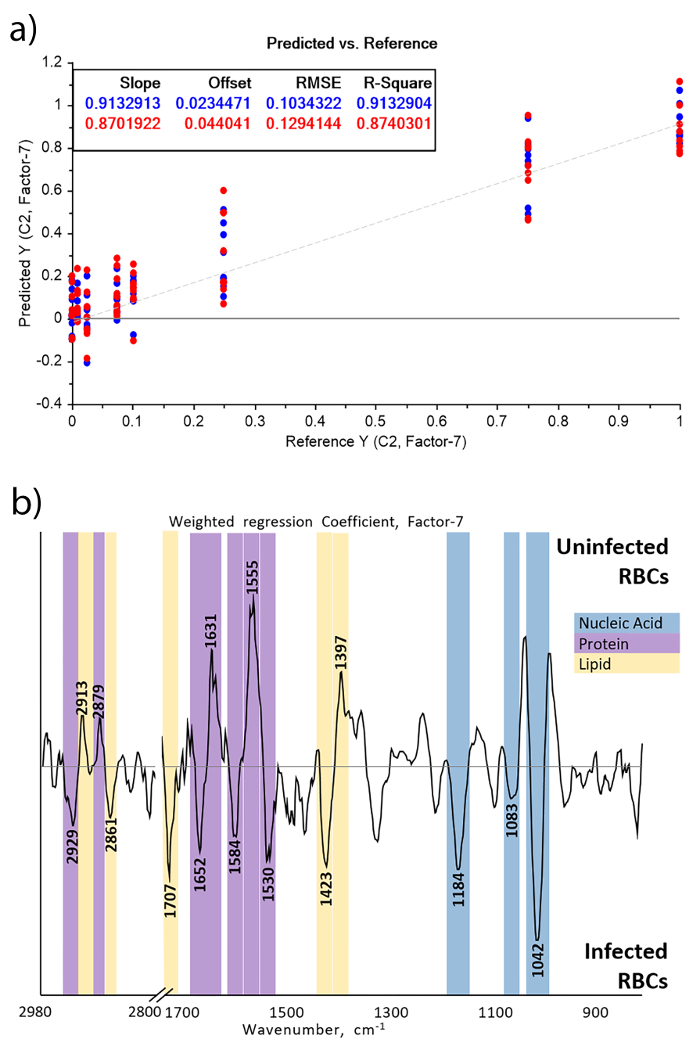
Figura 1: Trama di regressione PLS-R, (a), di globuli rossi a spillo tra 0 – 1% parassitemia e coefficiente di regressione associato, (b). La trama di regressione dimostra la capacità del modello di predire la parassitemia in ogni campione, con il nota parassitemia sull'asse x e la parassitemia prevista sull'asse y. Il coefficiente di regressione descrive il contributo di ciascuna banda per la capacità predittiva del modello, più intenso la band è più contribuisce al modello e così come il parassita altera la composizione chimica del sangue. Riprodotte e modificate da Martin, M. et al. 10 con il permesso della Royal Society of Chemistry. Clicca qui per visualizzare una versione più grande di questa figura.
| Banda di coefficiente di regressione (cm-1) | Assignment25-27 |
| 2929 | ΝaCH2 da lipidi, parassita |
| 2913 | ΝaCH2 da lipidi, rosso del globulo |
| 2879 | ΝsCH3 da lipidi, rosso del globulo |
| 2861 | ΝsCH2 da lipidi, parassita |
| 1707 | B-DNA/A-DNA base pair vibrazione νC = O & νC = N, parassita |
| 1652 | Ammide I da proteine α-elica, parassita |
| 1631 | Ammide I dal foglio β-pieghettato proteine, rosso del globulo |
| 1584 | ΝC = N da imidazoli negli acidi nucleici, parassita |
| 1555 | Ammide II da proteine, rosso del globulo |
| 1530 | Ammide II, parassita |
| 1423 | B-DNA deossiribosio, parassita |
| 1397 | Ν(COO-) da lipidi, rosso del globulo |
| 1226 | ΝaPO4-da B-DNA, parassita |
| 1184 | B-DNA deossiribosio, parassita |
| 1083 | ΝaPO4-dal DNA, parassita |
| 1042 | ΝC = O dal ribosio RNA, parassita |
Tabella 1: assegnazioni di bande di coefficiente di regressione di PLS-R su spettri di malaria a spillo RBCs. In PLSR, il valore di parassitemia associato con uno spettro viene calcolato moltiplicando lo spettro pre-elaborato per il vettore di regressione. Quindi, il vettore di regressione raffigura l'importanza e la direzione delle bande IR nella regressione. Perché gli spettri sono stati pre-elaborati utilizzando il secondo derivato, bande negativi sono associati con una parassitemia superiore mentre bande positivi sono associati con una bassa parassitemia. Riprodotte e modificate da Martin, M. et al. 10 con il permesso della Royal Society of Chemistry.
Discussione
Modello: PLS-R è un metodo multivariato supervisione che trova una relazione lineare, Y = bX + E, tra le variabili predittive X (qui, l'assorbanza a ciascun numero d'onda) e un continuo variabile Y (qui, il livello di parassitemia). In breve, il modello combina le variabili in X per creare un nuovo insieme di variabili latenti (LVs) che catturano la varianza su X correlato con Y e calcola un vettore di regressione (b) che moltiplicato per un nuovo risultati di spettro nella stima del valore y. La forza del modello può essere preso da due valori: il valore di2 R e il valore quadratico medio errore di Cross convalida (RMSECV). Il primo descrive la linearità del modello e quindi la robustezza (il più vicino è a 1 meglio) e dati per i modelli con un valore di2 R inferiore a 0,8 devono essere esaminati attentamente per valori erratici e dati rumorosi. Il valore RMSECV viene descritto l'errore in cui può essere eseguita la stima. È sempre meglio cercare di ridurre al minimo questo numero per quanto possibile facendo in modo da escludere valori erratici, trattando con attenzione i dati e assicurandosi che non ci sono repliche biologiche come molti come possibile.
È quindi imperativo che i dati di buona qualità sono acquisiti per la costruzione del modello. Preparazione della diluizione serie è critico; in particolare assicurandosi che ogni campione di RBC è più possibile omogeneo. Utilizzando la pipetta per elaborare ed espellere i globuli rossi un paio di volte mentre agitando o sfogliando delicatamente l'estremità del tubo del microcentrifuge 5 – 10 volte, saranno evitate incoerenze. In una vena simile, è anche fondamentale per pulire il cristallo tra ogni campione per evitare la contaminazione. Utilizzando acqua ultra-pura e lanugine gratis salviettine per pulire il cristallo e controllando che la linea di base è piatta garantisce che non vi siano residui che possono contaminare la misurazione del campione successiva.
Il numero di campioni biologici necessari per un modello che si applica nel campo è almeno 30 a condizione che viene convalidato con un set di dati di un numero simile. Questo notevole volume sarebbe prendere una quantità significativa di generare e quindi questo è il limite della tecnica. Tuttavia, come altri campioni vengono analizzati correttamente può essere aggiunto al modello predittivo e rafforzarlo. Così, una volta stabilito, il modello può diventare più preciso e può giustificare lo sforzo necessario per configurarlo.
A seconda dell'ambiente e la strumentazione in cui vengono prese le misure, possono verificarsi diversi problemi maggiori. Se la linea non è completamente racchiuso nello strumento, significato o le articolazioni non sono ermetiche o la finestra ATR è intercambiabile con altri scomparti di campione, etanolo e vapore acqueo possono contaminare gli spettri. Etanolo appare come una band forte forte intorno 1.070/cm 14. Questo può essere evitato misurando in uno spazio libero da etanolo e disinfezione dello strumento, solo una volta che tutte le misurazioni. Vapore acqueo è causato da cambiamenti nell'atmosfera e viene visualizzato come piccoli picchi positivi e negativi taglienti intorno 1400 – 1.800/cm e 3.200-3.600/cm. Questo può essere risolto aumentando la frequenza delle misurazioni di sfondo e l'eliminazione dello strumento con azoto. In caso di eliminazione non è disponibile e il vapore acqueo è ancora abbastanza forte, lisciatura algoritmi e compensazione vapor d'acqua possa essere aggiunti al pre-trattamento dei dati per rimuoverli.
Questa tecnica tiene un sacco di promesse per la futura diagnostica e risultati per il paziente. In primo luogo, il metodo nel complesso è molto semplice da implementare in quanto il campione ha bisogno di essere dispensato solo sul cristallo ATR e poi si può essere misurata direttamente ed essere inserito nel modello riducendo il rischio di errore dell'utente. Questo eliminerebbe la necessità di microscopista luce altamente qualificato, che attualmente in alcune regioni dell'Africa mancano2. Grazie alla semplicità, è eliminata anche la necessità di costosi reagenti ed apparecchiature e quindi la diagnostica sarebbe praticamente gratuito per i pazienti, che è il limite principale per la PCR. Questa combinazione porterebbe a un numero maggiore di pazienti arrivano per diagnosi molto più presto e così migliorando i risultati pazienti. La tecnica ha il potenziale di sensibilità estremamente elevati che consentirebbe di superare la bassa sensibilità di RDTs e sarebbe in grado di rilevare elementi portanti asintomatici prima e quindi possono essere utilizzati come una tecnica di screening. Inoltre, la tecnica ha il potenziale per essere utilizzato per altre malattie, sangue borne o altrimenti, dove pochi microlitri o microgrammi possono essere applicata al cristallo.
Tuttavia, nel modello di esempio ci sono molte cose che devono essere affrontati prima che possa essere applicata nel campo. Validazione incrociata è uno a causa del basso numero di replicati biologici e che non è l'ideale; invece piuttosto avere una convalida separata impostata per testare il modello è molto meglio. Inoltre, il RMSECV è abbastanza alto qui rendendo i livelli inferiori di quantificazione inaffidabile. Aumentando il numero delle ripetizioni migliorerà questo valore e deve essere fatto prima di impiegare questo metodo nel campo. Ciò è confermato in laboratorio prove9, dove RMSECV era molto inferiore a causa delle dimensioni del campione più grande. Oltre a questo, aumentando i punti e durante l'intervallo miglioreranno anche questo valore come bene. Per migliorare ulteriormente la capacità diagnostica, ceppi selvatici di falciparum del p. dovrebbero essere usati, come il 7 3 p. falciparum è un ceppo di laboratorio e possono presentare una composizione chimica differente a causa delle variazioni nel fenotipo.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Finanziamenti agli autori è stato fornito dall'Australian Research Council (futuro Fellowship FT120100926 a BRW), National Health and Medical Research Council of Australia (programma grant e Senior Research Fellowship di JGB; Inizio carriera Fellowships JSR; Infrastruttura per la ricerca istituti Grant Scheme supporto all'Istituto Burnet), e supporta l'infrastruttura operativa del governo di Victorian State Grant all'Istituto di Burnet. Riconosciamo il signor Finlay Shanks per sostegno strumentale.
Materiali
| Name | Company | Catalog Number | Comments |
| Donor blood | - | - | Must be collected by a trained medical practitioner |
| Stock blood | Australian Red Cross` | - | |
| 3D7 Plasmodium falciparum | The Burnet Institute | - | Laboratory strain wild type equivalent |
| RPMI-1640 media with L-hepes without Sodium Bicarbonate | Sigma Aldrich | R6504 | |
| Albumax (Gibco) | Thermofisher | E003000PJ | Aliquot into 50 mL falcon tubes and store in freezer |
| Sodium Bicarbonate | Sigma Aldrich | S5761 | |
| Giemsa Stain | Sigma Aldrich | 48900 | |
| Sorbitol | Sigma Aldrich | S1876 | Must be filtered before use in culture |
| Blood collection tubes (Vacutainers) | Becton, Dickonson and Company | 367671 | |
| Immersion Oil | Thermofisher | M3004 | |
| 50 mL culture dishes | Falcon | 353025 | |
| 25mL pipette tips | Falcon | 357515 | |
| 10mL pipette tips | Falcon | 357530 | |
| Microscope Slides | Sigma Aldrich | S8902 | |
| Centrifuge tubes 50 mL | Sigma Aldrich | T2318 | |
| Automated pipette controller | Integra-biosciences | 155 015 | |
| Sorvall Legend X1R Centrifuge | Thermofisher | 75004260 | |
| Forma™ 310 Direct Heat CO2 Incubators | Thermofisher | 310TS | |
| Nikon Eclipse E-100 Binocular Microscope | Nikon Instruments | E100_2CE-MRTK-1 | When purchasing ensure that the 100x lens is an oil immersion lens |
| Bruker Alpha Ft-IR Spectrometer with ATR Quick Snap Attachment | Bruker | 9308-3700 | Be sure to request "Eco-ATR" attachment when purchasing |
| Matlab | Mathworks Inc | Multivariate data analysis software | |
| The Unscrambler X | CAMO | Multivariate data analysis software | |
| PLS-Toolbox | Mathworks, Inc. | GUI for Matlab |
Riferimenti
- Hommel, M., Gilles, H., Collier, L., Balows, A., Sussman, M. Chapter 20. Topley & Wilson's Microbiology and Microbial Infections. Vol. 5 Parasitology. , 361 (1998).
- World Health Organization. . World Malaria Report 2016. , (2016).
- Rogers, W., Murray, P., Baron, E., Pflaller, M., Tenover, F., Yolken, R. Chapter 105. Manual of Clinical Microbiology. , 1355 (1999).
- White, N. J. Chapter 9. Manson's Tropical Infectious Diseases. 23, 532-600 (2014).
- Lindblade, K. A., Steinhardt, L., Samuels, A., Kachur, S. P., Slutsker, L. The silent threat: asymptomatic parasitemia and malaria transmission. Expert Rev Anti Infect Ther. 11 (6), 623-639 (2013).
- Hanscheid, T., Grobusch, M. How useful is PCR in the diagnosis of malaria. Trends Parasitol. 18 (9), 395-398 (2002).
- Joanny, F., Löhr, S. J., Engleitner, T., Lell, B., Mordmüller, B. Limit of blank and limit of detection of Plasmodium falciparum thick blood smear microscopy in a routine setting in Central Africa. Malar J. 13 (1), 234-241 (2014).
- World Health Organisation. . Malaria Rapid Diagnostic Test Performance: results of WHO product testing of malaria RDTs: round 6 (2014-2015). , (2014).
- Khoshmanesh, A., et al. Detection and quantification of early-stage malaria parasites in laboratory infected erythrocytes by attenuated total reflectance infrared spectroscopy and multivariate analysis. Anal Chem. 86, 4379-4386 (2014).
- Martin, M., et al. The effect of common anticoagulants in detection and quantification of malaria parasitemia in human red blood cells by ATR-FTIR spectroscopy. Anal. 142 (8), 1192-1199 (2017).
- Fabian, H., Mäntele, W. . Handbook of Vibrational Spectroscopy. , (2006).
- Whelan, D., et al. Monitoring the reversible B to A-like transition of DNA in eukaryotic cells using Fourier transform infrared spectroscoptMonitoring the reversible B to A-like transition of DNA in eukaryotic cells using Fourier transform infrared spectroscopy. Nucleic Acid Res. 39 (13), 5439-5448 (2011).
- Wood, B. The importance of hydration and DNA conformation in interpreting infrared spectra of cells and tissues. Chem Soc Rev. 45, 1980-1998 (1980).
- Plyler, E. K. Infrared spectra of methanol, ethanol, and rn-propanol. J Res Natl Bur Stand. 48 (4), (1952).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon