Method Article
Erkennung und Quantifizierung von Plasmodium Falciparum in wässrigen roten Blutkörperchen durch gedämpft totale Reflexion Infrarotspektroskopie und Multivariate Datenanalyse
In diesem Artikel
Zusammenfassung
Hier präsentieren wir Ihnen ein Protokoll für die Erkennung und Quantifizierung von Plasmodium Falciparum infizierten wässrigen roten Blutkörperchen mit einem abgeschwächten Totalreflexion Infrarot-Spektrometer und multivariate Datenanalyse.
Zusammenfassung
Wir zeigen eine Methode zur Quantifizierung und Nachweis von Parasiten in wässrigen roten Blutkörperchen (Erythrozyten) mithilfe einer einfachen Benchtop abgeschwächt totale Reflexion Fourier transformieren (ATR-FTIR) Infrarotspektrometer in Verbindung mit Multivariate Datenanalyse ( MVDA). 3 7 p. Falciparum wurden kultiviert, 10 % Parasitemia Ring Bühne Parasiten und verwendet frische Spender isoliert Erythrozyten erstellen eine Verdünnung Serie zwischen 0 – 1 Spike %. 10 µL jeder Probe wurden aufgestellt, auf die Mitte des Fensters ATR Diamant, das Spektrum zu erwerben. Die Beispieldaten wurde behandelt, um das Signal-Rausch-Verhältnis zu verbessern und den Beitrag des Wassers zu entfernen, und dann die zweite Ableitung wurde angewandt, um die spektrale Eigenschaften zu lösen. Die Daten wurden dann mit zwei Arten von MVDA analysiert: erste Principal Komponente Analysis (PCA), Ausreißer und dann Partial Least Squares Regression (PLS-R) für die Quantifizierung Modellerstellung zu bestimmen.
Einleitung
Malaria zählt zu den verheerendsten Krankheiten unserer Zeit; mehr als die Hälfte die Bevölkerung lebt in endemischen Gebieten gefährdet und es unverhältnismäßig belastet die Armen1,2,3,4. Ein großer Teil des Problems ist die asymptomatische Träger und frühen Stadium-Patienten, die als Reservoir für Moskito Vektoren5, Handeln verursacht Spikes von Infektion während der Regenzeit und in den Gemeinden bestehen bleiben soll, so dass. Malaria wird durch fünf Plasmodium -Parasiten, verursacht die tödlichste von denen p. Falciparum ist die schwerste Form der Krankheit2verursacht.
Derzeit sind Techniken für die Diagnose von Malaria weniger als perfekt. Optische Mikroskopie, der derzeitige Goldstandard erkennt nur 62-88 Parasiten/µL je nach der verwendeten Methode6. Darüber hinaus aufgrund der Intensität und hohen Geschick erforderlich, in vielen Regionen Mikroskopie Fehldiagnosen > 50 % der Fälle, vor allem solche mit niedrigem Parasitaemia Ebenen2, in dem der Mangel an Ressourcen im Bereich und führt direkt zugeordnet werden können der Missbrauch von Anti-Malaria-Medikamenten. Die anderen 2 wichtigsten diagnostischen Methoden sind Schnelltests (RDTs), die Antikörper für die Erkennung und die Polymerase-Kettenreaktion (PCR) Assays, die Diskriminierung und Parasiten von DNA quantifizieren zu nutzen. RDTs können derzeit nur zur Erkennung von p. Falciparum und p. Vivax von mindestens 100 Parasiten/µL Blut was in selteneren Formen der Krankheit nicht behandelt wird. 7 , 8 dagegen PCR-Assays diskriminieren und quantifizieren, verschiedene Arten von Plasmodium bei einer Empfindlichkeit von 0,0004-5 Parasiten/µL Blut. Aber es erfordert teure Reagenzien, Ausrüstung und technisches Geschick und so eignet sich nicht für den Feldeinsatz.
Eine höchst sensible, zuverlässige und erschwingliche Technik unbedingt verbessern Diagnose Mal Behandlungsergebnisse zu verbessern, und die Beseitigung der Krankheit ermöglichen. Abgeschwächte totale Reflexion Fourier Transform (ATR-FTIR) Spektroskopie bietet eine mögliche Lösung für dieses Problem. Bisherigen Arbeit hat gezeigt, dass war es möglich zu erkennen und zu quantifizieren, p. Falciparum in Methanol fixiert Blut Filme erreichen die Nachweisgrenzen der < 1 Parasit/µL Blut (< 0,0002 % Parasitemia)9, das PCR-Verfahren vergleichbar ist. Neuere Studien haben gezeigt, dass es möglich ist zu erkennen und zu quantifizieren Parasiten in wässrigen Proben und eliminieren die Fixierung Schritt. Allerdings müssen Faktoren wie Wasserdampf, spektrale Rauschen und Datenverarbeitung für optimale Ergebnisse10berücksichtigt werden.
Dieses Protokoll zielt darauf ab, neue Benutzer zeigen, wie Sie erwerben FTIR-ATR Spektren und bereiten ein Regressionsmodell für die Erkennung von p. Falciparum von wässrigen Erythrozyten (RBC) Proben10.
Protokoll
Bitte konsultieren Sie geeignete Material Sicherheit Data Sheets (MSDS) und suchen Sie entsprechenden Biosafety Level 2 (BSL-2) Ausbildung zu. Alle Kultivierung Schritte erfolgen in einem BSL-2 Schrank mit aseptischen Technik, d. h. es gibt ein Risiko einer Exposition gegenüber schädlichen UV-Strahlung von Dekontamination Treppe, Nadel-Stick Verletzungen und möglichen biologischen Exposition und Infektion, wenn die Parasiten-Kultur tritt keine Verletzungen. Darüber hinaus ist bestand Blut von Blutbanken nur für bestimmte Krankheiten und das Potenzial für die Verbreitung von Blut übertragbaren Krankheiten untersucht ein potenzielles Risiko. Suchen Sie sofort medizinischen Hilfe in dem Auftreten von Verletzungen.
1. Vorbereitung und Messung von 3 7 Plasmodium Falciparum Parasit Verdünnungsreihe
Hinweis: Plasmodium SP. Kultur ist sehr empfindlich. Frisch/noch nicht abgelaufenen Reagenzien zu verwenden und die Parasiten durch Ändern der Medien regelmäßig zu füttern. Futtermittel-Kulturen unter 5 % Parasitemia jeden zweiten Tag. Kulturen zwischen 6-10 % Parasitemia einmal oder zweimal täglich zu füttern; und Kulturen zwischen 11 und 20 % bis zu 4 mal täglich zu füttern. Parasiten, die anfangen zu hungern verlieren ihre Form und beginnen, sich zusammenzuziehen. In einem solchen Fall füttern Sie und verdünnen Sie sofort von den Medien ändern und Hinzufügen von Lager Erythrozyten sammeln das Spenderblut in Blutentnahmeröhrchen mit Heparin als Antigerinnungsmittel und Maßnahme innerhalb der ersten 6 h.
- Kultur insgesamt 30 mL von 3-7 Belastung Plasmodium Falciparum Parasiten nach dem Standardprotokoll, bis die Parasitemia 10 % Ringe erreicht.
-
Synchronisation von Parasiten
- 11 h nach Shizogeny in den Lebenszyklus des Parasiten, aufzuwirbeln, die Kultur und den Transfer in ein 50 mL konische Röhrchen mit einer automatisierten Pipette.
- Zentrifugieren Sie die Kultur bei 300 – 400 x g und standard Laborbedingungen (25° C und 100 kPa) für 5 min.
- Entfernen Sie den Überstand durch Ausarbeitung mit einer Pipette oder mit einer Pasteurpipette angebracht zu einem Vakuum ohne zu stören das Pellet. Entsorgen Sie Abfall Medien in einem 10 % Bleichmittel-Lösung.
- Langsam das Pellet mit einem automatisierten Pipette 12 – 15 mL 4 % Sorbit-Lösung hinzu und mischen Sie die Kultur durch Kappung und invertieren das Rohr, bis die Kultur homogen ist.
- Inkubieren Sie die Kultur bei 37 ° C für 15 min.
- Zentrifugieren Sie die Kultur bei 300 – 400 x g und standard Laborbedingungen für 5 min.
- Den Überstand zu entfernen, wie in Schritt 1.2.3 beschrieben.
- Das Pellet 10 – 15 mL 0,9 % Kochsalzlösung hinzu und mischen Sie die Lösung durch Kappung und invertieren das Rohr, bis die Kultur homogen ist.
- Wiederholen Sie die Schritte 1.2.6–1.2.8 zweimal mehr, alle Reste von Sorbit zu waschen.
-
Vorbereitung der Baureihe Parasitemia Verdünnung
- Waschen Sie Lager Erythrozyten wie in den Schritten 1.2.6–1.2.9.
- Die Kultur bei standard Laborbedingungen, Lager Erythrozyten bei 300 – 400 X g für 5 min Zentrifugieren und überstand wie in Schritt 1.2.3 zu verwerfen.
- Label Mikrozentrifugenröhrchen als 0 %, 0,010 %, 0,025 %, 0.075 %, 0,100 %, 0.250 %, 0.750 % und 1.000 % und 0 µL, 1 µL, 2,5 µL, 7,5 µL, 10 µL, 25 µL, 75 µL und 100 µL der Kultur, die jeweils mit einer 0,2-20 µL Pipette dazugeben.
- Hinzu kommen 100 µL, 99 µL, 97,5 µL, 92,5 µL, 80 µL, 75 µL, 0 und 25 µL Lager RBCs die Röhren beschriftet 0 %, 0,010 %, 0,025 %, 0.075 %, 0,100 %, 0.250 %, 0.750 % und 1.000 % bzw..
- Zentrifugieren Sie die frischen Spenderblut gesammelt in Antikoagulans Rohre bei 1.200 x g und standard Laborbedingungen für 10 min.
- Entfernen Sie das Plasma durch Ausarbeitung mit einer Pipette und verwerfen, wie in Schritt 1.2.3 beschrieben.
- 900 µL isolierte Erythrozyten in jeder Microcentrifuge Schlauch und gründlich mischen durch 10 X invertieren.
-
Spektralen Erwerb
- Mit dem zugehörigen spektralen Erwerb Programm, klicken Sie auf "Instrument Set-up" und die Daten Sammlung Parameter Folgendes: 128 Scans für Hintergrund; 32-Scans für Probe; und die Auflösung auf 8/cm über die maximale Sample-Bereich des Instruments.
- Stellen Sie die Temperatur des ATR-FTIR-Spektrometers pro Empfehlung des Herstellers oder über Nacht.
- Reinigen Sie den Kristall sanft in kreisenden Bewegungen mit Flusen frei wischt mit Reinstwasser gedämpft schrubben. Dann trocknen Sie gründlich, mit einem anderen Flusen frei wischen.
- Nehmen Sie eine Hintergrundmessung der Luft, indem Sie auf "Hintergrundmessung". Alle 20 min, reinigen Sie den Kristall wie in Schritt 1.4.3 und wiederholen Sie diese Messung.
- Öffnen Sie die live-Ansicht durch Klicken auf "Vorschau". Beobachten Sie eine flache, horizontale Grundlinie, die angibt, dass das Glas sauber ist.
- Pipette 10 µL deionisiertes Wasser direkt auf die Mitte des Kristalls und klicken Sie auf "Maßnahme Sample". Wiederholen Sie die Messung dieses Wasser nach jeder fünften Probe.
- Trocknen Sie den Kristall ein Fussel freien abwischen mit sanften kreisenden Bewegungen.
- Pipette 10 µL der Probe, und klicken Sie auf "Maßnahme Sample".
- Reinigen Sie den Kristall zwischen Proben wie in Schritt 1.4.3.
- Wiederholen Sie, bis 3 Wiederholungen erworben werden, und achten Sie auf die Reihenfolge der Proben und die Wiederholungen zu randomisieren.
2. Multivariate Datenanalyse (MVDA)
Hinweis: Datenbehandlung muss über das gesamte Dataset durchgeführt werden, um Lärm und die Zugabe von Gipfeln nicht biologischen Ursprungs zu vermeiden. Im Gegensatz dazu über die biologisch relevanten Regionen analysieren: 2980-2800/cm und 1750-850/cm.
-
Verarbeitung der Daten
Hinweis: Zwei Software, z. B. Matlab (im folgenden als Software 1 bezeichnet) und The Flaschenaufrichter X (im folgenden als Software 2 bezeichnet) dienen als Beispiele für MVDA. 1-Software hat die Fähigkeit der Ausführung MVDA ohne Zusatz von eine grafische Benutzeroberfläche (GUI). Es wird jedoch empfohlen, eine GUI (Table of Materials) zu kaufen. Die folgenden Anweisungen, wenn bezogen auf Software 1 annehmen, dass in Verbindung mit den im Handel erhältlichen GUI-Toolbox, und Behandlung Beispielskript Daten angehängt wurde.- Öffnen Sie die entsprechenden multivariate Datenanalyse-Software zu, klicken Sie auf "Daten importieren" und wählen Sie den Typ der Datei für die Analyse.
- In der Software 1 Eingang "Analyse" in das Befehlsfenster, die GUI zu öffnen. Rechten Maustaste die X-Box, um "Daten importieren" zu finden.
- Software 2 Klicken Sie auf die Registerkarte "Datei" "Importieren" zu finden.
- Importieren Sie Probe, Wasser und Basislinie Spektren als Datensätze in den Arbeitsbereich durch alle Spektren in jedem Satz separat auswählen und auf "Öffnen" klicken und geben Sie jedem Satz einen kurzen Namen, z. B. "Wat" für Dataset Wasser Spektren.
- Wählen Sie neue Tabelle/Matrix durch Klicken auf "neue Matrix/Vektor" und erzeugen Sie einen n × 1-Vektor, wobei n die Anzahl der Samples ist. Geben Sie die Parasitemia jeder Probe und geben Sie dem Vektor der Name "Parasitemia".
- 1 Software klicken Sie auf "Neue Variable" im Befehlsfenster.
- Software 2 Klicken Sie auf das Symbol "Neue Matrix".
- Plot-Daten durch Anklicken der "Plot-Daten". Inspizieren Sie die Spektren für Wasserdampf Effekte durch Klicken auf "Zoom" und Zoomen auf 1800-1400/cm; am deutlichsten beobachtete als kurze, scharfe, schmale Peaks entlang der Hänge der Amid ich und Amid II Bands.
- Öffnen Sie in Fällen von extremen Wasserdampf Registerkarte "Bearbeiten/pre-process Data" zu, und wählen Sie "Glättung". Reduzieren Sie die Geräusche bzw. der starken Wasserdampf Beiträge durch Glätten der Probe und Wasser Spektren mit bis zu 25 Punkten der Glättung oder verwenden Sie eine Wasserdampf-Korrektur-Methode.
- Richtig nicht-horizontalen Basislinie mithilfe des Grundlinie Korrektur Algorithmus gegebenenfalls unter der gleichen Registerkarte bearbeiten/pre-Prozess Daten in Schritt 2.1.4.
- Durchschnittliche Wasser Spektren und kopieren Sie die Zeilen in einer n × m Matrix entspricht der Beispiel-Dataset und reduzieren sie auf 70 % Intensität mit 0,7 multipliziert.
- In der Software 1 dazu im Befehlsfenster Eingabe das folgende Skript "AverageWater=mean(WaterDataset)". Dann kopieren und Einfügen der Zeilen entsprechend dem Beispieldatensatz. Um die Intensität zu reduzieren, geben Sie "AverageWater70 = AverageWater * 0,70".
- Durschnittliche Spektren von jedem Probenspektrum abziehen.
- In der Software 1, tun dies im Befehlsfenster durch Eingabe des folgenden Skripts "WaterCorrectedData = SampleDataset-AverageWater70".
- Öffnen Sie die Registerkarte bearbeiten/pre-Prozess Daten um eine zweite abgeleitete Funktion anwenden und dann die Daten durch Auswahl der einzelnen normalen Variate (SNV) Funktion zu normalisieren und Center-Daten bedeuten.
- In der Software 1 tun dies in einem Rutsch. Wählen Sie zuerst "Derivat" und Eingabe 25 Punkte der Glättung, Polynomgrad 3 und abgeleitete Ordnung auf das Sample-set mit 25 Punkten der Glättung und eine Savitzky-Golay-Funktion. Wählen Sie dann "SNV" und "Meine Mitte". Klicken Sie auf "OK/übernehmen".
- Öffnen Sie die Registerkarte "Bearbeiten/pre-Prozess-Daten" und wählen Sie im "Spalte Variablen", 2.980 – 2.800/cm und 1.750 – 850/cm indem sichergestellt wird, dass nur ihre Kästchen angekreuzt sind.
- Öffnen Sie die entsprechenden multivariate Datenanalyse-Software zu, klicken Sie auf "Daten importieren" und wählen Sie den Typ der Datei für die Analyse.
-
Datenanalyse
- Principal Component Analysis (PCA)
- Klicken Sie auf "Analyse | Decomposition"und wählen Sie PKA.
- 1 Software klicken Sie auf "Modell zu bauen".
- In 2-Software die Eingabemethoden Sie PCA. Die optimale Anzahl der wichtigsten Komponenten (PCs) - in dieser Arbeit, 7 - Eingang und einem Maximum von 100 Iterationen und ausgewählte Kreuzvalidierung für die Validierungsmethode. Klicken Sie auf "Ausführen".
- Beobachten Sie die 95 % Vertrauensgrenze, die gestrichelte Ring, auf dem Punkte-Grundstück zwischen PC1 und PC2.
- Markieren Sie die Probe-Spektren mit Noten, die außerhalb der Grenze als potenzielle Ausreißer mit dem "wählen Sie Spectra-Tool" auftreten.
- Wenn die markierte Spektren haben schließen Hotellings T2 Werte und hohe Q Residuen sie von der weiteren Analyse.
- Klicken Sie auf "Analyse | Decomposition"und wählen Sie PKA.
- Partial Least Squares Regression (PLS-R)
- Klicken Sie auf "Analyse | Regression"und wählen Sie"PLS-R".
- In Software 1, Y-Block rechts klicken und wählen Sie den Vektor "Parasitemia | Bauen Sie Modell".
- In 2-Software die Eingabemethoden Sie PLS-R. Wählen Sie den Vektor "Parasitemia" als "Y-Referenz" und die Beispiel-Dataset als "X Data Set" und wählen Sie Kreuzvalidierung als die Validierungsmethode. Klicken Sie auf "Ausführen".
- Analysieren Sie das Regressionsmodell. R2 Werte über 0,80 und Root-Mean-Square-Fehler der Kreuzvalidierung (RMSECV) Werte, die kleiner als 0,1 % Parasitemia sind akzeptabel.
- Analysieren Sie den Vektor der Regression und identifizieren Sie die biologische Bands.
- Klicken Sie auf "Analyse | Regression"und wählen Sie"PLS-R".
- Principal Component Analysis (PCA)
Ergebnisse
Partial Least Squares (PLS-R) plot und seiner damit verbundenen Regression Vektor, Abbildung 1a und 1 b bzw. zeigen, dass das Signal vom Parasiten unterscheidet die Erythrozyten, die sie verwendet werden können, bilden eine lineare Regression Modell für verwendet werden die Vorhersage des Parasiten in zukünftigen Datensätzen.
Eine robuste, lineare PLS-R-Modell wurde mit einem Bestimmtheitsmaß von 0,87 generiert und ein Root Mean squared Kreuzvalidierung Fehler (RMSECV) von 0,13 % Parasitemia, Abbildung 1a. Parasitemia mit zunehmender sind vor allem in erster Linie mit Nukleinsäure-Bands, 1042, 1083 und 1226/cm, die die ν(C=O) von der RNA Ribose zugeordnet sind gruppieren, νs(PO4 -) und νein(PO4 -) bzw. und bilden der größte negative Teil der Regression-Vektor. Dies ist da Erythrozyten einen Kern fehlt und Nukleinsäuren somit starke Indikatoren für die Parasiten sind. Darüber hinaus zeigt die 1226/cm Band, dass die Struktur eine B-DNA-Form die unterstreicht die Bedeutung hat des Messens in der wässrigen Zustand12. Im Gegensatz dazu Parasiten RBC Komponenten, in erster Linie Hämoglobin, nutzen und somit einen höheren Proteingehalt Lipid-Verhältnis dürfte in nicht infizierten Erythrozyten wie angegeben durch Lipid Streifen 2913, 2879 und 1397 cm-1 und 1631 und 1555 cm-1 aus dem Amid ich und Amid II Modi12,13.
Die unterschiedlichen Bands zeigen deutlich, dass die Signale wahr biologischen Bands sind eher als Differenzierung unechte Bands nicht biologischen Ursprungs wie zum Beispiel Wasserdampf, zugrunde, die als enge und intensive Bands präsentieren. Das Vorhandensein von DNA-Bänder, Tabelle 1, kann insbesondere der Parasit11,12,13direkt zugeschrieben werden. Im Allgemeinen kann der Regression Vektor in einer ähnlichen Weise zu Belastungen im PKA interpretiert werden. Bands sollten gut mit Bands und Verschiebungen der ursprünglichen Spektren korrelieren.
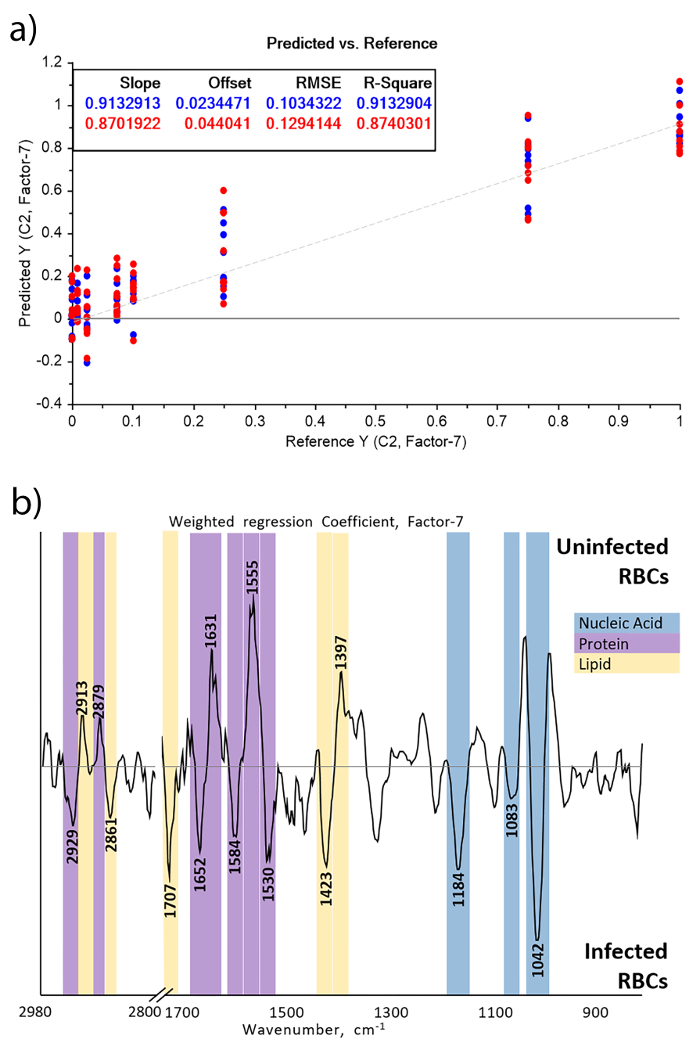
Abbildung 1: PLS-R Regression Grundstück, (a), der Erythrozyten versetzt zwischen 0-1 % Parasitemia und damit verbundenen Regressionskoeffizienten, (b). Die Regression-Plot zeigt die Fähigkeit des Modells zur Vorhersage der Parasitemia in jeder Probe, mit der bekannten Parasitemia auf der x-Achse und der vorhergesagten Parasitemia auf der y-Achse. Die Regressionskoeffizienten beschreibt den Beitrag der einzelnen Bänder um die Prognosefähigkeit des Modells, desto intensiver ist das Band, desto mehr trägt sie das Modell und damit, wie der Parasit die chemische Zusammensetzung des Blutes verändert. Reproduziert und modifiziert von Martin, M. Et al. 10 mit freundlicher Genehmigung von der Royal Society of Chemistry. Bitte klicken Sie hier für eine größere Version dieser Figur.
| Regression Koeffizient Band (cm-1) | Assignment25-27 |
| 2929 | ΝaCH2 von Lipiden, Parasit |
| 2913 | ΝaCH2 von Lipiden, rote Blutkörperchen |
| 2879 | ΝsCH3 von Lipiden, rote Blutkörperchen |
| 2861 | ΝsCH2 von Lipiden, Parasit |
| 1707 | B-DNA/A-DNA-Basenpaare Vibration νC = O & νC = N, Parasit |
| 1652 | Amid I von α-Helix-Proteine, Parasit |
| 1631 | Amid I von β-gefaltetes Blatt Proteine, rote Blutkörperchen |
| 1584 | ΝC = N von Imidazoles in Nukleinsäuren, Parasit |
| 1555 | Amid II von Proteine, rote Blutkörperchen |
| 1530 | Amid II, Parasit |
| 1423 | B-DNA Deoxyribose, Parasit |
| 1397 | Ν(COO-) von Lipiden, rote Blutkörperchen |
| 1226 | ΝaPO4-von der B-DNA, Parasit |
| 1184 | B-DNA Deoxyribose, Parasit |
| 1083 | ΝaPO4-aus DNA, Parasit |
| 1042 | ΝC = O aus RNA Ribose, Parasit |
Tabelle 1: Zuordnungen der Regression Koeffizient Bands der PLS-R auf Spektren von Malaria versetzt Erythrozyten. In PLSR wird ein Spektrum zugeordnete Parasitemia Wert berechnet durch Multiplikation der vorverarbeiteten Spektrums mit der Regression-Vektor. Daher zeigt der Regression-Vektor die Bedeutung und die Richtung der IR-Bands in der Regression. Da Spektren mit Hilfe der zweiten Ableitung vorverarbeitet wurden, sind negative Bands mit einer höheren Parasitemia assoziiert, während positive Bänder mit einer niedrigen Parasitemia verbunden sind. Reproduziert und modifiziert von Martin, M. Et al. 10 mit freundlicher Genehmigung von der Royal Society of Chemistry.
Diskussion
PLS-R-Modell ist eine betreute multivariate Methode, die findet eine lineare Beziehung, Y = bX + E, zwischen den prädiktiven Variablen X (hier: der Extinktion bei einzelnen Wellenzahl) und eine kontinuierliche Variable Y (hier: der Parasitemia-Ebene). Kurz gesagt, das Modell verbindet die Variablen X, um einen neuen Satz von latenten Variablen (LVs) erstellen, die die Varianz auf X korreliert mit Y zu erfassen, und einen Regression Vektor (b) berechnet, die ein neues Spektrum Ergebnisse bei der Schätzung der y-Wert multipliziert. Die Stärke des Modells zwei Werte entnommen werden kann: die R2 und dem Root Mean Square Error von Cross Validation (RMSECV) Wert. Erstere beschreibt die Linearität des Modells und somit die Robustheit (je näher es 1 desto besser) und Daten für Modelle mit einem R2 Wert weniger als 0,8 für Ausreißer und verrauschten Daten sorgfältig überprüft werden müssen. Der RMSECV-Wert beschreibt den Fehler in dem Vorhersage gemacht werden kann. Es ist immer am besten ausprobieren und diese Zahl so weit wie möglich zu minimieren, indem Sie sicherstellen, dass auszuschließende Ausreißer, die Daten sorgfältig zu behandeln und dafür sorgen, dass es so viele biologische Wiederholungen wie möglich sind.
Es ist daher zwingend notwendig, dass Daten von guter Qualität, zum Erstellen des Modells erworben werden. Vorbereitung der Baureihe Verdünnung ist entscheidend; speziell dafür sorgen, dass jede RBC Probe möglichst homogen ist. Mithilfe der Pipette zu erarbeiten und Auswerfen der Erythrozyten ein paar Mal während der wirbelnden oder sanft streichen Microcentrifuge Rohrende 5 – 10 Mal werden Inkonsistenzen vermieden werden. In ähnlicher Weise ist es auch wichtig, reinigen Sie den Kristall zwischen jeder Probe um Kontaminationen zu vermeiden. Mit Reinstwasser und Flusen frei Tücher reinigen das Glas und prüfen, ob die Grundlinie flach ist sorgt dafür, dass es gibt keine Rückstände, die die nächste Probenmessung verunreinigen werden.
Die Anzahl der biologischen Proben für ein Modell, das im Bereich anwendbar ist erforderlich ist mindestens 30 pro Zustand, der mit einem Dataset eine ähnliche Zahl überprüft wird. Diese erhebliche Mengen würde eine erhebliche Menge zu erzeugen und somit ist dies die Beschränkung der Technik. Jedoch da mehr Proben korrekt analysiert werden kann das Vorhersagemodell hinzugefügt werden und stärken. Also, einmal etabliert, das Modell kann genauer und rechtfertigt den Aufwand um es einzurichten.
Abhängig von der Umgebung und die Instrumentation auf die Messungen durchgeführt werden, können verschiedene weitere Probleme entstehen. Wenn das Strahlrohr nicht vollständig im Gerät beiliegt, können also entweder die Gelenke sind nicht luftdicht oder das ATR-Fenster ist austauschbar mit anderen Fächern Probe Ethanol und Wasserdampf die Spektren verunreinigen. Ethanol wird als starke scharfe Band um 1.070/cm 14. Dies kann vermieden werden durch die Messung in einem Raum frei von Ethanol und Desinfektion das Instrument nur einmal alle Messungen durchgeführt werden. Wasserdampf wird durch Veränderungen in der Atmosphäre verursacht und erscheint als kleine scharfe positive und negative Gipfel rund um 1400 – 1.800 /cm und 3.200 – 3.600/cm. Dies kann durch Erhöhung der Häufigkeit der Hintergrundmessungen und spülen das Gerät mit Stickstoff gelöst werden. In Fällen wo das leeren ist nicht verfügbar und der Wasserdampf ist immer noch ziemlich stark kann Glättung Algorithmen und Wasserdampf Entschädigung Vorverarbeitung der Daten zu entfernen hinzugefügt werden.
Diese Technik hält sehr viel versprechend für zukünftige Diagnostik und Behandlungsergebnisse. Erstens ist die Methode insgesamt sehr einfach zu implementieren, dass die Probe nur auf die ATR-Kristall pipettiert muss und anschließend direkt gemessen werden und in das Modell reduziert das Potenzial für Benutzerfehler eingegeben werden. Dies würde für hochqualifizierte leichte Mikroskopiker überflüssig machen, das derzeit in manchen Regionen Afrikas sind2fehlt. Aufgrund der Einfachheit, entfällt auch die Notwendigkeit für teure Reagenzien und Geräte und damit die Diagnose wäre praktisch Kosten-frei für Patienten, die die große Einschränkung für die PCR ist. Diese Kombination führt zu höheren Anzahl an Patienten kommt zur Diagnose schneller und damit Verbesserung der Behandlungsergebnisse. Die Technik hat das Potential für die extrem hohen Empfindlichkeiten, die die niedrigen Empfindlichkeiten der RDTs überwinden würde und wäre in der Lage zu erkennen, dass asymptomatische Träger früher und damit als ein Screening-Verfahren verwendet werden können. Darüber hinaus hat die Technik das Potenzial, für andere Krankheiten verwendet werden, Blut getragen oder anderweitig, wo wenige Mikroliter oder Mikrogramm auf den Kristall angewendet werden können.
Jedoch in der Beispielmodell gibt es viele Dinge, die müssen angegangen werden, bevor sie in das Feld angewendet werden kann. Kreuzvalidierung gehört aufgrund der geringen Anzahl von biologischen Wiederholungen und das ist nicht ideal; eher ist eine separate Bestätigung eingestellt, das Modell zu testen stattdessen viel besser. Darüber hinaus ist die RMSECV ziemlich hoch, hier machen die unteren Ebenen der Quantifizierung unzuverlässig. Erhöhung der Anzahl der Wiederholungen wird dieser Wert zu verbessern und vor dem Einsatz dieser Methode auf dem Gebiet getan werden muss. Dies wird im Labor Versuche9, bestätigt, wo RMSECV aufgrund der Stichprobengröße deutlich niedriger war. Darüber hinaus verbessert die Intervall-Palette und Punkte erhöhen auch dieser Wert auch. Zur weiteren Verbesserung der Diagnosefähigkeit, wilden Stämme von p. Falciparum sollte verwendet werden, als die 3-7 p. Falciparum ist ein Laborstamm und kann eine andere chemische Zusammensetzung aufgrund von Schwankungen im Phänotyp.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Zu den Autoren wurde gefördert vom Australian Research Council (Zukunft Fellowship FT120100926, BRW), National Health and Medical Research Council of Australia (Programm Grant und Senior Research Fellowship zu JGB; Frühe Karriere Stipendien JSR; Infrastruktur für die Forschung Institute Support Scheme Grant Burnet Institut), und die Victorian State Regierung betrieblichen Infrastruktur unterstützen Grant Burnet Institut. Wir bestätigen Herrn Finlay Shanks für instrumentale Unterstützung.
Materialien
| Name | Company | Catalog Number | Comments |
| Donor blood | - | - | Must be collected by a trained medical practitioner |
| Stock blood | Australian Red Cross` | - | |
| 3D7 Plasmodium falciparum | The Burnet Institute | - | Laboratory strain wild type equivalent |
| RPMI-1640 media with L-hepes without Sodium Bicarbonate | Sigma Aldrich | R6504 | |
| Albumax (Gibco) | Thermofisher | E003000PJ | Aliquot into 50 mL falcon tubes and store in freezer |
| Sodium Bicarbonate | Sigma Aldrich | S5761 | |
| Giemsa Stain | Sigma Aldrich | 48900 | |
| Sorbitol | Sigma Aldrich | S1876 | Must be filtered before use in culture |
| Blood collection tubes (Vacutainers) | Becton, Dickonson and Company | 367671 | |
| Immersion Oil | Thermofisher | M3004 | |
| 50 mL culture dishes | Falcon | 353025 | |
| 25mL pipette tips | Falcon | 357515 | |
| 10mL pipette tips | Falcon | 357530 | |
| Microscope Slides | Sigma Aldrich | S8902 | |
| Centrifuge tubes 50 mL | Sigma Aldrich | T2318 | |
| Automated pipette controller | Integra-biosciences | 155 015 | |
| Sorvall Legend X1R Centrifuge | Thermofisher | 75004260 | |
| Forma™ 310 Direct Heat CO2 Incubators | Thermofisher | 310TS | |
| Nikon Eclipse E-100 Binocular Microscope | Nikon Instruments | E100_2CE-MRTK-1 | When purchasing ensure that the 100x lens is an oil immersion lens |
| Bruker Alpha Ft-IR Spectrometer with ATR Quick Snap Attachment | Bruker | 9308-3700 | Be sure to request "Eco-ATR" attachment when purchasing |
| Matlab | Mathworks Inc | Multivariate data analysis software | |
| The Unscrambler X | CAMO | Multivariate data analysis software | |
| PLS-Toolbox | Mathworks, Inc. | GUI for Matlab |
Referenzen
- Hommel, M., Gilles, H., Collier, L., Balows, A., Sussman, M. Chapter 20. Topley & Wilson's Microbiology and Microbial Infections. Vol. 5 Parasitology. , 361 (1998).
- World Health Organization. . World Malaria Report 2016. , (2016).
- Rogers, W., Murray, P., Baron, E., Pflaller, M., Tenover, F., Yolken, R. Chapter 105. Manual of Clinical Microbiology. , 1355 (1999).
- White, N. J. Chapter 9. Manson's Tropical Infectious Diseases. 23, 532-600 (2014).
- Lindblade, K. A., Steinhardt, L., Samuels, A., Kachur, S. P., Slutsker, L. The silent threat: asymptomatic parasitemia and malaria transmission. Expert Rev Anti Infect Ther. 11 (6), 623-639 (2013).
- Hanscheid, T., Grobusch, M. How useful is PCR in the diagnosis of malaria. Trends Parasitol. 18 (9), 395-398 (2002).
- Joanny, F., Löhr, S. J., Engleitner, T., Lell, B., Mordmüller, B. Limit of blank and limit of detection of Plasmodium falciparum thick blood smear microscopy in a routine setting in Central Africa. Malar J. 13 (1), 234-241 (2014).
- World Health Organisation. . Malaria Rapid Diagnostic Test Performance: results of WHO product testing of malaria RDTs: round 6 (2014-2015). , (2014).
- Khoshmanesh, A., et al. Detection and quantification of early-stage malaria parasites in laboratory infected erythrocytes by attenuated total reflectance infrared spectroscopy and multivariate analysis. Anal Chem. 86, 4379-4386 (2014).
- Martin, M., et al. The effect of common anticoagulants in detection and quantification of malaria parasitemia in human red blood cells by ATR-FTIR spectroscopy. Anal. 142 (8), 1192-1199 (2017).
- Fabian, H., Mäntele, W. . Handbook of Vibrational Spectroscopy. , (2006).
- Whelan, D., et al. Monitoring the reversible B to A-like transition of DNA in eukaryotic cells using Fourier transform infrared spectroscoptMonitoring the reversible B to A-like transition of DNA in eukaryotic cells using Fourier transform infrared spectroscopy. Nucleic Acid Res. 39 (13), 5439-5448 (2011).
- Wood, B. The importance of hydration and DNA conformation in interpreting infrared spectra of cells and tissues. Chem Soc Rev. 45, 1980-1998 (1980).
- Plyler, E. K. Infrared spectra of methanol, ethanol, and rn-propanol. J Res Natl Bur Stand. 48 (4), (1952).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten