Method Article
Capture spettrometria di massa Compound - un potente strumento per identificare nuovi c-di-GMP effettori Proteine
In questo articolo
Riepilogo
The ubiquitous second messenger c-di-GMP controls growth and behavior of many bacteria. We have developed a novel Capture Compound Mass Spectrometry based technology to biochemically identify and characterize c-di-GMP binding proteins in virtually any bacterial species.
Abstract
Sono stati compiuti notevoli progressi nel corso degli ultimi dieci anni verso l'identificazione e la caratterizzazione di enzimi coinvolti nella sintesi (ciclasi diguanylate) e di degradazione (fosfodiesterasi) del secondo messaggero c-di-GMP. Al contrario, sono disponibili poche informazioni sui meccanismi molecolari e componenti cellulari attraverso cui questa molecola di segnalazione regolamenta una vasta gamma di processi cellulari. La maggior parte delle proteine effettrici noti appartengono alla famiglia Pilz o sono degenerati ciclasi diguanylate o fosfodiesterasi che hanno rinunciato a catalisi e hanno adottato la funzione effettrici. Così, per definire meglio la rete c-di-GMP cellulare in una vasta gamma di batteri metodi sperimentali sono necessari per identificare e convalidare nuovi effettori che affidabile e in silico previsioni FAIL.
Abbiamo recentemente sviluppato un nuovo Capture spettrometria di massa Compound (CCMS) tecnologia basata come un potente strumento perbiochimicamente identificare e caratterizzare le proteine di legame c-di-GMP. Questa tecnica è stata precedentemente riportato di essere applicabile ad una vasta gamma di organismi 1. Qui diamo una descrizione dettagliata del protocollo che utilizziamo per sondare tali componenti di segnalazione. Come esempio, si usa Pseudomonas aeruginosa, un patogeno opportunista in cui c-di-GMP gioca un ruolo critico nella virulenza e biofilm controllo. CCMS ha identificato il 74% (38/51) dei componenti note o previste della rete c-di-GMP. Questo studio spiega la procedura CCMS in dettaglio, e stabilisce come uno strumento potente e versatile per identificare nuovi componenti coinvolti in piccola molecola di segnalazione.
Introduzione
c-di-GMP è una chiave secondo messaggero utilizzato dalla maggior parte dei batteri per controllare vari aspetti della loro crescita e il comportamento. Per esempio, c-di-GMP regola la progressione del ciclo cellulare, la motilità e l'espressione di esopolisaccaridi e adesine superficiali 2-4. Attraverso il coordinamento di tali processi c-di-GMP promuove la formazione di biofilm, un processo che è associata con infezioni croniche di una gamma di batteri patogeni 5. c-di-GMP è sintetizzato da enzimi chiamati ciclasi diguanylate (DGCS) che ospitano un dominio catalitico GGDEF 4. Alcuni DGCS possiedono un sito inibitorio che regola le attività ciclasi su c-di-GMP vincolante. Il degrado di c-di-GMP è catalizzata da due classi distinte di fosfodiesterasi (PDE) che ospitano sia un EAL catalitico o HD-GYP dominio 6,7.
La maggior parte delle proteine effettrici noti che legano direttamente c-di-GMP appartengono a una delle tre classi di proteine: cataliticoGGDEF o EAL domini alleato inattivi e domini Pilz, piccoli interruttori molecolari che subiscono cambiamenti conformazionali su c-di-GMP vincolante 8. DGCS, PDE e Pilz proteine sono ben caratterizzati e loro domini possono essere previsti in silico relativamente sicuro. Un particolare interesse è ora focalizzata sulla identificazione di nuove classi di effettori c-di-GMP. Alcuni effettori c-di-GMP con diversi motivi di legame sono stati descritti recentemente come la CRP / FNR Bcam1349 famiglia di proteine in Burkholderia cenocepacia o FleQ regolatore trascrizionale in P. aeruginosa 9,10. Inoltre, riboswitches GMP-specifici c-di-stati recentemente identificati e indicati per controllare l'espressione genica in modo c-di-GMP-dipendente 11. I motivi c-di-GMP vincolanti di diversi effettori solo sono mal conservati fare previsioni bioinformatici di tali proteine difficile. Per risolvere questo problema, abbiamo sviluppato un metodo biochimico, che si basa sull'utilizzo di una spe c-di-GMPfica Compound Capture combinata con spettrometria di massa 1,12,13.
Abbiamo recentemente abbiamo progettato un nuovo trivalente c-di-GMP Compound Capture (CDG-CC, Figura 1) 1. Questo scaffold chimica è composto da: 1) un residuo c-di-GMP utilizzata come esca per catturare proteine leganti c-di-GMP, 2) un gruppo reattivo UV-fotoattivabile utilizzato per attraversare link CDG-CC alle proteine legate e 3) un biotina per isolare le proteine catturate utilizzando sfere magnetiche rivestite di streptavidina. CDG-CC può essere usato per catturare direttamente e specificamente effettori c-di-GMP dalla miscela complessa di macromolecole come lisati cellulari. Compound Capture basano e approcci proteomica basati chimici sono stati precedentemente segnalati per essere applicabile ad una vasta gamma di organismi, ad esempio Caulobacter crescentus, Salmonella enterica sierotipo typhimurium e P. aeruginosa 1,14.
In questo documento metodologico,forniamo una descrizione in profondità della procedura CCMS utilizzando estratti di P. aeruginosa come esempio. Questo studio stabilisce CCMS come uno strumento potente e versatile per identificare biochimicamente nuovi componenti che praticano la piccola molecola di segnalazione.
Protocollo
1. Preparazione del lisato
- Grow P. cellule aeruginosa in LB alla OD desiderata.
NOTA: Per la guida: usare ≈ 100 ml cultura / campione per le culture di fase stazionaria e ≈ 500 ml cultur / campione per le culture di fase log (OD 600nm = 0.5). - Pellet mediante centrifugazione per 20 min a 5000 x g.
- Risospendere 0.5-1 g di pellet in 1 ml di tampone di lisi (MES 6,7 mm, 6,7 mM Hepes, NaCl 200 mm, 6,7 mM Kac, DDT 1 mM, pH 7,5) e aggiungere inibitore della proteasi (completo mini, senza EDTA) e come DNaseI.
- Lyse le cellule 3 passaggi attraverso una cella di pressione francese, a 20.000 psi (vedi Materiali List).
- Ultra-centrifuga il lisato cellulare a 100.000 xg per 1 ora a 4 ° C.
- Salvare il surnatante (passare al punto 2).
- Lavare il pellet con 1 ml di tampone di lisi 1x pipettando su e giù.
- Ultracentrifuga a 100.000 x g per 1 ora a 4 ° C.
- Flash-congelare il pellet inazoto liquido e conservare a -20 ° C fino utilizzato per la cattura di proteine di membrana (vedi punto 3).
2. Rimozione dei nucleotidi liberi c-di-GMP e altri (frazione solubile Only)
- Lavare una colonna PD10 dissalazione (vedi Materiali List) con 10 ml di tampone di lisi freddo.
- Versare il supernatante (≈ 1 ml) sul PD10 per rimuovere nucleotidi.
- Eluire con 4 ml di tampone di lisi freddo (500 gradini microlitri).
- Selezionare le frazioni più concentrati, come determinato dal saggio Bradford, e piscina.
(Solo Frazione Membrane) 3. Pellet Resuspension e solubilizzazione
- Risospendere il pellet in 500 a 1.000 ml di buffer di acquisizione 1x (senza detersivo) (vedi tabella 1).
NOTA: Il pellet è difficile da riportare in sospensione. Si consiglia di prima pipetta su e giù con una pipetta per risospendere il pellet circa, poi utilizzare una siringa 27 G per omogeneizzare bene la soluzione. - Aggiungere 1% (w / v) di n-dodecil-β-D-maltopyranoside (DDM).
- Incubare a 4 ° C per almeno 2 ore (o O / N) su una ruota girevole.
- Ultracentrifuga a 100.000 x g per 1 ora a 4 ° C.
- Raccogliere il surnatante.
4. Protein misura della concentrazione
- Misurare la concentrazione di proteine mediante saggio Bradford (mediante saggio BCA per la frazione di membrana).
- Impostare la concentrazione totale di proteine a 10 mg / ml.
5. Capture
- Miscelare 300 mg di proteina con 1 mM di PIL, GTP, ATP, CTP, e 20 microlitri di tampone 5x cattura (HEPES 100 mM, 250 mM KAC, 50 MgAc mM, 5% glicerolo, pH 7,5) (Tabella 1). Regolare il volume di reazione di 100 microlitri con H 2 O.
NOTA: il volume totale comprende il volume del CDG-CC e c-di-GMP nel caso della concorrenza controllo (vedere fase 5.3 e tabella 2).
NOTA: tutti gli esperimenti sono stati eseguiti in 200 &# 181; l strisce PCR 12-tubo (Thermo Scientific).
NOTA: per la frazione di membrana, assicurarsi di mantenere sempre la concentrazione DDM sopra della concentrazione micellare critica (0,01% w / v) fino alla fase solubilizzazione delle proteine in 8 M Urea (punto 7.1). - Incubare a 4 ° C per 30 minuti su una ruota girevole.
- Aggiungere 10 mM CDG-CC (concentrazione finale).
NOTA: includere un controllo senza CDG-CC (indicato come "controllo tallone"), e un controllo integrato con 1 mM c-di-GMP ("controllo della concorrenza") (vedi tabella 2).
NOTA: la concentrazione di CDG-CC può essere regolato da 1 a 10 micron. - Incubare a 4 ° C per almeno 2 ore (/ N per la frazione di membrana O) su una ruota girevole, al buio.
- Dopo un breve giro, cross-link per l'attivazione della porzione reattiva della CDG-CC con la luce UV per 4 minuti, utilizzando un CaproBox (vedi lista materiali, λ = 310 nm, Irradiance ≥10 mW / cm², distanza dalla sorgente = 2 centimetri). NOTA: rimuovere il coperchio delle strisce prima cross-linking.
- Aggiungere 25 ml 5x tampone di lavaggio (5 M NaCl, 250 mM Tris, pH 7.5) e 50 ml di ben risospesa sfere magnetiche streptavidina, omogenizzare delicatamente.
- Incubare a 4 ° C per 1 ora su una ruota girevole.
6. lavaggio Steps
NOTA: (Magnet: vedi Materiali List). Iniziare con una cattura delle sfere magnetiche nel coperchio strip PCR, con il magnete. Quindi sostituire la striscia PCR con uno nuovo contenente la soluzione di lavaggio successivo. Rimuovere il magnete e risospendere le sfere, e incubare 2 min. Spin giù e sostituire il coperchio da un coperchio fresco.
- Fasi di lavaggio (frazione solubile solo)
- Lavare 6 volte in 200 microlitri tampone di lavaggio 1x.
- Lavare una volta in 200 ml HPLC H 2 O.
- Lavare 6 volte in 200 ml 80% acetonitrile.
- Lavare 2 volte in 200 microlitri HPLC H 2 O.
- Fasi di lavaggio (membraFrazione ne solo)
- Lavare 5 volte in 200 microlitri 1x tampone di lavaggio + 0,1% DDM.
- Lavare 2 volte in 200 microlitri 1x tampone di lavaggio + 0,05% DDM.
- Lavare una volta in 200 microlitri 1x tampone di lavaggio + 0,025% DDM.
- Lavare una volta in 200 microlitri 1x tampone di lavaggio + 0,0125% DDM.
- Lavare 3 volte in 200 ml di ABC 100 mm (di bicarbonato di ammonio, NH 4 CO 3) + 2 M Urea.
7. MS Preparazione del campione
- Risospendere le perline (direttamente nel coperchio) in 20 microlitri di ABC 100 mm (ABC 100 mm + 8 M Urea per la frazione di membrana) e trasferire in provette da 1,5 ml.
- Frazione di membrana solo: incubare a 60 ° C per 5 min, agitando a 500 rpm.
- Aggiungere 0,5 microlitri TCEP 200 mm (tris (2-carbossietil) fosfina) e incubare a 60 ° C per 1 ora, agitando a 500 rpm. Raffreddare a 25 ° C.
- Aggiungere 0,5 ml di preparati al momento 400 mm iodoacetamide e incubare a 25 ° C per 30 minuti, agitando unT 500 rpm e al buio.
- Aggiungere 0,5 ml di 0,5 M N-acetil cisteina e incubare a 25 ° C per 10 min agitando a 500 rpm.
- Soltanto frazione membrana: aggiungere 1 ml Lys-C e incubare a 37 ° C, O / N.
- Aggiungere 2 mg tripsina e incubare O / N a 37 ° C, agitando a 500 rpm (involucro in Parafilm per evitare l'essiccazione).
NOTA: i campioni possono essere conservati a -20 ° C in questa fase.
NOTA: frazione di membrana solo: aggiungi ABC 100 mM per regolare la concentrazione di urea per <2 M prima aggiunta di tripsina. - Brevemente centrifugare le provette e raccogliere perle con il magnete.
- Trasferire il surnatante in un nuovo tubo di 1,5 ml (ripetere questo passaggio se perle sono lasciati).
- Aggiungere 5 microlitri 5% TFA (acido trifluoroacetico) + 1 ml 2 M HCl (15 microlitri 5% TFA + 5 microlitri 2 M HCl per la frazione di membrana).
- Colonne Condizione C18 MicroSpin (Il Gruppo Nest, MA, USA) con 150 microlitri acetonitrile (spin 20 sec a 2.400 rpm).
- EquiLibrate le colonne C18 2 volte con 150 microlitri 0,1% TFA (spin 20 sec, 2.400 rpm).
- Caricare il campione e spin 2 min, 2.000 giri.
- Ricaricare il flow-through nella colonna e ripetere la fase di centrifuga (2 min, 2.000 rpm).
- Lavare 3 volte con 150 microlitri 0,1% TFA, 5% acetonitrile (20 sec, 2.400 rpm).
- Prendere una nuova provetta e eluire due volte con 150 microlitri 0,1% TFA, 50% acetonitrile (2 min, 2.000 rpm).
- Asciugare i peptidi in una velocità-vac.
- Risospendere in 40 microlitri 98% H 2 O, 2% acetonitrile, 0,15% di acido formico.
- Sonicare 20 sec (ciclo impulso 0.5, l'ampiezza del 100%, vedi Lista dei materiali) e spin down 5 sec, 12.000 rpm (da banco centrifuga). Vortex 10 sec, e spin down 5 sec, 12.000 rpm. Trasferire in un flaconcino HPLC per l'analisi LC-MS / MS.
- Congelare a -20 ° C.
NOTA: i campioni possono essere conservati a -20 ° C in questa fase.
8. LC-MS / MS Analysis
- Eseguire un nano-LC (sistemi nano-LC) equipped con una colonna RP-HPLC (75 um x 37 cm) imballato con resina C18 (Magic C18 AQ 3 micron) usando un gradiente lineare dal 95% di solvente A (acido formico al 0,15%, 2% acetonitrile) e 5% di solvente B ( 98% acetonitrile, 0,15% di acido formico) al 35% di solvente B oltre 60 minuti ad una portata di 0,2 ml / min.
- Analizzare i peptidi con LC-MS / MS (doppia pressione LTQ-Orbitrap Velos spettrometro di massa, collegato ad una sorgente ionica electrospray).
NOTA: La modalità di acquisizione dei dati è stato impostato per ottenere una alta risoluzione MS scansione nella parte FT dello spettrometro di massa ad una risoluzione di 60.000 FWHM seguita da scansioni MS / MS nella trappola ionica lineare delle 20 ioni più intensi. Per aumentare l'efficienza dei tentativi MS / MS, la carica dello stato di screening modus è stata attivata per escludere non assegnato e oneri singolarmente ioni. Collusione dissociazione indotta è stata innescata quando il precursore superato 100 conteggi ioni. La durata esclusione dinamico è stato impostato a 30 sec. Il tempo di accumulo di ioni è stato fissato a 300 msec (MS) e 50msec (MS / MS).
9. Database Search
- Scarica il P. aeruginosa NCBI database tramite la homepage NCBI ( http://www.ncbi.nlm.nih.gov/ ).
- Convertire gli spettri MS raw in file generici mascotte (MGF) utilizzando lo strumento di conversione MassMatrix ( http://www.massmatrix.net/mm-cgi/downloads.py ).
- Cerca questo file mgf utilizzando MASCOTTE versione 2.3 contro il P. aeruginosa NCBI database contenente in avanti e le voci di proteine reverse-esca.
- Eseguire un in silico tripsina digestione dopo lisina ed arginina (meno seguito da prolina) tollerando due spaccature nella peptidi completamente triptici perse.
- Impostare i parametri di ricerca database per consentire metionine ossidato (15,99491 Da) come modifiche variabile e carboxyamidomethylation (57,021464 Da) di residui di cisteina come modifica fisso. Per mascotte ci scrutaing scansioni ad alta risoluzione, impostare la tolleranza di massa precursore di 15 ppm e impostare la tolleranza di massa frammento di 0.6 Da. Infine, impostare la proteina FDR al 1%.
- Importare le ricerche mascotte di P. Esperimenti aeruginosa CCMS in Scaffold (Proteomesoftware, versione 3), impostare i parametri per ottenere una proteina FDR vicino a 1%, ed estrarre il totale conta spettrali.
- Per i risultati rappresentativi presentati in questo documento, abbiamo usato un test T accoppiato per confrontare l'esperimento con il controllo della concorrenza, e solo considerato colpi con un valore di p inferiore a 0,1, e un rapporto di conteggio spettrale sopra 2 (sperimentare conteggi spettrali / controllo della concorrenza spettrale conta).
- I dati possono essere esportati in qualsiasi software di foglio di calcolo per ulteriori analisi.
10. La quantificazione senza etichetta
- Importare i file raw in software progenesi LC-MS (Nonlinear Dynamics, versione 4.0).
- Eseguire l'allineamento LC-MS e la rilevazione in funzione Setti predefinitongs.
- Esportare i dati in formato mgf da progenesi LC-MS.
- Cerca gli spettri MS / MS con la mascotte contro il NCBI P. banca dati contenente aeruginosa in avanti e le voci di proteine reverse-esca.
- Importare i risultati di ricerca di database in progenesi LC-MS e mappare i peptidi identificazioni alle funzioni MS1.
- Per la valutazione dei dati (calcolo dei livelli di significatività, rapporti fold-change) è stato utilizzato lo script ProteinSQAnalysis di SafeQuant (soglia: valore q inferiore a 0,1, il rapporto di conteggio spettrale sopra 2).
Risultati
Per identificare nuovi c-di-GMP effettori in P. aeruginosa abbiamo usato sistematicamente CCMS per analizzare le frazioni solubili e di membrana di P. aeruginosa ceppo PAO1 da una cultura fase di log (OD 600 = 0,5). Qui riassumiamo e discutere i risultati rappresentativi di questa pesca. Sono stati utilizzati quattro repliche biologiche indipendenti. Per ogni esperimento sono stati utilizzati due differenti concentrazioni CDG-CC (5 micron e 10 micron). Per rilevare se la specificità, esperimenti sono stati condotti in presenza o assenza di 1 mM c-di-GMP come concorrente e, infine, con un controllo tallone (cioè senza CDG-CC) (Tabella 1).
Seguendo il metodo descritto in dettaglio in precedenza (figura 2) abbiamo prodotto un elenco di proteine catturate in un formato impalcatura. Probabili agenti contaminanti sono stati rimossi. Questa comprendeva proteine ribosomali, streptavidina, tripsina, albumina sierica, la cheratina ed altre proteine umane. La proteinaIdentificazione tasso di scoperta false (FDR) è stato fissato al 1% utilizzando il software Scaffold, ei dati esportati per eccellere. Il numero adesione fornita da impalcatura può essere convertito in numeri locus usando la funzione CERCA.VERT in Excel legata a una lista di P. numeri locus aeruginosa. In questa fase, l'elenco hit comprende 768 proteine per la frazione solubile e 433 proteine per la frazione di membrana. Tuttavia, la maggior parte delle proteine non sono significativamente arricchiti nell'esperimento cattura. Così, proteine che sono probabilmente catturati in modo non specifico (positivi nel controllo cordone o solo in presenza di c-di-GMP concorrente) sono stati rimossi. Abbiamo calcolato un rapporto conteggio spettrale tra l'esperimento di acquisizione ed il controllo della concorrenza e le proteine solo considerato con un rapporto maggiore di due. Inoltre abbiamo impiegato un t-test per dati appaiati sulla conta spettrali per fornire una misura significatività tra l'esperimento cattura e la concorrenza controllo, e impostare una soglia permissiva di 0,1. Infine, Abbiamo considerato solo colpi robusti con almeno quattro peptidi individuati nei quattro esperimenti per le concentrazioni composti 2 cattura presi tutto. Questi criteri devono essere regolati in base alle esigenze e con il verificato e predetto c-di-GMP proteine leganti come standard per impostare la soglia. Dopo la cernita, la lista è stata ridotta a 76 risultati per la frazione solubile e 133 proteine per la frazione di membrana. Ciò ha incluso 13 solubile e 21 proteine di membrana da P. aeruginosa che sono noti o previsto di impegnare c-di-GMP (Tabella 2). Gli altri 63 solubile e 112 proteine di membrana sono nuove proteine putative c-di-GMP vincolanti che non contengono una delle più conosciute domini di legame c-di-GMP. Questi successi hanno ora essere convalidato testando il loro legame c-di-GMP specifica.
In una schermata precedente abbiamo pescato GlyA2 (PA2444), GlyA3 (PA4602) e Gsp69 (PA1127) 1. Questi 3 proteine sono stati clonati, overexpressed, e purificati daE. coli e potrebbe essere validato per legare c-di-GMP in esperimenti di collegamento UV-cross con 33 P etichettati c-di-GMP 15. Le K d s sono stati determinati a 1.0, 2.0 e 6.9 mM, rispettivamente, indicando che effettivamente nuovi effettori possono essere identificati utilizzando CCMS.
In aggiunta a questo esempio rappresentativo, abbiamo usato CCMS con estratti di cellule raccolte da diverse condizioni di crescita e con varie concentrazioni di c-di-GMP intracellulari (n = 24). Nel complesso abbiamo catturato il 74% (38/51) delle note o previste P. componenti aeruginosa PAO1 c-di-GMP di segnalazione (24/32 proteine solubili, 14/19 proteine di membrana). Dato che almeno nove di questi geni hanno mostrato di essere trascritto a condizioni specifiche (stress ossidativo, rilevamento del quorum, biofilm) 16 e che alcuni non possono vincolare c-di-GMP a tutti, questo grado di copertura potrebbe essere vicino alla saturazione. Questo, insieme con l'osservazione che la maggior parte di questi componenti were catturato con alta specificità (tabella 2) sostiene con forza che questa tecnica è efficace e potente.
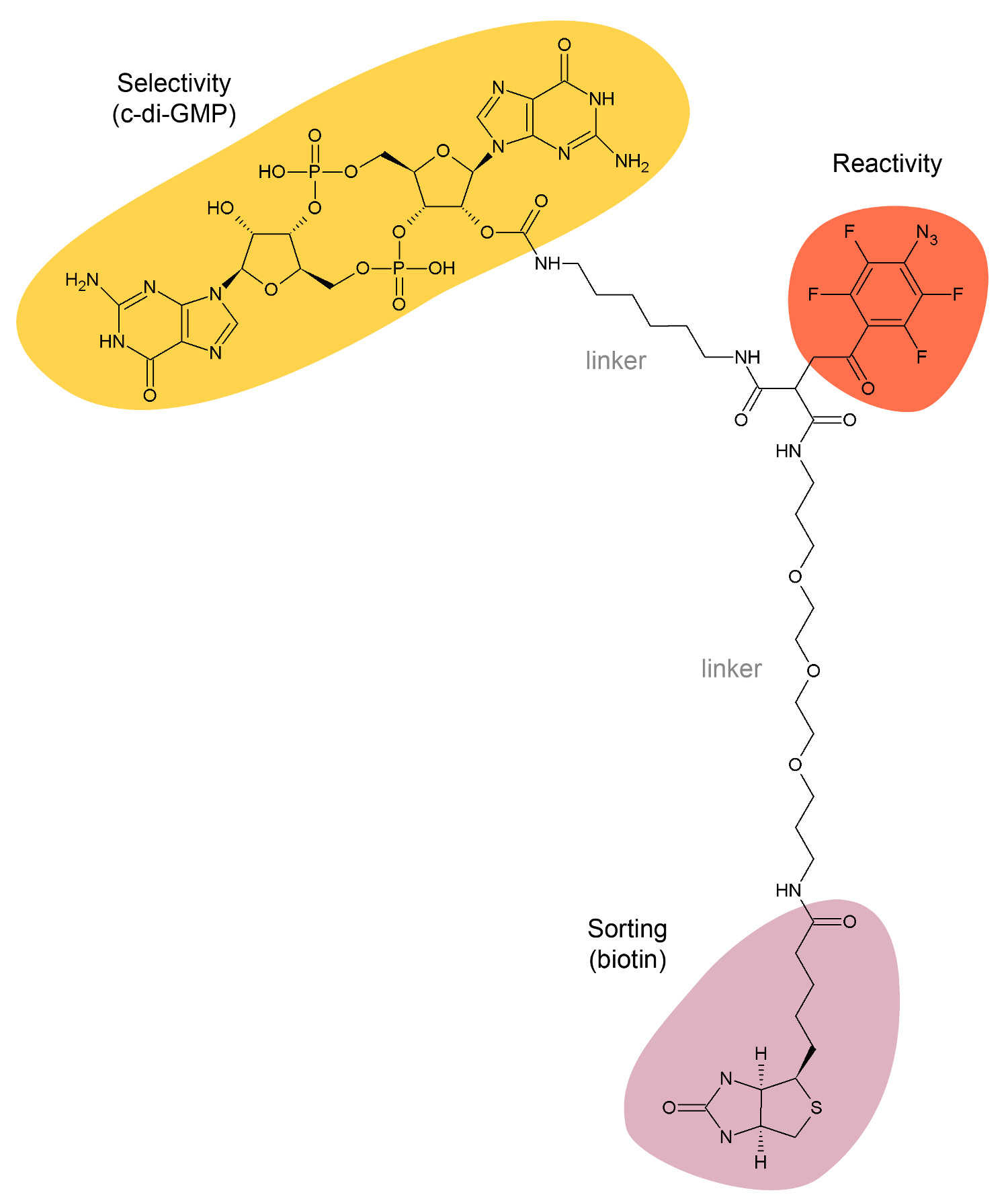
Figura 1: Struttura chimica del c-di-GMP Compound Capture.

Figura 2:. CCMS sintesi workflow Dopo lisi meccanica i nucleotidi liberi vengono rimossi con una colonna esclusione PD10. Le proteine provenienti dalle frazioni solubili o di membrana vengono incubati con il CDG-CC e la miscela è esposto a raggi UV per cross-link di proteine catturate. Passi di dura lavaggio sono svolte con composti legati alla streptavidina sfere magnetiche rivestite. On-bead digestione trittico fornisce peptidi,che vengono poi separate dalle perline e protonato per la loro identificazione spettrometria di massa. Cliccate qui per vedere una versione più grande della figura.
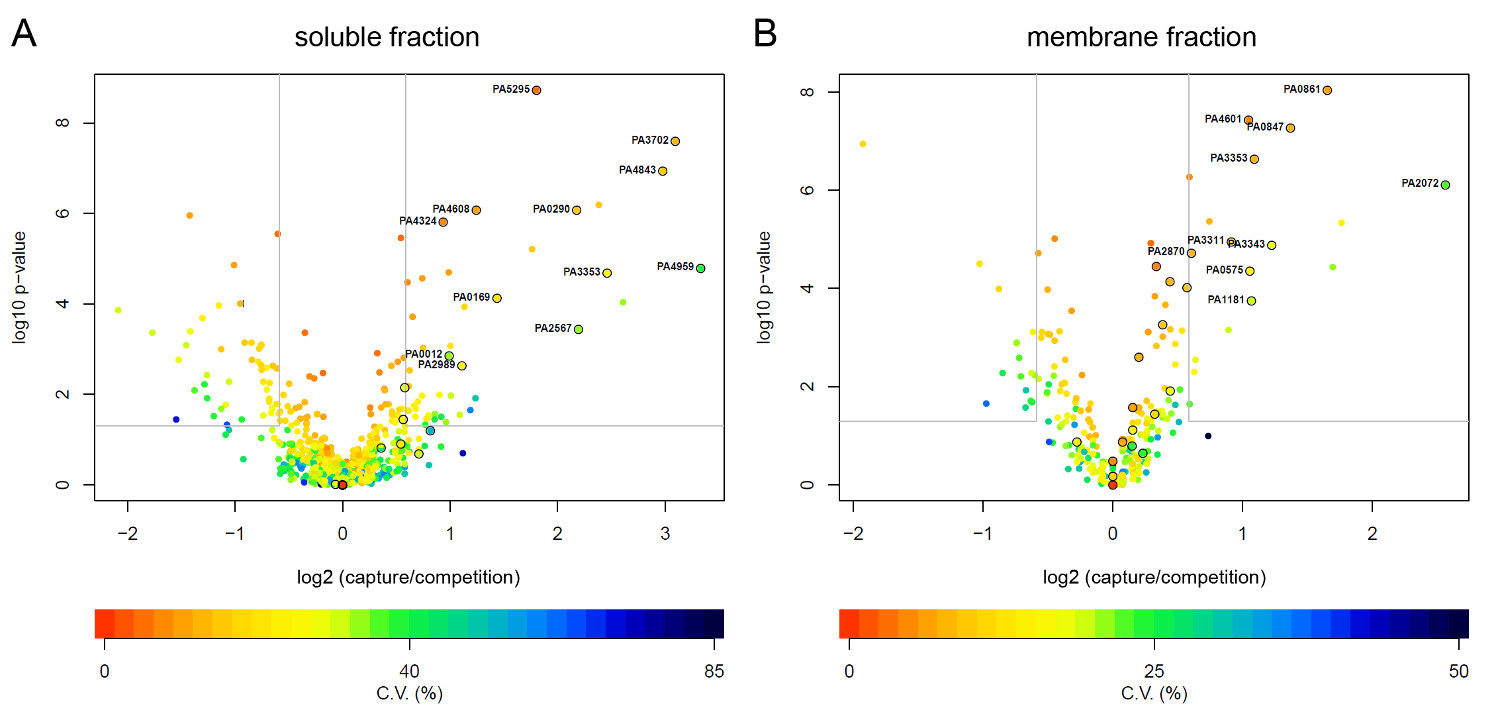
Figura 3: Volcanoplots di P. proteine aeruginosa significativamente arricchite di CCMS. Dopo l'analisi LC-MS / MS e quantificazione senza etichetta, proteine sono state classificate come descritto nel testo. Rapporto log2 intensità del peptide rilevato tra gli esperimenti di cattura e della concorrenza sono stati calcolati e tracciare la curva valori derivati dal significato analisi (t-statistica modificato, empirica metodo Bayes 17). Le proteine all'interno delle soglie di rilevanza per valori di p <0.05 e rapporti di intensità> 1,5 volte sono indicatcato in una casella grigia. Il 4 repliche per la frazione solubile (A) e la frazione di membrana (B) sono state eseguite in presenza di 10 mM c-di-GMP-CC, e l'esperimento concorso con 1 mM c-di-GMP. I punti cerchiati corrispondono a proteine leganti c-di-GMP noti. Cliccate qui per vedere una versione più grande della figura.
| Capture: | Buffer | Chimico | Fonte | Concentrazione |
| Batterica Lysis Buffer 10x | MES | Sigma | 67 mm | |
| pH 7.5 | HEPES | Sigma | 67 mm | |
| NaCl | MeRCK | 2 M | ||
| Na acetato | Merck | 67 mm | ||
| DTT | Fluka | 10 mM | ||
| DNaseI | Roche | 20 U / ml | ||
| Completa Protease Inhibitor Cocktail | Roche | 1 scheda / 10 ml | ||
| Cattura Buffer 5x | HEPES | Sigma | 100 MM | |
| Kac | Sigma | 250 mm | ||
| MgAc (anidro) | Sigma | 50 mm | ||
| Glicerina | Sigma | 50% (V / V) | ||
| PIL, GTP, ATP, CTP | sigma | 1 mm per | ||
| Lavare 5x tampone | Tris-HCl | Merck | 1 M | |
| (Per la frazione solubile) | EDTA | Sigma | 0.5 M | |
| pH 7.5 | NaCl | Sigma | 5 M | |
| n-ottil-β-D-glucopiranoside | Anagrade (Affymetrix) | 42.5 micron | ||
| Lavare 5x tampone | Tris-HCl | Merck | 1 M | |
| (Per la frazione di membrana) | EDTA | Sigma | 0.5 M | |
| pH 7.5 | NaCl | Sigma | 5 M | |
| Altri prodotti chimici: | Bicarbonato di ammonio (ABC) | Fluka | ||
| Urea | AppliChem | |||
| Preparazione del campione MS: | Buffer | Chimico | Fonte | Concentrazione |
| C18 Buffer A | TFA | Forare | 0,1% (V / V) | |
| H 2 O di grado HPLC | 99,9% (V / V) | |||
| C18 Buffer B | TFA | Forare | 0,1% (V / V) | |
| Acetonitrile | Biosolve | 49,9% (V / V) | ||
| H 2 O di grado HPLC | 50% (V / V) | |||
| C18 BufferC | TFA | Forare | 0,1% (V / V) | |
| Acetonitrile | Biosolve | 5% (V / V) | ||
| H 2 O di grado HPLC | 94,9% (V / V) | |||
| LC Buffer A | Acido formico | Sigma | 0,15% (V / V) | |
| Acetonitrile | Biosolve | 2% | ||
| H 2 O di grado HPLC | 97.85% | |||
| Altri prodotti chimici: | tris (2-carbossietil) fosfina (TCEP) | Sigma | ||
| iodoacetamide (IAA) | Sigma | |||
| N acetil-cisteina | Sigma | |||
| Endoproteinase Lys-C | Wako | |||
| Tripsina | Promega |
Tabella 1: buffers composizione Sintesi dei buffer di composizione, i prodotti chimici e fornitori.. Il-GMP-c-di cattura composto, c-di-GMP (per il controllo della concorrenza), sfere magnetiche rivestite streptavidina, buffer di cattura, e la soluzione di lavaggio sono inclusi nella caproKit.
| Controllo Bead | Esperimento Capture | Controllo della concorrenza | |
| Estratto proteico (10 mg / ml) | 30 ml | 30 ml | 30 ml |
| c-di-GMP (10 mM) | 0 microlitri | 0 microlitri | 10 microlitri |
| Nucleotidi (10 mm di ogni nucleotide) | 10 microlitri | 10 microlitri | 10 microlitri |
| 5x tampone Capture | 20 l | 20 l | 20 l |
| H 2 O | 42 microlitri | 32 microlitri | 22 microlitri |
| 30 min di incubazione | |||
| c-di-GMP ~ CC (magazzino 100 micron) | 0 microlitri | 5-10 microlitri | 5-10 microlitri |
| Concentrazioni finale: | Controllo Bead | Esperimento Capture | Controllo della concorrenza |
| c-di-GMP ~ CC (micron) | 0 micron | 5-10 micron | 5-10 micron |
| Concorrente c-di-GMP (micron) | 0 micron | 0 micron | 1.000 micron |
Tabella 2:. Miscela di reazione Capture Sintesi della miscela di reazione per il controllo tallone (cioè senza cattura composto), l'esperimento di cattura (con il c-di-GMP-CC), e la concorrenza controllo che contiene un grande eccesso di c- di-GMP. La concentrazione finale c-di-GMP-CC può essere regolata, ed è di solito impostato tra 5 e 10 micron.
| Nome Protein | Locus ID | Architettura di dominio | capture esperimento esperimento / concorrenza 1 | |||||||
| a) frazione solubile | CDG-CC = 5 micron | CDG-CC = 10 micron | ||||||||
| - | PA4843 | REC-REC-GGEEF * | 14/0 | 14/0 | 13/0 | 11/0 | 14/0 | 12/0 | 14/0 | 14/0 |
| WSPR | PA3702 | REC-GGEEF * | 9/0 | 9/0 | 10/0 | 9/0 | 11/0 | 10/0 | 11/0 | 11/0 |
| - | PA2567 | GAF-SPTRF-EAL | 8/0 | 4/0 | 9/0 | 0/0 | 7/0 | 3/0 | 8/0 | 8/0 |
| - | PA3353 | Pilz | 11/0 | 12/0 | 13/0 | 12/0 | 12/0 | 10/0 | 11/0 | 12/0 |
| - | PA0290 | PAS-GGDEF | 5/0 | 3/0 | 6/0 | 5/0 | 8/0 | 5/0 | 6/0 | 6/0 |
| - | PA5295 | GDDEF-EAL | 3/0 | 3/0 | 3/0 | 1/0 | 6/0 | 6/0 | 5/0 | 4/0 |
| Fimx | PA4959 | PAS-GDSIF-EVL | 23/1 | 21/0 | 21/0 | 11/0 | 24/3 | 23/2 | 22/0 | 20/0 |
| - | PA4608 | Pilz | 3/0 | 3/0 | 3/0 | 0/0 | 3/0 | 2/0 | 3/0 | 3/0 |
| - | PA0012 | Pilz | 3/0 | 2/0 | 2/0 | 2/0 | 2/0 | 2/0 | 4/0 | 2/0 |
| - | PA2989 | Pilz | 1/0 | 1/0 | 2/0 | 1/0 | 1/0 | 2/0 | 3/0 | 3/0 |
| - | PA4324 | Pilz | 2/0 | 2/0 | 1/0 | 1/0 | 2/0 | 2/0 | 1/0 | 2/0 |
| - | PA3177 | GGEEF | 2/0 | 1/0 | 3/0 | 0/0 | 1/0 | 1/0 | 3/0 | 1/0 |
| - | PA4396 | REC-DEQHF | 0/0 | 1/0 | 4/0 | 0/0 | 1/0 | 0/0 | 5/0 | 1/0 |
| - | PA0169 | GGEEF * | 3/0 | 2/0 | 6/0 | 7/0 | 7/1 | 6/2 | 9/1 | 7/1 |
| - | PA2799 | Pilz | 1/0 | 0/0 | 2/0 | 0/0 | 0/0 | 0/0 | 3/0 | 1/0 |
| - | PA5017 | PAS-GAF-PAS-ASNEF-EAL | 1/0 | 2/0 | 1/0 | 0/0 | 1/0 | 3/2 | 0/0 | 0/0 |
| - | PA5487 | GGEEF * | 0/0 | 0/0 | 0/0 | 1/0 | 1/0 | 0/0 | 1/0 | 1/0 |
| b) frazione di membrana | CDG-CC = 5 micron | CDG-CC = 10 micron | ||||||||
| - | PA2072 | CHASE4-TM-PAS-GGDEF-EAL | 13/1 | 25/0 | 27/0 | 19/0 | 36/0 | 36/0 | 31/0 | 23/0 |
| - | PA0861 | TM-PAS-GGDEF-ELL | 6/1 | 14/0 | 13/0 | 10/0 | 17/0 | 18/0 | 13/0 | 8/0 |
| - | PA3353 | Pilz | 6/0 | 10/0 | 9/0 | 7/0 | 10/0 | 10/0 | 6/0 | 5/0 |
| - | PA3343 | 5TM-GGDEF | 3/0 | 7/0 | 7/0 | 4/0 | 12/0 | 10/0 | 7/0 | 7/0 |
| - | PA1181 | MASE1-PAS-PAS-PAS-PAS-GGDEF-ELL | 3/0 | 6/0 | 9/0 | 3/0 | 12/0 | 12/0 | 5/0 | 2/0 |
| - | PA0847 | TM-CHASE4-HAMP-PAS-GGDEF | 0/0 | 4/0 | 4/0 | 1/0 | 15/0 | 13/0 | 8/0 | 6/0 |
| - | PA0575 | PBPb-TM-PAS-PAS-PAS-PAS-GGDEF-EAL | 1/0 | 7/0 | 6/0 | 3/0 | 10/0 | 10/0 | 6/0 | 2/0 |
| yfiN | PA1120 | 2TM-HAMP-GGDEF | 2/0 | 4/0 | 3/0 | 3/0 | 5/0 | 4/0 | 3/0 | 1/0 |
| - | PA0290 | PAS-GGDEF | 1/0 | 4/0 | 3/0 | 2/0 | 1/0 | 5/0 | 1/0 | 2/0 |
| - | PA4929 | 7TMR: DISMED2-7TMR: DISMED2-GGDEF | 2/0 | 4/0 | 2/0 | 2/0 | 2/0 | 2/0 | 2/0 | 1/0 |
| MORA | PA4601 | TM-TM-PAS-PAS-PAS-PAS-GGDEF-EAL | 3/0 | 7/0 | 7/3 | 5/0 | 9/0 | 10/0 | 5/0 | 4/0 |
| - | PA1851 | 5TM-GGDEF | 1/0 | 2/0 | 1/0 | 2/0 | 4/0 | 3/0 | 1/0 | 1/0 |
| - | PA2870 | TM-GGDEF | 0/0 | 0/0 | 1/0 | 0/0 | 4/0 | 4/0 | 4/0 | 2/0 |
| - | PA3311 | TM-MHYT-MHYT-MHYT-AGDEF-EAL | 1/1 | 5/0 | 7/1 | 4/0 | 8/0 | 8/0 | 5/1 | 3/0 |
| BIFA | PA4367 | TM-GGDQF-EAL | 1/0 | 2/0 | 1/0 | 2/0 | 1/0 | 2/0 | 2/1 | 1/0 |
| - | PA4608 | Pilz | 0/0 | 1/0 | 1/0 | 0/0 | 3/0 | 3/0 | 2/0 | 2/0 |
| - | PA4332 | 5TM-GGEEF | 1/0 | 3/0 | 2/0 | 1/0 | 1/0 | 1/0 | 2/0 | 0/0 |
| - | PA0012 | Pilz | 1/0 | 1/0 | 1/0 | 1/0 | 1/0 | 1/0 | 1/0 | 0/0 |
| - | PA2989 | Pilz | 4/0 | 8/0 | 7/0 | 7/0 | 5/2 | 11/2 | 7/2 | 8/3 |
| - | PA1433 | HAMP-RGGEF-KVL | 0/0 | 0/0 | 0/0 | 0/0 | 1/0 | 1/0 | 1/0 | 1/0 |
| - | PA4843 | REC-REC-GGEEF | 0/0 | 0/0 | 1/1 | 1/0 | 0/0 | 1/0 | 1/0 | 0/0 |
| * = GGDEF dominio contenente un sito I | ||||||||||
| 1 numero di conteggi spettrali di peptidi identificati | ||||||||||
Tabella 3: P. aeruginosa nota c-di-GMP segnalazione componenti appositamente catturati. proteine identificate sono stati allineati come descritto nel testo. Le proteine sono identificati con il loro nome e il luogo il numero, e ci indicano la loro architettura previsto con lo strumento online di database NCBI Conserve Domain (n = 4, CDG-CC = 5 micron o 10 micron) per mostrare la specificità cattura e la riproducibilità del metodo.
Discussione
Particolare attenzione dovrebbe essere presa in diverse fasi del protocollo. La concentrazione proteica è un parametro critico con una concentrazione di 10 mg / ml di essere difficile da raggiungere quando le cellule vengono coltivate in condizioni di crescita specifici (ad esempio biofilm o piccole varianti colonia). Così, la risospensione pellet deve essere eseguita in un basso volume di tampone di lisi. Le concentrazioni di proteine possono essere diminuiti di 8 mg / ml. Rispetto al metodo pubblicato da Nesper et al. 1, abbiamo aggiunto diversi nucleotidi alla reazione di cattura per ridurre al minimo non specifico cattura di proteine leganti nucleotidi. Sebbene l'aggiunta di nucleotidi migliorata specificità, può allo stesso tempo impedire la cattura di proteine che si legano diversi nucleotidi nello stesso sito. Ad esempio il FleQ effettore, che è stato recentemente dimostrato di legare ATP e c-di-GMP 18 è stato pescato particolare in assenza di ATP nella nostra precedente esperimento 1, ma non più nel data qui presentato in presenza di un eccesso di ATP.
CDG-CC deve essere attentamente protetto dalla luce. Anche se la luce ambiente contiene solo una piccola frazione di UV, si raccomanda di mantenere lo stock compound cattura avvolto in un foglio di alluminio, come pure la miscela cattura prima dell'attivazione mediante irraggiamento UV. Le fasi di lavaggio che seguono possono essere molto rigorosi per aumentare la specificità, come le proteine catturate sono covalentemente legati al CDG-CC. Per quanto riguarda l'analisi LC-MS / MS, gli esperimenti devono essere effettuate in un ambiente privo di cheratina pulito. Inoltre, tamponi compatibili HPLC devono essere utilizzati, specialmente dopo le fasi di lavaggio. La lista dei candidati comprende tipicamente tra 300 e 800 proteine (per un quadruplice), con bassi variazioni tra le repliche (vedi Tabella 2 come esempio).
Alcuni parametri, come la proteina e la concentrazione CDG-CC potrebbe aver bisogno di essere ottimizzate a seconda organismi analyzed. Dal basso proteine abbondanti o proteine espresse solo in determinate condizioni possono essere facilmente perdere, occorre prestare attenzione per quanto riguarda le condizioni di coltura utilizzate. Questo problema può essere superato confrontando la lista dei risultati con ATLAS proteine globali raccolti per le stesse condizioni di coltura. Infine, l'ottimizzazione del detersivo può essere difficile, in quanto deve essere ottimizzato per quanto riguarda la sua capacità di solubilizzare le proteine di membrana e deve anche essere compatibile con MS.
Occorre tenere presente che la molecola c-di-GMP della CC viene modificata chimicamente, come è collegata tramite il gruppo 2'OH di uno ribosio al resto del ponteggio. Questa modifica potrebbe alterare la sua capacità di legarsi ad alcuni effettori fornendo in tal modo i falsi-negativi. In questo contesto è da notare che non abbiamo mai catturato proteine che ospitano un dominio EAL, ma mancano di un dominio GGDEF in P. aeruginosa, anche se le proteine EAL sono stati catturati in altre specie, come Caulcrescentus obacter. Questo potrebbe essere dovuto a una scarsa accesso o una bassa affinità del CDG-CC al sito di legame, o la degradazione del CDG-CC da proteine EAL. Al contrario, molte proteine nucleotide binding stati catturati con una relativamente bassa specificità e, in larga misura, sono probabilmente falsi positivi. Ulteriore convalida del legame c-di-GMP utilizzando tecniche come DRaCALA 20 specifica, UV cross-linking 15, a scansione differenziale fluorimetria (DSF) 21, termoforesi microscala (MST) 22, isotermico calorimetria (ITC) 23 - 24 ... è pertanto necessario.
È anche possibile che una frazione della porzione c-di-GMP del composto cattura viene degradata dalla fosfodiesterasi dal lisato cellulare. Questo è uno dei motivi per cui la procedura deve essere eseguita a 4 ° C, quindi limitando l'attività fosfodiesterasi prima reticolazione.
Il Operazire può essere adattato a molte specie batteriche, ed è stato utilizzato con successo per 3 diverse specie batteriche con molto lievi modifiche 1. La tecnologia basata Compound Capture può ridurre i falsi positivi utilizzando accurato lavaggio (ad esempio 1 M di sale, ad alta concentrazione di detersivo, 2 M di urea nel caso di proteine di membrana, l'80% acetonitrile), rispetto ad altre tecniche che non si basano sul legame covalente. Dato che la convalida dei candidati può essere un processo noioso e che richiede tempo, questo è un grande vantaggio per i metodi alternativi, come proteomica chimici a base approcci 14.
Questo metodo di video illustrato stabilisce CCMS come uno strumento potente e versatile per identificare e caratterizzare nuovi componenti coinvolti in piccola molecola di segnalazione. In futuro, composti Capture simili ospitano altri gruppi di selettività potrebbero essere utilizzati per catturare le proteine coinvolte nella piccola molecola di segnalazione, come ad esempio i nuovi c-di-AMP effettori.
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
We thank Alberto Reinders for his work in optimizing the CCMS conditions for P. aeruginosa. We also thank Pablo Manfredifor the annotation of the P. aeruginosa proteins. This work was supported by the Swiss National Science Foundation (SNF) Sinergia grant CRSII3_127433.
Materiali
| Name | Company | Catalog Number | Comments |
| caproBox | caprotec bioanalytics | 1-5010-001 (220 V) | UV lamps coupled to a cooling 96-plate cooling block, for the photoactivation |
| caproMag | caprotec bioanalytics | included in the CCMS Starter Kit | For easy handling of magnetic particles without pipetting |
| c-di-GMP caproKit | caprotec bioanalytics | upon request | The kit contains the c-di-GMP-capture compound, c-di-GMP (for the competition control), streptavidin coated magnetic beads, capture buffer, and washing buffer |
| Disposable PD-10 Desalting Columns | GE Healthcare | 17-0851-01 | |
| 12-tube PCR strips | Thermo Scientific | AB-1114 | |
| UIS250v sonicator with VialTweeter | Hielscher ultrasound technology | UIS250v and VialTweeter | |
| Miniature French Pressure Cell | Thermo Electron Corporation | FA-003 |
Riferimenti
- Nesper, J., Reinders, A., Glatter, T., Schmidt, A., Jenal, U. A novel capture compound for the identification and analysis of cyclic di-GMP binding proteins. J Proteomics. 75, 4874-4878 (2012).
- Hengge, R. Principles of c-di-GMP signalling in bacteria. Nature reviews. Microbiology. 7, 263-273 (2009).
- Sondermann, H., Shikuma, N. J., Yildiz, F. H. You've come a long way: c-di-GMP signaling. Curr. Opin. Microbiol. 15, 140-146 (2012).
- Schirmer, T., Jenal, U. Structural and mechanistic determinants of c-di-GMP signalling. Nature reviews. Microbiology. 7, 724-735 (2009).
- Furukawa, S., Kuchma, S., O'Toole, G. Keeping their options open: acute versus persistent infections. J. Bacteriol. 188, 1211-1217 (2006).
- Christen, M., Christen, B., Folcher, M., Schauerte, A., Jenal, U. Identification and characterization of a cyclic di-GMP-specific phosphodiesterase and its allosteric control by GTP. J. Biol. Chem. 280, 30829-30837 (2005).
- Ryan, R. P., Fouhy, Y., Lucey, J. F., Dow, J. M. Cyclic di-GMP signaling in bacteria: recent advances and new puzzles. J. Bacteriol. 188, 8327-8334 (2006).
- Habazettl, J., Allan, M. G., Jenal, U., Grzesiek, S. Solution structure of the PilZ domain protein PA4608 complex with cyclic di-GMP identifies charge clustering as molecular readout. J. Biol. Chem. 286, 14304-14314 (2011).
- Fazli, M., et al. The CRP/FNR family protein Bcam1349 is a c-di-GMP effector that regulates biofilm formation in the respiratory pathogen Burkholderia cenocepacia. Molecular Microbiology. 82, 327-341 (2011).
- Hickman, J. W., Harwood, C. S. Identification of FleQ from Pseudomonas aeruginosa as a c-di-GMP-responsive transcription factor. Molecular Microbiology. 69, 376-389 (2008).
- Sudarsan, N., et al. Riboswitches in eubacteria sense the second messenger cyclic di-GMP. Science. 321, 411-413 (2008).
- Lenz, T., et al. Profiling of methyltransferases and other S-adenosyl-L-homocysteine-binding Proteins by Capture Compound Mass Spectrometry (CCMS). J Vis Exp. , (2010).
- Köster, H., et al. Capture compound mass spectrometry: a technology for the investigation of small molecule protein interactions. Assay Drug Dev Technol. 5, 381-390 (2007).
- Düvel, J., et al. A chemical proteomics approach to identify c-di-GMP binding proteins in Pseudomonas aeruginosa. Journal of Microbiological Methods. 88, 229-236 (2012).
- Christen, B., et al. Allosteric control of cyclic di-GMP signaling. J. Biol. Chem. 281, 32015-32024 (2006).
- Balasubramanian, D., Mathee, K. Comparative transcriptome analyses of Pseudomonas aeruginosa. Human genomics. 3, 349-361 (2009).
- Smyth, G. K. Linear models and empirical bayes methods for assessing differential expression in microarray experiments. Stat Appl Genet Mol Biol. 3 (3), (2004).
- Baraquet, C., Harwood, C. S. Cyclic diguanosine monophosphate represses bacterial flagella synthesis by interacting with the Walker A motif of the enhancer-binding protein FleQ. Proc. Natl. Acad. Sci. U.S.A. 110, 18478-18483 (2013).
- Nesper, J., Reinders, A., Glatter, T., Schmidt, A., Jenal, U. A novel capture compound for the identification and analysis of cyclic di-GMP binding proteins. J Proteomics. 75, 4874-4878 (2012).
- Roelofs, K. G., Wang, J., Sintim, H. O., Lee, V. T. Differential radial capillary action of ligand assay for high-throughput detection of protein-metabolite interactions. Proc. Natl. Acad. Sci. U.S.A. 108, 15528-15533 (2011).
- DeSantis, K., Reed, A., Rahhal, R., Reinking, J. Use of differential scanning fluorimetry as a high-throughput assay to identify nuclear receptor ligands. Nuclear receptor signaling. 10, e002(2012).
- Seidel, S. A., et al. Microscale thermophoresis quantifies biomolecular interactions under previously challenging conditions. Methods. 59, 301-315 (2013).
- Merighi, M., Lee, V. T., Hyodo, M., Hayakawa, Y., Lory, S. The second messenger bis-(3‘-5’)-cyclic-GMP and its PilZ domain-containing receptor Alg44 are required for alginate biosynthesis in Pseudomonas aeruginosa. Molecular Microbiology. 65, 876-895 (2007).
- Qi, Y., et al. Binding of cyclic diguanylate in the non-catalytic EAL domain of FimX induces a long-range conformational change. J. Biol. Chem. 286, 2910-2917 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon