Method Article
Capture Compound Mass Spectrometry - Ein leistungsfähiges Tool zum Identifizieren Novel c-di-GMP Effektorproteinen
In diesem Artikel
Zusammenfassung
The ubiquitous second messenger c-di-GMP controls growth and behavior of many bacteria. We have developed a novel Capture Compound Mass Spectrometry based technology to biochemically identify and characterize c-di-GMP binding proteins in virtually any bacterial species.
Zusammenfassung
Beachtliche Fortschritte wurden im letzten Jahrzehnt auf die Identifizierung und Charakterisierung von Enzymen bei der Synthese (diguanylate Cyclasen) und Abbau (Phosphodiesterasen) beteiligt der second messenger c-di-GMP hergestellt. Im Gegensatz dazu ist wenig Information über die molekularen Mechanismen und Zellkomponenten durch die dieses Signalmoleküls reguliert eine Vielfalt von zellulären Prozessen. Die meisten der bekannten Effektor-Proteine gehören zur Familie PilZ oder degeneriert diguanylate Cyclasen oder Phosphodiesterasen, die sich auf die Katalyse gegeben haben und Effektor-Funktion übernommen. So, um besser die zelluläre c-di-GMP-Netzwerk in einem weiten Bereich von Bakterien experimentelle Methodik zur Identifizierung und Validierung von neuartigen Effektoren für die zuverlässige in silico Vorhersage nicht definieren.
Wir haben vor kurzem eine neuartige Erfassung Verbindung Massenspektrometrie (CCMS) basierte Technologie als ein mächtiges Werkzeug, umbiochemisch zu identifizieren und zu charakterisieren, c-di-GMP-bindende Proteine. Diese Technik wurde bereits berichtet anwendbar auf einen weiten Bereich von Organismen 1 sein. Hier geben wir eine ausführliche Beschreibung des Protokolls, die wir verwenden, um solche Signalkomponenten zu untersuchen. Als ein Beispiel verwenden wir P. aeruginosa, ein opportunistisches Pathogen in dem c-di-GMP spielt eine kritische Rolle bei der Virulenz und Biofilmkontrolle. CCMS identifizierten 74% (38/51) der bekannten oder vorhergesagten Komponenten des c-di-GMP-Netzwerk. Diese Studie erklärt die CCMS-Verfahren im Detail und stellt es als ein leistungsfähiges und vielseitiges Werkzeug, um neue Komponenten in kleinen Molekülen Signalisierung beteiligt sind.
Einleitung
c-di-GMP ist ein wichtiger sekundärer Botenstoff von den meisten Bakterien verwendet, um verschiedene Aspekte des Wachstums und des Verhaltens zu steuern. Zum Beispiel regelt c-di-GMP Zellzyklusprogression, Motilität und die Expression von Exopolysacchariden und Oberflächen Adhäsine 2-4. Durch die Koordination solcher Prozesse c-di-GMP fördert Biofilmbildung, ein Verfahren, das mit einer chronischen Infektion einer Reihe von pathogenen Bakterien 5 zugeordnet ist. c-di-GMP wird durch Enzyme genannt diguanylate Cyclasen (DGCs), die eine katalytische Domäne GGDEF 4 Hafen synthetisiert. Einige DGCs besitzen eine hemmende Website, die nach unten reguliert die Cyclase-Aktivität bei der c-di-GMP verbindlich. Der Abbau von c-di-GMP wird durch zwei unterschiedliche Klassen von Phosphodiesterasen (PDEs) beherbergen entweder eine katalytische EAL oder HD-GYP Domain 6,7 katalysiert.
Die Mehrzahl der bekannten Effektorproteine, die direkt binden c-di-GMP gehören zu einer von drei Klassen von Proteinen: katalytischeVerbündeter inaktiv GGDEF oder EAL Domains und PilZ Domains, kleine molekulare Schalter, die Konformationsänderungen bei der c-di-GMP verbindlich 8 unterzogen werden. DGCs, PDEs und PilZ Proteine sind gut gekennzeichnet und ihre Domains können in silico relativ sicher vorhergesagt werden. Ein besonderes Interesse gilt nun auf der Identifizierung von neuen Klassen von c-di-GMP Effektoren konzentriert. Einige c-di-GMP Effektoren mit unterschiedlichen Bindungsmotiven wurden kürzlich beschrieben, wie die CRP / FNR-Protein-Familie Bcam1349 in Burkholderia cenocepacia oder der Transkriptionsregulator FleQ in P. aeruginosa 9,10. Zusätzlich wurden c-di-GMP-spezifische RNA-Schalter kürzlich identifiziert und gezeigt, um die Genexpression in einer c-di-GMP-abhängige Weise 11 steuern. Die c-di-GMP-Bindungsmotive von verschiedenen Effektoren nur schlecht konserviert machen bioinformatischen Vorhersagen solcher Proteine schwierig. Um dieses Problem anzugehen, ein biochemisches Verfahren, das auf der Verwendung eines c-di-GMP spe basiert entwickelten wirsche Erfassung Verbindung mit der Massenspektrometrie 1,12,13 kombiniert.
Wir haben vor kurzem entwickelt, eine neuartige dreiwertigen c-di-GMP-Capture Verbindung (CDG-CC, Abbildung 1) 1. Diese chemischen Gerüst besteht aus: 1) als Köder, um c-di-GMP erfassen bindenden Proteinen verwendet eine c-di-GMP-Einheit, 2) eine UV-photoaktivierbare reaktive Gruppe verwendet werden, um vernetzen CDG-CC zu den gebundenen Proteine und 3) einem Biotin, um die aufgenommenen Proteine unter Verwendung von Streptavidin-beschichteten magnetischen Kügelchen zu isolieren. Die CDG-CC kann verwendet werden, um direkt und spezifisch einzufangen c-di-GMP-Effektoren aus komplexen Mischung von Makromolekülen, wie Zell-Lysaten. Capture-Verbindung auf der Basis chemischer und Proteomik Ansätze wurde bereits berichtet Anwendbarkeit auf einen breiten Bereich von Organismen, wie zB Caulobacter crescentus, Salmonella enterica Serovar typhimurium und S. aeruginosa 1,14.
In diesem methodischen Papier,bieten wir eine eingehende Beschreibung des CCMS Verfahren unter Verwendung von Extrakten von P. aeruginosa als ein Beispiel. Diese Studie stellt CCMS als eine leistungsfähige und vielseitiges Werkzeug, um biochemisch identifiziert neue Komponenten in kleinen Molekülen Signalgebung beteiligt.
Protokoll
1. Lysatherstellung
- Wachsen P. aeruginosa-Zellen in LB an die gewünschte OD.
HINWEIS: Für Hinweise: Verwenden ≈ 100 ml Kultur / Probe für stationäre Phase Kulturen und ≈ 500 ml cultur / Probe für log-Phase Kulturen (OD 600nm = 0,5). - Pellet durch Zentrifugation für 20 min bei 5.000 x g.
- Resuspendieren 0,5-1 g Pellet in 1 ml Lyse-Puffer (6,7 mM MES, 6,7 mM HEPES, 200 mM NaCl, 6,7 mM KAc, 1 mM DDT, pH 7,5) und füge einen Proteaseinhibitor (vollständige Mini, EDTA-frei) sowie als DNaseI.
- Lyse der Zellen durch 3 Passagen durch einen Französisch Druckzelle bei 20.000 psi (siehe Materialliste).
- Ultrazentrifuge das Zelllysat bei 100.000 x g für 1 h bei 4 ° C.
- Speichern Sie den Überstand (gehen Sie zu Schritt 2).
- Durch Auf- und Abpipettieren Waschen des Pellets mit 1 ml 1x Lysepuffer.
- Ultrazentrifuge bei 100,000 x g für 1 h bei 4 ° C.
- Flash-einfrieren des Pellets inflüssigem Stickstoff und bei -20 ° C bis zur Erfassung von Membranproteinen (siehe Schritt 3).
2. Entfernung von Freier c-di-GMP und andere Nukleotide (lösliche Fraktion Only)
- Mit 10 ml kaltem Lysepuffer Waschen eine PD10-Entsalzungssäule (siehe Materialliste).
- Gießen Sie den Überstand (≈ 1 ml) auf die PD10, um Nukleotide zu entfernen.
- Eluiert mit 4 ml kaltem Lysepuffer (500 ul Stufen).
- Wählen Sie die am stärksten konzentrierten Fraktionen wie von Bradford bestimmt und sie gemeinsam.
3. Pellet Resuspension und Solubilisierung (Membranfraktion Only)
- Das Pellet in 500 bis 1000 ul 1x Sammlungspuffer (ohne Reinigungsmittel) (siehe Tabelle 1).
HINWEIS: Das Pellet ist schwierig, wieder zu suspendieren. Es ist mit der ersten Pipette auf und ab mit einer Pipette wird empfohlen, etwa das Pellet, dann, eine Spritze 27 G zu verwenden, um auch die Lösung zu homogenisieren. - 1% (w / v) n-Dodecyl-β-D-maltopyranosid (DDM).
- Inkubieren bei 4 ° C für mindestens 2 h (oder O / N) auf einem rotierenden Rad.
- Ultrazentrifuge bei 100,000 x g für 1 h bei 4 ° C.
- Ernten Sie den Überstand.
4. Proteinkonzentrationsmessung
- Messung der Proteinkonzentration durch Bradford-Assays (BCA-Assay für Membranfraktion).
- Stellen Sie die Gesamtproteinkonzentration auf 10 mg / ml.
5. Erfassen
- Mischungs 300 ug Protein und mit 1 mM GDP, GTP, ATP, CTP und 20 ul 5x Aufnahmepuffer (100 mM HEPES, 250 mM KAc, 50 mM MgAc, 5% Glycerin, pH 7,5) (Tabelle 1). Stellen Sie das Reaktionsvolumen auf 100 ul mit H 2 O.
HINWEIS: das Gesamtvolumen umfasst das Volumen des CDG-CC und c-di-GMP bei der Wettbewerbskontrolle (siehe Schritt 5.3 und Tabelle 2).
HINWEIS: Alle Experimente wurden in 200 & geführt# 181; l 12-Rohr-PCR-Streifen (Thermo Scientific).
HINWEIS: für die Membranfraktion, stellen Sie sicher, immer die DDM-Konzentration zu halten über der kritischen Mizellenkonzentration (0,01% w / v), bis das Protein Solubilisierungsschritt in 8 M Harnstoff (Schritt 7.1). - Inkubieren bei 4 ° C für 30 min auf einem sich drehenden Rad.
- In 10 uM CDG-CC (Endkonzentration).
HINWEIS: eine Steuer ohne CDG-CC (als "Perle Steuerung" bezeichnet), und eine Steuer mit 1 mM c-di-GMP ("Wettbewerbskontrolle") ergänzt (siehe Tabelle 2).
Hinweis: Die CDG-CC-Konzentration von 1 bis 10 & mgr; M eingestellt werden. - Inkubieren bei 4 ° C für mindestens 2 h (O / N für die Membranfraktion) auf einem rotierenden Rad, in der Dunkelheit.
- Nach einer kurzen Spin, vernetzen durch Aktivierung des reaktiven Rest des CDG-CC mit UV-Licht für 4 Minuten mit einem CaproBox (siehe Materialliste, λ = 310 nm, Bestrahlungsstärke ≥10 mW / cm², Entfernung von der Quelle = 2 cm). HINWEIS: Entfernen Sie den Deckel der Streifen vor der Vernetzung.
- Man gebe 25 & mgr; l 5x-Waschpuffer (5 M NaCl, 250 mM Tris, pH 7,5) und 50 ul gut resuspendierten Streptavidin-Magnetkügelchen, sanft zu homogenisieren.
- Inkubieren bei 4 ° C für 1 h auf einem rotierenden Rad.
6. Waschschritte
HINWEIS: (Magnet: siehe Materialliste). Beginnen Sie mit einer Erfassung der magnetischen Kügelchen in der PCR-Streifen Deckel, mit dem Magneten. Dann ersetzen Sie die PCR Streifen durch eine neue mit dem nächsten Waschlösung. Entfernen Sie den Magneten und resuspendieren die Perlen, und Inkubation 2 min. Spin down, und ersetzen Sie den Deckel durch eine frische Deckel.
- Waschschritte (lösliche Fraktion nur)
- 6-mal in 200 ul 1x Waschpuffer waschen.
- Einmal in 200 ul HPLC-Qualität H 2 O Waschen
- 6-mal in 200 ul 80% Acetonitril waschen.
- Waschen Sie 2-mal in 200 ul HPLC-Qualität H 2 O
- Waschschritte (membrane Bruch nur)
- 5 mal in 200 ul 1x Waschpuffer + 0.1% DDM waschen.
- In 200 ul 1x Waschpuffer + 0,05% DDM waschen 2 mal.
- Einmal in 200 ul 1x Waschpuffer + 0,025% DDM waschen.
- Einmal in 200 ul 1x Waschpuffer + 0,0125% DDM waschen.
- 3 Mal Waschen in 200 ul 100 mM ABC (Ammoniumbicarbonat, NH 4 CO 3) + 2 M Harnstoff.
7. MS Probenvorbereitung
- Resuspendieren der Beads (direkt im Deckel) in 20 ul 100 mM ABC (100 mM ABC + 8 M Harnstoff für die Membranfraktion) und Überführung in 1,5-ml-Röhrchen.
- Nur Membranfraktion: Inkubation bei 60 ° C für 5 min, bei 500 Upm Schütteln.
- In 0,5 ul 200 mM TCEP (Tris (2-Carboxyethyl) phosphin) und Inkubation bei 60 ° C für 1 h, bei 500 Upm schüttelnd. Abkühlen auf 25 ° C.
- In 0,5 ul frisch hergestelltes 400 mM Iodacetamid und Inkubation bei 25 ° C für 30 min, Schütteln einT 500 Umdrehungen pro Minute und in der Dunkelheit.
- Mit 0,5 ul 0,5 M N-Acetyl-Cystein und Inkubieren bei 25 ° C für 10 min, bei 500 Upm Schütteln.
- Nur Membranfraktion: fügen 1 ul Lys-C und bei 37 ° C, O / N.
- In 2 ug Trypsin und Inkubation O / N bei 37 ° C, bei 500 Upm (wrap in Parafilm Austrocknen zu verhindern) Schütteln.
HINWEIS: Die Proben können bei -20 ° C zu diesem Zeitpunkt gespeichert.
HINWEIS: Membranfraktion nur: mit 100 mM ABC, um die Harnstoffkonzentration auf <2 M vor der Zugabe von Trypsin anpassen. - Kurz Spin-Down der Rohre und sammeln Kügelchen mit dem Magneten.
- Den Überstand in ein neues 1,5-ml-Röhrchen (wiederholen Sie diesen Schritt, wenn Perlen gelassen werden).
- Mit 5 & mgr; l 5% TFA (Trifluoressigsäure) + 1 & mgr; l 2 M HCl (15 & mgr; l 5% TFA + 5 & mgr; l 2 M HCl für die Membranfraktion).
- Bedingung C18 Microspin-Säulen (The Nest Group, MA, USA) mit 150 & mgr; l Acetonitril (spin 20 sec bei 2400 rpm).
- Equilibrate der C18-Säulen 2 mal mit 150 ul 0,1% TFA (Spin 20 Sekunden, 2400 rpm).
- Laden Sie die Probe und Spin 2 min, 2000 Upm.
- Neuladen des Durchfluss auf die Säule und wiederholen den Spinnschritt (2 min, 2.000 rpm).
- 3x waschen mit 150 ul 0,1% TFA, 5% Acetonitril (20 sec, 2.400 rpm).
- Nehmen Sie eine neue Röhre und eluieren zweimal mit 150 ul 0,1% TFA, 50% Acetonitril (2 min, 2.000 rpm).
- Man trocknet die Peptide in einer Speed-vac.
- Resuspendieren in 40 & mgr; l 98% H 2 O, 2% Acetonitril, 0,15% Ameisensäure.
- Ultraschallbehandlung 20 s (Impulszyklus 0,5, Amplitude 100%, siehe Materialliste) und Spin-down 5 s, 12.000 min (Tischzentrifuge). Vortex 10 Sekunden, und Spin-down 5 s, 12.000 min. Übertragen Sie in einem HPLC-Ampulle für LC-MS / MS-Analyse.
- Bei -20 ° C.
HINWEIS: Die Proben können bei -20 ° C zu diesem Zeitpunkt gespeichert.
8. LC-MS / MS-Analyse
- Führen Sie einen Nano-LC (nano-LC-Systeme) equipped mit einer RP-HPLC-Säule (75 um x 37 cm) mit C18 Harz gepackt (Magic C18 AQ 3 & mgr; m) unter Verwendung eines linearen Gradienten von 95% Lösungsmittel A (0,15% Ameisensäure, 2% Acetonitril) und 5% Lösungsmittel B ( 98% Acetonitril, 0,15% Ameisensäure) bis 35% Lösungsmittel B über 60 min bei einer Flussrate von 0,2 ul / min.
- Analyse der Peptide mittels LC-MS / MS (Zweidruck LTQ-Orbitrap Velos Massenspektrometer, mit einem Elektrospray-Ionenquelle verbunden ist).
HINWEIS: Die Datenaufnahme-Modus wurde eingestellt, um eine hochauflösende MS-Scan in der FT Teil des Massenspektrometers mit einer Auflösung von 60.000 FWHM gefolgt von MS / MS-Scans in der linearen Ionenfalle der 20 intensivsten Ionen zu erhalten. Um die Effizienz der MS / MS-Versuche zu erhöhen, wurde der Ladezustand Screening Modus aktiviert werden, damit nicht zugeordnete auszuschließen und einzeln berechnet Ionen. Collusion induzierte Dissoziation wurde ausgelöst, als der Vorläufer über 100 Ionen zählt. Die dynamische Ausschluss Dauer wurde auf 30 Sekunden eingestellt. Der Ionenakkumulationszeit auf 300 ms (MS) und 50 eingestelltms (MS / MS).
9. Datenbanksuche
- Laden Sie die P. aeruginosa NCBI-Datenbank über die NCBI-Homepage ( http://www.ncbi.nlm.nih.gov/ ).
- Konvertieren Sie die MS Rohspektren in Maskottchen Generika-Dateien (mp) mit dem MassMatrix Konvertierungstool ( http://www.massmatrix.net/mm-cgi/downloads.py ).
- Suche auf der mp-Datei mit MASCOT Version 2.3 gegen die P. aeruginosa NCBI-Datenbank, die vorwärts und rückwärts-Köder-Protein-Einträge.
- Führen Sie ein in silico Trypsinverdau nach Lysin und Arginin (es sei denn, durch Prolin gefolgt) tolerieren zwei verpasst Spaltungen in voll tryptischen Peptide.
- Stellen Sie die Datenbanksuchparameter, um oxidierte Methionine (15,99491 Da) als variable Änderungen und Carboxyamidomethylierung (57,021464 Da) von Cysteinresten als feste Modifikation kann. Für MASCOT sucht unsing hochauflösende Scans, stellen Sie den Vorläufer Massentoleranz bis 15 ppm und stellen Sie die Fragmentmassentoleranz von 0,6 Da. Schließlich, stellen Sie den Protein FDR auf 1%.
- Importieren Sie die Maskottchen-Durchsuchungen von P. aeruginosa CCMS Experimente in Scaffold (Proteomesoftware, Version 3), stellen Sie die Parameter, um ein Protein FDR knapp 1% zu erhalten, und extrahieren Sie die gesamte spektrale zählt.
- Für in diesem Papier die repräsentativen Ergebnisse präsentiert, haben wir eine gepaarte T-Test, den Versuch mit der Wettbewerbskontrolle zu vergleichen und nur dann als Treffer mit einem p-Wert von weniger als 0,1 und einer spektralen Zahlverhältnis über 2 (experimentieren spektrale counts / Wettbewerbskontrolle Spektralbereich Aktivität).
- Die Daten können in jedem Tabellenkalkulations-Software zur weiteren Analyse exportiert werden.
10. Markierungsfreie Quantifizierung
- Importieren Sie die Raw-Dateien in Progenesis LC-MS-Software (Nichtlineare Dynamik, Version 4.0).
- Führen LC-MS Ausrichtung und Feature-Erkennung in Verzug settings.
- Exportieren Sie die Daten in mgf Format von Progenesis LC-MS.
- Durchsuchen Sie die MS / MS-Spektren mit Hilfe des MASCOT gegen die NCBI P. aeruginosa Datenbank, die vorwärts und rückwärts-Köder-Protein-Einträge.
- Importieren Sie die Datenbanksuchergebnissen in Progenesis LC-MS und Karte der Peptide Identifikationen zu MS1 Funktionen.
- Für die Datenauswertung (Berechnung der Signifikanzniveaus, klappbar Änderungsverhältnisse) die ProteinSQAnalysis Skript SafeQuant verwendet (Schwelle: q-Wert unter 0,1, spektrale Zahlverhältnis über 2).
Ergebnisse
Um neue c-di-GMP-Effektoren in P. identifizieren aeruginosa wir systematisch genutzt CCMS die löslichen und Membranfraktionen P. analysieren aeruginosa Stamm PAO1 aus einer log-Phase Kultur (OD 600 = 0,5). Hier werden wir zusammenfassen und diskutieren Vertreter Ergebnisse dieser Angelausflug. Vier unabhängige biologischen Replikate wurden verwendet. Für jeden Versuch zwei verschiedenen CDG-CC-Konzentrationen wurden verwendet (5 uM und 10 uM). Um die Spezifität zu untersuchen, wurden Experimente in der Gegenwart oder Abwesenheit von 1 mM c-di-GMP als Kompetitor und schließlich durchgeführt wird, mit einem Wulst-Steuerung (dh ohne CDG-CC) (Tabelle 1).
Wenn im Anschluss an die oben im Detail (Bild 2) beschriebenen Methode wir eine Liste der aufgenommenen Proteine produziert in einem Scaffold-Format. Wahrscheinlich Verunreinigungen entfernt wurden. Diese enthalten ribosomale Proteine, Streptavidin, Trypsin, Serumalbumin, Keratin und andere menschliche Proteine. Das ProteinIdentifizierung False Discovery Rate (FDR) wurde unter Verwendung der Software-Gerüst auf 1% gesetzt, und die Daten exportiert werden, um zu übertreffen. Die Zugangsnummer von Gerüst versehen können in Ortsnummern mit Hilfe der Funktion SVERWEIS in Excel umgewandelt werden verbunden, um eine Liste der P. aeruginosa Ort Zahlen. In diesem Stadium weist der Trefferliste 768 Proteine für die lösliche Fraktion und 433 Proteine, die für die Membranfraktion. Allerdings sind die meisten Proteine nicht wesentlich in der Capture-Experiments angereichert. So können Proteine, die wahrscheinlich nicht-spezifisch erfasst werden (positive im Wulst Steuerung oder nur in Anwesenheit von c-di-GMP Konkurrenten) wurden entfernt. Wir berechnen eine spektrale Zahlverhältnis zwischen der Capture-Experiment und die Wettbewerbskontrolle und nur als Proteine mit einem Verhältnis größer als zwei. Darüber hinaus beschäftigten wir gepaarter t-Test auf spektrale zählt, eine Bedeutung Maßnahme zwischen der Capture-Experiment und die Wettbewerbskontrolle bieten, und setzen Sie eine permissive Schwelle von 0,1. SchließlichBetrachteten wir nur robust Hits mit mindestens vier in den vier Versuchen für die 2 -Abscheidung Verbindungskonzentrationen zusammen genommen identifizierten Peptide. Diese Kriterien sind entsprechend den Bedürfnissen und unter Verwendung der verifizierte eingestellt werden und vorhergesagte c-di-GMP-bindende Proteine als Standard, um die Schwelle zu bestimmen. Nach dem Sortieren, wurde die Liste auf 76 Treffern zu der löslichen Fraktion und 133 Proteine der Membranfraktion abgenommen. Dazu gehörten 13 löslich und 21 Membranproteine aus P. aeruginosa, die bekannt sind oder voraussichtlich c-di-GMP (Tabelle 2) zu binden. Die anderen 63 löslich und 112 Membranproteine sind neu putative c-di-GMP-bindende Proteine, die eines der bekannten c-di-GMP-bindende Domänen enthalten. Diese Ergebnisse müssen nun durch Testen ihrer spezifischen Bindung an c-di-GMP validieren.
In einem vorherigen Bildschirm gefischt wir GlyA2 (PA2444), GlyA3 (PA4602) und Gsp69 (PA1127) 1. Diese 3-Proteine wurden kloniert, überexprimiert und gereinigt ausE. coli und konnte validiert, um c-di-GMP in UV-Vernetzung Experimente mit 33 P binden markierten c-di-GMP 15. Die K d s wurden zu 1,0, 2,0 und 6,9 & mgr; M bestimmt, was darauf hinweist, dass in der Tat neue Effektoren können mit CCMS identifiziert werden.
Zusätzlich zu diesem repräsentativen Beispiel CCMS verwendeten wir mit Extrakten aus Zellen, die von unterschiedlichen Wachstumsbedingungen geerntet und mit verschiedenen intrazellulärem c-di-GMP-Konzentrationen (n = 24). Insgesamt sind wir gefangen genommen 74% (38/51) der bekannte oder vorhergesagte P. aeruginosa PAO1 c-di-GMP-Signalkomponenten (24/32 lösliche Proteine, 14/19 Membranproteine). Sofern mindestens neun Genen wurde gezeigt, dass unter bestimmten Bedingungen (oxidativer Stress, Quorum Sensing Biofilme) 16 transkribiert werden, und dass einige nicht binden c-di-GMP überhaupt, kann diese Überdeckungsgrad nahe der Sättigung sein. Dies zusammen mit der Beobachtung, dass die meisten dieser Komponenten were mit hoher Spezifität (Tabelle 2) eingefangen stark argumentiert, dass diese Technik effektiv und leistungsstark ist.
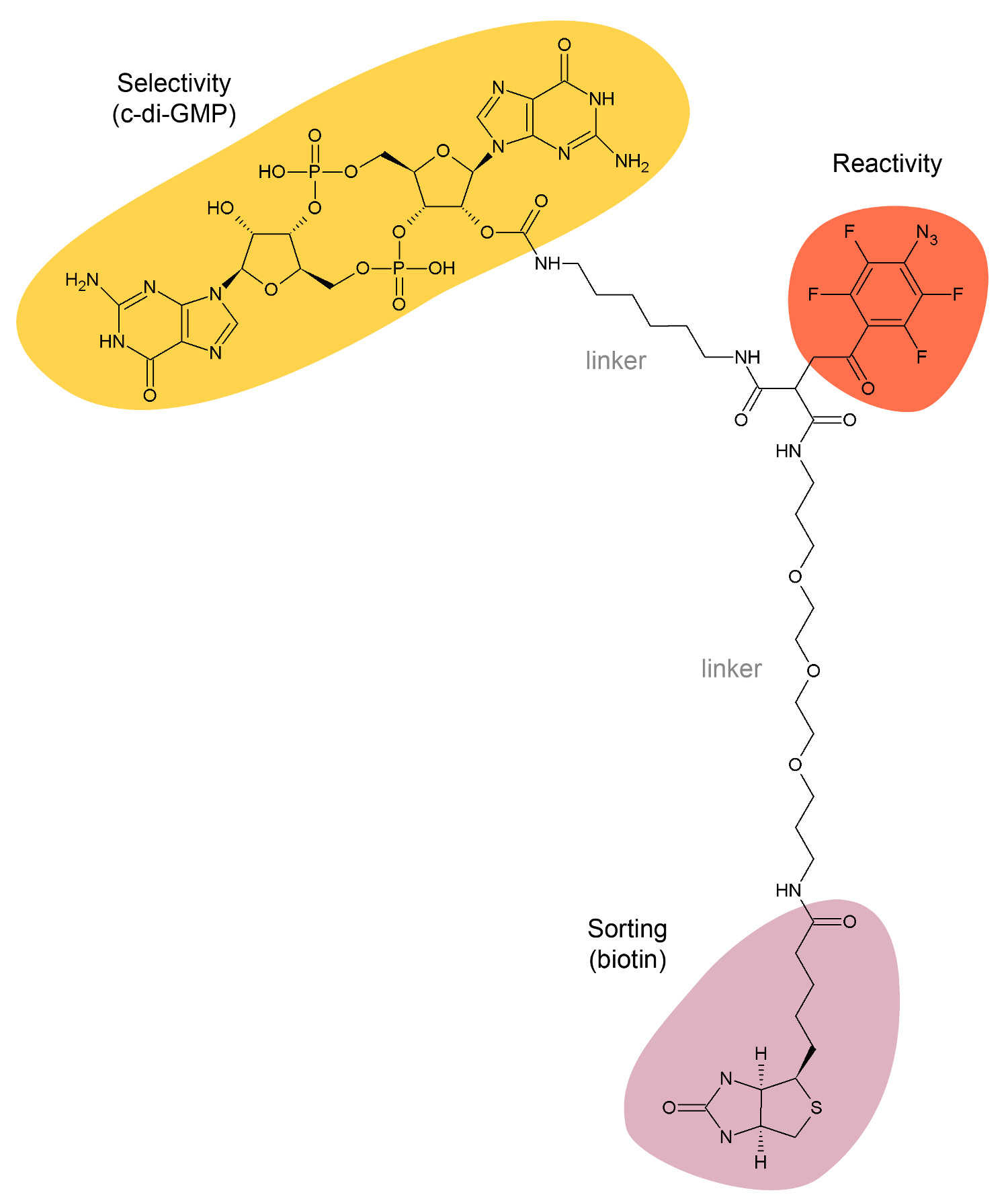
Abbildung 1: Chemische Struktur der c-di-GMP-Capture Compound.

Abb. 2: CCMS Workflow-Übersicht Nach der mechanischen Lyse die freien Nukleotide mit einer PD10 Ausschlusssäule entfernt. Proteine von den löslichen oder Membran-Fraktionen werden mit der CDG-CC inkubiert und die Mischung wird einer UV-Bestrahlung zu vernetzen aufgenommenen Proteine ausgesetzt. Schritte harten Waschen mit Verbindungen zu Streptavidin-beschichteten magnetischen Kügelchen gebunden durchgeführt. On bead Trypsinverdauung stellt Peptide,die dann von den Perlen abgetrennt werden und protonierten für ihre Massenspektrometrie Identifizierung. Bitte klicken Sie hier, um eine größere Version der Abbildung zu sehen.
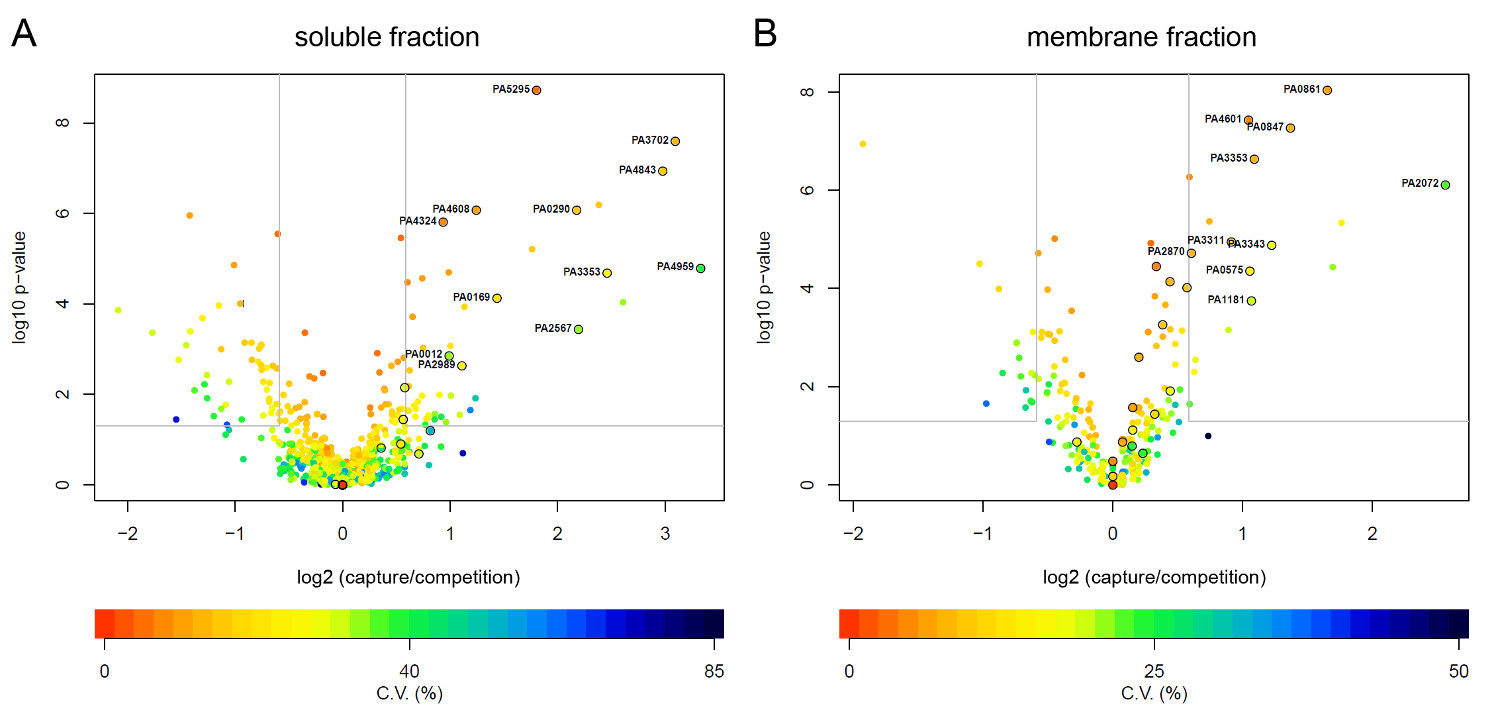
Abbildung 3: Volcanoplots P. aeruginosa Proteine signifikant durch CCMS bereichert. Folgende LC-MS / MS-Analyse und markierungsfreie Quantifizierung wurden Proteine, wie im Text beschrieben, sortiert. Log2-Intensitätsverhältnis ermittelt Peptid zwischen den Erfassung und Konkurrenzexperimente wurden berechnet und aufgetragen über Werte von Bedeutung Analyse (modifizierte t-Statistik, empirische Bayes-Methode 17) abgeleitet. Proteine innerhalb der Bedeutung Schwellenwerte für p-Werte <0,05 und Intensitätsverhältnisse> 1,5-fach sind indicatin einem grauen Kasten ed. Das 4 Wiederholungen für die lösliche Fraktion (A) und der Membranfraktion (B) wurden in Gegenwart von 10 uM c-di-GMP-CC durchgeführt wird und der Konkurrenzexperiment mit 1 mM c-di-GMP. Die eingekreisten Punkte entsprechen bekannten c-di-GMP-bindenden Proteinen. Bitte klicken Sie hier, um eine größere Version der Abbildung zu sehen.
| Erfassung: | Puffer | Chemikalie | Quelle | Konzentration |
| Bakterielle Lysis Buffer 10x | MES | Sigma | 67 mM | |
| pH 7,5 | HEPES | Sigma | 67 mM | |
| NaCl | Mirrck | 2 M | ||
| Na-Acetat | Merck | 67 mM | ||
| DTT | Fluka | 10 mM | ||
| DNaseI | Roche | 20 U / ml | ||
| Komplette Proteaseinhibitor-Cocktail | Roche | 1 Registerkarte / 10 ml | ||
| Sammlungspuffer 5x | HEPES | Sigma | 100 mM | |
| KAc | Sigma | 250 mM | ||
| MgAc (wasserfrei) | Sigma | 50 mM | ||
| Glycerol | Sigma | 50% (V / V) | ||
| BIP, GTP, ATP, CTP | Sigma- | Jeweils 1 mM | ||
| Waschpuffer 5x | Tris-HCl | Merck | 1 M | |
| (Für die lösliche Fraktion) | EDTA | Sigma | 0,5 M | |
| pH 7,5 | NaCl | Sigma | 5 M | |
| n-Octyl-β-D-glucopyranosid | Anagrade (Affymetrix) | 42,5 & mgr; | ||
| Waschpuffer 5x | Tris-HCl | Merck | 1 M | |
| (Für die Membranfraktion) | EDTA | Sigma | 0,5 M | |
| pH 7,5 | NaCl | Sigma | 5 M | |
| Andere Chemikalien: | Ammoniumbicarbonat (ABC) | Fluka | ||
| Harnstoff | Applichem | |||
| MS Probenvorbereitung: | Puffer | Chemikalie | Quelle | Konzentration |
| C18 Puffer A | TFA | Stechen | 0,1% (V / V) | |
| H 2 O für die HPLC | 99,9% (V / V) | |||
| C18 Puffer B | TFA | Stechen | 0,1% (V / V) | |
| Acetonitril | Biosolve | 49,9% (V / V) | ||
| H 2 O für die HPLC | 50% (V / V) | |||
| C18 BufferC | TFA | Stechen | 0,1% (V / V) | |
| Acetonitril | Biosolve | 5% (V / V) | ||
| H 2 O für die HPLC | 94,9% (V / V) | |||
| LC Puffer A | Ameisensäure | Sigma | 0,15% (V / V) | |
| Acetonitril | Biosolve | 2% | ||
| H 2 O für die HPLC | 97,85% | |||
| Andere Chemikalien: | Tris (2-carboxyethyl) phosphin (TCEP) | Sigma | ||
| Iodacetamid (IAA) | Sigma | |||
| N-Acetyl-Cystein | Sigma | |||
| Endoproteinase Lys-C | Wako | |||
| Trypsin | Promega |
Tabelle 1: Puffer Zusammensetzung Zusammenfassung der Puffer Zusammensetzung, Chemikalien und Lieferanten.. Die c-di-GMP-Capture-Verbindung, c-di-GMP (für die Wettbewerbskontrolle), Streptavidin beschichteten magnetischen Kügelchen, Sammlungspuffer und Waschpuffer sind in der caproKit enthalten.
| Bead Steuer | Einfangexperiment | Wettbewerb Kontrolle | |
| Proteinextrakt (10 mg / ml) | 30 ul | 30 ul | 30 ul |
| c-di-GMP (10 mM) | 0 & mgr; | 0 & mgr; | 10 & mgr; |
| Nukleotide (10 mM jedes Nukleotids) | 10 & mgr; | 10 & mgr; | 10 & mgr; |
| Sammlungspuffer 5x | 20 & mgr; | 20 & mgr; | 20 & mgr; |
| H 2 O | 42 & mgr; | 32 & mgr; | 22 & mgr; |
| Inkubation für 30 min | |||
| c-di-GMP ~ CC (Lager 100 uM) | 0 & mgr; | 5-10 & mgr; | 5-10 & mgr; |
| Endkonzentrationen: | Bead Steuer | Einfangexperiment | Wettbewerb Kontrolle |
| c-di-GMP ~ CC (uM) | 0 & mgr; | 5-10 & mgr; M | 5-10 & mgr; M |
| Wettbewerber c-di-GMP (uM) | 0 & mgr; | 0 & mgr; | 1,000 & mgr; M |
Tabelle 2:. Capture-Reaktionsmix Zusammenfassung der Reaktionsmischung für den Wulst Steuerung (dh ohne Fangverbindung), der Erfassungsversuch (mit c-di-GMP-CC), und der Konkurrenzsteuerung, die einen großen Überschuss an c- enthält di-GMP. Die c-di-GMP-CC Endkonzentration eingestellt werden kann, und wird typischerweise zwischen 5 und 10 um eingestellt.
| Protein Namen | Locus ID | Domain-Architektur | Capture-Experiment / Konkurrenzexperiment 1 | |||||||
| a) lösliche Fraktion | CDG-CC = 5 & mgr; | CDG-CC = 10 & mgr; | ||||||||
| - | PA4843 | REC-REC-GGEEF * | 14/0 | 14/0 | 13/0 | 11/0 | 14/0 | 12/0 | 14/0 | 14/0 |
| WSPR | PA3702 | REC-GGEEF * | 9/0 | 9/0 | 10/0 | 9/0 | 11/0 | 10/0 | 11/0 | 11/0 |
| - | PA2567 | GAF-SPTRF-EAL | 8/0 | 4/0 | 9/0 | 0/0 | 7/0 | 3/0 | 8/0 | 8/0 |
| - | PA3353 | PilZ | 11/0 | 12/0 | 13/0 | 12/0 | 12/0 | 10/0 | 11/0 | 12/0 |
| - | PA0290 | PAS-GGDEF | 5/0 | 3/0 | 6/0 | 5/0 | 8/0 | 5/0 | 6/0 | 6/0 |
| - | PA5295 | GDDEF-EAL | 3/0 | 3/0 | 3/0 | 1/0 | 6/0 | 6/0 | 5/0 | 4/0 |
| FIMX | PA4959 | PAS-GDSIF-EVL | 23/1 | 21/0 | 21/0 | 11/0 | 24/3 | 23/2 | 22/0 | 20/0 |
| - | PA4608 | PilZ | 3/0 | 3/0 | 3/0 | 0/0 | 3/0 | 2/0 | 3/0 | 3/0 |
| - | PA0012 | PilZ | 3/0 | 2/0 | 2/0 | 2/0 | 2/0 | 2/0 | 4/0 | 2/0 |
| - | PA2989 | PilZ | 1/0 | 1/0 | 2/0 | 1/0 | 1/0 | 2/0 | 3/0 | 3/0 |
| - | PA4324 | PilZ | 2/0 | 2/0 | 1/0 | 1/0 | 2/0 | 2/0 | 1/0 | 2/0 |
| - | PA3177 | GGEEF | 2/0 | 1/0 | 3/0 | 0/0 | 1/0 | 1/0 | 3/0 | 1/0 |
| - | PA4396 | REC-DEQHF | 0/0 | 1/0 | 4/0 | 0/0 | 1/0 | 0/0 | 5/0 | 1/0 |
| - | PA0169 | GGEEF * | 3/0 | 2/0 | 6/0 | 7/0 | 7/1 | 6/2 | 9/1 | 7/1 |
| - | PA2799 | PilZ | 1/0 | 0/0 | 2/0 | 0/0 | 0/0 | 0/0 | 3/0 | 1/0 |
| - | PA5017 | PAS-GAF-PAS-ASNEF-EAL | 1/0 | 2/0 | 1/0 | 0/0 | 1/0 | 3/2 | 0/0 | 0/0 |
| - | PA5487 | GGEEF * | 0/0 | 0/0 | 0/0 | 1/0 | 1/0 | 0/0 | 1/0 | 1/0 |
| b) Membranfraktion | CDG-CC = 5 & mgr; | CDG-CC = 10 & mgr; | ||||||||
| - | PA2072 | CHASE4-TM-PAS-GGDEF-EAL | 13/1 | 25/0 | 27/0 | 19/0 | 36/0 | 36/0 | 31/0 | 23/0 |
| - | PA0861 | TM-PAS-GGDEF-ELL | 6/1 | 14/0 | 13/0 | 10/0 | 17/0 | 18/0 | 13/0 | 8/0 |
| - | PA3353 | PilZ | 6/0 | 10/0 | 9/0 | 7/0 | 10/0 | 10/0 | 6/0 | 5/0 |
| - | PA3343 | 5TM-GGDEF | 3/0 | 7/0 | 7/0 | 4/0 | 12/0 | 10/0 | 7/0 | 7/0 |
| - | PA1181 | MASE1-PAS-PAS-PAS-PAS-GGDEF-ELL | 3/0 | 6/0 | 9/0 | 3/0 | 12/0 | 12/0 | 5/0 | 2/0 |
| - | PA0847 | TM-CHASE4-HAMP-PAS-GGDEF | 0/0 | 4/0 | 4/0 | 1/0 | 15/0 | 13/0 | 8/0 | 6/0 |
| - | PA0575 | PBPB-TM-PAS-PAS-PAS-PAS-GGDEF-EAL | 1/0 | 7/0 | 6/0 | 3/0 | 10/0 | 10/0 | 6/0 | 2/0 |
| yfiN | PA1120 | 2TM-HAMP-GGDEF | 2/0 | 4/0 | 3/0 | 3/0 | 5/0 | 4/0 | 3/0 | 1/0 |
| - | PA0290 | PAS-GGDEF | 1/0 | 4/0 | 3/0 | 2/0 | 1/0 | 5/0 | 1/0 | 2/0 |
| - | PA4929 | 7TMR: DISMED2-7TMR: DISMED2-GGDEF | 2/0 | 4/0 | 2/0 | 2/0 | 2/0 | 2/0 | 2/0 | 1/0 |
| Mora | PA4601 | TM-TM-PAS-PAS-PAS-PAS-GGDEF-EAL | 3/0 | 7/0 | 7/3 | 5/0 | 9/0 | 10/0 | 5/0 | 4/0 |
| - | PA1851 | 5TM-GGDEF | 1/0 | 2/0 | 1/0 | 2/0 | 4/0 | 3/0 | 1/0 | 1/0 |
| - | PA2870 | TM-GGDEF | 0/0 | 0/0 | 1/0 | 0/0 | 4/0 | 4/0 | 4/0 | 2/0 |
| - | PA3311 | TM-MHYT-MHYT-MHYT-AGDEF-EAL | 1/1 | 5/0 | 7/1 | 4/0 | 8/0 | 8/0 | 5/1 | 3/0 |
| BIfA | PA4367 | TM-GGDQF-EAL | 1/0 | 2/0 | 1/0 | 2/0 | 1/0 | 2/0 | 2/1 | 1/0 |
| - | PA4608 | PilZ | 0/0 | 1/0 | 1/0 | 0/0 | 3/0 | 3/0 | 2/0 | 2/0 |
| - | PA4332 | 5TM-GGEEF | 1/0 | 3/0 | 2/0 | 1/0 | 1/0 | 1/0 | 2/0 | 0/0 |
| - | PA0012 | PilZ | 1/0 | 1/0 | 1/0 | 1/0 | 1/0 | 1/0 | 1/0 | 0/0 |
| - | PA2989 | PilZ | 4/0 | 8/0 | 7/0 | 7/0 | 5/2 | 2.11 | 7/2 | 8/3 |
| - | PA1433 | HAMP-RGGEF-KVL | 0/0 | 0/0 | 0/0 | 0/0 | 1/0 | 1/0 | 1/0 | 1/0 |
| - | PA4843 | REC-REC-GGEEF | 0/0 | 0/0 | 1/1 | 1/0 | 0/0 | 1/0 | 1/0 | 0/0 |
| * = GGDEF Domäne, die ein I-Stelle | ||||||||||
| 1 Anzahl von spektralen Grafen von identifizierten Peptide | ||||||||||
Tabelle 3: P. aeruginosa bekannt c-di-GMP-Signalkomponenten spezifisch eingefangen. Identifizierte Proteine wurden zunächst sortiert, wie im Text beschrieben. Die Proteine werden mit ihrem Namen und Ortsnummer identifiziert, und wir zeigen ihre Architektur vorhergesagt mit der NCBI konservierte Domäne Datenbank Online-Tool (n = 4, CDG-CC = 5 uM oder 10 uM), um das Capture-Spezifität und die Reproduzierbarkeit zeigen des Verfahrens.
Diskussion
Besondere Sorgfalt sollte bei verschiedenen Stufen des Protokolls gefaßt. Die Proteinkonzentration ist ein kritischer Parameter bei einer Konzentration von 10 mg / ml, schwierig zu erreichen, wenn die Zellen unter bestimmten Wachstumsbedingungen (zB Biofilmen oder kleine Kolonie Varianten) gezüchtet. Somit sollte das Pellet resuspendiert in einem niedrigen Volumen Lysepuffer geführt werden. Proteinkonzentrationen können bis zu 8 mg / ml reduziert werden. Im Vergleich zu der durch Nespers et al. Veröffentlichten Methode 1, gaben wir verschiedene Nukleotide zur Einfangreaktion unspezifische Erfassung von Nukleotid-Bindungsproteinen zu minimieren. Obwohl die Zugabe von Nukleotiden, die Spezifität verbessert, kann sie gleichzeitig verhindern, dass die Erfassung der Proteine, die unterschiedliche Nukleotide binden an der gleichen Stelle. Zum Beispiel der Effektor FleQ, die kürzlich gezeigt wurde, dass ATP binden und c-di-GMP 18 wurde speziell in Abwesenheit von ATP in unseren früheren Versuch 1 nicht mehr in der d gefangen, aber nichtata hier vorgestellt, in Gegenwart eines Überschusses von ATP.
Das CDG-CC sorgfältig vor Licht geschützt werden. Auch wenn das Umgebungslicht nur einen kleinen Bruchteil der UV enthält, wird empfohlen, um die Erfassung Verbindung Lager in Aluminiumfolie eingewickelt, sowie die Erfassung Mischung vor der Aktivierung durch UV-Bestrahlung zu halten. Die Waschschritte, die folgen kann sehr streng sein, um die Spezifität zu erhöhen, wie die aufgenommenen Proteine kovalent an die CDG-CC gebunden. Bezüglich der LC-MS / MS-Analyse sollten die Versuche in einem sauberen Keratin freien Umgebung durchgeführt werden. Außerdem sollte HPLC kompatiblen Puffern verwendet werden, insbesondere nach den Waschschritten. Die Kandidatenliste enthält typischerweise zwischen 300 und 800 Proteine (für eine Vierfach), mit geringen Schwankungen zwischen den Replikaten (siehe Tabelle 2 als Beispiel).
Einige Parameter wie dem Protein und dem CDG-CC-Konzentration müssen möglicherweise abhängig von den Organismen eine zu optimierendennalyzed. Seit niedrigen reichlich Proteine oder nur unter bestimmten Bedingungen zum Ausdruck kann leicht übersehen werden, ist darauf zu den verwendeten Kulturbedingungen genommen werden. Dieses Problem lässt sich durch einen Vergleich der Trefferliste mit einer weltweiten Protein ATLAS für die gleichen Kulturbedingungen gesammelt überwunden werden. Schließlich kann die Optimierung der Waschmittel Herausforderung sein, wie es braucht, um in Bezug auf seine Fähigkeit, Membranproteine löslich zu optimieren und auch sein muss MS kompatibel.
Man muss beachten, dass die c-di-GMP-Molekül der CC chemisch modifiziert ist, wie es über die 2'-OH-Gruppe einer Ribose mit dem Rest des Gerüsts verbunden sind. Diese Veränderung könnte die Fähigkeit, einige Effektoren, wodurch falsch negativen binden ändern. In diesem Zusammenhang ist es bemerkenswert, dass wir nie eingefangen Proteine, die eine EAL Domain bergen aber es fehlt eine GGDEF Domain in P. aeruginosa, obwohl EAL Proteine in anderen Spezies eingefangen, wie Caulobacter crescentus. Dies könnte zu einem schlechten Zugang oder eine geringe Affinität des CDG-CC an die Bindungsstelle oder zu einer Verschlechterung des CDG-CC von EAL-Proteine sein. Im Gegensatz dazu wurden viele Nukleotid-bindenden Proteinen mit einem relativ geringen Spezifität fangen und zu einem großen Ausmaß, sind wahrscheinlich Fehlalarme. Weitere Validierung der spezifischen Bindung an c-di-GMP unter Verwendung von Techniken wie DRaCALA 20, UV-Vernetzungs 15. Differentialscanningkalorimetrie Fluorimetrie (DSF) 21 Micro Thermophorese (MST) 22, isotherme Kalorimetrie (ITC) 23 - 24 ... daher notwendig.
Es ist auch möglich, dass ein Teil des c-di-GMP-Einheit des Capture-Verbindung durch Phosphodiesterasen vom Zelllysat abgebaut. Dies ist einer der Gründe, warum das Verfahren muss bei 4 ° C durchgeführt werden, damit die Phosphodiesterasen Aktivität vor der Vernetzung einschränkend.
Die proceduWieder kann zu vielen Bakterienarten angepasst werden, und wurde erfolgreich für 3 verschiedene Bakterienarten mit sehr geringfügigen Änderungen 1 verwendet. Capture Compound basierte Technologie können Falsch-Positive mithilfe gründliches Waschen zu reduzieren (beispielsweise 1 M Salzgehalt, hoher Waschmittelkonzentration, 2 M Harnstoff bei Membranproteinen, 80% Acetonitril), verglichen mit anderen Techniken, die nicht auf kovalente Bindung angewiesen sind. Da die Validierung der Kandidaten kann eine mühsame und zeitraubender Vorgang sein kann, ist dies ein großer Vorteil gegenüber alternativen Methoden wie chemische Proteomik Ansätze 14.
Dies veranschaulicht Video Verfahren etabliert CCMS als leistungsfähiges und vielseitiges Werkzeug zur Identifizierung und Charakterisierung neuer Komponenten in kleinen Molekülen Signalgebung beteiligt. In Zukunft könnten ähnliche Fängerverbindungen beherbergen anderen Selektivität Gruppen verwendet werden, um Proteine in kleine Moleküle Signalisierung beteiligt sind, wie die neuartigen c-di-AMP-Effektoren zu erfassen.
Offenlegungen
The authors have nothing to disclose.
Danksagungen
We thank Alberto Reinders for his work in optimizing the CCMS conditions for P. aeruginosa. We also thank Pablo Manfredifor the annotation of the P. aeruginosa proteins. This work was supported by the Swiss National Science Foundation (SNF) Sinergia grant CRSII3_127433.
Materialien
| Name | Company | Catalog Number | Comments |
| caproBox | caprotec bioanalytics | 1-5010-001 (220 V) | UV lamps coupled to a cooling 96-plate cooling block, for the photoactivation |
| caproMag | caprotec bioanalytics | included in the CCMS Starter Kit | For easy handling of magnetic particles without pipetting |
| c-di-GMP caproKit | caprotec bioanalytics | upon request | The kit contains the c-di-GMP-capture compound, c-di-GMP (for the competition control), streptavidin coated magnetic beads, capture buffer, and washing buffer |
| Disposable PD-10 Desalting Columns | GE Healthcare | 17-0851-01 | |
| 12-tube PCR strips | Thermo Scientific | AB-1114 | |
| UIS250v sonicator with VialTweeter | Hielscher ultrasound technology | UIS250v and VialTweeter | |
| Miniature French Pressure Cell | Thermo Electron Corporation | FA-003 |
Referenzen
- Nesper, J., Reinders, A., Glatter, T., Schmidt, A., Jenal, U. A novel capture compound for the identification and analysis of cyclic di-GMP binding proteins. J Proteomics. 75, 4874-4878 (2012).
- Hengge, R. Principles of c-di-GMP signalling in bacteria. Nature reviews. Microbiology. 7, 263-273 (2009).
- Sondermann, H., Shikuma, N. J., Yildiz, F. H. You've come a long way: c-di-GMP signaling. Curr. Opin. Microbiol. 15, 140-146 (2012).
- Schirmer, T., Jenal, U. Structural and mechanistic determinants of c-di-GMP signalling. Nature reviews. Microbiology. 7, 724-735 (2009).
- Furukawa, S., Kuchma, S., O'Toole, G. Keeping their options open: acute versus persistent infections. J. Bacteriol. 188, 1211-1217 (2006).
- Christen, M., Christen, B., Folcher, M., Schauerte, A., Jenal, U. Identification and characterization of a cyclic di-GMP-specific phosphodiesterase and its allosteric control by GTP. J. Biol. Chem. 280, 30829-30837 (2005).
- Ryan, R. P., Fouhy, Y., Lucey, J. F., Dow, J. M. Cyclic di-GMP signaling in bacteria: recent advances and new puzzles. J. Bacteriol. 188, 8327-8334 (2006).
- Habazettl, J., Allan, M. G., Jenal, U., Grzesiek, S. Solution structure of the PilZ domain protein PA4608 complex with cyclic di-GMP identifies charge clustering as molecular readout. J. Biol. Chem. 286, 14304-14314 (2011).
- Fazli, M., et al. The CRP/FNR family protein Bcam1349 is a c-di-GMP effector that regulates biofilm formation in the respiratory pathogen Burkholderia cenocepacia. Molecular Microbiology. 82, 327-341 (2011).
- Hickman, J. W., Harwood, C. S. Identification of FleQ from Pseudomonas aeruginosa as a c-di-GMP-responsive transcription factor. Molecular Microbiology. 69, 376-389 (2008).
- Sudarsan, N., et al. Riboswitches in eubacteria sense the second messenger cyclic di-GMP. Science. 321, 411-413 (2008).
- Lenz, T., et al. Profiling of methyltransferases and other S-adenosyl-L-homocysteine-binding Proteins by Capture Compound Mass Spectrometry (CCMS). J Vis Exp. , (2010).
- Köster, H., et al. Capture compound mass spectrometry: a technology for the investigation of small molecule protein interactions. Assay Drug Dev Technol. 5, 381-390 (2007).
- Düvel, J., et al. A chemical proteomics approach to identify c-di-GMP binding proteins in Pseudomonas aeruginosa. Journal of Microbiological Methods. 88, 229-236 (2012).
- Christen, B., et al. Allosteric control of cyclic di-GMP signaling. J. Biol. Chem. 281, 32015-32024 (2006).
- Balasubramanian, D., Mathee, K. Comparative transcriptome analyses of Pseudomonas aeruginosa. Human genomics. 3, 349-361 (2009).
- Smyth, G. K. Linear models and empirical bayes methods for assessing differential expression in microarray experiments. Stat Appl Genet Mol Biol. 3 (3), (2004).
- Baraquet, C., Harwood, C. S. Cyclic diguanosine monophosphate represses bacterial flagella synthesis by interacting with the Walker A motif of the enhancer-binding protein FleQ. Proc. Natl. Acad. Sci. U.S.A. 110, 18478-18483 (2013).
- Nesper, J., Reinders, A., Glatter, T., Schmidt, A., Jenal, U. A novel capture compound for the identification and analysis of cyclic di-GMP binding proteins. J Proteomics. 75, 4874-4878 (2012).
- Roelofs, K. G., Wang, J., Sintim, H. O., Lee, V. T. Differential radial capillary action of ligand assay for high-throughput detection of protein-metabolite interactions. Proc. Natl. Acad. Sci. U.S.A. 108, 15528-15533 (2011).
- DeSantis, K., Reed, A., Rahhal, R., Reinking, J. Use of differential scanning fluorimetry as a high-throughput assay to identify nuclear receptor ligands. Nuclear receptor signaling. 10, e002(2012).
- Seidel, S. A., et al. Microscale thermophoresis quantifies biomolecular interactions under previously challenging conditions. Methods. 59, 301-315 (2013).
- Merighi, M., Lee, V. T., Hyodo, M., Hayakawa, Y., Lory, S. The second messenger bis-(3‘-5’)-cyclic-GMP and its PilZ domain-containing receptor Alg44 are required for alginate biosynthesis in Pseudomonas aeruginosa. Molecular Microbiology. 65, 876-895 (2007).
- Qi, Y., et al. Binding of cyclic diguanylate in the non-catalytic EAL domain of FimX induces a long-range conformational change. J. Biol. Chem. 286, 2910-2917 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten