Method Article
Utilizzando quantitativa real-time PCR per la determinazione cellulare attecchimento dei donatori in un modello murino competitivo trapianto di midollo osseo
In questo articolo
Riepilogo
Determinare l'attecchimento delle cellule del donatore rappresenta una sfida in modelli di trapianto di midollo osseo di topo che non hanno ben definiti marcatori fenotipici. Abbiamo descritto una metodologia per quantificare maschio attecchimento delle cellule del donatore nel topo femmina riceventi di trapianto. Questo metodo può essere utilizzato in tutti i ceppi di topo per lo studio delle funzioni HSC.
Abstract
Murini osseo modelli di trapianto di midollo fornire un importante strumento per la misurazione di cellule staminali ematopoietiche (HSC) funzioni e geni che determinano / molecole che regolano la CSE. In questi sistemi modello di trapianto, la funzione di CSE è determinato dalla capacità di queste cellule di topi riceventi attecchire e ricostituire letalmente irradiati. Comunemente, la cellula donatrice contributo / attecchimento si misura con gli anticorpi alle proteine delle cellule del donatore-specifici di superficie in citometria a flusso. Tuttavia, questo metodo dipende fortemente la specificità e la capacità del marcatore di superficie cellulare per differenziare donatore cellule derivate da cellule destinatario-origine, che possono non essere disponibili per tutti i ceppi di topo. Considerando i vari ambiti di provenienza di ceppi di topi geneticamente modificati sul mercato, questa superficie delle cellule / citometria a flusso a base di metodo presenta limitazioni significative soprattutto in ceppi di topi che mancano ben definiti marcatori di superficie di cellule del donatore separate da congenic destinatario ceLLS. Qui, abbiamo riportato un PCR-based tecnica per determinare l'attecchimento delle cellule del donatore / contributo in topi riceventi di trapianto. Abbiamo trapiantato maschi donatori cellule staminali emopoietiche del midollo osseo di topi irradiati letalmente congenici femminili. Campioni di sangue periferico sono stati raccolti in diversi tempi post-trapianto punti. Campioni di midollo osseo sono stati ottenuti al termine degli esperimenti. DNA genomico è stato isolato e il gene cromosoma Y specifico Zfy1, è stato amplificato mediante PCR quantitativa in tempo reale. L'attecchimento dei maschi donatori cellule derivate nei topi riceventi femminili è stato calcolato contro curva standard con percentuale nota di DNA maschile vs femminile. Bcl2 è stato usato come un gene di riferimento per normalizzare la quantità totale di DNA. I nostri dati suggeriscono che questo approccio determina affidabile attecchimento delle cellule del donatore e fornisce un metodo utile, ma semplice per misurare la ricostituzione delle cellule ematopoietiche in modelli murini di trapianto di midollo osseo. Il nostro metodo può essere eseguita di routine nella maggior parte dei laboratori perché noapparecchiature costose quali citometria di flusso è necessario.
Introduzione
Murine midollo osseo (BM) trapianto di modello è stato sviluppato nel 1960 1. Questo modello è stato ampiamente utilizzato per lo studio di cellule staminali ematopoietiche del donatore (HSC) biologia in un mouse destinatario host. Murine midollo osseo trapianto di modello ci ha fornito preziose conoscenze sulle funzioni HSC e la loro regolazione ed è indispensabile nella ricerca HSC. Trapianto allogenico di midollo osseo trapianto di modello come C57BL/6J H2b-Balb / C H2D o modello trapianto congenic come C56Bl/6J CD45.2-B6.SJL CD45.1 sono utilizzati in molti laboratori per studiare la funzione dei geni sull'attività HSC 2, effetto di trattamento farmacologico di patologie HSC funzione 3 o il trapianto correlati, come graft-versus-host disease (GvHD) 4.
Marcatori cellulari di superficie come aplotipo MHC o CD45.1 sono comunemente usati per distinguere i donatori cellule derivate da cellule destinatario-origine. C57BL/6J H2b, CD45.2 , Balb / C e H2D B6.SJL CD45.1 sono i ceppi di topi più comunemente utilizzate nel trapianto di midollo osseo in quanto il contributo cellula donatrice può essere facilmente valutata mediante citometria a flusso misurando CD45.1 vs CD45.2 o H2b vs H2D. Tuttavia, molti altri ceppi come FVB / NJ 5 e C3H sono spesso utilizzati per generare transgenici o geneticamente topi knockout. Questi topi possono essere re-incrociata a una linea inbred e mantenuto in un ambiente misto genetico / MHC sfondo. In questi casi, la determinazione delle cellule attecchimento donatore e funzione HSC potrebbe essere difficile da donatori specifici marcatori di superficie delle cellule può non essere disponibile.
Utilizzando Y-cromosoma-specifici test sul DNA per individuare le cellule del donatore di sesso maschile con macchia meridionale sesso non corrispondenti trapianto di midollo osseo è stato sviluppato dal gruppo del Dr. Miwa 6. Poi, una real-time PCR per l'determina il sesso Y regione è risultato essere un metodo accurato ed altamente specifico per quantificare maLe cellule fetali nel sangue materno sistema 7. Questo concetto è stato adattato da Dr. Schwarzenberger gruppo per lo sviluppo di una real-time PCR in un modello murino di trapianto di midollo osseo per determinare cellula donatrice attecchimento 8. Abbiamo ulteriormente modificato questo metodo per la misura di attecchimento delle cellule del donatore in FVB / NJ modello trapianto di midollo osseo del mouse. Questo metodo è attualmente ampiamente utilizzata nel nostro gruppo per studiare il ruolo della chinasi PIM1 in biologia HSC.
Protocollo
1. Midollo osseo di isolamento cellulare
- Eutanasia maschi donatori FVB / NJ topi e femmine FVB / NJ topi utilizzando CO 2 metodo seguito da dislocazione cervicale. Le femmine FVB / NJ cellule di midollo osseo saranno utilizzate come cellule competitivi.
- Utilizzare piccole forbici e pinze, sezionare femore e tibiaes da topi e metterli in un piatto 60 millimetri tessuto di coltura contenente 6 ml ghiacciata RPMI1640 con FBS di calore 5% inattivato. Usare tessuti Kimwipe per rimuovere muscolo e altri tessuti. Tagliare entrambe le estremità di ciascun albero osso nel piatto.
- Collegare l'estremità dell'osso con l'ago 23G in siringa da 3 cc, risciacquare midollo osseo con RPMI1640 con FBS di calore 5% inattivato nel piatto. Disaggregare i tessuti del midollo osseo da aspirazioni ripetute con lo stesso ago. Trasferire la sospensione cellulare a 15 ml provetta da centrifuga.
- Centrifugare le cellule per 5 min a 400 xg, rimuovere il surnatante, risospendere le cellule in 1 ml di temperatura ambiente rosso tampone di lisi delle cellule del sangue (155 mM potassium bicarbonato, cloruro di ammonio 10 mM, 0,1 mM di EDTA, pH = 7,4) e incubare a temperatura ambiente per 5 min quindi aggiungere 5-10 ml di RPMI 1640 con 5% di FBS inattivato al calore.
- Far passare le cellule attraverso un colino cellula. Raccogliere il flusso attraverso una nuova provetta. Centrifugare per 5 min a 400 x g. Rimuovere il surnatante, il pellet cellulare non dovrebbe contenere alcun colore rosso. L'assenza di colore rosso indica una rimozione completa di globuli rossi. Risospendere il pellet in 10 ml di RPMI1640 con FBS inattivato al calore 5%. Delicatamente vortex per assicurarsi che la sospensione cellulare è completamente uniforme. Prelevare un 'aliquota e contare le cellule in un emocitometro. Calcolare quanto il volume di cellule necessarie per il trapianto di midollo osseo e delle cellule aliquote abbastanza e mescolare cellule del donatore di sesso maschile con le cellule concorrenti femminili in un rapporto di 5:2. Centrifugare per 5 min a 400 xg, lavate con PBS, Risospendere in PBS alla concentrazione finale di cellule donatrici a 5 × 10 6 cellule / ml e concorrente a 2 x 10 6 / ml.
2. Competitivo di midollo osseo
- Irradiano topi riceventi femmine (8-12 settimane di età) con radiatore 137Cs raggi gamma alla dose singola di 11Gy 4-6 ore prima del trapianto di midollo osseo.
- Mettere i topi irradiati femminile in un dispositivo di immobilizzazione del mouse. Iniettare il donatore mista e cellule concorrenti con vena della coda in 0,1 ml di volume totale tale che ciascun topo riceve 5 x 10 5 cellule donatrici e 2 × 10 5 concorrente cellule di midollo osseo.
3. Raccolta dei campioni
Coppie settimane dopo il trapianto di midollo osseo, raccogliere campioni di sangue periferico (~ 50 ml) di topo ricevente femminile di retro-orbitale sanguinamento in condizioni di anestesia. I campioni vengono raccolti in tubi rivestiti EDTA. BM campioni possono essere raccolti alla fine della sperimentazione, di solito almeno 4 mesi dopo il trapianto, con modalità identiche a prima descritte (Fase 1);% di solito 20-401 tibia cellule BM sono abbastanza per fare una quantità sufficiente di DNA. Campioni di sangue o di BM di pari età topi normali femminili e maschili sono raccolti anche per preparare la curva standard.
4. Isolamento del DNA genomico
- Aggiungere 4 × volume della camera lisi temperatura del tampone RBC (~ 200 pl) per ogni campione di sangue. Mescolare bene e incubare a temperatura ambiente per 5 min. Aggiungere 1 ml di PBS, poi girare verso il basso per rimuovere la maggior parte del lisato RBC.
- Isolare DNA utilizzando un kit del DNA del sangue di estrazione (kit del DNA QIAmp Sangue estrazione). Pre-riscaldare il tampone di eluizione (AE) a 37 ° C per migliorare la resa di DNA eluito. DNA di sesso maschile e femminile per la curva standard sono isolati in modo simile.
- (Facoltativo) DNA genomico può essere ulteriormente purificato o concentrati usando EtOH metodo di precipitazione in presenza di 3 M acetato di sodio (pH = 5,5). I pellet di DNA vengono quindi risospese in 40-50 microlitri di acqua distillata per analisi immediatamente o conservato a -20 ° C per utilizzo futuro.
- Misurare la Concentrazione DNAzione con Nanodrop ND-1000 fotometro spettri. I campioni con OD 260/280 tra 1,8-2,0 vengono utilizzati per ulteriori analisi.
- Diluire con DNA DNAasi libera H 2 O a 4ng/μl in un volume totale di 50 microlitri.
5. Preparazione Curva standard
Diluire DNA maschio e femmina DNA ad una concentrazione di 4 ng / mL, e rendere la miscela di DNA secondo la Table1
| % Di DNA maschile in background Femmina | Volume (pl) di 4ng/μl DNA maschile | Volume (pl) di 4ng/μl DNA femminile | Volume totale (pl) |
| 0,2 | 1 | 499 | 500 |
| 0,5 | 1 | 199 | 200 |
| 2,5 | 5 | 195 | 200 |
| 12,5 | 25 | 175 | 200 |
| 50 | 100 | 100 | 200 |
| 87,5 | 175 | 25 | 200 |
| 100 | 200 | 0 | 200 |
Tabella 1. Preparazione del campione per la curva standard. Normali maschili e femminili FVB / NJ topi all'età di 8-12wks sono stati sacrificati. Cellule del sangue e BM sono stati raccolti. DNA da cellule maschili e femminili cellule sono state isolate e risospese ad una concentrazione di 4ng/μl. I DNA maschili e femminili sono stati miscelati in rapporti diversi per generare le miscele standard del campione di DNA.
6. Real-time PCR
- Impostare la piastra di reazione PCR mediante miscelazione dei reagenti SYBR Green Supermix con primer e del DNA genomico. Il volume di reazione (20 pl) contenente 400 nM di ciascun primer e 5 microlitri di DNA genomico delle cellule del sangue (4ng/μl pl x5 = 20 ng) in ciascuna reazione. DNA samples e gli standard sono stati istituiti in triplice copia. Le sequenze dei primers sono mostrati nella Tabella 2.
| Nome del gene | Avanti | Invertire |
| Bcl2 | 5'-AAGCTGTCACAGAGGGGCTA | 5'-CAGGCTGGAAGGAGAAGATG |
| Zfy1 | 5-TGGAGAGCCACAAGCTAACCA | 5'-CCCAGCATGAGAAAGATTCTTC |
Tabella 2. RT-PCR per la sequenza del primer Bcl2 murino e Zfy1.
- Effettuare reazione di PCR utilizzando Biorad iQ5 macchina PCR con le seguenti condizioni: 95 ° C 3 min, 42 cicli di amplificazione di 95 ° C per 10 sec, 58 ° C per 25 sec e 72 ° C per 15 secondi, seguito da una curva di fusione- passo.
- Ottenere ciclo soglia (Ct) i valori da Bio-Rad iQ5 2.1 Sistema Standard Edition ottico. Calcola ACT (Ct Zfy1 </ Sup>-Ct Bcl2) valore, e Zfy1 livello di espressione viene calcolato come valore di 2-δ Ct.
- Stabilire curve standard per ogni serie di reazione con 2-ACT lettura di note miscele standard maschio / femmina. Le curve standard sono generati tracciando la media di 2-ACT valore triplicati al DNA% noto maschio nella miscela con raccordo regressione lineare.
Risultati
Figura 1 e 2 mostrato esempi di curve standard tracciate con valori medi di 2-ACT contro percentuali di DNA maschile. Figura 1A mostrato specifica temperatura di fusione per Bcl2 e Zfy1 ampliconi localizzati a 78,5 ° C e 88,5 ° C, rispettivamente. Bcl2 è utilizzato come gene di riferimento per normalizzare la quantità totale di DNA caricato in ciascuna reazione PCR. Bcl2 curve di amplificazione (vedi Log) per ciascun campione standard fondersi tra loro indipendente dalla concentrazione di DNA maschile, indicando uguale quantità di DNA caricato (Figura 1B). Tuttavia, Zfy1 curva di amplificazione migrato verso sinistra con crescente quantità di DNA nel campione maschile (Figura 1C). Per validare il metodo, abbiamo testato maschio attecchimento delle cellule del donatore nei topi trapiantati che ricevono cellule BM da WT vs Pim knockout tripla (TKO) topi a diversi tempi post-trapianto punti. Sia il sangue periferico (6wks) e campioni di BM (16wks) w ere analizzato. CSE da Pim topi TKO hanno difetti di ricostituire topi letalmente irradiati (An, N et al., Manoscritto in corso di revisione). Come previsto, topi riceventi trapiantati con cellule Pim TKO una percentuale nettamente inferiore di cellule maschili rispetto ai topi trapiantati con cellule WT (Figura 3).
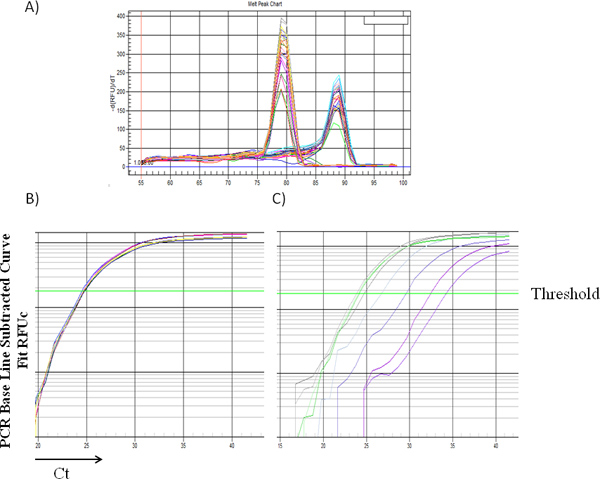
Figura 1. (A) della curva di fusione rappresentativa di campioni multipli con diverse percentuali di DNA maschile. Il picco a 78,5 indica amplificazione di Bcl2, mentre il picco a 88,5 ° C indica l'amplificazione di ZFY-1. Amplificazione curva rappresentativa di campioni multipli con diversa percentuale di DNA maschile per Bcl2 (B) e Zfy1 (C), Ct: ciclo soglia.= Target "https://www-jove-com.remotexs.ntu.edu.sg/files/ftp_upload/50193/50193fig1large.jpg" = "_blank"> Clicca qui per ingrandire la figura.

Figura 2. RT-PCR curva standard per percentuale DNA maschile. Campioni (20 ng di DNA in 20 pl di volume totale) di miscele di DNA maschili e femminili ottenuto da cellule di midollo osseo sono state preparate per la RT-PCR come descritto nel protocollo. I valori medi di ACT (Ct Zfy1-Ct Bcl2) per singoli campioni sono stati calcolati e 2-δ Ct (asse Y) sono state rilevate in percentuale DNA maschile (asse X) (da 0,2% a 100%) con la regressione lineare raccordo. R2 rappresenta il coefficiente di determinazione.
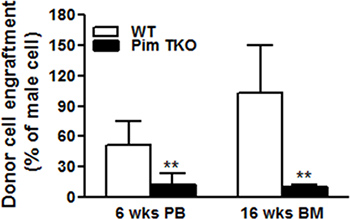
Figura 3. Competitivo trapianto di midollo osseo esperiments. 5 x 10 5 cellule totali BM di WT maschio o di pari età maschi Pim triplo KO (TKO) topi con 2 x 10 5 cellule femminili concorrenti di midollo osseo sono state trapiantate in irradiati ospiti femminili. Sangue periferico (PB) chimerismo a 6 settimane e chimerismo BM a 16 settimane post-trapianto sono stati stimati in base alla percentuale di DNA maschile mediante RT-PCR, come denunciato nel protocollo (* p <0.05, ** P <0.01).
Discussione
L'obiettivo del nostro studio è quello di fornire il pubblico una tecnica PCR per quantificare l'attecchimento delle cellule del donatore in un modello murino competitivo trapianto di midollo osseo. Diversi studi sono stati riportati con RT-PCR per rilevare le cellule del donatore in modelli di trapianto 9-10. Gruppo del Dr. Schwarzenberger primo sviluppato un modello murino di midollo osseo trapianto con real-time PCR per amplificare y-cromosoma-specifici 8. Questo metodo è stato utilizzato per studiare Gli-1 in funzione dell'attività HSC 11. Abbiamo poi ulteriormente modificato il protocollo e fornito una dettagliata, passo-passo protocollo sperimentale in formato visualizzato. La presentazione visualizzata del nostro protocollo permette al pubblico di seguire il nostro protocollo facilmente. Abbiamo anche confrontato i nostri risultati PCR utilizzando DNA genomico da campioni BM vs DNA da sangue periferico. I dati di attecchimento del donatore da BM e campioni di sangue sono sostanzialmente coerenti, tuttavia, variazione di solito meno è stato trovato in campioni di BM. Questo èprobabilmente a causa di emoglobina residua dopo lisi RBC nel sangue periferico che possono compromettere la purezza / qualità del DNA genomico nei campioni di sangue.
Ci sono molti vantaggi con la nostra tecnica basata sulla PCR per quantificare l'attecchimento delle cellule del donatore. Primo, la tecnica può essere eseguita nella maggior parte dei laboratori. Non vi è alcuna necessità di attrezzature specializzate e costose diverso tempo reale PCR macchina. Abbiamo ulteriormente modificato componente di reazione PCR e utilizzato SYBR Green supermix anziché Taqman. Questa modifica riduce ulteriormente i costi e le spese come eliminare la sonda fluorescente marcato. In secondo luogo, la nostra tecnica è affidabile e altamente riproducibile. Abbiamo utilizzato il gene Bcl2 riferimento per normalizzare la quantità totale di DNA a differenza di kit di quantificazione DNA. Gene Bcl2 è stato utilizzato perché: 1) questo gene non si trova sul cromosoma Y.. Pertanto, la sua espressione non è influenzata da attecchimento maschio cellula donatrice. 2). Bcl2 non è regolamentata nelle condizioni attuali di trapianto. Come shproprio in figura 1, Bcl2 gene produce un controllo altamente affidabile e coerente per caricamento DNA totale indipendente loaded quantità DNA maschile. Zfy1 è un gene dito di zinco nel testicolo determinano regione 12 e si trova sul cromosoma Y. E 'stato riportato che il livello di espressione genica di Zfy1 ben corrisponde alla quantità di DNA cellula maschile in un modello murino di trapianto 8. In terzo luogo, la nostra tecnica PCR è molto sensibile, e in grado di rilevare l'attecchimento delle cellule del donatore a <1%. Inoltre, i campioni possono essere conservati per lungo tempo per la successiva analisi. In contrasto, la citometria a flusso convenzionale basata metodo richiede un'elaborazione immediata campione. Infine, la nostra tecnica ceppo di topo è indipendente e può essere utilizzato per sfondo genetico che manca marcatori superficiali ben definite di cellule donatrici separate da cellule riceventi. Questo è il più importante vantaggio di questo metodo.
Sotto la nostra attuale condizione sperimentale (donatore / competitio rapporto è 5/2), abbiamo osservato intorno attecchimento donatore 80-90% a 16 settimane utilizzando il metodo basato su PCR. I nostri dati sono abbastanza confrontabili con convenzionale, citometria a flusso basato-metodo che ha mostrato ~ 95% l'attecchimento delle cellule del donatore a 16 settimane dopo il trapianto con donatore (CD45.2) / concorrente (CD45.1) rapporto a 4:1 2. Rispetto al metodo tradizionale che utilizza superficie cellulare marcatore per rilevare le cellule donatrici in topi riceventi, lo svantaggio principale del nostro metodo è l'incapacità di analizzare l'attecchimento in varie sottopopolazioni di cellule del sangue come nelle cellule B, cellule T o granulociti. Tuttavia, se necessario, è possibile raccogliere ogni popolazione di cellule usando la tecnica di separazione delle cellule e quindi soggetto i campioni per RT-PCR per determinare cella contributo donatore in ciascuna popolazione cellulare. Poiché l'attecchimento delle cellule del donatore è stato calcolato sulla base della curva standard, la lettura del campione è totalmente dipendente dalla pendenza e intercetta y della curva standard. E 'possibile che noi possiamo scendere gamma readout (vale a dire oltre il 100% di attecchimento). Tuttavia, questi "falsi positivi o falsi negativi" i risultati possono essere evitati se: 1) le procedure di isolamento del DNA per i campioni testati ed i campioni della curva standard sono identiche, e 2) la gamma di scelta curva standard è vicino a la lettura del campione previsto. Inoltre, poiché vi possono essere differenze in ciascuna reazione di PCR e in stato di taratura della macchina di PCR, i campioni curva standard devono essere inclusi in ciascuna piastra PCR per evitare variazioni tra ciascuna reazione PCR.
Infine, abbiamo convalidato la nostra tecnica con i nostri Pim triple topi KO. Coerentemente con la relazione precedente 13, abbiamo scoperto che le CSE del donatore di Pim triple topi KO sono difettosi nella ricostituzione topi riceventi letalmente irradiati.
Divulgazioni
Noi non abbiamo interessi finanziari concorrenti di divulgare.
Riconoscimenti
Ringraziamo il Dott. Carlo Greenberg per l'uso della sua macchina in tempo reale PCR. Questo lavoro è supportato da MUSC Hollings Cancer Center di avvio del fondo, Hollings Cancer Center ACS IRG, ASCO Conquer Cancer Premio alla Carriera della Fondazione per lo sviluppo, NIH 1K08HL 103780-01A1, e NIH 3P30CA138313-01S3. Il contenuto è di esclusiva responsabilità degli autori e non necessariamente rappresentano le opinioni ufficiali del National Institutes of Health o agenti di finanziamento.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | Commenti (opzionale) |
| RPMI 1640 | Hyclone | SH30255.01 | |
| FBS | Invitrogen | 16140-017 | Inattivato al calore |
| Cloruro di ammonio | MP Biomedicaals | 194806 | |
| Bicarbonato di potassio | Pescatore | P184-500 | |
| QIAamp DNA sangue minikit | Qiagen | 51106 | |
| Cella filtro | Bioscienze BD | ||
| iQ SYBR Green Supermix | Biorad | 170-8882 | |
| iQ5 real time PCR macchina | Biorad | ||
| Spectra fotometro | Nanodrop ND-1000 | ND-1000 |
Riferimenti
- McCulloch, E. A., Till, J. E. The radiation sensitivity of normal mouse bone marrow cells, determined by quantitative marrow transplantation into irradiated mice. Radiat. Res. 13, 115-125 (1960).
- Xiao, N., et al. Hematopoietic stem cells lacking Ott1 display aspects associated with aging and are unable to maintain quiescence during proliferative stress. Blood. 119, 4898-4907 (2012).
- Kang, Y., Chen, B. J., Deoliveira, D., Mito, J., Chao, N. J. Selective enhancement of donor hematopoietic cell engraftment by the CXCR4 antagonist AMD3100 in a mouse transplantation model. PLoS One. 5, e11316(2010).
- Sadeghi, B., et al. GVHD after chemotherapy conditioning in allogeneic transplanted mice. Bone Marrow Transplant. 42, 807-818 (2008).
- Taketo, M., et al. FVB/N: an inbred mouse strain preferable for transgenic analyses. Proc. Natl. Acad. Sci. U.S.A. 88, 2065-2069 (1991).
- Morisaki, H., et al. Genotypic analysis using a Y-chromosome-specific probe following bone marrow transplantation. Am. J. Hematol. 27, 30-33 (1988).
- Lo, Y. M., et al. Quantitative analysis of fetal DNA in maternal plasma and serum: implications for noninvasive prenatal diagnosis. Am. J. Hum. Genet. 62, 768-775 (1998).
- Byrne, P., et al. Chimerism analysis in sex-mismatched murine transplantation using quantitative real-time PCR. Biotechniques. 32, 279-280 (2002).
- Ma, X., et al. Contribution of recipient-derived cells in allograft neointima formation and the response to stent implantation. PLoS One. 3, e1894(2008).
- Bosio, E., et al. A comparison between real-time quantitative PCR and DNA hybridization for quantitation of male DNA following myoblast transplantation. Cell Transplant. 13, 817-821 (2004).
- Merchant, A., Joseph, G., Wang, Q., Brennan, S., Matsui, W. Gli1 regulates the proliferation and differentiation of HSCs and myeloid progenitors. Blood. 115, 2391-2396 (2010).
- Nagamine, C. M., Chan, K., Hake, L. E., Lau, Y. F. The two candidate testis-determining Y genes (Zfy-1 and Zfy-2) are differentially expressed in fetal and adult mouse tissues. Genes & development. 4, 63-74 (1990).
- Grundler, R., et al. Dissection of PIM serine/threonine kinases in FLT3-ITD-induced leukemogenesis reveals PIM1 as regulator of CXCL12-CXCR4-mediated homing and migration. J. Exp. Med. 206, 1957-1970 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon