Method Article
Studiare Checkpoint mitotica illustrando un comportamento dinamico delle proteine cinetocoro e Motion cromosoma in Living
In questo articolo
Riepilogo
Il cinetocore è dove il SAC inizia il suo segnale di controllo della segregazione mitotica dei cromatidi fratelli. Un metodo è descritto per visualizzare il reclutamento e il fatturato di una delle proteine cinetocoro e il suo coordinamento con il movimento dei cromosomi in Drosophila Embrioni usando una Leica sistema laser confocale a scansione.
Abstract
L'assemblaggio del mandrino checkpoint (SAC) meccanismo è un segnale attivo, che controlla l'interazione tra cinetocori cromosomiche e microtubuli del fuso per prevenire l'insorgenza anafase fino a quando i cromosomi sono collegati correttamente. Le cellule utilizzare questo meccanismo per impedire l'instabilità genomica o di aneuploidia, e quindi tumori e altre malattie umane, come difetti alla nascita e l'Alzheimer 1. Un certo numero di componenti SAC come Mad1, Mad2, Bub1, BubR1, Bub3, Mps1, Zw10, Rod e Aurora B chinasi sono stati identificati e sono tutte le proteine cinetocoro dinamiche 2. L'evidenza suggerisce che il cinetocore è dove il segnale SAC viene avviata. L'obiettivo primario SAC normativo Cdc20. Cdc20 è uno dei principali APC / C (A naphase P ROMUOVERE omplex C o C yclosome) attivatori 3 ed è anche una proteina cinetocoro dinamico 4-6. Quando attivato, il SAC inibisce l'attività di APC / C per evitare la distruzione dei due substrati chiave, ciclina B e securin, impedendo così la metafase anafase di transizione 7,8. Esattamente come il segnale SAC è avviato e montato sulle cinetocori e trasmesso sul APC / C per inibire la sua funzione rimane ancora sfuggente.
Drosophila è un sistema estremamente docile sperimentale, un organismo molto più semplice e più comprensibile rispetto a quella umana, ma che condivide i processi fondamentali in comune. Si tratta, forse, uno dei migliori organismi da utilizzare per studi di bio-imaging in cellule viventi, in particolare per la visualizzazione degli eventi mitotici nello spazio e nel tempo, come l'embrione precoce passa attraverso 13 cicli rapidi divisione nucleare sincrono (8-10 minuti per ogni ciclo a 25 ° C) e organizza gradualmente i nuclei in un monostrato unico appena sotto la corteccia 9.
Ecco, vi presento un bio-imaging metodo che utilizza espressioni Drosophila transgenicosing GFP (Green Fluorescent Protein) o la sua variante con targeting per proteine di interesse e una Leica TCS SP2 sistema di microscopio confocale a scansione laser per studiare la funzione SAC in mosche, mostrando immagini di proteine di fusione GFP di alcuni dei componenti del CAS, Cdc20 e Mad2 , come esempio.
Protocollo
1. Mosche transgeniche e manutenzione
- Il transgenico mosche usato in questa dimostrazione: UBQ-GFP-Cdc20 (II), UBQ-RFP-His2B (X *); UBQ-GFP-Cdc20 (II *), UBQ-GFP-Cdc20 (II *); Mad2 EY ( III *) e UBQ-GFP-Cdc20 mosche transgeniche sono state precedentemente generato in laboratorio attraverso una norma P-element approccio mediato transgenico 10,11 e UBQ-RFP-His2B è un gentile dono da Yohanns Belaïche a 144 UMR CNRS / Istituto Curie, Parigi, Francia. Essi sono stati introdotti in uno sfondo Mad2 mutante attraverso standard Drosophila genetica. La linea di Mad2 EY originale mutante è stato acquistato dal magazzino Bloomington centro. Non discutere la procedura utilizzata per l'allevamento dei trasformanti in questo protocollo.
Nota: * rappresenta il numero dei cromosomi. - Manutenzione: mosche transgeniche sono state mantenute a 25 ° C in fiale di plastica containing volare il cibo e con l'aggiunta di lievito in polvere secca sulla parte superiore. La fiala è stata regolarmente sostituiti ogni 3-4 settimane a seconda delle condizioni di crescita (figura 1).
2. Vola Preparazione del cibo (scala Lab)
- Una quantità appropriata di miscela alimentare mosca è stata riscaldata con agitazione costante per sciogliere i componenti.
- A proposito di 8-10 ml di questo mezzo è stato distribuito come impasto in ogni fiala di plastica (2,5 cm di diametro x 8 cm di lunghezza) con un Jencons Scientific Ltd pompa peristaltica.
- Quando l'impasto alimentare è stato impostato e si è raffreddata a temperatura ambiente, la fiala viene poi tappato con un tappo di cotone schiuma. Queste fiale alimenti sono collocati in un vassoio che viene quindi confezionato in un sacchetto di plastica e mantenuto a 4 ° C per un uso successivo.
3. Piccola Egg Collection
- Circa 50 coppie di 2-3 mosche adulte giorno vecchi sono stati trasferiti in una fiala di alimenti freschi fly fornito con altra polvere di lievito secco sulla sua superficie a 25 ° C per layIng embrioni.
- Le mosche sono poi trasferiti in una fiala fresca ogni ora e lasciare gli embrioni nel flacone per 30 minuti per garantire alcuni degli embrioni raccolti sono di età compresa tra circa 8-10 nucleari ciclo di divisione, quando i nuclei sono migrando gradualmente verso la corteccia e organizzata come una monostrato singolo. La collezione prima ora viene normalmente scartato in quanto spesso contiene embrioni di età che sono stati conservati nei corpi femminili quando le condizioni non erano adatte per la posa.
4. Preparazione Coprivetrini e diapositive
- Estrarre uno x 50 22 mm e coprioggetto leggermente bagnare suoi quattro angoli su un lato con una quantità molto piccola di acqua utilizzando una spazzola umida penna fine e messo su un vetrino da microscopio in modo che il vetrino non muoversi a causa della tensione superficiale capillare causato dalla pellicola sottile di liquido.
- Applicare una sottile striscia di colla eptano in tutto il centro del vetrino, l'eptano dovrebbe evaporare in pochi secondi per lasciare la colla su vetrino.
- Tagliare un altro vetrino con una penna di diamante in piccoli quadrati (~ 1,5 mm 2), prendere uno e appoggiarla su una delle estremità della striscia di colla (questo è utilizzato per aprire la punta dell'ago microiniezione quando è necessario) e salvare gli altri per il futuro utilizzare.
- Prendete un altro vetrino da microscopio e bastone un pezzo di nastro biadesivo adesivo di circa 2 cm ad esso e staccare la carta della copertina. (Vedi Figura 2A & B).
5. Embrioni Dechorionate
- Trasferire le mosche in una fiala di cibi freschi e utilizzare un pennello inumidito penna sottile di trasferire circa 50 uova sul nastro biadesivo adesivo sul vetrino con una quantità appropriata di liquido per permettere alle uova di essere distribuiti. Quando il liquido è evaporato le uova si attacchi al nastro.
- Sotto un tocco microscopio da dissezione le uova più volte lungo il loro asse lungo usando il lato esterno di una metà di un paio di pinzette delicatamente fino corion è rotto e lasciare aperta ma l'embrione in Choragli ioni di shell per prevenire la disidratazione rapida. Continuare questo processo finché 10-20 delle uova è stato trattato (a seconda del numero delle uova richieste per un singolo esperimento).
- Gli embrioni possono essere raccolte e rimossi dal guscio corion e delicatamente posto sulla striscia di colla uno per uno. Le uova possono essere posizionati e allineati sia con il lato lungo parallelo dell'embrione o anti-parallelo al lato lungo della striscia di collante dipende se devono essere microiniettati.
- Gli embrioni possono essere coperto immediatamente con una quantità appropriata di olio Voltalef 10S (una miscela 05:01 di Holocarbon 700 olio e olio 27, Sigma) per prevenire l'essiccamento ulteriormente per l'imaging immediato o collocato in una camera di essiccazione per 3-8 minuti fino disidratare ulteriormente a seconda dell'umidità della camera e lo scopo dell'esperimento. Gli embrioni disidratati dovrebbero essere coperti con olio 10S, se del caso per evitare eccessiva disidratazione se microiniezione è richiesto. (Vedi Figura. 2C, D& E).
6. Imaging embrioni
- Time-lapse o singole immagini di embrioni viventi sono raccolti utilizzando una Leica TCS SP2 sistema invertito microscopia confocale con un PL APO HCX CS 40X 1,25 obiettivo a immersione in olio (60 o obiettivi 100X può essere utilizzato anche a seconda dello scopo dell'esperimento, maggiore l'ingrandimento utilizzato la foto più sbiancamento si verificherà).
- Quando immagini fluorofori multipli, che hanno spettri sovrapposti e sono co-espressi nell'embrione stesso, ogni lunghezza d'onda di emissione fluorescente proteina è stata prelevati sequenzialmente. Le lunghezze d'onda di eccitazione e di emissione disponibili sul sistema Leica TCS SP2 sono: GFP, λ Ex: 488 nm e λ Em: 500-600 nm. RFP, λ Ex: 543 nm e λ Em: 555-700 nm.
- Una singola immagine era comunemente ottenuto come media di 2 linee di scansione con una media di 2 telaio-scan. Questo richiede un minimo di 6,3 secondi per la scansione di un p 512x512Ixel immagine in una modalità di scansione simultanea e 15.44 secondi per una modalità di scansione sequenziale e produce un buon rapporto segnale-rumore. Questi tempi devono essere presi in considerazione nel fissare i tempi per la time-lapse raccolta di immagini.
- Il foro stenopeico è stato normalmente impostato a 1-2 AU (unità arioso), e la potenza del laser è stata regolata a livello più basso possibile per evitare qualsiasi photobleaching significativo.
7. Provocare la SAC da Microinjecting degli embrioni con reagenti appropriati
- Preparare l'ago prima dell'esperimento. Noi preferiamo estrarre l'ago dal vetro siliconato capillari (GC-100-10, Harvard) utilizzando il fiammeggiante / marrone micropipetta estrattore (Instrument Sutter, Modello: P-97) con parametri: il calore = 600, tirare = 90, vel = 70 , del = 150.
- In sostegno degli l'ago con 1-2 ml di una concentrazione appropriata del reagente in tampone iniezione usando una punta fine di caricamento (20 pl lotto Eppendorf: W215818J).
- Montare l'ago nel Ho agolder sul microscopio con il tubo di mandata collegato al sistema di controllo dell'iniezione Eppendorf.
- Trova un embrione sotto obiettivo 40X e trovare l'ombra dell'ago senza modificare il piano focale spostando l'ago nel centro del campo visivo e gradualmente si abbassa al piano focale destra.
- Spostare l'embrione dolcemente per trovare la piccola piazza del vetrino rotto pre-montato ad una estremità della striscia di colla. Molto delicatamente spostare la punta dell'ago fino a raggiungere il bordo del vetrino piazzetta e si rompe delicatamente aperto.
- Spostare gli embrioni di nuovo in vista e selezionare l'embrione l'età giusta per l'iniezione. Io di solito iniettare l'embrione al ciclo di divisione nucleare a 5-7 come questo è prima di migrare verso l'esterno i nuclei alla corteccia. Le fasi nucleare ciclo di divisione può essere determinata da una scansione rapida dei segnali fluorescenti del GFP-Cdc20 o il segnale nucleare di RFP-Histone 2B prima dell'iniezione 9.
- Muovere con cautela il Embryo vicino all'ago e regolare nuovamente il piano focale sia per l'embrione e l'ago. Spostare l'embrione in l'ago ed iniettare una goccia del reagente nella posizione desiderata e spostare l'embrione di distanza (o seguire le istruzioni per il sistema microiniezione). L'embrione può essere iniettata dal suo posteriore o dal lato a seconda dello scopo dell'esperimento.
- Una volta iniettata l'embrione è pronto per essere ripreso al momento opportuno. (Vedi Figura 2F & G)
8. Risultati rappresentativi

Figura 1. Materiale occorrente: a. pen pennello sottile, b. piazzetta rotti gli occhiali contenitore, c. 22 x 50 mm coprioggetti, d. vetrino da microscopio, e. nastro biadesivo appiccicoso, f. lievito in polvere secca provetta con fori sul coperchio, g. lievitogranuli utilizzato per la manutenzione fly, h. volare vial cibo, i. un mezzo di una pinzetta utilizzati per embrioni dechorionate, j. una pinzetta, k. eptano colla contenitore di liquido (matraccio).
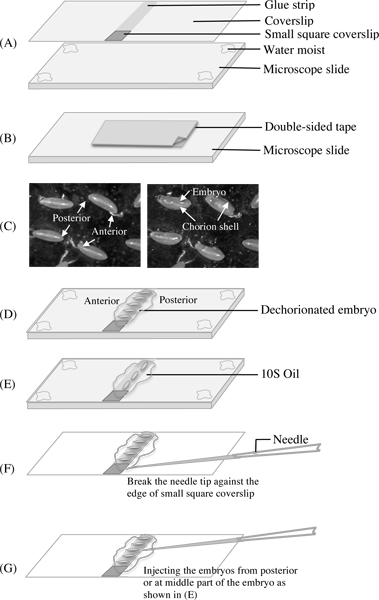
Figura 2. I diagrammi mostrano la preparazione dei vetrini coprioggetto e diapositive nel passaggio 4, dechorionation embrione nel passaggio 5 e preparati ad aghi per la microiniezione al punto 7. A. L'immagine in alto mostra un vetrino con una striscia di colla eptano in tutta la sua media e una piccola piazza di un vetrino rotto attaccato ad una estremità. L'immagine inferiore mostra un vetrino da microscopio con un sottile strato di acqua ai suoi quattro angoli per tenere un vetrino coprioggetto. La ragione per l'utilizzo di una diapositiva per tenere il vetrino è quello di mantenere o meno la stessa distanza focale che si trova quando si ha del nastro biadesivo sulle slitte e evitando ricentrare il microscopio mentre si are dissezione e trasferire gli embrioni tra il nastro e coprioggetto. B. Una diapositiva con un breve tratto di nastro biadesivo Scotch applicato prima che si staccava il suo documento di copertura utilizzato per dechorionating l'embrione. C. L'immagine a sinistra mostra embrioni con gusci coriali intatti contrassegnati con estremità anteriori e posteriori, l'immagine a destra mostra gli embrioni coriali i gusci rotti, prima che fossero trasferiti sul vetrino con la striscia di colla D & E diagrammi che mostrano due.. modi per il trasferimento, posizionamento e l'allineamento degli embrioni dechorionated su strisce di colla. Gli embrioni sono stati coperti da olio 10S dopo un periodo di essiccamento appropriato. Diagrammi F e G. mostrano come aprire la punta dell'ago e la posizione in modo da iniettare.
Immagini singole o time-lapse ottenuti dagli esperimenti sopra descritti possono essere analizzati direttamente con il software Leica o salvati in file TIF come un open FOdisponibili per la quantificazione ulteriore o le analisi di editing con altri comuni programmi di analisi delle immagini come immagine J, Photoshop e MetaMorph, ecc Due esempi per illustrare questo MATERIALE sono discussi di seguito:
Esempio 1: Un time-lapse movie (Figura 3) mostra il reclutamento dinamica cinetocoro di GFP-Cdc20 e il movimento dei cromosomi nel vivere embrioni di Drosophila sinciziale. La regione di interesse da parte originali time-lapse è stata determinata sequenza di immagini / a cura e automatizzato-lotto convertiti con il software Photoshop. Il film è stato assemblato utilizzando il software QuickTime.
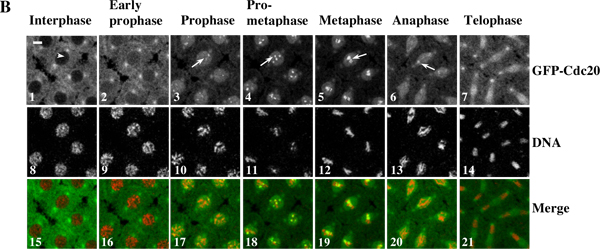
Figura 3. Un time-lapse filmato mostra il reclutamento dinamica cinetocoro di GFP-Cdc20 e il movimento dei cromosomi nel vivere embrioni di Drosophila sinciziale. (A) Time-lapse immagini sono state scattate da un embrione transgenico sinciziale co-esprimono GFP-Cdc20 (in verde)e RFP-Histone 2B (in rosso) e di proteine di fusione sono stati registrati usando un sistema confocale Leica TCS SP2 a 18 ° C durante i cicli di divisione nucleare 7-8. I frame sono stati prelevati ogni 10 secondi. Il telaio con le cellule già in profase viene considerato come il punto di tempo zero 10. Clicca qui per vedere il film per la figura 3A . (B) GFP-Cdc20 può essere facilmente osservata profase e prometafase cinetocori (B3 e 4, frecce bianche) e persiste su metafase e anafase cinetocori (B5 e 6, frecce bianche). GFP-Cdc20 è esclusa dalla interfase nucleo (freccia bianca), inserendo il nucleo di profase precoce. Morfologie cromatina sono stati determinati usando co-espressi His2BmRFP come marcatori (B8-14). Bar = 5mm.
Esempio 2: funzioni SAC può essere studiata attraverso la manipolazione degli embrioni da anticorpi, proteine microinjecting fluorescente o Chemical composti di interesse che potenzialmente innescano il SAC. Ad esempio, l'iniezione di colchicina depolimerizzano i microtubuli in modo da provocare la SAC come indicato in Figura 4 10.

Figura 4. Mad2 è essenziale per la colchicina-invocato la funzione SAC 10. GFP-cdc20; Mad2 + / +, GFP-cdc20; Mad2 EY (Mad2 - / - null mutante) e GFP-Mad2; Mad2 embrioni EY sono stati trattati con colchicina con microiniezione. Time-lapse immagini confocali sono state prese prima (01, 07 e 13) o dopo l'iniezione (02-06, 08-12; pannelli superiori, pannelli intermedi; 14-18, pannelli inferiori). GFP-Cdc20 (pannelli top e middle) o GFP-Mad2 (pannello inferiore) segnali cinetocoro sono state usate come marcatori di progressione del ciclo cellulare. Pannello superiore, le frecce indicano i cinetocori arrestati a 02-06. L'area contrassegnato da una freccia in 01 indica che prima del trattamento colchicina, GFP-Cdc20 viene escluso dal nucleo interfase tardi. Pannello centrale, in assenza di endogeno Mad2, GFP-Cdc20 segnali continuano ad oscillare in e fuori del nucleo, e dentro e fuori le cinetocori, sebbene citocinesi presenta difetti, come ci si aspetterebbe in embrioni privi microtubuli, in presenza di colchicina (indicato dalle frecce nella 07-12) suggerendo una funzione SAC fallito arrestare cellule in risposta al trattamento colchicina. La separazione del nucleo figlia fallito in figura 10. Pannello inferiore, punte di freccia indicano in 14-18 cinetocori arrestati con GFP-Mad2 accumulato proteine di fusione per suggerire funzionale GFP-Mad2 in soccorso il fenotipo difetto SAC in Mad2 embrione mutante. Arrowhead in 13 indica GFP-Mad2 accumulo in un nucleo interfase tardi. Bar = 5mm. Gli embrioni sono stati microiniettati con ~ volume di uovo 1% di una soluzione 100mg/ml magazzino colchicina in 1 x PBS.
Discussione
Il protocollo qui descritto è un metodo generico per l'imaging intero embrioni sinciziale utilizzando un Leica TCS SP2 microscopio confocale a scansione laser e può essere modificata per adattarsi altri sistemi microscopio e possono anche essere adattate per studiare le funzioni geniche altre utilizzano embrioni Drosophila sinciziale. Abbiamo usato questo protocollo per studiare molti aspetti del checkpoint di assemblaggio del mandrino, la dinamica delle proteine e proteolisi delle proteine con la mosca transgenici o anticorpi policlonali per visualizzare la localizzazione delle proteine nei campioni viventi o fissi 4,6,12-14.
E 'più semplice per mantenere le mosche in fiale, fresco, cibo mosca al momento del ritiro numeri piccoli (~ 10 - 100) di uova. Questo riduce il fastidio di fare e la preparazione di ulteriori piastre di agar succo di mela e le camere di raccolta delle uova, come descritto in altre pubblicazioni 15,16. L'aggiunta di un piccolo quadrato coprioggetto di vetro rotto ad una estremità della striscia di colla sul vetrino rende molto più facileper aprire l'ago e determinare il volume di iniezione regolando le impostazioni microiniezione di sistema, mentre l'ago è immergere nell'olio 10S. Il grado di essiccazione dell'embrione è importante per il successo della iniezione di evitare perdite e mantenere la vitalità embrioni soprattutto per gli esperimenti che richiedono un lungo periodo di tempo durante il sollevamento o trasformanti dopo l'iniezione. Tuttavia, non vi è alcuna regola d'oro esattamente per quanto tempo di essiccazione è richiesto. Questo varia da esperimento a esperimento e dipende dalla quantità di soluzione iniettata e l'umidità dell'ambiente di lavoro.
Le funzioni di una specifica proteina di interesse può essere manipolato da microinjecting inibitori della proteina specifici, mono-o poli-clonale gli anticorpi contro una proteina specifica, l'RNA messaggero o peptidi fluorescente sotto wild type o sfondi mutazione genetica. Molte di queste linee mutanti o GFP-tagged linee transgeniche sono accessibili al pubblico available dalla Drosophila centri di magazzino e la maggior parte di essi sono elencati sul Flybase ( http://flybase.org/~~V ) e possono essere consultate online.
Divulgazioni
Non abbiamo nulla da rivelare.
Riconoscimenti
Questo protocollo è stato sviluppato nell'ambito di un Wellcome Trust sovvenzione. Ringraziamo la signora Maureen Sinclair per il mantenimento degli stock mosca e preparare il cibo volo nel corso degli anni. Vorremmo anche ringraziare il Sig. Michael Aitchison per il suo aiuto e il supporto tecnico nello sviluppo di questo protocollo.
Materiali
Attrezzature essenziali e reagenti:
Sistema di imaging confocale: Il sistema di imaging descritto in questo protocollo è un Leica TCS SP2 sistema di scansione laser confocale microscopio invertito. Il metodo descritto è adatto anche forse con alcune piccole modifiche per altri sistemi di imaging.
Dissezione microscopio: Usiamo un SZX7 Olympus con DF PLAPO 1X-4 lenti.
Needle puller: Flaming / marrone micropipetta estrattore (da Sutter Instrument, Modello: P-97).
Microiniezione sistema: Un sistema microiniezione Eppendorf è descritto ma altro sistema idoneo può essere utilizzato.
Fly distributore alimentare: Jencons Scientific Ltd pompa peristaltica.
Reagenti:
| Name | Company | Catalog Number | Comments |
| Ingredienti | Pesos | Risorse | Lot No. |
| Farina di mais | 100.0g | SUMA, UK | |
| Di zucchero di canna | 50.0g | Billington, il Regno Unito | |
| Lievito in polvere | 25.0g | DCL LIEVITO Ltd. UK | |
| Agar | 12.5g | Fisher Scientific | 106556 |
| L'acido sorbico | 0.4g | BDH, VWR International Ltd. UK | 8829310 |
| Acido benzoico | 2.9g | Fisher Scientific | 1019599 |
| Nipagina (Metil-4-Hydro xybenzoate) | 0,9 g | BDH, VWR International Ltd. UK | K35969015 |
| H 2 O fino a 1L | |||
Holocarbon 700 e 27 oli acquistati da Sigma.
Lievito secco: Allison Thomas, UK.
Preparare la colla eptano: Prendere circa 1,5 metri di nastro biadesivo Scotch, metterlo in un tubo Falcon da 15 ml con 5 ml di eptano e ruotare per 3-5 ore o durante la notte. Dopo quel tempo diviso in 4 aliquote uguali in 1,5 ml provette Eppendorf e centrifugare per 5 min a velocità elevata in una centrifuga da banco per rimuovere eventuali detriti. Conservare la soluzione di eptano colla in un pallone tarato da 10 ml e mantenere per l'uso. La forza colla varierà a seconda della concentrazione e le quantità di colla utilizzata. Normalmente, la colla più sottile produce meno rumore di fondo durante la scansione al microscopio confocale.
- Coverslip: BDH, VWR International Ltd. UK
- Vetrino da microscopio: BDH, VWR International Ltd. UK
- Siliconato capillari di vetro: GC-100-10, Harvard
- Caricamento a punta sottile: 20μl molto eppendorf: W215818J
- Pinzetta
- Pen pennello sottile
Riferimenti
- Holland, A. J., Cleveland, D. W. Boveri revisited: chromosomal instability, aneuploidy and tumorigenesis. Nat. Rev. Mol. Cell Biol. 10, 478-4787 (2009).
- Zekanowski, C., Wojda, U. Aneuploidy, chromosomal missegregation, and cell cycle reentry in Alzheimer's disease. Acta Neurobiol. Exp. (Wars). 69, 232-253 (2009).
- Buffin, E., Emre, D., Karess, R. E. Flies without a spindle checkpoint. Nat. Cell Biol. 9, 565-572 (2007).
- Tang, Z., Shu, H., Oncel, D., Chen, S., Yu, H. Phosphorylation of Cdc20 by Bub1 provides a catalytic mechanism for APC/C inhibition by the spindle checkpoint. Mol. Cell. 16, 387-397 (2004).
- Huang, J., Raff, J. W. The disappearance of cyclin B at the end of mitosis is regulated spatially in Drosophila cells. EMBO Journal. 18, 2184-2195 (1999).
- Xia, G. Conformation-specific binding of p31(comet) antagonizes the function of Mad2 in the spindle checkpoint. Embo. J. 23, 3133-3143 (2004).
- Lorca, T. Fizzy is required for activation of the APC/cyclosome in Xenopus egg extracts. Embo. J. 17, 3565-3575 (1998).
- Howell, B. J. Cytoplasmic dynein/dynactin drives kinetochore protein transport to the spindle poles and has a role in mitotic spindle checkpoint inactivation. J. Cell Biol. 155, 1159-1172 (2001).
- Foe, V. E., Alberts, B. M. Studies of nuclear and cytoplasmic behaviour during the five mitotic cycles that precede gastrulation in Drosophila embryogenesis. Journal of cell science. 61, 31-70 (1983).
- Li, D., Morley, G., Whitaker, M., Huang, J. Y. Recruitment of Cdc20 to the kinetochore requires BubR1 but not Mad2 in Drosophila melanogaster. Mol. Cell Biol. 30, 3384-3395 (2010).
- Karess, R. E., Glover, D. P element mediated germ line transformation of Drosophila. DNA cloning: A practical approach. 2, 121-141 (1985).
- Wakefield, J. G., Huang, J. Y., Raff, J. W. Centrosomes have a role in regulating the destruction of cyclin B in early Drosophila embryos. Current Biology. 10, 1367-1370 (2000).
- Huang, J. Y., Raff, J. W. The dynamic localisation of the Drosophila APC/C: evidence for the existence of multiple complexes that perform distinct functions and are differentially localised. Journal of Cell Science. 115, 2847-2856 (2002).
- Raff, J. W., Jeffers, K., Huang, J. Y. The roles of Fzy/Cdc20 and Fzr/Cdh1 in regulating the destruction of cyclin B in space and time. Journal of Cell Biology. 157, 1139-1149 (2002).
- Brust-Mascher, I., Scholey, J. M. Microinjection Techniques for Studying Mitosis in the Drosophila melanogaster Syncytial Embryo. J. Vis. Exp. (31), e1382 (2009).
- Figard, L., Sokac, A. M. Imaging Cell Shape Change in Living Drosophila Embryos. J. Vis. Exp. (49), e2503 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon
Utilizziamo i cookies per migliorare la tua esperienza sul nostro sito web.
Continuando a utilizzare il nostro sito web o cliccando “Continua”, accetti l'utilizzo dei cookies.
