Method Article
ניתוח דיגיטלי של מעטפת חיסונית של חלבון ZW10 אינטראקציה רקמות ריאות האדם
In This Article
Summary
ZW10 (ZWINT) משתתפת במחסום הציר הmitotic ובפתוגנזה של קרצינומה. כאן, אנו מציגים מתודולוגיה של כתמים חיסוני של ZWINT ברקמות סרטן ריאות האדם, ואחריו סריקה דיגיטלית של שקופיות שלמות וניתוח תמונה. מתודולוגיה זו יכולה לספק תמונות דיגיטליות באיכות גבוהה ותוצאות אמינות.
Abstract
מטרת מחקר זה היא להחדיר מתודולוגיה של הרקמה החיסונית של רקמות הריאה האנושית, ואחריו שקופית שלמה סריקה דיגיטלית ניתוח תמונה. סריקה דיגיטלית היא דרך מהירה לסריקת ערימת שקופיות ולהפקת תמונות דיגיטליות באיכות גבוהה. זה יכול לייצר תוצאות concordant עם מיקרוסקופ אור קונבנציונאלי (CLM) על ידי פתולוגים. יתר על כן, הזמינות של תמונות דיגיטליות מאפשרת כי אותה שקופית ניתן לציין בו על ידי אנשים מרובים. יתר על כן, תמונות דיגיטליות של שקופיות ניתן לאחסן במסד נתונים, מה שאומר את הידרדרות ארוכת טווח של שקופיות זכוכית נמנעת. המגבלות של טכניקה זו הן כדלקמן. ראשית, הוא זקוק לרקמה באיכות גבוהה המוכן ואת השקופיות המקורי אימונוהיסטוכימיה (IHC) ללא כל נזק או שאריות איטום עודף. שנית, הגידול או שטחים שאינם סרטניים צריך להיות מצוין על ידי פתולוגים מנוסים לפני ניתוח באמצעות תוכנה, כדי למנוע בלבול על הגידול או אזורים שאינם סרטניים במהלך הבקיע. שלישית, המפעיל צריך לשלוט על שכפול הצבע לאורך כל תהליך הדיגיטציה בהדמיה שקופית שלמה.
Introduction
ZW10 (zwint) הוא מרכיב הכרחי של מתחם קינטוכורה אשר מעורב במחסום mitotic צירים1,2,3. זה דווח כי דלדול של zwint מוביל הפרדה חריגה כרומוזום מוקדם מוקדמת1,2,3. מחקרים שנעשו לאחרונה הראו כי zwint מעורב בפתוגנזה של גידולים מרובים על ידי קידום התפשטות של תאים סרטניים4,5. בעבר דיווחו על ביטוי יתר של ZWINT בסרטן הריאות5. זה התקבל באופן נרחב כי ניתוח של שקופיות על ידי פתולוגים באמצעות clm היא גוזלת זמן ולא כמותית6,7,8. יתרה מזאת, ההידרדרות של שקופיות הזכוכית המאוחסנות עלולה לגרום לנסיגה של שקופיות שנוצרו בעבר. השיטה המתהווה של הדמיה מבוססת-מחשב, שקופית שלמה דיגיטלית (wsi) עלולה להתגבר על מגבלות אלה6,7,8.
לשם כך, אנו מתארים מתודולוגיה של התרופה החיסונית של ZWINT ברקמות סרטן ריאות האדם, בשילוב עם שקופית שלמה סריקה דיגיטלית וניתוח תמונה מבוסס תוכנה. היתרון העיקרי של מתודולוגיה זו הוא הפקת תוצאות concordant עם CLM. טכנולוגיה זו ניתן להשתמש באופן נרחב באזורים של הבקיע פתולוגי של המטאוקסילין-אאוזין כתמים (H & E) ו-IHC, הזריחה ב היברידיזציה באתרו (דג), מיקרו מערכי רקמות (tma), וגילוי סמים ופיתוח.
Protocol
כל השיטות המתוארות כאן אושרו על ידי הוועדה האתית של בית החולים Zhongnan של ווהאן והחולים רנמין באוניברסיטת ווהאן.
1. הכנת שקופיות IHC
- לתקן את דגימת רקמת הריאה על ידי האישר רסיס רקמת הריאה האנושית (כ 3 x 3 ס מ) ב 4% פאראפורמלדהיד באגירה פוספט מתכלה (PBS) עבור 24 h בטמפרטורת החדר (RT).
- מייבשים את הרקמה ב 80%, 95%, ו 100% אתנול עבור 15 דקות, 20 דקות, ו 20 דקות, בהתאמה, בטמפרטורת החדר. לבסוף, לטבול את הרקמה 100% קסילן 3x עבור 20 דקות כל אחד בטמפרטורת החדר.
- להטבעת, לטבול את הרקמה בפרפין (63 ° c) עבור 30 דקות. שינוי פרפין לטבול את הרקמה שוב עבור 45 דקות נוספות. לבסוף, לשנות את הפרפין שוב ולטבול את הרקמה בו עבור 1 h.
- השתמש מיקרוטומה רוטרי לחתוך את הרקמה לתוך 5 μm-עובי מקטעים ולמקם אותם על 20 μg/mL פולי-L-ליזין מצופה מגלשות.
- עבור הסרת שעווה, למקם את השקופיות בתנור 65 ° c עבור 2 h ולאחר מכן, לטבול את הסעיפים diמתילבנזן עבור 15 דקות, ואחריו הטבילה ב 100% קסילן עבור 15 דקות.
- מימה את השקופיות עם סעיפים הרקמה 2x עבור 5 דקות ב 100% אתנול, ואחריו טיפול עם אתנול מדולל באופן סדרתי של 5 דקות ב 95% אתנול, 5 דקות ב 80% אתנול, 5 דקות ב 70% אתנול, ו 5 דקות ב 50% אתנול.
- בצע תיקון אנטיגן.
- לדלל חומצת לימון לתקן נוזל (20x) ל-1x עם מזוקקים פעמיים H2o (ddh2o).
- מניחים את השקופיות עם מקטעי הרקמה ואת חומצת לימון 1 x לתקן נוזל (300 mL) בתיבת תיקון אנטיגן ולאחר מכן, לחמם אותם במיקרוגל בחשמל גבוה עבור 2 דקות, ואחריו חימום בחשמל נמוך כדי לקיים הרתיחה עבור 6 דקות.
- אפשר לקטעים להתקרר באופן טבעי לטמפרטורת החדר.
- להחליף את חומצת לימון לתקן נוזל עם 0.01 M PBS, לשטוף את סעיפים 3x, בכל פעם עבור 5 דקות, ב שייקר רוטרי בטמפרטורת החדר.
- . סלק את הperoxidase האנדודוגני
- לדלל 30% של H2או2 כדי 3% עם ddh2o, להוסיף אותו לשקופיות (לוודא שהוא מכסה את כל הרקמות), ו מודיית את השקופיות בתיבה רטובה בטמפרטורת החדר עבור 30 דקות.
הערה: התיבה הרטובה היא קופסת פלסטיק של 10 x 15 ס מ והיא יכולה להכיל מים. - לשטוף את השקופיות 3x, בכל פעם עבור 5 דקות, עם 0.01 M PBS ב שייקר רוטרי בטמפרטורת החדר.
- לדלל 30% של H2או2 כדי 3% עם ddh2o, להוסיף אותו לשקופיות (לוודא שהוא מכסה את כל הרקמות), ו מודיית את השקופיות בתיבה רטובה בטמפרטורת החדר עבור 30 דקות.
- חסום את האנטיגן הלא ספציפי.
- לדלל את סרום העז הרגיל מרוכז 10% סרום עז עם PBS עם 0.1% רצף 20 (PBST).
- הוסף 10% סרום עז לשקופיות ומודחים אותם בקופסה רטובה ב 37 ° c עבור 30 דקות.
הערה: סרום העז צריך לכסות את כל הרקמות בשקופיות.
- . להכתים את הרקמות
- מאסטר השקופיות עם הארנב אנטי-ZWINT נוגדן נגד האדם (1:50) ב 4 ° c הלילה.
- לשטוף את השקופיות 3x, בכל פעם עבור 5 דקות, עם 0.01 M PBST (1 מ ל של 0.1% רצף 20 ב 1,000 mL) ב שייקר רוטרי בטמפרטורת החדר.
- דגירה את השקופיות עם נוגדן משני (מערכת איתור הארנב, 1:200) עבור 30 דקות ב 37 ° c.
- לשטוף את השקופיות 3x, בכל פעם עבור 5 דקות, עם 0.01 M PBST ב שייקר רוטרי.
- כדי להמחיש את הנוגדנים, להוסיף את השקופיות 300 μL של טרי 3, 3-diaminobidine ולאחר מכן, לשטוף את השקופיות עם מים ברז בטמפרטורת החדר.
הערה: מידת הצביעה מפוקחת תחת מיקרוסקופ (הגדלה 10X-40X). - . להכתים את הרקמה הכתם את השקופיות עם המטאוקסילין מכתים פתרון עבור 2 דקות בטמפרטורת החדר ולאחר מכן, שטוף את השקופיות עם מי ברז עבור 15 דקות.
הערה: הגרעין נראה כחול תחת המיקרוסקופ (הגדלה 10X-40X). אם המידה של כתמים כחולים הוא חזק יותר, בידול אלכוהול הידרוכלורית עבור 3-5 s יכול לשמש, ואחריו שטיפה את רקמת בחזרה כחול עם מים ברז עבור 15 דקות כדי להקל על כתמים בולטים של הרקמה. - מייבשים את השקופיות עבור 5 דקות ב 50% אתנול, 5 דקות ב 70% אתנול, 2x ב 80% אתנול עבור 5 דקות, 5 דקות ב 95% אתנול, ולבסוף, 2x עבור 5 דקות כל אחד ב 100% אתנול בטמפרטורת החדר.
- עבור שקיפות, לטבול את מקטעי הרקמה במיכל המכיל 100% קסילן עבור 15 דקות ולהעביר את הסעיפים למיכל אחר המכיל 100% קסילן עבור 15 דקות בטמפרטורת החדר.
- עבור איטום, לקחת 50-100 μL של מסטיק נייטרלי ולהוסיף 5% קסילן לתערובת (להימנע ערבוב בועות). הוסף 15 μL של התערובת הנ ל לסעיפים רקמת הריאה. חותם את הסרט עם זכוכית כיסוי ובעדינות ללחוץ את הבועות החוצה.
2. השלמה אוטומטית-סריקת שקופיות של IHC
- הניחו לשקופיות החתומות להתייבש במשך 12 שעות בטמפרטורת החדר כדי להתייבש.
- בחר באפשרות השדה הבהיר באופן ידני והזן את ממשק הסריקה הידני.
- שנה את שם השקופיות ובחר נתיב אחסון עבור התמונות.
- הגדר את אזור הסריקה ובחר את האפשרויות סריקת דוגמאות באמצעות ספי הגדרת משתמש. הסף הוא על 50 עם ערך הרחבה סריקה שטח של 200 μm.
- הגדר את ערך מוקד הסריקה, בחר את המוקד באופן אוטומטי — הוא נע בין 1050 ל-2650-ולבסוף, בחר במצב בשכבה אחת.
- טען את שקופיות IHC בתחנת העבודה והפעל את הסריקה האוטומטית.
הערה: ודא שהשקופיות אינן פגומות. שמירה על שקופיות נקיות ושקופות והימנעו מאבק, קונפטי ושאריות איטום עודפים על השקופיות. שקופית הזכוכית והכיסויים. צריכים להיות נטולי סימנים אל תנקה את השקופיות עם קסילן. גודל השקופיות מוענק בטבלה 1. - סרוק באופן אוטומטי את כל מקטע הרקמה בשקופית עם הגדלה של 20X, 40X או 100X.
- אסוף ואחסן את התמונות בסוף סריקת השקופיות כולה.
3. ניתוח השקופיות באמצעות תוכנת דימות
- הפעל את תוכנת ניתוח התמונה histopathological ובחר מחשב מקומי.
- פתח את הקובץ שמאחסן את נתוני הסריקה.
- בחר את רמת הזום המתאימה מהטווח 2X עד 40X.
- כוונן את הצבע כדי לקבוע את הניגודיות המיטבית באמצעות התאמת צבעדו-מצבי.
- עיגול ידני ושמו את החלקים המעניינים בכל תמונה.
הערה: למשל, אתה יכול להקיף אזור אחד ולשם אותו כמו אזור הגידול. - בחר תוספים ו- QC. . בשלב זה , בונה התרחישים צץ
- בחר quant צפיפות והתאם את סרגל הזיהוי.
הערה: יש לבצע תהליך זה בזהירות. בר הסובלנות הכחולה משמש להתאמת הצבע של הגרעין התאי. סרגל העמידות החום משמש להתאמת הרוויה של צבע המילוי בתמונה. תוצאות התמונות לאחר ההתאמות צריכות להיות עקביות עם הקבצים המקבילים המקוריים. - כוונן את רמות הניקוד כדי להפוך את עוצמת הצביעה של האזורים המוקפים בעיגול בדומה לזו של התמונות המקוריות.
הערה: חום כהה מציין חיובי חזק, צהוב חום הוא חיובי למדי, צהוב בהיר הוא חלש, ולבן הוא שלילי. - אחסן והשם את המודל כקובץ. החל את המודל על שקופיות אחרות ובחר ביאור נבחר".
- תן לתוכנה לחשב באופן אוטומטי את תוצאת ה-H של האזורים המוקפים בתמונה.
4. קוונפיקציה של הציונים באמצעות מערכת הניקוד H9,10
- חישוב אחוז הנוגדנים ועוצמת הצביעה (0: שלילי, 1 +: חלש, 2 +: בינוני, 3 +: חזק).
- השתמש במערכת הבקיע (H-score) כדי לחשב את הניקוד הפתולוגי של הגידול ואזורים סמוכים שאינם סרטניים.
הערה: H-score = (% של תאים עם עוצמה חלשה x 1) + (% של תאים עם עוצמה מתונה x 2) + (% של תאים עם עוצמה חזקה x 3). לכן, ה-H-score המקסימלי הוא 300 אם 100% מהתאים מתבכמת בעוצמה חזקה.
תוצאות
מדדנו את רמות הביטוי של ZWINT ב 28 זוגות של שאינם קטנים-תאים סרטן הריאות (NSCLC) דגימות (הגידול הסמוך רקמות שאינם סרטניים), כולל 14 תאים קשקשיים קרצינומה (SCCs) ו 14 אדנוקרצינומות (ADCs), על ידי IHC. הסריקה הדיגיטלית השלמה של השקופיות המסופקות תמונות דיגיטליות באיכות גבוהה (איור 1A). התוצאות הראו כי הציון ה-H של סרטן הריאות היה גבוה משמעותית מזה של רקמות סמוכות שאינן סרטן (P < 0.0001, דו-זנבית t-Test) (איור 1A ו- 1a). בנוסף, מצאנו כי רמת הביטוי של ZWINT ב SCCs היה גבוה משמעותית מזה ב-ADCs (איור 1C).
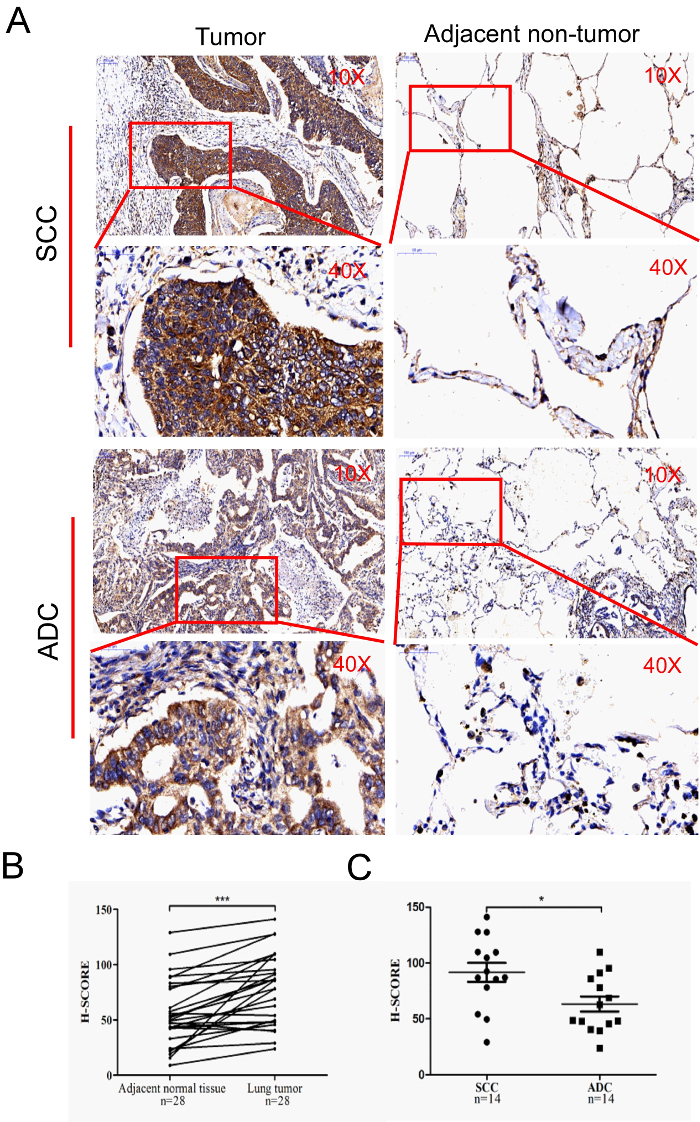
איור 1 : מכתים אימונוהיסטוכימיה לרקמות סרטן ריאות. דמות זו שונתה מיואן ואח אל. 5 עם אישור מעיתונות דאב. (א) פאנל זה מציג את מייצגת הדמויות האימונוהיסטוכימיה של חולי סרטן הריאות (המאצות: 10x ו-40x). (ב) פאנל זה מציג את ה-H-ציון של סרטן ריאות ורקמות סמוכות שאינן סרטן. (ג) פאנל זה מציג את הציון הגבוה של adcs ו-sccs. קווי השגיאה מציינים את סטיית התקן. * P < 0.05; . פי < 0.001 אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
| MIN (ממ) | מקס (ממ) | |
| רוחב | 25 | 26 |
| אורך | 75 | 76 |
| עובי | 0.9 | 1.2 |
טבלה 1: גודל שקופית.
Discussion
סריקת שקופית שלמה הופכת לנושא חם עבור סריקה והפקה איתנה של תמונות באיכות גבוהה למטרות קליניות ומחקר11,12,13. ניתן לייצר תמונות על ידי מיקרוסקופים סריקה שקופיות בתוך דקות11,12,13. על-ידי החלת מתודולוגיה זו, הצלחנו להשיג תמונות באיכות גבוהה עבור שקופיות של ZWINT IHC ולהשוות את ה-H-ניקוד בין רקמות הגידול לבין שאינם סרטניים. בפרוטוקול המוצג כאן, השלבים הקריטיים ביותר הם הכנת מגלשות ה-ihc באיכות גבוהה, רכישת התמונה והמפרט של אזורי הגידול והגידול לפני הניקוד הפתולוגי13,14 , מיכל בן 15 , . שישה עשרה
לשיטה הנוכחית יש כמה יתרונות לעומת (CLM). סריקה דיגיטלית מהירה מספיק (~ 20 שקופיות/שעה) כדי לסרוק ערימת שקופיות ולהפיק תמונות דיגיטליות באיכות גבוהה. זה יכול לייצר תוצאות concordant עם CLM אבל לוקח פחות זמן יחסית. הזמינות של תמונות דיגיטליות מאפשרת שניתן לצפות באותה שקופית בו על-ידי אנשים מרובים. תמונות דיגיטליות של שקופיות יכולות להיות מאוחסנות במסד נתונים, כלומר הידרדרות לטווח ארוך של שקופיות זכוכית נמנעת.
המגבלות של טכניקה זו כוללות את הצורך ברקמות באיכות גבוהה ו-ihc שקופיות6,8,11 ומומחיות טכנית כדי לציין את דגימות הגידול או רקמות שאינן גידולים לפני ניתוח באמצעות תוכנת הדמיה , על מנת למנוע כל בלבול על הגידול או האזורים שאינם סרטניים במהלך הבקיע6,7. המפעיל גם צריך לפקח על שכפול הצבע לאורך כל תהליך הדיגיטציה בהדמיה שקופית שלמה17.
שיטה זו ניתן להחיל באופן פעיל דגים, tma, וגילוי התרופה ופיתוח6,18. Tabata ואח ' חקרו את השימוש ב-wsi באבחנות פתולוגיים מרכזיות19. הם ניתחו באופן מיוחד 1070 WSI דגימות מתשעה בתי חולים ביפן ואישר את האימות של השימוש WSI באבחונים הראשוניים. אחד היתרונות העיקריים של WSI הוא שהוא מאפשר צפייה בו של השקופיות על ידי תלמידים מרובים20. לפיכך, האימות של תמונות סריקה של השקופיות השלמה עשוי לתרום לפיתוח אבחון דיגיטלי למטרות חינוכיות ומחקר6,18,21.
Disclosures
. למחברים אין מה לגלות
Acknowledgements
פרויקט זה היה נתמך על ידי הקרן הלאומית הטבעית של סין (No. 81500151, 81400121, 81270607, 81541027, ו 81501352) ואת היסוד הטבעי של מחוז חוביי (סין) (No 2017CFB631). המחברים מבטאים את הערכתם לגואיי קין, צ'אנג מין, לי הוי, ועמיתיו בחברת ווהאן Google ביולוגית טכנולוגיה ושות, בע מ עבור התמיכה הטכנית שלהם. המחברים גם מודים למוחמד ג'מאל על עריכת השפה.
Materials
| Name | Company | Catalog Number | Comments |
| Pannoramic MIDI | 3D HISTECH | Cat: PMIDI-040709 | An automated digital slide scanner with a remarkable feature set :12-slide capacity, fluorescence scanning, and many more. |
| QuantCenter | 3D HISTECH | Downloaded from the official website of the company | The framework for 3DHISTCH's image analysis applications. |
| LEICA RM2235 | Leica Microsystems | Cat: 14050038604 | The enhanced precision of the new accessories will add convenience to block to knife approach as well as specimen orientation. |
| Rabbit anti-human Anti-ZWINT antibody | Abcam | Cat: ab197794 | Immunohistochemical analysis of ZWINT in human lung tissue. |
| Anti-rabbit secondary antibody | Wuhan Goodbio Technology | Cat:GB23303-1 | Secondary antibody for IHC staining. |
| Phosphate-buffered saline | Wuhan Goodbio Technology | Cat:G0002 | A solution containing a phosphate buffer. |
| OLYMPUS CX23 | OLYMPUS | Cat:6M87620 | Microscope for detection of H&E or IHC slides. |
| Dimethylbenzene | Shanghai Lingfeng Chemical Reagent | Cat:1330-20-7 | A colorless, flammable fluid used as a solvent and clarifying agent in the preparation of tissue sections for microscopic study. |
| Hematoxylin Staining Solution | Wuhan Servicebio technology | Cat:G1039 | It is commonly used for histologic studies, oftern colors the nuclei of cells blue. |
| Tween 20 | Baitg | Cat:2005-64-5 | It is a polysorbate-type nonionic surfactant formed by the ethoxylation of sorbitan before the addition of lauric acid. It is used as a deterent and emulsifier in pharmacological applications. |
| Citric acid repair liquid | Wuhan Servicebio technology | Cat:G1202 | Is is used to repair antigen after fixation during IHC procedure. |
| LEICA ASP200s | Leica | Cat: 14048043626 | It was designed for routine and research histopathology of up to 200 cassettes. |
| LEICA Arcadia H | Leica | Cat: 14039354103 | It is a heated paraffin embedding station and allows for simple operation and precise control, resulting in improved quality, a smooth workflow and reliability. |
| LEICA Arcadia C | Leica | Cat: 14039354102 | It is a cold plate holding more than 60/65 cassettes on its large working surface. It was designed with an environment adaptive control module to make sure the operating temperature is always stabilized at -6°C. |
| CaseViewer Software | 3DHISTECH |
References
- Endo, H., Ikeda, K., Urano, T., Horie-Inoue, K., Inoue, S. Terf/TRIM17 stimulates degradation of kinetochore protein ZWINT and regulates cell proliferation. The Journal of Biochemistry. 151 (2), 139-144 (2012).
- Wang, H., et al. Human Zwint-1 specifies localization of Zeste White 10 to kinetochores and is essential for mitotic checkpoint signaling. Journal of Biological Chemistry. 279 (52), 54590-54598 (2004).
- Lin, Y. T., Chen, Y., Wu, G., Lee, W. H. Hec1 sequentially recruits Zwint-1 and ZW10 to kinetochores for faithful chromosome segregation and spindle checkpoint control. Oncogene. 25 (52), 6901-6914 (2006).
- Ying, H., et al. Overexpression of Zwint predicts poor prognosis and promotes the proliferation of hepatocellular carcinoma by regulating cell-cycle-related proteins. OncoTargets and Therapy. 11, 689-702 (2018).
- Yuan, W., et al. Bioinformatic analysis of prognostic value of ZW10 interacting protein in lung cancer. OncoTargets and Therapy. 11, 1683-1695 (2018).
- Higgins, C. Applications and challenges of digital pathology and whole slide imaging. Biotechnic & Histochemistry. 90 (5), 341-347 (2015).
- Webster, J. D., Dunstan, R. W. Whole-slide imaging and automated image analysis: considerations and opportunities in the practice of pathology. Veterinary Pathology. 51 (1), 211-223 (2014).
- Al-Janabi, S., Huisman, A., van Diest, P. J. Digital pathology: current status and future perspectives. Histopathology. 61 (1), 1-9 (2012).
- Bonomi, P. D., et al. Predictive biomarkers for response to EGFR-directed monoclonal antibodies for advanced squamous cell lung cancer. Annals of Oncology. 29 (8), 1701-1709 (2018).
- Villalobos, M., et al. ERCC1 assessment in upfront treatment with and without cisplatin-based chemotherapy in stage IIIB/IV non-squamous non-small cell. Medical Oncology. 35 (7), 106 (2018).
- Griffin, J., Treanor, D. Digital pathology in clinical use: where are we now and what is holding us back?. Histopathology. 70 (1), 134-145 (2017).
- Huisman, A., Looijen, A., van den Brink, S. M., van Diest, P. J. Creation of a fully digital pathology slide archive by high-volume tissue slide scanning. Human Pathology. 41 (5), 751-775 (2010).
- Gray, A., Wright, A., Jackson, P., Hale, M., Treanor, D. Quantification of histochemical stains using whole slide imaging: development of a method and demonstration of its usefulness in laboratory quality control. Journal of Clinical Pathology. 68 (3), 192-199 (2015).
- Hofman, F. M., Taylor, C. R. Immunohistochemistry. Current Protocols in Immunology. 103, (2013).
- Ramos-Vara, J. A. Principles and Methods of Immunohistochemistry. Methods in Molecular Biology. 1641, 115-128 (2017).
- Otali, D., Fredenburgh, J., Oelschlager, D. K., Grizzle, W. E. A standard tissue as a control for histochemical and immunohistochemical staining. Biotechnic & Histochemistry. 91 (5), 309-326 (2016).
- Clarke, E. L., Treanor, D. Colour in digital pathology: a review. Histopathology. 70 (2), 153-163 (2017).
- Potts, S. J. Digital pathology in drug discovery and development: multisite integration. Drug Discovery Today. 14 (19-20), 935-941 (2009).
- Tabata, K., et al. Whole-slide imaging at primary pathological diagnosis: Validation of whole-slide imaging-based primary pathological diagnosis at twelve Japanese academic institutes. Pathology International. 67 (11), 547-554 (2017).
- Saco, A., Bombi, J. A., Garcia, A., Ramírez, J., Ordi, J. Current Status of Whole-Slide Imaging in Education. Pathobiology. 83 (2-3), 79-88 (2016).
- Griffin, J., Treanor, D. Digital pathology in clinical use: where are we now and what is holding us back?. Histopathology. 70 (1), 134-145 (2017).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved