Test de la plaque : méthode de détermination de la charge virale exprimée en unités formant des plaques
Vue d'ensemble
Source: Tilde Andersson1, Rolf Lood1
1 Département des sciences cliniques Lund, Division of Infection Medicine, Biomedical Center, Lund University, 221 00 Lund, Suède
Les virus qui infectent les organismes procaryotes, appelés bactériophages ou simplement phages, ont été identifiés au début duXXe siècle par Twort (1) et d'Hérelle (2) indépendamment. Les phages ont depuis été largement reconnus pour leur valeur thérapeutique (3) et leur influence sur les écosystèmes humains (4), ainsi que sur les écosystèmes mondiaux (5). Les préoccupations actuelles ont alimenté un regain d'intérêt pour l'utilisation des phages comme alternative aux antibiotiques modernes dans le traitement des maladies infectieuses (6). Essentiellement, toutes les recherches sur les phages reposent sur la capacité de purifier et de quantifier les virus, également connu sous le nom de titre viral. Initialement décrit en 1952, c'était le but de l'assiduité de la plaque (7). Des décennies et de multiples progrès technologiques plus tard, l'analyse de la plaque demeure l'une des méthodes les plus fiables pour la détermination du titre viral (8).
Les bactériophages subsisent en injectant leur matériel génétique dans les cellules hôtes, en détournant les machines pour la production de nouvelles particules de phage, et finalement en provoquant l'hôte de libérer de nombreuses virions progéniture par lyse cellulaire. En raison de leur taille infime, les bactériophages ne peuvent pas être observés à l'aide uniquement de microscopie légère; par conséquent, la microscopie électronique de balayage est nécessaire (figure 1). En outre, les phages ne peuvent pas être cultivés sur les plaques d'agar nutritionnelles comme les bactéries, car ils ont besoin de cellules hôtes pour s'en prendre à eux.

Figure 1 : La morphologie d'un bactériophage, ici illustrée par un phage E. coli, peut être étudiée à l'aide de la microscopie électronique à balayage. La plupart des bactériophages appartiennent aux Caudovirales (bactériophages à queue). Ce phage particulier a une structure de queue très courte et une tête icosaèdre, le plaçant dans la famille des Podovirus.
L'essai de plaque (figure 2) est basé sur l'incorporation des cellules hôtes, préférentiellement dans la croissance de phase de journal, dans le milieu. Cela crée une couche dense et turbide de bactéries capables de soutenir la croissance virale. Un phage isolé peut ensuite infecter, se répliquer et lyser une cellule. Avec chaque cellule lysée, plusieurs cellules adjacentes deviennent immédiatement infectées. Plusieurs cycles dans, une zone claire (une plaque) peut être observée dans la plaque autrement turbide (figure 2B/figure 3A), indiquant la présence de ce qui était initialement une particule bactériophage unique. Le nombre d'unités de formation de plaques par volume(c.-à-d. PFU/mL) d'un échantillon, peut donc être déterminé à partir du nombre de plaques générées.
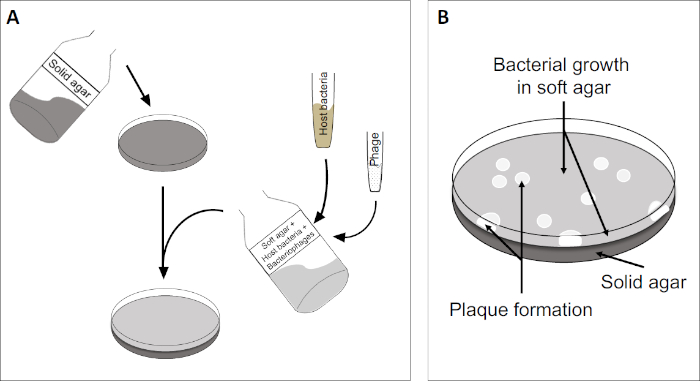
Figure 2 : Le dépistage des unités de formation de plaque (PFU) est une méthode courante pour déterminer le nombre de bactériophages dans un échantillon. (A) La base d'un plat stérile Petri est recouverte d'un milieu nutritif solide approprié, suivie d'un mélange de milieux mous, de cellules hôtes sensibles et d'une dilution de l'échantillon de bactériophage d'origine. Notez que la suspension de phage pourrait, dans certains cas, également être répartie uniformément sur la surface de l'agar mou déjà solidifié. (B) La croissance des bactéries hôtes forme une pelouse de cellules dans la couche supérieure d'agar. La réplication des bactériophages génère des zones claires, ou plaques, causées par la lyse des cellules hôtes.
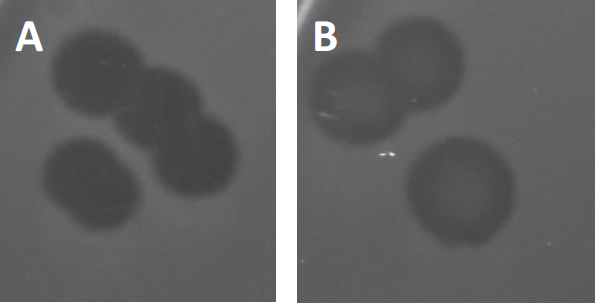
Figure 3 : Les résultats des tests PFU montrent plusieurs plaques générées par des bactériophages. En raison de la lyse des cellules hôtes sensibles, les plaques peuvent être considérées comme des zones de dégagement dans la pelouse bactérienne, soit avec (A) dégagement complet, soit (B) re-croissance partielle causée par la génération de bactéries résistantes (ou peut-être par des phages tempérés dans le cycle lysogénique).
Certains phages tempérés peuvent adopter ce qu'on appelle un cycle de vie lysogénique, en plus de la croissance lytique précédemment décrite. En lysogénie, le virus assume un état latent par l'incorporation de son matériel génétique dans le génome de la cellule hôte (9), conférant souvent une résistance à d'autres infections phages. Cela se révèle parfois par un léger obscurcissement de la plaque (figure 3B). Il est intéressant de noter cependant, que les plaques peuvent également apparaître floues en raison de la repousse des bactéries qui ont évolué résistance au phage indépendamment des infections phage précédente.
Les virus peuvent se fixer, ou adsorb, à seulement une gamme limitée de bactéries hôtes (10). Les plages d'hôtes sont encore limitées par des stratégies antivirales intracellulaires telles que le système CRISPR-Cas (11). La résistance/sensibilité à l'égard de phages spécifiques affichés par les sous-groupes bactériens a toujours été utilisée pour classer les souches bactériennes en différents types de phages (figure 4). Bien que l'efficacité de cette méthode ait maintenant été surpassée par de nouvelles techniques de séquençage, la dactylographie peut encore fournir des informations précieuses sur les interactions bactéries-phage, par exemple, en facilitant la conception d'un cocktail de phage pour une utilisation clinique .

Figure 4 : Sensibilité aux phages de différentes souches bactériennes. Des plaques d'agar molles avec la souche d'acné de Cutibacterium (A) AD27 et (B) AD35, ont été repérées avec 21 bactériophages différents de C. acnes. Seul phage 11 a été en mesure d'infecter et de tuer AD27 tandis que la souche AD35 a montré une sensibilité envers tous les phages. Cette technique, appelée dactylographie phage, peut être utilisée pour diviser les espèces bactériennes et les souches en différents sous-groupes basés sur la susceptibilité au phage.
Procédure
1. Mise en place
- Avant de commencer tout travail impliquant des microbes, assurez-vous que l'espace de travail est stérilisé(p. ex. essuyé avec 70 % d'éthanol). Portez toujours une blouse de laboratoire et des gants, gardez les cheveux longs attachés en arrière et assurez-vous que les plaies sont particulièrement bien protégées.
- Une fois terminé, stériliser toutes les surfaces et bien laver /stériliser les mains et les poignets.
2. Protocole
- Préparation de LB Media
Note: Selon la souche bactérienne hôte et le bactériophage, un milieu liquide différent peut être plus approprié pour la culture initiale de la souche bactérienne hôte ou un milieu solide différent peut être plus approprié pour la croissance ultérieure. Le bouillon de lysogénie (LB) est utilisé dans ce protocole pour le bouillon et l'agar.- Mélanger 4 g LB dans de l'eau distillée de 200 ml, en triplicate, pour le bouillon LB, l'agar solide en fond et l'agar doux. Toutes les solutions doivent être préparées dans des conteneurs capables de contenir deux fois le volume final pour éviter les débordements pendant l'autoclacage.
Note: Si vous effectuez l'assay en triple, préparez le double de la quantité d'agar de fond LB. - Ajustez le pH des trois solutions à 7.4 en utilisant NaOH ou HCl le cas échéant.
- Ajouter 3 g de puissance d'agar à la bouteille d'agar inférieure pour faire une solution d'agar de 1,5% pour l'agar solide
- Ajoutez 1,2 g de puissance d'agar à la bouteille d'agar supérieure pour faire une solution d'agar de 0,6 % pour l'agar doux.
- Placer les bouteilles, avec des bouchons semi-serrés, dans un autoclave réglé à 121 oC pendant 20 min pour stériliser les solutions. Fermez les bouchons dès que la course est terminée pour éviter la contamination.
- Lorsque le support LB a atteint une température d'environ 45-50 oC, ajouter 450 l de stérile 1 M CaCl2 aux trois solutions lb de 200 ml pour faire une concentration finale de 2,25 mM.
- Verser 15 ml d'aliquots du milieu d'agar solide dans des plats petri stériles (éviter de secouer pour éviter l'écume) et laisser l'agar se solidifier pendant quelques heures ou toute la nuit à température ambiante (figure 4A). Les plaques peuvent être stockées à l'envers à 4 oC pendant plusieurs jours.
- Mélanger 4 g LB dans de l'eau distillée de 200 ml, en triplicate, pour le bouillon LB, l'agar solide en fond et l'agar doux. Toutes les solutions doivent être préparées dans des conteneurs capables de contenir deux fois le volume final pour éviter les débordements pendant l'autoclacage.
- Cultiver des cellules hôtes
- Un jour avant l'assiduité, ajouter 10 l de culture E. coli à 10 ml de bouillon LB.
- Incuber la bactérie à 37oC pendant la nuit à 160 tr/min dans un incubateur secouant.
- Le matin de l'astodonte, ajouter 0,5 ml de la culture de nuit à 10 ml de LB frais.
Note: Si vous effectuez l'assay en triple, préparez le double de cette culture. - Incuber le 37oC à 160 tr/min dans un incubateur secouant jusqu'à ce que la culture bactérienne soit en phase de croissance. Cela peut être déterminé spectrophotométriquement par un OD600 de 0,5-0,7.
- Gardez la culture à température ambiante jusqu'à ce que les bactéries soient ajoutées à l'agar LB supérieur.
- 10 fois dilution en série du bactériophage
- Ajouter 180 l de bouillon LB à sept puits dans la première rangée d'une assiette de 96 puits
Note: Il est suggéré d'effectuer la dilution en triplicate afin d'augmenter sa fiabilité statistique. Pour ce faire, préparer des dilutions supplémentaires du bactériophage dans les deuxième et troisième rangées de la plaque. - Vortex soigneusement l'échantillon de bactériophage d'origine pour assurer l'homogénéité et transférer 20 L dans le premier puits.
- Bien mélanger l'échantillon par pipetting et vers le bas.
- Transférer 20 ll de la suspension résultante dans le deuxième puits.
- Continuer la dilution en série en transférant 20 l l de la solution dans le second puits dans le troisième puits, et ainsi de suite, jusqu'à ce que le sixième puits, laissant le septième bien comme un contrôle négatif à laquelle aucune suspension de phage ne sera ajouté. Cela créera une plage de dilution de 10-1-10-6.
- Ajouter 180 l de bouillon LB à sept puits dans la première rangée d'une assiette de 96 puits
- Placage
- Étiquetez la base des plats Petri (précédemment préparés à l'étape 2.1.7) avec le nom, la date et une courte description de l'échantillon (y compris le facteur de dilution de l'échantillon de phage). Préchauffer les plats Petri dans un incubateur à 37oC une heure avant l'assidu.
- Faire fondre le milieu d'agar mou solidifié (généralement fait à l'aide d'un four à micro-ondes; l'agar solidifié fond à 85 oC), et laisser venir à environ 45 oC. Les supports chauffés peuvent être placés dans un bain d'eau de 45 oC pendant 1h pour atteindre une température appropriée. Il doit être assez chaud pour rester sous forme liquide, mais assez frais pour ne pas tuer les bactéries ajoutées.
- Mélanger la culture bactérienne de 4 ml (à partir de l'étape 2,2) avec une gélose molle de 35 ml lb (à 45 oC). Tourbillonner pour répartir uniformément les cellules, mais éviter de secouer pour éviter l'écume (Figure 4A).
- Étiquetez un tube à essai stérile pour chacune des étapes de dilution en série et un pour l'échantillon témoin, pour un total de 7 tubes à essai étiquetés. Placer 5 mL d'aliquots de la culture bactérienne/mélange d'agar doux de l'étape 2.4.3 dans les 7 tubes. Travaillez rapidement à l'étape 2.4.6 parce que ces petits volumes de supports à base d'agar se solidifieront rapidement à température ambiante.
- Ajouter 100 l de l'échantillon témoin (à partir de l'étape 2.3) au tube d'essai de commande et tourbillonner soigneusement. Jetez la pointe de pipette usagée et transférez le même volume de chacun des échantillons de bactériophage dilués en série (étape 2.3) à leurs tubes à essai respectifs, tourbillonnant pour mélanger.
- Transférer immédiatement les mélanges de 5 ml sur les plaques d'agar étiquetées, préchauffées et solides (Figure 4A). Faites tourbillonner doucement les assiettes pour même étendre les mélanges.
Note: Si vous effectuez l'assay en triple, répétez deux fois de plus les étapes 2.4.3-6 deux fois de plus. - Sceller chaque plaque avec du film de laboratoire et laisser les deux couches se solidifier correctement à température ambiante (environ 15 minutes) avant de les placer vers le bas d'un incubateur de 37 oC, stimulant la croissance des bactéries et du phage, pendant 24 heures ou jusqu'à ce que les plaques développer, Il faut généralement environ 1-5 jours pour les plaques à apparaître (Figure 4B), mais le calendrier varie considérablement en fonction des conditions d'incubation, moyen, et les espèces bactériennes.
3. Analyse des données et résultats
-
Compter les plaques
- Assurez-vous qu'aucune plaque n'est visible dans les plaques marquées « contrôle », car cela indiquerait une contamination virale.
- Commencez par les plaques étiquetées 10-6, contenant l'échantillon de bactériophage le plus dilué. Comptez les plaques sans enlever le couvercle, en les marquant avec un stylo que vous allez pour indiquer quelles plaques ont déjà été comptés.
- Comptez les plaques restantes. Certaines plaques peuvent avoir trop peu ou trop de plaques à compter. Utilisez les plaques avec 10-150 plaques pour une analyse plus approfondie.
-
Calcul de l'UF
- Diviser le nombre de plaques par le facteur de dilution (ex. 10-6 pour l'échantillon le plus dilué) pour obtenir le nombre d'unités de formation de plaque (PFU) dans 100 l de mélange de phage.
Note: Si vous effectuez le récarthèse en triple, utilisez le nombre moyen de plaques des trois plaques. - Pour déterminer la concentration (dans l'UL/mL) de l'échantillon original, multipliez par un facteur de dilution supplémentaire de 10, puisque seulement 100 l'échantillon a été plaqué. (c'est à dire
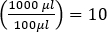 )
) - Calculez la valeur moyenne du PFU/mL pour toutes les dilutions qui avaient entre 10 et 150 plaques pour atteindre un résultat plus fiable.
- Diviser le nombre de plaques par le facteur de dilution (ex. 10-6 pour l'échantillon le plus dilué) pour obtenir le nombre d'unités de formation de plaque (PFU) dans 100 l de mélange de phage.
Applications et Résumé
Malgré les multiples progrès technologiques, les essais de plaque demeurent l'étalon-or pour la détermination du titre viral (comme PFU) et essentiels à l'isolement des populations de bactériophages purs. Les cellules hôtes sensibles sont cultivées dans la couche supérieure d'une plaque d'agar à deux couches, formant un lit homogène permettant une réplication virale. L'événement initial où un bactériophage isolé dans le cycle de vie lytique infecte une cellule, se reproduit à l'intérieur, et éventuellement la lyse, est trop petit pour l'observer. Cependant, les virions libérées infecteront les cellules adjacentes, donnant par la suite lieu à une zone de dégagement, ou plaque, dénotant la présence d'un seul PFU.
References
- Twort, F. An investigation on the nature of ultra-microscopic viruses. Lancet. 186 (4814): 1241-1243. (1915)
- d'Hérelle, F. An invisible antagonist microbe of dysentery bacillus. Comptes Rendus Hebdomadaires Des Seances De L Academie Des Sciences. 165: 373-375. (1917)
- Cisek AA, Dąbrowska I, Gregorczyk KP, Wyżewski Z. Phage Therapy in Bacterial Infections Treatment: One Hundred Years After the Discovery of Bacteriophages. Current Microbiology. 74 (2):277-283. (2017)
- Mirzaei MK, Maurice CF. Ménage à trois in the human gut: interactions between host, bacteria and phages. Nature Reviews Microbiology. 15 (7):397. (2017)
- Breitbart M, Bonnain C, Malki K, Sawaya NA. Phage puppet masters of the marine microbial realm. Nature Microbiology. 3 (7):754-766. (2018)
- Leung CY, Weitz JS. Modeling the synergistic elimination of bacteria by phage and the innate immune system. Journal of Theoretical Biology. 429:241-252. (2017)
- Dulbecco R. Production of Plaques in Monolayer Tissue Cultures by Single Particles of an Animal Virus. Proceedings of the National Academy of Sciences of the United States of America. 38 (8):747-752. (1952)
- Juarez D, Long KC, Aguilar P, Kochel TJ, Halsey ES. Assessment of plaque assay methods for alphaviruses. J Virol Methods. 187 (1):185-9. (2013)
- Clokie MRJ, Millard AD, Letarov AV, Heaphy S. 2011. Phages in nature. Bacteriophage. 1 (1):31-45. (2011)
- Moldovan R, Chapman-McQuiston E, Wu XL. On kinetics of phage adsorption. Biophys J. 93 (1):303-15. (2007)
- Garneau JE, Dupuis M-È, Villion M, Romero DA, Barrangou R, Boyaval P, Fremaux C, Horvath P, Magadán AH, Moineau S.. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320):67. (2010)
Tags
Passer à...
Vidéos de cette collection:

Now Playing
Test de la plaque : méthode de détermination de la charge virale exprimée en unités formant des plaques
Microbiology
185.8K Vues

Création d'une colonne de Winogradsky : une méthode pour enrichir les espèces microbiennes dans un échantillon de sédiments.
Microbiology
128.7K Vues

Dilutions en série et ensemencement des géloses : numération des micro-organismes
Microbiology
314.8K Vues

Cultures enrichies : Cultiver des micro-organismes aérobies et anaérobies dans des milieux sélectifs et différentiels
Microbiology
131.9K Vues

Cultures pures et ensemencement des géloses : isolement des colonies bactériennes pures à partir d'un échantillon mixte
Microbiology
165.9K Vues

Séquençage de l'ARNr 16S : une technique basée sur la PCR pour l'identification d'espèces bactériennes
Microbiology
188.2K Vues

Courbes de croissance : Générer des courbes de croissance en comptant les unités formant colonies (UFC) et en mesurant l'absorbance
Microbiology
294.1K Vues

Tests de sensibilité aux antibiotiques : Utilisation du ETEST pour déterminer la CMI de deux antibiotiques et évaluer la synergie des antibiotiques
Microbiology
93.6K Vues

Microscopie et coloration : Gram, Capsule et endospores.
Microbiology
362.7K Vues

Transformation des cellules E. coli en utilisant le chlorure de calcium
Microbiology
86.4K Vues

Conjugaison : méthode de transfert de la résistance à l'ampicilline du donneur à l'hôte E. coli
Microbiology
38.1K Vues

Transduction via bactériophage : méthode de transfert de la résistance à l'ampicilline du donneur au receveur E. coli
Microbiology
29.0K Vues