Ensayo de placa: Un método para determinar los títulos virales como unidades formadoras de placas (UFP)
Visión general
Fuente: Tilde Andersson1, Rolf Lood1
1 Departamento de Ciencias Clínicas Lund, División de Medicina de Infecciones, Centro Biomédico, Universidad de Lund, 221 00 Lund, Suecia
Los virus que infectan los organismos procaloicos, llamados bacteriófagos o simplemente fagos, fueron identificados a principiosdel siglo XX por Twort (1) y d'Hérelle (2) de forma independiente. Desde entonces, los fagos han sido ampliamente reconocidos por su valor terapéutico (3) y su influencia en los ecosistemas humanos (4), así como en los ecosistemas globales (5). Las preocupaciones actuales han impulsado un renovado interés en el uso de fagos como alternativa a los antibióticos modernos en el tratamiento de enfermedades infecciosas (6). Esencialmente toda la investigación de fagose se basa en la capacidad de purificar y cuantificar virus, también conocido como un titer viral. Descrito inicialmente en 1952, este fue el propósito del ensayo de placa (7). Décadas y múltiples avances tecnológicos más tarde, el ensayo de placa sigue siendo uno de los métodos más fiables para la determinación del titer viral (8).
Los bacteriófagos subsisten inyectando su material genético en las células huésped, secuestrando las máquinas para la producción de nuevas partículas de fago, y eventualmente causando que el huésped libere numerosos viriones de progenie a través de la lisis celular. Debido a su tamaño minúsmico, los bacteriófagos no se pueden observar utilizando únicamente microscopía de luz; por lo tanto, se requiere microscopía electrónica de barrido (Figura 1). Además, los fagos no se pueden cultivar en placas de agar nutricionales como bacterias, ya que necesitan células huésped para abeste.

Figura 1: La morfología de un bacteriófago, aquí ejemplificada por un fago E. coli, se puede estudiar mediante microscopía electrónica de barrido. La mayoría de los bacteriófagos pertenecen a Caudovirales (bacteriófagos de cola). Este fago en particular tiene una estructura de cola muy corta y una cabeza icosahedral, colocándola en la familia de Podovirus.
El ensayo de placa (Figura 2) se basa en la incorporación de células huésped, preferentemente en el crecimiento de la fase log, en el medio. Esto crea una capa densa y turbia de bacterias capaces de mantener el crecimiento viral. Un fago aislado puede infectar, replicar y oxidar una célula. Con cada célula lysed, varias de las adyacentes se infectan inmediatamente. Varios ciclos en, una zona clara (una placa) se pueden observar en la placa de otro modo turbia (Figura 2B/Figura 3A),lo que indica la presencia de lo que inicialmente era una sola partícula de bacteriófago. Por lo tanto, el número de unidades formadoras de placas por volumen(es decir, PFU/ml) de una muestra puede determinarse a partir del número de placas generadas.
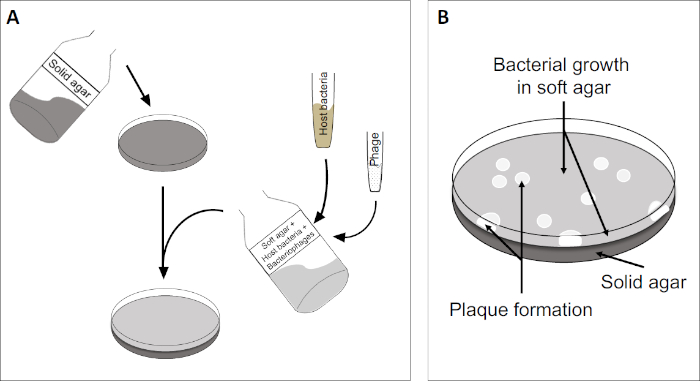
Figura 2: Las pruebas para las unidades formadoras de placas (PFU) son un método común para determinar el número de bacteriófagos en una muestra. (A) La base de una placa estéril de Petri está cubierta con un medio de nutrientes sólido adecuado, seguido de una mezcla de medios blandos, células huésped susceptibles y una dilución de la muestra original de bacteriófagos. Tenga en cuenta que la suspensión del fago podría, en algunos casos, también extenderse uniformemente a través de la superficie del agar blando ya solidificado. (B) El crecimiento de las bacterias anfitrionas forma un césped de células en la capa superior de agar. La replicación de bacteriófagos genera zonas claras, o placas, causadas por la lisis de la célula huésped.
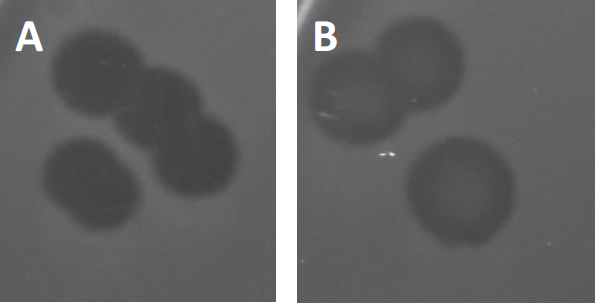
Figura 3: Los resultados de las pruebas de PFU muestran múltiples placas generadas por bacteriófagos. Debido a la lisis de las células huésped susceptibles, las placas pueden ser vistas como zonas de despeje en el césped bacteriano, ya sea con (A) aclaramiento completo, o (B) re-crecimiento parcial causado por la generación de bacterias resistentes (o posiblemente por fagos templados en el lisogénico).
Ciertos fagos templados pueden adoptar lo que se conoce como un ciclo de vida lisogénico, además del crecimiento lítico descrito anteriormente. En la lisógena, el virus asume un estado latente mediante la incorporación de su material genético en el genoma de la célula huésped (9), a menudo confiriendo resistencia a más infecciones por fagos. Esto a veces se revela a través de una ligera opacidad de la placa (Figura 3B). Vale la pena señalar sin embargo, que las placas también pueden aparecer borrosas debido al re-crecimiento de bacterias que han evolucionado resistencia al fago independiente de las infecciones anteriores de fago.
Los virus pueden adjuntarse, o adsorgar, a sólo un rango limitado de bacterias anfitrionas (10). Los rangos de acogida están aún más limitados por estrategias antivirales intracelulares como el sistema CRISPR-Cas (11). La resistencia/sensibilidad hacia fagos específicos mostrados por subgrupos bacterianos se ha utilizado históricamente para clasificar las cepas bacterianas en diferentes tipos de fagos (Figura 4). Aunque la eficacia de este método ha sido ahora superada por nuevas técnicas de secuenciación, la tipificación de fagos todavía puede proporcionar información valiosa sobre las interacciones entre bacterias y fagos, por ejemplo, facilitando el diseño de un cóctel de fago para uso clínico .

Figura 4: Sensibilidad del fago de diferentes cepas bacterianas. Las placas de agar blandas con cepa Cutibacterium acnes (A) AD27 y (B) AD35, fueron manchadas con 21 bacteriófagos Diferentes de C. acnes. Sólo el fago 11 fue capaz de infectar y matar AD27 mientras que la cepa AD35 mostró sensibilidad hacia todos los fagos. Esta técnica, que se denomina tipificación de fagos, se puede utilizar para dividir especies bacterianas y cepas en diferentes subgrupos basados en la susceptibilidad del fago.
Procedimiento
1. Configuración
- Antes de empezar cualquier trabajo que involucre microbios, asegúrese de que el espacio de trabajo esté esterilizado(por ejemplo, limpiado con 70% de etanol). Use siempre una capa de laboratorio y guantes, mantenga el cabello largo atado hacia atrás y asegúrese de que las heridas estén particularmente bien protegidas.
- Cuando haya terminado, esterilizar todas las superficies y lavar/esterilizar a fondo las manos y las muñecas.
2. Protocolo
- Preparación de LB Media
Nota: Dependiendo de la cepa bacteriana del huésped y del bacteriófago, un medio líquido diferente puede ser más adecuado para el cultivo inicial de la cepa bacteriana huésped o un medio sólido diferente puede ser más adecuado para el crecimiento posterior. El caldo de lysabagenia (LB) se utiliza en este protocolo para el caldo y el agar.- Mezclar 4 g LB en 200 ml de agua destilada, en triplicado, para el caldo LB, el agar inferior sólido y el agar superior suave. Todas las soluciones deben prepararse en recipientes capaces de contener el doble del volumen final para evitar el desbordamiento durante el autoclave.
Nota: Si realiza el ensayo en triplicado, prepare el doble de agar inferior LB. - Ajuste el pH de las tres soluciones a 7.4 utilizando NaOH o HCl según corresponda.
- Añadir 3 g de potencia de agar a la botella de agar inferior para hacer una solución de agar del 1,5% para el agar sólido
- Añadir 1,2 g de potencia de agar a la botella de agar superior para hacer una solución de agar 0.6% para agar suave.
- Colocar las botellas, con tapas semiapretadas, en un autoclave ajustado a 121oC durante 20 min para esterilizar las soluciones. Cierre las tapas tan pronto como finalice la carrera para evitar la contaminación.
- Cuando el medio LB haya alcanzado una temperatura de aproximadamente 45-50 oC, agregue 450 ml de 1 M CaCl2 estéril a las tres soluciones LB de 200 ml para realizar una concentración final de 2,25 mM.
- Vierta las alícuotas de 15 ml del medio de agar sólido en platos estériles de Petri (evitar agitar para evitar la formación de espuma) y deje que el agar se solidifique durante unas horas o durante la noche a temperatura ambiente(Figura 4A). Las placas se pueden almacenar boca abajo a 4oC durante varios días.
- Mezclar 4 g LB en 200 ml de agua destilada, en triplicado, para el caldo LB, el agar inferior sólido y el agar superior suave. Todas las soluciones deben prepararse en recipientes capaces de contener el doble del volumen final para evitar el desbordamiento durante el autoclave.
- Cultivo de células huésped
- Un día antes del ensayo, añadir 10 ml de cultivo de E. coli a 10 ml de caldo LB.
- Incubar las bacterias a 37oC durante la noche a 160 rpm en una incubadora de temblores.
- La mañana del ensayo, añadir 0,5 ml del cultivo nocturno a 10 ml de LB fresco.
Nota: Si usted está realizando el ensayo en triplicado, preparar el doble de la cantidad de esta cultura. - Incubar a 37oC a 160 rpm en una incubadora de agitación hasta que el cultivo bacteriano se encuentre en crecimiento en fase log. Esto se puede determinar espectrofotométricamente por un OD600 de 0.5-0.7.
- Mantenga el cultivo a temperatura ambiente hasta que las bacterias se añadan al agar LB superior.
- 10 veces dilución serial de bacteriófagos
- Añadir 180 l de caldo LB a siete pocillos en la primera fila de a a un plato de 96 pocillos
Nota: Se sugiere realizar la dilución en triplicado con el fin de aumentar su fiabilidad estadística. Para ello, preparar diluciones adicionales del bacteriófago en la segunda y tercera filas de la placa. - Reórse cuidadosamente la muestra original del bacteriófago para asegurar la homogeneidad y transferir 20 ml al primer pozo.
- Mezcle bien la muestra pipeteando y hacia abajo.
- Transfiera 20 s de la suspensión resultante al segundo pozo.
- Continúe la dilución en serie transfiriendo 20 l de la solución en el segundo pozo al tercer pozo, y así sucesivamente, hasta el sexto pozo, dejando el séptimo así como un control negativo al que no se añadirá ninguna suspensión de fago. Esto creará un rango de dilución de 10-1-10-6.
- Añadir 180 l de caldo LB a siete pocillos en la primera fila de a a un plato de 96 pocillos
- Galjanoplastia
- Etiquetar la base de los platos de Petri (previamente preparados en el paso 2.1.7) con nombre, fecha y una breve descripción de la muestra (incluido el factor de dilución de la muestra de fago). Precalienta los platos de Petri en una incubadora de 37oC una hora antes del ensayo.
- Derretir el agar-medio solidificado (típicamente hecho usando un microondas; el agar solidificado se derrite a 85 oC), y dejar que llegue a alrededor de 45 oC. El medio calentado se puede colocar en un baño de agua de 45oC durante 1 h para alcanzar una temperatura adecuada. Debe estar lo suficientemente caliente como para permanecer en forma líquida, pero lo suficientemente fresco como para no matar bacterias añadidas.
- Mezclar el cultivo bacteriano de 4 ml (del paso 2.2) con el agar blando de 35 ml LB (a 45oC). Gire para distribuir uniformemente las células, pero evite agitar para evitar la formación de espuma(Figura 4A).
- Etiquetar un tubo de ensayo estéril para cada uno de los pasos de dilución en serie y otro para la muestra de control, para un total de 7 tubos de ensayo etiquetados. Coloque 5 alícuotas ml del cultivo bacteriano/mezcla de agar suave del paso 2.4.3 en los 7 tubos. Trabaje rápidamente a través del paso 2.4.6 porque estos pequeños volúmenes de medios a base de agar se solidificarán rápidamente a temperatura ambiente.
- Añadir 100 l de la muestra de control (del paso 2.3) al tubo de ensayo de control y girar con cuidado. Deseche la punta de pipeta usada y transfiera el mismo volumen de cada una de las muestras de bacteriófagos diluidos en serie (paso 2.3) a sus respectivos tubos de ensayo, girando para mezclar.
- Transfiera inmediatamente las mezclas de 5 ml a las placas de agar sólidas etiquetadas, precalentadas(Figura 4A). Gire suavemente las placas para extender las mezclas.
Nota: Si está realizando el ensayo por triplicado, repita los pasos 2.4.3-6 dos veces más. - Sellar cada placa con película de laboratorio, y permitir que ambas capas se solidifiquen correctamente a temperatura ambiente (aproximadamente 15 minutos) antes de colocarlas de lado abajo una incubadora de 37oC, estimulando el crecimiento tanto de las bacterias como del fago, durante 24 horas o hasta que las placas desarrollar, Por lo general toma alrededor de 1-5 días para que aparezcan las placas(Figura 4B),pero el momento varía considerablemente dependiendo de las condiciones de incubación, medio, y las especies bacterianas.
3. Análisis de datos y resultados
-
Contar placas
- Asegúrese de que no haya placas visibles en las placas marcadas como "control", ya que esto indicaría contaminación viral.
- Comience con las placas etiquetadas 10-6, que contengan la muestra de bacteriófago más diluida. Cuente las placas sin quitar la tapa, marcándolas con un bolígrafo a medida que vaya para indicar qué placas ya se han contado.
- Cuente las placas restantes. Algunas placas pueden tener muy pocas o demasiadas placas para contar. Utilice las placas con 10-150 placas para su posterior análisis.
-
Cálculo de PFU
- Divida el número de placas por el factor de dilución(porejemplo, 10-6 para la muestra más diluida) para obtener el número de unidades formadoras de placas (PFU) en 100 ml de mezcla de fago.
Nota: Si realiza el ensayo en triplicado, utilice el número medio de placas de las tres placas. - Para determinar la concentración (en PFU/ml) de la muestra original, multiplique por un factor de dilución adicional de 10, ya que sólo se celado 100 ml de la muestra. (es
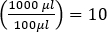 decir)
decir) - Calcular el valor medio de la PFU/ml para todas las diluciones que tenían entre 10 y 150 placas para lograr un resultado más confiable.
- Divida el número de placas por el factor de dilución(porejemplo, 10-6 para la muestra más diluida) para obtener el número de unidades formadoras de placas (PFU) en 100 ml de mezcla de fago.
Aplicación y resumen
A pesar de los múltiples avances tecnológicos, los ensayos de placas siguen siendo el estándar de oro para la determinación del titer viral (como PFU) y esenciales para el aislamiento de las poblaciones de bacteriófagos puros. Las células huésped susceptibles se cultivan en la capa superior de una placa de agar de dos capas, formando un lecho homogéneo que permite la replicación viral. El evento inicial donde un bacteriófago aislado en el ciclo de vida lítico infecta una célula, se replica dentro de ella, y finalmente la lyses, es demasiado pequeño para observar. Sin embargo, los viriones liberados infectarán las células adyacentes, dando lugar posteriormente a una zona de limpieza, o placa, que denota la presencia de una sola PFU.
Referencias
- Twort, F. An investigation on the nature of ultra-microscopic viruses. Lancet. 186 (4814): 1241-1243. (1915)
- d'Hérelle, F. An invisible antagonist microbe of dysentery bacillus. Comptes Rendus Hebdomadaires Des Seances De L Academie Des Sciences. 165: 373-375. (1917)
- Cisek AA, Dąbrowska I, Gregorczyk KP, Wyżewski Z. Phage Therapy in Bacterial Infections Treatment: One Hundred Years After the Discovery of Bacteriophages. Current Microbiology. 74 (2):277-283. (2017)
- Mirzaei MK, Maurice CF. Ménage à trois in the human gut: interactions between host, bacteria and phages. Nature Reviews Microbiology. 15 (7):397. (2017)
- Breitbart M, Bonnain C, Malki K, Sawaya NA. Phage puppet masters of the marine microbial realm. Nature Microbiology. 3 (7):754-766. (2018)
- Leung CY, Weitz JS. Modeling the synergistic elimination of bacteria by phage and the innate immune system. Journal of Theoretical Biology. 429:241-252. (2017)
- Dulbecco R. Production of Plaques in Monolayer Tissue Cultures by Single Particles of an Animal Virus. Proceedings of the National Academy of Sciences of the United States of America. 38 (8):747-752. (1952)
- Juarez D, Long KC, Aguilar P, Kochel TJ, Halsey ES. Assessment of plaque assay methods for alphaviruses. J Virol Methods. 187 (1):185-9. (2013)
- Clokie MRJ, Millard AD, Letarov AV, Heaphy S. 2011. Phages in nature. Bacteriophage. 1 (1):31-45. (2011)
- Moldovan R, Chapman-McQuiston E, Wu XL. On kinetics of phage adsorption. Biophys J. 93 (1):303-15. (2007)
- Garneau JE, Dupuis M-È, Villion M, Romero DA, Barrangou R, Boyaval P, Fremaux C, Horvath P, Magadán AH, Moineau S.. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320):67. (2010)
Saltar a...
Vídeos de esta colección:

Now Playing
Ensayo de placa: Un método para determinar los títulos virales como unidades formadoras de placas (UFP)
Microbiology
185.8K Vistas

¿Qué son los virus?
Microbiology
111.0K Vistas

La estructura viral
Microbiology
60.8K Vistas

El ciclo lítico de los bacteriófagos
Microbiology
69.7K Vistas

El ciclo Lisogénico de Bacteriófagos
Microbiology
61.3K Vistas

Los Ciclos de vida del retrovirus
Microbiology
45.3K Vistas

La recombinación viral
Microbiology
23.1K Vistas

Las mutaciones virales
Microbiology
32.0K Vistas

Infección
Microbiology
6.2K Vistas

Respuesta inmunitaria frente a patógenos virales
Microbiology
439 Vistas

Ensayo de placa: Un método para determinar los títulos virales como unidades formadoras de placas (UFP)
Microbiology
185.8K Vistas
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados