Research Article
Identification de gènes liés à la ferroptose et à l’hypoxie dans des cellules précurseurs d’oligodendrocytes dérivées d’iPSC de patients atteints de sclérose en plaques
Dans cet article
Résumé
Cette étude fournit de nouvelles perspectives sur les interactions entre l’hypoxie, la ferroptose et l’infiltration immunitaire dans la pathogenèse de la sclérose en plaques (SEP) grâce à l’analyse bioinformatique. En utilisant l’analyse de réseau de coexpression génique pondérée (WGCNA) et l’analyse d’interaction protéine-protéine (IPP), nous avons identifié trois gènes pivots (ITGB1, ITGB8 et VIM).
Résumé
La sclérose en plaques (SEP) est une maladie inflammatoire chronique caractérisée par une démyélinisation, l’échec de la remyélinisation entraînant une perte progressive des axones aux stades chroniques. Les cellules précurseurs des oligodendrocytes (OPC) sont essentielles à la remyélinisation. Des études récentes suggèrent que l’hypoxie et la ferroptose jouent toutes deux un rôle crucial dans la différenciation dysfonctionnelle des OPC. Cette recherche vise à identifier les gènes clés liés à l’hypoxie et à la ferroptose et les caractéristiques d’infiltration immunitaire dans les OPC dérivés de cellules souches pluripotentes induites (iPSC) de patients atteints de SEP et à construire un modèle diagnostique centré sur ces gènes pivots.
Nous avons analysé les données d’expression génique des ensembles de données GSE196575 et GSE147315 et comparé des patients atteints de SEP à des individus en bonne santé. À l’aide de l’analyse du réseau de coexpression génique pondérée (WGCNA), nous avons identifié les gènes du module primaire et les gènes essentiels associés à l’hypoxie, à la ferroptose et à la SEP. Le score Z de la ferroptose et le score Z de l’hypoxie calculés par l’analyse de la variation de l’ensemble des gènes (GSVA) étaient plus élevés dans les OPC dérivés de l’iPSC des patients atteints de SEP que dans ceux du groupe témoin. Les gènes impliqués sont principalement liés à la voie PI3K/Akt/mTOR, telle qu’identifiée par les analyses d’enrichissement de la voie Gene Ontology (GO) et Kyoto Encyclopedia of Genes and Genomes (KEGG).
Un réseau d’interaction protéine-protéine (IPP) de gènes cruciaux a révélé 10 gènes centraux (COL4A1, COL4A2, ITGB5, ITGB1, ITGB8, ITGAV, VIM, FLNA, VCL et SPARC). L’expression robuste d’ITGB1, d’ITGB8 et de VIM a été validée dans l’ensemble de données GSE151306, soutenant leur rôle de gènes clés du hub. De plus, un réseau d’interaction entre les facteurs de transcription (TF) et les gènes pivots a été établi via TRRUST (Transcriptional Regulatory Relationships Unraveled by Sentence-based Text), qui a identifié cinq TF clés. Les résultats de cette étude pourraient aider à élucider de nouveaux biomarqueurs ou cibles thérapeutiques pour la SP.
Introduction
La sclérose en plaques (SEP) est une maladie inflammatoire chronique caractérisée par une démyélinisation qui touche environ 2,5 millions de personnes dans le monde. La majorité des personnes diagnostiquées avec la SEP présentent une évolution cyclique de la maladie. Au cours de la phase récurrente, l’inflammation aiguë entraîne la perte inévitable de myéline et d’axones. À l’inverse, pendant la rémission, les lésions de démyélinisation peuvent être réparées par la remyélinisation, fournissant un soutien trophique aux axones et empêchant la perte progressive d’axones1. L’échec de la remyélinisation survient aux stades chroniques de la SP et entraîne une dégénérescence axonale progressive2.
Le processus de remyélinisation est fortement corrélé avec les cellules précurseurs d’oligodendrocytes (OPC), impliquant la prolifération et la migration des OPC pour se différencier en oligodendrocytes (OL) matures, qui sont les cellules formant la myéline dans le système nerveux central (SNC)3. Dans les premiers stades de la maladie, le nombre de nouveaux OL générés par les OPC autour des lésions démyélinisées est relativement préservé et peut favoriser avec succès la remyélinisation4. Cependant, au cours des stades avancés de la SEP, la migration et la différenciation inadéquates des OPC entraînent une réduction des nouvelles OL et une altération de la remyélinisation5, entraînant ainsi une dégénérescence nerveuse et une accumulation d’invalidité.
Deux hypothèses ont été proposées pour expliquer la neurodégénérescence dans la SP. L’hypothèse extrinsèque suggère que la réponse immunitaire initiée par les lymphocytes T activés provoque une démyélinisation ainsi qu’une neurodégénérescence6. Le modèle intrinsèque, cependant, suggère que les anomalies intrinsèques dans les OPC7, les OL8 et d’autres cellules du SNC peuvent contribuer à la neurodégénérescence. Auparavant, on considérait que le modèle intrinsèque ne s’appliquait qu’aux stades plus avancés de la SP, comme la SP progressive primaire ou secondaire (SPPP et SPPS). Néanmoins, une neurodégénérescence indépendante de l’inflammation ou de la rechute a récemment été observée dans la SEP-RR 9,10, ce qui suggère que des anomalies cellulaires intrinsèques peuvent être impliquées à tous les stades de la maladie, y compris la SEP-RR.
De plus, la ferroptose, une voie de mort cellulaire distinctive liée à des perturbations métaboliques lipidiques médiées par le fer, joue un rôle central dans la neurodégénérescence. Cette voie implique un déséquilibre des états redox intracellulaires induit par un excès de fer, conduisant à l’accumulation de peroxyde lipidique et à la production d’espèces réactives de l’oxygène (ROS), entraînant finalement la mort cellulaire oxydative11. Les maladies neurodégénératives telles que la maladie d’Alzheimer, la maladie de Parkinson et la maladie de Huntington proviennent souvent de dommages oxydatifs aux cellules neuronales, qui sont fréquemment déclenchés par des concentrations de fer inhabituellement élevées dans les lésions. Dans le cas de la SP, la vulnérabilité accrue aux dommages oxydatifs, combinée à un dysfonctionnement mitochondrial dû à la teneur élevée en lipides et à la consommation d’oxygène des cellules du SNC, favorise la peroxydation des lipides, un facteur critique de la ferroptose. Les OL sont sensibles à la peroxydation lipidique, une caractéristique essentielle de la ferroptose12. Le dépôt de fer près des lésions inflammatoires de la colonne vertébrale13 et la vulnérabilité des OL à la peroxydation lipidique14 et aux radicaux libres15 mettent en évidence la sensibilité de la SEP à la ferroptose.
L’hypoxie est un autre facteur critique de la pathogenèse de la SP, qui contribue à la perte d’oligodendrocytes. Les signes de lésions semblables à l’hypoxie et la production de ROS et d’oxyde d’azote (NO) dans les lésions aiguës de la SEP indiquent que ces facteurs de stress peuvent précipiter le dysfonctionnement mitochondrial et les déficits énergétiques qui en découlent16. Ce stress métabolique affecte non seulement les OL, mais altère également les axones voisins par le biais d’un transfert d’énergie perturbé17, car les canaux myéliniques transmettent l’énergie entre la gaine de myéline et les espaces péri-axonaux.
Les OPC et les OL primaires du SNC sont extrêmement difficiles d’accès chez les patients atteints de SEP. Par conséquent, les OPC et les OL humains dérivés de cellules souches pluripotentes induites (CSPi) sont apparus comme des outils prometteurs pour étudier les troubles intrinsèques de la SP. À la lumière des rôles cruciaux de la ferroptose et de l’hypoxie dans la pathogenèse de la SP et de leur impact sur la lignée des oligodendrocytes, cette étude a utilisé l’analyse pondérée des réseaux de coexpression génique (WGCNA) pour extraire des informations de module18 et pour élucider les modèles d’expression génique associés à ces phénomènes dans la SP. En examinant les coefficients de corrélation entre les gènes, nous sommes en mesure d’identifier des réseaux ou des modules de coexpression identiques ou similaires, ce qui met en lumière de nouveaux biomarqueurs ou des cibles thérapeutiques potentielles pour la SP. De plus, en mettant l’accent sur les facteurs de transcription (TF) qui régulent les gènes critiques, cette étude jette les bases d’une exploration plus approfondie des mécanismes et des stratégies d’intervention potentielles dans le domaine de la SP.
Protocole
1. Téléchargement et prétraitement des données
- Téléchargez les ensembles de données GSE196575 et GSE147315 à partir du Gene Expression Omnibus (GEO).
- Fusionnez les deux ensembles de données et supprimez les effets de lot, à l’aide d’une plateforme d’analyse en ligne (voir Tableau des matériaux).
- Choisissez le module Module d’expression | fusion de données. Téléchargez les deux fichiers de matrice d’expression en tant que fichiers d’entrée et cliquez sur Exécuter pour générer automatiquement la matrice fusionnée.
- Créez un exemple de fichier d’annotation sous forme de feuille de calcul.
- Choisissez le module Expression | Retirez le module d’effet par lots. Entrez le fichier d’annotation et la matrice fusionnée et cliquez sur Exécuter pour créer la matrice fusionnée avec l’effet de lot supprimé (matrice fusionnée 2).
- Normalisez les données d’expression à l’aide des fonctionnalités de fusion des jeux de données, d’effet de lot et de normalisation disponibles sur une plate-forme d’analyse en ligne. Choisissez le module d’expression et le module de normalisation. Entrez la matrice fusionnée 2 créée à l’étape 1.2.3. Cliquez sur le bouton Exécuter . (voir la table des matériaux).
- Générez le fichier de matrice d’expression après avoir exécuté l’ensemble du processus décrit ci-dessus. Extraire les informations de regroupement en fonction de l’intervention fournie sur le site web du GEO. Entrez les informations ci-dessus dans un document au format « .txt » pour créer le fichier d’informations de regroupement pour l’ensemble de données fusionné.
2. Analyse différentielle des gènes exprimés
- Effectuez une analyse différentielle de l’expression génique à l’aide du package limma dans R. Entrez le fichier de matrice d’expression et le fichier d’informations de regroupement. Définissez les critères de filtrage sur |logFC| = 1 et p < 0,05 pour identifier les gènes exprimés différentiellement (DEG) liés à la sclérose en plaques.
3. Analyse d’enrichissement fonctionnel (GO et KEGG)
- Effectuer une analyse d’enrichissement fonctionnel pour les DEG via les modules GO et KEGG sur des plateformes en ligne.
- L’ensemble de l’organisme modèle a été défini sur Homo sapiens, avec la version Ensemble_109 comme fichier de gènes de base et les gènes comme type de données.
- Évitez les termes généraux de GO ; affiner l’analyse à l’aide de ToppFun. Appliquez des filtres pour limiter les termes à 500 et 1 000 gènes associés.
REMARQUE : Les résultats du filtre de 500 gènes sont présentés dans le manuscrit principal. Les résultats complets de GO sont disponibles dans la figure supplémentaire S2. La plateforme en ligne KEGG est illustrée à la figure supplémentaire S3.
4. Analyse de la variation des ensembles de gènes pour la ferroptose et l’hypoxie (GSVA)
- Récupérez un total de 538 gènes liés à la ferroptose à partir de FerrDb et 200 gènes liés à l’hypoxie à partir de MsigDB.
- Créez des fichiers au format GMT pour les deux ensembles de gènes.
- Utilisez le module GSVA pour calculer le score Z de la ferroptose et le score Z de l’hypoxie pour chaque échantillon de l’ensemble de données fusionné, et utilisez le fichier de matrice d’expression, le fichier d’informations de regroupement et les fichiers GMT comme entrées.
- Comparez les scores Z de différents groupes (figure supplémentaire S4).
5. Analyse pondérée du réseau de co-expression génique (WGCNA)
- Extrayez le score Z de ferroptose et d’hypoxie de chaque échantillon calculé à l’étape 4.4. Utilisez les scores Z ci-dessus de la ferroptose et de l’hypoxie comme fichiers de traits à côté des fichiers de matrice d’expression pour identifier les modules de gènes clés liés à la ferroptose et à l’hypoxie.
- Utilisez le module WGCNA et définissez R2 = 0,9 et un seuil doux β = 18 pour construire un réseau pondéré de coexpression génique. Focus sur les modules significativement associés à la ferroptose et à l’hypoxie (Figure supplémentaire S4).
6. Identification des gènes exprimés de manière différentielle liés à la ferroptose et à l’hypoxie chez les patients atteints de SEP
- Intersection des DEG liés à la maladie avec les modules de gènes liés à la ferroptose et à l’hypoxie à l’aide d’un outil de diagramme de Venn sur une plateforme d’analyse en ligne (figure supplémentaire S3). Identifier les gènes associés à la ferroptose et à l’hypoxie dans la SP.
7. Analyse du réseau d’interaction protéine-protéine (IPP)
- Entrez les gènes intersectés dans le module Multiple Proteins de la base de données STRING.
- Définissez l’organisme sur Homo sapiens et cliquez sur Rechercher.
- Utilisez l’option CONTINUER au centre de la page Web pour générer le réseau PPI.
- Exporter les informations du réseau d’interaction au format TSV et les importer dans un logiciel d’analyse de réseau bio-informatique (figure supplémentaire S5).
- Utilisez le plugin Cytohubba pour identifier les 10 principaux gènes pivots sur la base de l’algorithme MCC.
8. Validation avec le jeu de données GSE151306
- Téléchargez le jeu de données GSE151306 à partir de GEO.
- Utilisez le package limma dans le logiciel R studio (les codes de langage R sont fournis dans GitHub : https://github.com/Drxiazhang/Identification-of-Ferroptosis--and-Hypoxia-Related-Genes-in-iPSC-Derived-OPCs-from-MS) pour valider l’expression des gènes hub dans différents groupes, et entrez le fichier de matrice d’expression, le fichier de regroupement de l’ensemble de validation et la liste des gènes hub.
9. Tracé de la courbe ROC pour les gènes de moyeu
- Utilisez le package pROC dans R pour tracer des courbes ROC pour les gènes de hub exprimés de manière différentielle.
- Validez la valeur de diagnostic de ces gènes de hub dans le jeu de données de validation (les codes de langue R sont fournis dans GitHub : https://github.com/Drxiazhang/Identification-of-Ferroptosis--and-Hypoxia-Related-Genes-in-iPSC-Derived-OPCs-from-MS).
10. Prédiction des réseaux de régulation des gènes du facteur de transcription
- Accédez à la base de données TRRUST v2 à l’https://www.grnpedia.org/trrust/.
- Utilisez le module Trouver des régulateurs clés pour interroger les gènes pour identifier les facteurs de transcription régulant les 10 gènes pivots.
- Définissez Humain pour l’espèce. Entrez les facteurs de transcription et les gènes centraux dans le module Multiple Proteins de STRING, définissez Homo sapiens pour les organismes et cliquez sur CONTINUER pour générer un réseau de régulation.
Résultats
L’ensemble de données fusionné, composé de quatre personnes en bonne santé comme témoins et de neuf personnes atteintes de SP (SP-TP), a été analysé, puis validé dans un autre ensemble de données composé de quatre témoins atteints de SP et de quatre témoins sains. Le protocole d’analyse est illustré à la figure 1, et les informations détaillées de tous les échantillons sont énumérées dans le tableau supplémentaire S1. L’analyse a permis d’identifier 706 gènes exprimés différentiellement (DEG, p < 0,01), dont 378 gènes étaient régulés à la hausse et 328 gènes étaient régulés à la baisse chez les patients atteints de SEP par rapport aux témoins (Figure 2). L’analyse de l’enrichissement fonctionnel a révélé l’implication significative de ces DEG dans divers processus biologiques. Notamment, les termes GO enrichis les plus affinés par 500 gènes comprenaient l’adhésion focale, la jonction cellule-substrat, la voie de signalisation médiée par l’intégrine, la liaison à l’intégrine et la liaison à la cadhérine (Figure 3A-C). L’analyse de la voie KEGG a également révélé que ces gènes sont principalement impliqués dans la voie de signalisation PI3K-Akt et la voie de signalisation Hippo (figure 3D), qui sont essentielles aux processus cellulaires pertinents à la pathogenèse de la SP.
Les scores Z de la ferroptose et de l’hypoxie, calculés par GSVA, étaient significativement plus élevés chez les cellules précurseurs d’oligodendrocytes (CPO) dérivées d’iPSC chez des patients atteints de SEP que chez celles de témoins sains (p = 0,0028 et 0,075, respectivement, figure 4), ce qui indique le rôle central de ces voies dans cette maladie. Cette constatation a été appuyée par la WGCNA, qui a identifié deux modules génétiques clés (figure 5A). Parmi ceux-ci, le module turquoise, comprenant 3 671 gènes, était significativement associé à la ferroptose et à l’hypoxie (Figure 5B). Il est important de noter que 71 gènes ont été identifiés à l’intersection des gènes liés à la maladie exprimés de manière différentielle et de ceux du module WGCNA, ce qui suggère une forte corrélation avec la ferroptose (bêta = 0,94, p = 2e-06) et l’hypoxie (bêta = 0,92, p = 7e-06) dans la SEP (Figure 6).
Une enquête plus approfondie sur le réseau PPI a révélé 10 gènes centraux, à savoir COL4A1, COL4A2, ITGB5, ITGB1, ITGB8, ITGAV, VIM, FLNA, VCL et SPARC (Figure 7). Ces gènes pivots ont été notés pour leur rôle crucial dans la médiation des processus cellulaires liés à la ferroptose et à l’hypoxie.
Nous avons ensuite validé l’ensemble de données de validation (GSE151306). Les résultats suggèrent que trois gènes pivots (ITGAV, ITGB8 et VIM) étaient régulés à la hausse dans le PwMS (figure 8), ce qui était cohérent avec les résultats de l’ensemble de données fusionnées (figure supplémentaire S1). Parmi eux, ITGB8 était le gène le plus significativement régulé à la hausse. Ces résultats suggèrent que l’ITGB8, un membre de la famille des intégrines, pourrait jouer un rôle critique dans la pathogenèse de la SP, particulièrement en ce qui a trait aux mécanismes de ferroptose et d’hypoxie. De plus, l’analyse de la courbe ROC (Receiver Operating Characteristic) a confirmé que l’ITGAV, l’ITGB8 et le VIM ont une bonne sensibilité et spécificité pour le diagnostic de la maladie (AUC = 58,3 %, 100 % et 91,7 %, respectivement ; Figure 9A-C), ITGB8 présentant une ASC de 100 %, démontrant son potentiel en tant que biomarqueur robuste de la SEP.
Enfin, un réseau de régulation du gène pivot du facteur de transcription a été établi, et trois facteurs de transcription clés – HOXD3, SP1 et VHL – ont été identifiés comme régulateurs des gènes pivots (Figure 10). Ces facteurs de transcription peuvent jouer un rôle essentiel dans la régulation de l’expression des gènes en réponse à l’hypoxie et à la ferroptose, ce qui met en évidence leur implication potentielle dans la progression de la SP.
Vidéo 1 : Vue coronale obtenue avec un transducteur basse fréquence centré sur l’hémidiaphragme droit montrant une excursion diaphragmatique qualitativement normale pendant la respiration de la capacité vitale. Veuillez cliquer ici pour télécharger cette vidéo.
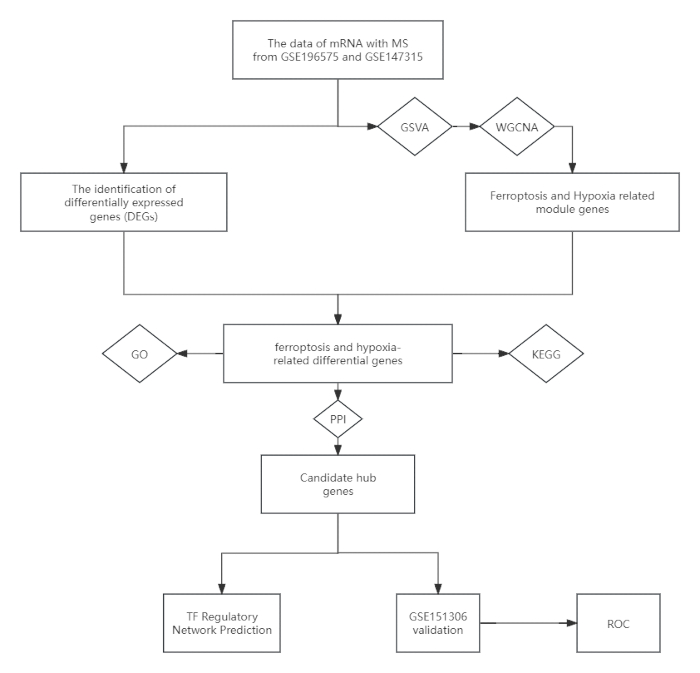
Figure 1 : Organigramme montrant la conception de l’étude. Abréviations : SP = sclérose en plaques ; GSVA = Analyse de la variation de l’ensemble des gènes ; WGCNA = analyse pondérée du réseau de co-expression génique ; KEGG = Encyclopédie de Kyoto des gènes et des génomes ; GO = Ontologie des gènes ; IPP = Interaction Protéine-Protéine ; ROC = Receiver Operating Characteristic. Veuillez cliquer ici pour voir une version agrandie de cette figure.
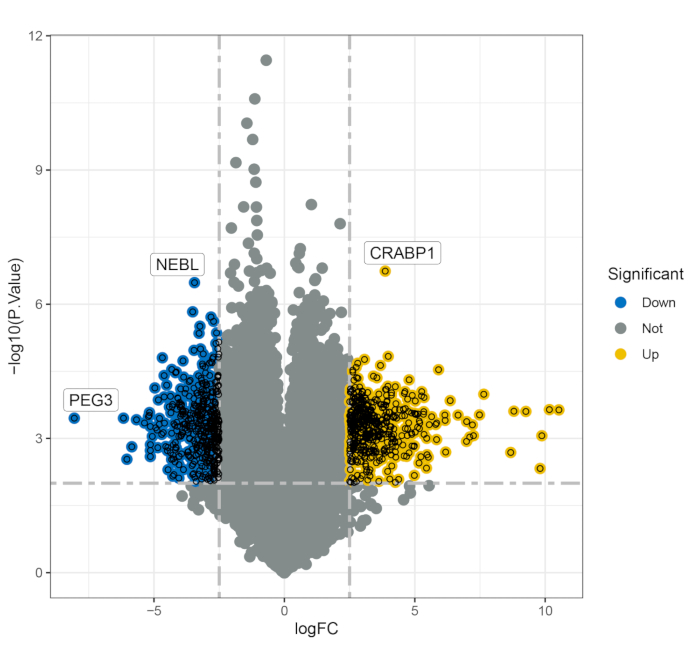
Figure 2 : Graphique du volcan illustrant les gènes exprimés de manière différentielle entre le groupe témoin sain et le groupe MS. Abréviations : FC = critères de filtrage ; PEG3 = gène 3 exprimé par le père ; NEBL = nébulette ; CRABP1 = protéine de liaison à l’acide rétinoïque cellulaire 1. Veuillez cliquer ici pour voir une version agrandie de cette figure.
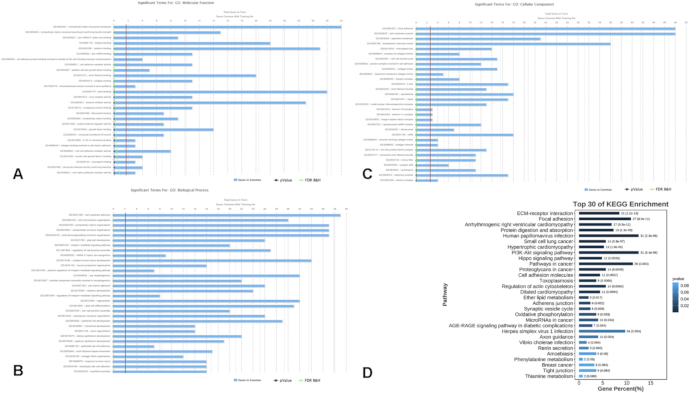
Figure 3 : Analyses d’enrichissement fonctionnel de gènes exprimés différentiellement. (A-C) Les catégories d’ontologie génétique ont été affinées en filtrant 500 gènes, notamment (A) la fonction moléculaire, (B) le processus biologique et (C) le composant cellulaire. (D) Analyse de l’enrichissement de la voie KEGG indiquant les voies enrichies parmi les DEG. Abréviations : DEG = gènes exprimés de manière différentielle ; GO = Ontologie des gènes ; MF = fonction moléculaire ; BP = processus biologique ; CC = composant cellulaire ; KEGG = Encyclopédie des gènes et des génomes de Kyoto. Veuillez cliquer ici pour voir une version agrandie de cette figure.
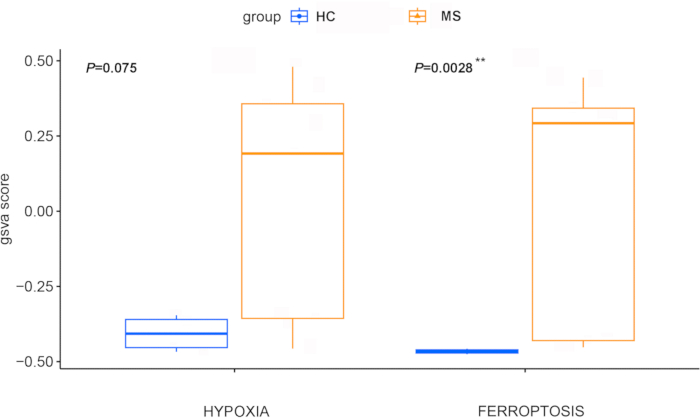
Figure 4 : Boîte à moustaches comparant les scores Z de la ferroptose et de l’hypoxie entre le groupe témoin sain et le groupe atteint de SEP. Les scores Z de la ferroptose et de l’hypoxie ont été calculés via GSVA. Le groupe de contrôle est représenté en bleu et le groupe MS est indiqué en rouge. Des comparaisons statistiques ont été effectuées via le test de Wilcoxon. ** Indique des valeurs p comprises entre 0 et 0,01. Abréviations : SP = sclérose en plaques ; HC = contrôle sain ; GSVA = Analyse de la variation des ensembles de gènes. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Résultats de l’analyse du réseau pondéré de coexpression génique. (A) Dendrogramme en grappes de gènes catégorisés en modules. (B) Corrélations entre les modules et les traits liés à l’hypoxie et à la ferroptose. Abréviation : ADJ = ajusté. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 6 : Diagramme de Venn révélant 71 gènes qui se croisent. Le diagramme illustre le chevauchement des DEG des patients atteints de sclérose en plaques avec les gènes des modules liés à l’hypoxie et à la ferroptose. Abréviations : WGCNA = analyse pondérée du réseau de co-expression génique ; DEG = gènes exprimés de manière différentielle. Veuillez cliquer ici pour voir une version agrandie de cette figure.
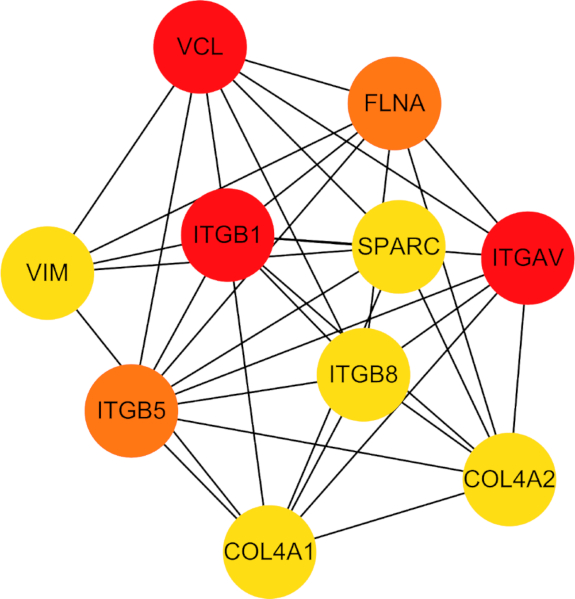
Figure 7 : Schéma de réseau des interactions protéine-protéine entre les 10 gènes pivots identifiés. Abréviations : VCL = vinculine ; FLNA = filamine A ; ITGAV = sous-unité d’intégrine Alpha V ; GOL4A2 = Chaîne alpha 2 du collagène de type IV ; COL4A1 = Chaîne alpha 1 du collagène de type IV ; ITGB5 = Sous-unité d’intégrine bêta 5 ; VIM = Vimentin ; SPARC = protéine sécrétée acide et riche en cystéine ; ITGB8 = Sous-unité d’intégrine bêta 8 ; ITGB1 = Sous-unité d’intégrine bêta 1. Veuillez cliquer ici pour voir une version agrandie de cette figure.
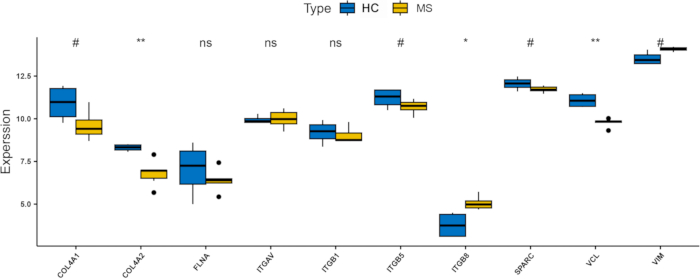
Figure 8 : Diagramme à barres montrant les niveaux d’expression des gènes pivots dans l’ensemble de données de validation (GSE151306). * indique des valeurs p comprises entre 0,01 et 0,05, et # indique des valeurs p comprises entre 0,05 et 0,2. ns indique des valeurs p comprises entre 0,2 et 1. Abréviations : SC : témoins sains ; SEP : sclérose en plaques. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 9 : Courbes ROC pour l’évaluation de la sensibilité et de la spécificité diagnostiques des gènes de moyeu exprimés différentiellement chez GSE151306. (A) Courbe ROC pour ITGAV. (B) Courbe ROC pour ITGB8. (C) Courbe ROC pour VIM. Abréviations : ROC = Receiver Operating Characteristic ; ITGAV = sous-unité d’intégrine alpha V ; ITGB8 = sous-unité d’intégrine bêta 8 ; VIM = vimentine. Veuillez cliquer ici pour voir une version agrandie de cette figure.
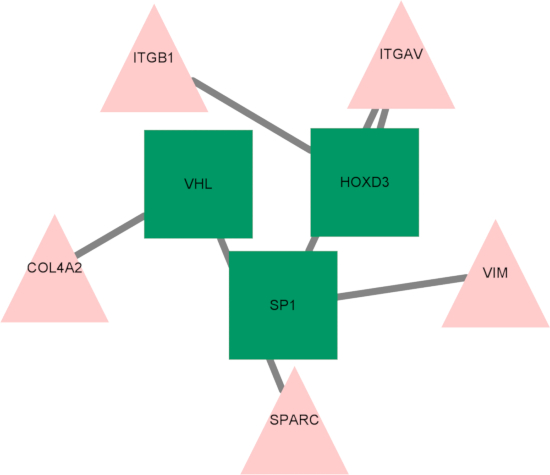
Figure 10 : Schéma du réseau de régulation du gène facteur de transcription -hub. Les triangles représentent les facteurs de transcription ; Les carrés représentent les gènes du hub. Abréviations : ITGB1 = sous-unité d’intégrine bêta 1 ; ITGAV = sous-unité d’intégrine alpha V ; VIM = Vimentin ; SPARC = protéine sécrétée, acide et riche en cystéine ; COL4A2 = chaîne alpha 2 du collagène de type IV ; HOXD3 = homéobox D3 ; VHL = suppresseur de tumeur de von Hippel-Lindau ; SP1 = Spécificité Protéine 1. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 11 : L’impact de l’intégrine β1 sur la voie PI3K-Akt. La forme d’extension avec une tête ouverte (état actif, haute affinité) intégrine β1 pourrait former une « adhésion focale » et des « complexes focaux » et interagir avec la kinase FAK et Scr. Le complexe FAK/Src actif recrute des molécules adaptatrices pour activer la voie PI3K-Akt. Cette figure a été adaptée de Yan et Cui37. Abréviations : PI3K = phosphatidylinositide-3-kinase ; FAK = kinase d’adhésion focale. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Figure supplémentaire S1 : Les tendances d’expression des gènes clés dans l’ensemble de données fusionnées. Wilcox.test du package R « limma » a été utilisée pour les tests statistiques. « * » indique des valeurs p comprises entre 0,01 et 0,05, et « # » indique des valeurs p comprises entre 0,05 et 0,2. Veuillez cliquer ici pour télécharger cette figure.
Figure supplémentaire S2 : Mise en page originale de l’analyse de l’ontologie génétique des gènes exprimés différentiellement. Veuillez cliquer ici pour télécharger cette figure.
Figure supplémentaire S3 : Page d’accueil de la plateforme d’analyse en ligne Omicshare. Ce site web est un hub bioinformatique chinois sur lequel KEGG a été réalisé. Veuillez cliquer ici pour télécharger cette figure.
Figure supplémentaire S4 : Page d’accueil de la plateforme d’analyse en ligne de Biowinford sur laquelle WGCNA a été réalisée. Veuillez cliquer ici pour télécharger cette figure.
Figure supplémentaire S5 : Page d’accueil de la base de données STRING sur laquelle l’IPP a été effectué. Veuillez cliquer ici pour télécharger cette figure.
Tableau supplémentaire S1 : Caractéristiques des échantillons et des ensembles de données regroupés et analysés. Veuillez cliquer ici pour télécharger ce tableau.
Discussion
Étant donné leur rôle central dans le processus de remyélinisation, la migration, la différenciation et la mort des COP sont considérées depuis longtemps comme des facteurs cruciaux dans la pathogenèse de la SP et ses cibles thérapeutiques. Une dégénérescence nerveuse progressive indépendante de l’inflammation a été observée dans les trois types de SP19, et une perte prononcée d’oligodendrocytes a été observée au centre des lésions démyélinisées20, ce qui suggère que les troubles primaires des cellules de la lignée des oligodendrocytes pourraient accélérer la progression de la SP.
Dans cette étude, nous avons effectué une analyse bioinformatique de séquences d’ARNm ciblées en nous concentrant sur les gènes associés à la ferroptose et à l’hypoxie dans les OPC induits par les iPSC provenant de PwMS et de témoins sains via des ensembles de données fusionnés provenant de quatre témoins sains et de neuf PwMS. Nous avons découvert 706 DEG, dont 378 étaient régulées à la hausse et 328 à la baisse. Nous avons ensuite identifié des gènes liés à la ferroptose et à l’hypoxie et comparé leur expression dans les OPC induits par les iPSC de patients atteints de SEP et de témoins sains en calculant le score Z de chaque échantillon. Les scores Z de la ferroptose et de l’hypoxie étaient significativement plus élevés chez les personnes atteintes de SP que chez les témoins sains.
Des études pathologiques ont démontré une oligodendrogliopathie distale dans les lésions de SEP de type III ressemblant à un accident vasculaire cérébral ischémique de la substance blanche16 , ainsi que des lésions de type hypoxique associées à des taux élevés de ROS et de NO dans les lésions aiguës de SEP21. La perturbation du complexe IV de la chaîne respiratoire mitochondriale altère la formation et la viabilité des OPC et des oligodendrocytesmatures 15, bien que les mécanismes exacts sous-jacents à cette altération restent à élucider.
De plus, la ferroptose, une forme de mort cellulaire programmée dépendante du fer, joue un rôle médiateur dans la perte d’oligodendrocytes et la progression de la démyélinisation dans des modèles animaux de SP induits par lacuprizone22. Le séquençage unicellulaire a révélé une diminution de l’expression du gène de l’antiferroptose dans les OPC et les OL dans le tissu cérébral de PwMS23. Dans la phase chronique de la SP, la carence en fer dans les oligodendrocytes peut perturber la remyélinisation et compromettre l’intégrité axonale de la substance blanche, entraînant une progression de la maladie24. De plus, le séquençage de l’ARNsn des lésions de la SEP dans la substance blanche suggère que la ferroptose est principalement active dans les lésions de la substance blanche des patients atteints de SEP et qu’elle est associée à l’activation du système phagocytaire25. Chez les souris expérimentales atteintes d’encéphalomyélite auto-immune (EAE), les niveaux de GPX4, un régulateur important de la ferroptose, sont réduits26. La progression de la maladie de l’EAE peut être accélérée par la ferroptose induite par ACSL4, qui améliore la signalisation du récepteur des lymphocytes T (TCR). Le blocage de la ferroptose et de l’ACSL4 ralentit significativement la progression de l’invalidité dans l’EAE27.
La ferroptose est impliquée dans le déclin cognitif causé par l’hypoxie chez les modèles de rats28. L’hypoxie et le facteur 1α induit par l’hypoxie favorisent la prolifération et la migration des ganglions de la racine dorsale29. Ainsi, la ferroptose et l’hypoxie peuvent agir en synergie dans la pathogenèse de la SP. Grâce à WGCNA, notre étude a identifié deux modules importants ; le module turquoise, en particulier, était fortement corrélé avec le statut de SEP, l’hypoxie et la ferroptose. D’après les résultats de l’étude WGCNA et des DEG, nous avons identifié 71 gènes clés qui étaient simultanément corrélés avec la ferroptose, l’hypoxie et la SP.
Les analyses d’enrichissement fonctionnel GO et KEGG des 706 DEG ont révélé que la voie de signalisation PI3K-Akt est une voie cruciale dans le développement de la SEP. L’inhibition de CXCR2 améliore la différenciation des OPC et favorise la remyélinisation dans les modèles murins de SEP en activant la signalisation PI3K/AKT/mTOR30. Un déséquilibre dans l’approvisionnement en oxygène pourrait affecter les voies de signalisation MAPK/PI3K-Akt, déclenchant des mécanismes clés impliqués dans le stress oxydatif31.
L’analyse de l’IPP a révélé que 10 gènes pivots, à savoir COL4A1, COL4A2, ITGB5, ITGB1, ITGB8, ITGAV, VIM, FLNA, VCL et SPARC, étaient simultanément corrélés avec l’hypoxie, la ferroptose et la SEP, avec une surexpression notable chez les patients atteints de SEP par rapport aux témoins. Ces gènes ont été validés dans GSE151306, une base de données de lignées d’OPC induites par des iPSC dérivées de patients atteints de SPPS. Nous avons constaté que l’ITGAV, l’ITGB8 et le VIM étaient également régulés à la hausse dans l’ensemble de validation. En particulier, ITGB8, qui fait partie de la famille des intégrines, était significativement surexprimé dans la SEP, ce qui était cohérent avec les ensembles de tests combinés GSE147315 et GSE196575.
Le gène ITGB8 est un membre de la famille des intégrines (ITG) et code pour l’intégrine bêta 8, qui a déjà été signalée comme un régulateur de la croissance tumorale et des métastases32,33. L’intégrine bêta 8 est fortement exprimée par les OPC et les oligodendrocytes matures. Une étude a montré que l’ostéopontine protège les OPC de l’apoptose induite par H2O2 en régulant à la hausse les intégrines, y compris ITGB834. Ces résultats indiquent que ITGB8 joue un rôle dans la survie cellulaire dans des conditions de stress oxydatif. Dans la sclérose en plaques, où le stress oxydatif est répandu, ITGB8 peut aider les OPC à survivre et à se différencier en oligodendrocytes pour augmenter la remyélinisation.
ITGAV code pour la sous-unité de l’intégrine alpha V, qui régule l’angiogenèse et la progression du cancer. L’ITGAV, en association avec le bêta-5 (formant l’intégrine αvβ5), médie l’activation microgliale en réponse au dépôt de fibronectine et de vitronectine, qui sont élevés dans les lésions de SEP. Dans le modèle de rat MS de l’EAE, l’intégrine avβ5 a activé la microglie, entraînant une expression accrue de la métalloprotéinase matricielle-9 (MMP-9), une enzyme qui dégrade la matrice extracellulaire et les protéines de myéline, contribuant ainsi à la démyélinisation35.
Notamment, l’un des gènes pivots qui a été exprimé de manière différentielle dans notre ensemble fusionné, ITGB1, s’est avéré être régulé à la hausse dans les lymphocytes T CD4+ extraits du liquide céphalo-rachidien des patients atteints de SEP36. L’intégrine β1 a eu un effet notable sur la voie de signalisation PI3K-Akt, ce qui est cohérent avec nos résultats (Figure 11)37. De plus, une augmentation de l’expression de la β1-intégrine a été détectée sur les cellules Th17 chez les patients atteints de SEP par rapport aux témoins sains. L’activation de la β1-intégrine dans les cellules Th17 peut augmenter considérablement les niveaux de glutamate et favoriser la neuroinflammation38.
Nous avons effectué une analyse de la courbe ROC pour évaluer le potentiel de l’expression d’ITGAV, d’ITGB8 et de VIM dans les OPC en tant que biomarqueurs pour la prédiction du risque de SEP. L’AUC de l’ITGB8 était de 100 %, suivi de 91,7 % pour VIM et de 58,3 % pour l’ITGAV. À l’heure actuelle, aucune étude n’a révélé d’association entre l’expression d’ITGB8 dans les oligodendrocytes et la SP. D’autres études devraient être menées pour explorer l’utilisation d’ITGB8 comme biomarqueur pour la prédiction de la SEP.
Enfin, nous avons construit un réseau de gènes de carrefour de facteurs de transcription et identifié trois facteurs de transcription clés : HOXD3, SP1 et VHL. SP1 est un facteur de transcription qui se lie à de nombreux promoteurs. Il a été démontré que SP1, connu pour sa large liaison promoteur, régule l’expression de Prdx6, inhibe les ROS et élimine la ferroptose dans les environnements à forte teneur en glucose39. L’expression de SP1 a également été liée à l’activité du facteur de nécrose tumorale (TNF), qui joue un rôle crucial dans le développement de laSEP-RR 40. Le récepteur des androgènes (AR), un autre facteur de transcription identifié, est activé par les hormones stéroïdes et est détecté dans les astrocytes dans les zones de remyélinisation, mettant en évidence son implication potentielle dans la fonction des oligodendrocytes41.
Les voies immunitaires et les cellules jouent un rôle important dans la pathogenèse et la neurodégénérescence de la SP et sont possiblement associées à la ferroptose, à l’hypoxie et aux COP. Des observations faites sur des souris intoxiquées à la cuprizone ainsi que sur des lésions progressives de SP ont montré une prévalence et des taux de prolifération élevés de lymphocytes T CD8+ avec surexpression des granules cytotoxiques42. Les lymphocytes T suffisamment activés sont capables d’interagir avec les oligodendrocytes présentateurs d’antigènes, ce qui peut entraîner la destruction des oligodendrocytes médiés par les lymphocytes T CD8+ . Dans le microenvironnement tumoral, l’interféron-γ sécrété par les lymphocytes T CD8+ réduit l’expression de l’échangeur de glutamate cystéine (xCT), améliorant ainsi la ferroptose43. De plus, il a été suggéré que la ferroptose active les lymphocytes T via la signalisation du récepteur des lymphocytes T dans les modèles EAE24.
Les recherches actuelles sur les OPC sont considérablement limitées en raison de la rareté de ces cellules. Cette limitation peut être partiellement résolue en utilisant des OPC induits par les iPSC. Cependant, la plupart des études sur les OPC induites par les iPSC impliquent des échantillons de petite taille, généralement moins de 10 donneurs, en raison du processus long et coûteux d’induction cellulaire. Notre étude fournit des résultats plus robustes en intégrant des échantillons provenant de plusieurs études.
Nous avons affiné davantage le pipeline bioinformatique en divisant l’ensemble de données disponible en ensembles d’entraînement et de validation. Cette approche minimise les biais potentiels induits par les différences dans les méthodes d’induction cellulaire et les techniques de séquençage entre les laboratoires. La plupart des études existantes sur la sclérose en plaques se concentrent sur la neurodégénérescence induite par l’inflammation ou utilisent une analyse isolée de la ferroptose. Les analyses traditionnelles de l’expression génique sont utiles pour identifier les gènes individuels exprimés de manière différentielle, mais elles ne permettent souvent pas de révéler les réseaux d’interaction complexes et les modèles de coexpression associés à la progression de la SEP. En revanche, notre méthodologie s’appuie sur WGCNA pour construire un réseau de coexpression génique, permettant l’identification de modules de gènes fortement corrélés à la ferroptose et à l’hypoxie. Cela permet de comprendre à l’échelle du système comment ces voies interagissent dans la pathologie de la SP, ce qui n’est pas réalisable avec les seules méthodes d’expression différentielle standard.
Cette méthode peut être appliquée à de nouveaux traitements de la SP en identifiant des cibles génétiques pour des thérapies visant à réduire la neurodégénérescence. Il pourrait également être utilisé dans d’autres maladies neurodégénératives impliquant la ferroptose et l’hypoxie, telles que la maladie de Parkinson ou la maladie d’Alzheimer, pour développer des interventions sur mesure et prédire des réponses thérapeutiques.
Notre recherche présente plusieurs limites. Tout d’abord, une validation in vivo et in vitro supplémentaire est nécessaire. Deuxièmement, nos résultats proviennent de deux bases de données et ont été validés dans une base de données couvrant un nombre limité de patients. Des cohortes plus importantes devraient être recueillies pour une exploration plus approfondie. Troisièmement, compte tenu de la difficulté d’accès aux COP humains, la base de données que nous avons adoptée était basée sur les COP induits par les CSPi, qui peuvent ne pas ressembler entièrement aux tissus humains. D’autres études axées sur l’étude de l’impact de l’hypoxie et de la ferroptose sur la différenciation et la mort cellulaire des cellules de la lignée des oligodendrocytes dans la SEP devraient être menées à l’avenir.
Une autre limite de cette étude est l’incapacité d’analyser les interactions entre les cellules immunitaires en raison de l’utilisation de COP dérivés de CSPi, qui n’ont pas les cellules immunitaires présentes dans les lésions de SP. Bien que les cellules immunitaires jouent un rôle crucial dans la pathogenèse de la SP, cette limite reflète les défis que pose l’utilisation de cultures dérivées d’iPSC. Les études futures devraient porter sur la diaphonie entre les cellules immunisées contre les OPC par le biais de systèmes de coculture ou d’organoïdes, qui peuvent mieux modéliser les interactions cellulaires dans l’environnement de la SP.
Déclarations de divulgation
Les auteurs n’ont pas d’intérêts concurrents à déclarer qui soient pertinents pour le contenu de cet article.
Remerciements
Cette étude a été soutenue et financée par le National High Level Hospital Clinical Research Funding (2022-PUMCH-B-103). Les auteurs tiennent à remercier la Dre Shuang Song, du Département de biostatistique, Harvard T. H. Chan School of Public Health, de l’Université Harvard, pour ses précieux conseils et orientations à l’étape de la révision de cet article.
matériels
| Name | Company | Catalog Number | Comments |
| BioInfoTools | / | online analysis website http://biowinford.site:3838/patrick_wang87/ | |
| Cytoscape | / | bioinformatics network analysis software | |
| GSE196575,GSE147315 and GSE151306 | / | RNA-seq from GEO dataset | |
| Omicshare | GENE DENOVO | online analysis tools https://www.omicshare.com/tools/Home/Soft/getsoft | |
| R-studio | RStudio, Inc | R integrated development environment software |
Références
- Fünfschilling, U., et al. Glycolytic oligodendrocytes maintain myelin and long-term axonal integrity. Nature. 485 (7399), 517-521 (2012).
- Nishiyama, A., et al. Polydendrocytes (NG2 cells): multifunctional cells with lineage plasticity. Nat Rev Neurosci. 10 (1), 9-22 (2009).
- Franklin, R. J. M., Ffrench-Constant, C. Remyelination in the CNS: from biology to therapy. Nat Rev Neurosci. 9 (11), 839-855 (2008).
- Lucchinetti, C., et al. A quantitative analysis of oligodendrocytes in multiple sclerosis lesions. A study of 113 cases. Brain. 122 (Pt 12), 2279-2295 (1999).
- Patrikios, P., et al. Remyelination is extensive in a subset of multiple sclerosis patients. Brain. 129 (Pt 12), 3165-3172 (2006).
- Starost, L., et al. Extrinsic immune cell-derived, but not intrinsic oligodendroglial factors contribute to oligodendroglial differentiation block in multiple sclerosis. Acta Neuropathol. 140 (5), 715-736 (2020).
- Niu, J., et al. Aberrant oligodendroglial-vascular interactions disrupt the blood-brain barrier, triggering CNS inflammation. Nat Neurosci. 22 (5), 709-718 (2019).
- Plastini, M. J., et al. Transcriptional abnormalities in induced pluripotent stem cell-derived oligodendrocytes of individuals with primary progressive multiple sclerosis. Front Cell Neurosci. 16, 972144 (2022).
- Pisa, M., et al. Subclinical anterior optic pathway involvement in early multiple sclerosis and clinically isolated syndromes. Brain. 144 (3), 848-862 (2021).
- Vidal-Jordana, A., et al. Optical coherence tomography measures correlate with brain and spinal cord atrophy and multiple sclerosis disease-related disability. Eur J Neurol. 27 (11), 2225-2232 (2020).
- Stockwell, B. R. Ferroptosis turns 10: Emerging mechanisms, physiological functions, and therapeutic applications. Cell. 185 (14), 2401-2421 (2022).
- Thamizhoviya, G., Vanisree, A. J. Enriched environment enhances the myelin regulatory factor by mTOR signaling and protects the myelin membrane against oxidative damage in rats exposed to chronic immobilization stress. Neurochem Res. 46 (12), 3314-3324 (2021).
- Williams, R., et al. Pathogenic implications of iron accumulation in multiple sclerosis. J Neurochem. 120 (1), 7-25 (2012).
- Stojkovic, L., et al. Targeted RNAseq revealed the gene expression signature of ferroptosis-related processes associated with disease severity in patients with multiple sclerosis. Int J Mol Sci. 25 (5), 3016 (2024).
- Ziabreva, I., et al. Injury and differentiation following inhibition of mitochondrial respiratory chain complex IV in rat oligodendrocytes. Glia. 58 (15), 1827-1837 (2010).
- Aboul-Enein, F., et al. Preferential loss of myelin-associated glycoprotein reflects hypoxia-like white matter damage in stroke and inflammatory brain diseases. J Neuropathol Exp Neurol. 62 (1), 25-33 (2003).
- Halder, S. K., Milner, R. Hypoxia in multiple sclerosis; is it the chicken or the egg. Brain. 144 (2), 402-410 (2021).
- Langfelder, P., Horvath, S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 9, 559 (2008).
- Pisa, M., et al. Subclinical neurodegeneration in multiple sclerosis and neuromyelitis optica spectrum disorder revealed by optical coherence tomography. Mult Scler. 26 (10), 1197-1206 (2020).
- Lucchinetti, C., et al. Heterogeneity of multiple sclerosis lesions: implications for the pathogenesis of demyelination. Ann Neurol. 47 (6), 707-717 (2000).
- Aboul-Enein, F., Lassmann, H. Mitochondrial damage and histotoxic hypoxia: a pathway of tissue injury in inflammatory brain disease. Acta Neuropathol. 109 (1), 49-55 (2005).
- Jhelum, P., et al. Ferroptosis mediates cuprizone-induced loss of oligodendrocytes and demyelination. J Neurosci. 40 (48), 9327-9341 (2020).
- Zhang, D., et al. Evidence of pyroptosis and ferroptosis extensively involved in autoimmune diseases at the single-cell transcriptome level. J Transl Med. 20 (1), 363 (2022).
- Luoqian, J., et al. Ferroptosis promotes T-cell activation-induced neurodegeneration in multiple sclerosis. Cell Mol Immunol. 19 (8), 913-924 (2022).
- Wu, T., et al. Role of ferroptosis in neuroimmunity and neurodegeneration in multiple sclerosis revealed by multi-omics data. J Cell Mol Med. 28 (10), e18396 (2024).
- Zeng, L., et al. Advances in research on immunocyte iron metabolism, ferroptosis, and their regulatory roles in autoimmune and autoinflammatory diseases. Cell Death Dis. 15 (7), 481 (2024).
- Neel, D. V., et al. Catching a killer: Mechanisms of programmed cell death and immune activation in Amyotrophic Lateral Sclerosis. Immunol Rev. 311 (1), 130-150 (2022).
- Liu, L., et al. Ferritinophagy-mediated hippocampus ferroptosis is involved in cognitive impairment in immature rats induced by hypoxia combined with propofol. Neurochem Res. 49 (7), 1703-1719 (2024).
- An, S., et al. HIF-1α induced by hypoxia promotes peripheral nerve injury recovery through regulating ferroptosis in DRG neuron. Mol Neurobiol. 61 (9), 6300-6311 (2024).
- Wang, L., et al. CXCR2 antagonism promotes oligodendrocyte precursor cell differentiation and enhances remyelination in a mouse model of multiple sclerosis. Neurobiol Dis. 134, 104630 (2020).
- Merelli, A., et al. Understanding the role of hypoxia inducible factor during neurodegeneration for new therapeutics opportunities. Curr Neuropharmacol. 16 (10), 1484-1498 (2018).
- Huang, L., et al. miR19b-3p promotes the growth and metastasis of colorectal cancer via directly targeting ITGB8. Am J Cancer Res. 7 (10), 1996-2008 (2017).
- Li, J., et al. Exosomal circDNER enhances paclitaxel resistance and tumorigenicity of lung cancer via targeting miR-139-5p/ITGB8. Thorac Cancer. 13 (9), 1381-1390 (2022).
- Mazaheri, N., et al. Ameliorating effect of osteopontin on H2O2-induced apoptosis of human oligodendrocyte progenitor cells. Cell Mol Neurobiol. 38 (4), 891-899 (2018).
- Milner, R., et al. Fibronectin- and vitronectin-induced microglial activation and matrix metalloproteinase-9 expression is mediated by integrins alpha5beta1 and alphavbeta5. J Immunol. 178 (12), 8158-8167 (2007).
- Hrastelj, J., et al. CSF-resident CD4+ T-cells display a distinct gene expression profile with relevance to immune surveillance and multiple sclerosis. Brain Commun. 3 (3), fcab155 (2021).
- Yan, L., Cui, Z. Integrin β1 and the repair after nervous system injury. Eur Neurol. 86 (1), 2-12 (2023).
- Birkner, K., et al. β1-Integrin- and KV1.3 channel-dependent signaling stimulates glutamate release from Th17 cells. J Clin Invest. 130 (2), 715-732 (2020).
- Zhang, Q., et al. Sp1-mediated upregulation of Prdx6 expression prevents podocyte injury in diabetic nephropathy via mitigation of oxidative stress and ferroptosis. Life Sci. 278, 119529 (2021).
- Hadi, N., et al. Study of the correlation between miR-106a, miR-125b, and miR-330 on multiple sclerosis patients by targeting TNFSF4 and SP1 in NF-кb/TNF-α pathway: A case-control study. Cell J. 24 (7), 403-409 (2022).
- Asbelaoui, N., et al. Interplay between androgen and CXCR4 chemokine signaling in myelin repair. Acta Neuropathol Commun. 12 (1), 18 (2024).
- Kaddatz, H., et al. Cuprizone-induced demyelination triggers a CD8-pronounced T cell recruitment. Glia. 69 (4), 925-942 (2021).
- Wang, W., et al. CD8+ T cells regulate tumour ferroptosis during cancer immunotherapy. Nature. 569 (7755), 270-274 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon