Method Article
Enrichissement des vésicules extracellulaires natives et recombinantes de mycobactéries
Dans cet article
Résumé
Ce protocole détaille l’enrichissement des vésicules extracellulaires mycobactériennes natives (mEVs) à partir de cultures axéniques de Mycobacterium smegmatis (Msm) et comment les MsmEVs recombinants contenant mCherry (un rapporteur fluorescent rouge) peuvent être conçus et enrichis. Enfin, il vérifie la nouvelle approche avec l’enrichissement de MsmEVs contenant la protéine EsxA de Mycobacterium tuberculosis.
Résumé
La plupart des bactéries, y compris les mycobactéries, génèrent des vésicules extracellulaires (VE). Étant donné que les VE bactériennes (bEV) contiennent un sous-ensemble de composants cellulaires, notamment des métabolites, des lipides, des protéines et des acides nucléiques, plusieurs groupes ont évalué les versions natives ou recombinantes des bEV pour leur pouvoir protecteur en tant que candidats vaccins sous-unitaires. Contrairement aux VE natifs, les VE recombinants sont génétiquement modifiés pour contenir un ou plusieurs immunogènes d’intérêt. Au cours de la dernière décennie, différents groupes ont exploré diverses approches pour générer des bEV recombinants. Cependant, nous rapportons ici la conception, la construction et l’enrichissement des VE mycobactériennes recombinantes (mEV) dans les mycobactéries. Pour ce faire, nous utilisons Mycobacterium smegmatis (Msm), une mycobactérie avirulente du sol comme système modèle. Nous décrivons d’abord la génération et l’enrichissement des EV natifs de Msm. Ensuite, nous décrivons la conception et la construction de mEVs recombinants qui contiennent soit mCherry, une protéine rapporteure fluorescente rouge, soit EsxA (Esat-6), un immunogène important de Mycobacterium tuberculosis. Nous y parvenons en fusionnant séparément les N-terminaisons mCherry et EsxA avec l’extrémité C d’une petite protéine Msm, Cfp-29. Cfp-29 est l’une des rares protéines abondamment présentes chez les MsmEVs. Le protocole de génération et d’enrichissement des mEVs recombinants à partir de Msm reste identique à celui de génération et d’enrichissement des EVs natifs de Msm.
Introduction
Malgré le développement et l’administration d’une large gamme de vaccins contre les maladies infectieuses, encore aujourd’hui, ~30 % de tous les décès humains sont dus à des maladies transmissibles1. Avant l’avènement du vaccin antituberculeux (TB) - Bacillus Calmette Guérin (BCG) - la tuberculose était la première cause de mortalité (~10 000 à 15 000/100 000 habitants)2. Grâce à l’administration du BCG et à l’accès facile aux médicaments antituberculeux de première et de deuxième intention, en 2022, le nombre de décès liés à la tuberculose a considérablement chuté à ~1 million/an en 2022 (c’est-à-dire ~15-20/100 000 habitants1). Cependant, dans les populations endémiques de tuberculose dans le monde, les décès liés à la tuberculose continuent de se situer entre ~100 et 550/100 000 habitants1. Bien que les experts reconnaissent plusieurs raisons conduisant à ces chiffres faussés, la protection médiée par le BCG ne dure même pas pendant la première décennie de la vie semble être la principale raison 3,4,5,6,7. Par conséquent, compte tenu des « Objectifs de développement durable » renouvelés de l’ONU et de la « Stratégie pour mettre fin à la tuberculose » de l’OMS, il y a un effort mondial concerté pour développer une alternative vaccinale bien supérieure au BCG qui offre peut-être une protection à vie contre la tuberculose.
Pour atteindre cet objectif, plusieurs groupes évaluent actuellement des souches modifiées/recombinantes du BCG, des espèces mycobactériennes non pathogènes et atténuées autres que le BCG, et des sous-unités candidates 8,9,10,11,12,13,14,15,16,17,18 . En règle générale, les vaccins sous-unitaires sont des liposomes chargés sélectivement avec peu de protéines immunogènes purifiées (~1-6) de longueur complète ou tronquée de l’agent pathogène. Cependant, en raison de leur repliement parasite en conformations non natives et/ou d’interactions aléatoires non fonctionnelles entre les protéines chargées, les sous-unités manquent souvent d’épitopes natifs et germaniques et, par conséquent, ne parviennent pas à amorcer suffisamment le système immunitaire14,19,20.
Par conséquent, les vésicules extracellulaires (VE) des bactéries ont pris de l’ampleur en tant qu’alternative prometteuse 21,22,23,24,25,26. En règle générale, les VE bactériennes (bEV) contiennent un sous-ensemble de leurs composants cellulaires, y compris certaines portions d’acides nucléiques, de lipides et des centaines de métabolites et de protéines27,28. Contrairement aux liposomes où quelques protéines purifiées sont chargées artificiellement, les bEV contiennent des centaines de protéines naturellement chargées, repliées nativement, avec une meilleure propension à amorcer le système immunitaire, en particulier sans le coup de pouce / l’aide d’adjuvants et d’agonistes des récepteurs de type Toll (TLR)27,28,29. C’est dans cette ligne de recherche que nous et d’autres avons exploré l’utilité des VE mycobactériennes en tant que boosters potentiels de sous-unités du BCG30. Malgré les inquiétudes selon lesquelles les bEV n’ont pas de charges antigéniques uniformes, les VE de Neisseria meningitidis atténué ont réussi à protéger les humains contre le méningocoque du sérogroupeB 31,32.
Du moins en théorie, les meilleurs VE qui pourraient bien booster le BCG sont les VE enrichis en bactéries pathogènes. Cependant, l’enrichissement des VE générées par des mycobactéries pathogènes est coûteux, long et risqué. De plus, les VE générées par des agents pathogènes peuvent être plus virulentes que protectrices. Compte tenu des risques potentiels, nous rapportons ici un protocole bien testé pour l’enrichissement des VE générées par le Msm cultivé axéniquement, une mycobactérie avirulente.
Cependant, bien qu’elles codent pour plusieurs orthologues de protéines pathogènes, les mycobactéries avirulentes manquent de plusieurs antigènes vaccinaux/épitopes protéiques pathogènes nécessaires pour préparer suffisamment le système immunitaire à la protection33. Par conséquent, nous avons également exploré la construction et l’enrichissement des VE recombinantes de Msm par ingénierie moléculaire, de sorte qu’une partie importante de toute protéine pathogène d’intérêt exprimée et traduite en Msm, doit atteindre ses EV. Nous avons émis l’hypothèse qu’une ou plusieurs des 10 protéines les plus abondantes des VE Msm, lorsqu’elles sont fusionnées à la protéine d’intérêt, aideront à une telle translocation.
Alors que nous commencions à standardiser l’enrichissement des VE mycobactériennes (mEVs) dans notre laboratoire, en 2011, Prados-Rosales et al. ont rapporté pour la première fois la visualisation et l’enrichissement des mEVs in vitro30. Plus tard, en 2014, le même groupe a publié une version modifiée de sa méthode34 de 2011. En 2015, Lee et al. ont également fait état d’une méthode standardisée indépendante pour l’enrichissement en mEV à partir de cultures axéniques de mycobactéries35. En combinant les deux protocoles34,35 et en incorporant quelques-unes de nos modifications après une standardisation approfondie, nous décrivons ici un protocole qui permet d’enrichir régulièrement les mEVs à partir de cultures axéniques de mycobactéries 36.
Ici, nous détaillons particulièrement l’enrichissement des VE spécifiques au MSM, qui est une extension d’un protocole publié36 pour l’enrichissement des VE mycobactériennes en général. Nous détaillons également comment construire des mEVs recombinants (R-mEVs) qui contiennent la protéine mCherry (en tant que rapporteur fluorescent rouge) et EsxA (Esat-6)37,38,39, un immunogène prédominant et une sous-unité potentielle de vaccinogène de Mycobacterium tuberculosis. Le protocole d’enrichissement des R-mEVs reste identique à celui que nous avons décrit pour l’enrichissement des VE natifs à partir de Msm.
Protocole
1. Conditions de croissance de Mycobacterium smegmatis, Escherichia coli et de leurs dérivés
- Média
- Bouillon liquide Middlebrook 7H9
- Préparez la solution mère Tween-80 à 20 % en préchauffant le volume requis d’eau doublement distillée (ddw) dans un bécher en verre à ~45-50 °C au micro-ondes, ajoutez le volume requis de Tween-80 à l’aide d’une éprouvette de mesure appropriée et remuez continuellement sur un petit agitateur magnétique pour amener les 20 % Tween-80 dans une solution uniforme. Filtrer le Tween-80 à 20 % à travers un filtre d’élimination de 0,22 um et conserver la solution mère jaune pâle à 4 °C.
REMARQUE : Toutes les traces de Tween-80 dans l’éprouvette de mesure doivent être transférées dans le bécher pour une concentration finale précise. Ne pas autoclaver le bouillon Tween-80 préparé. Filtrer (utiliser 0,22 μM), stériliser et conserver à 4 °C. - Suivez les instructions du fabricant pour la préparation du bouillon 7H9. Suspendre 4,7 g de poudre 7H9 dans 900 mL de ddw. Ajouter 2 mL de glycérol, mélanger le contenu et cuire à l’autoclave à 121 °C, 15 psi pendant 20 min.
REMARQUE : N’ajoutez pas d’enrichissement ADC Middlebrook (ADC) et Tween-80 avant l’autoclavage. - Une fois que le milieu autoclavé a refroidi à température ambiante (RT, c’est-à-dire ~25 °C), dans une enceinte de sécurité biologique standard de type A2, ajouter de manière aseptique 10 fois 10 ml de stock d’ADC (1 x final) et 2,5 ml (0,05 % final) de 20 % Tween-80. Filtrer le mélange à travers une unité de filtration jetable de 0,22 μm et stocker la solution mère transparente vert jaunâtre très clair à 4 °C.
REMARQUE : Le pH du substrat doit être de ~6,6 à 6,8 (si <6,4 ou >7,0, jetez-le et faites-le frais). Conserver à 4 °C (stable pendant 3 à4 semaines). Il est fortement recommandé de filtrer le média 7H9 (après l’ajout de l’ADC et du Tween-80) deux fois à travers deux unités de filtration jetables indépendantes de 0,22 μm.
- Préparez la solution mère Tween-80 à 20 % en préchauffant le volume requis d’eau doublement distillée (ddw) dans un bécher en verre à ~45-50 °C au micro-ondes, ajoutez le volume requis de Tween-80 à l’aide d’une éprouvette de mesure appropriée et remuez continuellement sur un petit agitateur magnétique pour amener les 20 % Tween-80 dans une solution uniforme. Filtrer le Tween-80 à 20 % à travers un filtre d’élimination de 0,22 um et conserver la solution mère jaune pâle à 4 °C.
- Bouillon liquide de Sáton (milieu minimal)
- Dissoudre la L-asparagine (0,4 % p/v) et l’acide citrique (0,2 % p/v) dans 950 mL de ddw. Ajouter 1 mL de chacun des bouillons 1 000 x (en poids jj) de phosphate de potassium dibasique fraîchement préparés (incolores ; bouillon 10 g/20 mL ; 1 x 0,5 g/L final) ; sulfate de magnésium heptahydraté (incolore ; bouillon 10 g/20 mL ; final 1x 0,5 g/L) ; et le citrate d’ammonium ferrique (brun très clair ; bouillon 1,6 g/40 mL ; final 1x 0,04 g/L) et bien mélanger sur un agitateur magnétique.
REMARQUE : Au mieux, les stocks 1 000x peuvent avoir 2 semaines ; si vous êtes plus âgé, préparez des bouillons frais ; rangez-les à RT dans l’obscurité (par exemple, à l’intérieur d’une armoire ou d’une étagère). Si le citrate d’ammonium ferrique est brun foncé, jetez-le et rendez-le frais. Il est préférable d’ajouter les trois sels dans l’ordre mentionné à l’étape 1.1.2.1. Faites tourbillonner la solution à chaque fois avant d’ajouter chaque solution saline. - Mesurez et notez le pH à l’aide d’un pH-mètre ; Assurez-vous qu’il se situe autour de 3,1 à 3,2. Si le pH est supérieur à 3,7, jetez les bouillons et préparez-les frais. Pour ajuster le pH à 7,4, utilisez autant de gouttes d’hydroxyde de sodium 10 N que nécessaire. Surveillez le pH final tout en agitant continuellement la solution/le milieu sur un agitateur magnétique.
- Ajouter 4,76 mL de glycérol, 0,25 mL de Tween-80 à 20 % (0,005 % final, voir aussi la note de l’étape 2.2.1.3), et seulement ensuite, porter le volume à 1 L. Ensuite, filtrez-stérilisez deux fois 1 L de milieu avec deux filtres d’élimination séparés de 0,22 μm. Conservez le milieu clair et incolore à 4 °C (stable pendant 2 semaines).
REMARQUE : Ce n’est qu’après avoir ajusté le pH à 7,4 que vous ajoutez du glycérol. Sinon, le support deviendra blanc trouble. S’il est trouble, jetez-le (n’essayez pas de le chauffer) et préparez-le frais. Pour toutes les préparations de mEVs, utilisez du Sauton’s fraîchement préparé.
- Dissoudre la L-asparagine (0,4 % p/v) et l’acide citrique (0,2 % p/v) dans 950 mL de ddw. Ajouter 1 mL de chacun des bouillons 1 000 x (en poids jj) de phosphate de potassium dibasique fraîchement préparés (incolores ; bouillon 10 g/20 mL ; 1 x 0,5 g/L final) ; sulfate de magnésium heptahydraté (incolore ; bouillon 10 g/20 mL ; final 1x 0,5 g/L) ; et le citrate d’ammonium ferrique (brun très clair ; bouillon 1,6 g/40 mL ; final 1x 0,04 g/L) et bien mélanger sur un agitateur magnétique.
- Base gélosée Middlebrook 7H11
- Suivez les instructions du fabricant pour la préparation. Suspendre 19 g de poudre 7H11 dans 900 mL de ddw. Ajouter 5 mL de glycérol, agiter à l’aide d’un agitateur magnétique pour obtenir une suspension uniforme (vert pâle ; pH 6,6 à 6,8 ; si >7,2, jeter et préparer frais), et autoclaver à 121 °C, 15 psi et pendant 20 min.
REMARQUE : N’ajoutez pas d’ADC et 0,05% Tween-80 avant l’autoclavage. - Lorsque le milieu refroidit à ~50 °C, de manière aseptique dans une enceinte de sécurité biologique de type A2, ajouter 100 mL d’ADC (amené à RT) et 2,5 mL de Tween-80 à 20 % (final de 0,05 %) (amené à RT). Distribuer immédiatement de manière aseptique dans des boîtes de Pétri.
REMARQUE : Les plaques sont stables à 4 °C pendant au moins 4 à6 semaines. Assurez-vous que les plaques sont à RT pendant la nuit avant d’emballer les plaques pour un stockage à 4 °C. Sinon, l’humidité sera piégée dans les plaques pendant l’incubation à 37 °C.
- Suivez les instructions du fabricant pour la préparation. Suspendre 19 g de poudre 7H11 dans 900 mL de ddw. Ajouter 5 mL de glycérol, agiter à l’aide d’un agitateur magnétique pour obtenir une suspension uniforme (vert pâle ; pH 6,6 à 6,8 ; si >7,2, jeter et préparer frais), et autoclaver à 121 °C, 15 psi et pendant 20 min.
- Bouillon Miller Luria Bertani (LB) et base d’agar
- Suivez les instructions du fabricant pour la préparation. Pour préparer le bouillon LB, suspendez 25 g de poudre dans 1 000 mL de ddw et remuez doucement pendant 5 minutes dans un bécher en verre sur un agitateur magnétique. Lors de la suspension uniforme, aliquote les volumes requis dans des flacons de milieu (p. ex., 300 mL dans un flacon de 500 mL) et un autoclave. Pour préparer la gélose LB, suspendre 40 g de poudre dans 1 000 mL de ddw et autoclaver (12 g de poudre dans 300 mL de ddw dans un flacon en verre de 500 mL).
- Bouillon liquide Middlebrook 7H9
- Conditions de croissance
REMARQUE : Toutes les étapes du travail de culture bactérienne doivent être effectuées dans une enceinte de sécurité biologique (type A2). Toutes les cultures doivent être traitées avec des tubes, des flacons et des pointes de pipette stériles.- Jour 1
- Ajouter 1 mL de bouillon glycérol de Msm (du congélateur à -80 °C) à 2 x 10 mL (dans des tubes centrifugés coniques stériles de 50 mL) de bouillon 7H9 fraîchement autoclavé, refroidi et préchauffé (~37 °C), remuer trois fois, fermer les couvercles et incuber les tubes pendant la nuit à 37 °C et 200-220 tr/min (agitateur d’incubateur).
REMARQUE : Pour cultiver Mycobacterium tuberculosis (Mtb), suivez des étapes similaires, mais incubez les tubes de 50 mL pendant 4 à 6 jours à37 °C et 120-150 tr/min (agitateur d’incubateur) dans des réglages BSL3. Suivre TOUTES les directives internationales et les pratiques de biosécurité des agents pathogènes BSL3 et du groupe de risque 3 lors de la manipulation et de la mise au rebut du Mtb et de ses cultures. Utilisez une enceinte de sécurité biologique de type B2 pour la manipulation du Mtb et de ses cultures.
- Ajouter 1 mL de bouillon glycérol de Msm (du congélateur à -80 °C) à 2 x 10 mL (dans des tubes centrifugés coniques stériles de 50 mL) de bouillon 7H9 fraîchement autoclavé, refroidi et préchauffé (~37 °C), remuer trois fois, fermer les couvercles et incuber les tubes pendant la nuit à 37 °C et 200-220 tr/min (agitateur d’incubateur).
- Jour 2
- Lorsque OD 600 (A 600nm ; densitomètre cellulaire) atteint ~1,0, centrifugez les cultures Msm pendant 10 min à 3200 × g et RT (centrifugeuse de paillasse). Jeter les surnageants à l’aide d’embouts de pipette stériles de 1 ml.
REMARQUE : L’étape ci-dessus reste la même pour les cultures Mtb, sauf que le nombre de jours est de 4 à 7. Veillez à ne pas toucher la pastille bactérienne avec l’embout de la pipette. - Lavage : À chaque pastille de Msm, ajouter 1 mL de milieu Sauton’s préchauffé (à l’étape 1.1.2) et remettre délicatement en suspension avec des pointes de pipette de 1 mL pour obtenir une suspension uniforme. À l’aide d’embouts de pipette stériles, composez les volumes jusqu’à 10 ml (dans chacun) avec le même milieu. Centrifuger les suspensions pendant 10 min à 3 200 × g et RT et éliminer les surnageants. Répétez cette étape une fois de plus.
REMARQUE : L’étape ci-dessus reste la même pour les cultures Mtb. - Remettre en suspension les cellules Msm lavées deux fois dans 20 ml (chacune) de Sauton préchauffées (préchauffées dans un incubateur à plaques ou à agitateur) et mesurer la densité optique des cellules à 600 nm. Inoculer le volume requis de cultures Msm dans des erlenmeyers stériles de 1 L contenant ~330 mL de Sauton stérile de sorte que le diamètre extérieur final de600 soit de ~0,05.
REMARQUE : Les resuspensions doivent toujours commencer dans un petit volume. Si le volume final est directement ajouté à la pastille en une seule étape, les cellules resteront sous forme de pastilles diffuses (un indicateur d’une mauvaise remise en suspension). La seule façon de résoudre le problème est de faire tourner les cultures vers le bas et de refaire la suspension comme recommandé. L’étape ci-dessus reste la même pour les cultures Mtb.
- Lorsque OD 600 (A 600nm ; densitomètre cellulaire) atteint ~1,0, centrifugez les cultures Msm pendant 10 min à 3200 × g et RT (centrifugeuse de paillasse). Jeter les surnageants à l’aide d’embouts de pipette stériles de 1 ml.
- Jour 2/3
- Incuber la culture de 330 mL dans l’agitateur de l’incubateur à 200 tr/min et à 37 °C jusqu’à ce que le diamètre extérieur de la culture600 atteigne ~0,3. Ensuite, lavez les cellules une fois (comme à l’étape 1.2.2.2 mais avec un volume égal), puis remettez la pastille en suspension dans le même volume. Répartir 50 mL des cultures remises en suspension chacune dans six erlenmeyers stériles de 1 L, contenant chacun 280 mL de Sauton stérile préchauffé avec 1/10ème de Tween-80 normalement utilisé (0,05 %) soit 0,005 % (voir également la note de l’étape 2.2.1.3. pour comprendre pourquoi 1/10ème). Le diamètre extérieur final de600 doit être d’environ 0,05.
REMARQUE : Pour les cultures Mtb, au lieu de flacons Erlenmeyer, utilisez des bouteilles à rouleau (d’une capacité de 1/2/4 L). Ajustez le volume de culture par bouteille de manière à ce que, lorsqu’elle est conservée sur l’appareil à rouleaux, la culture n’atteigne pas l’embouchure de la bouteille. Le volume réel de la bouteille à rouleau dépendra de la capacité de la bouteille à rouleau. - Incuber chacune des cultures de 330 mL dans l’agitateur de l’incubateur à 200 tr/min et à 37 °C jusqu’à ce que la DO600 atteigne 2,0 à 2,5 (~15-18 h).
REMARQUE : Pour les cultures Mtb, assurez-vous que le diamètre extérieur final600 est de ~1,0-1,2 (prend ~5-8 jours).
- Incuber la culture de 330 mL dans l’agitateur de l’incubateur à 200 tr/min et à 37 °C jusqu’à ce que le diamètre extérieur de la culture600 atteigne ~0,3. Ensuite, lavez les cellules une fois (comme à l’étape 1.2.2.2 mais avec un volume égal), puis remettez la pastille en suspension dans le même volume. Répartir 50 mL des cultures remises en suspension chacune dans six erlenmeyers stériles de 1 L, contenant chacun 280 mL de Sauton stérile préchauffé avec 1/10ème de Tween-80 normalement utilisé (0,05 %) soit 0,005 % (voir également la note de l’étape 2.2.1.3. pour comprendre pourquoi 1/10ème). Le diamètre extérieur final de600 doit être d’environ 0,05.
- Jour 1
2. Enrichissement des mEV Msm par l’utilisation de la centrifugation à gradient de densité
- Jour 4
- Centrifuger les ~2 L de cultures Msm au stade exponentiel moyen dans 6 flacons à centrifuger stériles de 400 ml à 4 °C pendant 20 min à ~8 000 × g (centrifugeuse au sol). Recueillir le surnageant de milieux usés ou de culture dans deux erlenmeyers de 1 L pré-réfrigérés et autoclavés et conserver une aliquote de la pastille pour toute procédure d’analyse (telle que le SDS-PAGE et le western blot - non détaillé ici).
REMARQUE : Toutes les étapes suivantes doivent être effectuées à froid (~ 4 °C) pour mieux maintenir l’intégrité des mEV. Les mEV sont assez stables à RT, mais la réfrigération est indispensable pour une stabilité à long terme (la congélation et la décongélation répétées ne sont pas recommandées). Au cours de la croissance de la culture axénique, les mEV se dissocient de la surface du MSM/Mtb et s’accumulent dans le surnageant de culture/milieu épuisé. - Filtrer le surnageant de culture Msm d’abord à travers la ou les unités de filtration d’élimination de 0,45 μm, puis à travers l’unité de filtration d’élimination de 0,22 μm pour éliminer toute trace de bactéries.
REMARQUE : La filtration directe à travers les filtres de 0,22 μm étouffe souvent les unités de filtration (car la pastille bactérienne peut être perturbée lors de l’exécution de l’étape 2.1.1.). Pour générer des surnageants de culture de Mtb, effectuer trois filtrations de cultures de Mtb (c’est-à-dire la première étape de filtration avec une unité de filtration jetable de 0,45 μm ; deux étapes de filtration consécutives avec des unités de filtration jetables de 0,22 μm) avant de déplacer les filtrats de culture dans des réglages BSL-2.
- Centrifuger les ~2 L de cultures Msm au stade exponentiel moyen dans 6 flacons à centrifuger stériles de 400 ml à 4 °C pendant 20 min à ~8 000 × g (centrifugeuse au sol). Recueillir le surnageant de milieux usés ou de culture dans deux erlenmeyers de 1 L pré-réfrigérés et autoclavés et conserver une aliquote de la pastille pour toute procédure d’analyse (telle que le SDS-PAGE et le western blot - non détaillé ici).
- Jour 4/5
- Utilisez les concentrateurs à membrane de 30 kDa pour concentrer le filtrat de culture Msm (~2 L) jusqu’à ~ 38 mL en centrifugeant le filtrat de culture à 4 °C, 20 min et à 3 200 × g.
- Prélavez d’abord les concentrateurs avec de l’eau froide stérile (~15 mL) (laver à 4 °C, 5 min et à 3 200 × g).
- Laver avec ~15 mL de Sauton’s préfiltré et froid (mêmes conditions que pour l’eau (2.2.1.1.)) pour éliminer toute trace de produits chimiques (utilisés lors de la fabrication).
- Étant donné que ~130 centricons (si une seule utilisation) sont techniquement nécessaires pour concentrer ~2 L de filtrat de culture, réutilisez les centricons au moins 3 à 4 fois si nécessaire. Suivez ces étapes : concentrez 15 mL à 0,5 à 1,0 mL (suivez l’étape 2.2.1), transférez le concentré dans un tube d’ultracentrifugation propre, autoclavé et froid de 38 mL, puis transférez à nouveau le filtrat de culture non concentré restant dans les centricons utilisés pour une concentration répétée.
REMARQUE : L’utilisation de concentrateurs de 24 et 30 kDa pour concentrer 2 L de filtrat de culture peut prendre jusqu’à 6 h. Lors de la concentration du filtrat de culture, le Tween-80 se concentre également et peut bloquer le concentrateur. L’utilisation de Tween-80 à 0,005 % final (plutôt que 0,05 %) dans les milieux de Sauton permet d’éviter ce blocage. La concentration réduite de Tween-80 n’affecte pas la suspension uniforme de Msm pendant la croissance et ne provoque pas d’agglutination des cellules Msm. Cependant, comme les cellules Mtb s’agglutinent à 0,005 % Tween-80, utilisez 0,05 % Tween-80 pour les VE spécifiques à Mtb.
- Transvaser le filtrat de culture concentré de Msm (~38 ml) dans un tube à centrifuger en polypropylène propre, lavé (avec ddw) et pré-refroidi de 40 à 50 mL et le soumettre à une centrifugation en deux étapes, d’abord à 4 000 × g, puis à 15 000 × g, les deux étapes à 4 °C pendant 20 minutes (pour éliminer tous les débris). Utilisez une centrifugeuse au sol pour la même chose.
- Utilisez les concentrateurs à membrane de 30 kDa pour concentrer le filtrat de culture Msm (~2 L) jusqu’à ~ 38 mL en centrifugeant le filtrat de culture à 4 °C, 20 min et à 3 200 × g.
- Jour 5/6
- Transvaser le surnageant de culture dans un tube à ultracentrifuger en polypropylène pré-réfrigéré de 38,5 mL et le faire tourner dans une ultracentrifugeuse à 100 000 × g pendant 4 h à 4 °C.
REMARQUE : Un godet pivotant fonctionne mieux à cette vitesse. Assurez-vous de remplir le tube d’ultracentrifugation à ras bord et d’avoir un tube d’ultracentrifugeuse d’équilibre de poids équivalent. Si l’un des tubes est collé au godet pivotant après la centrifugation (ce qui se produit en raison de la condensation), à l’aide d’une pince, retirez-le doucement du rotor. L’essuyage de l’humidité condensée présente sur la surface extérieure de l’ultracentrifugeuse avant l’ultracentrifugation empêche le collage. - Conservez le surnageant dans un tube préréfrigéré de 50 ml (voir note). Retournez le tube de l’ultracentrifugeuse sur un papier absorbant frais non pelucheux pour éliminer les traces du surnageant. Remettre le granulé en suspension dans 600 μL de solution tampon HEPES (50 mM HEPES et 150 mM NaCl, pH 7,4 ; filtrer-stériliser avant utilisation).
REMARQUE : Enregistrez le surnageant uniquement en tant que sauvegarde. La pastille mEV native se présente sous la forme d’une tache gélatineuse de 5 à 7 mm de diamètre, jaune grisâtre terne à translucide. Si aucune pastille n’est visible, répétez l’étape 2.3.1 en réutilisant le surnageant enregistré. Si aucune pastille n’apparaît après avoir répété l’étape 2.3.1, jetez-la et recommencez à partir de l’étape 1.2. La pastille prend du temps à se remettre en suspension. Il est recommandé d’ajouter le tampon HEPES et de le laisser toute la nuit à 4 °C pour faciliter la remise en suspension. Remettre en suspension doucement, mais avec des pipetages répétés (utiliser des embouts P200 pour une meilleure remise en suspension) jusqu’à ce que la remise en suspension soit uniforme.
- Transvaser le surnageant de culture dans un tube à ultracentrifuger en polypropylène pré-réfrigéré de 38,5 mL et le faire tourner dans une ultracentrifugeuse à 100 000 × g pendant 4 h à 4 °C.
- Jour 6
- Soumettre la pastille remise en suspension à une centrifugation à gradient de densité à base d’iodixanol.
- Étalez la pastille remise en suspension au fond du tube d’ultracentrifugation en polypropylène ultra-clair de 13 mL propre, lavé (avec ddw) et pré-refroidi et mélangez doucement (utilisez une pipette de 1 mL) avec ~4 mL de solution d’iodixanol à gradient de densité inerte (disponible dans le commerce sous forme de solution à ~60 % p/v). Après avoir superposé la pastille remise en suspension au fond du tube (jusqu’à un maximum de 5 mL), recouvrir de 1 mL (p/v) de sous-stocks de 40 %, 30 %, 20 % et 10 % de 'iodixanol' dans l’ordre respectif (préparer les sous-stocks (avec tampon HEPES) à partir de 60 % de stock). Ensuite, ajoutez 4 mL de sous-stock à 6 % (préparé à partir de 60 % de bouillon avec tampon HEPES) en haut pour remplir le tube.
REMARQUE : Préparez le dégradé juste avant l’utilisation ; Ne jamais stocker et utiliser. - Pesez soigneusement (sans secouer) le tube dans un bécher en verre et transférez-le doucement dans le rotor à godets oscillants.
REMARQUE : La pesée est nécessaire pour équilibrer avec un tube factice (également pesé). - Soumettez-le à l’ultracentrifugation à 141 000 × g pendant 16 h à 4 °C.
- Étalez la pastille remise en suspension au fond du tube d’ultracentrifugation en polypropylène ultra-clair de 13 mL propre, lavé (avec ddw) et pré-refroidi et mélangez doucement (utilisez une pipette de 1 mL) avec ~4 mL de solution d’iodixanol à gradient de densité inerte (disponible dans le commerce sous forme de solution à ~60 % p/v). Après avoir superposé la pastille remise en suspension au fond du tube (jusqu’à un maximum de 5 mL), recouvrir de 1 mL (p/v) de sous-stocks de 40 %, 30 %, 20 % et 10 % de 'iodixanol' dans l’ordre respectif (préparer les sous-stocks (avec tampon HEPES) à partir de 60 % de stock). Ensuite, ajoutez 4 mL de sous-stock à 6 % (préparé à partir de 60 % de bouillon avec tampon HEPES) en haut pour remplir le tube.
- Soumettre la pastille remise en suspension à une centrifugation à gradient de densité à base d’iodixanol.
- Jour 7
- Retirez délicatement le tube (voir la note de l’étape 2.3.1) et prélevez des fractions de 1 mL dans des tubes de microcentrifugation fraîchement autoclavés ; faites attention aux fractions 4à 6, qui contiennent généralement les mEV Msm.
REMARQUE : Les mEV de ces fractions se fractionnent normalement en trois ou quatre bandes de mEVs (l’une est la bande principale) qui sont de couleur blanc terne. Les R-mEVs contenant mCherry se fractionnent dans les 5 e à 7e fractions et apparaissent violet foncé à magenta. Les véhicules électriques VTT se fractionnent généralement en 5à 7e fraction. La séparation des mEV dans le gradient dépend de la concentration de gradient utilisée et de la qualité de la couche du gradient. Si les mEV se rompent partiellement, ils peuvent se fractionner en fractions antérieures. Alternativement, si la pastille obtenue après l’étape 2.3.2 n’est pas bien remise en suspension, les vésicules forment des micropastilles denses qui se fractionnent sous forme de fractions ultérieures. Nous recommandons une collecte précise des fractions uniquement lorsque l’utilisateur souhaite évaluer laquelle des fractions de 1 mL contient les mEV. Les utilisateurs peuvent souhaiter aliquoter en fractions plus ou moins grandes en fonction de leur convenance. Lorsque nous les utilisons pour certaines applications, par exemple, en les testant en tant que rappel potentiel d’un vaccin sous-unitaire au BCG, nous limitons notre collecte des mEV à moins de 250 μL des fractions (où les mEV se fractionnent) afin de ne pouvoir collecter spécifiquement que les bandes et traiter les mEV comme indiqué en 2.7.5. Cela permet d’éliminer plus efficacement toutes les traces d’iodixanol qui pourraient interférer avec nos expériences en aval.
- Retirez délicatement le tube (voir la note de l’étape 2.3.1) et prélevez des fractions de 1 mL dans des tubes de microcentrifugation fraîchement autoclavés ; faites attention aux fractions 4à 6, qui contiennent généralement les mEV Msm.
- Jour 8
- Regrouper les fractions contenant des mEVs, diluer avec un tampon HEPES à 38 mL et répéter l’ultracentrifugation à 4 °C pendant 16 h à 100 000 × g. Remettre la pastille en suspension (comme à l’étape 2.3.2 avec les mêmes précautions) soit dans un tampon HEPES, soit dans un tampon que les expériences en aval (non détaillées ici) telles que l’estimation des protéines, les analyses de nano-suivi, la coloration négative, la microscopie électronique à transmission, le transfert Western et le marquage immuno-or nécessitent.
REMARQUE : Pour une meilleure remise en suspension, sonnez le tube contenant du mEV pendant 10 minutes à l’aide d’un sonicateur à bain-marie à ultrasons. La sonisation pendant une période plus longue peut provoquer la rupture et la perte de mEV intacts. S’il est bien suspendu, la sonication n’est pas nécessaire. Toutes les étapes de 2.1 à 2.6 sont identiques tout en enrichissant les VE générés par le VTT.
- Regrouper les fractions contenant des mEVs, diluer avec un tampon HEPES à 38 mL et répéter l’ultracentrifugation à 4 °C pendant 16 h à 100 000 × g. Remettre la pastille en suspension (comme à l’étape 2.3.2 avec les mêmes précautions) soit dans un tampon HEPES, soit dans un tampon que les expériences en aval (non détaillées ici) telles que l’estimation des protéines, les analyses de nano-suivi, la coloration négative, la microscopie électronique à transmission, le transfert Western et le marquage immuno-or nécessitent.
3. Construction et enrichissement de mEVs recombinants.
NOTE : L’une des 10 protéines les plus abondantes (identifiées par spectrométrie de masse) des VE Msm est Cfp-2930. Compte tenu de sa petite taille (29 kDa), de sa structure secondaire simple 40, de sa localisation dans la membrane 41 et de sa propension à être sécrété dans les milieux épuisés dans les cultures axéniques (par exemple, en tant que protéine de filtrat de culture ; sécrétée à la fois par Msm et Mtb42,43, ici, elle a été exploitée pour délivrer un rapporteur fluorescent rouge et une protéine d’intérêt (EsxAMtb) dans les mEVs. Pour y parvenir,
- Utiliser des amorces directes et inverses appropriées (Tableau 1 ; compatibles avec le clonage direct dans un vecteur navette tel que pMV261) pour amplifier le cfp-29 à partir de Msm. En utilisant ~50 ng d’ADN génomique Msm de haut poids moléculaire (~>20 kb) comme modèle, la PCR amplifie le fragment du gène cfp-29 avec une ADN polymérase de relecture haute fidélité (telle que Phusion ou Q5). Suivez les recommandations du fabricant pour la PCR.
REMARQUE : Concevez les sites de restriction nécessaires en amorces pour faciliter le clonage dans n’importe quel autre vecteur de navette d’intérêt. Les conditions et le volume de la PCR varient en fonction du type et de la marque de l’ADN polymérase de relecture utilisée. Le volume du mélange réactionnel varie en fonction de la quantité de matrice d’ADN et de la quantité d’ADN polymérase relusée. Suivez les recommandations du fabricant pour les conditions de PCR, le succès de l’amplification et l’élimination du recuit non spécifique des amorces. - La PCR purifie l’amplicon élué avec n’importe quel kit de purification PCR disponible dans le commerce et vérifie la longueur de l’amplicon par électrophorèse sur gel d’agarose standard. Digérer l’amplicon purifié.
- Estimez la quantité d’amplicon purifié à l’aide d’un spectrophotomètre. Utilisez au moins 2 μg d’amplicon cfp-29 pour la digestion.
- Digérer d’abord avec BstB1 à 65 °C pendant 1 h (type et quantité de tampon et d’enzyme - selon les recommandations du fabricant), ramener la température de réaction à RT, puis digérer avec HindIII pendant 1 h à 37 °C (type et quantité de tampon et d’enzyme - selon les recommandations du fabricant).
- La PCR purifie et élue l’amplicon digéré dans 50 μL de ddw sans nucléase autoclavée.
- Estimer la concentration et le rendement de l’amplicon digéré à l’aide d’un spectrophotomètre. Conserver à -20 °C jusqu’à la mise en place de la ligature. REMARQUE : Après PCR, vérifier la longueur de l’amplicon (~798 + 50 pb) et le rendement en électrophoresant 10 μL du mélange de réaction PCR sur un gel d’agarose à 1%. Bien que la double digestion ne soit pas possible avec cette combinaison d’enzymes, un tampon compatible empêchera la purification répétée par PCR et la perte subséquente de l’amplicon digéré. La concentration d’amplicon digéré varie selon le kit utilisé pour la purification par PCR. Il varie également en fonction de la longueur (en pb) des vecteurs alternatifs de votre choix.
- Utiliser les amorces directes et inverses (Tableau 1), une relecture de l’ADN polymérase et ~50 ng d’ADN génomique Mtb, amplifier par PCR l’amplicon spécifique à l’étiquette FLAG esxA ou esxA-3X. Utilisez ~5 ng d’ADN plasmidique approprié pour amplifier mCherry par PCR. Les détails du plasmide et de la séquence se trouvent dans le fichier supplémentaire 1.
REMARQUE : Utilisez un linker Glycine, Glycine, Glycine, Glycine, Serine 44,45(G4S) entre cfp-29 et mCherry/esxA/esxA-3X FLAG ; avant le démarrage de mCherry, le linker G4S aide mCherry à ne pas subir de plis non fonctionnels parasites (y compris ceux entraînés par Cfp-29). Reportez-vous aux séquences cfp-29, hsp60 promoter, mCherry et esxA dans le fichier supplémentaire 1. Les plasmides servant de modèles pour mCherry sont disponibles dans différents dépôts/banques de plasmides. Différentes versions (séquences légèrement modifiées) de mCherry sont disponibles qui nécessiteront des séquences d’amorçage avant et arrière modifiées. Les amorces (tableau 1) aident à amplifier le mCherry mentionné dans le fichier supplémentaire 1. La fusion de l’extrémité N de mCherry/esxA/esxA ::3XFLAG à l’extrémité C-terminale de Cfp-29 fonctionne bien. - Digérer 1 μg d’ADN des amplicons mCherry/esxA/esxA ::3XFLAG et purifier l’ADN digéré.
- Digérer deux fois chaque amplicon avec HindIII et HpaI pendant 1 h à 37 °C (ou selon les recommandations du fabricant).
- Utilisez un kit de purification PCR disponible dans le commerce et les recommandations du fabricant pour purifier l’amplicon digéré. Éluer l’amplicon digéré dans 50 μL de ddw sans nucléase autoclavée.
- Estimer la concentration et le rendement de l’amplicon digéré à l’aide d’un spectrophotomètre. Conserver à -20 °C jusqu’à la mise en place de la ligature.
- Traiter 2 μg de pMV261-KanR (fichier supplémentaire 1) ou un vecteur de clonage approprié avec l’enzyme ou les enzymes de votre choix.
- Digérer d’abord avec BstB1 à 65 °C pendant 1 h (type et quantité de tampon et d’enzyme - selon les recommandations du fabricant), ramener la température de réaction à RT, puis digérer avec HindIII pendant 1 h à 37 °C (type et quantité de tampon et d’enzyme - selon les recommandations du fabricant).
- Gel purifiant et éluant dans 50 μL de ddw sans nucléase autoclavée.
- Estimez la concentration et le rendement du vecteur digéré à l’aide d’un spectrophotomètre. Conserver à -20 °C jusqu’à la mise en place de la ligature.
REMARQUE : Le clonage en une étape à deux fragments est possible avec les amorces ci-dessus pour le pMV261. N’importe quel vecteur navette alternatif qui survit en tant qu’épisome fonctionnera. Les plasmides intégratifs fonctionneront également, mais le rendement en protéines recombinantes sera relativement inférieur par cellule.
- Mettez en relation et transformez-vous en une souche compatible d’E. coli.
- Pour la ligature, utilisez 125 ng du vecteur. Utilisez des amplicons mCherry/esxA/esxA ::3XFLAG correctement digérés dans un rapport molaire de 1 :3. Effectuer la ligase pendant la nuit à l’aide de T4 DNA Ligase (quantité conforme aux recommandations du fabricant) à 16 °C dans un bain-marie réfrigéré à circulation.
REMARQUE : Utiliser des témoins appropriés, tels que le vecteur uniquement avec et sans T4 DNA Ligase, pour évaluer l’efficacité de la digestion et prédire l’efficacité du succès du clonage. - Transformation
- Décongeler des aliquotes de cellules chimiquement compétentes NEB5α (~100 μL par transformation) sur de la glace pendant 15 min. Mélangez délicatement deux fois à l’aide d’un embout de pipette stérile. Ajouter le mélange de ligature (jusqu’à 20 μL) aux cellules froides compétentes.
- Mélangez délicatement les cellules de pipette + l’ADN ligaturé. Incubez le mélange sur de la glace pendant 30 minutes supplémentaires.
- Fournir un choc thermique à 42 °C (dans un bain-marie à circulation) pendant 60 s et transférer immédiatement sur la glace pendant 15 minutes supplémentaires.
- Récupérer les cellules transformées en ajoutant 1 mL de bouillon SOC (2 % de tryptone, 0,5 % d’extrait de levure, 10 mM de NaCl, 2,5 mM de KCl, 10 mM de MgCl2, 10 mM de MgSO4 et 20 mM de glucose) et incuber à 37 °C pendant 1 h à 200 tr/min.
- Essorez les bactéries récupérées dans un tube de microcentrifugation de 1,5 mL (3000 × g, RT et 10 min), jetez le surnageant, remettez la pastille en suspension dans 200 μL de milieux LB stériles, préchauffés et fraîchement préparés, et étalez la suspension sur des plaques de gélose LB Miller fraîchement versées contenant les antibiotiques requis à des concentrations appropriées.
- Incuber les boîtes de Pétri dans un incubateur à plaques préréglé à 37 °C.
- Pour la ligature, utilisez 125 ng du vecteur. Utilisez des amplicons mCherry/esxA/esxA ::3XFLAG correctement digérés dans un rapport molaire de 1 :3. Effectuer la ligase pendant la nuit à l’aide de T4 DNA Ligase (quantité conforme aux recommandations du fabricant) à 16 °C dans un bain-marie réfrigéré à circulation.
- Rechercher (non détaillé ici) les clones potentiels, vérifier (par PCR/enzymes de restriction de la colonie)46 et les séquencer (séquençage de Sanger) pour confirmer la fusion.
- Extraire l’ADN plasmidique (~200 ng/1-2 μL ; n’importe quel kit disponible dans le commerce) du clone confirmé (fond E. coli ) et le transformer en cellules électro-compétentes de Msm fraîchement fabriquées.
- Préparation de cellules électrocompétentes Msm
- Cultivez fraîchement Msm (comme dans les étapes 1.2.1 et 1.2.2). Lavez le Msm fraîchement cultivé comme aux étapes 1.3.3 et 1.3.4 (sauf qu’utilisez 7H9 + ADC + Tween-80 (riche) au lieu de Sauton).
- Ajouter une aliquote des cellules Msm lavées à un OD final de 600 de ~0,05 dans un erlenmeyer stérile de500 ml contenant 150 ml de milieux riches.
- Incuber dans l’agitateur de l’incubateur à 200 tr/min et 37 °C jusqu’à ce que le diamètre d’exposition de la culture600 atteigne ~0,8 à 1,0 (~12-14 h).
- Transférez la culture dans une bouteille à centrifuger préréfrigérée de 400 ml et incubez-la sur de la glace pendant 60 à 90 minutes. Ensuite, pelletez les cellules à 4 °C pendant 15 minutes à 4 000 × g.
- Lavez les cellules deux fois (chaque fois avec 150 ml, à 4 000 × g, 4 °C et 15 min) avec du glycérol à 10 % stérile (autoclavé) glacé.
REMARQUE : À chaque lavage, le granulé se détache. Soyez prudent lorsque vous jetez le surnageant entier (après chaque lavage). Sinon, la majorité des cellules seront perdues dans le surnageant mis au rebut. - Lavez les cellules une fois de plus avec 75 mL de glycérol stérile glacé à 10 % contenant 0,005 % de Tween-80.
- Remettre en suspension la pastille cellulaire dans 7,5 mL de glycérol à 10 % avec 0,005 % de Tween-80 et l’aliquote dans des aliquotes de 400 μL.
REMARQUE : Bien que les cellules électrocompétentes Msm soient compétentes pendant au moins 4 mois, les cellules électrocompétentes fraîchement préparées donnent les meilleurs résultats. Lors de l’utilisation de vieilles cellules compétentes, quelques colonies non roses (blanches) apparaissent comme de faux transformants. Plus les cellules compétentes sont anciennes, plus les colonies blanches sont nombreuses.
- Transformation de Msm
- Décongeler les cellules compétentes Msm aliquote sur de la glace.
REMARQUE : La décongélation à RT et la transformation de ces cellules donnent moins d’efficacité. - Ajouter 1 à 2 μL d’ADN plasmidique (~200 ng au total) aux cellules froides compétentes, mélanger doucement avec un embout de pipette stérile de 1 mL et transférer dans une cuvette d’électroporation stérile pré-refroidie de 2 mm.
- Transférez la cuvette fermée avec le mélange de cellules compétentes Msm + ADN plasmidique dans la souris de l’elctroporator, fermez doucement le couvercle et appliquez une impulsion (type de désintégration exponentielle) à 2,5 kV (tension), 25 μF (capacité) et 1000 Ω (résistance).
- Ajouter immédiatement 1 mL de milieu riche stérile préchauffé (7H9 + ADC + Tween-80) dans la cuvette, mélanger délicatement avec un embout de pipette stérile de 1 mL et transférer tout le contenu dans un tube stérile de 10 mL.
- Incuber le contenu pendant 3 h dans un agitateur incubateur réglé à 37 °C et 200 tr/min. Essorer le contenu dans un tube de microcentrifugation (4 000 × g, RT et 10 min), jeter le surnageant, remettre la pastille en suspension dans 200 μL de milieu riche stérile préchauffé et étaler la suspension sur des plaques de gélose 7H11 fraîchement versées contenant de l’ADC, du Tween-80 et les antibiotiques requis à des concentrations appropriées.
REMARQUE : Pour le Msm, si nécessaire, utiliser l’hygromycine, la kanamycine et l’apramycine aux concentrations finales de 50 μg/mL, 25 μg/mL et 50 μg/mL, respectivement. Le MSM en soi n’est PAS résistant à ces antibiotiques. N’utilisez ces antibiotiques que lorsque vous utilisez des plasmides avec les gènes de résistance appropriés pour la sélection/croissance de colonies de MSM transformantes/recombinantes. - Incuber les boîtes de Pétri dans un incubateur à plaques préréglé à 37 °C. En règle générale, les transformants apparaissent entre 3 et5 jours.
NOTE : Si une protéine d’intérêt est toxique pour le Msm, les transformants peuvent émerger plus tard ou ne pas émerger. Dans ce cas, clonez les versions tronquées de la longueur complète. - Faire des stocks de glycérol des colonies émergentes de Msm après avoir vérifié la présence de clones positifs (comme à l’étape 3.7)
- Décongeler les cellules compétentes Msm aliquote sur de la glace.
- Pour enrichir les R-mEVs contenant des protéines mCherry ou EsxA, cultivez d’abord R-Msm exprimant le FLAG mCherry ou exsA ou esxA ::3X en suivant les étapes 1.2.1 à 1.2.3, puis suivez les étapes 2.1 à 2.6. pour enrichir les R-mEVs. Les R-mEVs éluent dans les fractions 4 ème-7ème après le spin du gradient de densité (étape 2.5). Les pastilles R-mEVs après la première étape d’ultracentrifugation (2.3.1). Après avoir effectué la même chose qu’à l’étape 2.3.1, vérifiez que les R-mEV sont visibles sous la forme d’une pastille violet foncé à magenta de 5 à 7 mm de diamètre en bas au centre de l’ultracentrifugeuse.
- Effectuer des analyses occidentales47 (non détaillées ici) pour détecter la ou les protéines d’intérêt dans les R-mEVs enrichis.
Résultats
Nous utilisons M. smegmatis (Msm) comme mycobactérie modèle pour démontrer l’enrichissement des mEVs natives et recombinantes (R-mEVs). Ce protocole d’enrichissement des mEVs résumé schématiquement (Figure 1) fonctionne également pour l’enrichissement des R-mEVs de Msm et des EVs natifs de Mtb (avec des modifications mineures comme dans les notes de protocole de 1.2). La visualisation des mEVs enrichis nécessite de les colorer négativement au microscope électronique à transmission36 (Figure 2A). En règle générale, les VE spécifiques au Msm se séparent dans les fractions 4 e-6e 1 mL du gradient de densité de 6 à 60 % de 13 mL (figure 2B). Environ 80 à 100 μg d’équivalent protéique des VE sont couramment obtenus à partir de 2 L de cultures axéniques mi-logarithmiques de Msm. Leur diamètre varie généralement entre 20 nm et 250 nm (figure 2C).
L’un des objectifs à long terme de notre laboratoire est d’évaluer si les mEV de différentes mycobactéries pourraient potentiellement agir comme une sous-unité de rappel du vaccin existant, le BCG. Étant donné que l’enrichissement des mEV générés par des bactéries pathogènes est long, risqué et coûteux, l’exploitation des VE natives et recombinantes à partir de mycobactéries avirulentes peut constituer une alternative appropriée. Par conséquent, nous visons non seulement à standardiser le protocole pour enrichir les mEVs de Msm, mais aussi à construire et enrichir ses R-mEVs.
Pour construire les R-mEVs, nous avons d’abord sélectionné les 10 protéines les plus abondantes des VE Msm (Tableau 1 ; nous les avons identifiées à partir d’analyses détaillées par spectrométrie de masse des EVs Msm)30. Nous avons émis l’hypothèse que si une protéine étrangère d’intérêt est fusionnée par translation avec l’une d’entre elles, elle devrait être capable de se localiser en mEV. Ensuite, nous avons présélectionné Cfp-29 parmi les 10 parce que c’est le plus petit d’entre eux (~29 kDa), qu’il est localisé par membrane et qu’il s’agit d’une protéine de filtrat de culture avec une structure secondaire relativement simple40,41,42,43. Nous avons fusionné translationnellement l’extrémité N-terminale de mCherry (protéine fluorescente) à l’extrémité C-terminale de Cfp-29 et évalué son chargement/livraison dans les mEVs. Une partie des mEV enrichis de Msm est devenue rose (Figure 3), ce qui indique la capacité de Cfp-29 à transporter une protéine étrangère d’intérêt dans les VE Msm.
Compte tenu de cette capacité de Cfp-29 (Figure 3), nous avons ensuite évalué EsxA (Esat-6), une importante protéine immunogène 37,38,39 délivrée dans les VE Msm. Encore une fois, nous avons généré deux fusions translationnelles indépendantes vers l’extrémité C de l’EsxA Cfp-29 uniquement et la balise EsxA + 3X FLAG (3X FLAG fusionné à l’extrémité C de l’EsxA. Comme on pouvait s’y attendre, nous avons observé l’EsxA de Mtb dans les VE de Msm (voies 5, 6 et 10, Figure 4A,B), bien qu’en faible quantité. Il est intéressant de noter que Cfp-29 ::EsxA ::3XFLAG était beaucoup plus stable (voies 3 et 4 plus 8 et 9 ; Graphique 4A) et accumulés à des niveaux plus élevés dans les mEV (voies 3 et 4 plus 8 et 9 ; Graphique 4B). En résumé, nous démontrons la conception, la construction et l’enrichissement des R-mEVs qui contiennent une protéine étrangère d’intérêt (Figure 3 et Figure 4).

Figure 1 : Représentation schématique de l’enrichissement des vésicules extracellulaires mycobactériennes. « Jours » (en rouge, en haut de la figure) fait référence aux jours nécessaires à l’enrichissement des mEV à partir de Msm (en particulier pour les étapes de protocole 1 et 2). Les « étapes » (en noir, en bas de la figure) font référence aux étapes du protocole telles que décrites dans la section sur le protocole. Pour l’enrichissement des mEV à partir de Mtb, bien que toutes les étapes soient similaires, il faut compter au moins 10 jours pour les différentes étapes de la culture (étape 1) à la première étape de centrifugation (étape 2). Les étapes suivantes nécessitent une durée identique (comme indiqué en rouge). Avant le gradient de densité, les complexes granulés mEVs + extracellulaires semblent incolores lorsqu’ils enrichissent à la fois les mEVs natifs et les R-mEVs. Cependant, il apparaîtrait rosâtre à bleu (voir Figure 3) lors de l’enrichissement de mEVs contenant mCherry. Après le gradient de densité, les mEV apparaîtraient en blanc (natifs et R-mEVs) ou roses (s’ils contiennent des mCherry) dans le tube de gradient de densité. Les couleurs des mEV dans la figure ne servent qu’à indiquer la clarté et ne représentent pas les couleurs exactes. Voir la figure 3 pour plus de clarté. Abréviations : Msm = M. smegmatis ; Mtb = M. tuberculosis ; 0,45 et 0,22 μm = taille des pores des unités de filtration d’élimination. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Les VE mycobactériennes sont circulaires, se concentrent avec des gradients de densité et varient en dimensions. (A) Une image représentative des VE Msm lors de leur visualisation avec coloration négative et visualisation au microscope électronique à transmission. Barre d’échelle = 200 nm. (B) Une image représentative de la façon dont les mEV apparaissent dans le tube d’ultracentrifugation lors de l’exécution d’un gradient de densité de 6 à 60 %. Si le gradient de 6 à 60 % n’est pas superposé avec précision, le positionnement des bandes principales (flèche ouverte en haut) et mineures (flèche ouverte en bas) des mEV peut changer considérablement, modifiant ainsi le nombre de fractions de 1 mL. (C) Une image représentative de l’analyse de nanotracking des mEVs enrichis de Msm. Les analyses de nanotracking révèlent les proportions et les concentrations de mEVs de différentes tailles et le nombre total de mEVs dans la préparation. En moyenne, avec le protocole détaillé ici, ~1-3 ×10 10 EV sont enrichis à partir de 2 L de Msm et Mtb. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Différentes étapes indicatives de l’enrichissement des mEVs exprimant mCherry. (A) Image représentative montrant la culture axénique Msm exprimant Cfp-29 ::mCherry. (B) Image représentative de la pastille bactérienne post-centrifugation de la culture axénique Msm exprimant Cfp-29 ::mCherry. (C) Image représentative d’un concentré de filtrat de culture obtenue après concentration des milieux épuisés de culture axénique Msm exprimant Cfp-29 ::mCherry à travers des concentrateurs centricon. (D) Image représentative des mEVs + complexes extracellulaires après ultracentrifugation d’un concentré de filtrat de culture obtenu à partir d’une culture axénique de Msm exprimant Cfp-29 ::mCherry. La pastille resterait cependant incolore si les mEV étaient natifs ou recombinants (lors de la fusion avec le Cfp-29) pour toute protéine étrangère, à l’exception de mCherry. (E) Images représentatives de mCherry contenant des mEVs. Notez que tous les mEV ne sont pas roses, ce qui indique que tous les mEV ne contiennent pas de Cfp-29. Bon : Une image représentative indiquant les différentes bandes de mEVs généralement obtenues après enrichissement pour les EVs Cfp-29 ::mCherry. Étant donné que les VE contenant mCherry sont plus denses, ils se séparent en fractions ultérieures. Médiocre : Une image représentative indiquant une mauvaise séparation (les mEVs blancs se séparent dans la première/deuxième fraction et les mEVs contenant mCherry se séparent dans la 10e fraction (peut-être en raison d’une mauvaise remise en suspension de la pastille de mEV, c’est-à-dire l’étape de protocole 2.3.2). Veuillez cliquer ici pour voir une version agrandie de cette figure.
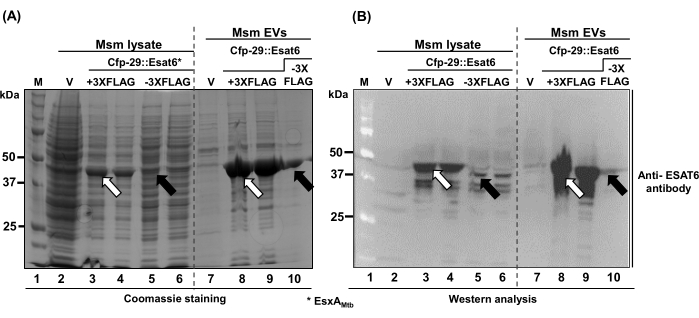
Figure 4 : Cellules souches recombinantes contenant de l’EsxA (ESAT-6), un immunogène codé par Mtb dans le lysat de cellules totales de Msm et des VE générées par le Msm. (A) gel de Coomassie et (B) analyse occidentale (détectée avec des anticorps polyclonaux spécifiques d’ESAT6) images montrant l’accumulation d’ExsA fusionné dans le lysat de cellules totales Msm et les mEVs exprimant soit cfp-29 ::esxA ::3XFLAG (+ 3XFLAG, voies 3 et 4 (lysat total) et voies 8 et 9 (mEVs)) ou cfp-29 : esxA (- 3XFLAG, voies 5 et 6 (lysat total) et voie 10 (mEVs)). Les flèches ouvertes et remplies indiquent respectivement les Cfp-29 ::ExsA ::3XFLAG et Cfp-29 ::ExsA accumulés. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Amorces: | ||||||||||
| Sl # | Gène | Amorce | Séquence (5' à 3') | Source | Pour cloner dans | |||||
| 1 | Le CFP-29 | En avant | CAGTTCGAA(BstBI)ATGAACAACCTCTATCGC | ADN génomique Msm | Réf. pMV261 | |||||
| Inverse | GAAAAGCTT(HindIII)GGGGGTCAGCGCGACAG | ADN génomique Msm | Réf. pMV261 | |||||||
| 2 | mCerise | En avant | GAAAAGCTT(HindIII) ggcggcggtggctcg (Lien G4S)ATGGTGAGCAAGGGCGAGGAGG | Collection de laboratoire | Réf. pMV261 | |||||
| Inverse | TGTGTTAAC(HpaI)CTACTTGTACAGCTCGTCC | Collection de laboratoire | Réf. pMV261 | |||||||
| 3 | esxA | En avant | GAAAAGCTT(HindIII)ggcggcggtggctcg (éditeur de liens G4S) ATGACAGAGCAGCAGTGGAATTTCGCGGGTATCGAG | ADN génomique Mtb | Réf. pMV261 | |||||
| Inverse | TGTGTTAAC(HpaI)TCATGCGAACA TCCCAGTGACGTTGCCTTCGGTCG | ADN génomique Mtb | Réf. pMV261 | |||||||
| 4 | DRAPEAU exsA-3X | En avant | GAAAAGCTT(HindIII)ggcggcggtggctcg (éditeur de liens G4S) ATGACAGAGCAGCAGTGGAATTTCGCGGGTATCGAG | ADN génomique Mtb | Réf. pMV261 | |||||
| Inverse | TGTGTTAAC(HpaI)TCA cttgtcgtcgtcgtccttgtagtcgatgtcgtg gtccttgtagtcaccgtcgtggtccttgt CCTTCGGTCGAAGCCATTGCCTGACC | ADN génomique Mtb | Réf. pMV261 | |||||||
Tableau 1 : Séquences d’amorces. Les amorces directes et inverses pour l’amplification des FLAG cfp-29, mCherry, esxA et esxA ::3X et le clonage dans le vecteur de navette pMV261 sont répertoriées.
Fichier supplémentaire 1 : A : pMV261 , sa carte circulaire, ses principales caractéristiques et sa séquence nucléotidique complète. Séquence nucléotidique du promoteur hsp60 en surbrillance jaune ; Sites de restriction de surbrillance verts et cyan dans lesquels les amplicons mCherry, esxA et esxA ::3X FLAG ont été clonés. B, C et D : séquences nucléotidiques de cfp-29, mCherry et esxA, respectivement. Codon vert de cfp-29, mCherry et esxA. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Étant donné que la mise au point d’un nouveau vaccin antituberculeux supérieur au BCG et capable de le remplacer reste un défi de taille, plusieurs groupes poursuivent la découverte de différents vaccins antituberculeux sous-unitaires capables d’augmenter la puissance du BCG et de prolonger sa durée de protection48,49. Compte tenu de l’attention croissante portée aux VE bactériennes (bEV) en tant que sous-unités potentielles et adjuvants naturels50,51, l’enrichissement constant de quantités suffisantes de mEV pour leur test/analyse en aval est devenu une étape importante. C’est en tenant compte de ces questions de recherche que ce protocole vise à enrichir les mEVs à partir de cultures axéniques et de leurs versions recombinantes.
Au cours d’analyses détaillées du protéome par spectrométrie de masse des VE Msm, Prados-Rosales et al. ont identifié les 10 protéines les plus abondantes30. Nous avons en outre émis l’hypothèse qu’avec une ingénierie moléculaire suffisante, l’un d’entre eux devrait suffire à transporter une protéine étrangère d’intérêt dans les mEV. Il est intéressant de noter que le Cfp-29 s’est imposé comme la meilleure option possible en raison de ses diverses caractéristiques 39,40,41,42. Nos données montrent en effet qu’il est suffisant pour transporter mCherry et EsxA (bien qu’EsxA ait besoin d’une petite balise à son extrémité C pour être plus stable) et les accumuler dans les mEVs. Récemment, en 2021, Tang et al. ont démontré que le Cfp-29 est une encapsuline capable de transporter des peroxydases de type peroxydase décolorante (DyP)40. Peut-être que le Cfp-29 peut également transporter d’autres protéines étrangères.
Nous avons exploré EsxA parce qu’il s’agit d’un immunogène Mtb proéminent 37,38,39, qu’il peut être protecteur en tant que vaccin sous-unitaire dans le modèle animal37,38,39, qu’il est de petite taille ; et son orthologue (MSMEG_0066) est étonnamment absent dans les EV Msm. Bien que nous n’en discutions pas ici, nous avons réussi à générer des R-mEVs pour quelques autres protéines Mtb (uniquement présentes dans Mtb et non codées par Msm), y compris l’antigène 85B (Rv 1886c). Dans le même temps, il est intéressant de noter que nous n’avons pas non plus réussi à générer des R-mEVs pour quelques autres, y compris Rv2660c et Rv0288 pleine longueur, peut-être parce que les protéines sont toxiques pour Msm. Nous concluons ainsi parce que, malgré le clonage des séquences nucléotidiques correctes et l’exécution de transformations répétées, aucun transformatant de Msm n’a émergé. Étant donné qu’EsxA nécessitait une balise 3XFLAG à son extrémité C-terminale pour une meilleure accumulation dans les mEVs, nous avons ajouté une balise 3XFLAG à toutes les autres protéines codées par Mtb que nous avons évaluées. Malgré l’étiquette, Msm n’a pas survécu, ce qui indique que certaines protéines Mtb restent toxiques malgré la fusion de l’étiquette. Nous supposons que dans ces cas, le clonage des régions d’épitopes prédites ou l’assemblage de plusieurs épitopes peut s’avérer payant (actuellement en cours d’évaluation dans notre laboratoire). Nous avons utilisé un petit liant de cinq acides aminés (quatre glycine et un sérine) entre Cfp-29 et mCherry/EsxA pour minimiser l’influence négative sur le repliement de la protéine d’intérêt44,45. Nous prévoyons que sans ce linker, le repliement de la protéine d’intérêt serait fortement influencé par le repliement de Cfp-29.
Ce protocole d’enrichissement est facilement extensible à l’enrichissement des véhicules électriques générés par VTT. Nous supposons également que l’enrichissement des mEV à partir de mycobactéries environnementales devrait également être réalisable avec ce protocole. Bien que le protocole soit simple et direct, il faut encore 7 à 8 jours pour enrichir les EV et les R-mEV générés par Msm. En revanche, il faut 15 à 20 jours pour l’enrichissement des véhicules électriques générés par le VTT. La concentration des VE sur un volume plus petit prend du temps et coûte cher, et nous explorons actuellement la filtration tangentielle et d’autres approches pour résoudre le problème du « temps ».
Enfin, nous utilisons le Sauton’s et non le 7H9 pour enrichir les mEV car le supplément 'ADC' contient de grandes quantités d’albumine sérique bovine qui peut interférer avec toute utilisation en aval des mEVs. Ce protocole peut être facilement étendu à tout autre milieu spécifique (par exemple, un milieu à faible teneur en fer) qui doit être utilisé pour évaluer la composition des mEV. Alternativement, pour certaines applications, à la dernière étape (c’est-à-dire à l’étape de protocole 2.7.5), au lieu du tampon HEPES, on pourrait utiliser une solution saline stérile lors de l’injection de la même chose à des souris ou à des cobayes pour diverses analyses in vivo.
Déclarations de divulgation
Tous les auteurs déclarent que ce travail de recherche a été mené en l’absence de toute relation/intérêt commercial ou financier qui pourrait être interprété comme un conflit d’intérêts potentiel.
Remerciements
Les auteurs remercient sincèrement le professeur Sarah M. Fortune d’avoir aimablement partagé le stock de M. smegmatis mc2155. Ils remercient également Servier Medical Art (smart.servier.com) d’avoir fourni quelques éléments de base pour la figure 1. Ils remercient sincèrement le soutien des autres membres du laboratoire pour leurs ajustements patients pendant la longue utilisation des agitateurs d’incubateur, des centrifugeuses et des ultracentrifugeuses pour l’enrichissement en mEV. Ils remercient également M. Surjeet Yadav, l’assistant de laboratoire, de toujours s’assurer que la verrerie et les consommables nécessaires étaient toujours disponibles et à portée de main. Enfin, ils remercient les équipes administratives, d’achat et financières de THSTI pour leur soutien constant et leur aide dans la bonne exécution du projet.
matériels
| Name | Company | Catalog Number | Comments |
| A2 type Biosafety Cabinet | Thermo Fisher Scientific, USA | 1300 series | |
| Bench top Centrifuge | Eppendorf, USA | 5810 R | |
| BstB1, HindIII, HpaI | NEB, USA | NEB | |
| Cell densitometer | GE Healthcare, USA | Ultraspec 10 | |
| Citric Acid | Sigma-Aldrich, Merck, USA | Sigma Aldrich | |
| Dibasic Potassium Phosphate | Sigma-Aldrich, Merck, USA | Sigma Aldrich | |
| Double Distilled Water | Merck, USA | ~18.2 MW/cm @ 25 oC | |
| Electroporation cuvettes | Bio-Rad, USA | 2 mm | |
| Electroporator | Bio-Rad, USA | Electroporator | |
| EsxA-specific Ab | Abcam, UK | Rabbit polyclonal | |
| Ferric Ammonium Citrate | Sigma-Aldrich, Merck, USA | Sigma Aldrich | |
| Floor model centrifuge | Thermo Fisher Scientific, USA | Sorvall RC6 plus | |
| Glassware | Borosil, INDIA | 1 L Erlenmeyer flasks | |
| Glycerol | Sigma-Aldrich, Merck, USA | Sigma Aldrich | |
| HEPES and Sodium Chloride | Sigma-Aldrich, Merck, USA | Sigma Aldrich | |
| Incubator shakers | Thermo Fisher Scientific, USA | MaxQ 6000 & 8000 | |
| L-Asparagine | Sigma-Aldrich, Merck, USA | Sigma Aldrich | |
| Luria Bertani Broth and Agar, Miller | Hi Media, INDIA | Hi Media | |
| Magnesium Sulfate Heptahydrate | Sigma-Aldrich, Merck, USA | Sigma Aldrich | |
| Magnetic stirrer | Tarsons, INDIA | Tarsons | |
| mCherry-specific Ab | Abcam, UK | Rabbit monoclonal | |
| Microwave | LG, INDIA | MC3286BLT | |
| Middlebrook 7H9 Broth | BD, USA | Difco Middlebrook 7H9 Broth | |
| Middlebrook ADC enrichment | BD, USA | BBL Middlebrook ADC enrichment | |
| Nanodrop | Thermo Fisher Scientific, USA | Spectronic 200 UV-Vis | |
| NEB5a | NEB, USA | a derivative of DH5a | |
| Optiprep (Iodixanol) | Merck, USA | Available as 60% stock solution (in water) | |
| PCR purification kit | Hi Media, INDIA | Hi Media | |
| pH Meter | Mettler Toledo, USA | Mettler Toledo | |
| Plasmid DNA mini kit | Hi Media, INDIA | Hi Media | |
| Plate incubator | Thermo Fisher Scientific, USA | New Series | |
| Plasmid pMV261 | Addgene, USA * *The plasmid is no more available in this plasmid bank | Shuttle vector | |
| Proof-reading DNA Polymerase | Thermo Fisher Scientific, USA | Phusion DNA Plus Polymerase | |
| Q5 Proof-reading DNA Polymerase | NEB, USA | NEB | |
| Refrigerated circulating water bath | Thermo Fisher Scientific, USA | R20 | |
| Middlebrock 7H11 Agar base | BD, USA | BBL Seven H11 Agar base | |
| SOC broth | Hi Media, INDIA | Hi Media | |
| Sodium Hydroxide | Sigma-Aldrich, Merck, USA | Sigma Aldrich | |
| T4 DNA Ligase | NEB, USA | NEB | |
| Tween-80 | Sigma-Aldrich, Merck, USA | Sigma Aldrich | |
| Ultracentrifuge | Beckman Coulter, USA | Optima L100K | |
| Ultracentrifuge tubes - 14 mL | Beckman Coulter, USA | Polyallomer type – ultra clear type in SW40Ti rotor | |
| Ultracentrifuge tubes - 38 mL | Beckman Coulter, USA | Polypropylene type– cloudy type for SW28 rotor | |
| Ultrasonics cleaning waterbath sonicator | Thermo Fisher Scientific, USA | Sonicator - bench top model | |
| 0.22 µm Disposable filters | Thermo Fisher Scientific, USA | Nunc-Nalgene | |
| 30-kDa Centricon concentrators | Merck, USA | Amicon Ultra centrifugal filters - Millipore | |
| 3X FLAG antibody | Sigma-Aldrich, Merck, USA | Sigma Aldrich | |
| 400 mL Centrifuge bottles | Thermo Fisher Scientific, USA | Nunc-Nalgene | |
| 50 mL Centrifuge tubes | Corning, USA | Sterile, pre-packed | |
| Bacteria | |||
| Strain | |||
| Escherichia coli | NEB, USA | NEB 5-alpha (a derivative of DH5α). | |
| Msm expressing cfp29::mCherry | This study | MC2 155 | |
| Msm expressing cfp29::esxA | This study | MC2 155 | |
| Msm expressing cfp29::esxA::3X FLAG | This study | MC2 155 | |
| Mycobacterium smegmatis (Msm) | Prof. Sarah M. Fortune, Harvard Univ, USA | MC2 155 |
Références
- . Global Tuberculosis Report 2022. , (2022).
- Luca, S., Mihaescu, T. History of BCG vaccine. Mædica. 8 (1), 53-58 (2013).
- Palmer, C. E., Long, M. W. Effects of infection with atypical mycobacteria on BCG vaccination and tuberculosis. The American Review of Respiratory Disease. 94 (4), 553-568 (1966).
- Brandt, L., et al. Failure of the Mycobacterium bovis BCG vaccine: some species of environmental mycobacteria block multiplication of BCG and induction of protective immunity to tuberculosis. Infection and Immunity. 70 (2), 672-678 (2002).
- Andersen, P., Doherty, T. M. The success and failure of BCG - implications for a novel tuberculosis vaccine. Nature Reviews. Microbiology. 3 (8), 656-662 (2005).
- Kumar, P. A perspective on the success and failure of BCG. Frontiers in Immunology. 12, 778028 (2021).
- Fine, P. E. Variation in protection by BCG: implications of and for heterologous immunity. Lancet. 346 (8986), 1339-1345 (1995).
- Triccas, J. A. Recombinant BCG as a vaccine vehicle to protect against tuberculosis. Bioengineered Bugs. 1 (2), 110-115 (2010).
- Dietrich, G., Viret, J. -. F., Hess, J. Mycobacterium bovis BCG-based vaccines against tuberculosis: novel developments. Vaccine. 21 (7-8), 667-670 (2003).
- Singh, V. K., Srivastava, R., Srivastava, B. S. Manipulation of BCG vaccine: a double-edged sword. European Journal of Clinical Microbiology & Infectious Diseases. 35 (4), 535-543 (2016).
- Bastos, R. G., Borsuk, S., Seixas, F. K., Dellagostin, O. A. Recombinant Mycobacterium bovis BCG. Vaccine. 27 (47), 6495-6503 (2009).
- Kaufmann, S. H. E., Gengenbacher, M. Recombinant live vaccine candidates against tuberculosis. Current Opinion in Biotechnology. 23 (6), 900-907 (2012).
- Yuan, X., et al. A live attenuated BCG vaccine overexpressing multistage antigens Ag85B and HspX provides superior protection against Mycobacterium tuberculosis infection. Applied Microbiology and Biotechnology. 99 (24), 10587-10595 (2015).
- Vartak, A., Sucheck, S. J. Recent advances in subunit vaccine carriers. Vaccines. 4 (2), 12 (2016).
- Lindenstrøm, T., et al. Tuberculosis subunit vaccination provides long-term protective immunity characterized by multifunctional CD4 memory T cells1. The Journal of Immunology. 182 (12), 8047-8055 (2009).
- Liu, Y., et al. A subunit vaccine based on rH-NS induces protection against Mycobacterium tuberculosis infection by inducing the Th1 immune response and activating macrophages. Acta Biochimica et Biophysica Sinica. 48 (10), 909-922 (2016).
- Ning, H., et al. Subunit vaccine ESAT-6:c-di-AMP delivered by intranasal route elicits immune responses and protects against Mycobacterium tuberculosis infection. Frontiers in Cellular and Infection Microbiology. 11, 647220 (2021).
- Woodworth, J. S., et al. A Mycobacterium tuberculosis-specific subunit vaccine that provides synergistic immunity upon co-administration with Bacillus Calmette-Guérin. Nature Communications. 12 (1), 6658 (2021).
- Moyle, P. M., Toth, I. Modern subunit vaccines: development, components, and research opportunities. ChemMedChem. 8 (3), 360-376 (2013).
- Baxter, D. Active and passive immunity, vaccine types, excipients and licensing. Occupational Medicine. 57 (8), 552-556 (2007).
- Lee, W. -. H., et al. Vaccination with Klebsiella pneumoniae-derived extracellular vesicles protects against bacteria-induced lethality via both humoral and cellular immunity. Experimental & Molecular Medicine. 47 (9), e183-e183 (2015).
- Micoli, F., et al. Comparative immunogenicity and efficacy of equivalent outer membrane vesicle and glycoconjugate vaccines against nontyphoidal Salmonella. Proceedings of the National Academy of Sciences. 115 (41), 10428-10433 (2018).
- Obiero, C. W., et al. A phase 2a randomized study to evaluate the safety and immunogenicity of the 1790GAHB generalized modules for membrane antigen vaccine against Shigella sonnei administered intramuscularly to adults from a shigellosis-endemic country. Frontiers in Immunology. 8, 1884 (2017).
- Sedaghat, M., et al. Evaluation of antibody responses to outer membrane vesicles (OMVs) and killed whole cell of Vibrio cholerae O1 El Tor in immunized mice. Iranian Journal of Microbiology. 11 (3), 212-219 (2019).
- Adriani, R., Mousavi Gargari, S. L., Nazarian, S., Sarvary, S., Noroozi, N. Immunogenicity of Vibrio cholerae outer membrane vesicles secreted at various environmental conditions. Vaccine. 36 (2), 322-330 (2018).
- Roier, S., et al. Intranasal immunization with nontypeable Haemophilus influenzae outer membrane vesicles induces cross-protective immunity in mice. PLOS ONE. 7 (8), e42664 (2012).
- Furuyama, N., Sircili, M. P. Outer membrane vesicles (OMVs) produced by gram-negative bacteria: structure, functions, biogenesis, and vaccine application. BioMed Research International. 2021, e1490732 (2021).
- Buzas, E. I. The roles of extracellular vesicles in the immune system. Nature Reviews Immunology. 23 (4), 236-250 (2022).
- Cai, W., et al. Bacterial outer membrane vesicles, a potential vaccine candidate in interactions with host cells based. Diagnostic Pathology. 13 (1), 95 (2018).
- Prados-Rosales, R., et al. Mycobacteria release active membrane vesicles that modulate immune responses in a TLR2-dependent manner in mice. The Journal of Clinical Investigation. 121 (4), 1471-1483 (2011).
- Bai, X., Findlow, J., Borrow, R. Recombinant protein meningococcal serogroup B vaccine combined with outer membrane vesicles. Expert Opinion on Biological Therapy. 11 (7), 969-985 (2011).
- Nagaputra, J. C., et al. Neisseria meningitidis native outer membrane vesicles containing different lipopolysaccharide glycoforms as adjuvants for meningococcal and nonmeningococcal antigens. Clinical and Vaccine Immunology: CVI. 21 (2), 234-242 (2014).
- Echeverria-Valencia, G., Flores-Villalva, S., Espitia, C. I. Virulence factors and pathogenicity of Mycobacterium. Mycobacterium - Research and Development. IntechOpen. , (2017).
- Prados-Rosales, R., Brown, L., Casadevall, A., Montalvo-Quirós, S., Luque-Garcia, J. L. Isolation and identification of membrane vesicle-associated proteins in Gram-positive bacteria and mycobacteria. MethodsX. 1, 124-129 (2014).
- Lee, J., et al. Proteomic analysis of extracellular vesicles derived from Mycobacterium tuberculosis. Proteomics. 15 (19), 3331-3337 (2015).
- Das, S., et al. Development of DNA aptamers to visualize release of mycobacterial membrane-derived extracellular vesicles in infected macrophages. Pharmaceuticals. 15 (1), 45 (2022).
- Brandt, L., Elhay, M., Rosenkrands, I., Lindblad, E. B., Andersen, P. ESAT-6 subunit vaccination against Mycobacterium tuberculosis. Infection and Immunity. 68 (2), 791-795 (2000).
- Kim, W. S., Kim, H., Kwon, K. W., Cho, S. -. N., Shin, S. J. Immunogenicity and vaccine potential of InsB, an ESAT-6-like antigen identified in the highly virulent Mycobacterium tuberculosis Beijing K strain. Frontiers in Microbiology. 10, 220 (2019).
- Valizadeh, A., et al. Evaluating the performance of PPE44, HSPX, ESAT-6 and CFP-10 factors in tuberculosis subunit vaccines. Current Microbiology. 79 (9), 260 (2022).
- Tang, Y., et al. Cryo-EM structure of Mycobacterium smegmatis DyP-loaded encapsulin. Proceedings of the National Academy of Sciences. 118 (16), (2021).
- Rosenkrands, I., et al. Identification and characterization of a 29-kilodalton protein from Mycobacterium tuberculosis culture filtrate recognized by mouse memory effector cells. Infection and Immunity. 66 (6), 2728-2735 (1998).
- Gu, S., et al. Comprehensive proteomic profiling of the membrane constituents of a Mycobacterium tuberculosis strain. Molecular & cellular proteomics: MCP. 2 (12), 1284-1296 (2003).
- Xiong, Y., Chalmers, M. J., Gao, F. P., Cross, T. A., Marshall, A. G. Identification of Mycobacterium tuberculosis H37Rv integral membrane proteins by one-dimensional gel electrophoresis and liquid chromatography electrospray ionization tandem mass spectrometry. Journal of Proteome Research. 4 (3), 855-861 (2005).
- Chen, X., Zaro, J. L., Shen, W. -. C. Fusion protein linkers: property, design and functionality. Advanced Drug Delivery Reviews. 65 (10), 1357-1369 (2013).
- Klein, J. S., Jiang, S., Galimidi, R. P., Keeffe, J. R., Bjorkman, P. J. Design and characterization of structured protein linkers with differing flexibilities. Protein Engineering, Design and Selection. 27 (10), 325-330 (2014).
- Green, M. R., Sambrook, J. . Molecular cloning, a laboratory manual, 4th Edition. , (2012).
- Atmakuri, K., Ding, Z., Christie, P. J. VirE2, a Type IV secretion substrate, interacts with the VirD4 transfer protein at cell poles of Agrobacterium tumefaciens. MolecularMicrobiology. 49 (6), 1699-1713 (2003).
- Woodworth, J. S., et al. A Mycobacterium tuberculosis-specific subunit vaccine that provides synergistic immunity upon co-administration with Bacillus Calmette-Guérin. Nature Communications. 12 (1), 6658 (2021).
- Khademi, F., Derakhshan, M., Yousefi-Avarvand, A., Tafaghodi, M., Soleimanpour, S. Multi-stage subunit vaccines against Mycobacterium tuberculosis: an alternative to the BCG vaccine or a BCG-prime boost. Expert Review of Vaccines. 17 (1), 31-44 (2018).
- Prior, J. T., et al. Bacterial-derived outer membrane vesicles are potent adjuvants that drive humoral and cellular immune responses. Pharmaceutics. 13 (2), 131 (2021).
- Tan, K., Li, R., Huang, X., Liu, Q. Outer membrane vesicles: current status and future direction of these novel vaccine adjuvants. Frontiers in Microbiology. 9, 783 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon