Method Article
Quantification au niveau global des modifications post-traductionnelles des histones dans un modèle de culture cellulaire 3D de tissu hépatique
Dans cet article
Résumé
Ce protocole décrit comment un système de culture cellulaire tridimensionnel peut être utilisé pour modéliser, traiter et analyser les modifications de la chromatine dans un état quasi physiologique.
Résumé
Les cultures plates de cellules de mammifères sont une approche in vitro largement utilisée pour comprendre la physiologie cellulaire, mais ce système est limité dans la modélisation des tissus solides en raison de la réplication cellulaire anormalement rapide. Ceci est particulièrement difficile lors de la modélisation de la chromatine mature, car les cellules à réplication rapide sont fréquemment impliquées dans la réplication de l’ADN et ont une population polyploïde hétérogène. Vous trouverez ci-dessous un flux de travail pour la modélisation, le traitement et l’analyse des modifications de la chromatine au repos à l’aide d’un système de culture cellulaire tridimensionnelle (3D). En utilisant ce protocole, les lignées cellulaires de carcinome hépatocellulaire sont cultivées sous forme de sphéroïdes 3D reproductibles dans un incubateur fournissant une diffusion active des nutriments et de faibles forces de cisaillement. Le traitement au butyrate de sodium et au succinate de sodium a induit une augmentation de l’acétylation des histones et de la succinylation, respectivement. Les augmentations des niveaux d’acétylation des histones et de succinylation sont associées à un état de chromatine plus ouvert. Les sphéroïdes sont ensuite collectés pour isoler les noyaux cellulaires, à partir desquels les protéines d’histones sont extraites pour l’analyse de leurs modifications post-traductionnelles. L’analyse des histones est réalisée par chromatographie liquide couplée en ligne à la spectrométrie de masse en tandem, suivie d’un pipeline de calcul interne. Enfin, des exemples de représentation de données pour étudier la fréquence et l’occurrence des marques combinatoires d’histones sont montrés.
Introduction
Depuis la fin du19e siècle, les systèmes de culture cellulaire ont été utilisés comme modèle pour étudier la croissance et le développement des cellules en dehors du corps humain 1,2. Leur utilisation a également été étendue pour étudier le fonctionnement des tissus et des organes dans des contextes sains et malades 1,3. Les cellules en suspension (p. ex. les cellules sanguines) se développent dans des boîtes de Pétri ou des flacons de façon transparente et interchangeable, car elles ne s’assemblent pas dans des structures tridimensionnelles (3D) in vivo. Les cellules dérivées d’organes solides peuvent se développer dans des systèmes de culture bidimensionnels (2D) ou 3D. En culture 2D, les cellules sont cultivées dans une monocouche qui adhère à une surface plane 2,4. Les systèmes de culture cellulaire 2D sont caractérisés par une croissance exponentielle et un temps de doublement rapide, généralement de 24 h à quelques jours5. Les cellules des systèmes 3D se développent pour former des interactions cellules-cellules complexes en modélisant plus étroitement des conglomérats tissulaires, et elles se caractérisent par leur capacité à atteindre un équilibre dynamique où leur temps de doublement peut atteindre 1 mois ou plus5.
Présenté dans cet article est une méthodologie innovante pour cultiver des sphéroïdes 3D dans des systèmes de culture cellulaire rotatifs qui simulent une gravité réduite6. Il s’agit d’un dérivé simplifié d’un système de culture cellulaire introduit par la NASA dans les années 19907. Cette approche minimise les forces de cisaillement, qui se produisent dans les méthodes existantes comme la filature des flacons, et augmente la reproductibilité des sphéroïdes6. En outre, le bioréacteur rotatif augmente la diffusion active des nutriments, minimisant la formation nécrotique qui se produit dans des systèmes tels que la culture cellulaire suspendue où l’échange de milieux n’est pas pratique6. De cette façon, les cellules se développent la plupart du temps sans être dérangées, ce qui permet la formation de caractéristiques structurelles et physiologiques associées à la croissance des cellules dans les tissus. Les hépatocytes C3A (HepG2/C3A) cultivés de cette manière avaient non seulement des organites ultrastructuraux, mais produisaient également des quantités d’ATP, d’adénylate kinase, d’urée et de cholestérol comparables aux niveaux observés in vivo 1,2. En outre, les cellules cultivées en 2D par rapport aux systèmes de culture cellulaire .3D présentent des modèles d’expression génique différents8. L’analyse de l’expression génique des hépatocytes C3A cultivés sous forme de sphéroïdes 3D a montré que ces cellules exprimaient un large éventail de protéines spécifiques au foie, ainsi que des gènes impliqués dans des voies clés qui régulent la fonction hépatique8. Des publications antérieures ont démontré les différences entre les protéomes de cellules à croissance exponentielle en culture 2D et les cellules à l’équilibre dynamique dans les cultures sphéroïdes 3D5. Ces différences incluent le métabolisme cellulaire, qui à son tour affecte la structure, la fonction et la physiologie de la cellule5. Le protéome des cellules cultivées en culture 2D était plus enrichi en protéines impliquées dans la réplication cellulaire, tandis que le protéome des sphéroïdes 3D était plus enrichi en fonctionnalité hépatique5.
Le taux de réplication plus lent des cellules cultivées sous forme de sphéroïdes 3D modélise plus précisément des phénomènes spécifiques associés à l’état et aux modifications de la chromatine (par exemple, coupure d’histone9). L’écrêtage des histones est une modification post-traductionnelle irréversible des histones (PTM) qui provoque un clivage protéolytique d’une partie de la queue N-terminale de l’histone. Bien que sa fonction biologique soit encore en discussion 10,11,12,13, il est clair que sa présence dans les cellules primaires et les tissus hépatiques est modélisée par des cellules HepG2/C3A cultivées sous forme de sphéroïdes, mais pas sous forme de cellules plates 9. Ceci est essentiel, car l’état et les modifications de la chromatine régulent la lecture de l’ADN principalement en modulant l’accessibilité aux gènes et donc leur expression14. Les PTM d’histone influencent soit directement l’état de la chromatine en affectant la charge nette des nucléosomes où les histones sont assemblées, soit indirectement en recrutant des écrivains, des lecteurs et des gommes à base de chromatine14. Des centaines de PTM d’histones ont été identifiés à ce jour15, renforçant l’hypothèse selon laquelle la chromatine héberge un « code d’histone » utilisé par la cellule pour interpréter l’ADN16. Cependant, l’identification d’une myriade de combinaisons ptm15 et la découverte que les combinaisons de PTM d’histones ont souvent des fonctions biologiques différentes des PTM présents isolément (par exemple, Fischle et al.17), soulignent que davantage de travail est nécessaire pour déchiffrer le « code des histones ».
À l’heure actuelle, l’analyse ptm des histones est soit basée sur des techniques utilisant des anticorps (par exemple, transferts de Western, immunofluorescence ou immunoprécipitation de la chromatine suivie d’un séquençage [ChIP-seq]) ou la spectrométrie de masse (SEP). Les techniques à base d’anticorps ont une sensibilité élevée et peuvent fournir des informations détaillées sur la localisation des marques d’histones à l’échelle du génome, mais sont souvent limitées dans l’étude des PTM rares ou des PTM présents dans des combinaisons 18,19,20. La SEP est plus adaptée à l’identification et à la quantification à haut débit et non biaisées des modifications protéiques uniques et coexistantes, en particulier les protéines histones 18,19,20. Pour ces raisons, ce laboratoire et plusieurs autres ont optimisé le pipeline de SEP pour l’analyse des peptides d’histones (SEP ascendante), des queues d’histones intactes (SEP moyennement descendante) et des protéines d’histones pleine longueur (MS descendante)21,22,23.
Vous trouverez ci-dessous un flux de travail pour la culture des sphéroïdes HepG2 / C3A et leur préparation à l’analyse peptidique des histones (MS ascendante) par chromatographie nano-liquide, couplée en ligne à la spectrométrie de masse en tandem (nLC-MS / MS). Une culture cellulaire 2D a été cultivée et les cellules ont été récoltées et transférées dans un bioréacteur où elles ont commencé à former des sphéroïdes (Figure 1). Après 18 jours de culture, les sphéroïdes ont été traités avec du butyrate de sodium ou du succinate de sodium pour augmenter les abondances relatives d’acétylation et de succinylation des histones. Notamment, les cultures 3D peuvent être traitées avec des composés génotoxiques tout aussi bien que leurs équivalents de culture plate; en fait, des publications récentes soulignent que la réponse toxicologique des cellules en culture 3D est plus similaire aux tissus primaires que celles en culture plate 2D24,25. Les cellules ont ensuite été collectées à des moments précis et l’isolement nucléaire a été effectué. Ensuite, les histones ont été extraites et dérivées avec de l’anhydride propionique avant et après la digestion de la trypsine selon un protocole développé pour la première fois par Garcia et al.26. Cette procédure génère des peptides d’une taille appropriée pour la séparation en ligne avec chromatographie en phase inversée (C18) et la détection avec MS. Enfin, les peptides d’histones ont été identifiés et quantifiés, et les données générées ont été représentées de multiples façons pour une interprétation biologique plus complète.
Protocole
1. Préparation des tampons et des réactifs
- Milieux de croissance cellulaire (pour les cellules HepG2/C3A) : Ajouter le sérum fœtal bovin (FBS) (10 % v/v), les acides aminés non essentiels (1 % v/v), le supplément de L-glutamine (1 % v/v) et la pénicilline/streptomycine (0,5 % v/v) au milieu d’aigle modifié de Dulbecco (DMEM, contenant 4,5 g/L de glucose). Les milieux de croissance sont conservés à 4 °C pendant un maximum de 2 semaines.
- Solution de butyrate de sodium (NaBut) de 200 mM : Pour préparer 10 mL, remettre en suspension 220,18 mg de NaBut dans 10 mL de ddH2O. Conserver 1 mL d’aliquotes à -20 °C. Avant le traitement cellulaire, filtrer la solution à l’aide d’un filtre à seringue de 0,45 mm et ajouter 1 mL de la solution filtrée à 9 mL de milieu de croissance cellulaire pour une concentration de travail de 20 mM.
- Solution de succinate de sodium (NaSuc) de 100 mM : Pour préparer 10 mL, remettre en suspension 162,05 mg de NaSuc dans 10 mL de ddH2O. Conserver 1 mL d’aliquotes à -20 °C. Avant le traitement cellulaire, filtrer la solution à l’aide d’un filtre à seringue de 0,45 mm et ajouter 1 mL de la solution filtrée à 9 mL de milieu de croissance cellulaire pour une concentration de travail de 10 mM.
- Froid 0,2 M H2SO4 : Pour préparer 1 L, ajouter 10 mL deH2SO4 concentré à 990 mL d’eau de qualité CLHP. Conserver à 4 °C.
- Acétone froide + 0,1 % d’acide chlorhydrique (HCl) : Ajouter du HCl concentré (0,1 % v/v) à l’acétone. Conserver à 4 °C.
- Solution NH4HCO3 à 100 mM, pH 8,0 : Pour préparer 1 L, remettre en suspension 7,91 g de NH4HCO3 dans 1 L d’eau de qualité CLHP. Conserver 50 mL d’aliquotes à -20 °C.
- Solution d’acide trifluoroacétique (TFA) à 0,1 % : Ajouter de l’AGT concentré (0,1 % v/v) à l’eau de qualité CLHP. Conserver à 4 °C.
- Solution d’acétonitrile à 60 % / solution de TFA à 0,1 % : Ajoutez de l’acétonitrile de qualité HPLC (60 % v/v) à de l’eau de qualité HPLC. Ensuite, ajoutez du TFA concentré (0,1 % v/v) à cette solution. Conserver à 4 °C.
- Acétonitrile de qualité HPLC à 2 % + 0,1 % d’acide formique : Ajoutez de l’acétonitrile de qualité HPLC (2 % v/v) à de l’eau de qualité HPLC. Ensuite, ajoutez de l’acide formique concentré (0,1% v/v) à cette solution.
- 80 % d’acétonitrile de qualité HPLC + 0,1 % d’acide formique : Ajoutez de l’acétonitrile de qualité HPLC (80 % v/v) à de l’eau de qualité HPLC. Ensuite, ajoutez de l’acide formique concentré (0,1% v/v) à cette solution.
2. Préparation du système de culture 3D
REMARQUE: Différentes cellules, primaires ou immortalisées, ont des propriétés de culture différentes, de sorte que la formation de sphéroïdes peut différer d’un type de cellule à l’autre. Ce protocole a été établi pour la formation de sphéroïdes HepG2/C3A à l’aide de bioréacteurs et d’un système innovant de culture cellulaire 3D.
- En utilisant des milieux de croissance standard, cultivez des cellules en monocouche jusqu’à ce qu’elles soient confluentes à 80%.
- Laver les cellules avec HBSS (5 mL pour une fiole de 75 cm2 ) et incuber les cellules avec 5 mL de trypsine-EDTA à 0,05 % diluées dans du HBSS (dilution 1:2) pendant 5 min à 37 oC avec 5 % de CO2.
- Vérifiez le détachement cellulaire au microscope et neutralisez la trypsine en ajoutant 3 mL de sérum bovin fœtal (FBS) ou de milieu de croissance (contenant 5% à 10% de FBS).
- Compter le nombre de cellules et diluer la suspension cellulaire pour obtenir 1 x 106 cellules dans un volume maximum de 1,5 mL.
- Équilibrager une plaque de fond ronde de 24 puits à fixation ultra-faible (contenant plusieurs micro-puits par puits) en lavant les puits avec 0,5 mL de milieu de croissance. Centrifugez la plaque pendant 5 min à 3 000 x g pour éliminer les bulles d’air de la surface du puits.
- Transférer la suspension de la cellule dans la plaque et centrifuger pendant 3 min à 120 x g.
- Incuber la plaque pendant 24 h à 37 oC avec 5% de CO2 pour la formation de sphéroïdes. Pendant ce temps, équilibrez le bioréacteur en remplissant la chambre d’humidité avec 25 mL d’eau stérile et la chambre cellulaire avec 9 mL de milieu de croissance.
- Incuber le bioréacteur, en tournant dans l’incubateur à clinostat, pendant 24 h à 37 oC avec 5% de CO2.
3. Croissance des sphéroïdes dans les bioréacteurs
REMARQUE: Pour préserver la structure des sphéroïdes, de larges pointes d’alésage sont utilisées pour manipuler les structures 3D.
- Détachez les sphéroïdes de la plaque de fixation ultra-basse en pipetant doucement de haut en bas avec des embouts d’alésage de 1 mL de large et transférez-les dans un plat traité par culture tissulaire.
- Laver la plaque avec 0,5 mL de milieu de croissance préchauffé et transférer dans le même plat.
- Évaluer la qualité des sphéroïdes par microscopie et sélectionner des sphéroïdes suffisamment formés. Les sphéroïdes de bonne qualité ont une taille, une compacité et une rondeur uniformes.
- Transférer les sphéroïdes dans des bioréacteurs équilibrés remplis de 5 mL de milieux de croissance frais. Après avoir transféré les sphéroïdes, remplissez complètement le bioréacteur avec des milieux de croissance frais.
- Placez le bioréacteur dans l’incubateur à clinostat et ajustez la vitesse de rotation à 10-11 tr / min.
- Échangez des supports de croissance tous les 2 à 3 jours en retirant 10 mL d’anciens supports et en les remplaçant par 10 mL de nouveaux supports.
- Ajustez la vitesse de rotation en fonction de la croissance des sphéroïdes, en augmentant à mesure que les sphéroïdes grandissent en taille et en nombre.
- Après 18 jours de culture, les sphéroïdes sont prêts pour le traitement et/ou la collecte
4. Traitement et collecte des sphéroïdes
REMARQUE: Dans ce protocole, les sphéroïdes HepG2 / C3A sont traités avec du butyrate de sodium (NaBut) et du succinate de sodium (NaSuc) pour évaluer les niveaux de marques d’histones contenant de l’acétylation et de la succinylation, respectivement.
- Préparer les milieux de croissance avec la concentration de travail appropriée du composé (p. ex., 20 mM de NaBut ou 10 mM de NaSuc). Échangez le milieu dans le bioréacteur avec le milieu traité.
REMARQUE: Pour établir une condition de contrôle, soit recueillir suffisamment de sphéroïdes pour les expériences avant d’ajouter le traitement, soit désigner un bioréacteur pour les sphéroïdes non traités. - Prélever les sphéroïdes pour l’analyse protéomique après un temps de traitement suffisant (p. ex., 48 h à 1 semaine pour le traitement NaSuc ou 48-72 h pour le traitement NaBut).
REMARQUE: Pour l’extraction des histones à l’aide de ce protocole, six à huit sphéroïdes contenant environ 1 x 106 cellules ont été collectés.- Retirez 3 à 5 mL de média du bioréacteur par l’orifice supérieur.
- Ouvrez l’orifice avant et utilisez une pointe d’alésage de 1 mL de large pour retirer les sphéroïdes et les placer dans des tubes de microcentrifugation.
- Centrifuger les sphéroïdes à 100 x g pendant 5 min et jeter le milieu.
REMARQUE: Les milieux dans le bioréacteur peuvent être changés et le bioréacteur peut être retourné à l’incubateur pour un temps de traitement ou de récupération supplémentaire. - Lavez les sphéroïdes avec 200 μL de HBSS pour éliminer le FBS. Centrifuger à 100 x g pendant 5 min et retirer le surnageant.
REMARQUE: Les sphéroïdes peuvent être stockés à -80 °C jusqu’au traitement.
5. Extraction des histones
REMARQUE: Les nombreux résidus d’acides aminés basiques présents dans les histones leur permettent d’interagir étroitement avec l’ADN, qui a une colonne vertébrale d’acide phosphorique. Parce que les histones sont parmi les protéines les plus basiques du noyau, lorsqu’elles sont extraites avec de l’acide sulfurique glacé (0,2 M H2SO4), la contamination est minimale. Les protéines non histones précipiteront dans l’acide fort. L’acide trichloroacétique (TCA) hautement concentré dilué à une concentration finale de 33% est ensuite utilisé pour précipiter les histones de l’acide sulfurique. Conservez tous les échantillons, tubes et réactifs sur la glace pendant toute l’extraction des histones.
- Ajouter cinq volumes (~100 μL) de froid 0,2 MH2SO4 à la pastille cellulaire (~10-20 μL), et pipeter de haut en bas pour perturber la pastille et libérer des histones.
- Incuber les tubes jusqu’à 4 h à rotation constante ou agitation à 4 °C.
REMARQUE: Pour les échantillons remis en suspension qui ont un volume supérieur à 500 μL, une incubation de 2 h est suffisante pour extraire les histones (une incubation plus longue peut entraîner l’extraction d’autres protéines nucléaires de base). Pour les échantillons remis en suspension d’un volume inférieur à 200 μL, une incubation de 4 h est nécessaire pour un meilleur rendement. - Centrifuger à 3 400 x g pendant 5 min à 4 °C. Collectez le surnageant dans un nouveau tube et jetez la pastille plus tard.
- Ajouter le TCA concentré à froid de telle sorte qu’il constitue un TCA final de 25% à 33% v / v (par exemple, 40-60 μL de TCA froid: 120 μL de surnageant) et mélanger en inversant le tube plusieurs fois.
- Incuber les tubes pendant au moins 1 h à rotation constante ou en agitant à 4 °C.
REMARQUE: Pour les granulés de départ de plus petite taille, une incubation de nuit est recommandée. - Centrifuger à 3 400 x g pendant 5 min à 4 oC. Jeter le surnageant par pipetage. Aspirer soigneusement le surnageant; ne touchez pas les côtés du tube ou de la pastille.
REMARQUE: Les histones se déposent à la fois sur les côtés et au fond du tube. La pastille blanche insoluble formée tout en bas du tube contient principalement des protéines non histones et d’autres biomolécules. - Laver le tube (parois et granulés) avec de l’acétone froide + 0,1% HCl à l’aide d’une pipette Pasteur en verre (~500 μL/tube).
- Centrifuger à 3 400 x g pendant 5 min à 4 oC. Jeter le surnageant en retournant le tube.
- Lavez le tube (parois et granulés) avec de l’acétone 100% froide à l’aide d’une pipette Pasteur en verre (~500 μL/tube). Centrifuger à 3 400 x g pendant 5 min à 4 oC.
- Jetez le surnageant en retournant le tube et pipettez l’acétone restante. Ouvrez le couvercle et séchez l’échantillon à l’air libre sur le banc pendant environ 20 min.
- Procéder à la propionylation ou conserver les échantillons à -80 °C jusqu’à leur utilisation.
6. Premier cycle de dérivatisation
REMARQUE: L’utilisation de la trypsine pour digérer les protéines d’histone conduit à des peptides excessivement petits qui sont difficiles à identifier en utilisant les configurations protéomiques traditionnelles. Pour cette raison, l’anhydride propionique est utilisé pour dévatiser chimiquement les groupes ɛ-amino des résidus de lysine non modifiée et monométhyl. Cela limite la protéolyse de la trypsine aux résidus d’arginine C-terminal. Pour les échantillons dans des plaques de 96 puits, l’utilisation de pipettes et de réservoirs multicanaux pour le ramassage des réactifs est recommandée (figure 2A). La dérivatisation est également réalisée après digestion pour marquer les N-termini libres des peptides augmentant l’hydrophobicité peptidique et donc la rétention chromatographique en phase inversée.
- Remettre en suspension des échantillons dans 20 μL d’acétonitrile à 15 % à 20 % dans du bicarbonate d’ammonium de 100 mM (pH 8,0). Vortex pendant 15 s, puis rotation à 1 000 x g pendant 30 s.
- S’il y a huit échantillons ou plus, transférer chaque échantillon remis en suspension dans une plaque de 96 puits.
REMARQUE : Si les échantillons n’ont pas été transférés sur une plaque de 96 puits, une pipette à canal unique peut être utilisée aux étapes 6.3-6.7, 7.1-7.5 et 8.1-8.5. - Sous le capot, ajouter 2 μL d’anhydride propionique à l’aide d’une pipette multicanal. Mélanger en pipetant de haut en bas 5x.
- Ajouter rapidement 10 μL d’hydroxyde d’ammonium à l’aide d’une pipette multicanal. Mélanger en pipetant de haut en bas 5x.
REMARQUE: L’acide propionique est un produit de la réaction entre l’anhydride propionique et les amines libres de peptides et peut diminuer le pH de l’échantillon. Le pH 8 peut être rétabli en ajoutant de l’hydroxyde d’ammonium à l’échantillon dans un rapport de 1:5 (v/v). - Assurez-vous que le pH est de 8 à l’aide d’un papier d’essai du pH. Si le pH < 8, ajuster en ajoutant 1 μL d’hydroxyde d’ammonium. Si le pH > 8, ajuster en ajoutant 1 μL d’acide formique ou acétique. Lorsque le pH > 10, il est possible de marquer d’autres résidus d’acides aminés avec un pKa plus élevé.
- Incuber à température ambiante pendant 10 min.
- Répétez les étapes 6.3 à 6.6. Un double tour de propionylation des histones assure une efficacité de réaction presque complète.
- Sécher la plaque sous vide jusqu’à ce que tous les puits soient complètement séchés (~9 h).
- Procéder à la digestion de la trypsine ou conserver les échantillons à -80 °C jusqu’à l’utilisation.
7. Digestion des histones
REMARQUE: Les histones sont digérées en peptides à l’aide de trypsine, qui coupe du côté carboxyle de l’arginine et des résidus de lysine. Cependant, comme la propionylation modifie les résidus de lysine, seuls les résidus d’arginine sont clivés (figure 2B).
- Préparer la solution de trypsine (25 ng/μL dans 50 mM NH4HCO3, pH 8,0). Ajouter 20 μL de trypsine (500 ng) à chaque échantillon.
- Pour préparer une solution de NH4HCO3 de 50 mM, diluer 100 mM de solution NH4HCO3 1:1 v/v avec de l’eau de qualité CLHP.
- Assurez-vous que le pH est de 8 à l’aide d’un papier test de pH. Si le pH < 8, ajuster en ajoutant 1 μL d’hydroxyde d’ammonium. Si le pH > 8, ajuster en ajoutant 1 μL d’acide formique ou acétique.
- Digérer à température ambiante pendant la nuit ou incuber à 37 °C pendant 6 à 8 h.
- Si possible, vérifiez le pH après environ 3 h de digestion, car il peut avoir diminué. Si le pH < 8, ajuster en ajoutant 1 μL d’hydroxyde d’ammonium.
- Ajouter 5 μL supplémentaires de solution de trypsine de 50 ng/μL (250 ng) et poursuivre la digestion.
- Passez à la deuxième série de propionylation ou conservez les échantillons à -80 °C jusqu’à l’utilisation.
8. Dérivatisation du peptide N-termini
REMARQUE: La propionylation des peptides d’histones à leur terminaison N améliore la rétention des peptides les plus courts par chromatographie liquide en phase inversée (par exemple, les acides aminés 3-8 de l’histone H3), car le groupe propionyle augmente l’hydrophobicité peptidique.
- Sous le capot, ajouter 2 μL d’anhydride propionique à l’aide de la pipette multicanal. Mélanger en pipetant de haut en bas 5x.
- Ajouter rapidement 10 μL d’hydroxyde d’ammonium à l’aide de la pipette multicanal. Mélanger en pipetant de haut en bas 5x.
REMARQUE: L’acide propionique est un produit de la réaction entre l’anhydride propionique et les amines libres de peptides, et peut diminuer le pH de l’échantillon. Le pH 8 peut être rétabli en ajoutant de l’hydroxyde d’ammonium à l’échantillon dans un rapport de 1:5 (v/v). - Assurez-vous que le pH est de 8 à l’aide d’un papier d’essai du pH. Si le pH < 8, ajuster en ajoutant 1 μL d’hydroxyde d’ammonium. Si le pH > 8, ajuster en ajoutant 1 μL d’acide formique ou acétique. Lorsque le pH > 10, il est possible de marquer d’autres résidus d’acides aminés avec un pKa plus élevé.
- Incuber à température ambiante pendant 10 min.
- Répétez les étapes 8.1 à 8.4. Un double tour de propionylation des histones assure une efficacité de réaction presque complète.
- Sécher la plaque sous vide jusqu’à ce que tous les puits soient complètement séchés (~9 h).
- Passez à l’étape de dessalement ou conservez les échantillons à -80 °C jusqu’à leur utilisation.
9. Dessalage et nettoyage des échantillons
REMARQUE: Les sels présents dans l’échantillon interfèrent avec l’analyse par spectrométrie de masse. Les sels sont également ionisés lors de l’électropulvérisation et peuvent supprimer les signaux des peptides. Les sels peuvent former des adduits ioniques sur les peptides, ce qui fait que le peptide adduit a une masse différente. Cela réduit l’intensité du signal du peptide et empêche une identification et une quantification appropriées. La configuration du dessalement est illustrée à la figure 2C.
- Commencez à mélanger la résine HLB (50 mg/mL dans 100% d’acétonitrile) sur une plaque d’agitation magnétique.
- Assurez-vous qu’une plaque de collecte de 96 puits est placée sous la plaque filtrante en polypropylène de 96 puits pour recueillir le débit.
- Ajouter 70 μL de suspension HLB par puits à la plaque filtrante. Allumez doucement l’aspirateur pour éviter les éclaboussures. Jetez le flux.
- Lavez la résine avec 100 μL de 0,1% TFA. Allumez doucement l’aspirateur pour éviter les éclaboussures. Jetez le flux.
- Remettre en suspension chaque échantillon dans 100 μL de 0,1 % de TFA. Vérifiez le pH; il devrait être ~ 2-3.
- Chargez chaque échantillon dans chaque puits. Allumez doucement l’aspirateur pour éviter les éclaboussures. Jetez le flux.
- Laver avec 100 μL 0,1% TFA. Allumez doucement l’aspirateur pour éviter les éclaboussures. Jetez le flux.
- Remplacez la plaque de collecte par une nouvelle plaque de collecte de 96 puits.
- Ajouter 60 μL d’acétonitrile à 60 % / 0,1 % de TFA par puits. Allumez doucement l’aspirateur pour éviter les éclaboussures. Collectez le flux et séchez-le dans un vide rapide.
- Passez à LC-MS/MS ou conservez les échantillons à -80 °C jusqu’à ce qu’ils soient utilisés.
10. Analyse peptidique des histones par chromatographie liquide couplée à la spectrométrie de masse
- Préparer les phases mobiles à fonctionner sur la chromatographie liquide à haute performance (CLHP). Phase mobile A (AMP) : 2 % d’acétonitrile de qualité CLHP + 0,1 % d’acide formique. Phase B mobile (MPB) : 80 % d’acétonitrile de qualité CLHP + 0,1 % d’acide formique.
- Programmez la méthode HPLC comme suit : (1) 4 % à 34 % de MPB sur 30 min; (2) 34% -90% MPB sur 5 min; et (3) isocratique 90% MPB pendant 5 min. Utilisez les propriétés de colonne recommandées suivantes : matériau d’emballage C18 3 μm, diamètre intérieur 75 μm, longueur 20-25 cm. Réglez le débit à 250-300 nL/min pour les nano-colonnes d’un diamètre interne de 75 μm.
- Dans le cas où la CLHP n’est pas programmée pour automatiser l’équilibrage des colonnes avant le chargement de l’échantillon, incluez (4) 90% -4% MPB sur 1 min et (5) isocratique 4% MPB sur 10 min.
- Programmer la méthode d’acquisition de la SEP.
- Assurez-vous que l’instrument effectue une analyse complète de la SEP au début de chaque cycle d’utilisation. L’instrumentation à haute résolution (p. ex., les analyseurs orbitrap ou à temps de vol) est recommandée, en raison de la précision de masse qui peut être utilisée lors de l’extraction du signal. Cependant, l’instrumentation basse résolution peut également être utilisée comme décrit précédemment27,28.
- Assurez-vous que l’analyse MS complète est suivie de 16 événements d’analyse MS/MS, chacun avec une largeur d’isolement de 50 m/z, couvrant la plage m/z de 300-1100. Par exemple, le premier balayage doit isoler les signaux à 300-350 m/z, le second à 350-400 m/z, etc. Si possible, acquérir des scans MS/MS en haute résolution également, mais une résolution inférieure par rapport au scan MS complet est suffisante (en raison des masses plus petites d’ions fragments par rapport aux ions intacts).
- La méthode HPLC générera des signaux chromatographiques avec des largeurs de crête d’environ 3-40 s; pour assurer une quantification adéquate du signal, assurez-vous que le spectromètre de masse effectue au moins 10 cycles d’utilisation par pic chromatographique (c.-à-d. un rapport cyclique de 3 s ou plus).
- Si vous utilisez un analyseur de masse de type piégeage (orbitrap, piège à ions), assurez-vous que la limite de temps d’injection d’ions est réglée sur <200 ms; pour les autres analyseurs (quadripôle, temps de vol), ce n’est pas un problème en raison de leur temps d’analyse plus rapide. Un test préliminaire pourrait être nécessaire.
REMARQUE: Plus de détails sur les méthodes MS pour l’analyse des peptides d’histones peuvent être trouvés dans les références suivantes 27,28,29.
- Remettre en suspension l’échantillon dans 10 μL de MPA, ce qui correspond à ~1 μg/μL d’échantillon d’histone digéré. La quantité exacte de chargement n’est pas critique (il n’est pas non plus trivial d’évaluer) si tous les échantillons du lot sont chargés en utilisant des dilutions et des volumes similaires.
- Chargez 1 μL d’échantillon sur la colonne HPLC.
- Exécutez la méthode LC-MS/MS programmée aux étapes 10.1 à 10.3.
11. Analyse des données
- Importez les fichiers de données brutes MS dans un logiciel conçu pour effectuer l’intégration de la zone de pointe.
REMARQUE: EpiProfile 30,31 est utilisé dans la présente étude et est généralement recommandé, car il est optimisé pour une extraction fiable de la zone de crête des peptides d’histones connus. Cependant, d’autres logiciels disponibles gratuitement pour la chromatographie ionique extraite tels que Skyline32,33 conviennent. - Calculer l’abondance relative d’un peptide (non)modifié donné comme l’aire d’un seul peptide divisée par l’aire totale du peptide sous toutes ses formes modifiées. Des logiciels tels que EpiProfile30,31 contiennent déjà des bibliothèques de peptides pour l’extraction de signaux. Sinon, générez une bibliothèque de peptides d’intérêt manuellement ou via l’identification des peptides à l’aide de pipelines protéomiques de routine.
Résultats
Dans ce protocole, les sphéroïdes HepG2/C3A ont été traités avec 20 mM de NaBut et 10 mM de NaSuc, qui ont tous deux affecté les niveaux globaux d’histones PTM (Figure 3A). Les PTM d’histones ont ensuite été identifiés et quantifiés au niveau de résidu unique par acquisition de MS/MS (Figure 3B).
Lorsque les échantillons sont exécutés en répétitions, une analyse statistique peut être effectuée pour évaluer l’enrichissement du changement de pli d’un PTM entre les échantillons, ainsi que la reproductibilité de l’observation. Les données présentées démontrent que les peptides modifiés par acétylations sont enrichis en sphéroïdes traités avec NaBut par rapport au contrôle (Figure 3C), tandis que les échantillons traités avec NaSuc ont une abondance relative plus élevée de peptides d’histones modifiés par succinylation de lysine (Figure 3D). Ces calculs ont été effectués dans un tableur tel que détaillé dans une publication distincte34. Une augmentation globale d’une modification d’histone donnée peut être mieux représentée dans les diagrammes radar, où l’observation d’une abondance globale plus élevée d’une certaine modification devient plus intuitive, tout en conservant des informations détaillées sur les sites de modification analysés (Figure 3E, F).
Ce protocole génère un peptide à partir des résidus d’acides aminés histone H3 9-17, qui comprennent les résidus fréquemment modifiés K9, S10 et K14. Les données présentées indiquent que le traitement par NaBut augmente les niveaux de H3K14ac, mais uniquement sur les histones co-modifiées avec H3K9me2 et non H3K9me3 (Figure 4A). La fréquence de coexistence entre deux modifications peut être représentée plus intuitivement sous la forme d’un graphique en anneau, où les nœuds représentent des modifications individuelles tandis que l’épaisseur des lignes de connecteur représente la fréquence de coexistence entre les deux PTM (Figure 4B). Parfois, la fréquence de coexistence n’est pas affectée, mais les données représentées sous forme de graphiques à barres peuvent être trompeuses. Par exemple, les données représentées à la figure 4A indiquent que la combinaison H3K9me2K14ac est plus abondante dans le traitement NaBut que dans le contrôle. C’est exact, mais cette combinaison donnée est la plus fréquente quel que soit le traitement. La figure 4B montre clairement que H3K9me2K14ac et H3K9me3K14ac sont les modèles combinatoires les plus fréquents quel que soit le traitement (épaisseur de ligne), mais que les niveaux globaux de H3K14ac (nœud) sont ce qui change vraiment dans l’expérience.
Ce protocole génère un peptide à partir des résidus d’histone H4 4-17, qui comprend des résidus modifiables aux positions K5, K8, K12 et K16 (principalement par acétylations). En comparant le traitement témoin et le traitement NaBut, il est possible d’observer une augmentation des combinaisons d’acétylations en représentant les données comme, par exemple, des nuages de mots (Figure 4C). Cette représentation souligne clairement que la version non modifiée de l’histone H4 est la plus abondante dans l’échantillon témoin, tandis que les sphéroïdes traités avec NaBut sont enrichis en protéoformes d’histone H4 doublement, triplement et quadruplement acétylées. Cependant, les nuages de mots sont limités dans l’affichage des valeurs exactes; l’abondance relative d’un code histone doit être alambiquée par la taille du texte, qui peut être estimée de manière inexacte. Par conséquent, des diagrammes de Venn ou des équivalents plus modernes tels que la représentation UpSetR35 peuvent être utilisés pour montrer la quantification exacte des PTM d’histones coexistants (Figure 4D, E). Les données montrées soulignent une fois de plus que certaines combinaisons d’acétylations sur l’histone H4 sont relativement plus abondantes dans le traitement NaBut que dans le contrôle.
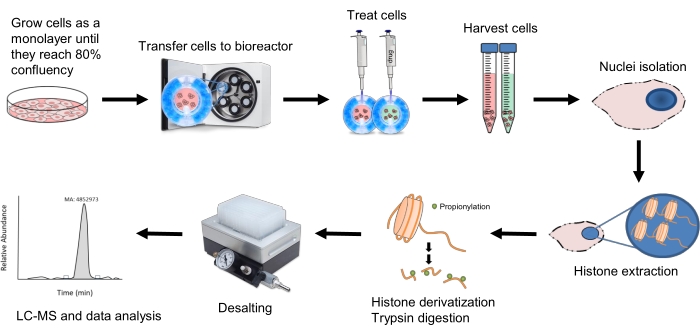
Figure 1 : Flux de travail pour l’analyse peptidique des histones des sphéroïdes 3D. Les cellules HepG2/C3A sont d’abord cultivées en culture 2D jusqu’à ce qu’elles atteignent 80% de confluence. Les cellules sont ensuite transférées dans un bioréacteur équilibré et placées dans l’incubateur de clinostat où elles tourneront à 10-11 tr / min pour former des sphéroïdes. Après 18 jours, les sphéroïdes sont traités avec 20 mM de NaBut ou 10 mM de NaSuc et sont récoltés après leurs points de temps correspondants. Les noyaux sont isolés des cellules et l’extraction des histones est réalisée avec 0,2 M H2SO4. La dérivatisation des histones est ensuite réalisée avec de l’anhydride propionique avant et après la digestion de la trypsine pour assurer la rétention des peptides courts résultants par chromatographie liquide. Les échantillons sont dessalés, puis exécutés à l’aide de la méthode LC-MS/MS mentionnée à l’étape 10, et les données résultantes sont analysées comme décrit à l’étape 11. Veuillez cliquer ici pour voir une version agrandie de cette figure.
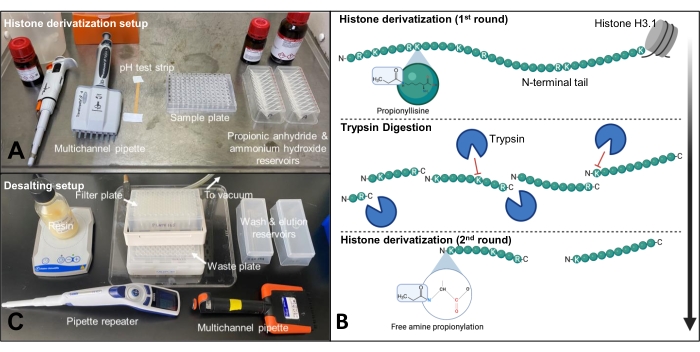
Figure 2: Configuration des étapes de propionylation et de dessalement. (A) La propionylation est effectuée dans une hotte et tous les composants sont disposés de manière à ce que les étapes puissent être effectuées en succession rapide. (B) Schéma de la première série de propionylation, de la digestion de la trypsine et de la deuxième série de propionylation sur la queue de l’histone H3.1. (C) Le dessalement est effectué sur le banc à l’aide d’un collecteur à vide de 96 puits et d’une plaque filtrante en polypropylène de 96 puits. Veuillez cliquer ici pour voir une version agrandie de cette figure.
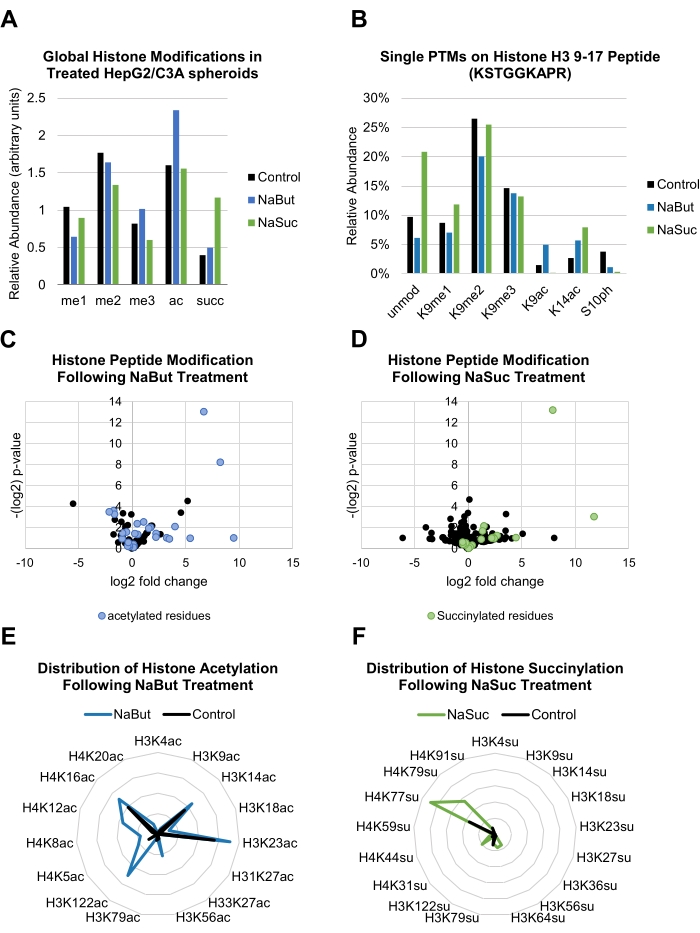
Figure 3 : Représentation des modifications individuelles des histones. (A) Graphique à barres montrant l’abondance relative des modifications globales courantes des histones chez les sphéroïdes HepG2/C3A témoins et traités (20 mM NaBut ou 10 mM NaSuc). (B) Graphique à barres montrant l’abondance des PTM à histone unique présents sur les résidus 9-17 du peptide d’histone H3 (KSTGGKAPR) dans les sphéroïdes HepG2/C3A témoins et traités (20 mM NaBut ou 10 mM NaSuc). (C,D) Diagrammes de volcan montrant le changement de pli et la signification de l’expression différentielle des PTM peptidiques d’histones après traitement avec 20 mM NaBut (C) ou 10 mM NaSuc (D). Les points bleus et verts surlignés représentent respectivement les résidus acétylés et succinylés. (E,F) Tracés radar montrant l’abondance de l’acétylation peptidique d’histone unique (E) ou de la succinylation (F) après traitement avec 20 mM NaBut ou 10 mM NaSuc respectivement par rapport au contrôle. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Représentation des modifications coexistantes des histones. (A) Graphique à barres montrant l’abondance des PTM combinatoires d’histones se produisant sur les résidus 9-17 d’histone H3 (KSTGGKAPR) dans les sphéroïdes HepG2/C3A témoins et traités (20 mM NaBut ou 10 mM NaSuc). (B) Graphiques en anneau montrant la relation entre les PTM d’histones combinatoires sur les résidus 9-17 d’histone H3 (KSTGGKAPR) en contrôle et traités (20 mM NaBut) HepG2/C3A sphéroïdes. L’intensité de la couleur du nœud correspond à l’abondance d’un seul PTM dans son groupe de traitement, tandis que l’épaisseur de la ligne correspond à la fréquence de cooccurrence ptm. (C) Nuages de mots montrant la fréquence des PTM d’histones combinatoires sur les résidus d’histone H4 en cours de contrôle et traités (20 mM NaBut) HepG2/C3A sphéroïdes. La taille du texte correspond à l’abondance du PTM combinatoire spécifié. (D,E) Diagramme de Venn représentant la fréquence des modifications coexistantes sur les résidus peptidiques d’histone H4 4-17 dans les échantillons témoins et traités par NaBut de 20 mM. Les données sont affichées à l’aide de ShinyApp UpSetR35. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau supplémentaire 1 : Liste des peptides détectés à l’aide de ce protocole. Veuillez cliquer ici pour télécharger ce tableau.
Discussion
L’analyse des PTM d’histones est fondamentalement différente du pipeline d’analyse protéomique typique. La plupart des PTM d’histones ont encore des fonctions biologiques énigmatiques; par conséquent, les annotations telles que l’ontologie des gènes ou les bases de données de voies ne sont pas disponibles. Il existe plusieurs ressources qui associent les modifications des histones à l’enzyme responsable de leur catalyse ou aux protéines contenant des domaines qui lient ces PTM (par exemple, HISTome36). De plus, il est possible de spéculer sur l’état général de la chromatine lorsque les niveaux globaux d’histones PTM sont régulés. Par exemple, une augmentation globale de l’acétylation des histones ou d’autres acylations comme la succinylation est normalement associée à la décondensation de la chromatine37,38.
L’analyse de la SEP fournit des informations plus détaillées sur ces modifications, telles que leur localisation exacte sur la séquence d’acides aminés. Dans ce protocole, l’acquisition de MS/MS est utilisée pour identifier et quantifier les PTM d’histones, ce qui peut être essentiel pour l’interprétation biologique. Par exemple, la triméthylation sur la lysine 4 de l’histone H3 (H3K4me3) est enrichie sur les promoteurs des gènesactivement transcrits 39, tandis que la même modification sur la lysine 9 (H3K9me3) repère l’hétérochromatineconstitutive 40. Les modifications des histones sont actuellement utilisées comme biomarqueurs dans des maladies spécifiques; en tant que tel, l’analyse des histones peut être utilisée pour étudier la pathologie de la maladie en plus de la réponse au traitement (par exemple, avec des médicaments épigénétiques)41,42.
Il est plus difficile de représenter visuellement les interactions entre plusieurs PTM par opposition à des PTM uniques. Bien que les graphiques existants tels que les graphiques en anneau puissent montrer la fréquence de coexistence de deux PTM, ils ne peuvent pas représenter la fréquence de coexistence entre plus de deux PTM à la fois, car cela nécessiterait une représentation tridimensionnelle du réseau. Pour cette raison, d’autres représentations pourraient être plus appropriées pour mettre en évidence les changements dans les codes d’histones lorsque trois PTM ou plus sont considérés. En général, la diversification de la représentation des données offre plus de chances d’observer des changements significatifs entre les échantillons. Ce protocole présente des exemples de différentes illustrations pour l’affichage des régulations des PTM histones et des PTM coexistants.
Bien que ce protocole génère des peptides d’histones relativement petits en raison de la digestion de la trypsine (environ 4 à 20 acides aminés), certains peptides contiennent de multiples résidus modifiables. L’analyse de ces peptides permet de quantifier les fréquences de coexistence des PTM, ce qui pourrait révéler des informations importantes sur les marques d’histones combinatoires régulées dans un ensemble de données donné. Notamment, il n’y a pas d’étapes lors de la préparation de l’échantillon où la quantification des histones est effectuée. Il y a quatre raisons à cela: (1) la trypsine a un large éventail d’activités et peut être utilisée à un large éventail de rapports enzymes / échantillons (1: 10-1: 200). Même lorsque le rendement expérimental des histones extraites diffère de celui attendu, des problèmes de digestion n’ont pas été rencontrés en utilisant ce protocole. (2) Ce protocole est destiné à des quantités infimes de matière, où la quantification des histones peut être difficile à effectuer en raison du manque de sensibilité. (3) En utilisant une concentration constante de trypsine quelle que soit la quantité de matériau d’histone, nous pouvons utiliser des peptides tryptiques (comme les autolyses de trypsine) pour comparer les performances de chromatographie. De légères variations dans le rendement de l’échantillon seront normalisées par un logiciel d’analyse de données (étape 11), qui utilise tous les signaux (non)modifiés pour un peptide donné comme dénominateur pendant le processus de normalisation. (4) Enfin, une sous-estimation dramatique de la quantité de matière première pourrait créer des problèmes de surcharge de la colonne chromatographique nanoLC. Toutefois, l’exécution de l’étape de dessalement comme indiqué dans ce protocole empêche ce problème de se produire. En cas de quantités excessives de matière première (par exemple >100 μg), la limite de la capacité de la résine de dessalement sera dépassée et tout excès d’échantillon sera emporté pendant l’étape de chargement.
Il est important de noter que tous les peptides détectés par cette analyse n’ont pas été mis en évidence dans les figures 3 et 4. De plus, toutes les modifications d’histones ne sont pas détectables à l’aide de ces méthodes spécifiques de préparation et d’acquisition d’échantillons. Le tableau supplémentaire 1 est fourni pour énumérer tous les signaux peptidiques qui sont extraits à l’aide du pipeline décrit. Quelques modifications bien connues ne sont pas répertoriées dans le tableau, car la préparation de l’échantillon décrite ne convient pas à leur détection. Des exemples notables sont les peptides ubiquitinylés de l’histone H2A et H2B, et la phosphorylation de l’histone H2A. X (un marqueur général des dommages à l’ADN). En effet, la propionylation des peptides associés à ces PTM conduit à des peptides excessivement longs, qui ne conviennent pas à la chromatographie en C18 et à la méthode de détection de la SEP décrite. D’autres modifications présentes dans la littérature mais non présentes dans le tableau supplémentaire 1 sont des modifications d’abondance très faibles (actuellement détectables uniquement à l’aide de la SEP après des stratégies d’enrichissement telles que l’immunoprécipitation ou un traitement cellulaire spécifique), telles que la lactylation43 ou la sérotonylation44. Les modifications des histones avec des changements de masse imprévisibles causés par la polymérisation ou la liaison covalente hétérogène à la séquence d’histones ne sont pas non plus prises en compte (par exemple, la poly-ADP-ribosylation45 et la glycation46). De plus, ce protocole utilise NaSuc et NaBut pour traiter les sphéroïdes HepG2 / C3A, mais il peut être modifié pour une utilisation avec d’autres médicaments / modificateurs épigénétiques et des types de culture cellulaire 2D / 3D.
Déclarations de divulgation
Les auteurs n’ont pas d’intérêts financiers concurrents.
Remerciements
Le laboratoire Sidoli remercie la Leukemia Research Foundation (Hollis Brownstein New Investigator Research Grant), l’AFAR (Sagol Network GerOmics award), Deerfield (Xseed award), Relay Therapeutics, Merck et le NIH Office of the Director (1S10OD030286-01).
matériels
| Name | Company | Catalog Number | Comments |
| 0.05% trypsin-EDTA solution | Gibco | 25300054 | |
| 0.5-20 µL pipet tips | BRAND | 13-889-172 (Fisher Scientific) | |
| 1.5 mL microcentrifuge tubes | Bio-Rad | 2239480 | |
| 10 µL multi-channel pipette | BRAND | BR7059000 (Millipore Sigma) | |
| 10 mL syringe | Henke Sass Wolf | 14-817-31 (Fisher Scientific) | Luer lock tip, graduated to 12 mL |
| 10, 20, 200, and 1000 µL single-channel pipettes | Eppendorf | 14-285-904 (Fisher Scientific) | |
| 1000 µL pipet tips | Rainin | 30389164 | |
| 18 G syringe needle | Air-Tite | 14-817-100 (Fisher Scientific) | 3" length, 0.05" diameter |
| 200 µL multi-channel pipette | Corning | 4082 | |
| 2-200 µL pipet tips | BRAND | Z740118 (Millipore Sigma) | |
| 24-well ultra-low attachment microplate | Corning | 07-200-602 | |
| 75 cm2 U-shaped cell culture flask | Corning | 461464U | Untreated, with vent cap |
| 96-well skirted plate | Axygen | PCR-96-FS-C (Corning) | |
| Acetone | Fisher Scientific | A949-1 | Acetone should be used cold |
| Ammonium bicarbonate (NH4HCO3) | Sigma-Aldrich | A6141-25G | |
| Ammonium hydroxide solution | Fisher Scientific | AC423300250 | |
| Cell culture grade water | Corning | 25-055-CV | |
| ClinoReactor | CelVivo | 10004-12 | Bioreactor for 3D cell culture |
| ClinoStar | CelVivo | N/A | Clinostat CO2 incubator for 3D cell culture |
| Control unit | CelVivo | N/A | Tablet for ClinoStar settings |
| Dulbecco's Modified Eagle's Medium (DMEM) | Corning | 17-205-CV | 1X solution with 4.5 g/L glucose and sodium pyruvate, without L-glutamine and phenol red |
| Fetal bovine serum (FBS) | Corning | 35-010-CV | |
| Formic acid | Thermo Scientific | 28905 | |
| Fume hood | Mott | N/A | Model 7121000 |
| Glass Pasteur pipette | Fisher Scientific | 13-678-8B | 9", cotton-plugged, borosilicate glass, non-sterile |
| Glutagro supplement | Corning | 25-015-CI | 200 mM L-ananyl-L-glutamine |
| Hank’s Balanced Salt Solution (HBSS) | Corning | 21-022-CV | 1X solution without calcium, magnesium, and phenol red |
| HPLC grade acetonitrile | Fisher Scientific | A955-4 | |
| HPLC grade water | Fisher Scientific | W6-1 | |
| Hydrochloric acid (HCl) | Fisher Scientific | A481-212 | |
| Ice | N/A | N/A | |
| MEM non-essential amino acids | Corning | 25-025-CI | 100X solution |
| Oasis HLB resin | Waters | 186007549 | Hydrophilic-Lipophilic-Balanced (HLB) Resin with 30µm particle size |
| Orbitrap Fusion Lumos Tribrid mass spectrometer | Thermo Fisher Scientific | IQLAAEGAAPFADBMBHQ | High resolution mass spectrometer |
| Oro-Flex I polypropylene filter plate | Orochem | OF1100 | 96-well polypropylene filter plate w/ 10 µM PE frit |
| Penicillin-Streptomycin | Corning | 30-002-CI | 100X solution |
| pH paper | Hydrion | Z111848 (Sigma-Aldrich) | 0-13 pH test paper |
| Pipette gun | Eppendorf | Z666467 (Millipore Sigma) | |
| Polymicro capillary | Molex | 50-110-7740 (Fisher Scientific) | Flexible fused silica capillary tubing with polymide coating, 75 µM ID x 363 µM OD |
| Polystyrene 10 mL serological pipets, sterile | Fisher Scientific | 1367549 | |
| Propionic anhydride | Sigma-Aldrich | 240311-50G | |
| Refrigerated centrifuge | Thermo Scientific | 75-217-420 | |
| Reprosil-Pur resin | MSWIL | R13.AQ.0003 | 120 Å pore size, C18-AQ phase, 3 µM bead size |
| Rotator | Clay Adams | 25477 (American Laboratory Trading) | Nutator Mixer 1105 |
| Sequencing grade modified trypsin | Promega | V5111 | |
| Sodium butyrate | Thermo Scientific | A11079 | |
| Sodium succinate dibasic | Sigma-Aldrich | 14160-100G | |
| SpeedVac vacuum concentrator (1.5 mL microcentrifuge tubes) | Savant | 20249 (American Laboratory Trading) | |
| SpeedVac vacuum concentrator (96-well) | Thermo Scientific | 15308325 | Savant SPD1010 |
| Sterile hood | Thermo Scientific | 1375 | Class II, Type A2 |
| Sulfuric acid (H2SO4) | Fisher Scientific | 02-004-375 | Baker Analyzed ACS reagent |
| Tissue-culture treated 100 mm x 20 mm dish | Fisher Scientific | 08-772-23 | |
| Trichloroacetic acid (TCA) | Thermo Scientific | AC421451000 | Resuspend 100% w/v in HPLC grade water |
| Trifluoroacetic acid (TFA) | Fisher Scientific | PI28904 | Sequencing grade |
| Vacuum manifold 96-well | Millipore | MAVM0960R | |
| Vortex | Sigma-Aldrich | Z258415 | |
| Water bath | Fisher Scientific | FSGPD10 | |
| Wide bore pipet tips 1000 µL | Axygen | 14-222-703 (Fisher Scientific) | |
| Wide bore pipet tips 200 µL | Axygen | 14-222-730 (Fisher Scientific) |
Références
- Kapalczynska, M., et al. 2D and 3D cell cultures - a comparison of different types of cancer cell cultures. Archives of Medical Science. 14 (4), 910-919 (2018).
- Breslin, S., O'Driscoll, L. Three-dimensional cell culture: the missing link in drug discovery. Drug Discovery Today. 18 (5-6), 240-249 (2013).
- Kim, J. B. Three-dimensional tissue culture models in cancer biology. Seminars in Cancer Biology. 15 (5), 365-377 (2005).
- Duval, K., et al. Modeling physiological events in 2D vs. 3D cell culture. Physiology (Bethesda). 32 (4), 266-277 (2017).
- Wrzesinski, K., et al. The cultural divide: exponential growth in classical 2D and metabolic equilibrium in 3D environments. PLoS One. 9 (9), 106973(2014).
- Wrzesinski, K., Fey, S. J. Metabolic reprogramming and the recovery of physiological functionality in 3D cultures in micro-bioreactors. Bioengineering (Basel). 5 (1), 22(2018).
- Gonda, S. R., et al. Three-dimensional transgenic cell model to quantify genotoxic effects of space environment. Advances in Space Research. 27 (2), 421-430 (2001).
- Yamada, K. M., Cukierman, E. Modeling tissue morphogenesis and cancer in 3D. Cell. 130 (4), 601-610 (2007).
- Tvardovskiy, A., et al. Top-down and middle-down protein analysis reveals that intact and clipped human histones differ in post-translational modification patterns. Molecular and Cellular Proteomics. 14 (12), 3142-3153 (2015).
- Azad, G. K., et al. Modifying chromatin by histone tail clipping. Journal of Molecular Biology. 430 (18), 3051-3067 (2018).
- Kragesteen, B. K., Amit, I. Heads or tails: histone tail clipping regulates macrophage activity. Nature Immunology. 22 (6), 678-680 (2021).
- Dhaenens, M. Histone clipping: the punctuation in the histone code. EMBO Reports. 22 (8), 53440(2021).
- Anderson, L. C., et al. Analyses of histone proteoforms using front-end electron transfer dissociation-enabled orbitrap instruments. Molecular and Cellular Proteomics. 15 (3), 975-988 (2016).
- Morgan, M. A. J., Shilatifard, A. Reevaluating the roles of histone-modifying enzymes and their associated chromatin modifications in transcriptional regulation. Nature Genetics. 52 (12), 1271-1281 (2020).
- Chan, J. C., Maze, I. Nothing is yet set in (hi)stone: novel post-translational modifications regulating chromatin function. Trends in Biochemical Sciences. 45 (10), 829-844 (2020).
- Jenuwein, T., Allis, C. D. Translating the histone code. Science. 293 (5532), 1074-1080 (2001).
- Fischle, W., et al. Molecular basis for the discrimination of repressive methyl-lysine marks in histone H3 by Polycomb and HP1 chromodomains. Genes and Development. 17 (15), 1870-1881 (2003).
- Onder, O., et al. Progress in epigenetic histone modification analysis by mass spectrometry for clinical investigations. Expert Review of Proteomics. 12 (5), 499-517 (2015).
- Sidoli, S., Cheng, L., Jensen, O. N. Proteomics in chromatin biology and epigenetics: Elucidation of post-translational modifications of histone proteins by mass spectrometry. Journal of Proteomics. 75 (12), 3419-3433 (2012).
- Egelhofer, T. A., et al. An assessment of histone-modification antibody quality. Nature Structural and Molecular Biology. 18 (1), 91-93 (2011).
- Moradian, A., et al. The top-down, middle-down, and bottom-up mass spectrometry approaches for characterization of histone variants and their post-translational modifications. Proteomics. 14 (4-5), 489-497 (2014).
- Zheng, Y., Huang, X., Kelleher, N. L. Epiproteomics: quantitative analysis of histone marks and codes by mass spectrometry. Current Opinion in Chemical Biology. 33, 142-150 (2016).
- Sidoli, S., Garcia, B. A. Middle-down proteomics: a still unexploited resource for chromatin biology. Expert Review of Proteomics. 14 (7), 617-626 (2017).
- Stampar, M., et al. Hepatocellular carcinoma (HepG2/C3A) cell-based 3D model for genotoxicity testing of chemicals. Science of the Total Environment. 755, 143255(2021).
- Calitz, C., et al. Toxicity and anti-prolific properties of Xysmalobium undulatum water extract during short-term exposure to two-dimensional and three-dimensional spheroid cell cultures. Toxicology Mechanisms and Methods. 28 (9), 641-652 (2018).
- Garcia, B. A., et al. Chemical derivatization of histones for facilitated analysis by mass spectrometry. Nature Protocols. 2 (4), 933-938 (2007).
- Sidoli, S., et al. Sequential window acquisition of all theoretical mass spectra (SWATH) analysis for characterization and quantification of histone post-translational modifications. Molecular and Cellular Proteomics. 14 (9), 2420-2428 (2015).
- Sidoli, S., et al. Low resolution data-independent acquisition in an LTQ-orbitrap allows for simplified and fully untargeted analysis of histone modifications. Analytical Chemistry. 87 (22), 11448-11454 (2015).
- Karch, K. R., Sidoli, S., Garcia, B. A. Identification and quantification of histone PTMs using high-resolution mass spectrometry. Methods in Enzymology. 574, 3-29 (2016).
- Yuan, Z. F., et al. EpiProfile quantifies histone peptides with modifications by extracting retention time and intensity in high-resolution mass spectra. Molecular and Cellular Proteomics. 14 (6), 1696-1707 (2015).
- Yuan, Z. F., et al. EpiProfile 2.0: a computational platform for processing epi-proteomics mass spectrometry data. Journal of Proteome Research. 17 (7), 2533-2541 (2018).
- MacLean, B., et al. Skyline: an open source document editor for creating and analyzing targeted proteomics experiments. Bioinformatics. 26 (7), 966-968 (2010).
- Pino, L. K., et al. The Skyline ecosystem: Informatics for quantitative mass spectrometry proteomics. Mass Spectrometry Reviews. 39 (3), 229-244 (2020).
- Aguilan, J. T., Kulej, K., Sidoli, S. Guide for protein fold change and p-value calculation for non-experts in proteomics. Molecular Omics. 16 (6), 573-582 (2020).
- Lex, A., et al. UpSet: Visualization of intersecting sets. IEEE Transactions on Visualization and Computer Graphics. 20 (12), 1983-1992 (2014).
- Shah, S. G., et al. HISTome2: a database of histone proteins, modifiers for multiple organisms and epidrugs. Epigenetics and Chromatin. 13 (1), 31(2020).
- Xie, Z., et al. Lysine succinylation and lysine malonylation in histones. Molecular and Cellular Proteomics. 11 (5), 100-107 (2012).
- Liu, J., et al. Histone succinylation and its function on the nucleosome. Journal of Cellular and Molecular Medicine. 25 (15), 7101-7109 (2021).
- Howe, F. S., et al. Is H3K4me3 instructive for transcription activation. Bioessays. 39 (1), 1-12 (2017).
- Nicetto, D., Zaret, K. S. Role of H3K9me3 heterochromatin in cell identity establishment and maintenance. Current Opinion in Genetics and Development. 55, 1-10 (2019).
- Wojcik, J. B., et al. Histone H3K27 dimethyl loss is highly specific for malignant peripheral nerve sheath tumor and distinguishes true PRC2 loss from isolated H3K27 trimethyl loss. Modern Pathology. 32 (10), 1434-1446 (2019).
- Sellers, W. R., et al. Next-generation characterization of the cancer cell line encyclopedia. Nature. 569 (7757), 503-508 (2019).
- Zhang, D., et al. Metabolic regulation of gene expression by histone lactylation. Nature. 574 (7779), 575-580 (2019).
- Farrelly, L. A., et al. Histone serotonylation is a permissive modification that enhances TFIID binding to H3K4me3. Nature. 567 (7749), 535-539 (2019).
- Chen, Q., et al. ADP-ribosylation of histone variant H2AX promotes base excision repair. The EMBO Journal. 40 (2), 104542(2021).
- Zheng, Q., et al. Reversible histone glycation is associated with disease-related changes in chromatin architecture. Nature Communications. 10 (1), 1289(2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon