Method Article
Méthode de PCR par gouttelettes numériques pour la quantification de l’efficacité de la transduction de l’AAV dans la rétine murine
Dans cet article
Résumé
Ce protocole montre comment quantifier l’efficacité de la transduction de l’AAV dans la rétine de souris en utilisant la PCR numérique par gouttelettes (dd-PCR) ainsi que la production d’AAV à petite échelle, l’injection intravitréenne, l’imagerie rétinienne et l’isolement de l’ADN génomique rétinien.
Résumé
De nombreux laboratoires de biologie cellulaire de la rétine utilisent maintenant régulièrement des virus associés à l’adéno (AAV) pour l’édition de gènes et des applications réglementaires. L’efficacité de la transduction de l’AAV est généralement critique, ce qui affecte les résultats expérimentaux globaux. L’un des principaux déterminants de l’efficacité de la transduction est le sérotype ou la variante du vecteur AAV. Actuellement, divers sérotypes et variantes artificiels de l’AAV sont disponibles avec différentes affinités pour les récepteurs de surface des cellules hôtes. Pour la thérapie génique rétinienne, il en résulte différents degrés d’efficacité de transduction pour différents types de cellules rétiniennes. En outre, la voie d’injection et la qualité de la production d’AAV peuvent également affecter l’efficacité de la transduction de l’AAV rétinien. Par conséquent, il est essentiel de comparer l’efficacité de différentes variantes, lots et méthodologies. La méthode de PCR par gouttelettes numériques (dd-PCR) quantifie les acides nucléiques avec une grande précision et permet d’effectuer une quantification absolue d’une cible donnée sans étalon ni référence. En utilisant la dd-PCR, il est également possible d’évaluer l’efficacité de la transduction des AAV par quantification absolue du nombre de copies du génome AAV dans une rétine injectée. Ici, nous fournissons une méthode simple pour quantifier le taux de transduction des AAV dans les cellules rétiniennes en utilisant la dd-PCR. Avec des modifications mineures, cette méthodologie peut également servir de base à la quantification du nombre de copies de l’ADN mitochondrial ainsi qu’à l’évaluation de l’efficacité de l’édition de base, essentielle pour plusieurs maladies de la rétine et applications de thérapie génique.
Introduction
Les virus associés à l’adéno (AAV) sont maintenant couramment utilisés pour diverses études de thérapie génique rétinienne. Les AAV offrent un moyen sûr et efficace d’administration de gènes avec moins d’immunogénicité et moins d’intégrations du génome. L’entrée de l’AAV dans la cellule cible se produit par endocytose, ce qui nécessite la liaison des récepteurs et des co-récepteurs à la surface de la cellule1,2. Par conséquent, l’efficacité de transduction des AAV pour différents types de cellules dépend principalement de la capside et de ses interactions avec les récepteurs de la cellule hôte. Les AAV ont des sérotypes et chaque sérotype peut avoir des tropismes cellulaires / tissulaires distincts et des efficacités de transduction. Il existe également des sérotypes et des variantes artificiels de l’AAV générés par la modification chimique de la capside virale, la production de capsides hybrides, l’insertion peptidique, le brassage de la capside, l’évolution dirigée et la mutagénèse rationnelle3. Même des changements mineurs dans la séquence d’acides aminés ou la structure de la capside peuvent avoir une influence sur les interactions avec les facteurs de la cellule hôte et entraîner différents tropismes4. En plus des variantes de capside, d’autres facteurs tels que la voie d’injection et la variation de lot à lot de la production d’AAV peuvent affecter l’efficacité de transduction des AAV dans la rétine neuronale. Par conséquent, des méthodes fiables de comparaison des taux de transduction pour différentes variantes sont nécessaires.
La majorité des méthodes permettant de déterminer l’efficacité de la transduction de l’AAV reposent sur l’expression du gène rapporteur. Il s’agit notamment de l’imagerie fluorescente, de l’immunohistochimie, du transfert western ou de l’analyse histochimique du produit du gène rapporteur5,6,7. Cependant, en raison de la contrainte de taille des AAV, il n’est pas toujours possible d’inclure des gènes rapporteurs pour surveiller l’efficacité de la transduction. L’utilisation de promoteurs puissants comme l’activateur hybride CMV/bêta-actine de poulet ou l’élément régulateur post-transcriptionnel de l’hépatite Woodchuck (WPRE) comme séquence stabilisatrice d’ARNm complique encore le problème de taille8. Par conséquent, il sera également avantageux de définir le taux de transduction des AAV injectés avec une méthodologie plus directe.
La PCR numérique par gouttelettes (dd-PCR) est une technique puissante pour quantifier l’ADN cible à partir de quantités infimes d’échantillons. La technologie dd-PCR dépend de l’encapsulation de l’ADN cible et du mélange réactionnel PCR par des gouttelettes d’huile. Chaque réaction dd-PCR contient des milliers de gouttelettes. Chaque gouttelette est traitée et analysée comme une réaction PCR indépendante9. L’analyse des gouttelettes permet de calculer le nombre absolu de copies des molécules d’ADN cibles dans n’importe quel échantillon en utilisant simplement l’algorithme de Poisson. Étant donné que l’efficacité de transduction des AAV est corrélée avec le nombre de copies des génomes AAV dans la rétine neuronale, nous avons utilisé la méthode dd-PCR pour quantifier les génomes AAV.
Ici, nous décrivons une méthodologie dd-PCR pour calculer l’efficacité de transduction des vecteurs AAV à partir de l’ADNgénomique rétinien 6,10. Tout d’abord, les AAV qui expriment tdTomato reporter ont été générés en utilisant le protocole à petite échelle, et titrés par la méthode dd-PCR11. Deuxièmement, les AAV ont été injectés par voie intravitréenne dans la rétine neuronale. Pour démontrer l’efficacité de la transduction, nous avons d’abord quantifié l’expression de tdTomato à l’aide de la microscopie fluorescente et du logiciel ImageJ. Cela a été suivi par l’isolement de l’ADN génomique pour la quantification des génomes AAV dans les rétines injectées à l’aide de la dd-PCR. La comparaison des niveaux d’expression de tdTomato avec les génomes dAV transduilés quantifiés par la dd-PCR a montré que la méthode dd-PCR quantifiait avec précision l’efficacité de transduction des vecteurs AAV. Nos protocoles ont démontré une description détaillée d’une méthodologie basée sur la dd-PCR pour quantifier l’efficacité de la transduction de l’AAV. Dans ce protocole, nous montrons également le nombre absolu de génomes AAV qui sont transduis après des injections intravitréennes en utilisant simplement le facteur de dilution après l’isolement de l’ADN génomique et les résultats de la dd-PCR. Dans l’ensemble, ce protocole fournit une méthode puissante, qui serait une alternative à l’expression rapporteure pour quantifier l’efficacité de la transduction des vecteurs AAV dans la rétine.
Protocole
Tous les protocoles expérimentaux ont été acceptés par le comité d’éthique de l’Université Sabanci et les expériences ont été menées conformément à la déclaration de « L’Association pour la recherche en vision et ophtalmologie » pour l’utilisation des animaux dans la recherche
1. Production d’AAV à petite échelle12
- Culture de cellules HEK293T à l’aide de plaques de 15 cm dans 10 mL de DMEM/10% FBS jusqu’à 70-80% de confluence.
- Préparer le mélange de transfection avec 20 μg de plasmide auxiliaire (pHGT1-Adeno1), 7 μg de plasmide de capside et 7 μg de vecteur AAV2-CBA-tdTomato-WPRE et 136 μL de solution PEI (1 mg/mL) dans 5 mL de DMEM.
- Ajouter le mélange de transfection préparé de 5 mL dans une boîte de culture cellulaire contenant 10 mL de milieux de culture et incuber les cellules transfectées pendant 48 à 60 h à 37 °C.
- Recueillir le milieu à 48-60 h après la transfection et le digérer avec de la DNase I à une concentration finale de 250 U/mL pendant 30 min à 37 °C.
- Centrifuger le milieu digéré à 4000 x g,4 °C pendant 30 min, puis filtrer avec un filtre à seringue de 0,22 μm dans la membrane de cellulose régénérée pré-mouillée pendant 100 kDa.
- Centrifuger la membrane de cellulose régénérée à 4000 x g,4 °C pendant 30 min et jeter le support.
- Laver et centrifuger la membrane de cellulose régénérée avec du PBS contenant 0,001% de Pluronic F-68 trois fois à 4000 x g,4 °C pendant 30 min. Jetez le PBS à chaque étape.
- Recueillir les AAV concentrés de la partie supérieure de la membrane de cellulose régénérée et aliquote pour d’autres utilisations.
- Digester 5 μL d’AAV fraîchement préparé avec de la DNase I (0,2U/μl) pendant 15 min à 37 °C pour le titrage. Ceci est suivi de 10 min à 95 °C d’incubation pour à la fois l’inactivation de la DNase I et la dégradation des capsides virales.
- Préparer une dilution série 10 fois supérieure à partir d’AAV digérés à l’aide de Pluronic F-68 à 0,05 %. Utilisez les amorces AAV2-ITR et WPRE pour le titrage (Tableau 1). Les titres ont été calculés en multipliant les résultats de la dd-PCR avec les facteurs de dilution. Les résultats ont été convertis en copie du génome/mL (GC/mL).
REMARQUE: Les concentrations d’AAV pour la production d’AAV à petite échelle devraient être d’environ 1 x10 12 GC / mL. Ce protocole ne s’applique pas aux souches d’AAV qui ne libèrent pas efficacement les AAV dans des supports comme AAV213.
2. Injection intravitréenne d’AAV
-
Préparation de l’équipement
- Avant de commencer, préparez 10 μL de microsyringe avec une aiguille émoussée de 36 G. Rincez cinq fois avec 70% d’EtOH, cinq fois dans ddH2O, et enfin cinq fois avec PBS.
- Chargez la quantité appropriée d’AAV dans le microsyringe pour chaque injection.
- Préparez l’aiguille chirurgicale (suture, soie, 6/0), le ruban adhésif, la pince et les ciseaux.
-
Injection intravitréenne
- Appliquer 1 goutte de 0,5 % de tropicamide et de chlorhydrate de phényléphrine à 2,5 % (p. ex. Mydfrin) contenant du collyre avant l’anesthésie pour dilater les pupilles.
- Anesthésier les souris avec de l’isoflurane 5% (1 L / min) et continuer avec 1,5% d’isoflurane (1 L / min) pendant la procédure. Une petite chambre d’isoflurane est utilisée pour la première induction.
- Vérifiez la profondeur de l’anesthésie par la perte du réflexe de redressement, le réflexe de retrait et la réponse au pincement de la queue.
- Appliquer 0,3% de tobramycine et 0,1% de dexaméthasone solution ophtalmique stérile sur chaque œil avant la procédure d’injection. L’application de goutte oculaire prévient la sécheresse et exerce des effets anti-inflammatoires et antibactériens.
- Utilisez le crochet chirurgical pour stabiliser la paupière supérieure. Tirez légèrement en arrière pour exposer la partie dorsale de l’œil. Collez le crochet sur le banc pour le maintenir en position.
- Retirer la conjonctive (moins de 1 mm2)avec les ciseaux à iris incurvés pour exposer la sclérotique de l’œil sous le microscope à dissection. Utilisez une seringue à insuline fraîche et stérile (30 G) pour percer la sclérotique.
- Insérez l’aiguille du microsyringe à travers la même ponction à l’aide d’un micromanipulateur. Placez le bout de l’aiguille derrière la lentille au milieu de l’œillet.
- Injecter 1 μL d’AAV2/BP2 et d’AAV2/PHP. Vecteurs S ayant des concentrations de 1,63 x10 12 GC/mL et 1,7 x 1012 GC/mL lentement dans le vitré de l’œil. Le contrôle du volume d’injection se fait manuellement. Laissez l’aiguille en place pendant 1 min et retirez lentement l’aiguille.
- Relâchez la paupière et appliquez un traitement en gel topique anti-inflammatoire et antibactérien contenant 0,3% de tobramycine et 0,1% de dexaméthasone sur les oculaires. Surveillez et évaluez les souris dans les jours suivants en fonction de la feuille de score en termes d’apparence (yeux brillants, pelage soigné, intuition), de comportement (activité, immobilisation, automutilation) et de poids corporel sur une échelle de 0 à 3. Chaque condition a un score de 0-3 et un score total de 3 ou plus est un critère de terminaison.
- Placez les animaux dans la cage après la récupération.
3. Fluorescence et imagerie du fond d’œil
- Filtrer la souris et dilater la pupille en plaçant des gouttes de 0,5% de tropicamide et de 2,5% de chlorhydrate de phényléphrine dans chaque œil.
- Anesthésiez l’animal avec la procédure ci-dessus avec de l’isoflurane. Évaluez soigneusement la profondeur de l’anesthésie en pinçant les pattes.
- Placez la souris sur la scène d’imagerie et vérifiez la dilatation correcte en vérifiant à travers la caméra du fond d’œil. Appliquer un gel topique (0,2% carbomère 980) sur la surface des yeux pour protéger les yeux de la déshydratation sous anesthésie et l’utiliser comme gel de couplage pour l’imagerie.
- Manipulez l’étape d’imagerie pour l’aligner avec le nez de l’objectif. Ajustez la scène au besoin pour centrer l’œil de la souris. Une fois l’œil centré, déplacez lentement l’objectif jusqu’à ce qu’il entre en contact avec l’œil.
- Effectuer une imagerie du fond d’œil et de la fluorescence sur les deux yeux pour suivre l’expression de tdTomato à 1 semaine et 2 semaines après l’injection intravitréenne (Figure 3B)14. Prenez des images de fond d’œil et de fluorescence à des paramètres identiques pour comparer l’expression du rapporteur.
4. Isolement de la rétine
- Euthanasier les animaux après le fond d’œil et l’imagerie par fluorescence par inhalation de CO2 pour la collecte de la rétine à 2 semaines.
- Déplacez le globe oculaire vers l’avant à l’aide d’une pince fendeuse de 13,5 cm.
- Retirez la lentille avec les restes du vitré en appliquant une légère pression avec la pince.
- Coupez la connexion du globe oculaire au nerf optique avec une pince incurvée de précision et pressez doucement la rétine avec la même pince incurvée.
- Transférer les rétines disséquées dans un tube de microcentrifugation.
- Snap freeze rétine en plaçant les tubes dans l’azote liquide.
5. Isolement de l’ADN génomique tissulaire
- Isolez l’ADN génomique avec un kit d’ADN génomique tissulaire commercialisé à base de digestion de la protéinase K.
- Digérer la rétine à 55 °C, 200 x g pendant 30 min avec la protéinase K, qui est déjà fournie dans le kit.
- Effectuez l’étape d’incubation de la RNase, les étapes de lavage avec le tampon de lavage I et II, et enfin l’étape d’élution selon le protocole du fabricant.
- Ajouter une étape d’essorage supplémentaire après le dernier lavage pour éliminer l’éthanol résiduel.
- Mesurer la concentration d’ADN génomique et stocker les échantillons à -20 °C.
6. Analyse par PCR numérique par gouttelettes d’échantillons de rétine de souris pour la quantification des génomes viraux
- Diluer les ADN génomiques des rétines injectées dans une solution de Pluronic F-68 à 0,05 % pour atteindre une concentration finale de 1 ng/μL pour chaque échantillon.
- Effectuer des réactions distinctes pour WPRE et 18S à l’aide d’amorces spécifiques à la cible avec une concentration finale de 125 nM pour chaque amorce.
- Effectuer la génération de gouttelettes dans un mélange de réaction PCR de 20 μL comprenant 1 ng d’ADN génomique, des amorces de 125 nM et 2x supermélange evagreen.
- Chargez le mélange d’échantillons dans la partie centrale de la cartouche pour la génération de gouttelettes.
- Une fois le chargement de l’échantillon sur la cartouche terminé, ajoutez 70 μL d’huile de génération de gouttelettes dans la rangée inférieure de la cartouche.
- Placez le joint sur le dessus de la cartouche et placez-le dans le générateur de gouttelettes. Assurez-vous qu’il n’y a pas d’espace entre le joint et la cartouche.
- Prenez doucement environ 40 μL de gouttelettes générées dans le puits supérieur. Ajouter la solution de gouttelettes à la plaque de réaction PCR semi-jupe de 96 puits.
- Scellez la plaque PCR avec un scellant à plaque PCR à l’aide d’une feuille d’étanchéité en aluminium.
- Placez la plaque PCR dans un cycleur thermique thermoscellé à 96 puits. Utilisez le protocole PCR; 95 °C pendant 5 min, 40 cycles de 95 °C pendant 30 s, 60 °C pendant 1 min, 4 °C pendant 5 min, 90 °C pendant 5 min et 4 °C en tenue infinie. Appliquer une vitesse de rampe de 2 °C/s à chaque cycle pour s’assurer que les gouttelettes atteignent la température correcte pour chaque étape pendant le cycle
- Placez la plaque PCR dans le lecteur de gouttelettes pour quantifier les gouttelettes à l’aide du logiciel dd-PCR. Un modèle est configuré à l’aide de paramètres spécifiques pour evagreen.
- Analysez les données à l’aide d’un graphique 1D avec chaque échantillon pour l’intensité de fluorescence par rapport au nombre de gouttelettes. La valeur seuil est disposée de manière à ce que les gouttelettes au-dessus du seuil soient attribuées comme positives et les gouttelettes ci-dessous comme négatives. Ceci est suivi par l’application de l’algorithme de Poisson pour déterminer la concentration de départ de l’ADN cible en unités de copies/μL. Le rapport entre WPRE et 18S est calculé pour mesurer l’efficacité de la transduction de l’AAV.
Résultats
La production d’AAV à petite échelle est une méthode rapide et efficace qui fournit des vecteurs pour les injections intravitréennes (Figure 1). La production d’AAV à petite échelle donne généralement des titres dans la plage de 1 x 1012 GC / ml, ce qui est suffisant pour détecter l’expression du rapporteur dans la rétine (Figure 2). Le titrage de l’AAV à l’aide de dd-PCR donne des résultats cohérents. Des amorces spécifiques à ITR2 et WPRE sont couramment utilisées et la concentration de départ de chaque molécule cible a été calculée avec le logiciel dd-PCR par modélisation en tant que distribution de Poisson. Les calculs ont été finalisés par multiplication des facteurs de dilution et convertis en copie du génome par mL. Les AAV produits pour ce protocole avaient des titres de 1,63 x 1012 GC/mL et 1,7x 1012 GC/ml pour AAV2/BP2 et AAV2/PHP.S7,15, respectivement. À des fins de comparaison, des constructions d’expression tdTomato identiques ont été utilisées pour les deux souches. Nous avons injecté des titres à peu près égaux d’AAV pour la quantification de l’efficacité de transduction de l’AAV. Après injection de 1 μL à partir des concentrations susmentionnées d’AAV, les animaux ont été photographiés à des points de temps de 1 semaine et 2 semaines. Un système d’imagerie du fond d’œil et de la fluorescence a été utilisé pour AAV2/BP2 et AAV2/PHP. Rétines injectées S générant des profils d’expression rapporteurs similaires, à l’exception de la rétine#1 (Figure 3A,B). L’expression de TdTomato a été quantifiée à l’aide du logiciel ImageJ pour la valeur grise moyenne (intensité de fluorescence moyenne) afin de comparer l’efficacité de transduction et l’expression rapporteure (Figure 3A, B). Il s’agit essentiellement de la somme des valeurs grises de tous les pixels de la sélection divisée par le nombre de pixels16. Les images fluorescentes de Retina #1 n’ont montré qu’une petite zone d’expression de tdTomato, probablement en raison d’un reflux ou d’une fuite après des injections intravitréennes. Conformément à cette constatation, l’intensité de fluorescence de la rétine #1 était de 0,9, ce qui était la plus faible par rapport à toutes les rétines qui ont été injectées et imagées par voie intravitréenne. L’intensité moyenne de fluorescence était comprise entre 4 et 11. Cela a montré que la quantification des images fluorescentes de l’expression de tdTomato était réussie et corrélée avec les images qui ont été montrées (Figure 3A, B).
Après l’imagerie des rétines, l’ADN génomique a été isolé des rétines injectées à un moment de 2 semaines. La dd-PCR a été réalisée à l’aide d’amorces WPRE pour la quantification du génome AAV. La souris 18S a été utilisée pour la normalisation des génomes de l’AAV au génome de la souris (Figure 3C)17. Conformément à l’intensité moyenne de fluorescence et aux images, la rétine #1 avait un très faible nombre de copies du génome AAV par rapport à 18S, 0,011 fois inférieur à celui de la rétine #2. De plus, les rétines #2, 3, 4 et 5 donnent des différences de niveau de pli similaires. Les différences de pli par rapport à la rétine n ° 2 pour les rétines n ° 3, 4 et 5 étaient de 1,75, 0,55 et 0,99, respectivement. Parmi ceux-ci, la rétine n ° 3 a donné la plus haute intensité fluorescente et le nombre de copies du génome AAV. Cela a déjà montré que la quantification des génomes AAV transduis par dd-PCR est en corrélation avec l’intensité de fluorescence et donc avec l’expression de tdTomato observée. La méthode dd-PCR nous a également permis de faire une quantification absolue des AAV transduites. La quantification absolue du génome total de l’AAV par rétine a été calculée simplement en multipliant le nombre total identifié à partir de la dd-PCR et le facteur de dilution de l’ADN génomique. Cela a donné des résultats similaires par rapport à l’efficacité de transduction du génome AAV normalisée 18S (Figure 3D).

Figure 1: Organigramme des procédures expérimentales. La production d’AAV à petite échelle est suivie d’un titrage AAV basé sur dd-PCR. Les AAV sont injectés par voie intravitréenne dans la rétine adulte. Le suivi des animaux a été effectué à l’aide d’un fond d’œil et d’un système d’imagerie fluorescente afin d’analyser l’expression du rapporteur. Le fond d’œil et les images fluorescentes ont été prises à des points de temps de 1 semaine et de 2 semaines. L’ADN génomique est isolé des rétines injectées pour analyse avec dd-PCR afin de quantifier le génome total de l’AAV présent dans les rétines injectées. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2: Titrage AAV utilisant la méthode dd-PCR. ( A )Laméthode Dd-PCR bénéficie de réactions PCR encapsulées dans une gouttelette. Après une réaction de PCR utilisant la chimie evagreen, des gouttelettes positives et négatives pour le gène cible ont été analysées pour déterminer le nombre absolu d’ADN cibles dans une solution. (boîtes rouges en gouttelettes vertes). (B) Données dd-PCR représentatives pour le titrage AAV. Plusieurs lots d’AAV ont été titrés à l’aide d’amorces ITR2. Les gouttelettes positives et négatives sont séparées au-dessus et au-dessous du seuil (ligne violette). Veuillez cliquer ici pour voir une version agrandie de cette figure.
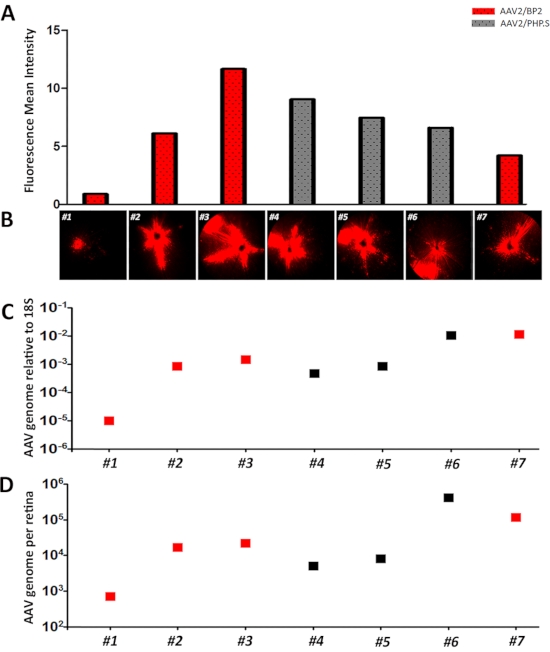
Figure 3: Quantification des génomes d’AAV transduites dans la rétine par dd-PCR. Des animaux de type sauvage âgés de 16 semaines ont été injectés par voie intravitréenne avec AAV2/BP2 et AAV2/PHP. Vecteurs S ayant des concentrations de 1,63 x10 12 GC/mL et 1,7 x 1012 GC/mL, respectivement. Les deux AAV avaient la même construction d’expression de tdTomato et le volume d’injection pour tous les AAV était de 1 μL. Les rétines injectées (1-7) ont été suivies d’une imagerie du fond d’œil et de la fluorescence. Les images prises ont été quantifiées à l’aide du logiciel Image J. Malgré la différence de capside, tous les animaux avaient une valeur d’intensité moyenne de fluorescence(valeur grise moyenne)comprise entre 4 et 11, à l’exception de la rétine #1 qui avait l’intensité la plus faible, 0,9, et une faible expression de tdTomato. Les colonnes rouges et grises sont AAV2/BP2 et AAV2/PHP. Rétines injectées S, respectivement (A-B). La dd-PCR a été réalisée à l’aide d’amorces WPRE pour le génome AAV et d’amorce 18S pour la normalisation à un moment de 2 semaines. Retina # 1 a également montré des nombres de copies AAV nettement inférieurs par copies 18S (C). Les génomes AAV totaux par rétine ont également été calculés en utilisant le nombre de copies WPRE et le facteur de dilution pour l’isolement génomique de l’ADN (D). Les carrés rouges et gris sont AAV2/BP2 et AAV2/PHP. Rétines injectées S, respectivement. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Amorces ddPCR | |
| ITR2 F | GGAACCCCTAGTGATGGAGTT |
| ITR2 R | CGGCCTCAGTGAGCGA |
| WPRE F | GGCTGTTGGGCACTGACAA |
| WPRE R | CCAAGGAAAGGACGATGATTTC |
| 18S F | GGCCGTTCTTAGTTGGTGGA |
| 18S R | CCCGGACATCTAAGGGCATC |
Tableau 1 : séquences d’amorce dd-PCR. Séquences d’amorces avant et arrière pour WPRE, ITR2 et souris 18S.
Discussion
Dans ce protocole, nous avons généré deux vecteurs AAV qui ont des protéines de capside différentes, puis nous les avons titrés en conséquence. L’une des étapes les plus cruciales de ce protocole est de produire des quantités suffisantes d’AAV qui produiront une expression de rapporteur détectable après la transduction12,13.
Le titrage des AAV est également un facteur important pour ajuster les doses d’AAV pour les injections intravitréennes. Une fois ces critères importants atteints, il est possible de quantifier l’efficacité de transduction des AAV par la méthodologie dd-PCR.
De nombreux laboratoires utilisent la méthode de PCR quantitative (qPCR) pour le titrage AAV. La méthode de quantification absolue basée sur la qPCR nécessite une courbe standard qui a des dilutions de quantités connues d’ADN cible18. Cependant, la dd-PCR ne nécessite pas de courbe standard car elle quantifie directement le nombre total de molécules cibles dans un échantillon donné en détectant les gouttelettes positives qui ont au moins une copie de l’ADN cible. Étant donné que la dd-PCR est une analyse de point final et qu’aucune courbe standard n’est requise, les problèmes d’efficacité survenus lors de la réaction qPCR sont moins préoccupants pour la dd-PCR comme les PCR à faible efficacité ou la qualité des courbes standard 9. La méthodologie dd-PCR peut être appliquée aux échantillons déjà préparés pour la qPCR. Comme il a déjà été mentionné dans la section Méthodes pour le titrage AAV, la dd-PCR nécessite des échantillons dilués. Il s’agit d’une étape critique puisque la concentration de l’échantillon doit être ajustée de manière à ce qu’il y ait suffisamment de gouttelettes négatives pour exécuter l’algorithme de Poisson. En d’autres termes, il n’est pas possible d’effectuer la réaction dd-PCR avec des échantillons avec des concentrations trop élevées d’ADN cible qui ne produisent pas de gouttelettes négatives.
Pour les mesures de titrage AAV et d’efficacité de transduction, différents ensembles de cibles sont possibles à utiliser. Pour le titrage AAV, nous utilisons principalement des amorces spécifiques À AAV2 ITR en raison de l’épine dorsale AAV2 dans nos constructions. Il est également possible d’utiliser WPRE et d’autres cibles spécifiques au gène en fonction de la construction AAV qui a été analysée.
Pour évaluer l’efficacité de transduction des AAV dans la rétine neuronale, nous avons appliqué la méthode dd-PCR en utilisant des échantillons entiers de la rétine pour quantifier la quantité totale de génomes AAV par rétine. Nous avons évalué l’exactitude de la méthodologie en utilisant un vecteur AAV qui exprime tdTomato reporter. Comparaison des résultats de dd-PCR avec les niveaux d’expression de tdTomato corrélés, à l’exception des deux valeurs aberrantes. Les rétines #6 et #7 ont donné des copies de génome AAV en excès malgré des intensités de fluorescence similaires à celles des rétines #2,3 4 ou 5. Cela peut être dû au fait que ces rétines ont des taux de transduction plus élevés avec des niveaux d’expression limités ou peuvent en partie être liées à des particules AAV durables ou à de l’ADN vecteur dans la rétine vitrée ou neuronale19,20. Dans l’ensemble, c’était le seul facteur limitant pour notre méthode et peut facilement être identifié parmi d’autres échantillons. Nous avons également quantifié le nombre total de génomes AAV par rétine, ce qui était l’un des points forts de cette méthodologie. Cela permet de comparer les données entre différents laboratoires et lots d’animaux. Par conséquent, cette méthode peut facilement être appliquée pour évaluer l’efficacité de transduction d’un nouveau sérotype, d’une variante ou d’une variation de lot à lot pour les vecteurs AAV qui n’ont pas d’expression rapporteure et nous aider à comparer les données à différents paramètres. Ceci est essentiel pour les vecteurs AAV qui ne permettent pas l’expression d’un rapporteur en raison de contraintes de taille.
Cette méthode fournit également une base pour d’autres types de tests sensibles qui utilisent l’ADN cible. En utilisant les mêmes protocoles, nous pouvons quantifier le nombre de copies du génome mitochondrial avec des amorces appropriées. D’autres améliorations sont également possibles, notamment la dissociation de la rétine et les étapes de tri par cytométrie en flux pour quantifier l’ADN cible dans des cellules individuelles ou des lots de cellules. De plus, il est également possible d’évaluer l’efficacité de correction de l’édition de base en utilisant cette méthode21,22, qui est également essentielle pour plusieurs maladies de la rétine et thérapies géniques.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous tenons à remercier Oezkan Keles, Josephine Jüttner et le professeur Botond Roska, Institute of Molecular and Clinical Ophthalmology Basel, Complex Viruses Platform pour leur aide et leur soutien à la production d’AAV. Nous tenons également à remercier le professeur Jean Bennett, de la Perelman School of Medicine de l’Université de Pennsylvanie, pour la souche AAV8/BP2. Le travail animal est effectué à l’installation pour animaux de l’Université technique de Gebze. Pour cela, nous remercions Leyla Dikmetas et le professeur Uygar Halis Tazebay pour leur assistance technique et leur soutien à l’élevage. Nous tenons également à remercier la Dre Fatma Ozdemir pour ses commentaires sur le manuscrit. Ce travail est soutenu par TUBITAK, les subventions 118C226 et 121N275, et la subvention d’intégration de l’Université Sabanci.
matériels
| Name | Company | Catalog Number | Comments |
| 96-Well Semi-Skirted ddPCR plates | BioRad | 12001925 | ddPCR |
| Amicon Filter | Millipore | UFC910096 | AAV |
| C1000 TOUCH 96 DEEP WELLS | BioRad | 1851197 | ddPCR |
| C57BL/6JRj mice strain | Janvier | C57BL/6JRj | Mice |
| DG8 gaskets | BioRad | 1863009 | ddPCR |
| DG8 Cartridges | BioRad | 1864008 | ddPCR |
| DMEM | Lonza | BE12-604Q | AAV |
| DPBS | PAN BIOTECH | L 1825 | AAV |
| Droplet generation oil eva green | BioRad | 1864006 | ddPCR |
| Droplet reader oil | BioRad | 1863004 | ddPCR |
| FBS | PAN BIOTECH | p30-3306 | AAV |
| Foil seals for PX1 PCR Plate sealer | BioRad | 1814040 | ddPCR |
| Insulin Syringes | BD Medical | 320933 | Intravitreal injection |
| Isoflurane | ADEKA ILAC SANAYI VE TICARET | N01AB06 | anesthetic |
| Microinjector MM33 | World Precision Instruments | 82-42-101-0000 | Intravitreal injection |
| Micron IV | Phoenix Research Labs | Micron IV | Microscopy system based on 3-CCD color camera, frame grabber, and off-the-shelf software enables researchers to image mouse retinas. |
| Mydfrin (%2.5 phenylephrine hydrochloride) | Alcon | S01FB01 | pupil dilation |
| Nanofil Syringe 10 μl | World Precision Instruments | NANOFIL | Intravitreal injection |
| Needle RN G36, 25 mm, PST 2 | World Precision Instruments | NF36BL-2 | Intravitreal injection |
| PEI-MAX | Polyscience | 24765-1 | AAV |
| Penicillin-Streptomycin | PAN BIOTECH | P06-07100 | AAV |
| Plasmid pHGT1-Adeno1 | PlasmidFactory | PF1236 | AAV |
| Pluronic F-68 | Gibco | 24040032 | AAV |
| PX1 PCR Plate Sealer system | BioRad | 1814000 | ddPCR |
| QX200 ddPCR EvaGreen Supermix | BioRad | 1864034 | ddPCR |
| QX200 Droplet Reader/QX200 Droplet Generator | BioRad | 1864001 | ddPCR |
| SPLITTER FORCEP WATCHER MAKER - LENGTH = 13.5 CM | endostall medical | EJN-160-0155 | Retina isolation |
| Steril Syringe Filter | AISIMO | ASF33PS22S | AAV |
| Tissue Genomic DNA Kit | EcoSpin | E1070 | gDNA isolation |
| Tobradex (0.3% tobramycin / 0.1% dexamethasone) | Alcon | S01CA01 | anti-inflammatory / antibiotic |
| Tropamid (% 0.5 tropicamide) | Bilim Ilac Sanayi ve Ticaret AS. | S01FA06 | pupil dilation |
| Turbonuclease | Accelagen | N0103L | AAV |
| Viscotears (carbomer 2 mg/g) | Bausch+Lomb | S01XA20 | lubricant eye drop |
Références
- Herrmann, A. K., Grimm, D. High-throughput dissection of AAV-host interactions: The fast and the curious. Journal of Molecular Biology. 430 (17), 2626-2640 (2018).
- Pillay, S., Carette, J. E. Host determinants of adeno-associated viral vector entry. Current Opinion in Virology. 24, 124-131 (2017).
- Castle, M. J., Turunen, H. T., Vandenberghe, L. H., Wolfe, J. H. Controlling AAV tropism in the nervous system with natural and engineered capsids. Methods in Molecular Biology. 1382, 133-149 (2016).
- Agbandje-McKenna, M., Kleinschmidt, J. AAV capsid structure and cell interactions. Methods in Molecular Biology. 807, 47-92 (2011).
- Han, I. C., et al. Retinal tropism and transduction of adeno-associated virus varies by serotype and route of delivery (intravitreal, subretinal, or suprachoroidal) in rats. Human Gene Therapy. 31 (23-24), 1288-1299 (2020).
- Muraine, L., et al. Transduction efficiency of adeno-associated virus serotypes after local injection in mouse and human skeletal muscle. Human Gene Therapy. 31 (3-4), 233-240 (2020).
- Cronin, T., et al. Efficient transduction and optogenetic stimulation of retinal bipolar cells by a synthetic adeno-associated virus capsid and promoter. EMBO Molecular Medicine. 6 (9), 1175-1190 (2014).
- Higashimoto, T., et al. The woodchuck hepatitis virus post-transcriptional regulatory element reduces readthrough transcription from retroviral vectors. Gene Therapy. 14 (17), 1298-1304 (2007).
- Pinheiro, L. B., et al. Evaluation of a droplet digital polymerase chain reaction format for DNA copy number quantification. Annals in Chemistry. 84 (2), 1003-1011 (2012).
- Albayrak, C., et al. Digital quantification of proteins and mrna in single mammalian cells. Molecular Cell. 61 (6), 914-924 (2016).
- Lock, M., Alvira, M. R., Chen, S. J., Wilson, J. M. Absolute determination of single-stranded and self-complementary adeno-associated viral vector genome titers by droplet digital PCR. Hum Gene Therapy Methods. 25 (2), 115-125 (2014).
- Juttner, J., et al. Targeting neuronal and glial cell types with synthetic promoter AAVs in mice, non-human primates and humans. Nature Neuroscience. 22 (8), 1345-1356 (2019).
- Vandenberghe, L. H., et al. Efficient serotype-dependent release of functional vector into the culture medium during adeno-associated virus manufacturing. Human Gene Therapy. 21 (10), 1251-1257 (2010).
- Barben, M., Schori, C., Samardzija, M., Grimm, C. Targeting Hif1a rescues cone degeneration and prevents subretinal neovascularization in a model of chronic hypoxia. Molecular Neurodegeneration. 13 (1), 12(2018).
- Chan, K. Y., et al. Engineered AAVs for efficient noninvasive gene delivery to the central and peripheral nervous systems. Nature Neuroscience. 20 (8), 1172-1179 (2017).
- Guiet, R., Burri, O., Seitz, A. Open source tools for biological image analysis. Methods Molecular Biology. 2040, 23-37 (2019).
- Parks, M. M., et al. Variant ribosomal RNA alleles are conserved and exhibit tissue-specific expression. Science Advances. 4 (2), (2018).
- Aurnhammer, C., et al. Universal real-time PCR for the detection and quantification of adeno-associated virus serotype 2-derived inverted terminal repeat sequences. Human Gene Therapy Methods. 23 (1), 18-28 (2012).
- Maclachlan, T. K., et al. Preclinical safety evaluation of AAV2-sFLT01- a gene therapy for age-related macular degeneration. Molecular Therapy. 19 (2), 326-334 (2011).
- Stieger, K., et al. Detection of intact rAAV particles up to 6 years after successful gene transfer in the retina of dogs and primates. Molecular Therapy. 17 (3), 516-523 (2009).
- Gyorgy, B., et al. Allele-specific gene editing prevents deafness in a model of dominant progressive hearing loss. Nature Medicine. 25 (7), 1123-1130 (2019).
- Cox, D. B. T., et al. RNA editing with CRISPR-Cas13. Science. 358 (6366), 1019-1027 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon