Method Article
Digitale Tropfen-PCR-Methode zur Quantifizierung der AAV-Transduktionseffizienz in der Murinen Netzhaut
In diesem Artikel
Zusammenfassung
Dieses Protokoll zeigt, wie die AAV-Transduktionseffizienz in der Netzhaut der Maus mittels digitaler Tröpfchen-PCR (dd-PCR) in Verbindung mit AAV-Produktion im kleinen Maßstab, intravitrealer Injektion, retinaler Bildgebung und retinaler genomischer DNA-Isolierung quantifiziert werden kann.
Zusammenfassung
Viele Labore für netzhautzellbiologie verwenden heute routinemäßig Adeno-assoziierte Viren (AAVs) für Gen-Editing und regulatorische Anwendungen. Die Effizienz der AAV-Transduktion ist in der Regel kritisch, was sich auf die gesamten experimentellen Ergebnisse auswirkt. Eine der Hauptdeterminanten für die Transduktionseffizienz ist der Serotyp oder die Variante des AAV-Vektors. Derzeit sind verschiedene künstliche AAV-Serotypen und -Varianten mit unterschiedlichen Affinitäten zu Wirtszelloberflächenrezeptoren verfügbar. Für die retinale Gentherapie führt dies zu unterschiedlichen Transduktionswirkungsgraden für verschiedene retinale Zelltypen. Darüber hinaus können der Injektionsweg und die Qualität der AAV-Produktion auch die retinale AAV-Transduktionseffizienz beeinflussen. Daher ist es wichtig, die Effizienz verschiedener Varianten, Chargen und Methoden zu vergleichen. Die digitale Tröpfchen-PCR-Methode (dd-PCR) quantifiziert die Nukleinsäuren mit hoher Präzision und ermöglicht die durchführung einer absoluten Quantifizierung eines bestimmten Ziels ohne Standard oder Referenz. Mit hilfe der dd-PCR ist es auch möglich, die Transduktionseffizienzen von AAVs durch absolute Quantifizierung der AAV-Genomkopienzahlen innerhalb einer injizierten Netzhaut zu bewerten. Hier stellen wir eine einfache Methode zur Quantifizierung der Transduktionsrate von AAVs in Netzhautzellen mittels dd-PCR zur Verfügung. Mit geringfügigen Modifikationen kann diese Methodik auch die Grundlage für die Kopienzahlquantifizierung der mitochondrialen DNA sowie für die Bewertung der Effizienz der Basenbearbeitung sein, die für mehrere Netzhauterkrankungen und Gentherapieanwendungen von entscheidender Bedeutung ist.
Einleitung
Adeno-assoziierte Viren (AAVs) werden heute häufig für eine Vielzahl von retinalen Gentherapiestudien verwendet. AAVs bieten eine sichere und effiziente Art der Genabgabe mit weniger Immunogenität und weniger Genomintegrationen. Der AAV-Eintritt in die Zielzelle erfolgt durch Endozytose, die eine Bindung von Rezeptoren und Co-Rezeptoren auf der Zelloberfläche erfordert1,2. Daher hängt die Transduktionseffizienz von AAVs für verschiedene Zelltypen hauptsächlich vom Kapsid und seinen Wechselwirkungen mit den Wirtszellrezeptoren ab. AAVs haben Serotypen und jeder Serotyp kann unterschiedliche Zell- / Gewebetropismen und Transduktionseffizienzen aufweisen. Es gibt auch künstliche AAV-Serotypen und -Varianten, die durch chemische Modifikation des Viruskapsids, Produktion von Hybridkasiden, Peptidinsertion, Kapsidmuffling, gerichtete Evolution und rationale Mutagenese erzeugt werden3. Selbst geringfügige Veränderungen der Aminosäuresequenz oder der Kapsidstruktur können einen Einfluss auf Wechselwirkungen mit Wirtszellfaktoren haben und zu unterschiedlichen Tropismen führen4. Neben Kapsidvarianten können andere Faktoren wie der Injektionsweg und die Batch-to-Batch-Variation der AAV-Produktion die Transduktionseffizienz von AAVs in der neuronalen Netzhaut beeinflussen. Daher sind zuverlässige Methoden für den Vergleich von Transduktionsraten für verschiedene Varianten notwendig.
Die Mehrheit der Methoden zur Bestimmung der AAV-Transduktionseffizienz beruht auf der Expression des Reportergens. Dazu gehören fluoreszierende Bildgebung, Immunhistochemie, Western Blot oder histochemische Analyse des Reportergenprodukts5,6,7. Aufgrund der Größenbeschränkung von AAVs ist es jedoch nicht immer möglich, Reportergene einzubeziehen, um die Transduktionseffizienz zu überwachen. Die Verwendung starker Promotoren wie dem hybriden CMV-Enhancer/Chicken Beta-Actin oder woodchuck hepatitis post-transcriptional regulatory element (WPRE) als mRNA-Stabilisatorsequenz verkompliziert das Größenproblemweiter 8. Daher wird es auch von Vorteil sein, die Transduktionsrate von injizierten AAVs mit einer direkteren Methodik zu definieren.
Die digitale Tröpfchen-PCR (dd-PCR) ist eine leistungsstarke Technik zur Quantifizierung der Ziel-DNA aus winzigen Probenmengen. Die dd-PCR-Technologie hängt von der Verkapselung der Ziel-DNA und der PCR-Reaktionsmischung durch Öltröpfchen ab. Jede dd-PCR-Reaktion enthält Tausende von Tröpfchen. Jedes Tröpfchen wird als eigenständige PCR-Reaktion aufbereitet und analysiert9. Die Analyse von Tröpfchen ermöglicht die Berechnung der absoluten Kopienzahl von Ziel-DNA-Molekülen in jeder Probe mithilfe des Poisson-Algorithmus. Da die Transduktionseffizienz der AAVs mit der Kopienzahl von AAV-Genomen in der neuronalen Netzhaut korreliert, verwendeten wir die dd-PCR-Methode, um AAV-Genome zu quantifizieren.
Hier beschreiben wir eine dd-PCR-Methodik zur Berechnung der Transduktionseffizienz von AAV-Vektoren aus retinaler genomischer DNA6,10. Erstens wurden AAVs, die tdTomato-Reporter exprimieren, unter Verwendung des Small-Scale-Protokolls generiert und durch die dd-PCR-Methode11titeriert. Zweitens wurden AAVs intravitreal in die neuronale Netzhaut injiziert. Um die Transduktionseffizienz zu demonstrieren, haben wir zunächst die tdTomato-Expression mit Fluoreszenzmikroskopie und ImageJ-Software quantifiziert. Es folgte die Isolierung genomischer DNA zur Quantifizierung von AAV-Genomen in injizierten Netzhäuten mittels dd-PCR. Der Vergleich der tdTomato-Expressionsniveaus mit den transduzierten AAV-Genomen, die durch die dd-PCR quantifiziert wurden, zeigte, dass die dd-PCR-Methode die Transduktionseffizienz von AAV-Vektoren genau quantifizierte. Unsere Protokolle zeigten eine detaillierte Beschreibung einer dd-PCR-basierten Methodik zur Quantifizierung der AAV-Transduktionseffizienz. In diesem Protokoll zeigen wir auch die absolute Anzahl der AAV-Genome, die nach intravitrealen Injektionen transduziert werden, indem einfach der Verdünnungsfaktor nach der genomischen DNA-Isolierung und die dd-PCR-Ergebnisse verwendet werden. Insgesamt bietet dieses Protokoll eine leistungsfähige Methode, die eine Alternative zur Reporterexpression wäre, um die Transduktionseffizienz von AAV-Vektoren in der Netzhaut zu quantifizieren.
Protokoll
Alle experimentellen Protokolle wurden von der Ethikkommission der Sabanci Universität akzeptiert und die Experimente wurden in Übereinstimmung mit der Erklärung der "Association for Research in Vision and Ophthalmology" für die Verwendung von Tieren in der Forschung durchgeführt
1. Kleine AAV-Produktion12
- Kultivieren Sie HEK293T-Zellen unter Verwendung von 15-cm-Platten in vollständigen 10 ml DMEM / 10% FBS bis zu 70-80% Konfluenz.
- Die Transfektionsmischung ist mit 20 μg Hilfsplasmid (pHGT1-Adeno1), 7 μg Kapsidplasmid und 7 μg AAV2-CBA-tdTomato-WPRE-Vektor und 136 μL PEI-Lösung (1 mg/ml) in 5 ml DMEM herzustellen.
- Die 5 ml hergestellte Transfektionsmischung in eine Zellkulturschale mit 10 ml Nährmedien geben und die transfizierten Zellen für 48-60 h bei 37 °C inkubieren.
- Sammeln Sie das Medium 48-60 h nach der Transfektion und verdauen Sie es mit DNase I in einer Endkonzentration von 250 U/ml für 30 min bei 37 °C.
- Zentrifugieren Sie das aufgeschlossene Medium bei 4000 x g,4 °C für 30 min und filtrieren Sie es dann mit einem 0,22 μm Spritzenfilter in die vorbenetzte regenerierte Cellulosemembran für 100 kDa.
- Zentrifugieren Sie die regenerierte Cellulosemembran bei 4000 x g,4 °C für 30 min und entsorgen Sie das Medium.
- Waschen und zentrifugieren Sie die regenerierte Cellulosemembran mit PBS, das 0,001% Pluronic F-68 enthält, dreimal bei 4000 x g,4 °C für 30 min. Verwerfen Sie den PBS bei jedem Schritt.
- Sammeln Sie konzentrierte AAVs aus dem oberen Teil der regenerierten Cellulosemembran und Aliquot für weitere Anwendungen.
- 5 μL frisch hergestelltes AAV mit DNase I (0,2 HE/μl) für 15 min bei 37 °C zum Titerieren aufbereiten. Es folgt eine Inkubation von 10 min bei 95 °C, um sowohl die DNase I zu inaktivieren als auch die viralen Kapside abzubauen.
- Bereiten Sie eine 10-fache serielle Verdünnung aus verdauten AAVs mit 0,05% Pluronic F-68 vor. Verwenden Sie AAV2-ITR- und WPRE-Primer für die Titration (Tabelle 1). Die Titer wurden berechnet, indem die dd-PCR-Ergebnisse mit den Verdünnungsfaktoren multipliziert wurden. Die Ergebnisse wurden in Genomkopie/ml (GC/ml) umgewandelt.
HINWEIS: Die AAV-Konzentrationen für die AAV-Produktion in kleinem Maßstab werden voraussichtlich etwa 1 x 1012 GC/ml betragen. Dieses Protokoll gilt nicht für AAV-Stämme, die AAVs nicht effizient in Medien wie AAV213 freisetzen.
2. Intravitreale Injektion von AAV
-
Vorbereitung der Ausrüstung
- Bevor Sie beginnen, bereiten Sie 10 μL Mikrospritze mit einer 36 G stumpfen Nadel vor. Spülen Sie fünfmal mit 70% EtOH, fünfmal in ddH2O und schließlich fünfmal mit PBS.
- Laden Sie für jede Injektion die entsprechende Menge AAV in die Mikrospritze.
- Bereiten Sie die chirurgische Nadel (Naht, Seide, 6/0), Klebeband, Pinzette und Schere vor.
-
Intravitreale Injektion
- Tragen Sie vor der Narkose 1 Tropfen 0,5% Tropicamid und 2,5% Phenylephrinhydrochlorid (z. B. Mydfrin) mit Augentropfen auf, um die Pupillen zu erweitern.
- Anästhesieren Sie Mäuse mit Isofluran 5% (1 L/min) und fahren Sie während des Eingriffs mit 1,5% Isofluran (1 L/min) fort. Für die erste Induktion wird eine kleine Isoflurankammer verwendet.
- Überprüfen Sie die Tiefe der Anästhesie durch den Verlust des Aufrichtungsreflexes, des Entzugsreflexes und der Schwanzquetschreaktion.
- Tragen Sie vor dem Injektionsvorgang 0,3% Tobramycin und 0,1% ige sterile ophthalmologische Lösung mit Dexamethason auf jedes Auge auf. Die Anwendung von Augentropfen verhindert Trockenheit und übt entzündungshemmende und antibakterielle Wirkungen aus.
- Verwenden Sie den Operationshaken, um das obere Augenlid zu stabilisieren. Ziehen Sie sich leicht zurück, um den dorsalen Teil des Auges freizulegen. Kleben Sie den Haken an die Bank, um ihn in Position zu halten.
- Entfernen Sie die Bindehaut (weniger als 1 mm2)mit der gekrümmten Irisschere, um die Sklera des Auges unter dem Seziermikroskop freizulegen. Verwenden Sie eine frische und sterile Insulinspritze (30 G), um die Sklera zu punktieren.
- Führen Sie die Nadel der Mikrospritze mit einem Mikromanipulator durch dieselbe Punktion. Legen Sie die Nadelspitze hinter die Linse in die Mitte der Augenmuschel.
- Injizieren Sie 1 μL AAV2/BP2 und AAV2/PHP. S-Vektoren mit Konzentrationen von 1,63 x10 12 GC/ml und 1,7 x10 12 GC/ml gelangen langsam in den Glaskörper des Auges. Die Einspritzvolumenregelung erfolgt manuell. Lassen Sie die Nadel für 1 Minute an Ort und Stelle und ziehen Sie die Nadel langsam zurück.
- Lassen Sie das Augenlid los und tragen Sie eine entzündungshemmende und antibakterielle topische Gelbehandlung mit 0,3% Tobramycin und 0,1% Dexamethason auf die Augenmuschel auf. Überwachen und bewerten Sie die Mäuse in den folgenden Tagen gemäß dem Scoresheet in Bezug auf Aussehen (helle Augen, gepflegtes Fell, Buckeln), Verhalten (Aktivität, Ruhigstellung, Selbstverstümmelung) und Körpergewicht auf einer Skala von 0 bis 3. Jede Bedingung hat eine Punktzahl von 0-3 und eine Gesamtpunktzahl von 3 oder höher ist ein Beendigungskriterium.
- Legen Sie die Tiere nach der Genesung in den Käfig.
3. Fluoreszenz- und Fundusbildgebung
- Die Maus abseihen und die Pupille erweitern, indem Sie Tropfen von 0,5% Tropicamid und 2,5% Phenylephrinhydrochlorid in jedes Auge geben.
- Betäuben Sie das Tier mit dem oben genannten Verfahren mit Isofluran. Beurteilen Sie die Tiefe der Anästhesie sorgfältig, indem Sie die Pfoten kneifen.
- Legen Sie die Maus auf die Bildgebungsstufe und überprüfen Sie die richtige Dilatation, indem Sie die Funduskamera überprüfen. Tragen Sie topisches Gel (0,2% Carbomer 980) auf die Oberfläche der Augen auf, um die Augen vor Austrocknung unter Narkose zu schützen und es als Kopplungsgel für die Bildgebung zu verwenden.
- Manipulieren Sie die Bildgebungsstufe so, dass sie sich an den Revolver des Objektivs anpasst. Passen Sie die Bühne nach Bedarf an, um das Mausauge zu zentrieren. Sobald das Auge zentriert ist, bewegen Sie das Ziel langsam, bis es Kontakt mit dem Auge hat.
- Führen Sie eine Fundus- und Fluoreszenzbildgebung an beiden Augen durch, um die tdTomato-Expression 1 Woche und 2 Wochen nach intravitrealer Injektion zu verfolgen (Abbildung 3B)14. Nehmen Sie Fundus- und Fluoreszenzbilder mit identischen Einstellungen zum Vergleich der Reporterexpression.
4. Isolierung der Netzhaut
- Einschläfern von Tieren nach Fundus- und Fluoreszenzbildgebung durch CO2-Inhalation zur Netzhautentnahme nach 2 Wochen.
- Verschieben Sie den Augapfel mit Hilfe einer 13,5 cm Splitterzange nach vorne.
- Entfernen Sie die Linse zusammen mit dem übrig gebliebenen Glaskörper, indem Sie sanften Druck mit der Pinzette ausüben.
- Schneiden Sie die Verbindung des Augapfels zum Sehnerv mit einer präzise gekrümmten Pinzette ab und drücken Sie die Netzhaut vorsichtig mit der gleichen gekrümmten Pinzette zusammen.
- Übertragen Sie sezierte Netzhäute in ein Mikrozentrifugenröhrchen.
- Snap Freeze Retina, indem Sie die Röhrchen in den flüssigen Stickstoff legen.
5. Genomische DNA-Isolierung von Gewebe
- Isolieren Sie genomische DNA mit einem kommerzialisierten Proteinase K-Verdauungs-basierten genomischen Gewebe-DNA-Kit.
- Verdauen Sie die Netzhaut bei 55 °C, 200 x g für 30 min mit Proteinase K, die bereits im Kit enthalten ist.
- Führen Sie den RNase-Inkubationsschritt, die Waschschritte mit dem Waschpuffer I und II und schließlich den Elutionsschritt gemäß dem Protokoll des Herstellers durch.
- Fügen Sie nach der letzten Wäsche einen zusätzlichen Schleuderschritt hinzu, um Den Restethanol zu entfernen.
- Messen Sie die Konzentration der genomischen DNA und lagern Sie die Proben bei -20 °C.
6. Digitale Tröpfchen-PCR-Analyse von Netzhautproben von Mäusen zur Quantifizierung viraler Genome
- Verdünnung genomischer DNAs aus injizierter Netzhaut in 0,05% pluronic F-68 Lösung, um eine Endkonzentration von 1 ng/μL für jede Probe zu erreichen.
- Führen Sie separate Reaktionen für WPRE und 18S mit zielspezifischen Primern mit einer Endkonzentration von 125 nM für jeden Primer durch.
- Führen Sie die Tröpfchengenerierung in einer 20 μL PCR-Reaktionsmischung durch, einschließlich 1 ng genomischer DNA, 125 nM-Primern und 2x Evagreen-Supermix.
- Laden Sie die Probenmischung in den mittleren Teil der Patrone, um Tröpfchen zu erzeugen.
- Nachdem die Probe in die Kartusche geladen wurde, geben Sie 70 μL des Tröpfchenerzeugungsöls in die untere Reihe der Kartusche.
- Setzen Sie die Dichtung auf die Patrone und legen Sie sie in den Tröpfchengenerator. Stellen Sie sicher, dass kein Spalt zwischen der Dichtung und der Kartusche besteht.
- Nehmen Sie vorsichtig etwa 40 μL Tröpfchen, die im oberen Brunnen erzeugt werden. Geben Sie die Tröpfchenlösung in die halbverzierte 96-Well-PCR-Reaktionsplatte.
- Versiegeln Sie die PCR-Platte mit einem PCR-Plattenversiegeler unter Verwendung von Aluminium-Dichtfolie.
- Legen Sie die PCR-Platte in einen 96-Well-Heißsiegel-Thermocycler. Verwenden Sie das PCR-Protokoll; 95 °C für 5 min, 40 Zyklen von 95 °C für 30 s, 60 °C für 1 min, 4 °C für 5 min, 90 °C für 5 min und 4 °C unendlich halten. Wenden Sie bei jedem Zyklus eine Rampenrate von 2 °C/s an, um sicherzustellen, dass die Tröpfchen während des Zyklus für jeden Schritt die richtige Temperatur erreichen
- Legen Sie die PCR-Platte in den Tröpfchenleser ein, um die Tröpfchen mit der dd-PCR-Software zu quantifizieren. Eine Vorlage wird mit spezifischen Einstellungen für evagreen eingerichtet.
- Analysieren Sie die Daten mithilfe eines 1D-Diagrammdiagramms mit jeder Probe für die Fluoreszenzintensität im Vergleich zur Tröpfchenzahl. Der Schwellenwert ist so angeordnet, dass die Tröpfchen oberhalb des Schwellenwerts als positiv und die darunter liegenden als negativ zugeordnet werden. Es folgt die Anwendung des Poisson-Algorithmus zur Bestimmung der Ausgangskonzentration der Ziel-DNA in Kopieneinheiten/μL. Das Verhältnis von WPRE zu 18S wird für die Messung der AAV-Transduktionseffizienz berechnet.
Ergebnisse
Die AAV-Produktion im kleinen Maßstab ist eine schnelle und effiziente Methode, die Vektoren für intravitreale Injektionen liefert (Abbildung 1). Die AAV-Produktion im kleinen Maßstab ergibt in der Regel Titer im Bereich von 1 x 1012 GC/ml, was ausreicht, um die Reporterexpression in der Netzhaut nachzuweisen (Abbildung 2). Die Titerierung von AAV mittels dd-PCR liefert konsistente Ergebnisse. ITR2- und WPRE-spezifische Primer werden routinemäßig verwendet und die Ausgangskonzentration jedes Zielmoleküls wurde mit der dd-PCR-Software durch Modellierung als Poisson-Verteilung berechnet. Die Berechnungen wurden durch Multiplikation der Verdünnungsfaktoren abgeschlossen und in Genomkopie pro ml umgewandelt. AAVs, die für dieses Protokoll hergestellt werden, hatten Titer von 1,63 x10 12 GC/ml bzw. 1,7x10 12 GC/ml für AAV2/BP2 bzw. AAV2/PHP.S7,15. Zum Vergleich wurden für beide Stämme identische tdTomato-Expressionskonstrukte verwendet. Wir injizierten ungefähr gleiche Titer von AAV für die Quantifizierung der AAV-Transduktionseffizienz. Nach der Injektion von 1 μL aus den oben genannten Konzentrationen von AAVs wurden die Tiere zu Zeitpunkten von 1 Woche und 2 Wochen abgebildet. Ein Fundus- und Fluoreszenzbildgebungssystem wurde sowohl für AAV2/BP2 als auch für AAV2/PHP verwendet. S injizierte Netzhaut, die ähnliche Reporterexpressionsprofile erzeugt, mit Ausnahme von Netzhaut #1 (Abbildung 3A, B). Die TdTomato-Expression wurde mit der ImageJ-Software für den durchschnittlichen Grauwert (mittlere Fluoreszenzintensität) quantifiziert, um die Transduktionseffizienz und die Reporterexpression zu vergleichen (Abbildung 3A,B). Dies ist im Grunde die Summe der Grauwerte aller Pixel in der Auswahl geteilt durch die Anzahl der Pixel16. Fluoreszierende Bilder von Retina #1 zeigten nur einen kleinen Bereich der tdTomato-Expression, höchstwahrscheinlich aufgrund von Rückfluss oder Leckage nach intravitrealen Injektionen. In Übereinstimmung mit diesem Befund betrug die Fluoreszenzintensität der Netzhaut Nr. 1 0,9, was im Vergleich zu allen Netzhäuten, die intravitreal injiziert und abgebildet wurden, am niedrigsten war. Die mittlere Fluoreszenzintensität lag zwischen 4 - 11. Dies zeigte, dass die Quantifizierung von Fluoreszenzbildern der tdTomato-Expression erfolgreich war und mit den gezeigten Bildern korrelierte (Abbildung 3A,B).
Nach der Bildgebung der Netzhaut wurde die genomische DNA nach 2 Wochen aus der injizierten Netzhaut isoliert. Die dd-PCR wurde unter Verwendung von WPRE-Primern zur AAV-Genomquantifizierung durchgeführt. Maus 18S wurde zur Normalisierung von AAV-Genomen auf das Mausgenom verwendet (Abbildung 3C)17. In Übereinstimmung mit der mittleren Fluoreszenzintensität und den Bildern hatte Netzhaut # 1 eine sehr niedrige AAV-Genomkopienzahl im Vergleich zu 18S, 0,011-mal niedriger im Vergleich zu Netzhaut # 2. Darüber hinaus ergeben die Netzhäute # 2, 3, 4 und 5 ähnliche Falzniveauunterschiede. Die Falzunterschiede im Vergleich zu Netzhaut # 2 für Netzhaut # 3, 4 und 5 betrugen 1,75, 0,55 bzw. 0,99. Unter diesen ergab retina # 3 sowohl die höchste Fluoreszenzintensität als auch die AAV-Genomkopienzahl. Dies zeigte bereits, dass die Quantifizierung von transduzierten AAV-Genomen mit dd-PCR mit der beobachteten Fluoreszenzintensität und damit mit der beobachteten tdTomato-Expression korreliert. Die dd-PCR-Methode ermöglichte es uns auch, eine absolute Quantifizierung von transduzierten AAVs durchzuführen. Die absolute Quantifizierung des gesamten AAV-Genoms pro Netzhaut wurde einfach berechnet, indem die aus der dd-PCR identifizierte Gesamtzahl und der Verdünnungsfaktor für genomische DNA multipliziert wurden. Dies führte zu ähnlichen Ergebnissen im Vergleich zur 18S-normalisierten AAV-Genomtransduktionseffizienz (Abbildung 3D).

Abbildung 1: Flussdiagramm der experimentellen Verfahren. Auf die AAV-Produktion im kleinen Maßstab folgt die dd-PCR-basierte AAV-Titerierung. AAVs werden intravitreal in die netzhaut des Erwachsenen injiziert. Die Nachsorge der Tiere wurde mit einem Fundus- und Fluoreszenzbildgebungssystem durchgeführt, um die Expression des Reporters zu analysieren. Fundus- und Fluoreszenzbilder wurden zu Zeitpunkten von 1 Woche und 2 Wochen aufgenommen. Genomische DNA wird aus injizierten Netzhäuten für die Analyse mit dd-PCR isoliert, um das gesamte AAV-Genom zu quantifizieren, das in der injizierten Netzhaut vorhanden ist. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: AAV-Titerierung mit der dd-PCR-Methode. (A) Die Dd-PCR-Methode profitiert von verkapselten PCR-Reaktionen innerhalb eines Tröpfchens. Nach der PCR-Reaktion mittels Evagreen-Chemie wurden positive und negative Tröpfchen für das Zielgen analysiert, um die absolute Anzahl der Ziel-DNAs innerhalb einer Lösung zu bestimmen. (rote Kästchen in grünen Tröpfchen). (B) Repräsentative dd-PCR-Daten für die AAV-Titerierung. Mehrere Chargen von AAVs wurden mit ITR2-Primern titeriert. Positive und negative Tröpfchen werden oberhalb und unterhalb des Schwellenwerts getrennt (violette Linie). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
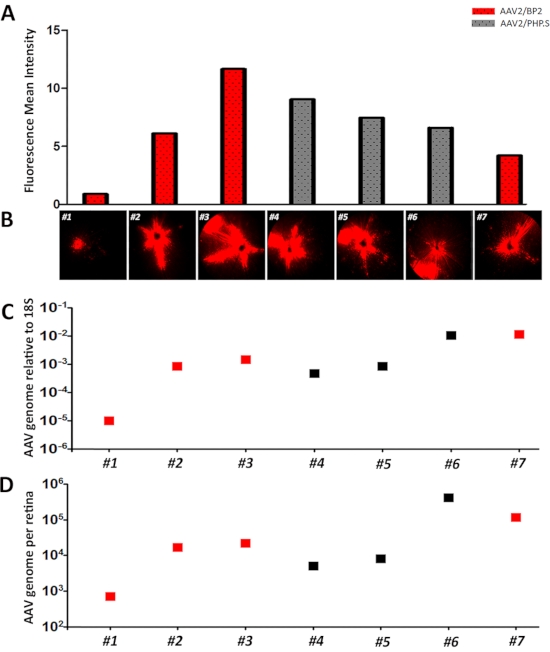
Abbildung 3: Quantifizierung von transduzierten AAV-Genomen in der Netzhaut mittels dd-PCR. 16 Wochen alte Wildtyptiere wurden intravitreal mit AAV2/BP2 und AAV2/PHP injiziert. S-Vektoren mit Konzentrationen von 1,63 x10 12 GC/ml bzw. 1,7 x10 12 GC/ml. Beide AAVs hatten das identische tdTomato-Expressionskonstrukt und das Injektionsvolumen für alle AAVs betrug 1 μL. Injizierte Netzhäute (1-7) wurden mit Fundus- und Fluoreszenzbildgebung verfolgt. Die aufgenommenen Bilder wurden mit der Image J-Software quantifiziert. Trotz des Kapsidunterschieds hatten alle Tiere einen mittleren Fluoreszenzintensitätswert(durchschnittlicher Grauwert)zwischen 4 und 11, mit Ausnahme der Netzhaut Nr. 1, die die niedrigste Intensität, 0,9, und eine schwache tdTomato-Expression aufwies. Rote und graue Spalten sind AAV2/BP2 und AAV2/PHP. S injizierte Netzhaut bzw. (A-B). Die dd-PCR wurde unter Verwendung von WPRE-Primern für das AAV-Genom und 18S-Primer für die Normalisierung zum Zeitpunkt von 2 Wochen durchgeführt. Retina # 1 zeigte auch deutlich niedrigere AAV-Kopienzahlen pro 18S-Kopien (C). Die gesamten AAV-Genome pro Netzhaut wurden auch unter Verwendung der WPRE-Kopienzahl und des Verdünnungsfaktors für die genomische DNA-Isolierung berechnet (D). Rote und graue Quadrate sind AAV2/BP2 und AAV2/PHP. S injizierte Netzhaut. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| ddPCR Primer | |
| ITR2 F | GGAACCCCTAGTGATGGAGTTT |
| ITR2 R | CGGCCTCAGTGAGCGAGA |
| WPRE F | GGCTGTTGGGCACTGACAA |
| WPRE R | CCAAGGAAAGGACGATGATTTC |
| 18S F | GGCCGTTCTTAGTTGGTGGA |
| 18S R | CCCGGACATCTAAGGGCATC |
Tabelle 1: dd-PCR-Primersequenzen. Sequenzen von Vorwärts- und Rückwärtsprimern für WPRE, ITR2 und Maus 18S.
Diskussion
In diesem Protokoll haben wir zwei AAV-Vektoren mit unterschiedlichen Kapsidproteinen generiert und diese dann entsprechend titeriert. Einer der wichtigsten Schritte dieses Protokolls besteht darin, ausreichende Mengen an AAVs zu erzeugen, die nach der Transduktion12,13eine nachweisbare Reporterexpression ergeben.
Die Titerierung von AAVs ist auch ein wichtiger Faktor, um die Dosierung von AAV für intravitreale Injektionen anzupassen. Sobald diese wichtigen Kriterien erreicht sind, ist es möglich, die Transduktionseffizienz von AAVs mit der dd-PCR-Methodik zu quantifizieren.
Viele Labore verwenden die quantitative PCR -Methode (qPCR) für die AAV-Titerierung. Die qPCR-basierte absolute Quantifizierungsmethode erfordert eine Standardkurve, die Verdünnungen bekannter Mengen der Ziel-DNA18 aufweist. Die dd-PCR erfordert jedoch keine Standardkurve, da sie die Gesamtzahl der Zielmoleküle in einer bestimmten Probe direkt quantifiziert, indem sie die positiven Tröpfchen nachweist, die mindestens eine Kopie der Ziel-DNA aufweisen. Da es sich bei der dd-PCR um eine Endpunktanalyse handelt und keine Standardkurve erforderlich ist, sind die Effizienzprobleme, die während der qPCR-Reaktion aufgetreten sind, für die dd-PCR weniger besorgniserregend wie pcR mit niedrigem Wirkungsgrad oder die Qualität der Standardkurven 9. Die dd-PCR-Methodik kann auf Proben angewendet werden, die bereits für die qPCR vorbereitet sind. Wie bereits im Abschnitt Methoden für die AAV-Titerierung erwähnt, erfordert die dd-PCR verdünnte Proben. Dies ist ein kritischer Schritt, da die Probenkonzentration so eingestellt werden sollte, dass genügend negative Tröpfchen vorhanden sind, um den Poisson-Algorithmus auszuführen. Mit anderen Worten, es ist nicht möglich, die dd-PCR-Reaktion mit Proben mit zu hohen Konzentrationen von Ziel-DNA durchzuführen, die keine negativen Tröpfchen ergeben.
Sowohl für AAV-Titerierungs- als auch für Transduktionseffizienzmessungen können unterschiedliche Zielsätze verwendet werden. Für das AAV-Titering verwenden wir hauptsächlich AAV2 ITR-spezifische Primer aufgrund des AAV2-Backbones in unseren Konstrukten. Es ist auch möglich, WPRE und andere genspezifische Ziele in Abhängigkeit von dem analysierten AAV-Konstrukt zu verwenden.
Um die Transduktionseffizienz von AAVs in der neuronalen Netzhaut zu bewerten, haben wir die dd-PCR-Methode unter Verwendung ganzer Netzhautproben angewendet, um die Gesamtmenge an AAV-Genomen pro Netzhaut zu quantifizieren. Wir bewerteten die Genauigkeit der Methodik anhand eines AAV-Vektors, der den tdTomato-Reporter ausdrückt. Vergleich der dd-PCR-Ergebnisse mit den tdTomato-Expressionsniveaus korreliert mit Ausnahme der beiden Ausreißer. Die Netzhäute #6 und #7 lieferten überschüssige AAV-Genomkopien, obwohl sie im Vergleich zu den Netzhäuten #2,3 4 oder 5 ähnliche Fluoreszenzintensitäten aufwiesen. Dies kann daran liegen, dass diese Netzhaut höhere Transduktionsraten mit begrenzten Expressionsniveaus aufweisen oder teilweise mit dauerhaften AAV-Partikeln oder Vektor-DNA innerhalb der Glaskörper- oder neuronalen Netzhautzusammenhängen 19,20. Insgesamt war dies der einzige limitierende Faktor für unsere Methode und kann leicht unter anderen Proben identifiziert werden. Wir quantifizierten auch die Gesamtzahl der AAV-Genome pro Netzhaut, was eine der Stärken dieser Methodik war. Dies ermöglicht den Vergleich von Daten zwischen verschiedenen Labors und Chargen von Tieren. Daher kann diese Methode leicht angewendet werden, um die Transduktionseffizienz eines neuen Serotyps, einer neuen Variante oder einer Batch-zu-Batch-Variation für AAV-Vektoren zu bewerten, die keinen Reporterausdruck haben, und uns hilft, Daten bei verschiedenen Einstellungen zu vergleichen. Dies ist entscheidend für AAV-Vektoren, die aufgrund von Größenbeschränkungen den Ausdruck eines Reporters nicht zulassen.
Diese Methode bietet auch eine Grundlage für andere Arten von sensitiven Assays, die Ziel-DNA verwenden. Mit den identischen Protokollen können wir mitochondriale Genomkopienzahlen mit geeigneten Primern quantifizieren. Weitere Verbesserungen sind ebenfalls möglich, einschließlich der Dissoziation der Netzhaut und der Durchflusszytometrie-Sortierschritte, um die Ziel-DNA entweder in einzelnen Zellen oder in Zellchargen zu quantifizieren. Darüber hinaus ist es auch möglich, die Korrektureffizienz der Basenbearbeitung mit dieser Methode21,22zubewerten,die auch für mehrere Netzhauterkrankungen und Gentherapien kritisch ist.
Offenlegungen
Die Autoren haben nichts preiszugeben.
Danksagungen
Wir danken Oezkan Keles, Josephine Jüttner und Prof. Botond Roska, Institut für Molekulare und Klinische Ophthalmologie Basel, Complex Viruses Platform für ihre Hilfe und Unterstützung bei der AAV-Produktion. Wir möchten uns auch bei Prof. Jean Bennett, Perelman School of Medicine, der University of Pennsylvania, für den AAV8/BP2-Stamm bedanken. Tierarbeiten werden in der Tieranlage der Technischen Universität Gebze durchgeführt. Dafür danken wir Leyla Dikmetas und Prof. Uygar Halis Tazebay für die technische Hilfe und Unterstützung bei der Tierhaltung. Wir danken auch Dr. Fatma Özdemir für ihre Anmerkungen zum Manuskript. Diese Arbeit wird durch TUBITAK, Fördernummern 118C226 und 121N275, und Sabanci University Integration Grant unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 96-Well Semi-Skirted ddPCR plates | BioRad | 12001925 | ddPCR |
| Amicon Filter | Millipore | UFC910096 | AAV |
| C1000 TOUCH 96 DEEP WELLS | BioRad | 1851197 | ddPCR |
| C57BL/6JRj mice strain | Janvier | C57BL/6JRj | Mice |
| DG8 gaskets | BioRad | 1863009 | ddPCR |
| DG8 Cartridges | BioRad | 1864008 | ddPCR |
| DMEM | Lonza | BE12-604Q | AAV |
| DPBS | PAN BIOTECH | L 1825 | AAV |
| Droplet generation oil eva green | BioRad | 1864006 | ddPCR |
| Droplet reader oil | BioRad | 1863004 | ddPCR |
| FBS | PAN BIOTECH | p30-3306 | AAV |
| Foil seals for PX1 PCR Plate sealer | BioRad | 1814040 | ddPCR |
| Insulin Syringes | BD Medical | 320933 | Intravitreal injection |
| Isoflurane | ADEKA ILAC SANAYI VE TICARET | N01AB06 | anesthetic |
| Microinjector MM33 | World Precision Instruments | 82-42-101-0000 | Intravitreal injection |
| Micron IV | Phoenix Research Labs | Micron IV | Microscopy system based on 3-CCD color camera, frame grabber, and off-the-shelf software enables researchers to image mouse retinas. |
| Mydfrin (%2.5 phenylephrine hydrochloride) | Alcon | S01FB01 | pupil dilation |
| Nanofil Syringe 10 μl | World Precision Instruments | NANOFIL | Intravitreal injection |
| Needle RN G36, 25 mm, PST 2 | World Precision Instruments | NF36BL-2 | Intravitreal injection |
| PEI-MAX | Polyscience | 24765-1 | AAV |
| Penicillin-Streptomycin | PAN BIOTECH | P06-07100 | AAV |
| Plasmid pHGT1-Adeno1 | PlasmidFactory | PF1236 | AAV |
| Pluronic F-68 | Gibco | 24040032 | AAV |
| PX1 PCR Plate Sealer system | BioRad | 1814000 | ddPCR |
| QX200 ddPCR EvaGreen Supermix | BioRad | 1864034 | ddPCR |
| QX200 Droplet Reader/QX200 Droplet Generator | BioRad | 1864001 | ddPCR |
| SPLITTER FORCEP WATCHER MAKER - LENGTH = 13.5 CM | endostall medical | EJN-160-0155 | Retina isolation |
| Steril Syringe Filter | AISIMO | ASF33PS22S | AAV |
| Tissue Genomic DNA Kit | EcoSpin | E1070 | gDNA isolation |
| Tobradex (0.3% tobramycin / 0.1% dexamethasone) | Alcon | S01CA01 | anti-inflammatory / antibiotic |
| Tropamid (% 0.5 tropicamide) | Bilim Ilac Sanayi ve Ticaret AS. | S01FA06 | pupil dilation |
| Turbonuclease | Accelagen | N0103L | AAV |
| Viscotears (carbomer 2 mg/g) | Bausch+Lomb | S01XA20 | lubricant eye drop |
Referenzen
- Herrmann, A. K., Grimm, D. High-throughput dissection of AAV-host interactions: The fast and the curious. Journal of Molecular Biology. 430 (17), 2626-2640 (2018).
- Pillay, S., Carette, J. E. Host determinants of adeno-associated viral vector entry. Current Opinion in Virology. 24, 124-131 (2017).
- Castle, M. J., Turunen, H. T., Vandenberghe, L. H., Wolfe, J. H. Controlling AAV tropism in the nervous system with natural and engineered capsids. Methods in Molecular Biology. 1382, 133-149 (2016).
- Agbandje-McKenna, M., Kleinschmidt, J. AAV capsid structure and cell interactions. Methods in Molecular Biology. 807, 47-92 (2011).
- Han, I. C., et al. Retinal tropism and transduction of adeno-associated virus varies by serotype and route of delivery (intravitreal, subretinal, or suprachoroidal) in rats. Human Gene Therapy. 31 (23-24), 1288-1299 (2020).
- Muraine, L., et al. Transduction efficiency of adeno-associated virus serotypes after local injection in mouse and human skeletal muscle. Human Gene Therapy. 31 (3-4), 233-240 (2020).
- Cronin, T., et al. Efficient transduction and optogenetic stimulation of retinal bipolar cells by a synthetic adeno-associated virus capsid and promoter. EMBO Molecular Medicine. 6 (9), 1175-1190 (2014).
- Higashimoto, T., et al. The woodchuck hepatitis virus post-transcriptional regulatory element reduces readthrough transcription from retroviral vectors. Gene Therapy. 14 (17), 1298-1304 (2007).
- Pinheiro, L. B., et al. Evaluation of a droplet digital polymerase chain reaction format for DNA copy number quantification. Annals in Chemistry. 84 (2), 1003-1011 (2012).
- Albayrak, C., et al. Digital quantification of proteins and mrna in single mammalian cells. Molecular Cell. 61 (6), 914-924 (2016).
- Lock, M., Alvira, M. R., Chen, S. J., Wilson, J. M. Absolute determination of single-stranded and self-complementary adeno-associated viral vector genome titers by droplet digital PCR. Hum Gene Therapy Methods. 25 (2), 115-125 (2014).
- Juttner, J., et al. Targeting neuronal and glial cell types with synthetic promoter AAVs in mice, non-human primates and humans. Nature Neuroscience. 22 (8), 1345-1356 (2019).
- Vandenberghe, L. H., et al. Efficient serotype-dependent release of functional vector into the culture medium during adeno-associated virus manufacturing. Human Gene Therapy. 21 (10), 1251-1257 (2010).
- Barben, M., Schori, C., Samardzija, M., Grimm, C. Targeting Hif1a rescues cone degeneration and prevents subretinal neovascularization in a model of chronic hypoxia. Molecular Neurodegeneration. 13 (1), 12(2018).
- Chan, K. Y., et al. Engineered AAVs for efficient noninvasive gene delivery to the central and peripheral nervous systems. Nature Neuroscience. 20 (8), 1172-1179 (2017).
- Guiet, R., Burri, O., Seitz, A. Open source tools for biological image analysis. Methods Molecular Biology. 2040, 23-37 (2019).
- Parks, M. M., et al. Variant ribosomal RNA alleles are conserved and exhibit tissue-specific expression. Science Advances. 4 (2), (2018).
- Aurnhammer, C., et al. Universal real-time PCR for the detection and quantification of adeno-associated virus serotype 2-derived inverted terminal repeat sequences. Human Gene Therapy Methods. 23 (1), 18-28 (2012).
- Maclachlan, T. K., et al. Preclinical safety evaluation of AAV2-sFLT01- a gene therapy for age-related macular degeneration. Molecular Therapy. 19 (2), 326-334 (2011).
- Stieger, K., et al. Detection of intact rAAV particles up to 6 years after successful gene transfer in the retina of dogs and primates. Molecular Therapy. 17 (3), 516-523 (2009).
- Gyorgy, B., et al. Allele-specific gene editing prevents deafness in a model of dominant progressive hearing loss. Nature Medicine. 25 (7), 1123-1130 (2019).
- Cox, D. B. T., et al. RNA editing with CRISPR-Cas13. Science. 358 (6366), 1019-1027 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten