Method Article
Imagerie confocale à haute résolution de la barrière hémato - encéphalique : imagerie, Reconstruction 3D et la Quantification de la transcytose
Dans cet article
Résumé
Nous présentons ici un protocole microscopie d’imagerie à haute résolution et une reconstruction tridimensionnelle de la souris, unité neurovasculaire et la barrière hémato - encéphalique, en utilisant des sections flottant de cerveau. Cette méthode permet la visualisation, l’analyse et quantification des organites intracellulaires à la BHE.
Résumé
La barrière sang - encéphalique (BHE) est une interface multicellulaire dynamique qui réglemente le transport des molécules entre le cerveau et la circulation. Transcytose à travers la BHE réglemente la fourniture des anticorps thérapeutiques, métabolites et hormones pour le parenchyme du cerveau. Nous présentons ici un protocole qui allie immunofluorescence des sections flottantes à balayage laser microscopie confocale et analyse d’images pour visualiser les organites subcellulaires au sein des cellules endothéliales à la BHE. La combinaison de cet ensemble de données avec logiciel d’analyse image 3D permet la segmentation semi-automatique et quantification du volume capillaire et superficie, ainsi que le nombre et l’intensité des organites intracellulaires à la BHE. La détection des immunoglobulines endogènes de la souris (IgG) au sein de vésicules intracellulaires et leur quantification à la BHE est utilisée pour illustrer la méthode. Ce protocole peut potentiellement être appliqué à l’étude des mécanismes contrôlant la transcytose BBB de différentes molécules in vivo.
Introduction
La barrière sang - encéphalique (BHE) est une barrière cellulaire continue formée par les cellules endothéliales, péricytes, neurones et les astrocytes qui sépare le système nerveux central (CNS) de la circulation de sang1. La réglementation du transport à travers la BHE joue un rôle crucial dans le maintien de l’homéostasie du cerveau et est véhiculée par des propriétés spécialisées des cellules endothéliales du cerveau (BECs). La présence de jonctions intercellulaires entre les BECs et un faible taux basal de transcytose limitent le transport paracellulaire et transcellulaire de molécules par voie sanguine, respectivement2. Récemment, la voie de la transcytose à BECs a été mobilisée pour améliorer la prestation des thérapeutiques grosses molécules au cerveau3,4. Cependant, les mécanismes de la transcytose à travers la BHE n’ont pas encore été complètement caractérisé5,6.
Beaucoup de travail a été fait in vitro afin de décrypter les mécanismes cellulaires et moléculaires régissant le transport intracellulaire à travers BECs7,8,9,10, 11, mais ces systèmes ne parviennent pas à récapituler l’architecture complexe et la physiologie de l’unité neurovasculaire (UNV). En revanche, des études in vivo12,13 fournir des informations quantitatives détaillées sur les taux de transport à travers la BHE mais ne fournissent pas de mieux comprendre les mécanismes intracellulaires de transport. Par conséquent, étudie les composants cellulaires et intracellulaires de l’UNV in vivo et ex vivo reste très difficile14. Seulement un nombre limité de techniques est susceptible d’analyser des structures subcellulaires dans les cellules de l’UNV. La plupart des études utilisent la microscopie électronique, mais cette technique est limitée par les protocoles complexes nécessaires à la préparation du tissu adéquate et la manipulation des échantillons. Par conséquent, nous avons établi une méthodologie basée sur la microscopie confocal de haute résolution qui faciliterait le traitement des échantillons de cerveau, l’analyse et la quantification des compartiments subcellulaires dans les cellules de l’UNV.
Nous décrivons ici un protocole qui utilise souris cerveau flottant sections pour effectuer l’imagerie quantitative du BBB et NVU au niveau cellulaire et subcellulaire. Nous avons testé et validé un certain nombre d’anticorps d’image et de reconstruire l’UNV en trois dimensions. En outre, ce protocole permet l’imagerie à la résolution optique maximale de diffraction limitée des organelles dans les capillaires du cerveau. Ainsi que l’analyse d’image, ce protocole peut être utilisé pour étudier le transport intracellulaire des macromolécules à travers la BHE dans des conditions expérimentales différentes, par exemple dans les modèles souris de maladies de la neurodégénérescence.
Protocole
approbation déontologique pour cette étude a été fournie par le Federal Food Safety and Office vétérinaire de la Suisse. Toutes les expériences sur des animaux ont été menées dans le strict respect de l’ordonnance sur la protection animale et le bien-être, ainsi que selon les règles de l’Association pour l’évaluation et l’accréditation du laboratoire Animal Care International (AAALAC).
1. génération de cerveau mobiles Sections
- pour assurer une qualité optimale d’échantillon, préparer une nouvelle solution de 2 % de paraformaldéhyde (PFA) dans une solution saline tampon de phosphate (PBS) le jour de la perfusion de.
ATTENTION : PFA est modérément toxique par contact avec la peau et probablement cancérogène. Gants en nitrile permet de gérer la PFA et de préparer la solution sous une hotte chimique.- Préparer 60 mL de solution PFA par animal.
- Bring PFA en solution en augmentant le pH à 180-200 µL d’une solution KOH de 5 M pour chaque 100 mL de PBS et de chauffage de la solution jusqu'à 60 ° C.
NOTE : NaOH ne doit pas être utilisé car elle influence négativement préservation des tissus lors de la fixation. - Laisser refroidir les solution à température ambiante et amener le pH à 7.4 à l’aide de HCl filtrer la solution à l’aide de papier filtre (voir la Table des matières).
- Effectuer une fixation toute souris par perfusion de transcardial comme décrit précédemment 15 avec quelques modifications. Tout d’abord, rincer le sang de la vascularisation avec 20 mL de PBS puis perfuse avec 40 mL de 2 % PFA.
- Enlever le cerveau du crâne comme décrit précédemment 15.
- Plonger un fraîchement 2 % PFA et sous perfusion cérébrale dans 20 mL de 2 % PFA pendant 7 h à 4 ° C d’après fixation.
NOTE : Augmenter l’incubation en PFA est déconseillée car elle peut empêcher la détection des structures intracellulaires. - Abondamment laver le cerveau avec du PBS glacee.
- Embed fixé des cerveaux dans l’agarose.
- Préparer un gel d’agarose 3 % solution de PBS à l’aide d’un four à micro-ondes pour le chauffage. Agiter doucement la solution pour refroidir mais éviter de solidification.
- Sécher l’excès de PBS autour du cerveau avant l’immersion dans la solution de gel d’agarose dans un récipient en plastique. Faire pivoter le cerveau à l’intérieur de l’agarose pour enlever les bulles d’air. Permettre l’agarose à se solidifier en refroidissant sur glace
- Retirer le bloc d’agarose dans le conteneur en plastique soigneusement et couper un cube autour du cerveau à l’aide d’une lame de rasoir. Monter le cerveau sur un porte-échantillon vibratome (voir Table des matières) à l’aide de colle cyanoacrylate (voir Table des matières). Prévoir un délai suffisant pour la colle se solidifie avant de procéder à la découpe.
- Transfert le cerveau dans le porte-échantillon dans le plateau de tampon vibratome rempli avec du PBS. Utilisez le vibratome à l’article coupes de cerveau de 100 µm (sagittales ou coronales). Recueillir des sections de cerveau dans une plaque de 6 puits précédemment rempli de PBS.
- Dès la finition de cerveau de sectionnement, soigneusement sortir PBS et les remplacer par une solution de 1:1 de PBS/glycérol.
- Conserver les sections en PBS/glycérol à -20 ° C.
2. Cellules ou organites étiquetage par Immunofluorescence souillant
- soigneusement transfert une partie du cerveau dans un puits d’une plaque 24 puits rempli précédemment avec 500 µL de PBS / puits. La section restera dans le même puits jusqu'à la fin de la procédure.
- Rincer la section deux fois avec 500 µL de PBS pendant 5 min sous agitation modérée.
- Enlever le PBS et effectuer le blocage simultané et perméabilisation des tranches de cerveau.
- Préparer une solution de blocage et la perméabilisation avec 0,3 % le sérum âne Triton-X et 10 % dans du PBS.
NOTE : Sérum de l’âne peut être remplacé par sérum de chèvre pour correspondre à l’espèce hôte spécifique anticorps secondaire utilisé. - Incuber des sections avec 250 µL de solution de blocage et de perméabilisation pendant 1 h à température ambiante sous agitation modérée.
- Préparer une solution de blocage et la perméabilisation avec 0,3 % le sérum âne Triton-X et 10 % dans du PBS.
- Enlever la solution et ajouter une solution de PBS contenant 5 % de sérum d’âne et l’anticorps primaire à une dilution appropriée (p. ex., 1/100 à 1 / 1 000). Incuber une nuit à 4 ° C sous agitation modérée.
Remarque : Consultez la Table des matières pour obtenir une liste des anticorps qui étiquette avec succès les différents types cellulaires de l’UNV avec ce protocole. La dilution optimale des anticorps primaires doit être déterminée empiriquement. Pour un étiquetage simultanée des antigènes différents, diluer tous les anticorps primaires dans la même solution. Tous les anticorps primaires doivent être déclenchés dans différentes espèces. Pour améliorer la pénétration de l’anticorps, des échantillons peuvent être incubés jusqu'à 72 h à 4 ° C. - Remove anticorps solution lavage sections et trois fois pendant 10 min dans du PBS sous agitation modérée à température ambiante. Retirez les PBS et ajouter 250 µL PBS solution contenant 5 % âne du sérum et l’anticorps secondaire au fluorescent étiquetés spécifiques appropriées. Incuber les sections à température ambiante pendant 1 h sous agitation modérée.
- Voir la Table des matières liste des anticorps secondaires utilisés avec ce protocole.
- Supprimer les sections de solution et lavage des anticorps trois fois pendant 10 min dans du PBS sous agitation modérée à température ambiante.
- PBS de supprimer et ajouter 250 µL d’un 4 ', 6-diamidino-2-phénylindole (DAPI) solution avec une concentration finale de 1 µg/mL. Incuber les sections à température ambiante pendant 10 minutes sous agitation modérée. Retirer les sections de solution et lavage DAPI 3 fois pendant 5 min avec du PBS.
- Monter la section du cerveau sur le collage des lames de microscope (p. ex., les lames de verre histo-bond). Retirez soigneusement les PBS excès autour de la section sur la diapositive. Ajouter une goutte de milieu de montage (voir Table des matières) sur le dessus de la section de cerveau et soigneusement le couvrir avec une lamelle de verre borosilicaté de 0,17 mm (n ° 1,5).
- Conserver les échantillons à 4 ° C, abri de la lumière jusqu'à l’exécutant de l’acquisition de l’image.
3. Imagerie confocale à haute résolution de la barrière hémato - encéphalique
microscope confocal- Perform acquisition d’images avec un laser à balayage adapté (voir Table des matières) avec lignes laser à 405, 488, 561 et 633 nm pour l’excitation des fluorophores dans les régions de bleues, vertes, orange et rouges du spectre lumineux. Pour l’acquisition de l’image, utilisez un 63 X objectif à huile avec une ouverture numérique de 1,4.
- Dans le cadre de l’acquisition, le menu du logiciel qui contrôle le microscope, définir l’image paramètres d’acquisition. Dans le menu déroulant de ‘ taille de l’Image ’, sélectionnez une valeur de 1024 x 1024 pixels. Modifier la taille du pixel à une valeur comprise entre 200 et 300 nm en ajustant la valeur de la ‘ Zoom ’ menu. ”
Remarque : pour l’analyse des structures intracellulaires, réduire la taille du pixel à 75 nm. Ce paramètre de taille d’image sensiblement augmente le temps d’acquisition et peut être réduit à 512 x 512 pixels pour diminuer le temps d’acquisition par un petit champ de vision d’imagerie. - Dans la ' vitesse d’Acquisition ' menu déroulant, sélectionnez une valeur de 400 Hz, c'est-à-dire 400 lignes par seconde. Dans le ' paramètres systèmes & #39; menu, sélectionnez le ' Echantillonnage ' du menu déroulant, puis changez la valeur en 12 bits.
- Dans le logiciel de microscope, au sein de la ' acquisition ' onglet, activer/désactiver l’option pour l’acquisition de trame séquentielle. Définir l’épaisseur de coupe optique à une valeur comprise entre 0,75 et 1 µm pour chaque canal en modifiant la valeur dans la ' trou d’épingle ' menu.
- Dans le panneau pour l’excitation fluorescente, activer le laser pour mieux exciter les fluorophores dans l’échantillon, par exemple 488 nm laser ligne pour un fluorophore émettant vert.
Remarque : Utilisez une visionneuse de spectres fluorophore (voir Table des matières) pour sélectionner le laser d’excitation suffisante. - Dans le panneau pour détection par fluorescence, déplacez le curseur pour sélectionner les longueurs d’onde qui se mesurera dans chaque canal, par exemple entre 510 et 550 nm pour un fluorophore émettant vert.
Remarque : Utilisez une visionneuse de spectres fluorophore (voir Table des matières) pour sélectionner les longueurs d’onde de détection adéquats. - Ajouter une goutte d’huile à immersion avec un indice de réfraction de 1,52 sur le dessus de la lamelle avec la section du cerveau en fonction de l’indice de réfraction du verre couvre-objet et objectif. Placer l’échantillon dans le microscope et allumez la lampe épifluorescence pour visualiser l’exemple à l’aide de jumelles microscope.
- En utilisant les boutons sur le statif, changer la roue de filtre pour sélectionner un filtre approprié pour la visualisation de noyaux colorés au DAPI. L’accent grossier permet de mettre le signal des noyaux colorés au DAPI focus sur.
- En utilisant les boutons sur le statif, changer les filtres de visualiser le signal à partir des marqueurs vasculaires (par exemple, CollagenIV ou CD31) et centrer le champ de vision sur un segment capillaire.
- Appuyez sur le bouton pour lancer le mode balayage direct. Lors de la numérisation, réglez l’intensité de laser et de gain pour chaque canal afin de maximiser la plage dynamique des images et éviter la saturation des pixels.
Remarque : Utiliser une table de correspondance qui étiquète saturés pixels pour évaluer visuellement la sursaturation. Pour éviter la sursaturation signal, ajustez les réglages à l’échantillon qui est censé avoir le plus haut signal fluorescent. - Définir la valeur de ligne avec une moyenne de 2.
Remarque : Lorsque vous utilisez des vitesses plus élevées acquisition, augmentez cette valeur pour réduire le bruit. - Établir commence et termine les sections pour l’exécution optiques z-piles qui s’étendent sur l’ensemble du volume du capillaire. Utilisez une taille d’étape de 0,45 µm.
Remarque : Si des images seront utilisés pour la quantification ultérieure, conserver les mêmes paramètres d’acquisition pour l’ensemble de données complet. - Pour la quantification, acquérir z-piles de 10 à 20 par section d’au moins trois différentes souris pour les comparaisons statistiques.
NOTE : Acquérir l’ensemble de données complet dans la même session de réduire la variabilité découlant des fluctuations d’intensité de blanchissement et laser échantillon.
4. Image Processing Reconstruction 3D de l’unité neurovasculaire
- (facultatif) pour augmenter la résolution axiale des z-piles acquises, effectuer la déconvolution de l’ensemble de données d’images à l’aide des logiciels appropriés (voir Table des matières ).
- Dans le logiciel de déconvolution, modifiez les paramètres pour effectuer " aveugle " déconvolution, (c'est-à-dire, en utilisant une fonction de point-diffusion adaptative).
- Dans les paramètres de logiciel de déconvolution, activer/désactiver l’option pour l’élimination de l’arrière-plan ; cette option va déterminer la plus petite valeur de l’intensité dans la pile d’image et il en soustraire les valeurs d’intensité de tous les pixels de l’image.
- Dans les paramètres de logiciel de déconvolution, désactiver les options de redimensionnement d’intensité et de redimensionnement à 16 bits de profondeur ; ces options conservera les valeurs d’intensité origine et la plage dynamique des images.
- Dans le menu du ' l’indice de réfraction ', définissez la valeur sur 1,52. Dans le ' définir la longueur d’onde ' menu, sélectionnez l’émission de valeurs pour chaque canal de l’image. Par exemple, sélectionnez 520 pour un fluorophore émettant vert.
- Ouvrir le jeu de données d’image à l’aide de logiciels adaptés pour la visualisation et l’analyse des ensembles de données 3-dimensionnelle (voir Table des matières). Dans le logiciel d’analyse de l’image, appuyez sur la " Surpass " bouton pour restituer le volume 3D de capillaires.
- Dans la presse de logiciel image analyse la " Ajouter Nouvelle surface " bouton pour segmenter la surface capillaire. Utilisez les flèches bas pour vous déplacer entre les étapes de l’Assistant de création de surface.
- Dans la ' Create ' onglet, régler le canal de la source à la chaîne avec le marqueur capillaire, par exemple CollagenIV et définir le détail de l’aire de la surface d’une valeur de 0,5 µm.
Remarque : Cette dernière option applique un filtre gaussien à l’image et détermine la finesse de la surface. - Dans les ' créer ' onglet, activer/désactiver l’option pour le seuil d’intensité absolue.
Remarque : Cette option crée une surface en générant un masque basé sur l’intensité absolue de la chaîne sélectionnée dans l’image. - Sur la prochaine étape de l’Assistant de création de surface, ajuster le seuil d’intensité pour la surface en déplaçant la fenêtre coulissante sur l’histogramme d’intensité jusqu'à ce que le masque rendu couvre le capillaire entier dans l’image. Déterminer la plage de valeurs pour ce paramètre empirique pour chaque ensemble de données.
- Lors de la dernière étape de l’Assistant de création de surface, cliquez sur le menu déroulant sous " filtre Type " et sélectionnez l’option ' nombre de Voxels '. Définit le nombre minimal de voxels sur une valeur comprise entre 1.0e5 à 2.0e5.
Remarque : Cette option exclura les surfaces avec un petit nombre de voxels, par exemple, de petits segments de capillaires au bord de l’armature. - Terminez l’Assistant de création de surface et d’enregistrer les paramètres de création en cliquant sur " n’oubliez pas de paramètres " dans l’onglet de la reconstruction du menu surface.
- Répéter la procédure pour l’ensemble de jeu de données d’imagerie en chargeant le jeu de paramètres utilisé pour la première image. Ajustez les paramètres pour l’intensité, le seuil et le nombre minimum de voxels pour chaque image à tenir compte des différences dans l’intensité de l’arrière-plan et morphologie capillaire, respectivement.
- Dans la ' Create ' onglet, régler le canal de la source à la chaîne avec le marqueur capillaire, par exemple CollagenIV et définir le détail de l’aire de la surface d’une valeur de 0,5 µm.
- Au segment des structures vésiculaires intracellulaires (par exemple, rempli d’immunoglobuline endosomes), tout d’abord créer un nouveau canal qui inclut uniquement un signal fluorescent de dans le capillaire.
Remarque : Ce processus définira une région d’intérêt en créant un masque à l’aide de la surface capillaire, créée à l’étape 4.3- Sélectionnez le ' edit ' menu dans la ' surface capillaire ' panneau. Sous ' masquer les propriétés ', cliquez " masque tous les ". Sélectionnez la chaîne qui contient le signal de vésicules intracellulaires comme les ' Source canal ' et bascule sur l’option " dupliquer le canal avant d’appliquer le masque ".
- Dans la ' masquer les paramètres ' colonne, sélectionnez " constante à l’intérieur/extérieur " et " voxels en dehors de la surface à la valeur : " et affectez-lui la valeur 0,00.
Remarque : Ce processus va créer un canal supplémentaire de l’image où tous les voxels en dehors du masque capillaire aura une valeur d’intensité de 0. Pour quantifier les événements en dehors de la capillaire (c'est-à-dire, dans le parenchyme du cerveau), choisissez " voxels à l’intérieur de la surface à la valeur : " et affectez-lui la valeur 0,00. - à segmenter les structures vésiculaires, appuyez sur le bouton pour démarrer la " ajouter de nouveaux Spots " Assistant. Utilisez les flèches bas pour vous déplacer entre les étapes de l’Assistant de création de surface.
- Dans l’onglet créer, utilisez le menu déroulant sous " Source canal " pour sélectionner le canal masqué intracellulaire, créé à l’étape 4.4.4.
- Dans l’onglet créer, dans le " Spot detection " section, d’une valeur comprise entre 0,5 et 1 µm, selon la taille moyenne des vésicules observée dans la valeur du diamètre estimé de XY l’expérience. Cette option détermine la plus petite taille qui sera détectée par l’algorithme de segmentation.
- Dans l’onglet créer, dans le " Spot detection " section, activer/désactiver l’option pour " fond soustraction ".
Remarque : Cette option va lisser l’image avec un filtre gaussien de 0,75 rayon spot (à partir de la valeur sélectionnée à l’étape 4.4.7) et puis en soustrayant l’intensité de l’image originale gaussienne filtrée par rayon spot 0.88. - Dans les ' créer ' l’onglet, appuyez sur le menu déroulant sous ' filtre Type ' et sélectionnez " qualité ". Notez que ceci va segmenter l’image par l’application des seuils basés sur l’intensité au centre des taches. Réglez le seuil d’intensité en déplaçant la fenêtre coulissante à travers la ' qualité ' histogramme jusqu'à ce que la majorité des vésicules identifiables est étiquetée.
Remarque : La plage de valeurs pour ce paramètre doit être déterminées empiriquement pour chaque ensemble de données. - Terminez l’Assistant de création de spot et enregistrer les paramètres de création en appuyant sur le bouton " n’oubliez pas de paramètres " dans l’onglet de la reconstruction du menu surface.
- Répéter la procédure pour l’ensemble de jeu de données d’imagerie en chargeant le jeu de paramètres utilisé pour la première image. Ajustez les paramètres pour la ' seuil d’intensité de qualité ' pour chaque image tenir compte des différences dans l’intensité de l’arrière-plan.
5. Quantification du Transport intracellulaire à la BHE
- quantifier le volume (en µm 3) et la zone (µm 2) du segment capillaire. Dans le logiciel d’analyse, accéder au panneau de statistiques de l’aire vasculaire créé à l’étape 4.3. Sélectionnez le ' Detailed ' onglet, puis utilisez le menu déroulant pour sélectionner " toutes les valeurs " pour trouver les valeurs de volume et de la zone.
- Quantifier le nombre de vésicules dans le segment des capillaire. Dans le logiciel d’analyse, accéder au panneau de statistiques des taches créé à l’étape 4.4. Sélectionnez le ' globale ' onglet pour trouver la valeur du nombre total de points dans l’image.
- Pour calculer le nombre de vésicules par volume capillaire, pour chaque image de normaliser le nombre de places par le volume calculé à l’étape 5.1. Multipliez cette valeur par 1 000 pour obtenir le nombre de vésicules par 1 000 µm 3 du volume capillaire.
- Pour quantifier l’intensité totale dans le capillaire crée une surface qui couvre ce qui suit une image toute les instructions décrites dans les étapes 4.3 avec les modifications suivantes. Sélectionnez la chaîne qui contient le signal pertinent (p. ex., mIgG) comme canal source et définissez la valeur du niveau de détail de la superficie à 5 µm. Pour créer une surface qui couvre l’ensemble de l’image, définissez la valeur du seuil plus faible intensité 0.
- Dans le logiciel d’analyse, accéder au panneau de statistiques de la surface créée à l’étape 5.4. Sélectionnez le " Detailed " onglet, puis utilisez le menu déroulant pour sélectionner " toutes les valeurs ". Enregistrez les valeurs pour ' intensité somme ' pour les canaux d’intérêt ; ce paramètre correspond à la somme des valeurs d’intensité différentes pour tous les pixels.
Remarque : Pour un signal intracellulaire, cela correspond au canal dupliqué avec voxels en dehors de la surface de la valeur 0.0. Pour un signal dans le parenchyme du cerveau, ce qui correspond au canal dupliqué avec voxels à l’intérieur de la surface de la valeur 0.0. - Pour calculer l’intensité de fluorescence par volume capillaire, pour chaque image de normaliser l’intensité par le volume calculé à l’étape 5.1. Multipliez cette valeur par 1 000 pour obtenir la valeur d’intensité de fluorescence par 1 000 µm 3 du volume capillaire.
Remarque : Pour les signaux fluorescents dans le parenchyme du cerveau, normaliser les valeurs par le volume de l’image totale moins le volume capillaire et multiplier par 1000 pour obtenir l’intensité totale par 1 000 µm 3 du parenchyme cerveau. - Répéter la procédure pour l’ensemble des données et utiliser un logiciel approprié pour effectuer une analyse statistique des données.
Résultats
Comme exemples représentatifs d’images obtient à partir du protocole décrit ici, sections de cerveau de souris ont été colorées avec les anticorps reconnaissant les différentes composantes de l’UNV dont la membrane basale, les astrocytes, les péricytes et les cellules endothéliales (voir Table des matières pour les anticorps spécifiques utilisés) (Figure 1 a, D et E). Avec cette résolution, il est possible de distinguer les différents processus astrocytaires et fin-pieds qui sont en contact direct avec les capillaires.
Pour mettre en évidence les qualités du présent protocole pour la détection des structures intracellulaires, sections de cerveau d’animaux injectés en périphérie de l’humain anti-Tau mAb86 anticorps16 ont été colorées avec un anticorps fluorescent anti-humain ( Figure 1B). mAb86 sait précisément les neurones cibles exprimant une forme pathologique de Tau16. En utilisant le protocole décrit dans les présentes, mAb86 a été détectée avec une résolution limitée par la diffraction au sein des structures vésiculaires individuels dans les neurones (Figure 1B-C). En outre, souris endogène IgG a été détectée dans des structures intracellulaires dans les cellules endothéliales mais pas dans les péricytes (D-E de laFigure 1et Figure 2).
L’acquisition de haute résolution confocal z-cheminées de la vascularisation du cerveau permet une segmentation en trois dimensions des capillaires et des vésicules intracellulaires à la BHE. La figure 2 montre un exemple du processus de rendu et de segmentation d’un capillaire avec CollagenIV et souris IgG positif intracellulaire vésicules. En quantifiant un ensemble complet de données images segmentées, par exemple en mesurant le nombre de vésicules par volume capillaire, il est possible d’étudier les changements dans les processus de transport intracellulaire dans des conditions différentes. Figure 3 B-C montre les différences de mIgG vésicule nombre et fluorescence intensité correspondant à mIgG dans le parenchyme du cerveau, respectivement, à l’appauvrissement dans le modèle murin pdgf-bret/ret pericyte comme indiqué précédemment 17. la même approche a été récemment utilisée pour analyser les changements dans le transport intracellulaire à la BHE entre cerveau différentes régions18.

Figure 1 : Etiquetage des plusieurs types de cellules et des structures subcellulaires de la neurovasculaire unité Images représentatives de l’unité neurovasculaire (A et D) et de vésicules intracellulaires contenus dans les neurones (B) ou de cellules endothéliales (E) obtenus avec ce protocole. L’image projetée en intensité maximale à A (haut) montre la répartition des astrocytes GFAP-positive (rouges) qui entourent les capillaires labellisés par le CollagenIV (verts). Flèches pointent vers des processus astrocytaires individuels. Echelle = 20 µm. Avec cette résolution, les processus individuels astrocyte et fin-pieds sont clairement visibles, comme illustré dans l’image zoomée de la région en boîte (en bas). Pointes de flèche pointent vers fin-pieds astrocytaires. Echelle = 10 µm. L’image b montre l’accumulation d’un anticorps injectés en périphérie, mAb86 (vert), au sein d’un neurone hippocampe. Pointes de flèche pointent vers les vésicules mAb86 positifs individuels. Echelle = 10 µm. Le graphique en C indique l’intensité de profil de ligne d’une vésicule simple. La vésicule a été estimée de la pleine largeur à mi-hauteur d’un ajustement gaussien (ligne noire épaisse) de la courbe d’intensité (ligne verte et des cercles). Les images d montrent une reconstruction tridimensionnelle d’une cellule endothéliale (verte) entourée d’un pericyte (rouge) au sein de la couche basale (CollagenIV, gris). Les panneaux inférieurs montrent les canaux individuels de fluorescence. Echelle = 10 µm. Les images en E montrent la localisation du mIgG (rouge) dans les vésicules intracellulaires dans les cellules endothéliales (panneau, CD31 en vert à gauche) mais pas en péricytes (panneau de droite, CD13 en vert). Dans toutes les images, DAPI teinté noyaux sont indiqués en bleu. Echelle = 5 µm. panneaux D et E ont été modifiés de référence17. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 2 : Rendu tridimensionnel des capillaires et des vésicules intracellulaires à la barrière hémato - encéphalique. Avec le protocole décrit, haute résolution images confocales de capillaires CollagenIV séropositifs (verts) et de la souris IgG vésicules intracellulaires (rouge) ont été acquises (A). Le panneau de gauche montre une coupe optique unique avec les coupes. Les flèches pointent vers les vésicules mIgG positifs individuels dans les cellules endothéliales du cerveau. Le groupe d’experts sur la reconstruction de droite montre la 3D de la z-pile complète à l’aide logiciel de traitement d’image (voir la Table des matières). Le volume capillaire (B) et des vésicules (C) ont été rendus en trois dimensions et quantifiées à l’aide de logiciels de traitement d’image (voir la Table des matières). Dans toutes les images, DAPI teinté noyaux sont indiqués en bleu. Barreaux de l’échelle = 5 µm. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
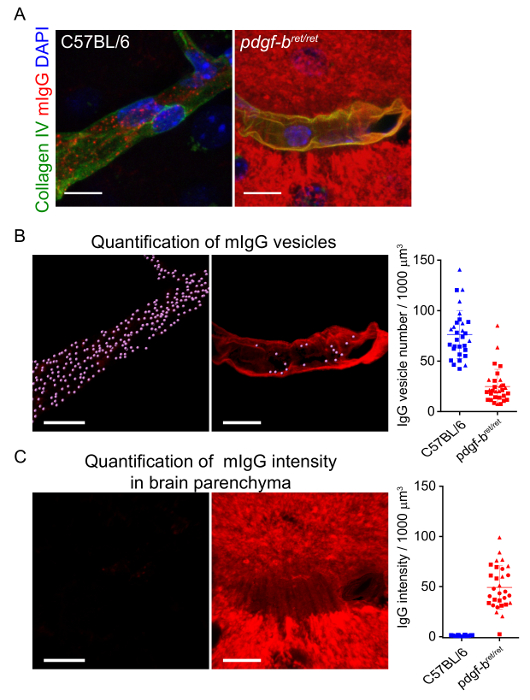
Figure 3 : Quantification de la localisation intracellulaire mIgG à la BHE. Les images représentatives démontrant reconstructions tridimensionnelles (A) des capillaires (étiquetés avec collagenIV, vert) et la distribution de mIgG (rouge) chez les souris C57BL/6 et pdgf-bret/ret pericyte appauvri souris précédemment décrits dans19. Barreaux de l’échelle = 10 µm. Les images b montrent la segmentation des vésicules intracellulaires dans le masque de CollagenIV. Thgraphique de e à B présente la quantification et la comparaison du nombre de vésicules mIgG par volume capillaire. Chaque point représente les mesures des segments individuels de capillaires. La ligne continue indique la moyenne et les barres d’erreur représentent l’écart type des données. Les images du spectacle C le mIgG signal de fluorescence à l’extérieur du masque de CollagenIV. De même, le graphique en C montre la quantification et la comparaison de l’intensité de fluorescence mIgG dans le parenchyme du cerveau entre les souris C57BL/6 et le pdgf-bret/ret pericyte appauvri souris. Unités d’intensité de fluorescence ont été normalisées par l’intensité de parenchyme mIgG moyenne mesurée dans toutes les souris C57BL/6. Ce chiffre a été modifié par référence17. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Discussion
Le protocole décrit ci-dessus décrit la préparation du cerveau sections flottant, immunofluorescence, acquisition d’images et paramètres d’analyse pour la microscopie à haute résolution de la BHE. Cette méthode a été récemment utilisée pour étudier la localisation des anticorps livraison plates-formes3, le transport des IgG endogène à travers le BBB17et l’hétérogénéité de la BHE à perte de pericyte18. Différentes étapes dans le protocole peuvent être modifiés pour s’adapter à l’objectif spécifique de l’expérience. Tout d’abord, l’utilisation d’épaisseur importante (100 µm) facilite leur manipulation pendant les procédures de montage et immunostaining. Il permet également pour la reconstruction 3D du réseau capillaire, l’unité neurovasculaire et pour la génération de capillaire et les coupes NVU. Cependant, pénétration des anticorps au sein des sections de tissu peut varier et la coloration de certains anticorps peut être limitée à la couche superficielle du tissu proche de la lamelle couvre-objet. Le protocole peut être modifié en augmentant la concentration de détergent pendant l’étape de la perméabilisation et/ou la longueur de l’étape de la perméabilisation pour améliorer la pénétration des anticorps dans le tissu. En second lieu, la qualité d’image peut être compromise lors d’une tentative d’acquisition d’images plus profondément dans les tissus (généralement 20 à 30 µm sous la surface) en raison de la diffusion de la lumière ainsi que les aberrations optiques de l’incompatibilité de l’indice de réfraction. Pour contourner ce problème, nouvelles méthodes de compensation des tissus et active en anticorps pénétration20 peuvent être associés à ce protocole aux volumes plus grandes image du tissu. En troisième lieu, déconvolution s’effectue après l’acquisition d’images pour améliorer la résolution axiale de l’image. Le choix de l’algorithme de déconvolution aveugle utilisé dans le présent protocole est fondé sur (i) sa facilité d’utilisation, comme aucun calcul préalable de la fonction de l’écart de points n’est requis, (ii) sa robustesse pour améliorer l’image qualité21et (iii) l’absence d’objets mIgG structures intracellulaires après la mise en œuvre. Selon les structures intracellulaires visualisés dans l’échantillon, autres algorithmes de déconvolution peuvent entraîner une meilleure qualité d’image. Les références suivantes21,22 fournissent une large discussion sur les avantages et les limitations des algorithmes supplémentaires pour la déconvolution d’images. Enfin, l’utilisation d’un progiciel d’analyse image autorisé la segmentation des capillaires et des structures intracellulaires en trois dimensions. Clairement, analyse d’image n’est pas limitée au logiciel décrit dans le protocole et les paquets, par exemple celles mentionnées dans la référence23, peut être utilisé pour des images de segment. L’adéquation des différents logiciels d’analyse des structures intracellulaires à travers la BHE devrait être vérifiée empiriquement par l’évaluation de l’exactitude de la segmentation de l’image.
Étant donné que cette méthode est basée sur des échantillons de fixes, il ne fournit pas des informations directes sur la dynamique de la transcytose à travers la BHE. Toutefois, il peut être associé des expériences de temps24, par exemple par voie intraveineuse par injection de la molécule d’intérêt et en mesurant son accumulation dans les BECs à des moments différents après l’injection, pour reconstruire la cinétique de transport intracellulaire. L’avantage de cette approche est qu’elle permet l’analyse des régions du cerveau profond, comme illustré dans le18, qui sont actuellement inaccessibles aux approches d’imagerie live intravitale. Une étape critique pendant le protocole est le suivi attentif de la fixation du tissu. Fixation avec 4 % PFA réduit considérablement l’immunogénicité des organites intracellulaires et des immunoglobulines endogènes ou périphériquement gérés17 (Figure 1 b). Une limitation du présent protocole est son exigence d’anticorps de haute qualité(p. ex., une coloration non spécifique faible, faible réactivité croisée) appropriés pour l’immunofluorescence. Sous réserve que ces réactifs sont disponibles, la méthode peut être appliquée afin d’étudier la localisation intracellulaire d’une protéine d’intérêt. Par exemple, le protocole servait à identifier les lysosomes dans le cerveau des cellules endothéliales17. Il devrait également noter que, étant donné que ce protocole est basé sur la microscopie confocale, la résolution latérale est limitée par la diffraction et ne peut pas résoudre des structures inférieures à environ 175 à 250 nm (Figure 2).
Des études antérieures ont effectué une analyse détaillée de la composition cellulaire de l’unité neurovasculaire, à l’aide de la microscopie confocale25,26. Cependant, enquête sur le transport intracellulaire à la BHE repose principalement sur l’utilisation de la microscopie électronique de transmission19,27,28. Bien que cette méthode offre la plus haute résolution latérale des structures intracellulaires, microscopie électronique reste une technique difficile avec un faible débit. En outre, le nombre de cibles moléculaires différentes qui peuvent être visualisées par EM est très limité. Ce protocole offre une alternative accessible afin d’étudier le transport intracellulaire à la BHE. La procédure complète, de la collection du cerveau à l’analyse de l’image, peut être effectuée en 5 à 6 jours. Si les anticorps appropriés sont disponibles, immunofluorescence permet la détection simultanée de plusieurs molécules/types des cellules au sein d’un même échantillon. En outre, ce protocole pourrait être associé à des techniques de microscopie de Super-résolution pour surmonter les limitations en résolution spatiale29. Dans l’ensemble, le protocole décrit ci-dessus permet la quantification des changements dans la localisation intracellulaire des protéines d’intérêt au sein de l’unité neurovasculaire. Son application des différentes perturbations génétiques ou pharmacologiques permettra d’enquêter sur les fonctions de structure et de transport intracellulaires du BBB en vivo.
Déclarations de divulgation
Les auteurs sont sous un emploi rémunéré par Roche.
Remerciements
R.V. travail a été soutenu par une bourse postdoctorale de Roche (2014-2017).
matériels
| Name | Company | Catalog Number | Comments |
| Rat monoclonal antibody clone ER-MP12 against CD31/PECAM | Novus Biologicals | MCA2388 | Labels Brain Endothelial Cells Use at dilution 1:100 detect with donkey polyclonal anti-rat IgG (H+L) coupled with suitable AlexaFluor dye, used at dilution 1:400 |
| Rat monoclonal antibody against Podocalyxin | R&D Systems | MAB1556 | Labels Lumen of capillaries Use at dilution 1:100 detect with donkey polyclonal anti-rat IgG (H+L) coupled with suitable AlexaFluor dye, used at dilution 1:400 |
| Rabbit polyclonal antibody against CollagenIV | Biotrend | BT21-5014-70 | Labels Basement membrane of capillaries Use at dilution 1:100 detect with donkey polyclonal anti-rabbit IgG (H+L) coupled with suitable AlexaFluor dye, used at dilution 1:400 |
| Goat polyclonal antibody against CD13 | R&D Systems | AF2335 | Labels pericytes Use at dilution 1:100 detect with donkey polyclonal anti-goat IgG (H+L) coupled with suitable AlexaFluor dye, used at dilution 1:400 |
| Mouse monoclonal antibody clone G-A-5 against GFAP coupled to Cy3 | Abcam | ab49874 | Labels Astrocytes Use at dilution 1:100 |
| Rabbit polyclonal antibody against Iba-1 | Wako | 019-19741 | Labels Microglia Use at dilution 1:100 detect with donkey polyclonal anti-rat IgG (H+L) coupled with suitable AlexaFluor dye, used at dilution 1:400 |
| Rat monoclonal antibody ABL93 gainst LAMP2 | Fitzgerald | 10R-CD107BBMSP | Labels Lysosomes Use at dilution 1:100 detect with donkey polyclonal anti-rat IgG (H+L) coupled with suitable AlexaFluor dye, used at dilution 1:400 |
| donkey polyclonal antibody against mouse IgG (H+L) AlexaFluor594 | LifeTechnologies | A21203 | Use at dilution 1:400 |
| donkey polyclonal antibody against mouse IgG (H+L) AlexaFluor488 | LifeTechnologies | A21202 | Use at dilution 1:400 |
| goat polyclonal antibody against mouse IgG (H+L) AlexaFluor555 | LifeTechnologies | A21422 | Use at dilution 1:400 |
| Filter paper | Sigma-Aldrich | WHA10334347 | Whatman prepleated qualitative filter paper Grade 0858 1/2, grained |
| Cyanoacrylate glue | Roth Carl | 258.1 | Roti Coll 1 |
| Fluorescent Mounting medium | Dako | S3023 | Dako fluorescent mounting medium |
| Adult mice | The Jackson Laboratory | 000664 | C57BL/6J male or female mice between 9 and 19 months of age |
| Vibratome | Leica Biosystems | 14047235612 | Leica VT100S vibrating blade microtome |
| Laser Scanning Confocal microscope | Leica Microsystems | NA | Leica TCS SP8 X with HyD detectors and White light laser |
| Image processing software | Leica Microsystems | NA | Leica Application Suite AF version 3.1.0 build 8587 |
| Image analysis software | Bitplane scientific software | NA | Imaris version 7.6.5 build 31770 for x64 |
| Fluorescence SpectraViewer | ThermoFisher Scientific | NA | https://www.thermofisher.com/ch/en/home/life-science /cell-analysis/labeling-chemistry/fluorescence-spectraviewer.html |
Références
- Blanchette, M., Daneman, R. Formation and maintenance of the BBB. Mech Dev. 138, Pt 1 8-16 (2015).
- Chow, B. W., Gu, C. The Molecular Constituents of the Blood-Brain Barrier. Trends Neurosci. 38 (10), 598-608 (2015).
- Niewoehner, J., et al. Increased Brain Penetration and Potency of a Therapeutic Antibody Using a Monovalent Molecular Shuttle. Neuron. 81 (1), 49-60 (2014).
- Yu, Y. J., et al. Therapeutic bispecific antibodies cross the blood-brain barrier in nonhuman primates. Sci Transl Med. 6 (261), 154(2014).
- Preston, J. E., Joan Abbott, N., Begley, D. J. Transcytosis of macromolecules at the blood-brain barrier. Adv Pharmacol. 71, 147-163 (2014).
- De Bock, M., et al. Into rather unexplored terrain-transcellular transport across the blood-brain barrier. Glia. 64 (7), 1097-1123 (2016).
- Bien-Ly, N., et al. Transferrin receptor (TfR) trafficking determines brain uptake of TfR antibody affinity variants. J Exp Med. 211 (2), 233-244 (2014).
- Sade, H., et al. A human blood-brain barrier transcytosis assay reveals antibody transcytosis influenced by pH-dependent receptor binding. PLoS One. 9 (4), 96340(2014).
- Tian, X., et al. LRP-1-mediated intracellular antibody delivery to the Central Nervous System. Sci Rep. 5, 11990(2015).
- Hsu, J., Rappaport, J., Muro, S. Specific binding, uptake, and transport of ICAM-1-targeted nanocarriers across endothelial and subendothelial cell components of the blood-brain barrier. Pharm Res. 31 (7), 1855-1866 (2014).
- Siupka, P., et al. Bidirectional apical-basal traffic of the cation-independent mannose-6-phosphate receptor in brain endothelial cells. J Cereb Blood Flow Metab. , (2017).
- Zlokovic, B. V., et al. A saturable mechanism for transport of immunoglobulin G across the blood-brain barrier of the guinea pig. Exp Neurol. 107 (3), 263-270 (1990).
- Deane, R., et al. IgG-assisted age-dependent clearance of Alzheimer's amyloid beta peptide by the blood-brain barrier neonatal Fc receptor. J Neurosci. 25 (50), 11495-11503 (2005).
- Cabezon, I., et al. Serial block-face scanning electron microscopy applied to study the trafficking of 8D3-coated gold nanoparticles at the blood-brain barrier. Histochem Cell Biol. , (2017).
- Gage, G. J., Kipke, D. R., Shain, W. Whole animal perfusion fixation for rodents. J Vis Exp. (65), (2012).
- Collin, L., et al. Neuronal uptake of tau/pS422 antibody and reduced progression of tau pathology in a mouse model of Alzheimer's disease. Brain. 137 (10), 2834-2846 (2014).
- Villasenor, R., et al. Trafficking of Endogenous Immunoglobulins by Endothelial Cells at the Blood-Brain Barrier. Sci Rep. 6, 25658(2016).
- Villasenor, R., et al. Region-specific permeability of the blood-brain barrier upon pericyte loss. J Cereb Blood Flow Metab. , (2017).
- Armulik, A., et al. Pericytes regulate the blood-brain barrier. Nature. 468 (7323), 557-561 (2010).
- Richardson, D. S., Lichtman, J. W. Clarifying Tissue Clearing. Cell. 162 (2), 246-257 (2015).
- Sibarita, J. B. Deconvolution Microscopy. 95, 201-243 (2005).
- Shaw, P. J. Comparison of Widefield/Deconvolution and Confocal Microscopy for Three-Dimensional Imaging. , 453-467 (2006).
- Chenouard, N., et al. Objective comparison of particle tracking methods. Nat Methods. 11 (3), 281-289 (2014).
- Foret, L., et al. A general theoretical framework to infer endosomal network dynamics from quantitative image analysis. Curr Biol. 22 (15), 1381-1390 (2012).
- Daneman, R., Zhou, L., Kebede, A. A., Barres, B. A. Pericytes are required for blood-brain barrier integrity during embryogenesis. Nature. 468 (7323), 562-566 (2010).
- Bell, R. D., et al. Pericytes control key neurovascular functions and neuronal phenotype in the adult brain and during brain aging. Neuron. 68 (3), 409-427 (2010).
- Ben-Zvi, A., et al. Mfsd2a is critical for the formation and function of the blood-brain barrier. Nature. 509 (7501), 507-511 (2014).
- Stewart, P. A. Endothelial vesicles in the blood-brain barrier: are they related to permeability. Cell Mol Neurobiol. 20 (2), 149-163 (2000).
- Follain, G., Mercier, L., Osmani, N., Harlepp, S., Goetz, J. G. Seeing is believing - multi-scale spatio-temporal imaging towards in vivo cell biology. J Cell Sci. 130 (1), 23-38 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon