Method Article
Microarray Anticorps chimiquement bloqué pour multiplexée à haut débit de profilage de glycosylation des protéines spécifiques dans les échantillons complexes
Dans cet article
Résumé
Dans cette étude, nous décrivons un protocole amélioré pour une puce anticorps à haut débit multiplexées avec la méthode de détection lectine qui peut être utilisé dans le profilage de glycosylation des protéines spécifiques. Ce protocole propose de nouveaux réactifs fiables et réduit considérablement le temps, le coût et les besoins en équipement de laboratoire par rapport à la procédure précédente.
Résumé
In this study, we describe an effective protocol for use in a multiplexed high-throughput antibody microarray with glycan binding protein detection that allows for the glycosylation profiling of specific proteins. Glycosylation of proteins is the most prevalent post-translational modification found on proteins, and leads diversified modifications of the physical, chemical, and biological properties of proteins. Because the glycosylation machinery is particularly susceptible to disease progression and malignant transformation, aberrant glycosylation has been recognized as early detection biomarkers for cancer and other diseases. However, current methods to study protein glycosylation typically are too complicated or expensive for use in most normal laboratory or clinical settings and a more practical method to study protein glycosylation is needed. The new protocol described in this study makes use of a chemically blocked antibody microarray with glycan-binding protein (GBP) detection and significantly reduces the time, cost, and lab equipment requirements needed to study protein glycosylation. In this method, multiple immobilized glycoprotein-specific antibodies are printed directly onto the microarray slides and the N-glycans on the antibodies are blocked. The blocked, immobilized glycoprotein-specific antibodies are able to capture and isolate glycoproteins from a complex sample that is applied directly onto the microarray slides. Glycan detection then can be performed by the application of biotinylated lectins and other GBPs to the microarray slide, while binding levels can be determined using Dylight 549-Streptavidin. Through the use of an antibody panel and probing with multiple biotinylated lectins, this method allows for an effective glycosylation profile of the different proteins found in a given human or animal sample to be developed.
Introduction
Glycosylation of protein, which is the most ubiquitous post-translational modification on proteins, modifies the physical, chemical, and biological properties of a protein, and plays a fundamental role in various biological processes1-6. Because the glycosylation machinery is particularly susceptible to disease progression and malignant transformation, aberrant glycosylation has been recognized as early detection biomarkers for cancer and other diseases 7-12. In fact, most current cancer biomarkers, such as the L3 fraction of α-1 fetoprotein (AFP) for hepatocellular carcinoma 13-15, and CA199 for pancreatic cancer 16, 17 are all aberrant glycan moieties on glycoproteins. However, methods to study protein glycosylation have been complicated, and not suitable for routine laboratory and clinical settings. Chen et al. has recently invented a chemically blocked antibody microarray with a glycan-binding protein (GBP) detection method for high-throughput and multiplexed profile glycosylation of native glycoproteins in a complex sample 18. In this affinity based microarray method, multiple immobilized glycoprotein-specific antibodies capture and isolate glycoproteins from the complex mixture directly on the microarray slide, and the glycans on each individual captured protein are measured by GBPs. Because all normal antibodies contain N-glycans which could be recognized by most GBPs, the critical step of this method is to chemically block the glycans on the antibodies from binding to GBP. In the procedure, the cis-diol groups of the glycans on the antibodies were first oxidized to aldehyde groups by using NaIO4 in sodium acetate buffer avoiding light. The aldehyde groups were then conjugated to the hydrazide group of a cross-linker, 4-(4-N-MaleimidoPhenyl)butyric acid Hydrazide HCl (MPBH), followed by the conjugation of a dipeptide, Cys-Gly, to the maleimide group of the MPBH. Thus, the cis-diol groups on glycans of antibodies were converted into bulky none hydroxyl groups, which hindered the lectins and other GBPs bindings to the capture antibodies. This blocking procedure makes the GBPs and lectins bind only to the glycans of captured proteins. After this chemically blocking, serum samples were incubated with the antibody microarray, followed by the glycans detection by using different biotinylated lectins and GBPs, and visualized with Cy3-streptavidin. The parallel use of an antibody panel and multiple lectin probing provides discrete glycosylation profiles of multiple proteins in a given sample 18-20. This method has been used successfully in multiple different labs 1, 7, 13, 19-31. However, stability of MPBH and Cys-Gly, complicated and extended procedure in this method affect the reproducibility, effectiveness and efficiency of the method. In this new protocol, we replaced both MPBH and Cys-Gly with one much more stable reagent glutamic acid hydrazide (Glu-hydrazide), which significantly improved the reproducibility of the method, simplified and shorten the whole procedure so that the it can be completed within one working day. In this new protocol, we describe the detailed procedure of the protocol which can be readily adopted by normal labs for routine protein glycosylation study and techniques which are necessary to obtain reproducible and repeatable results.
Protocole
1. Imprimer une Microarray Anticorps pour le test
- Diluer tous les anticorps à 0,5 mg / ml dans une solution saline de tampon phosphate, pH 7,2 (PBS).
- Aliquoter 40 ul de chaque anticorps dans la plaque de source de 384 puits.
- Charger la plaque de source de 384 puits sur le microarrayer sciFLEXARRAYER Scienion.
- Chargez 20 diapositives microarray PATH sur le microarrayer en tant que cible.
- Réglez le microarrayer d'imprimer 48 sous-réseaux identiques, dans lequel 27 anticorps et les protéines de contrôle sont repérés en triple exemplaire dans un modèle 9x9 (figure 1E, 1F).
- Démarrez le microarrayer d'imprimer les diapositives biopuces d'anticorps.
- Recueillir les diapositives biopuces d'anticorps, et de les stocker dans la cassette diapositives avec déshydratant. Passez l'aspirateur sceller la cassette dans un sac en plastique à l'aide de vide scellant (Foodsaver).
- Rangez les lames microarray scellées à 4 ° C au réfrigérateur.
2. Chimiquement Bloquer le Microarray Anticorps visant à prévenir GBPLier aux anticorps de capture
Le test commence une fois que les puces à ADN microarray diapositives sont chimiquement bloqué et dure environ 8 heures. Une fois démarré l'essai microarray doit être complété (étapes 2 à 8).
- Prenez la puce se glisse hors du réfrigérateur, et de les amener à température ambiante pendant 30 minutes.
- Retirer la lame de la boîte de rangement et brièvement les rincer à pH tampon phosphate salin à 7,2 avec 0,1% de Tween 20 (PBST0.1) une fois dans une diapositive lavabo, puis dans 15 mM, pH tampon acétate de sodium 5,0 avec 0,1% (Tween CBT0 0.1) d'une manière séquentielle. Incuber les lames dans CBT0.1 pendant 10 minutes dans le bassin de lavage diapositive.
- Préparer une nouvelle solution 150 mM NaIO 4 à 15 mM de tampon acétate de sodium pH 5,0 (CB), et conservez-le dans dans une diapositive lavabo dans un réfrigérateur tout en évitant la lumière avant de l'utiliser.
- Retirer la lame de la CB, et le mettre dans le bassin contenant frais NaIO 4 avec le côté d'anticorpsvers le haut. Couvrez le bassin avec une feuille d'aluminium pour éviter la lumière, et incuber le bassin diapositive pendant 2 heures avec agitation douce à 4 ° C dans un réfrigérateur.
- Préparer 300 ml de 10 mM d'acide glutamique hydrazide (le bloqueur) dans CB.
- Retirer la lame du bassin, et brièvement le rincer à CB 3 fois pendant 5 minutes à chaque fois dans le bassin de lavage diapositive.
- Incuber les lames dans le bloqueur dans un bassin de lavage pour 2 heures à température ambiante sous agitation douce.
- Retirer les lames du bassin, et les laver avec PBST0.1 pendant 3 minutes.
3. Bloc non-spécifiques de liaisons pour la micropuce de la sérumalbumine bovine (BSA)
- Préparer 300 ml de BSA 1% dans du tampon phosphate pH 7,2 avec une solution saline à 0,5% de Tween (PBST0.5) dans un bassin de lavage coulisseau, et incuber le coulisseau microréseau dans le bassin pendant 1 heure à température ambiante avec agitant doucement.
- Rincer les lames dans PBST0.1 trois fois pendant 3 minutes à chaque fois.
- Mettre la diapositivesur une grille coulissante, et tournent à 1200 xg sur une centrifugation pendant 2 minutes pour sécher la lame puces à ADN.
4. Grille de cire Mentions légales sur la lame de puces à ADN pour séparer chaque sous-réseau
- Préchauffer le dispositif d'impression de cire à 70 ° C pendant 5 minutes.
- Charger le coulisseau microréseau bloquée dans le dispositif d'impression de la cire avec le côté faisant face à l'anticorps de la cire. Tirez doucement sur la poignée à la cire empreinte sur la lame régulièrement.
5. Appliquer échantillons de sérum sur la lame de puces à ADN
- Lors de l'étape 2.4, la préparation des échantillons de sérum pour dosage soit profilage glyco dans un échantillon (5.1.1), ou simple glyco epiptope mesures parmi plusieurs échantillons (5.1.2).
- Dans une expérience pour les profilages glycanniques de plusieurs glycoprotéines sériques dans un échantillon de sérum en utilisant plusieurs Gbps (voir 1 expérience de l'échantillon), un des échantillons de sérum sera appliquée sur tous les sous-réseaux. Dans ce cas, 40 pl amplement le sérum est dilué dans pl 360 de PBS contenant 0,1%Tween-20, 0,1% Brij 35, 100 ug / ml de IgG de souris, 100 ug / ml de IgG de rat, 100 ug / ml de IgG de lapin, 100 ug / ml de IgG de chèvre et de 100 pg / ml d'âne IgG. Ce volume est suffisant pour l'application de 6 pi de solution de sérum dilué sur chaque sous-réseau.
- Dans une expérience pour la mesure d'une glycane sur plusieurs protéines sériques entre plusieurs échantillons de sérum en utilisant un détections GBP (voir 2 l'expérience de l'échantillon). Dans ce cas, 1 pl amplement sérum est dilué dans 9 ul de PBS contenant 0,1% de Tween-20, 0,1% Brij 35, 100 ug / ml de IgG de souris, 100 ug / ml de IgG de rat, 100 ug / ml de IgG de lapin , 100 ug / ml de IgG de chèvre et 100 pg / ml d'âne IgG. Ce volume est suffisant pour l'application de 6 pi de solution de sérum dilué sur chaque sous-réseau.
- Après empreinte de cire à l'étape 4, appliquez soigneusement 6 pi d'échantillon dilué ou des échantillons de contrôle (PBST0.1) à chaque sous-tableau de la diapositive. Incuber la lame dans une cassette humidifié avec du papier absorbant humide à la température ambiantependant 1 heure.
- Rincer la lame avec PBST0.1 trois fois pendant 3 minutes à chaque fois.
- Sécher la lame en le tournant à 1200 xg pendant 2 minutes.
6. Appliquer biotinylé GBP (Anticorps anti-lectine ou glycane) sur la glissière
- Lors de l'étape 2.4, préparer des lectines biotinylées 10μg/ml ou Gbps en PBST0.1.
- Dans l'expérience de profilage glycane que la sonde un échantillon avec des lectines multiples (1 expérience de l'échantillon), préparer 350 pi de lectine biotinylée qui est suffisant pour tous les sous-réseaux.
- En simple glycane épitope / biomarqueur de dépistage dans de multiples échantillons en utilisant des lectines multiples, préparer 10 ul de chaque lectine biotinylée qui est suffisant pour un sous-réseau.
- Appliquer 6 pl de la lectine biotinylée dilué (s) à chaque sous-tableau de la diapositive, et incuber dans la boîte de diapositive humidifié avec du papier absorbant humide à température ambiante pendant 1 heure.
- Rincer les lames avec PBST0.1 trois fois pendant 3 minutes de chaque timoi.
- Sécher la lame en le tournant à 1200 xg dans une centrifugeuse pour 2 minutes.
7. Appliquer de la teinture neutravidine Étiqueté pour la détection par fluorescence
- Préparer 350 pi de Dylight 549 neutravidine étiqueté qui est suffisant pour tous les sous-réseaux.
- Appliquer 6 pi de Dylight 549 neutravidine marqué sur chaque sous-réseau, et incuber la lame dans la cassette diapositive humide à température ambiante pendant 1 heure.
- Rincer la lame avec PBST0.1 trois fois pendant 3 minutes à chaque fois.
- Sécher la lame en faisant tourner la il à 1200 xg dans une centrifugeuse pour 2 minutes.
8. Obtenir image de la diapositive Microarray par balayage de la diapositive
- Numérisation de la diapositive en utilisant un scanner de fluorescence à puce une résolution de 10 um. Les paramètres du laser et PMT devrait être aussi fort que possible, mais pas de point de saturation est observée.
9. Extraction et analyse des données
- Ouvrez l'image dans ArrayPro 3.2.
- Mettre en place le modèle réseau selon la carte réseau qui montre les taches d'anticorps endroits. Alignez soigneusement chaque cercle gabarit sur l'endroit correspondant dans l'image.
- Extrait de l'intensité de chaque spot dans un fichier Excel pour une analyse plus approfondie.
10. Les résultats représentatifs
1 Exemple d'expérience
La glycosylation de profilage plusieurs glycoprotéines sériques dans l'échantillon du patient carcinome hépatocellulaire sérum en utilisant des anticorps microarray chimiquement bloqué avec détection des lectines multiples.
Le but de cette expérience est d'explorer le profil de glycosylation des glycoprotéines individuelle de 20 dans le carcinome hépatocellulaire (HCC) de l'échantillon de sérum du patient à l'aide de puces à ADN d'anticorps chimiquement bloqué avec détection de lectine. Une puce à ADN d'anticorps, qui contient 48 sous-réseaux identiques qui comprennent 26 anticorps et la biotine-BSA, a été conçu et fabriqué comme décrit dans Step 1. Ces anticorps étaient 26 contre 20 glycoprotéines sériques que identifiés comme prometteurs valeur diagnostic précoce pour les patients HCC en utilisant la lectine immunoprécipitation combinée avec des l'identification des protéines par spectrométrie de masse 12, 32 comme indiqué dans le tableau 1. Le motif et l'agencement des points imprimés en triple anticorps dans un sous-matrice représentant sont représentés à la figure 1E et 1F, respectivement. Deux lames microarray identiques, on n'était pas bloqué chimiquement (figure 1A), tandis que l'autre a été (figure 1B), ont été utilisés pour réaliser l'expérience de glycosylation même profilage afin de démontrer l'importance de la procédure de blocage chimiquement à l'analyse. Pour la diapositive chimiquement bloqué (figure 1B), l'expérience a commencé à l'étape 2, car il n'en diapositive chimiquement bloqué (figure 1A), l'expérience a débuté à l'étape 3. L'expérience a été réalisée par following toutes les étapes décrites dans le protocole, sauf pour l'étape 5.1.2 et 6.1.2. Dans l'étape 5.2, un échantillon de contrôle PBST0.1 a été appliqué sur sous-réseaux dans la colonne 1 et 3, et une mise en commun HCC échantillon de sérum a été appliqué sur sous-réseaux dans la colonne 2 et 4, respectivement (comme le montre la figure 1G). Cette comparaison est de montrer l'efficacité, l'efficience de la procédure, ainsi que l'affinité de liaison d'antigène des anticorps après chimiquement blocage. 22 lectines biotinylées (comme indiqué dans le tableau 1) que spécifique à différents glycanes 18, 20 ont été appliquées sur chaque sous-réseau comme le montre la figure 1G pour le profilage de glycosylation. Images des chimiquement bloqués (figure 1B) et non-chimiquement bloqué (figure 1A) puces après le dosage de profilage glycosylation en suivant le protocole. Comme indiqué dans les sous-réseaux dans la colonne 1 et 3 dans les pays non-chimiquement puces bloquées (figure 1A et figure 1C), sur lequelseulement PBST0.1 a été appliqué, la plupart des lectines lié à anticorps de capture, et a montré de fond très élevé que ces sous-réseaux comparables à la colonne 2 et 4, sur lequel l'échantillon de sérum a été appliquée. Il est impossible d'obtenir des informations de profil glycane à partir de cette diapositive puces à ADN. Au contraire, lorsque la même expérience a été faite sur une diapositive microréseau d'anticorps chimiquement bloqué, les sous-réseaux dans la colonne 1 et 3, sur lequel a été appliqué seulement PBST0.1, la plupart des lectines ont montré les liaisons pas ou très faible pour capturer les anticorps, tandis que l'antigène de haute liaisons étaient encore observées dans les sous-réseaux dans la colonne 2 et 4, sur lequel un échantillon de sérum a été appliquée (figure 1B et 1D). Ces résultats ont montré la procédure de blocage chimiquement était une étape essentielle avant la mesure de glycanes sur des anticorps capturés glycoprotéines. En suivant le protocole, profils de glycosylation des glycoprotéines dans 22 HCC sérum peut être obtenu.
Expérience 2
Écran pour fucos modifiésylation sur glycoprotéines sériques spécifiques comme biomarqueurs pour la cirrhose du foie et de discrimination patients atteints de carcinome hépatocellulaire.
Le but de cette expérience est à l'écran pour fucosylation altérée sur glycoprotéines sériques spécifiques en tant que biomarqueurs qui sont discriminatoires à la cirrhose du foie et le carcinome hépatocellulaire (CHC) patients. Différent de l'expérience 1, dans lequel un seul échantillon de sérum a été appliqué sur tous les sous-réseaux et sondé avec différentes lectines, dans cet essai, le total des 40 différents échantillons de sérum de patients CHC et la cirrhose ont été appliquées sur chaque sous-réseau, et sondé avec une lectine (AAL ). L'analyse statistique, tels que le test T, le récepteur-caractéristique de fonctionnement (ROC), a été fait pour évaluer les distributions ou les performances diagnostiques de l'epiptope glycane / biomarqueur sur chaque protéine individuelle dans tous les échantillons de sérum. Nous avons utilisé la puce même anticorps fabriqués dans l'expérience 1, sauf pour l'anti-CA19-9 et anti-Lewis anticorps X dans cette étude. Le expertiseiment a été réalisée à partir de Sept 2 à l'étape 9, sauf pour l'étape 5.1.1 et 6.1.1. Total des 40 échantillons de sérum à partir de 20 cirrhose et 20 patients HCC ont été appliqués à sous-réseau aléatoire des sous-réseaux 48 ainsi que les échantillons de contrôle CPE comme contrôle négatif. Fucosylation de chaque protéines des saisies a ensuite été détecté en utilisant biotinylé Fucose-lectine spécifique. L'image microarray illustré à la figure 1 a démontré l'AAL lectine ne se lie aux protéines sériques capturés sur la puce (figure 2D) au lieu d'anticorps capturés (figure 2E). Les intensités AAL de liaison de tous les points ont ensuite été extraites et analysées en utilisant le test T et les courbes ROC pour évaluer la performance de la fucosylation (AAL intensité contraignante) de chaque protéine du sérum sur la discrimination entre le HCC et les groupes de cirrhose. Les résultats ont montré que la protéine GP73 fucosylation de donné la meilleure discrimination entre les deux groupes avec un p = 0,03 et l'aire sous-courbe de la courbe ROC est égal à 0,72. Cette expérience a démontré cette procédure est une méthode rapide, efficace pour le glycane épitope / biomarqueur de dépistage sur des échantillons multiples au sein de multiples protéines.
| ID | Nom du réactif | Abréviation | Entreprise | Catalogue # |
| L1 | Biotinylé concanavaline A | ConA | Vector Laboratories | BK-1000 |
| L2 | Biotinylé Sambucus Nigra Lectin | SCN | Vector Laboratories | B-1305 |
| L3 | Biotinylé Lens culinaris Agglutinin | LCA | Vector Laboratories | BK-2000 |
| L4 | Biotinylés agglutinine de Ricinus communis, je | RCA | Vector Laboratories | BK-1000 |
| L5 | Biotinylé Aleuria Aurantia Lectin | AAD | Vector Laboratories | B-1395 |
| L6 | Biotinylé Erythrina cristagalli Lectin | ECL | Vector Laboratories | BK-3000 |
| L7 | Biotinylé Griffonia (Bandeiraea) Lectin Simplicifolia II | GSL II | Vector Laboratories | BK-3000 |
| L8 | Biotinylé de germe de blé Agglutinin | WGA | Vector Laboratories | BK-1000 |
| L9 | Biotinylé Erythroagglutinin Phaseolus vulgaris | PHA-E | Vector Laboratories | BK-2000 |
| L10 | Biotinylé Leucoagglutinin Phaseolus vulgaris | PHA-L | Vector Laboratories | BK-2000 |
| L11 | BioAgglutinin arachide tinylated | PNA | Vector Laboratories | BK-1000 |
| L12 | Biotinylés Pisum sativum Agglutinin | PSA | Vector Laboratories | BK-2000 |
| L13 | Biotinylé Dolichos biflorus Agglutinin | DBA | Vector Laboratories | BK-1000 |
| L14 | Biotinylé Lectin Datura stramonium | DSL | Vector Laboratories | BK-3000 |
| L15 | Biotinylé Sophora japonica Agglutinin | ASJ | Vector Laboratories | BK-2000 |
| L16 | Agglutinine du soja biotinylé | SBA | Vector Laboratories | BK-1000 |
| L17 | Biotinylé Solanum tuberosum (pomme de terre) Lectin | STL | Vector Laboratories | BK-3000 |
| L18 | Biotinylé Lectin Griffonia Simplicifolia (Bandeiraea) Je | GSL, je | Vector Laboratories | BK-2000 |
| L19 | Biotinylé Vicia villosa Lectin | VVL | Vector Laboratories | BK-2000 |
| L20 | Biotinylé Lycopersicon esculentum (tomate) Lectin | LIE | Vector Laboratories | BK-3000 |
| L21 | Ulex biotinylé Europaeus Agglutinin je | UEA I | Vector Laboratories | BK-1000 |
| L22 | Biotinylé jacaline | Jacaline | Vector Laboratories | BK-3000 |
| A1 | F de chèvre (ab ') 2 Fragment IgM anti-humain, un anticorps Fc5μ | IgM | Jackson Immuno Research | 109-006-129 |
| A2 | Ane F (ab ') 2Frag anti-IgG humaine (H + L) antibody | AB1 | Jackson Immuno Research | 709-006-149 |
| A3 | Souris anti-IgG humaine F (ab ') 2 d'anticorps monoclonal | AB3 | Jackson Immuno Research | 209-005-097 |
| A4 | De chèvre anti-anticorps humain alpha macroglobuline 2 polyclonal | A2M | GeneTex | GTX62924 |
| A5 | De lapin anti-alpha humain-1-antitrypsine d'anticorps polyclonaux | A1AT | Lee Biosiences | CA1T-80A |
| A6 | Souris anti-humaine alpha-1-antitrypsine anticorps monoclonal | A1AT | Sigma-Aldrich | SAB4200198 |
| A7 | De lapin anti-alpha humain-1-antitrypsine d'anticorps polyclonaux | AGIR | NeoMarkers | RB-367-A1 |
| A8 | De lapin anti-humain alpha-1-antiquehymotrypsin anticorps polyclonal | AGIR | Fisher Scientific | RB9213R7 |
| A9 | Souris anti-transferrine humaine d'anticorps monoclonal | La transferrine | GeneTex | GTX101035 |
| A10 | De lapin anti-transferrine humaine d'anticorps polyclonaux | La transferrine | GeneTex | GTX77130 |
| A11 | De chèvre anti-humain apolipoprotéine J anticorps polyclonaux | ApoJ | Abcam | ab7610 |
| A12 | De souris anti-humaine GP73 anticorps monoclonal | GP73 | Abbott | 14H4-23 |
| A13 | De souris anti-humaine GP73 anticorps monoclonal | GP73 | Santa Cruz Biotechnology INC | sc-101275 |
| A14 | De lapin anti-humain alpha-1 foetoprotéine anticorps polyclonaux | AFP | GenWay | GWB-41C966 |
| A15 | Souris anti-humaine alpha-1 foetoprotéine anticorps monoclonal | AFP | Fitzgerald | 10-A05A |
| A16 | Souris anti-humaine hémopexine anticorps monoclonal | Hemopexin | Assaypro | 60190-05011 |
| A17 | Souris anti-humaine glypicane-3 (1G12) anticorps monoclonal | GPL3 | Santa Cruz Bio | sc-65443 |
| A18 | Souris anti-humaine kininogène (LMW) anticorps monoclonal | Kininogène | Assaypro | 20333-05011 |
| A19 | De lapin anti-humain MMP-21 anticorps monoclonal | MMP21 | Epitomic | 1955-1 |
| A20 | Souris anti-humaine CEACAM-1 anticorps monoclonal | CEACAM | R & D Systems | MAB1180 |
| Une21 | Rat anti-humaine DPPIV/cd26 anticorps monoclonal | DPPIV | R & D Systems | MAB22441 |
| A22 | Souris anti-humaine PIVKA II anticorps monoclonal | PIVICA | Cristal chem | 8040 |
| A23 | Souris anti-antigène carcino-embryonnaire | CEA | US biologique | C1300 |
| A24 | Antigène de souris anti-CA125 cancer | CA125 | US biologique | C0050-01D |
| A25 | Souris anti-CA19-9 du cancer antigène | CA19-9 | US biologique | C0075-18 |
| A26 | Souris anti-Lewis x anticorps monoclonal | X Lewis | Calbiochem | 434631 |
| bio | Biotinylé BSA (contrôle positif) | Bio | Home-made | N / A |
Tableau 1. Liste des lectines et les anticorps utilisés dans ce protocole.
| Nom de / du réactif s équipements | Entreprise | Numéro de catalogue |
| Microarrayer sans contact | BioDot Inc | sciFLEXARRAYER |
| 384 microplaques | Pêcheur | 14-230-243 |
| FoodSaver | FoodSaver | V3835 |
| Ultrathin nitrocellulose Coate microarray glisse | Gentel | PATH |
| Diapositive impression (en option) | La Société Gel | WSP60-1 |
| Shaker | Pêcheur | 15-453-211 |
| Centrifuger | Eppendorf | 5804 000.013 |
| Faites glisser lavabo / Slide bac à coloration wie Rack | Pêcheur | 08-812 |
| Incubation des lames diapositive boîte de chambre / microscope | Pêcheur | 03-448-5 |
| Brij 35, solution à 30% p / v dans l'eau | Acros Organics | AC32958-0025 |
| Tween-20 | Pêcheur | P337-100 |
| Periodate de sodium (NaIO 4) | Sigma | 311448 |
| L-acide glutamique γ-hydrazide | Sigma | G-7257 |
| Acétate de sodium anhydre (CH 3 COONa) | Sigma | S2889 |
| Bovine Serum Albumin (BSA) | Lampire Labs biologique | 7500804 |
| Tampon phosphate salin (PBS) (10X) | Denville scientifique | CP4390-48 |
| Dylight 549 neutravidine conjugué | Thermo | 22837 |
| Comprimés inhibiteurs de la protéase à cocktails | Roche | 4693159001 |
| ChromPure IgG humaine, fragment Fc | Jackson Immunoresearch | 009-000-008 |
| ChromPure IgG humaine, molécule entière | Jackson Immunoresearch | 009-000-003 |
| ChromPure IgG de souris, molécule entière | Jackson Immunoresearch | 015-000-003 |
| ChromPure IgG de souris, le fragment Fc | Jackson Immunoresearch | 015-000-008 |
| ChromPure IgG de lapin, molécule entière | Jackson Immunoresearch | 011-000-003 |
| ChromPure âne IgG, molécule ensemble | Jackson Immunoresearch | 017-000-003 |
| Microarray Scanner | Tecan | LS Reloaded |
Tableau 2. Listedes équipements et des réactifs utilisés dans le présent protocole.

Schéma 1 Un schéma montrant la puce anticorps lectine fondée processus de découverte de biomarqueurs glycane 1 (étape 2 à 4): Bloquer le microréseau d'anticorps avec le bloqueur (Glu-hydrazide) et de la BSA, 2 (étape 5):.. S'appliquent échantillons de sérum et de capturer glycoprotéines spécifiques avec des anticorps spécifiques, 3 (étape 6): appliquer lectine biotinylée (s), 4 (étape 7): la sonde de l'AAL biotinylé avec Dylight 549 neutravidine étiqueté pour microréseau d'imagerie.
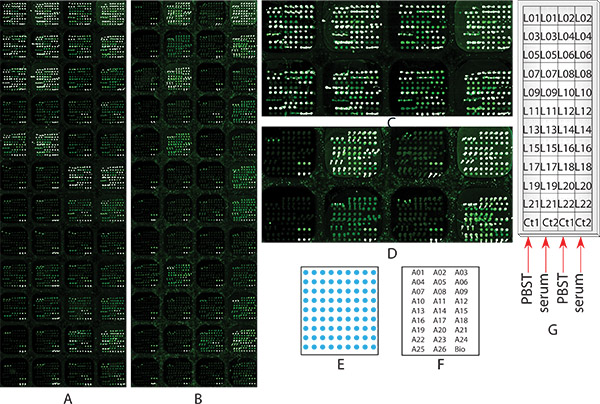
Figure 1. Images de puces à ADN de l'échantillon le profilage Expérience glycosylation 1 de multiples glycoprotéines sériques dans l'échantillon de sérum du patient CHC en utilisant chemicallié bloquée microréseau d'anticorps avec détection lectine multiples. Deux lames microarray identiques, (A) aucune chimiquement bloqué, ou (B) chimiquement bloqué comme décrit dans l'étape 2, à la fois est passé par toutes les étapes de 2 à 9 pour le profilage de glycosylation, ainsi que des fins de comparaison. (A) et (B) sont les images de microréseaux scanné à l'étape 8, dans une résolution de 10 microns. (C) le zoom dans l'image des deux premières lignes de la diapositive bloquée chimiquement aucune puce à ADN (A); (D) le zoom dans l'image des deux premières rangées de la diapositive non chimiquement puces bloqué (B)); (E) le schéma de l'agencement d'anticorps au sein de chaque sous-réseau; (F) des cartes du tableau: l'emplacement de chaque anticorps dans le sous-tableau, chaque nom d'anticorps représente 3 points; (G) et l'emplacement échantillon de sérum lectine: une montre le schéma qui sous-tableau de chaque échantillon de sérum et lectine a été appliqué sur.

Figure 2. Images de puces à ADN de laexpérience échantillon de 2 écran pour fucosylation altérée sur glycoprotéines sériques spécifiques en tant que biomarqueurs qui sont discriminatoires à la cirrhose du foie et les patients HCC. Le dosage microarray a été réalisée comme décrit dans l'expérience l'échantillon 2 de l'article. (A) L'image diapositive ensemble de la diapositive microréseau de l'étape 8; (B) le schéma de l'agencement d'anticorps au sein de chaque sous-réseau; (C) des cartes de type tableau: l'emplacement de chaque anticorps dans le sous-tableau, chaque nom d'anticorps représente 3 points; (D) un zoom avant sur l'image d'un sous-matrice qui ont été mis à incuber avec un échantillon de sérum; (E) un zoom en une image de sous-matrice qui ont été incubés avec du PBS de commande.

Figure 3. Résultats du profilage des glycanes de 1 expérience de l'échantillon. Chaque diagramme représentent le profil de liaison de lectine (ou profilés glycane) de l'un de la protéine 20 testée. Total des 22 lectines différentes ont été utilisées pour analyser les ee glycane le profil de chaque protéine.
Discussion
1. Protéine cible et la sélection d'anticorps de capture
Avant le dosage d'anticorps microarray, certains réactifs et matériels sont nécessaires pour être considéré et préparé. Pour concevoir un microréseau d'anticorps pour le profilage ou le dépistage des biomarqueurs glycane glycane, un panel d'anticorps spécifiques aux candidats glycoprotéine devrait être déterminée en fonction de la littérature ou à partir des résultats précédents. Ces anticorps ont été généralement achetés auprès de différents fournisseurs tels que la R & D Systems IgG etc sont préférables anticorps de capture depuis nos tests précédents ont montré que certains IgM et IgE peuvent perdre complètement leurs affinités de liaison d'antigène après la modification chimique.
2. Conception et fabrication de puces à ADN d'anticorps
Fabrication du microréseau d'anticorps est une étape facultative qui doit microarrayer professionnelle et coûteux, et un personnel bien formé pour l'opération. Toutefois, personnalisé anticorps microarray fabricationture peut être facilement fait à partir d'un fournisseur de services tels que Serome Biosciences Inc Pour un résultat robuste, nous recommandons une microarrayer non-contact, tels que le volume Scienion ultra sciFLEXARRAYER bas sans contact microarrayer qui a été utilisé dans notre fabrication microréseau d'anticorps. De liaison élevée diapositives microarray capacités, telles que PATH (Gentel Bio Inc WI) ou Diapositive H (Shott, PA).
3. Choisir et préparer glycanniques protéines de liaison (Gbps) pour le profilage de glycosylation
Gbps qui cible différente mono-ou oligosaccharides peuvent être trouvés dans la littérature et à travers un moteur de recherche développé par le laboratoire Haab 29 et maintenue à l'Institut de génomique translationnelle Pour sélectionner Gbps avec une forte spécificité et affinité pourles épitopes glycanniques ciblées, sélectionnez le motif (épitope) à partir de dans le menu déroulant, puis cliquez sur "Recherche". Le spécifique Gbps à ce motif (épitope) seront classés selon leur valeur logP de haut à l'ordre faible. Supérieur logP indique une plus forte affinité de liaison et la spécificité au motif glycane / épitope. En raison de la non-spécifique inhérente question contraignant de la lectine, cette méthode n'est pas encore aussi optimale que le dosage des anticorps base. Ainsi, nous recommandons fortement que les anticorps anti-glycanes sont utilisés, tels que des anti-Lewis x ou anti-sialyl Lewis A anticorps, si elles sont disponibles. Utilisation de multiples Gbps pour la détection est une autre stratégie afin de recouper les différents profils de liaison et d'obtenir des données fiables contraignantes.
4. L'analyse des données
Parce que cette méthode est utilisée pour détecter les protéines sériques natives, complexes protéine-protéine peuvent être capturés et détectés. Western blot ou spectrométrie de masse est de bonnes méthodes pour valider les données de biopuces. Dans l'intervalle, La détection des niveaux de protéines en utilisant la puce même est une autre méthode pour apprendre les détails de la modification de glycosylation de la protéine, comme si les changements étaient dus à la variation du niveau de protéines totales ou tout simplement que le niveau de glycosylation accrue sur chacune des protéines.
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Ce travail a été soutenu par l'Institut pour l'hépatite et de la recherche de virus.
matériels
| Name | Company | Catalog Number | Comments | |
| ID | Nom du réactif | Abréviation | Entreprise | Catalogue # |
| L1 | Biotinylé concanavaline A | ConA | Vector Laboratories | BK-1000 |
| L2 | Biotinylé Sambucus Nigra Lectin | SCN | Vector Laboratories | B-1305 |
| L3 | Biotinylé Lens culinaris Agglutinin | LCA | Vector Laboratories | BK-2000 |
| L4 | Biotinylés agglutinine de Ricinus communis, je | RCA | Vector Laboratories | BK-1000 |
| L5 | Biotinylé Aleuria Aurantia Lectin | AAD | Vector Laboratories | B-1395 |
| L6 | Biotinylé Erythrina cristagalli Lectin | ECL | Vector Laboratories | BK-3000 |
| L7 | Biotinylé Griffonia (Bandeiraea) Lectin Simplicifolia II | GSL II | Vector Laboratories | BK-3000 |
| L8 | Biotinylé de germe de blé Agglutinin | WGA | Vector Laboratories | BK-1000 |
| L9 | Biotinylé Erythroagglutinin Phaseolus vulgaris | PHA-E | Vector Laboratories | BK-2000 |
| L10 | Biotinylé Leucoagglutinin Phaseolus vulgaris | PHA-L | Vector Laboratories | BK-2000 |
| L11 | Agglutinin arachide biotinylé | PNA | Vector Laboratories | BK-1000 |
| L12 | Biotinylés Pisum sativum Agglutinin | PSA | Vector Laboratories | BK-2000 |
| L13 | Biotinylé Dolichos biflorus Agglutinin | DBA | Vector Laboratories | BK-1000 |
| L14 | Biotinylé Lectin Datura stramonium | DSL | Vector Laboratories | BK-3000 |
| L15 | Biotinylé Sophora japonica Agglutinin | ASJ | Vector Laboratories | BK-2000 |
| L16 | Agglutinine du soja biotinylé | SBA | Vector Laboratories | BK-1000 |
| L17 | Biotinylé Solanum tuberosum (pomme de terre) Lectin | STL | Vector Laboratories | BK-3000 |
| L18 | Biotinylé Griffonia (bandeeiraea) Simplicifolia Lectin je | GSL, je | Vector Laboratories | BK-2000 |
| L19 | Biotinylé Vicia villosa Lectin | VVL | Vector Laboratories | BK-2000 |
| L20 | Biotinylé Lycopersicon esculentum (tomate) Lectin | LIE | Vector Laboratories | BK-3000 |
| L21 | Ulex biotinylé Europaeus Agglutinin je | UEA I | Vector Laboratories | BK-1000 |
| L22 | Biotinylé jacaline | Jacaline | Vector Laboratories | BK-3000 |
| A1 | F de chèvre (ab ') 2 Fragment IgM anti-humain, un anticorps Fc5μ | IgM | Jackson Immuno Research | 109-006-129 |
| A2 | Ane F (ab ') 2 Frag anti-IgG humaine (H + L) unetibody | AB1 | Jackson Immuno Research | 709-006-149 |
| A3 | Souris anti-IgG humaine F (ab ') 2 d'anticorps monoclonal | AB3 | Jackson Immuno Research | 209-005-097 |
| A4 | De chèvre anti-anticorps humain alpha macroglobuline 2 polyclonal | A2M | GeneTex | GTX62924 |
| A5 | De lapin anti-alpha humain-1-antitrypsine d'anticorps polyclonaux | A1AT | Lee Biosiences | CA1T-80A |
| A6 | Souris anti-humaine alpha-1-antitrypsine anticorps monoclonal | A1AT | Sigma-Aldrich | SAB4200198 |
| A7 | De lapin anti-alpha humain-1-antitrypsine d'anticorps polyclonaux | AGIR | NeoMarkers | RB-367-A1 |
| A8 | De lapin anti-humainl'alpha-1-antichymotrypsine anticorps polyclonal | AGIR | Fisher Scientific | RB9213R7 |
| A9 | Souris anti-transferrine humaine d'anticorps monoclonal | La transferrine | GeneTex | GTX101035 |
| A10 | De lapin anti-transferrine humaine d'anticorps polyclonaux | La transferrine | GeneTex | GTX77130 |
| A11 | De chèvre anti-humain apolipoprotéine J anticorps polyclonaux | ApoJ | Abcam | ab7610 |
| A12 | De souris anti-humaine GP73 anticorps monoclonal | GP73 | Abbott | 14H4-23 |
| A13 | De souris anti-humaine GP73 anticorps monoclonal | GP73 | Santa Cruz Biotechnology INC | sc-101275 |
| A14 | De lapin anti-humain alpha-1 fetoprotein anticorps polyclonal | AFP | Genway | GWB-41C966 |
| A15 | Souris anti-humaine alpha-1 foetoprotéine anticorps monoclonal | AFP | Fitzgerald | 10-A05A |
| A16 | Souris anti-humaine hémopexine anticorps monoclonal | Hemopexin | Assaypro | 60190-05011 |
| A17 | Souris anti-humaine glypicane-3 (1G12) anticorps monoclonal | GPL3 | Santa Cruz Bio | sc-65443 |
| A18 | Souris anti-humaine kininogène (LMW) anticorps monoclonal | Kininogène | Assaypro | 20333-05011 |
| A19 | De lapin anti-humain MMP-21 anticorps monoclonal | MMP21 | Epitomic | 1955-1 |
| A20 | Souris anti-humaine-1 CEACAM antibod monoclonaly | CEACAM | R & D Systems | MAB1180 |
| A21 | Rat anti-humaine DPPIV/cd26 anticorps monoclonal | DPPIV | R & D Systems | MAB22441 |
| A22 | Souris anti-humaine PIVKA II anticorps monoclonal | PIVICA | Cristal chem | 8040 |
| A23 | Souris anti-antigène carcino-embryonnaire | CEA | US biologique | C1300 |
| A24 | Antigène de souris anti-CA125 cancer | CA125 | US biologique | C0050-01D |
| A25 | Souris anti-CA19-9 du cancer antigène | CA19-9 | US biologique | C0075-18 |
| A26 | Souris anti-Lewis x anticorps monoclonal | X Lewis | Calbiochem | 434631 |
| bio | Biotinylé BSA (contrôle positif) | Bio | Home-made | N / A |
Tableau 1. Liste des lectines et les anticorps utilisés dans ce protocole.
| Nom de / du réactif s équipements | Entreprise | Numéro de catalogue |
| Microarrayer sans contact | BioDot Inc | sciFLEXARRAYER |
| 384 microplaques | Pêcheur | 14-230-243 |
| FoodSaver | FoodSaver | V3835 |
| Ultrathin nitrocellulose Coate microarray glisse | Gentel | PATH |
| Diapositive impression (en option) | La Société Gel | WSP60-1 |
| Shaker | Pêcheur | 15-453-211 |
| Centrifuger | Eppendorf | 5804 000.013 |
| Glissez à laver la vaisselle coloration bassin / Slide avec rack amovible | Pêcheur | 08-812 |
| Incubation des lames diapositive boîte de chambre / microscope | Pêcheur | 03-448-5 |
| Brij 35, solution à 30% p / v dans l'eau | Acros Organics | AC32958-0025 |
| Tween-20 | Pêcheur | P337-100 |
| Periodate de sodium (NaIO 4) | Sigma | 311448 |
| L-acide glutamique γ-hydrazide | Sigma | G-7257 |
| Acétate de sodium anhydre (CH 3 COONa) | Sigma | S2889 |
| Bovine Serum Albumin (BSA) | Lampire Labs biologique | 7500804 |
| Tampon phosphate salin (PBS) (10X) | Denville scientifique | CP4390-48 |
| Dylight 549 neutravidine conjugué | Thermo | 22837 |
| Comprimés inhibiteurs de la protéase à cocktails | Roche | 4693159001 |
| ChromPure IgG humaine, fragment Fc | Jackson Immunoresearch | 009-000-008 |
| ChromPure IgG humaine, molécule entière | Jackson Immunoresearch | 009-000-003 |
| ChromPure IgG de souris, molécule entière | Jackson Immunoresearch | 015-000-003 |
| ChromPure IgG de souris, le fragment Fc | Jackson Immunoresearch | 015-000-008 |
| ChromPure IgG de lapin, molécule entière | Jackson Immunoresearch | 011-000-003 |
| ChromPure âne IgG, molécule ensemble | Jackson Immunoresearch | 017-000-003 |
| Microarray Scanner | Tecan | LS Reloaded |
Tableau 2. Liste des équipements et des réactifs utilisés dans le présent protocole.
Références
- Fang, M. The ER UDPase ENTPD5 promotes protein N-glycosylation, the Warburg effect, and proliferation in the PTEN pathway. Cell. 143, 711-724 (2010).
- Marino, K., Bones, J., Kattla, J. J., Rudd, P. M. A systematic approach to protein glycosylation analysis: a path through the maze. Nat. Chem. Biol. 6, 713-723 (2010).
- Shental-Bechor, D., Levy, Y. Effect of glycosylation on protein folding: a close look at thermodynamic stabilization. Proc. Natl. Acad. Sci. U. S. A. 105, 8256-8261 (2008).
- Hossler, P., Khattak, S. F., Li, Z. J. Optimal and consistent protein glycosylation in mammalian cell culture. Glycobiology. 19, 936-949 (2009).
- Nothaft, H., Szymanski, C. M. Protein glycosylation in bacteria: sweeter than ever. Nat. Rev. Microbiol. 8, 765-778 (2011).
- Sola, R. J., Griebenow, K. Effects of glycosylation on the stability of protein pharmaceuticals. J. Pharm. Sci. 98, 1223-1245 (2009).
- Li, C., Lubman, D. M. Analysis of serum protein glycosylation with antibody-lectin microarray for high-throughput biomarker screening. Methods Mol. Biol. 723, 15-28 (2011).
- Dwek, M. V., Jenks, A., Leathem, A. J. A sensitive assay to measure biomarker glycosylation demonstrates increased fucosylation of prostate specific antigen (PSA) in patients with prostate cancer compared with benign prostatic hyperplasia. Clin. Chim. Acta. 411, 1935-1939 (2010).
- Drake, P. M. Sweetening the pot: adding glycosylation to the biomarker discovery equation. Clin. Chem. 56, 223-236 (2010).
- Kim, Y. -. P., Park, S., Oh, E., Oh, Y. -. H., Kim, H. -. S. On-chip detection of protein glycosylation based on energy transfer between nanoparticles. Biosensors & Bioelectronics. 24, 1189-1194 (2009).
- Boland, M., Rudd, P. M. Disease related glycosylation changes and biomarker discovery: challenges and possibilities in an emerging field. Editorial. Dis. Markers. 25, 189-192 (2008).
- Norton, P. A. N-linked glycosylation of the liver cancer biomarker GP73. J. Cell Biochem. 104, 136-149 (2008).
- Nakagawa, T. Glycomic analysis of alpha-fetoprotein L3 in hepatoma cell lines and hepatocellular carcinoma patients. J. Proteome Res. 7, 2222-2233 (2008).
- Durazo, F. A. Des-gamma-carboxyprothrombin, alpha-fetoprotein and AFP-L3 in patients with chronic hepatitis, cirrhosis and hepatocellular carcinoma. J. Gastroenterol Hepatol. 23, 1541-1548 (2008).
- Kobayashi, M. Fucosylated fraction of alpha-fetoprotein, L3, as a useful prognostic factor in patients with hepatocellular carcinoma with special reference to low concentrations of serum alpha-fetoprotein. Hepatol. Res. 37, 914-922 (2007).
- Maisey, N. R. CA19-9 as a prognostic factor in inoperable pancreatic cancer: the implication for clinical trials. Br. J. Cancer. 93, 740-743 (2005).
- Talar-Wojnarowska, R. Clinical value of serum neopterin, tissue polypeptide-specific antigen and CA19-9 levels in differential diagnosis between pancreatic cancer and chronic pancreatitis. Pancreatology. 10, 689-694 (2010).
- Chen, S. Multiplexed analysis of glycan variation on native proteins captured by antibody microarrays. Nat. Methods. 4, 437-444 (2007).
- Shao, C. Antibody microarray analysis of serum glycans in esophageal spuamous cell carcinoma cases and controls. Proteomics Clinical Applications. 3, 923-931 (2009).
- Chen, S., Haab, B. B. Analysis of glycans on serum proteins using antibody microarrays. Methods Mol. Biol. 520, 39-58 (2009).
- Yue, T. The Prevalence and Nature of Glycan Alterations on Specific Proteins in Pancreatic Cancer Patients Revealed Using Antibody-Lectin Sandwich Arrays. Molecular & Cellular Proteomics. 8, 1697-1707 (2009).
- Wolf-Yadlin, A., Sevecka, M., MacBeath, G. Dissecting protein function and signaling using protein microarrays. Current Opinion in Chemical Biology. 13, 398-405 (2009).
- Richard, E. Proteomics as Applied to Inherited Metabolic Diseases. Current Proteomics. 6, 140-153 (2009).
- Nolen, B., Winans, M., Marrangoni, A., Lokshin, A. Aberrant tumor-associated antigen autoantibody profiles in healthy controls detected by multiplex bead-based immunoassay. Journal of Immunological Methods. 344, 116-120 (2009).
- Kuno, A. Focused Differential Glycan Analysis with the Platform Antibody-assisted Lectin Profiling for Glycan-related Biomarker Verification. Molecular & Cellular Proteomics. 8, 99-108 (2009).
- Hsu, K. -. L., Mahal, L. K. Sweet tasting chips: microarray-based analysis of glycans. Current Opinion in Chemical Biology. 13, 427-432 (2009).
- Borrebaeck, C. A. K., Wingren, C. High-throughput proteomics using antibody microarrays: an update. Expert Review of Molecular Diagnostics. 7, 673-686 (2007).
- Sanchez-Carbayo, M. Antibody array-based technologies for cancer protein profiling and functional proteomic analyses using serum and tissue specimens. Tumor Biology. 31, 103-112 (2010).
- Porter, A. A motif-based analysis of glycan array data to determine the specificities of glycan-binding proteins. Glycobiology. 20, 369-380 (2010).
- Maupin, K. A. Glycogene Expression Alterations Associated with Pancreatic Cancer Epithelial-Mesenchymal Transition in Complementary Model Systems. Plos One. 5, (2010).
- Sevecka, M., Wolf-Yadlin, A., MacBeath, G. Lysate Microarrays Enable High-throughput, Quantitative Investigations of Cellular Signaling. Molecular & Cellular Proteomics. 10, (2011).
- Wang, M. Novel fucosylated biomarkers for the early detection of hepatocellular carcinoma. Cancer Epidemiol. Biomarkers Prev. 18, 1914-1921 (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon