Method Article
Chemisch-Antikörper blockiert Microarray für Multiplex-High-Throughput-Profiling von spezifischen Protein-Glykosylierung bei Complex Samples
In diesem Artikel
Zusammenfassung
In dieser Studie beschreiben wir ein verbessertes Protokoll für eine Multiplex-Hochdurchsatz-Antikörper Mikroarray mit Lektin Detektionsverfahren, das in der Glykosylierung Profilierung spezifischer Proteine verwendet werden können. Dieses Protokoll bietet zuverlässige neue Reagenzien und reduziert den Zeitaufwand, Kosten und Laborgeräte Anforderungen an das vorherige Verfahren verglichen.
Zusammenfassung
In this study, we describe an effective protocol for use in a multiplexed high-throughput antibody microarray with glycan binding protein detection that allows for the glycosylation profiling of specific proteins. Glycosylation of proteins is the most prevalent post-translational modification found on proteins, and leads diversified modifications of the physical, chemical, and biological properties of proteins. Because the glycosylation machinery is particularly susceptible to disease progression and malignant transformation, aberrant glycosylation has been recognized as early detection biomarkers for cancer and other diseases. However, current methods to study protein glycosylation typically are too complicated or expensive for use in most normal laboratory or clinical settings and a more practical method to study protein glycosylation is needed. The new protocol described in this study makes use of a chemically blocked antibody microarray with glycan-binding protein (GBP) detection and significantly reduces the time, cost, and lab equipment requirements needed to study protein glycosylation. In this method, multiple immobilized glycoprotein-specific antibodies are printed directly onto the microarray slides and the N-glycans on the antibodies are blocked. The blocked, immobilized glycoprotein-specific antibodies are able to capture and isolate glycoproteins from a complex sample that is applied directly onto the microarray slides. Glycan detection then can be performed by the application of biotinylated lectins and other GBPs to the microarray slide, while binding levels can be determined using Dylight 549-Streptavidin. Through the use of an antibody panel and probing with multiple biotinylated lectins, this method allows for an effective glycosylation profile of the different proteins found in a given human or animal sample to be developed.
Introduction
Glycosylation of protein, which is the most ubiquitous post-translational modification on proteins, modifies the physical, chemical, and biological properties of a protein, and plays a fundamental role in various biological processes1-6. Because the glycosylation machinery is particularly susceptible to disease progression and malignant transformation, aberrant glycosylation has been recognized as early detection biomarkers for cancer and other diseases 7-12. In fact, most current cancer biomarkers, such as the L3 fraction of α-1 fetoprotein (AFP) for hepatocellular carcinoma 13-15, and CA199 for pancreatic cancer 16, 17 are all aberrant glycan moieties on glycoproteins. However, methods to study protein glycosylation have been complicated, and not suitable for routine laboratory and clinical settings. Chen et al. has recently invented a chemically blocked antibody microarray with a glycan-binding protein (GBP) detection method for high-throughput and multiplexed profile glycosylation of native glycoproteins in a complex sample 18. In this affinity based microarray method, multiple immobilized glycoprotein-specific antibodies capture and isolate glycoproteins from the complex mixture directly on the microarray slide, and the glycans on each individual captured protein are measured by GBPs. Because all normal antibodies contain N-glycans which could be recognized by most GBPs, the critical step of this method is to chemically block the glycans on the antibodies from binding to GBP. In the procedure, the cis-diol groups of the glycans on the antibodies were first oxidized to aldehyde groups by using NaIO4 in sodium acetate buffer avoiding light. The aldehyde groups were then conjugated to the hydrazide group of a cross-linker, 4-(4-N-MaleimidoPhenyl)butyric acid Hydrazide HCl (MPBH), followed by the conjugation of a dipeptide, Cys-Gly, to the maleimide group of the MPBH. Thus, the cis-diol groups on glycans of antibodies were converted into bulky none hydroxyl groups, which hindered the lectins and other GBPs bindings to the capture antibodies. This blocking procedure makes the GBPs and lectins bind only to the glycans of captured proteins. After this chemically blocking, serum samples were incubated with the antibody microarray, followed by the glycans detection by using different biotinylated lectins and GBPs, and visualized with Cy3-streptavidin. The parallel use of an antibody panel and multiple lectin probing provides discrete glycosylation profiles of multiple proteins in a given sample 18-20. This method has been used successfully in multiple different labs 1, 7, 13, 19-31. However, stability of MPBH and Cys-Gly, complicated and extended procedure in this method affect the reproducibility, effectiveness and efficiency of the method. In this new protocol, we replaced both MPBH and Cys-Gly with one much more stable reagent glutamic acid hydrazide (Glu-hydrazide), which significantly improved the reproducibility of the method, simplified and shorten the whole procedure so that the it can be completed within one working day. In this new protocol, we describe the detailed procedure of the protocol which can be readily adopted by normal labs for routine protein glycosylation study and techniques which are necessary to obtain reproducible and repeatable results.
Protokoll
1. Drucken Sie einen Antikörper Microarray für die Assay-
- Die Serum-Antikörper bis 0,5 mg / ml in phosphatgepufferter Kochsalzlösung, pH 7,2 (PBS).
- 40 ul Aliquot jedes Antikörpers in die 384-Well-Platte Quelle.
- Laden Sie das 384-Well-Platte auf die Quelle Scienion sciFLEXARRAYER Microarrayer.
- Legen Sie 20 PATH Microarray Slides auf den Microarrayer als Ziel.
- Stellen Sie die Microarrayer zu 48 identische Untergruppen, in denen 27-Antikörpern und Kontroll-Proteinen in dreifacher Ausfertigung in einer 9x9-Muster (Abbildung 1E, 1F) gesichtet werden zu drucken.
- Starten Sie den Microarrayer, um die Antikörper Microarray-Slides zu drucken.
- Sammeln Sie die Antikörper Microarray Slides, und speichern sie in Dias Kassette mit Trockenmittel. Vakuumieren Sie die Kassette in einer Plastiktüte mit Folienschweißgerät (FoodSaver).
- Lagern Sie die versiegelte Microarray Slides bei 4 ° C im Kühlschrank.
2. Chemisch blockieren Antikörper Microarray GBP zu verhindernBindung an die Fänger-Antikörper
Die Mikroarray-Test beginnt, sobald die Microarray-Slides sind chemisch blockiert und dauert ca. 8 Stunden. Nach dem Start der Microarray-Assay wurde abgeschlossen werden (Schritte 2 bis 8).
- Nehmen Sie die Microarray-Slides aus dem Kühlschrank, und sie ins Gleichgewicht auf Raumtemperatur für 30 Minuten.
- Entfernen der Folie aus dem Aufbewahrungsbehälter und kurz spülen in phosphatgepufferter Kochsalzlösung pH 7,2 mit 0,1% Tween 20 (PBST0.1) einmal in einer Folie Waschbecken, und dann in 15 mM Natriumacetat, pH 5,0 mit 0,1% Tween (CBT0 .1) in einer sequenziellen Weise. Die Objektträger in CBT0.1 für 10 Minuten in Folie Waschbecken.
- Bereiten Sie frischen 150 mM NaIO 4 in 15 mM Natriumacetat-Puffer pH 5,0 (CB), und halten Sie sie in in eine Folie Waschbecken in einem Kühlschrank bei gleichzeitiger Vermeidung Licht vor dem Gebrauch.
- Entfernen Sie die Folie aus der CB, und steckte es in das Becken mit frischem NaIO 4 mit dem Antikörper Seitenach oben zeigt. Decken Sie die Schüssel mit Alufolie, um Licht zu vermeiden, und inkubieren Sie die Folie Becken für 2 Stunden unter leichtem Schütteln bei 4 ° C im Kühlschrank aufbewahrt.
- Bereiten Sie 300 ml 10 mM Hydrazid Glutaminsäure (der Blocker) in CB.
- Entfernen Sie die Folie aus dem Becken, und spülen Sie ihn kurz in CB 3 mal für jeweils 5 min in Folie Waschbecken.
- Objektträger in der Blocker in ein Waschbecken für 2 Stunden bei Raumtemperatur unter leichtem Schütteln.
- Entfernen Sie die Dias aus dem Becken, und waschen Sie sie mit PBST0.1 für 3 Minuten.
3. Sperren nicht-spezifische Bindungen zu dem Mikroarray mit Rinderserumalbumin (BSA)
- Planen 300 ml 1% BSA in phosphatgepufferter Kochsalzlösung pH 7,2 mit 0,5% Tween (PBST0.5) in einem Schlitten Waschbecken, und Inkubieren des Mikroarrays Folie in dem Becken für 1 Stunde bei Raumtemperatur mit sanftem Schütteln.
- Spülen Sie die Folien in PBST0.1 dreimal für jeweils 3 Minuten Zeit.
- Legen Sie die Folieauf einer Folie Rack, und drehen sich mit 1.200 xg auf einer Zentrifugation für 2 Minuten, um die Dia-Mikroarray trocknen.
4. Impressum Wax Gitter auf den Microarray-Slide an jede Untergruppe Trennen
- Vorwärmen das Wachs Imprinter bei 70 ° C für 5 Minuten.
- Laden Sie das blockierte Microarray gleiten in den Wachs-Imprinter mit Antikörper Seite nach dem Wachs. Ziehen Sie den Griff zum Impressum Wachs gleichmäßig auf den Objektträger.
5. Bewerben Serumproben auf den Microarray-Slide
- Während der Schritt 2.4, bereiten Serumproben entweder für Glyco-Profiling-Test in einer Probe (5.1.1), oder einzelne Glyco epiptope Messung von mehreren Proben (5.1.2).
- In einem Experiment für die Glykan Profilierungen mehrerer Serumglycoproteinen in einer Serumprobe, indem Sie mehrere Euros (siehe Probe Experiment 1), wird ein Serum-Proben auf alle Untergruppen angewendet werden. In diesem Fall wird 40 ul Serum ausreichend in 360 ul PBS, enthaltend 0,1% verdünntTween-20, 0,1% Brij 35, 100 ug / ml Maus-IgG, 100 ug / ml Ratten-IgG, 100 ug / ml Kaninchen-IgG, 100 ug / ml Ziegen IgG und 100 ug / ml IgG Esel. Diese Menge ist ausreichend für die Anwendung von 6 ul verdünnte Serum-Lösung auf jedes Sub-Arrays.
- In einem Experiment für die eine Glykan Messung an mehreren Serumproteine unter mehreren Serumproben unter Verwendung eines GBP Erkennungen (siehe Beispiel Experiment 2). In diesem Fall wird 1 ul Serum ausreichend in 9 l PBS, enthaltend 0,1% Tween-20 verdünnt, 0,1% Brij 35, 100 ug / ml Maus-IgG, 100 ug / ml Ratten-IgG, 100 ug / ml Kaninchen-IgG , 100 ug / ml Ziegen IgG und 100 ug / ml IgG Esel. Diese Menge ist ausreichend für die Anwendung von 6 ul verdünnte Serum-Lösung auf jedes Sub-Arrays.
- Nach Wachs Impressum in Schritt 4, sorgfältig anwenden 6 ul der verdünnten Proben bzw. Kontrollproben (PBST0.1) zu jedem Subarray des Schlittens. Inkubieren Sie den Objektträger in einer feuchten Kassette mit feuchten Papiertüchern bei Raumtemperaturfür 1 Stunde.
- Spülen Sie den Schlitten mit PBST0.1 dreimal für jeweils 3 Minuten Zeit.
- Trocknen Sie die Folie, durch Drehen der bei 1200 xg für 2 Minuten.
6. Bewerben Biotinyliertes GBP (Lektin oder Anti-Glykan Antikörper) auf den Objektträger
- Während der Schritt 2.4, bereiten 10μg/ml von biotinylierten Lektine / Gbps in PBST0.1.
- In Glykan Profiling-Experiment, dass die Sonde eine Probe mit mehreren Lektine (Probe Experiment 1), bereiten 350 ul biotinylierten Lektin, das reicht für alle Teilfelder ist.
- In einzelnen Glykan Epitop / Biomarker-Screening in mehreren Proben durch die Verwendung mehrerer Lektine, bereiten 10 ul jeder biotinylierten Lektin, das reicht für eine Subarray ist.
- Bewerben 6 ul der verdünnten biotinylierten Lektin (s) zu jeder Teilmatrix von der Folie, und bebrüten in der befeuchteten schiebeschachtel mit feuchten Papiertüchern bei Raumtemperatur für 1 Stunde.
- Spülen Sie die Dias mit PBST0.1 dreimal für jeweils 3 Minuten timich.
- Trocknen Sie die Folie, durch Drehen der bei 1200 xg in der Zentrifuge für 2 Minuten.
7. Bewerben Farbstoff markiert NeutrAvidin für Fluoreszenzdetektion
- Bereiten Sie 350 ul DyLight 549 beschriftet NeutrAvidin, die ausreichend für alle Teilfelder ist.
- Bewerben 6 &mgr; l DyLight 549 beschriftet NeutrAvidin auf jede Untergruppe, und inkubieren Sie die Folie in der befeuchteten Objektträger Kassette bei Raumtemperatur für 1 Stunde.
- Spülen Sie den Schlitten mit PBST0.1 dreimal für jeweils 3 Minuten Zeit.
- Trocknen Sie die Folie durch Spinnen der es bei 1200 xg in der Zentrifuge für 2 Minuten.
8. Erhalten Microarray Slide Bild durch Scannen des Slide
- Scannen Sie das Bild mit Hilfe eines Fluoreszenz-Mikroarray-Scanner mit 10 mm Auflösung. Die Laser-und PMT-Einstellungen sollten so stark sein wie möglich, aber keine Sättigung vor Ort beobachtet wird.
9. Datenextraktion und-analyse
- Öffnen Sie das Bild in ArrayPro 3.2.
- Richten Sie den Array-Vorlage nach dem Array Karte, die die Antikörper Flecken Standorten zeigt. Richten Sie jede Vorlage Kreis auf die entsprechende Stelle im Bild.
- Entpacken Sie die Intensität jedes Spots in eine Excel-Datei zur weiteren Analyse.
10. Repräsentative Ergebnisse
Probe Experiment 1
Glykosylierung Profilierung von mehreren Serumglycoproteinen in hepatozellulären Karzinoms Patientenserum Beispiel mithilfe von chemisch blockiert Antikörper-Mikroarray mit mehreren Lektine Erkennung.
Das Ziel dieses Experiments ist es, die einzelnen Glycosylierungsprofil von 20 Glykoproteine in hepatozellulären Karzinoms (HCC) Patientenproben untersuchen mit Hilfe chemisch blockiert Antikörper-Mikroarray mit Lektin-Erkennung. Ein Antikörper-Mikroarray, der 48 identischen Untergruppen, die 26-Antikörpern und Biotin-BSA zählen enthält, wurde entwickelt und gebaut wie in S beschriebenTEP-1. Diese 26 Antikörper wurden gegen 20 Serumglycoproteinen, die als erste vielversprechende Diagnose Wert für HCC-Patienten unter Verwendung Lectin basierten Immunpräzipitation mit massenspektrometrischen Proteinidentifizierung 12, 32, wie in Tabelle 1 gezeigt kombiniert identifiziert. Das Muster und die Anordnung der Antikörper Stellen in dreifacher Ausfertigung in einer repräsentativen Subarray gedruckt werden in 1E und 1F gezeigt. Zwei identische Microarray Substrate, war eine nicht chemisch blockiert (1A), während die andere wurde (1B), wurden verwendet, um die Durchführung der gleichen Glykosylierung Profilierung Experiment, um die Bedeutung der chemisch blockiert Verfahren zur Analyse nachweisen. Für die chemisch blockiert Schieber (Abbildung 1B), begann das Experiment an der Stufe 2, für die keine chemisch blockiert Schieber (Abbildung 1A), begann das Experiment ab Schritt 3. Das Experiment wurde durchgeführt, indem following alle Schritte in dem Protokoll mit Ausnahme von Schritt 5.1.2 und 6.1.2 beschrieben. Im Schritt 5.2 wurde eine Kontrollprobe PBST0.1 auf Subarrays in Spalte 1 und 3 angelegt und eine gepoolte HCC Serumprobe wurde auf Teilfelder in Spalte 2 bzw. 4 aufgetragen (wie in Figur 1G gezeigt). Dieser Vergleich ist es, die Effektivität, Effizienz des Verfahrens, sowie die Antigen-Bindungsaffinität der Antikörper nach der chemisch blockiert zeigen. 22 biotinylierten Lektine (wie in Tabelle 1 gezeigt), die speziell für verschiedene Glykane 18, 20 auf jeder Teilmatrix angewendet wurden, wie in 1G für die Glycosylierung Profilierung gezeigt. Bilder der chemisch blockiert (Abbildung 1B) und nicht-chemisch blockiert (Abbildung 1A) Microarrays nach der Glykosylierung Profiling-Test, indem Sie das Protokoll. Wie in den Subarrays in Spalte 1 und 3 in nicht-chemisch blockiert Mikroarrays (1A und 1C), gezeigt, auf demnur PBST0.1 angelegt wurde, die meisten Lektine gebunden an Antikörper zu erfassen, und zeigten eine sehr hohe Hintergrund vergleichbar mit den Sub-Arrays in Spalte 2 und 4, auf dem der Serum-Probe wurde aufgetragen. Es ist unmöglich, Glykan Profilinformationen ins Mikroarray Folie zu erhalten. Im Gegenteil, wenn das gleiche Experiment auf einem chemisch blockiert Antikörper Microarray Objektträger wurde durchgeführt, die Sub-Arrays in Spalte 1 und 3, auf denen nur PBST0.1 angelegt wurde, zeigten die meisten Lektine keine oder nur geringe Bindungen an Antikörper zu erfassen, während hohe Antigen Bindungen wurden noch in Untergruppen in Spalte 2 und 4 beobachtet, an dem Serumprobe angewandt wurde (1B und 1D). Diese Ergebnisse zeigten, das chemisch blockiert Verfahren war ein entscheidender Schritt Vordergrund der Messung der Antikörper-Glykane auf Glykoproteinen eingefangen. Mit dem Anschluss an das Protokoll, kann Glycosylierungsprofilen von 22 Glykoproteine in HCC Serum gewonnen werden.
Experiment 2
Screen für veränderte fucoslierung auf spezifische Serum-Glykoproteine als Biomarker für diskriminiert Leberzirrhose und hepatozelluläres Karzinom-Patienten.
Das Ziel dieses Experiments ist es, Bildschirm für veränderte Fucosylierung auf spezifische Serum-Glykoproteine als Biomarker, die Leberzirrhose und hepatozelluläres Karzinom (HCC) Patienten zu diskriminieren. Verschiedene aus dem Experiment 1, die nur ein Serumprobe auf alle Untergruppen angewendet wurde und sondiert mit verschiedenen Lectinen, in diesem Assay, insgesamt 40 verschiedene Serumproben von Patienten mit Leberzirrhose und HCC wurden auf jede Untergruppe angelegt, und sondiert mit einem Lektin (AAL ). Die statistische Analyse, wie T-Test, Receiver-Operating Characteristic (ROC)-Kurve, wurde durchgeführt, um die Verteilungen oder diagnostische Leistung des Glycans epiptope / Biomarker für jedes einzelne Protein in allen Serumproben zu bewerten. Wir verwendeten die gleichen Antikörper Mikroarray in Versuch 1 hergestellt, außer für anti-CA19-9 und anti-Lewis-X-Antikörper in dieser Studie. Das Knowiment wurde vom 2. September bis 9 durchgeführt, außer für den Schritt 5.1.1 und 6.1.1 fort. Insgesamt 40 Serumproben von 20 Leberzirrhose und 20 Patienten mit HCC wurden zufällig Untermatrix der 48 Subarrays zusammen mit den Steuerdaten PBS Proben als negative Kontrolle eingesetzt. Fukosylierung jedes der erfassten Proteine wurde dann durch Verwendung von biotinylierten Fucose-spezifischen Lektin detektiert. Die Mikroarray-Bild in 1 gezeigt nachgewiesen das Lektin AAL nur Serumproteinen, auf dem Mikroarray (2D) anstelle von gebundenen Antikörper (2E) aufgenommen gebunden. Die AAL Bindung Intensitäten aller Punkte wurden dann extrahiert und analysiert unter Verwendung von T-Test und ROC-Kurven, um die Leistung des Fucosylierung (AAL Bindungsintensität) von jedem Serum-Protein auf der Diskriminierung zwischen den HCC und Zirrhose evaluieren. Die Ergebnisse zeigten, dass die Fucosylierung von GP73-Protein die beste Diskriminierung zwischen den beiden Gruppen gab mit einem p = 0,03 und die Fläche unter-Kurve der ROC-Kurve ist gleich 0,72. Dieses Experiment zeigte, dieses Verfahren ist eine schnelle, effiziente Methode zur Glycan Epitop / Biomarker-Screening auf mehrere Proben innerhalb mehrerer Proteine.
| Identifikation | Name des Reagenzes | Abkürzung | Firma | Katalog # |
| L1 | Biotinyliertes Concanavalin A | ConA | Vector Laboratories | BK-1000 |
| L2 | Biotinyliertes Sambucus nigra-Lectin | SNA | Vector Laboratories | B-1305 |
| L3 | Biotinyliertes Lens culinaris Agglutinin | LCA | Vector Laboratories | BK-2000 |
| L4 | Biotinylierte Ricinus communis-Agglutinin I | RCA | Vector Laboratories | BK-1000 |
| L5 | Biotinyliertes Aleuria aurantia-Lectin | AAL | Vector Laboratories | B-1395 |
| L6 | Biotinyliertes Erythrina cristagalli Lektin | ECL | Vector Laboratories | BK-3000 |
| L7 | Biotinyliertes Griffonia (Bandeiraea) simplicifolia Lektin II | GSL II | Vector Laboratories | BK-3000 |
| L8 | Biotinyliertes Weizenkeim-Agglutinin | WGA | Vector Laboratories | BK-1000 |
| L9 | Biotinyliertes Phaseolus vulgaris Erythroagglutinin | PHA-E | Vector Laboratories | BK-2000 |
| L10 | Biotinyliertes Phaseolus vulgaris Leucoagglutinin | PHA-L | Vector Laboratories | BK-2000 |
| L11 | Biobiotinylierte Erdnußagglutinin | PNA | Vector Laboratories | BK-1000 |
| L12 | Biotinylierte Pisum sativum Agglutinin | PSA | Vector Laboratories | BK-2000 |
| L13 | Biotinyliertes Dolichos biflorus Agglutinin | DBA | Vector Laboratories | BK-1000 |
| L14 | Biotinyliertes Datura Stramonium Lektin | DSL | Vector Laboratories | BK-3000 |
| L15 | Biotinyliertes Sophora Japonica Agglutinin | SJA | Vector Laboratories | BK-2000 |
| L16 | Biotinyliertes Sojabohnen Agglutinin | SBA | Vector Laboratories | BK-1000 |
| L17 | Biotinyliertes Solanum tuberosum (Kartoffel) Lektin | STL | Vector Laboratories | BK-3000 |
| L18 | Biotinyliertes Griffonia (Bandeiraea) simplicifolia Lektin I | GSL ich | Vector Laboratories | BK-2000 |
| L19 | Biotinylierte Vicia villosa-Lectin | VVL | Vector Laboratories | BK-2000 |
| L20 | Biotinyliertes Lycopersicon esculentum (Tomate) Lektin | UEG | Vector Laboratories | BK-3000 |
| L21 | Biotinyliertes Ulex europaeus Agglutinin I | UEA I | Vector Laboratories | BK-1000 |
| L22 | Biotinyliertes Jacalin | Jacalin | Vector Laboratories | BK-3000 |
| A1 | Ziegen-F (ab ') 2 Fragment Anti-Human-IgM, Antikörper Fc5μ | IgM | Jackson Immuno Research | 109-006-129 |
| A2 | Esel F (ab ') 2Frag Anti-Human-IgG (H + L) antibody | AB1 | Jackson Immuno Research | 709-006-149 |
| A3 | Maus Anti-Human-IgG F (ab ') 2-Antikörpers | AB3 | Jackson Immuno Research | 209-005-097 |
| A4 | Anti-Human-Alpha-2-Makroglobulin polyklonalen Antikörper | A2M | GeneTex | GTX62924 |
| A5 | Kaninchen anti-Human-alpha-1-Antitrypsin polyklonaler Antikörper | A1AT | Lee Biosiences | CA1T-80A |
| A6 | Maus Anti-Human-alpha-1-Antitrypsin monoklonalen Antikörper | A1AT | Sigma Aldrich | SAB4200198 |
| A7 | Kaninchen anti-Human-alpha-1-Antitrypsin polyklonaler Antikörper | HANDELN | NeoMarkers | RB-367-A1 |
| A8 | Kaninchen anti-human alpha-1-antichymotrypsin polyklonaler Antikörper | HANDELN | Fisher Scientific | RB9213R7 |
| A9 | Maus Anti-Human-Transferrin monoklonalen Antikörper | Transferrin | GeneTex | GTX101035 |
| A10 | Kaninchen anti-Human-Transferrin polyklonaler Antikörper | Transferrin | GeneTex | GTX77130 |
| A11 | Anti-Human-Apolipoprotein J polyklonaler Antikörper | ApoJ | Abcam | ab7610 |
| A12 | Maus Anti-Human-GP73 monoklonalen Antikörper | GP73 | Abbott | 14H4-23 |
| A13 | Maus Anti-Human-GP73 monoklonalen Antikörper | GP73 | Santa Cruz Biotechnology INC | sc-101275 |
| A14 | Kaninchen anti-Human-alpha-1 Fetoprotein polyklonaler Antikörper | AFP | GenWay | GWB-41C966 |
| A15 | Maus Anti-Human-alpha-1 Fetoprotein monoklonalen Antikörper | AFP | Fitzgerald | 10-A05A |
| A16 | Maus Anti-Human-Hämopexin monoklonalen Antikörper | Hämopexin | Assaypro | 60190-05011 |
| A17 | Maus Anti-Human-Glypican-3 (1G12) monoklonaler Antikörper | GPL3 | Santa Cruz Bio | sc-65443 |
| A18 | Maus Anti-Human-Kininogen (LMW) monoklonaler Antikörper | Kininogen | Assaypro | 20333-05011 |
| A19 | Kaninchen anti-Human-MMP-21 monoklonalen Antikörpers | MMP21 | Epitomic | 1955-1 |
| A20 | Maus Anti-Human-CEACAM-1 monoklonalen Antikörper | CEACAM | R & D Systems | MAB1180 |
| Ein21 | Ratten-Anti-Human-DPPIV/CD26 monoklonalen Antikörper | DPPIV | R & D Systems | MAB22441 |
| A22 | Maus Anti-Human-PIVKA-II-monoklonalen Antikörpers | PIVICA | Kristall-chem | 8040 |
| A23 | Maus anti-carcinoembryonales Antigen | CEA | US biologischen | C1300 |
| A24 | Maus anti-CA125 Cancer Antigen | CA125 | US biologischen | C0050-01D |
| A25 | Maus anti-CA19-9 Cancer Antigen | CA19-9 | US biologischen | C0075-18 |
| A26 | Maus Anti-Lewis-x monoklonalen Antikörper | Lewis-X | Calbiochem | 434631 |
| Bio | Biotinyliertes BSA (positive Kontrolle) | Bio | Hausgemachte | N / A |
Tabelle 1. Liste der Lektine und Antikörper in diesem Protokoll verwendet.
| Name des Reagenzes s / Ausrüstungen | Firma | Katalog-Nummer |
| Berührungslose Microarrayer | BioDot Inc | sciFLEXARRAYER |
| 384 Mikroplatte | Fischer | 14-230-243 |
| FoodSaver | FoodSaver | V3835 |
| Ultradünne Nitrocellulose Coate Microarray-Slides | Gentel | PATH |
| Slide Imprinter (optional) | Das Gel Unternehmen | WSP60-1 |
| Shaker | Fischer | 15-453-211 |
| Zentrifugieren | Eppendorf | 5804 000.013 |
| Schieben Waschbecken / Glaszylinder, Schale with Wechselrahmen | Fischer | 08-812 |
| Slide Inkubationskammer / Objektträger-Box | Fischer | 03-448-5 |
| Brij 35, 30 w / v% igen Lösung in Wasser | Acros Organics | AC32958-0025 |
| Tween-20 | Fischer | P337-100 |
| Natriumperiodat (NaIO 4) | Sigma | 311448 |
| L-Glutaminsäure-γ-hydrazid | Sigma | G-7257 |
| Natriumacetat wasserfrei (CH 3 COONa) | Sigma | S2889 |
| Rinderserumalbumin (BSA) | Lampire Biologische Labs | 7500804 |
| Phosphatgepufferte Kochsalzlösung (PBS) (10X) | Scientific Denville | CP4390-48 |
| DyLight 549 konjugierten NeutrAvidin | Thermo | 22837 |
| Protease Inhibitor Cocktail Tablets | Roche | 4693159001 |
| ChromPure Human-IgG, Fc-Fragment | Jackson Immunoresearch | 009-000-008 |
| ChromPure Human-IgG, ganze Molekül | Jackson Immunoresearch | 009-000-003 |
| ChromPure Maus-IgG, ganze Molekül | Jackson Immunoresearch | 015-000-003 |
| ChromPure Mouse IgG, Fc-Fragment | Jackson Immunoresearch | 015-000-008 |
| ChromPure Rabbit IgG, ganze Molekül | Jackson Immunoresearch | 011-000-003 |
| ChromPure Donkey IgG, ganze Molekül | Jackson Immunoresearch | 017-000-003 |
| Microarray Scanner | Tecan | LS Reloaded |
Tabelle 2. Listevon Geräten und Reagenzien in diesem Protokoll verwendet.

Schema 1 ein Schema, das Lektin Antikörper Microarray-Basis Glykan Biomarkern Prozess 1 (Schritt 2 bis 4): Blockieren Sie die Antikörper mit der Microarray-Blocker (Glu-hydrazid) und BSA; 2 (Schritt 5):.. Gelten Serumproben und erfassen spezifische Glykoproteine mit spezifischen Antikörpern; 3 (Schritt 6): gelten biotinylierten Lektin (s); 4 (Schritt 7): Probe der biotinylierte AAL DyLight mit 549 beschriftet NeutrAvidin für Microarray-Bildgebung.
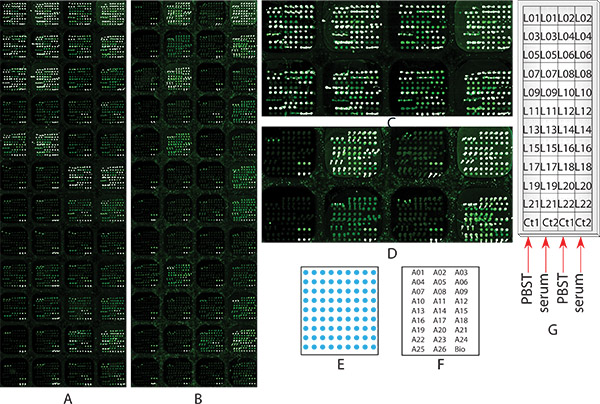
Abbildung 1. Microarray-Bilder der Probe Experiment 1 Glykosylierung Profilierung von mehreren Serumglycoproteinen in HCC Patienten Serumprobe mit ChemicVerbündeter blockiert Antikörper Microarray mit mehreren Lectin Detektion. Zwei identische Microarray Slides, (A) keine chemisch blockiert, oder (B) chemisch blockiert, wie in Schritt 2 beschrieben, beide gingen durch alle Schritte 2 bis 9 für die Glykosylierung Profiling, sowie Vergleich Zweck. (A) und (B) werden die Microarray-Bildern bei Schritt 8 in einer Auflösung von 10 Mikrometern gescannt. (D) die Zoom-Bild der ersten beiden Zeilen des nicht chemisch blockiert Mikroarray Folie (B));, (C) die Zoom-Bild der ersten zwei Zeilen der keine chemisch Mikroarray Folie (A) blockiert (E) Das Diagramm des Antikörpers Anordnung innerhalb jedes Sub-Arrays; (F)-Array-Karten: Die Lage der einzelnen Antikörper innerhalb der Subarray stellt jedes Antikörper-Name 3 Spots, (g) Serumprobe und Lektin Lage: Ein Diagramm zeigt, welche Subarray jeder Serumprobe und Lectin wurde auf aufgebracht.

Abbildung 2. Microarray-Bildern von derProbe Experiment 2 Bildschirm für veränderte Fucosylierung auf spezifische Serum-Glykoproteine als Biomarker dass diskriminieren Leberzirrhose und HCC-Patienten. Die Microarray-Assay wurde wie im Sample-Experiment 2 beschrieben durchgeführt. (A) Die gesamte Folie Bild des Mikroarrays Folie aus Schritt 8, (B) die Darstellung der Antikörper-Anordnung innerhalb jedes Sub-Arrays, (C)-Array-Karten: Die Lage der einzelnen Antikörper innerhalb der Subarray stellt jedes Antikörper-Name 3 Spots; (D) ein Zoom-in-Bild einer Untermatrix, die mit Serumprobe inkubiert wurden, (E) ein Zoom-in-Bild einer Untermatrix, die mit PBS-Kontrolle inkubiert wurden.

Abbildung 3. Glycan Profiling-Ergebnisse von Probe-Experiment ein. Jedes Balkendiagramm darstellen Lectin Profil (oder Glykan Profile) von einem der 20 getesteten Proteins. Insgesamt 22 verschiedene Lektine wurden verwendet, um th zu analysierene Glykan Profil jedes Protein.
Diskussion
1. Zielprotein und Fängerantikörper Auswahl
Vor der Antikörper-Mikroarray-Test werden einige Reagenzien und Materialien benötigt, um in Betracht gezogen werden und vorbereitet. Um einen Antikörper Microarray-Profiling für Glykan oder Glykan Biomarker-Screening, ein Gremium von spezifischen Antikörpern gegen Glykoprotein Kandidaten entwerfen sollte nach der Literatur oder von früheren Ergebnissen festgestellt werden. Diese Antikörper wurden in der Regel von verschiedenen Anbietern wie R & D Systems usw. IgGs gekauft werden bevorzugt Fänger-Antikörper, da unsere bisherigen Tests haben gezeigt, dass einige IgM und IgE kann vollständig verlieren ihre Antigen Bindungsaffinitäten nach der chemischen Modifizierung.
2. Entwurf und Herstellung von Antikörper-Mikroarray
Herstellung des Antikörpers Microarray ist ein optionaler Schritt, die professionelle und teure Microarrayer und gut ausgebildetes Personal für den Betrieb benötigt. Allerdings kundenspezifische Antikörper-Mikroarray Herstellertur leicht von einem Dienstleister wie Serome Biosciences Inc. Für ein robustes Ergebnis durchgeführt werden, empfehlen wir eine berührungslose Druckverfahren, wie der Scienion sciFLEXARRAYER Ultra Low Volume berührungslose Microarrayer, die in unserer Antikörper Microarray-Herstellung verwendet wurde. Hohe Bindungskapazität Microarray Slides, wie PATH (Gentel Bio Inc. WI) oder Slide H (Schott, PA).
3. Auswahl und Vorbereitung Glycan bindende Proteine (-Euros) für die Glykosylierung Profiling
Ziel-Euros, dass verschiedene Mono-oder Oligosaccharide sind in der Literatur und über eine Suchmaschine durch den Haab Labor 29 entwickelt und gepflegt am Translational Genomics Institute gefunden werden So wählen Sie Gbps mit hoher Spezifität und Affinität zudie gezielte Glykan Epitope, wählen Sie das Motiv (Epitop) aus dem Dropdown-Menü, und klicken Sie auf "Suchen". Die-Euros spezifisch für dieses Motivs (Epitop) werden nach ihrer logP Wert von hohen zu niedrigen Reihenfolge aufgeführt werden. Höhere logP deutet auf eine stärkere Affinität und Spezifität an die Glykan Motiv / Epitop. Aufgrund der inhärenten nicht-spezifische Bindung Problem von Lectin, ist dieses Verfahren immer noch nicht so optimal wie der auf Antikörpern basierende Assay. Daher empfehlen wir dringend, dass die Anti-Glykan Antikörper verwendet werden, wie z. B. Anti-Lewis-x-oder Anti-Sialyl-Lewis-A-Antikörpern, wenn sie verfügbar sind. Verwendung von mehreren Gbps für Erkennung ist eine weitere Strategie zum Abgleich verschiedener Profile und verbindliche, verlässliche Daten zu erhalten bindend.
4. Datenanalyse
Da diese Methode wird verwendet, um nativen Serum-Proteine erkennen können Protein-Protein-Komplexe erfasst und erkannt werden. Western-Blot oder Massenspektrometrie ist eine gute Methoden, um die Microarray-Daten zu validieren. Inzwischen, Nachweis von Protein-Spiegel unter Verwendung des gleichen Microarray ist eine weitere Methode, um die Details der Glykosylierung Veränderung des Proteins, z. B. ob die Änderungen waren aufgrund des gesamten Proteins zu ändern oder nur, dass die Glykosylierung auf jeder Ebene der Proteine erhöht lernen.
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Diese Arbeit wurde durch das Institut für Hepatitis-Virus und Forschung gefördert.
Materialien
| Name | Company | Catalog Number | Comments | |
| Identifikation | Name des Reagenzes | Abkürzung | Firma | Katalog # |
| L1 | Biotinyliertes Concanavalin A | ConA | Vector Laboratories | BK-1000 |
| L2 | Biotinyliertes Sambucus nigra-Lectin | SNA | Vector Laboratories | B-1305 |
| L3 | Biotinyliertes Lens culinaris Agglutinin | LCA | Vector Laboratories | BK-2000 |
| L4 | Biotinylierte Ricinus communis-Agglutinin I | RCA | Vector Laboratories | BK-1000 |
| L5 | Biotinyliertes Aleuria aurantia-Lectin | AAL | Vector Laboratories | B-1395 |
| L6 | Biotinyliertes Erythrina cristagalli Lektin | ECL | Vector Laboratories | BK-3000 |
| L7 | Biotinyliertes Griffonia (Bandeiraea) simplicifolia Lektin II | GSL II | Vector Laboratories | BK-3000 |
| L8 | Biotinyliertes Weizenkeim-Agglutinin | WGA | Vector Laboratories | BK-1000 |
| L9 | Biotinyliertes Phaseolus vulgaris Erythroagglutinin | PHA-E | Vector Laboratories | BK-2000 |
| L10 | Biotinyliertes Phaseolus vulgaris Leucoagglutinin | PHA-L | Vector Laboratories | BK-2000 |
| L11 | Biotinyliertes Erdnußagglutinin | PNA | Vector Laboratories | BK-1000 |
| L12 | Biotinylierte Pisum sativum Agglutinin | PSA | Vector Laboratories | BK-2000 |
| L13 | Biotinyliertes Dolichos biflorus Agglutinin | DBA | Vector Laboratories | BK-1000 |
| L14 | Biotinyliertes Datura Stramonium Lektin | DSL | Vector Laboratories | BK-3000 |
| L15 | Biotinyliertes Sophora Japonica Agglutinin | SJA | Vector Laboratories | BK-2000 |
| L16 | Biotinyliertes Sojabohnen Agglutinin | SBA | Vector Laboratories | BK-1000 |
| L17 | Biotinyliertes Solanum tuberosum (Kartoffel) Lektin | STL | Vector Laboratories | BK-3000 |
| L18 | Biotinyliertes Griffonia (Bandeiraea) simplicifolia Lektin I | GSL ich | Vector Laboratories | BK-2000 |
| L19 | Biotinylierte Vicia villosa-Lectin | VVL | Vector Laboratories | BK-2000 |
| L20 | Biotinyliertes Lycopersicon esculentum (Tomate) Lektin | UEG | Vector Laboratories | BK-3000 |
| L21 | Biotinyliertes Ulex europaeus Agglutinin I | UEA I | Vector Laboratories | BK-1000 |
| L22 | Biotinyliertes Jacalin | Jacalin | Vector Laboratories | BK-3000 |
| A1 | Ziegen-F (ab ') 2 Fragment Anti-Human-IgM, Antikörper Fc5μ | IgM | Jackson Immuno Research | 109-006-129 |
| A2 | Esel F (ab ') 2 Frag Anti-Human-IgG (H + L) einkörper- | AB1 | Jackson Immuno Research | 709-006-149 |
| A3 | Maus Anti-Human-IgG F (ab ') 2-Antikörpers | AB3 | Jackson Immuno Research | 209-005-097 |
| A4 | Anti-Human-Alpha-2-Makroglobulin polyklonalen Antikörper | A2M | GeneTex | GTX62924 |
| A5 | Kaninchen anti-Human-alpha-1-Antitrypsin polyklonaler Antikörper | A1AT | Lee Biosiences | CA1T-80A |
| A6 | Maus Anti-Human-alpha-1-Antitrypsin monoklonalen Antikörper | A1AT | Sigma Aldrich | SAB4200198 |
| A7 | Kaninchen anti-Human-alpha-1-Antitrypsin polyklonaler Antikörper | HANDELN | NeoMarkers | RB-367-A1 |
| A8 | Kaninchen anti-humanAlpha-1-Antichymotrypsin polyklonaler Antikörper | HANDELN | Fisher Scientific | RB9213R7 |
| A9 | Maus Anti-Human-Transferrin monoklonalen Antikörper | Transferrin | GeneTex | GTX101035 |
| A10 | Kaninchen anti-Human-Transferrin polyklonaler Antikörper | Transferrin | GeneTex | GTX77130 |
| A11 | Anti-Human-Apolipoprotein J polyklonaler Antikörper | ApoJ | Abcam | ab7610 |
| A12 | Maus Anti-Human-GP73 monoklonalen Antikörper | GP73 | Abbott | 14H4-23 |
| A13 | Maus Anti-Human-GP73 monoklonalen Antikörper | GP73 | Santa Cruz Biotechnology INC | sc-101275 |
| A14 | Kaninchen anti-human alpha-1 fetoprotein polyklonaler Antikörper | AFP | Genway | GWB-41C966 |
| A15 | Maus Anti-Human-alpha-1 Fetoprotein monoklonalen Antikörper | AFP | Fitzgerald | 10-A05A |
| A16 | Maus Anti-Human-Hämopexin monoklonalen Antikörper | Hämopexin | Assaypro | 60190-05011 |
| A17 | Maus Anti-Human-Glypican-3 (1G12) monoklonaler Antikörper | GPL3 | Santa Cruz Bio | sc-65443 |
| A18 | Maus Anti-Human-Kininogen (LMW) monoklonaler Antikörper | Kininogen | Assaypro | 20333-05011 |
| A19 | Kaninchen anti-Human-MMP-21 monoklonalen Antikörpers | MMP21 | Epitomic | 1955-1 |
| A20 | Maus Anti-Human-CEACAM-1-monoklonalen Antiköry | CEACAM | R & D Systems | MAB1180 |
| A21 | Ratten-Anti-Human-DPPIV/CD26 monoklonalen Antikörper | DPPIV | R & D Systems | MAB22441 |
| A22 | Maus Anti-Human-PIVKA-II-monoklonalen Antikörpers | PIVICA | Kristall-chem | 8040 |
| A23 | Maus anti-carcinoembryonales Antigen | CEA | US biologischen | C1300 |
| A24 | Maus anti-CA125 Cancer Antigen | CA125 | US biologischen | C0050-01D |
| A25 | Maus anti-CA19-9 Cancer Antigen | CA19-9 | US biologischen | C0075-18 |
| A26 | Maus Anti-Lewis-x monoklonalen Antikörper | Lewis-X | Calbiochem | 434631 |
| Bio | Biotinyliertes BSA (positive Kontrolle) | Bio | Hausgemachte | N / A |
Tabelle 1. Liste der Lektine und Antikörper in diesem Protokoll verwendet.
| Name des Reagenzes s / Ausrüstungen | Firma | Katalog-Nummer |
| Berührungslose Microarrayer | BioDot Inc | sciFLEXARRAYER |
| 384 Mikroplatte | Fischer | 14-230-243 |
| FoodSaver | FoodSaver | V3835 |
| Ultradünne Nitrocellulose Coate Microarray-Slides | Gentel | PATH |
| Slide Imprinter (optional) | Das Gel Unternehmen | WSP60-1 |
| Shaker | Fischer | 15-453-211 |
| Zentrifugieren | Eppendorf | 5804 000.013 |
| Slide Waschbecken / Glaszylinder, Schale mit Wechselrahmen | Fischer | 08-812 |
| Slide Inkubationskammer / Objektträger-Box | Fischer | 03-448-5 |
| Brij 35, 30 w / v% igen Lösung in Wasser | Acros Organics | AC32958-0025 |
| Tween-20 | Fischer | P337-100 |
| Natriumperiodat (NaIO 4) | Sigma | 311448 |
| L-Glutaminsäure-γ-hydrazid | Sigma | G-7257 |
| Natriumacetat wasserfrei (CH 3 COONa) | Sigma | S2889 |
| Rinderserumalbumin (BSA) | Lampire Biologische Labs | 7500804 |
| Phosphatgepufferte Kochsalzlösung (PBS) (10X) | Scientific Denville | CP4390-48 |
| DyLight 549 konjugierten NeutrAvidin | Thermo | 22837 |
| Protease Inhibitor Cocktail Tablets | Roche | 4693159001 |
| ChromPure Human-IgG, Fc-Fragment | Jackson Immunoresearch | 009-000-008 |
| ChromPure Human-IgG, ganze Molekül | Jackson Immunoresearch | 009-000-003 |
| ChromPure Maus-IgG, ganze Molekül | Jackson Immunoresearch | 015-000-003 |
| ChromPure Mouse IgG, Fc-Fragment | Jackson Immunoresearch | 015-000-008 |
| ChromPure Rabbit IgG, ganze Molekül | Jackson Immunoresearch | 011-000-003 |
| ChromPure Donkey IgG, ganze Molekül | Jackson Immunoresearch | 017-000-003 |
| Microarray Scanner | Tecan | LS Reloaded |
Tabelle 2. Liste der Geräte und Reagenzien in diesem Protokoll verwendet.
Referenzen
- Fang, M. The ER UDPase ENTPD5 promotes protein N-glycosylation, the Warburg effect, and proliferation in the PTEN pathway. Cell. 143, 711-724 (2010).
- Marino, K., Bones, J., Kattla, J. J., Rudd, P. M. A systematic approach to protein glycosylation analysis: a path through the maze. Nat. Chem. Biol. 6, 713-723 (2010).
- Shental-Bechor, D., Levy, Y. Effect of glycosylation on protein folding: a close look at thermodynamic stabilization. Proc. Natl. Acad. Sci. U. S. A. 105, 8256-8261 (2008).
- Hossler, P., Khattak, S. F., Li, Z. J. Optimal and consistent protein glycosylation in mammalian cell culture. Glycobiology. 19, 936-949 (2009).
- Nothaft, H., Szymanski, C. M. Protein glycosylation in bacteria: sweeter than ever. Nat. Rev. Microbiol. 8, 765-778 (2011).
- Sola, R. J., Griebenow, K. Effects of glycosylation on the stability of protein pharmaceuticals. J. Pharm. Sci. 98, 1223-1245 (2009).
- Li, C., Lubman, D. M. Analysis of serum protein glycosylation with antibody-lectin microarray for high-throughput biomarker screening. Methods Mol. Biol. 723, 15-28 (2011).
- Dwek, M. V., Jenks, A., Leathem, A. J. A sensitive assay to measure biomarker glycosylation demonstrates increased fucosylation of prostate specific antigen (PSA) in patients with prostate cancer compared with benign prostatic hyperplasia. Clin. Chim. Acta. 411, 1935-1939 (2010).
- Drake, P. M. Sweetening the pot: adding glycosylation to the biomarker discovery equation. Clin. Chem. 56, 223-236 (2010).
- Kim, Y. -. P., Park, S., Oh, E., Oh, Y. -. H., Kim, H. -. S. On-chip detection of protein glycosylation based on energy transfer between nanoparticles. Biosensors & Bioelectronics. 24, 1189-1194 (2009).
- Boland, M., Rudd, P. M. Disease related glycosylation changes and biomarker discovery: challenges and possibilities in an emerging field. Editorial. Dis. Markers. 25, 189-192 (2008).
- Norton, P. A. N-linked glycosylation of the liver cancer biomarker GP73. J. Cell Biochem. 104, 136-149 (2008).
- Nakagawa, T. Glycomic analysis of alpha-fetoprotein L3 in hepatoma cell lines and hepatocellular carcinoma patients. J. Proteome Res. 7, 2222-2233 (2008).
- Durazo, F. A. Des-gamma-carboxyprothrombin, alpha-fetoprotein and AFP-L3 in patients with chronic hepatitis, cirrhosis and hepatocellular carcinoma. J. Gastroenterol Hepatol. 23, 1541-1548 (2008).
- Kobayashi, M. Fucosylated fraction of alpha-fetoprotein, L3, as a useful prognostic factor in patients with hepatocellular carcinoma with special reference to low concentrations of serum alpha-fetoprotein. Hepatol. Res. 37, 914-922 (2007).
- Maisey, N. R. CA19-9 as a prognostic factor in inoperable pancreatic cancer: the implication for clinical trials. Br. J. Cancer. 93, 740-743 (2005).
- Talar-Wojnarowska, R. Clinical value of serum neopterin, tissue polypeptide-specific antigen and CA19-9 levels in differential diagnosis between pancreatic cancer and chronic pancreatitis. Pancreatology. 10, 689-694 (2010).
- Chen, S. Multiplexed analysis of glycan variation on native proteins captured by antibody microarrays. Nat. Methods. 4, 437-444 (2007).
- Shao, C. Antibody microarray analysis of serum glycans in esophageal spuamous cell carcinoma cases and controls. Proteomics Clinical Applications. 3, 923-931 (2009).
- Chen, S., Haab, B. B. Analysis of glycans on serum proteins using antibody microarrays. Methods Mol. Biol. 520, 39-58 (2009).
- Yue, T. The Prevalence and Nature of Glycan Alterations on Specific Proteins in Pancreatic Cancer Patients Revealed Using Antibody-Lectin Sandwich Arrays. Molecular & Cellular Proteomics. 8, 1697-1707 (2009).
- Wolf-Yadlin, A., Sevecka, M., MacBeath, G. Dissecting protein function and signaling using protein microarrays. Current Opinion in Chemical Biology. 13, 398-405 (2009).
- Richard, E. Proteomics as Applied to Inherited Metabolic Diseases. Current Proteomics. 6, 140-153 (2009).
- Nolen, B., Winans, M., Marrangoni, A., Lokshin, A. Aberrant tumor-associated antigen autoantibody profiles in healthy controls detected by multiplex bead-based immunoassay. Journal of Immunological Methods. 344, 116-120 (2009).
- Kuno, A. Focused Differential Glycan Analysis with the Platform Antibody-assisted Lectin Profiling for Glycan-related Biomarker Verification. Molecular & Cellular Proteomics. 8, 99-108 (2009).
- Hsu, K. -. L., Mahal, L. K. Sweet tasting chips: microarray-based analysis of glycans. Current Opinion in Chemical Biology. 13, 427-432 (2009).
- Borrebaeck, C. A. K., Wingren, C. High-throughput proteomics using antibody microarrays: an update. Expert Review of Molecular Diagnostics. 7, 673-686 (2007).
- Sanchez-Carbayo, M. Antibody array-based technologies for cancer protein profiling and functional proteomic analyses using serum and tissue specimens. Tumor Biology. 31, 103-112 (2010).
- Porter, A. A motif-based analysis of glycan array data to determine the specificities of glycan-binding proteins. Glycobiology. 20, 369-380 (2010).
- Maupin, K. A. Glycogene Expression Alterations Associated with Pancreatic Cancer Epithelial-Mesenchymal Transition in Complementary Model Systems. Plos One. 5, (2010).
- Sevecka, M., Wolf-Yadlin, A., MacBeath, G. Lysate Microarrays Enable High-throughput, Quantitative Investigations of Cellular Signaling. Molecular & Cellular Proteomics. 10, (2011).
- Wang, M. Novel fucosylated biomarkers for the early detection of hepatocellular carcinoma. Cancer Epidemiol. Biomarkers Prev. 18, 1914-1921 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten