Method Article
Whole mont In Situ de E8.5 à E11.5 embryons de souris
Dans cet article
Résumé
Ce support entière In situ décrit les étapes essentielles qui assurent des résultats reproductibles de haute qualité pour les études d'expression génique dans des embryons de jour E8.5-E11.5 vieille souris.
Résumé
Monter toute l'hybridation in situ est une approche très instructive pour définir les profils d'expression génique dans des embryons. L'hybridation in situ dans les procédures sont longues et techniquement exigeant avec plusieurs étapes importantes qui contribuent collectivement à la qualité du résultat final. Ce protocole décrit en détail plusieurs étapes clés de contrôle de qualité pour l'étiquetage de sonde d'optimisation et de performance. Globalement, notre protocole fournit une description détaillée des étapes critiques nécessaires pour obtenir des résultats reproductibles de grande qualité. Premièrement, nous décrivons la génération de digoxygénine (DIG) des sondes ARN marqués par transcription in vitro de matrices d'ADN générés par PCR. Nous décrivons trois critiques des tests de contrôle qualité pour déterminer le montant, l'intégrité et l'activité spécifique de la DIG sondes marquées. Ces étapes sont importantes pour générer une sonde d'une sensibilité suffisante pour détecter des ARNm endogène dans un embryon de souris ensemble. En outre, nous décrivons les méthodes de fixation et le stockage d'embryons jour E8.5-E11.5 vieille souris pour l'hybridation in situ. Ensuite, nous décrivons des méthodes détaillées pour limiter la protéinase K digestion des embryons réhydratés suivie par les détails des conditions d'hybridation, lavages post-hybridation et le traitement RNase pour enlever hybridation de sonde non spécifique. Un anticorps AP-conjugué est utilisé pour visualiser la sonde marquée et de révéler le profil d'expression de la transcription endogène. Résultats représentatifs sont présentés à partir d'expériences réussies et les expériences typiques sous-optimale.
Protocole
1. La génération intermédiaire ribosonde transcription in vitro
- Préparation des produits de PCR dans des modèles de transcription in vitro.
- Conception des amorces PCR avec des séquences promotrices de la transcription dans le phage leurs extrémités 5 '.
Remarque: La séquence du promoteur ajouté à l'extrémité 5 'de l'amorce sens brin PCR sera utilisée pour transcrire la sonde sens, et la séquence du promoteur ajouté à l'extrémité 5' de l'amorce antisens PCR sera utilisée pour synthétiser la sonde anti-sens 1-3. Nous n'avons détecté aucune différence dans l'efficacité de transcription entre les modèles qui ne contiennent que les séquences promoteur du core vs modèles qui contiennent le promoteur du core plus 5 supplémentaires "séquences. Cette méthode a été utilisée avec le T3, T7 et SP6 promoteurs de phages.
Les séquences qui sont très conservés entre les membres de la famille des gènes ou des séquences hautement répétitives doivent être évités car ils peuvent entraîner une hybridation non spécifique et d'augmenter ainsi la coloration de fond. Sondes à haute teneur en GC aura limitée digoxigénine rUTP constitution et des séquences ADN matrice avec des pistes de résidus T va limiter l'incorporation de DIG-UTP dû à une interférence stérique entre les molécules de digoxigénine. La longueur de sonde peut varier de 300 pb à 1kb. Mais tant que les critères ci-dessus sont remplies, les sondes ont généralement plus élevé des activités spécifiques. - Le modèle pour la PCR peut être un clone plasmidique, clone génomique ou d'ADN génomique. Typiquement nous achetons toute la longueur des clones EST (par exemple auprès de l'ATCC ou Open Biosystems) à utiliser comme modèles PCR. Amplifier la matrice d'ADN en utilisant les procédures standard de PCR pour produire un produit de PCR contenant les séquences du promoteur. Pour faire des modèles de sondes suffisamment pour supporter plusieurs réactions de synthèse de sonde nous avons généralement mis en place 8 x 50 pl PCR. Les réactions sont mises en commun pour la prochaine étape. En général, une PCR quelques grands volumes ou des réactions plus petits (comme les réactions que nous utilisons 50 pl) sont suffisantes pour générer assez de template pour plusieurs réactions de synthèse de la sonde.
- Pour éliminer les protéines contaminantes, notamment des traces de RNase dans les préparations plasmidiques, de digérer 100 pi de chaque réaction de PCR avec 1 microlitre de 20mg/ml protéinase K à 55 ° C pendant au moins 30 minutes. Tous les réactifs et autres objets utilisés après cette digestion protéinase K doit être libre et RNase spécifiquement dédié au travail d'ARN.
- Pendant la digestion protéinase K, d'exécuter un échantillon de la réaction PCR sur un gel de vérifier l'intégrité du produit de PCR. Nous utilisons des gels d'acrylamide pour résoudre mini petits produits de PCR (moins de 600 à 700 pb) et des gels d'agarose pour résoudre les grands produits de PCR (> 600 à 700 pb) pour l'analyse de contrôle de la qualité. Si vous voyez plusieurs bandes sur le gel, nous vous conseillons d'optimiser la réaction de PCR afin que vous obteniez un seul produit PCR de la taille correcte.
- Bien extraire la protéinase K digérés réaction PCR avec un volume égal d'un phénol / chloroforme (1:1), suivie par une extraction avec un volume égal de chloroforme. Mélanger à chaque étape par le tube vortex pendant au moins 30 secondes. Ces étapes doivent être effectuées dans une hotte.
- Précipiter le produit PCR purifié en ajoutant 0,1 volume d'acétate de sodium 3M et 2 volumes d'éthanol 100%. Laissez-les dans -20 ° C pendant au moins 30 minutes.
- Spin pendant 5 minutes à 13000 rpm dans une microcentrifugeuse, enlever le surnageant et laver avec de l'éthanol à 70%. Autoriser le culot d'ADN de sécher complètement.
- Resuspendre l'ADN dans le volume approprié (par exemple 50 pl) de 1xTE. Mesurer la concentration d'ADN en utilisant un fluorimètre.
- Déterminer la séquence nucléotidique de la préparation de chaque modèle PCR utilisant des amorces qui correspondent aux séquences du promoteur phage ou d'une séquence interne au sein de la sonde. Ceci est une étape cruciale de la qualité de contrôle qui garantit que le modèle de sonde est de l'ordre correct.
- L'ADN est prêt à servir de modèle pour la transcription in vitro. Magasin de la matrice d'ADN à 4 ° C.
- Conception des amorces PCR avec des séquences promotrices de la transcription dans le phage leurs extrémités 5 '.
- transcription in vitro en utilisant le produit de PCR comme un modèle.
- Mettre en place la réaction une transcription de l'ARN dans un volume final de 50 pl. La réaction de transcription comprend 500 - 1000 ng d'ADN matrice, 5uL de tampon de transcription 10X (avec la TNT 100mm), 10 ul 2,5 mM creusez-NTP mélanger, 3μl (50 ~ 90 unités) de l'ARN polymérase, 1 microlitre Alpha-32 P CTP (jusqu'à à 6 mois) et le reste du volume est constituée par adjonction d'eau diéthylpyrocarbonate traités. La fouille 2,5 mM-NTP mix est généralement constituée comme un stock de travail contenant 10 ul 40μl 10mM CTP, GTP 10 ul 10mM, 10 ul 10 mM ATP, UTP 6.5μl 10mM, 3.5μl 10mM digoxigénine-11 UTP. En assemblant la réaction de transcription, assurez-vous que tous les composants de la réaction (à l'exception de l'enzyme) sont chauffés à température ambiante. Mélanger l'ADN et de l'eau en premier, puis ajouter le tampon de réaction, mélanger, puis ajouter les autres composantssinon la spermidine dans le tampon de transcription va précipiter l'ADN. Incuber pendant 2 heures à la température recommandée pour l'ARN polymérase utilisée.
Remarque: le P α-32 est ajouté à la réaction pour vous permettre de déterminer la proportion de la piscine à partir de nucléotides qui est incorporé dans la sonde ARN. Ceci est une mesure importante de l'efficacité de la réaction. Toutes les étapes ci-dessous suivent les procédures de traitement 32 p.
Une méthode alternative pour mesurer la quantité de sonde ARN synthétisé est d'utiliser un spectrophotomètre petit volume. Cela évite l'utilisation de 32 à étiqueter P (voir les notes à l'étape 1.3.1) - Pendant les dernières minutes d'incubation, préparer la colonne de centrifugation rapide (Roche) selon les instructions du fabricant.
- Après incubation, ajouter 1 microlitre de la RNase DNase I et incuber à 37 ° C pendant 10 min.
- Diluer à 100 ul avec 50 pl de tampon de dilution de réaction (20 mM Tris pH 7,5, SDS 1%, 20mm EDTA, 100 mM NaCl (RNase, faire un bouillon de 50ml et stocker à température ambiante)). Prenez 1 microlitre (habituellement dans des 4μl 1x TE) pour le comptage à scintillation des comtes de départ total et pour l'analyse de gel.
- Appliquer le reste de la réaction sur le centre du lit de colonne et centrifuger à 1100 xg pendant 4 min dans une centrifugeuse clinique de table haut. Après le spin votre sonde ARN est dans le tube collecteur.
- Ajouter 2 volumes d'éthanol à 100% dans la réaction éluées. Mélangez bien et de garder sur la glace pendant 5min (ou -20 ° C ou 30 min).
- Spin le tube dans une microcentrifugeuse au régime maximum pendant 5 minutes pour culotter la sonde ARN.
- Supprimer complètement le surnageant à la pipette et laisser le culot de l'air sec. Ne laissez pas le culot plus sec, car il sera très difficile de le dissoudre.
- Dissoudre la sonde ARN avec 50 ul - 100 pi ou 1xTE DEPC-H 2 O. Prenez un échantillon de 1 ul pour déterminer l'incorporation pour cent et pour une analyse sur un gel d'acrylamide dénaturant. Ajustez votre concentration de sonde à 100 ng / ul selon le rendement estimé.
- Sondes qui ont passé tous les contrôles de qualité les trois peuvent être ensuite stockés dans -20 ° C pendant 6 à 12 mois. En général, nous épuiser le stock de sonde dans les 12 mois. Il est très probable que les sondes peuvent être stockés pendant des périodes très étendu au-20C.
- Mettre en place la réaction une transcription de l'ARN dans un volume final de 50 pl. La réaction de transcription comprend 500 - 1000 ng d'ADN matrice, 5uL de tampon de transcription 10X (avec la TNT 100mm), 10 ul 2,5 mM creusez-NTP mélanger, 3μl (50 ~ 90 unités) de l'ARN polymérase, 1 microlitre Alpha-32 P CTP (jusqu'à à 6 mois) et le reste du volume est constituée par adjonction d'eau diéthylpyrocarbonate traités. La fouille 2,5 mM-NTP mix est généralement constituée comme un stock de travail contenant 10 ul 40μl 10mM CTP, GTP 10 ul 10mM, 10 ul 10 mM ATP, UTP 6.5μl 10mM, 3.5μl 10mM digoxigénine-11 UTP. En assemblant la réaction de transcription, assurez-vous que tous les composants de la réaction (à l'exception de l'enzyme) sont chauffés à température ambiante. Mélanger l'ADN et de l'eau en premier, puis ajouter le tampon de réaction, mélanger, puis ajouter les autres composantssinon la spermidine dans le tampon de transcription va précipiter l'ADN. Incuber pendant 2 heures à la température recommandée pour l'ARN polymérase utilisée.
- Évaluation de la qualité de la ribosonde - sonde de mesure de rendement.
- Pour estimer le rendement de sonde, divisez le compte récupéré après la colonne de centrifugation par les comtes de la réaction avant que la colonne spin (mesuré avec les échantillons prélevés dans le protocole de synthèse de la sonde). Pour cette réaction, l'incorporation 100% = 33 pg. Une réaction réussie donne habituellement un rendement 15-50%. Sondes avec l'incorporation de moins de 15% (rendement
- Évaluation de la qualité de la ribosonde - mesurer l'activité spécifique.
- Pour mesurer l'ampleur de la digoxigénine - UTP incorporation effectuer un essai à la tache. Ceci est un test de qualité essentielle de contrôle.
Ajuster la concentration de la sonde à 100 ng / ul. Spot 1 ul de dilutions en série (10 -2 à 10 -5) de la sonde sur un diamètre 82mm d'Hybond-N + (GE Healthcare) ou d'autres filtre en nylon équivalent. Inclure un échantillon d'ADN non étiquetés comme contrôle négatif. Crosslink le filtre humide immédiatement dans un réticulant UV à 125mJoules ou selon les instructions du fabricant pour le filtre en nylon que vous utilisez.
Toutes les étapes suivantes (1.4.2 - 1.4.6) sont effectués dans une boîte de Pétri sur une plate-forme à bascule à la température ambiante. Vous n'avez pas à vous soucier de la RNase après avoir repéré les dilutions de sonde sur le filtre - Bloquer le filtre pendant 30 minutes à température ambiante dans 10 ml de réactif de blocage de 1% (Roche) dans 1X TN (10X TN = tampon Tris 1 M pH 7,5, 1,5 M de NaCl)
- Lavez 1 X 15 min dans 10 ml de tampon 1X TN.
- Incuber avec le fragment Fab anti digoxigénine à une dilution de 1 / 5000 dans du tampon 1X TN pendant 30 minutes.
- Lavez 2 X 15 min dans 10 ml de tampon 1X TN.
- La réaction colorée est réalisée avec le substrat mauve BM (besoin d'environ 5 ml dans le plat). La couleur ne doit être visible sur l'endroit de la dilution 10 -4 en 30 minutes. À ce moment, arrêter la réaction de couleur par le lavage deux fois pendant 5 minutes chacune avec 5X TE. Si le signal n'est pas visible à l'endroit qui correspond à la dilution 10 -4 alors la sonde doit être jeté.
Remarque: Dans notre expérience, un manque d'activité spécifiques ne se trouve avec des sondes courtes. Augmenter la taille du modèle de sonde (jusqu'à 1kb) tout en respectant les autres critères de conception spécifiés dans ce protocole devrait augmenter l'activité spécifique de la sonde. Faible activité spécifique conduit à des niveaux faibles ou indétectables de signal.
- Pour mesurer l'ampleur de la digoxigénine - UTP incorporation effectuer un essai à la tache. Ceci est un test de qualité essentielle de contrôle.
- Test de l'intégrité de la sonde par électrophorèse sur un gel d'acrylamide dénaturant à 5%.
Ceci est un autre test important de la qualité de contrôle. Ce protocole suppose que la sonde a été marquée au 32 P lors de la réaction de synthèse (voir étape 1.2.1). Nous utilisons des gels d'acrylamide dénaturant pour fournir une très haute résolution, nous permettant de détecter facilement les sondes dégradés ou incomplets. Généralement, un 5% d'acrylamide dénaturant minigel (Urée 7.5g, l'acrylamide 1.9ml 40%, 1.9ml 2% de bis-acrylamide (ou utiliser un acrylamide 20h01: bis-acrylamide solutions), 1,5 ml de tampon 10X gel de MOPS (0,2 M morpholinopropanesulfonique acide, pH 7,0, 50 mM d'acétate de sodium 5 mM EDTA) et H 2 O pour un volume final de 15 ml) fonctionne très bien pour les sondes de la taille résoudre les gammes utilisées dans ce protocole.
Remarque: Comme alternative, TBE-urée gels préfabriqués peuvent être achetés auprès de divers fournisseurs. Suivez les instructions du fabricant pour faire fonctionner les gels préfabriqué.- Mélanger l'échantillon avec un volume égal de tampon de chargement de gel (Ambion) et bien mélanger.
- Chauffer à 95 ° C pendant 5 minutes pour dénaturer toute structure secondaire.
- Immédiatement refroidir sur glace avant le chargement sur le gel pour prévenir la ré-recuit.
- Exécutez l'avant et après les échantillons colonne ensemble à 200V jusqu'à ce que le colorant bleu court à la fin du gel. Le gel est exécuté en 1X tampon de gel MOPS.
- Envelopper le gel dans une pellicule plastique et l'exposer à des rayons X du film ou de phosphore imageur d'écran afin de détecter l'ARN radiomarqué. Si 32 à étiqueter P n'est pas utilisé vous pouvez teindre le gel avec un colorant fluorescent conventionnel pour détecter l'ARN
- Une synthèse réussie de sonde se traduira par une des sondes d'ARN qui migre comme une bande nette très proche de la partie supérieure du gel.
2. Collecte et stockage des embryons de souris
- Embryons de souris devraient être recueillis dans la glace froide PBST (PBS + 0,1% Tween 20, (diéthylpyrocarbonate (DEPC) traité)) et conservés sur la glace lors de la dissection.
- Les embryons sont traités dans quatre flacons à vis ml en verre plafonné. Plusieurs embryons peut être fixé dans un flacon. Comme beaucoup de 10 à 15 E9.5 ou 5 embryons E10.5 peut être fixé dans un flacon unique. En raison de l'augmentation de la taille de l'embryon, les embryons doivent être E11.5 hemisected avec une lame de rasoir avant la fixation de l'aide à l'accès de sonde lors de l'hybridation. Jusqu'à 3 hemisected embryons E11.5 peut être placé dans un flacon unique.
- Pour maximiser l'efficacité des étapes de ce protocole et à réduire les dommages à l'embryon, la fixation embryon et tous les lavages sont effectués dans des flacons remplis d'échantillon avec des solutions au niveau inférieur du cou flacon de sorte que seule une petite bulle d'air reste, sauf indication contraire spécifié. Pour toutes les étapes de lavage des flacons sont fixées sur leur côté sur une plate-forme à bascule.
- Correction pour les 6 heures pour une nuit à 4 ° C sur une table à bascule avec du paraformaldéhyde 4% (PFA) dans le PBST. Les flacons doivent être placés horizontalement et bercé sur une plateforme à bascule à 4 ° C.
- Après fixation, les embryons sont lavés deux fois pendant 5 minutes chacun dans PBST sur la glace et ensuite par étapes déshydratées à travers une série de méthanol / PBST (25% de méthanol dans du PBST, 50% de méthanol dans du PBST et du méthanol à 75% dans le PBST) dans le méthanol à 100% en remplaçant des solutions dans le flacon de verre à l'aide de pipettes Pasteur. Les embryons peuvent être conservés à-20C dans le méthanol à 100% pendant 8 mois et peut-être plus.
3. L'hybridation de la sonde marquée à la digoxigénine embryons de souris toute
- Crevaison coeurs et des chefs de E10.5 embryons et plus avec un couteau de microdissection tandis que les embryons sont encore dans le méthanol (si cela n'a pas été fait lors de la dissection). Cette étape simple permet les solutions de lavage pour entrer dans les vésicules du cerveau et les cavités cardiaques réduisant ainsi considérablement la coloration de fond.
- Réhydrater les embryons par des lavages successifs dans une série de méthanol / PBST (75% de méthanol dans du PBST, 50% de méthanol dans du PBST puis 25% de méthanol dans du PBST) pendant 5-10 minutes à chaque concentration de méthanol. Laver deux fois pendant 5 minutes chaque lavage en PBST 100%.
- Incuber dans PBST 04h01: 30% H 2 O 2pendant 1 heure sur la glace. Ensuite laver avec 3 changements 5 min chacune dans 1X PBST.
- Retirez le lavage PBST dernier et le remplacer par 1ml 10 ug / ml de protéinase K dans le PBST. Pendant la place protéinase K digestion des tubes à la verticale dans un rack dans un bain d'eau à 25 ° C.
Remarque: Les solutions de protéinase K perdre son activité au cours du temps en raison de l'auto-digestion. Pour assurer une activité maximale et constante de la solution de protéinase K doit être préparée immédiatement avant chaque utilisation. Pour chaque utilisation en font une petite quantité d'un stock 1mg/ml, puis le diluer. Le temps d'incubation doit être déterminée pour chaque lot de poudre de protéinase K et pour chaque âge embryon. Par exemple, pour les embryons de E9.5 un bon moment est habituellement 10-15 minutes à 25 ° C alors que pour E10.5 il est généralement 20-30 min à 25 ° C. Le temps d'incubation de protéinase K doivent également être mesurés avec précision au cours d'expériences pour permettre des comparaisons valables entre les flacons et entre les expériences. La température doit être soigneusement entretenus lors de la digestion d'augmenter la reproductibilité. Différences aussi petites que 1 ° C peut modifier le montant de la digestion entre les expériences. Il est fortement recommandé que les digestions protéinase K être fait dans un incubateur ou un bain-marie pour le contrôle de température précis. Cette étape est essentielle pour permettre à la sonde à pénétrer dans le tissu générant ainsi un signal fort. Cependant, la sur-digestion endommager les embryons et ils se désintègrent graduellement au cours des étapes restantes. - Arrêtez la digestion par la protéinase K laver deux fois pendant 5 minutes chaque lavage à température ambiante avec fraîchement préparé 2 mg / ml de glycine dans le PBST. Puis laver deux fois pendant 5 minutes chacun dans du PBST.
- Fixer les embryons digérés pendant 20 minutes à température ambiante dans 4% PFA / PBST, glutaraldéhyde à 0,2% (Polysciences). Ne pas sur-ou underfix. Lavez 3 fois pendant 5 minutes chacun dans du PBST.
- À ce stade, les embryons doivent être divisés en groupes pour l'hybridation. Retirez le PBST. Ajouter 1 ml de tampon d'hybridation (formamide ultrapur à 50% (Invitrogen), 5x SSC, pH 5, 1% SDS, 50 pg / ml d'héparine (Acros), 50 pg / ml d'ARN Torula (Sigma)) qui a été chauffé à 65 ° C (sans sonde). Autoriser les embryons de s'installer, puis retirez tout le tampon hybrization et la remplacer par une hybridation ml tampon frais réchauffé sans sonde.
- Prehybridize pendant 1 heure à 65 ° C dans un bain d'eau agité ou dans un four sur un plateau oscillant (par exemple Four le Shake Boekle 'n' Bake, voir tableau 1). Si vous incuber les flacons dans un bain d'eau être sûr de l'eau monte, mais pas plus, les sommets des flacons. La température d'hybridation peut être augmentée jusqu'à 70 ° C. L'hybridation températures supérieures à 65 ° C ne touchent pas l'intensité du signal ou de fond avec la plupart des sondes, nous avons testé. Les embryons deviennent translucides, collant et fragile dans les tampons formamide (tampon d'hybridation, par exemple) être prudent lors de leur manipulation.
- Remplacer le tampon de préhybridation avec le tampon d'hybridation contenant la sonde 0.4ml à une concentration de 0.25 à 1 ug / ml. Utiliser 0,5 pg / ml de sonde pour des embryons jusqu'à environ E8.5. Pour les plus vieux embryons, les sondes doivent être titrés de 1 à 0,1 pg / ml pour déterminer la concentration optimale pour un bon signal avec faible bruit de fond (généralement 0,25 à 0,5 pg / ml ou plus). Notez que l'hybridation des volumes plus importants peuvent être nécessaires pour les embryons plus grands. Hybrider une nuit à 65 ° C.
- Si les embryons ont été plus traitée par la protéinase K. ils apparaissent extrêmement transparent. Ils peuvent aussi coller aux parois de la fiole, ou s'effondrer. Les embryons qui apparaissent de cette façon doivent être jetés à ce point dans le protocole. Leur traitement ultérieur ne sera pas aboutir à des données interprétables.
- Remplacer le tampon d'hybridation avec la solution chauffée I (50% formamide, 5x SSC pH de 5, 1% SDS) à 70 ° C. Laver deux fois pendant 30 minutes chaque lavage pour E8.5 embryons, trois fois pendant 30 minutes par lavage pour E9.5 et plus.
- Laver une fois avec préchauffé solution à 50% I: solution à 50% II (0,5 M de NaCl, 10 mM Tris HCl pH 7,5, 0,1% Tween 20) à 70 ° C pendant 10 minutes.
- Laver trois fois pendant 5 minutes par lavage avec la solution II à température ambiante.
- Remplacer mémoire tampon avec la solution II contenant de la RNase A 100 ug / ml, la RNase T1 à 100 unités / ml. Incuber sur un rocker dans la salle chaude à 37 ° C à deux reprises pendant 30 minutes par lavage. Le lavage RNase est essentiel pour réduire de fond non spécifique résultant de sondes hybridées.
- Laver dans une solution III (50% de formamide, 2x SSC pH5) à 65 ° C pendant 30 minutes deux fois par chaque lavage pour E8.5, 3 fois pendant 30 minutes par lavage pour E9.5 et plus.
- Lavez 3 fois pendant 5 minutes par lavage avec 1X TBST (100 ml de TBS 10X contient 8g de NaCl, KCl 0,2 g, 12,5 ml 2M Tris HCl pH 7,6 dans de l'eau. Pour faire 1X 10X TBST diluer le SCT dans un volume approprié d'eau et Ajouter Tween concentration de 20 à 0,1% final)
- Sauf indication contraire, tous les lavages sont effectués dans des flacons d'échantillons avec des solutions rempli au niveau inférieur de lacol du flacon. Pour toutes les étapes de lavage, les flacons sont fixées sur leur côté sur une plate-forme à bascule.
4. La détection d'anticorps de l'digoxygénine sonde marquée
- Les embryons sont pré-bloqués dans 0,5 ml de 1X TBST + chaleur de 10% du sérum de mouton traitée. Puis les flacons à la verticale de roche à la température ambiante (~ 20 ° C) pendant au moins 2,5 heures.
- Remplacer la solution de blocage avec 1X TBST frais + 10% de sérum contenant un 1:2000 - 1:5000 dilution des fragments alcalines moutons conjugué à la phosphatase Fab anti-digoxigénine (soustraite par l'incubation avec de la poudre d'acétone embryon, voir ci-dessous). Utiliser des dilutions supérieures pour les embryons de E9.5 et plus, jusqu'à 1:5000 pour minimiser fond. Incuber à la verticale sur rocker à 4 ° C pendant la nuit.
Note: Nous avons constaté que l'incubation des fragments Fab anti-digoxigénine avec une poudre d'acétone embryon réduit les niveaux de bruit de fond dans ce protocole. Pour préparer la poudre d'acétone embryon d'homogénéiser commun des embryons de souris E12.5-14.5, dans un volume minimum de PBS. Ajouter 4 volumes de glace acétone, enrobé à froid et incuber sur glace pendant 30 minutes. Retirer le surnageant par centrifugation à 10000 xg pendant 10 minutes. Laver le culot avec de la glace acétone froide et de spin à nouveau. Fais le culot sortir et moudre en poudre sur une feuille de papier filtre et le laisser sécher à l'air. La poudre peut alors être stocké dans un tube étanche à 4 ° C pendant des années.
Pour la soustraction d'anticorps, l'anticorps doit être pré-absorbé à la poudre d'acétone embryons pour réduire la liaison non-spécifique pendant ou avant la pré-bloquant. Incuber anticorps à 1 / 100 avec une petite quantité de poudre de l'embryon (le montant n'est pas critique) dans 1xTBST/10% de sérum pendant au moins 1h à 4 ° C avec bascule. Enlevez la poudre en tournant dans une microcentrifugeuse pendant 10 minutes à 4 ° C. D'anticorps soustrait peut être conservé à 4 ° C pendant au moins 6 mois. - Laver trois fois 5 minutes chacune avec TBST 1X, puis 5-6 fois pendant 1 heure chacune à température ambiante pour éliminer excès d'anticorps. Si vous préférez, faire un lavage prolongé nuit à 4 ° C pour réduire de fond. Le lavage du jour au lendemain peut réduire considérablement les niveaux de fond augmentant ainsi le rapport signal sur bruit.
- Laver deux fois pendant 20 minutes par lavage avec le tampon de la phosphatase alcaline fraîchement préparé (100mm Tris, pH 9,5, 50 mM MgCl2, 100 mM NaCl, 0,1% Tween 20. Ajouter lévamisole 0.5mg/ml, un inhibiteur endogène AP, pour E9.5 et plus embryons).
- Remplacer le dernier lavage avec BM Violet AP substrat (Roche) préchauffée à la température ambiante.
- Laisser la réaction colorée à incuber à température ambiante, et de regarder pour un signal périodique à travers une loupe binoculaire. Pour les réactions de couleurs étendue (plusieurs heures pour la nuit) le flacon doit être protégé de la lumière. Signal pour les ARNm abondants devraient être visibles dans les 15 minutes. Réactions doit être arrêté lorsque vous êtes satisfait avec le signal, ou lorsque le fond commence à être un problème. Si la solution elle-même commence à tourner sombre, vous pouvez prolonger la réaction colorée en remplaçant la solution de substrat.
- Arrêtez la réaction en deux lavages de 5 minutes par lavage dans 1X PBST/5mM EDTA.
- Transfert à 4% de paraformaldéhyde / PBST de re-fixer, de plusieurs heures à une nuit. Ou d'embryons peuvent être conservés dans PBST paraformaldehyde/1X 4% pour les années ou peut-être immédiatement traités pour la coupe. Cette étape de fixation finale est crucial pour la préservation à long terme du modèle de coloration.
5. Inclusion en paraffine et coupes d'embryons tachés
- Avant les embryons sont fixés après la réaction de la couleur (étape 4.8) des photos de l'ensemble du montage embryons colorées sont prises lorsque le niveau de coloration est à l'intensité désirée. Après avoir documenté les profils d'expression dans les embryons entiers ils sont incubés dans le substrat BM mauve nuit à 24 heures à température ambiante jusqu'à l'embryon tout entier devient bleu foncé. Cette étape est de s'assurer que la coloration sera suffisamment sombre pour apparaître dans les coupes de tissus. S'il vous plaît noter que la couleur bleue de l'embryon est dû au dépôt d'une couche très mince de pigment sur toute la surface embryonnaire. Ce bruit de fond ne sera pas détectable dans le tissu d'intérêt, après la coupe.
- Laver les embryons colorés avec du PBS à trois reprises pour enlever le substrat en excès précipite et fixer la nuit dans PFA 4% à 4 ° C.
- Laver avec du PBS à trois reprises, puis déshydrater les embryons dans 100% de méthanol (comme décrit au point 2.4).
- Remplacer le méthanol dernier avec le xylène. Laver dans du xylène, 2 à 3 fois pendant 2 min à chaque lavage jusqu'à ce que les embryons deviennent claires. Ne pas trop se laver dans le xylène, car il fera le tissu très fragile, après il est intégré ce qui rend très difficile de récupérer des sections intactes. Surveiller visuellement chaque incubation xylène faire embryons que sont juste au point d'être clair et être très prudent pour ne pas permettre l'incubation de xylène pour s'étendre au-delà ce point.
- Transfert des embryons (E10.5 et plus) Dans une biopsie Sure-Tek cassettes (Fisherbrand), et incuber la cassette entière dans de la cire (Fisherbrand) pour 15 ~ 30 min à 65 ° C. Changement dans la cire fraîche pour un autre 15 ~ 30min puis changer de nouveau. Les petits embryons / troncs tissus, requièrent moins de temps d'incubation de la cire. E9.5 embryons peuvent être placés dans des moules intégration tissulaire de tous les changements de cire. Dans ces cas, vous pouvez enlever la cire du moule au lieu de transférer les embryons.
- Après trois changements de cire, le transfert des embryons dans les moules intégration tissulaire (Polysciences) et l'enrober avec de la cire fraîche. Soigneusement la position de l'embryon pour le plan désiré de sectionnement. Laissez la cire refroidir jusqu'à solidification.
- Section de la cire blocs en 8 ~ 14μm tranches utilisant des microtome (Leica RM2155 par exemple). Recueillir sections sur des lames de microscope (Fisherbrand).
- Laissez les diapositives à plat et sec sur un slidewarmer. Les diapositives peuvent être conservés à 4 ° C pendant des mois.
- Sections sèches sont déparaffinées par deux lavages de 5 min xylène et réhydratés dans l'eau par série méthanol / eau lave (100% de méthanol 2 min, 100% de méthanol 2 min, 90% méthanol / eau 2min, 70% de méthanol / eau 2min, eau déminéralisée 5 min).
- Colorez les coupes avec des armes nucléaires solution de coloration au rouge rapide (Sigma) pendant 1 minute. Pour générer le nucléaire solution rapide coloration rouge, diluez un stock d'armes nucléaires concentrées rouge rapide (0,1% de nucléaire rouges rapide dans le sulfate d'aluminium de 5% dans de l'eau, dissoudre par la chaleur et filtre à travers papier filtre Whatman) 1:5 dans une solution aqueuse de 5 sulfate d'aluminium%. Après coloration laver les lames à l'eau courante jusqu'à ce que l'eau soit claire. Le rouge nucléaire rapide agit comme une contre-coloration et révèle la structure du tissu global dans les sections. Sa couleur contraste bien avec la coloration bleu-violet qui marque la localisation de la sonde ARN. Nucléaire rouges rapide est une tache et saturer l'intensité de coloration est contrôlée par la concentration de la solution de coloration. Si l'intensité de la coloration nucléaire rouges rapide est insuffisante lorsque vous utilisez cette solution de coloration vous pouvez répéter cette étape avec un plus concentrée solution nucléaire rouges rapide.
- Préparer les lames pour les lamelles de montage par des lavages dans chacune des solutions dans les séries suivantes: 90% de méthanol / eau, suivie de 2 lavages dans du méthanol 100%, suivi par deux lavages en xylènes. Incuber la lame pendant 2 minutes dans chacun des lavages.
- Monter les préparations avec des Cytoseal 60 milieu de montage (Richard-Allan scientifique). S'il vous plaît noter que les milieux aqueux de montage va dissoudre le rouge nucléaire rapide tache.
Remarque: nous avons également utilisé une résine d'inclusion plastique (Immuno-Bed, Polysciences Inc) pour générer des sections d'embryons colorés avec d'excellents résultats 4.
6. Les résultats représentatifs:
La figure 1 illustre les résultats obtenus représentatifs en utilisant ce protocole. La figure 1A montre le profil d'expression des GATA3 dans des embryons de souris E10.5. Ce panneau illustre un bon résultat avec un signal élevé par rapport arrière-plan. Figure 1C, en revanche, montre un échec d'hybridation in situ pour les GATA3 ARNm qui a utilisé une sonde ARN partiellement dégradé. Une sonde de faible activité spécifique se traduira par des résultats similaires avec un signal faible à contraste de fond. Une comparaison des panneaux A et C illustre l'importance d'effectuer des évaluations de contrôle de qualité minutieuse de la qualité de la sonde pour assurer des résultats de haute qualité. Le résultat obtenu avec la sonde FOXG1 (figure 1B) illustre également un bon résultat avec une coloration spécifique dans le télencéphale avec faible bruit de fond dans d'autres parties du cerveau. Le cerveau et le cœur sont des sites communs de la coloration de fond provoqué par le piégeage de la sonde. Perforer le cerveau et le cœur avec une pince permet solutions de lavage dans l'embryon et la coloration de fond minimise (voir étape 3.1). La figure 1D illustre le type de fond bleu clair trouvés lorsque le cerveau n'est pas percé. Cela montre un résultat sous-optimal obtenu en utilisant une sonde Pax1. Pax1 n'est pas exprimé dans n'importe quelle partie du cerveau et de l'ensemble des taches observées dans le cerveau de cet embryon est due à fond non spécifique.
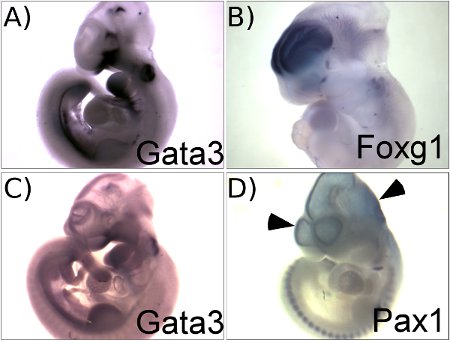
Figure 1. Les résultats représentatifs montrant des exemples de réussite (A, B) et des sous-optimale (C, D) les résultats. A. E10.5 embryons hybrides avec des GATA3 sonde. B. E11.5 embryons hybrides avec des FOXG1 sonde. C. E10. 5 embryons hybrides avec des GATA3 sonde qui est partiellement dégradée, montrant signal très faible bruit de fond élevé, mais grâce à l'embryon tout entier. D. E10.5 embryons hybrides avec des sondes Pax1 sans perforation de la tête, montrant une coloration de fond dans les ventricules (têtes de flèche).
Discussion
Les méthodes décrites dans ce protocole ont été adaptés à partir d'un certain nombre de sources différentes et optimisé pour le montage toute E8.5-E11.5 embryons de souris âgés jour. Méthodes de montage toute l'hybridation in situ des embryons de vertébrés est apparu au début des années 1990 2,5-12. Ce protocole a été adapté principalement à partir des méthodes développées pour les embryons de Xénope 7,8 ainsi que la souris 2,11. Dans notre protocole beaucoup d'accent est mis sur l'évaluation de qualité vigilant de la sonde. Une attention particulière à générer une haute qualité et haute activité spécifique de sonde ARN tel que déterminé par les premières étapes de ce protocole permettra d'accroître considérablement la qualité des données et économiser des quantités considérables de temps et d'effort dans le long terme. Notre protocole inclut également l'utilisation de modèles de PCR ADN générés pour la transcription d'ARN polymérase du phage. Cette approche est rapide et très évolutive. Il peut permettre la génération d'un grand nombre de sondes d'ARN pour les écrans à grande échelle en hybridation in situ 13-15.
Plusieurs étapes de ce protocole devrait être considérée lorsque le fond est élevé mais le signal est faible. Une considération est la sonde. Nous n'avons jamais eu de fond élevé avec des sondes d'ARN qui répondaient aux critères de conception précisés dans ce protocole et ce qui s'était passé les trois tests de contrôle qualité (rendement, l'activité spécifique et de longueur de la sonde). Un autre facteur qui peut affecter les niveaux de fond est la capacité de la sonde à pénétrer dans les tissus embryonnaires. Des précautions doivent être prises lors de la dissection d'enlever complètement les membranes extra-embryonnaires qui couvrent l'embryon. En outre, l'étape de digestion protéinase K est absolument essentiel pour les embryons de plus de E9.5. Plus protéinase K donne les temps de digestion inférieurs de fond et un signal plus fort, cependant, les temps de digestion excessive peut conduire à des dommages aux tissus accrue et peut entraîner la désintégration de l'embryon au cours de la procédure. Optimisation du temps de digestion pour chaque lot de protéinase K est nécessaire pour obtenir les meilleurs résultats. Une autre étape qui peut être optimisé pour obtenir le signal haute à des ratios de fond est l'incubation de RNase (étape 3.14). Notre protocole utilise un mélange de RNAse A et RNase T1. Les deux enzymes digèrent l'ARN simple brin, mais chaque enzyme a une spécificité de base différents. La combinaison des résultats des enzymes de digestion deux étendues d'ARN simple brin d'oligoribonucléotides petits. Cela permet à la plupart des sondes non hybridées spécifiquement pour être enlevée par les lavages post-hybridation résultant en une diminution importante de la coloration de fond. L'utilisation de la RNase A ou les résultats T1 seul en tâche de fond accrue et devrait être évitée. Nous avons également constaté que la qualité de la réaction de couleur finale est améliorée par l'utilisation du tampon AP frais, faite immédiatement avant l'utilisation.
Notre protocole peut être adapté pour les personnes âgées d'embryons ou de tissus adultes avec un traitement supplémentaire. Par exemple, nous avons aussi utilisé ce protocole pour effectuer les hybridations in situ sur les épais (100 um) sections vibratome du cerveau de souris adulte (données non présentées). Nous avons également utilisé ce protocole pour étudier l'expression des gènes dans les vieux embryons. Dans certains cas, ont utilisé ce protocole de visualiser l'expression des gènes à la surface de E13.5 E14.5 et embryons, comme GAD1 expression dans les follicules des vibrisses 4. Dans d'autres cas nous avons utilisé une lame de rasoir ou un scalpel pour couper E12.5 E14.5-embryons pour fournir un accès de sonde à des tissus spécifiques au sein de l'embryon. Fragments d'embryon préparé de cette manière peuvent être traitées en utilisant ce protocole avec quelques modifications. Dans ces cas, la longueur de tous les traitements et étapes de lavage doit être ajusté selon la taille des tissus et optimisé de manière empirique.
Notre protocole inclut également des méthodes de post-hybridation de sectionnement et d'analyse des profils d'expression. Combiné avec la visualisation monter l'ensemble de l'expression génique de cette étape supplémentaire peut ajouter un grand nombre d'informations supplémentaires concernant le profil d'expression d'un gène. Nous avons intégré des embryons après avoir subi une hybridation in situ dans de la paraffine ainsi que dans de la résine de plastique 4,16. Les articles tachés d'embryons peuvent être utilisés pour générer une reconstruction en trois dimensions du patron d'expression révélée par toute la montagne dans le processus d'hybridation in situ. Nous avons utilisé un logiciel de reconstruction à partir du logiciel SURFdriver à cet effet.
Déclarations de divulgation
Aucun conflit d'intérêt déclaré.
Remerciements
Ce travail a été soutenu par des subventions du NIH R21MH082360 (BGC) et R01HD056315 (GRN), ainsi que l'Université de Géorgie.
matériels
| Name | Company | Catalog Number | Comments | |
| Nom | Tapez | Société | Numéro de catalogue | Commentaires |
|---|---|---|---|---|
| Digoxigénine-11-uridine-5'-triphosphate | Réactifs | Roche | 11209256910 | |
| Réglez Triphosphate ribonucléoside | Réactifs | Roche | 11277057001 | |
| Colonnes Spin rapide pour la purification d'ARN radiomarqué | Alimentation | Roche | 11274015001 | |
| T7 RNA polymérase | Réactifs | Roche | 10881767001 | |
| SP6 ARN polymérase | Réactifs | Roche | 10810274001 | |
| T3 ARN polymérase | Réactifs | Roche | 11031163001 | |
| CTP, [α-32P] - 800Ci/mmol 10mCi/ml, 250 pCi | Réactifs | Perkin Elmer | BLU008X250UC | |
| DNase I recombinante, sans RNase | Réactifs | Roche | 4716728001 | |
| Urée, incolore à blanc cristaux ou poudre cristalline | Réactifs | Fisher | BP169-500 | |
| Tampon de chargement de gel | Réactifs | Ambion | AM8547 | |
| Hybond-N +, Amersham | Alimentation | GE Healthcare | RPN82B | |
| UV Stratalinker 2400 | Équipement | Stratagene | ||
| Diéthyl pyrocarbonate | Réactifs | Sigma | D5758-100ML | |
| Héparine sodique | Réactifs | Acros | 41121-0010 | |
| peroxyde d'hydrogène à 30% dans de l'eau | Réactifs | Fisher Scientific | BP2633-500 | |
| Glutaraldéhyde, 8% de grade EM | Réactifs | Polysciences | 0 / 710 | À utiliser avec prudence selon les instructions du fabricant |
| Réactif de blocage | Réactifs | Roche | 11096176001 | Faire en stock 5% et conserver à -20 ° C |
| Protéinase K | Réactifs | Roche | 3115852001 | |
| RNase A | Réactifs | Roche | 10109142001 | Faire que 10 mg / ml d'actions et de stocker à -20 ° C. |
| RNase T1 | Réactifs | Roche | 10109193001 | |
| Ultrapure Formamide | Réactifs | Invitrogen | 15515-026 | |
| Chlorhydrate de lévamisole | Réactifs | ICN Biomedicals. Inc | 155228 | |
| L'acide ribonucléique de la levure Torula, type VI | Réactifs | Sigma | R6625 | phénol / chloroforme extrait plusieurs fois et précipité, remis en suspension dans DEPC dH2O et conservés à -20 ° C |
| Anti-digoxigénine-AP fragments Fab | Réactifs | Roche | 11093274910 | |
| BM Violet AP Substrat, précipitant. Prêt-à-l'emploi. | Réactifs | Roche | 11 442 074 001 | |
| 4 ml, transparent, fermé Top, Kit commodité de stockage Vial | Alimentation | Scientifique nationale | B7800-2 | |
| Shake N four à hybridation Cuire | Équipement | Boekel Scientific | 136400 | |
| Biopsie Sure-Tek | Alimentation | Fisherbrand | 15 à 200-402C | |
| Paraplast plus le milieu tissulaire intégration | Réactifs | Fisherbrand | 23-021-400 | |
| Peel-A-Way jetables en plastique moules intégration tissulaire | Alimentation | Polysciences | 18646A | |
| Microscope diapositives Colorfrost plus | Alimentation | Fisherbrand | 12-550-17 | |
| Nucléaire Rouge Rapide | Réactifs | Sigma | N8002 | |
| Cytoseal 60 | Réactifs | Richard-Allan scientifique | 8310-16 |
Références
- Divjak, M., Glare, E. M., Walters, E. H. Improvement of non-radioactive in situ hybridization in human airway tissues: use of PCR-generated templates for synthesis of probes and an antibody sandwich technique for detection of hybridization. J. Histochem. Cytochem. 50, 541-548 (2002).
- Wilkinson, D. G., Nieto, M. A. Detection of messenger RNA by in situ hybridization to tissue sections and whole mounts. Methods Enzymol. 225, 361-373 (1993).
- Logel, J., Dill, D., Leonard, S. Synthesis of cRNA probes from PCR-generated DNA. Biotechniques. 13, 604-610 (1992).
- Maddox, D. M., Condie, B. G. Dynamic expression of a glutamate decarboxylase gene in multiple non-neural tissues during mouse development. BMC Dev Biol. 1, 1-1 (2001).
- Conlon, R. A., Rossant, J. Exogenous retinoic acid rapidly induces anterior ectopic expression of murine Hox-2 genes in vivo. Development. 116, 357-368 (1992).
- Krauss, S. Zebrafish pax[zf-a]: a paired box-containing gene expressed in the neural tube. EMBO J. 10, 3609-3619 (1991).
- Hemmati-Brivanlou, A. Localization of specific mRNAs in Xenopus embryos by whole-mount in situ hybridization. Development. 110, 325-330 (1990).
- Harland, R. M. In situ hybridization: an improved whole-mount method for Xenopus embryos. Methods Cell Biol. 36, 685-695 (1991).
- Herrmann, B. G. Expression pattern of the Brachyury gene in whole-mount TWis/TWis mutant embryos. Development. 113, 913-917 (1991).
- Conlon, R. A., Herrmann, B. G. Detection of messenger RNA by in situ hybridization to postimplantation embryo whole mounts. Methods Enzymol. 225, 373-383 (1993).
- Parr, B. A., Shea, M. J., Vassileva, G., McMahon, A. P. Mouse Wnt genes exhibit discrete domains of expression in the early embryonic CNS and limb buds. Development. 119, 247-261 (1993).
- Rosen, B., Beddington, R. S. Whole-mount in situ hybridization in the mouse embryo: gene expression in three dimensions. Trends Genet. 9, 162-167 (1993).
- Quiring, R. Large-scale expression screening by automated whole-mount in situ hybridization. Mech. Dev. 121, 971-976 (2004).
- Thut, C. J., Rountree, R. B., Hwa, M., Kingsley, D. M. A large-scale in situ screen provides molecular evidence for the induction of eye anterior segment structures by the developing lens. Dev. Biol. 231, 63-76 (2001).
- Neidhardt, L. Large-scale screen for genes controlling mammalian embryogenesis, using high-throughput gene expression analysis in mouse embryos. Mech. Dev. 98, 77-94 (2000).
- Gordon, J., Bennett, A. R., Blackburn, C. C., Manley, N. R. Gcm2 and Foxn1 mark early parathyroid- and thymus-specific domains in the developing third pharyngeal. 103, 141-143 (2001).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon