Method Article
Ganze Berge In-situ Hybridisierung von E8.5 bis E11.5 Maus Embryonen
In diesem Artikel
Zusammenfassung
Das ganze Berg In situ Hybridisierung Protokoll diskutiert kritische Schritte, die reproduzierbar hohe Qualität der Ergebnisse zur Genexpression Studien in E8.5-E11.5 Tage alten Maus-Embryonen zu gewährleisten.
Zusammenfassung
Whole mount in situ Hybridisierung ist eine sehr informative Ansatz für die Definition Genexpressionsmuster in Embryonen. Die in-situ-Hybridisierung Verfahren sind langwierig und technisch anspruchsvolle mit mehreren wichtigen Schritten, die gemeinsam die Qualität des Endergebnisses beitragen. Dieses Protokoll beschreibt im Detail einige wichtige Qualitätskontrolle Schritte zur Optimierung Sondenmarkierung und Leistung. Insgesamt bietet unser Protokoll eine detaillierte Beschreibung der kritischen Schritte notwendig, um reproduzierbar zu erhalten qualitativ hochwertige Ergebnisse. Zunächst beschreiben wir die Erzeugung von Digoxigenin (DIG) markierten RNA-Sonden über In-vitro-Transkription der DNA-Templates mittels PCR generiert. Wir beschreiben drei kritischen Qualitätskontrolle Assays, um die Menge, die Integrität und die spezifische Aktivität des DIG-markierten Sonden zu bestimmen. Diese Schritte sind für die Erzeugung einer Sonde eine ausreichende Empfindlichkeit, um endogene mRNAs in einer ganzen Maus-Embryo erkennen wichtig. Darüber hinaus beschreiben wir für die Fixierung und Lagerung von E8.5-E11.5 Tage alten Maus-Embryonen für die in-situ-Hybridisierung. Dann beschreiben wir detailliert Methoden für begrenzte Proteinase K Verdau der rehydriert Embryonen durch die Details der Hybridisierungsbedingungen, post-Hybridisierung wäscht und RNase-Behandlung, um nicht-spezifische Sonden-Hybridisierung zu entfernen. Ein AP-konjugierte Antikörper verwendet, um die markierten Sonde visualisieren und zeigen die Expressionsmuster der endogenen Transkript. Repräsentative Ergebnisse sind aus der erfolgreichen Experimente und typische suboptimal Experimente gezeigt.
Protokoll
1. Riboprobe Generation über In-vitro-Transkription
- Vorbereitung PCR-Produkte für in-vitro-Transkription Vorlagen.
- Designing PCR-Primer mit Phagen Transkription Promotor-Sequenzen in ihren 5'-Enden.
Hinweis: Die Promotorsequenz hinzugefügt, um der Sense-Strang-PCR-Primer 5'-Ende wird zum Aufzeichnen der Sense-Sonde verwendet werden, und die Promotorsequenz hinzugefügt, um der Antisense-PCR-Primer 5'-Ende wird für die Synthese des anti-sense-Sonde verwendet werden 1-3. Wir haben keinen Unterschied in der Transkription Effizienz zwischen Vorlagen, die nur der Kern Promotorsequenzen vs Vorlagen, die die Kern-Promotor plus zusätzliche 5'-Sequenzen enthalten, enthalten erkannt. Diese Methode hat mit dem T3, T7 und SP6-Phagen-Promotoren verwendet worden.
Sequenzen, die sehr unter-Gen Familienmitglieder oder stark repetitiven Sequenzen konserviert sind zu vermeiden, da sie in nicht-spezifische Hybridisierung führen kann und dadurch Hintergrundfärbung werden. Probes mit hoher GC-Gehalt begrenzt Digoxigenin-rUTP Einarbeitung und DNA-Template-Sequenzen mit Auflagen von T-Reste wird der Einbau von DIG-UTP aufgrund sterischer Interferenz zwischen dem Digoxigenin-Moleküle zu begrenzen. Die Länge der Sonden kann von 300bp bis 1kb Bereich. Aber solange die oben genannten Kriterien erfüllt sind, haben in der Regel längere Sonden höhere spezifische Aktivitäten. - Die Vorlage für die PCR kann ein Plasmid-Klon, genomischen Klon oder genomischen DNA werden. In der Regel kaufen wir in voller Länge EST-Klone (zB von ATCC oder Open Biosystems) als PCR-Vorlagen verwenden. Amplify die Template-DNA mittels Standard-PCR-Verfahren zu einem PCR-Produkt mit den Promotorsequenzen zu produzieren. Um genügend Sonde Vorlagen an mehrere Sonden Synthesereaktionen wir in der Regel einzurichten 8 x 50 ul PCR zu unterstützen. Die Reaktionen sind für den nächsten Schritt zusammengefasst. In der Regel ein paar große Volumen PCRs oder mehrere kleine Reaktionen (wie z. B. die 50 ul Reaktionen, die wir benutzen) ausreicht, um genügend Vorlage für mehrere Sonden Synthesereaktionen zu erzeugen.
- Um verunreinigende Proteine, vor allem Spuren von RNase in Plasmid-Präparationen zu entfernen, zu verdauen jeweils 100 &mgr; l der PCR-Reaktion mit 1μl von 20mg/ml Proteinase K bei 55 ° C für mindestens 30 Minuten. Alle Reagenzien und andere Gegenstände nach dieser Proteinase K-Verdau verwendet werden sollte RNase frei und spezifisch für RNA Arbeit gewidmet.
- Während der Proteinase K-Verdau, führen Sie eine Probe der PCR-Reaktion auf ein Gel, um die Integrität des PCR-Produkts zu überprüfen. Wir verwenden Acrylamid Minigele zu kleineren PCR-Produkte (weniger als 600-700 bp) und Agarosegelen zu lösen, um größere PCR-Produkte (> 600-700 bp) für die Qualitätskontrolle Analyse zu lösen. Wenn Sie mehrere Banden auf dem Gel zu sehen, empfehlen wir eine Optimierung der PCR-Reaktion, so dass Sie ein einzelnes PCR-Produkt der richtigen Größe zu erhalten.
- Gründlich extrahieren Sie die Proteinase K verdaute PCR-Reaktion mit einem gleichen Volumen einer Phenol / Chloroform (1:1)-Gemisch, durch eine Extraktion mit einem gleichen Volumen Chloroform. Mix bei jedem Schritt durch Vortexen das Rohr für mindestens 30 Sekunden. Diese Maßnahmen müssen in einem Laborabzug durchgeführt werden.
- Man fällt das gereinigte PCR-Produkt durch Zugabe von 0,1 Volumen 3 M Natriumacetat und 2 Volumen 100% Ethanol. Lassen Sie in -20 ° C für mindestens 30 Minuten.
- Spin für 5 Minuten bei 13.000 rpm in einer Mikrozentrifuge, entfernen Sie den Überstand und wäscht mit 70% Ethanol. Lassen Sie das DNA-Pellet vollständig trocknen.
- Resuspendieren DNA in geeigneten Volumen (zB 50 ul) von 1xTE. Messen Sie die DNA-Konzentration mit einem Fluorometer.
- Bestimmen Sie die Nukleotidsequenz der einzelnen PCR-Template prep Verwendung von Primern, die Phagen-Promotor-Sequenzen oder auf eine interne Sequenz innerhalb der Sonde entspricht. Dies ist eine kritische Qualitätskontrolle Schritt, dass die Sonde Vorlage der richtigen Reihenfolge ist gewährleistet.
- Die DNA ist bereit, als Vorlage für die in vitro Transkription dienen. Bewahren Sie die Template-DNA bei 4 ° C.
- Designing PCR-Primer mit Phagen Transkription Promotor-Sequenzen in ihren 5'-Enden.
- In-vitro-Transkription unter Verwendung des PCR-Produktes als Vorlage.
- Richten Sie die eine RNA-Transkription in einem Endvolumen von 50 ul. Die Transkription umfasst 500 - 1000 ng Template-DNA, 5μl 10X Transcription Buffer (mit 100mm DTT), 10 &mgr; l 2,5 dig-NTP-Mix, 3μl (50 ~ 90 Einheiten) der RNA-Polymerase, 1μl Alpha-32 P CTP (bis bis 6 Monate alt) und der Rest des Volumens wird durch Zugabe von Diethylpyrocarbonat behandeltem Wasser hergestellt. Die 2,5-mm-dig-NTP-Mix ist in der Regel bis als 40 ul arbeitet Lager mit 10 &mgr; l 10 mM CTP, 10 &mgr; l 10 mM GTP, 10 &mgr; l 10 mM ATP, 10 mM 6.5μl UTP, 3.5μl 10mM Digoxigenin-11 UTP hergestellt. Bei der Zusammenstellung der Transkription, stellen Sie sicher, dass alle der Reaktionskomponenten (außer für das Enzym) auf Raumtemperatur erwärmt werden. Mischen Sie die DNA und Wasser zuerst, dann die Reaktion, mischen und fügen Sie dann die anderen Komponentensonst die Spermidin in der Transkription Puffer Fällung der DNA. Inkubieren für 2 Stunden bei der Temperatur für die RNA-Polymerase verwendet empfohlen.
Hinweis: Die α-32 P ist zu der Reaktion gegeben, damit Sie den Anteil der ab Nukleotid-Pool, der in die RNA-Sonde integriert ist zu bestimmen. Dies ist ein wichtiges Maß für die Effizienz der Reaktion. Alle folgenden Schritte Verfahren für die Behandlung 32 S.
Eine alternative Methode zur Messung der Menge an RNA-Sonde synthetisiert wird auf ein kleines Volumen Spektralphotometer verwenden. Dies vermeidet die Verwendung von 32 P Kennzeichnung (siehe Hinweise in Schritt 1.3.1) - In den letzten paar Minuten der Inkubation, bereiten Sie die Quick Spin Column (Roche) nach den Anweisungen des Herstellers.
- Nach der Inkubation, fügen 1μl RNase freie DNase I und Inkubation bei 37 ° C für 10 min.
- Verdünnen, um 100 &mgr; mit 50 ul Reaction Verdünnungspuffer (20 mM Tris pH 7,5, 1% SDS, 20 mM EDTA, 100 mM NaCl (RNase frei, machen Sie eine 50ml Lager und bei Raumtemperatur lagern)). Nehmen 1μl (in der Regel in 4μl von 1x TE) für Szintillationszählung der gesamten beginnend zählt und für die Gel-Analyse.
- Wenden Sie den Rest der Reaktion auf die Mitte der Säule Bett und Spin bei 1100 X g für 4 min in einer Tischplatte klinischen Zentrifuge. Nach dem Spin Ihre RNA-Sonde wird in das Sammelrohr.
- Add 2 Volumen 100% Ethanol in den eluierten Reaktion. Gut mischen und halten auf Eis für 5min (oder in -20 ° C oder 30 min).
- Drehen Sie das Rohr in einer Mikrozentrifuge bei maximaler Drehzahl für 5 Minuten, um Pellets der RNA-Sonde.
- Vollständig zu entfernen der Überstand mit einer Pipette und lassen Sie das Pellet an der Luft trocknen. Lassen Sie sich nicht das Pellet over-trocken, weil es sehr schwierig sein wird, um es aufzulösen.
- Lösen Sie die RNA-Sonde mit 50 ul - 100 ul 1xTE oder DEPC-H 2 O. Nehmen Sie einen 1 ul Probe zur Bestimmung der prozentualen Einbau und zur Analyse auf einem denaturierenden Acrylamid-Gel. Passen Sie Ihre Sonde Konzentration auf 100 ng / ul nach dem geschätzten Ertrag.
- Probes, dass alle drei Qualitätskontrollen bestanden haben, können dann in -20 ° C für 6 bis 12 Monate gelagert. Wir in der Regel erschöpft Sonde Lager innerhalb von 12 Monaten. Es ist sehr wahrscheinlich, dass Sonden für sehr lange Zeiträume bei -20 ° C gelagert werden kann.
- Richten Sie die eine RNA-Transkription in einem Endvolumen von 50 ul. Die Transkription umfasst 500 - 1000 ng Template-DNA, 5μl 10X Transcription Buffer (mit 100mm DTT), 10 &mgr; l 2,5 dig-NTP-Mix, 3μl (50 ~ 90 Einheiten) der RNA-Polymerase, 1μl Alpha-32 P CTP (bis bis 6 Monate alt) und der Rest des Volumens wird durch Zugabe von Diethylpyrocarbonat behandeltem Wasser hergestellt. Die 2,5-mm-dig-NTP-Mix ist in der Regel bis als 40 ul arbeitet Lager mit 10 &mgr; l 10 mM CTP, 10 &mgr; l 10 mM GTP, 10 &mgr; l 10 mM ATP, 10 mM 6.5μl UTP, 3.5μl 10mM Digoxigenin-11 UTP hergestellt. Bei der Zusammenstellung der Transkription, stellen Sie sicher, dass alle der Reaktionskomponenten (außer für das Enzym) auf Raumtemperatur erwärmt werden. Mischen Sie die DNA und Wasser zuerst, dann die Reaktion, mischen und fügen Sie dann die anderen Komponentensonst die Spermidin in der Transkription Puffer Fällung der DNA. Inkubieren für 2 Stunden bei der Temperatur für die RNA-Polymerase verwendet empfohlen.
- Bewertung der Qualität des Ribosonde - Messsonde zu erhalten.
- Zur Abschätzung der Sonde ergeben, teilen sich die Grafen nach dem Spin-Säule von den Grafen in der Reaktion vor dem Spin-Säule (gemessen mit den Proben in der Sonde Syntheseprotokoll genommen) erholt. Für diese Reaktion, 100% Einbau = 33 pg. Eine erfolgreiche Reaktion in der Regel gibt eine 15-50% Ausbeute. Probes mit weniger als 15% Einbau (<5 ug Ausbeute) sollten nicht verwendet werden.
Hinweis: Eine alternative Methode zur Messung der Sonde Ertrag ist auf ein kleines Volumen Spektralphotometer oder Fluorometer verwenden, um direkt zu messen die Menge an RNA vorhanden, nachdem Spin Säulenchromatographie. Dieser alternative Ansatz vermeidet die Verwendung von 32 P Spur Kennzeichnung. Wir haben Ribosonde Konzentration mit einem BioTek Epoch Mikroplatten-Spektrophotometer gemessen. Ein Vergleich der Werte aus dem Spektralfotometer den Schätzungen der Ribosonde erhaltene Masse aus dem 32 P Inkorporation zeigten, dass die beiden Methoden vergleichbare Ergebnisse mit einer ähnlichen kleinen Volumen (2 ul) Probe aus dem letzten Sonde Vorbereitung zu geben.
Fast immer einen sehr geringen oder gar keinen Ertrag ist aufgrund der RNase Kontamination aus der Plasmid-Präparation als Matrize für die PCR verwendet. Achten Sie darauf, Ihre Plasmid-DNA und PCR-Produkt mit Proteinase K behandelt, wie in Schritt 1.1.3 beschrieben.
- Zur Abschätzung der Sonde ergeben, teilen sich die Grafen nach dem Spin-Säule von den Grafen in der Reaktion vor dem Spin-Säule (gemessen mit den Proben in der Sonde Syntheseprotokoll genommen) erholt. Für diese Reaktion, 100% Einbau = 33 pg. Eine erfolgreiche Reaktion in der Regel gibt eine 15-50% Ausbeute. Probes mit weniger als 15% Einbau (<5 ug Ausbeute) sollten nicht verwendet werden.
- Bewertung der Qualität des Ribosonde - Messung der spezifischen Aktivität.
- Um das Ausmaß der Digoxigenin messen - UTP Einarbeitung führen Sie eine Stelle zu testen. Dies ist ein entscheidender Qualitätskontrolle.
Stellen Sie die Sonde Konzentration auf 100 ng / ul. Spot 1 ul von Verdünnungsreihen (10 -2 bis 10 -5) der Sonde auf eine 82mm Durchmesser Hybond-N + (GE Healthcare) oder andere gleichwertige Nylonfilter. Fügen Sie eine unmarkierte DNA-Probe als negative Kontrolle. Crosslink den feuchten Filter sofort in einem UV-Crosslinker bei 125mJoules oder nach den Anweisungen des Herstellers für die Nylon-Filter Sie verwenden.
Alle folgenden Schritte (1.4.2 - 1.4.6) werden in einer Petrischale auf einem Wippschüttler bei Raumtemperatur durchgeführt. Sie müssen sich keine Gedanken über RNase Sorgen nach dem Spotten der Sonde Verdünnungen auf dem Filter - Blockieren Sie die Filter für 30 Minuten bei Raumtemperatur in 10 ml 1% Blocking Reagenz (Roche) in 1X TN (10X TN-Puffer = 1M Tris pH 7,5, 1,5 M NaCl)
- Wash 1 x 15 min in 10 ml 1X TN-Puffer.
- Inkubieren mit anti Digoxigenin Fab-Fragment zu 1 / 5000 Verdünnung in 1X TN-Puffer für 30 Minuten.
- Wash 2 X 15 Minuten in 10 ml 1X TN-Puffer.
- Die Farbreaktion wird mit BM purple Substrat (benötigen etwa 5 ml in der Schüssel) durchgeführt. Farbe sichtbar sein soll auf der Stelle aus dem 10 -4 Verdünnung in 30 Minuten. An diesem Punkt, stoppen Sie die Farbreaktion durch Waschen 2 mal für jeweils 5 Minuten mit 5fach-TE. Wenn das Signal nicht in die Stelle, an der 10 -4 Verdünnung entspricht sichtbaren dann die Sonde muss verworfen werden.
Hinweis: Nach unserer Erfahrung, unzureichende spezifische Aktivität ist nur mit kurzen Sonden gefunden. Die Erhöhung der Größe der Sonde Vorlage (bis 1kb) unter Einhaltung der anderen Design-Kriterien in diesem Protokoll festgelegten erhöhen sollte die spezifische Aktivität der Sonde. Mit geringer spezifischer Aktivität führt zu einer geringen oder nicht nachweisbare Signal.
- Um das Ausmaß der Digoxigenin messen - UTP Einarbeitung führen Sie eine Stelle zu testen. Dies ist ein entscheidender Qualitätskontrolle.
- Testen Sie die Integrität der Sonde durch Elektrophorese durch einen 5% igen denaturierenden Acrylamid-Gel.
Dies ist ein weiteres wichtiges Qualitätsmerkmal Kontrollassay. Dieses Protokoll setzt voraus, dass die Sonde mit 32 P wurde bei der Synthese-Reaktion (siehe Schritt 1.2.1) gekennzeichnet. Wir verwenden denaturierenden Acrylamid-Gelen auf eine sehr hohe Auflösung bieten, so dass wir leicht erkennen, abgebaut oder unvollständige Sonden. Typischerweise wird eine 5% denaturierenden Acrylamid Minigel (7,5 g Harnstoff, 1.9ml 40% Acrylamid, 1.9ml 2% Bisacrylamid (oder verwenden Sie ein 20:1 Acrylamid: Bisacrylamid-Lösungen), 1,5 ml 10x MOPS-Gel-Puffer (0,2 M Morpholinopropansulfonsäure Säure, pH 7,0, 50 mM Natriumacetat 5mM EDTA) und H 2 O auf ein Gesamtvolumen von 15 ml) funktioniert sehr gut für die Lösung Sonden in der Größe reicht in diesem Protokoll verwendet.
Hinweis: Als Alternative TBE-Harnstoff Fertiggelen können von verschiedenen Lieferanten bezogen werden. Folgen Sie den Anweisungen des Herstellers für den Betrieb Fertiggelen.- Mischen Sie die Probe mit dem gleichen Volumen Gel-Ladepuffer (Ambion) und gut mischen.
- Hitze bis 95 ° C für 5 Minuten keine Sekundärstruktur denaturieren.
- Unmittelbar kühl auf Eis vor dem Laden auf das Gel wieder Glühen zu verhindern.
- Führen Sie die vor und nach der Spalte Proben zusammen bei 200 V, bis der blaue Farbstoff läuft bis zum Ende des Gels. Das Gel wird in 1X MOPS-Gel-Puffer laufen.
- Wickeln Sie das Gel in Frischhaltefolie und schützen Sie es vor einem Röntgenfilm oder Phosphor-Imager Bildschirm, um radioaktiv markierte RNA zu erkennen. Wenn 32 P Kennzeichnung nicht verwendet wird, können Sie das Gel mit einem herkömmlichen Fluoreszenz-Farbstoff Fleck um die RNA zu erkennen
- Eine erfolgreiche Sonde Synthese wird in einer RNA-Sonden, die als scharfe Bande wandert sehr nah an der Spitze des Gels führen.
2. Sammlung und Speicherung von Maus-Embryonen
- Maus-Embryonen sollten in eiskaltem PBST (PBS + 0,1% Tween 20, (Diethylpyrocarbonat (DEPC) behandelt)) gesammelt und auf Eis gehalten während der Präparation.
- Die Embryonen werden in 4 ml verschraubbaren Glasfläschchen verarbeitet. Multiple Embryonen können in einem Fläschchen befestigt werden. So viele wie 10 bis 15 E9.5 oder 5 E10.5 Embryonen können in einer einzigen Flasche befestigt werden. Aufgrund der zunehmenden Größe des Embryos, sollte E11.5 Embryonen mit einer Rasierklinge hemisected vor der Fixierung auf staatliche Beihilfen in der Sonde Zugang während der Hybridisierung. Bis zu 3 hemisected E11.5 Embryonen können in einem einzigen Gefäß platziert werden.
- Um die Wirksamkeit der Schritte in diesem Protokoll zu maximieren und um eine Beschädigung des Embryos, Embryos Fixierung und alle Waschmittel sind in Probenfläschchen mit Lösungen gefüllt, um die untere Ebene des Fläschchens Hals durchgeführt, so dass nur eine kleine Luftblase bleibt zu reduzieren, sofern nicht anders angegeben wird. Für alle Waschschritte Fläschchen werden sich auf ihrer Seite auf einer Wippe Plattform gelegt.
- Fix für 6 Stunden bis über Nacht bei 4 ° C auf einem Schaukelstuhl Tisch mit 4% Paraformaldehyd (PFA) in PBST. Vials sollten horizontal platziert werden und auf einer Wippe Plattform rockten bei 4 ° C.
- Nach der Fixierung sind Embryonen zweimal für jeweils 5 Minuten in PBST auf Eis gewaschen und anschließend entwässert schrittweise durch ein Methanol / PBST-Serie (25% Methanol in PBST, 50% Methanol in PBST und 75% Methanol in PBST) in 100% Methanol durch den Austausch Lösungen in der Durchstechflasche mit Glas Pasteur Pipetten. Embryonen können bei -20 ° C in 100% Methanol für 8 Monate und vielleicht auch länger gelagert werden.
3. Die Hybridisierung der Digoxigenin markierten Sonde, ganze Mäuse-Embryonen
- Pannen-Herzen und Köpfe der E10.5 und älteren Embryonen mit einer Mikrodissektion Messer, während die Embryonen noch in Methanol (wenn dies nicht während der Dissektion getan). Dieses einfache Schritt ermöglicht die Waschlösungen an das Gehirn Vesikel und die Herzkammern damit stark reduziert Hintergrundfärbung geben.
- Rehydrieren die Embryonen durch aufeinanderfolgende Waschungen in einem Methanol / PBST-Serie (75% Methanol in PBST, 50% Methanol in PBST und dann 25% Methanol in PBST) für 5-10 Minuten an jedem Methanol-Konzentration. Zweimal waschen für 5 Minuten jeder Wäsche in 100% PBST.
- Inkubieren in 4:1 PBST: 30% H 2 O 2für 1 Stunde auf Eis. Dann mit 3 Änderungen waschen jeweils 5 min in 1X PBST.
- Entfernen Sie die letzten PBST waschen und ersetzen mit 1ml 10 pg / ml Proteinase K in PBST. Während der Proteinase K-Verdau Ort die Rohre vertikal in einem Rack in einem 25 ° C Wasserbad.
Hinweis: Die Lösungen von Proteinase K Aktivität verlieren im Laufe der Zeit wegen Selbst-Verdauung. Um sicherzustellen, maximale und konsistente Aktivität der Proteinase K-Lösung muss frisch zubereitet werden unmittelbar vor jedem Gebrauch. Für jeden Einsatz eine kleine Menge einer 1mg/ml Lager dann verdünnen. Die Inkubationszeit sollte für jede Charge von Proteinase K Pulver und für jeden Embryo Alter bestimmt werden. Zum Beispiel für E9.5 Embryonen eine gute Zeit ist in der Regel 10-15 min bei 25 ° C, während für E10.5 es in der Regel 20-30 min bei 25 ° C. Die Proteinase K Inkubationszeit sollte auch gerade in Experimenten gemessen werden, um aussagekräftige Vergleiche zwischen den Flaschen und zwischen den Experimenten zu ermöglichen. Die Temperatur sollte während der Verdauung, um die Reproduzierbarkeit zu erhöhen beibehalten werden. Unterschiede so gering wie 1 ° C die Menge der Verdauung zwischen den Experimenten verändern kann. Es wird dringend empfohlen, dass die Proteinase K Verdau in einem Inkubator oder Wasserbad für eine genaue Temperaturregelung möglich. Dieser Schritt ist wichtig für die Zulassung der Sonde mit dem Gewebe um dadurch starkes Signal zu durchdringen. Allerdings wird über-Verdauung Beschädigung des Embryos, und sie werden nach und nach im Laufe der verbleibenden Schritte zerfallen. - Stoppen Sie die Proteinase K-Verdau durch zweimaliges Waschen für jeweils 5 Minuten Waschen bei Raumtemperatur mit frisch zubereiteten 2 mg / ml Glycin in PBST. Dann zweimal waschen für jeweils 5 Minuten in PBST.
- Befestigen Sie das verdaut Embryonen für 20 Minuten bei Raumtemperatur in 4% PFA / PBST, 0,2% Glutaraldehyd (Polysciences). Nicht über-oder underfix. Wash 3 mal für jeweils 5 Minuten in PBST.
- An dieser Stelle sollte Embryonen in Gruppen für die Hybridisierung aufgeteilt werden. Entfernen Sie die PBST. 1 ml Hybridisierungspuffer (50% Reinstwasser Formamid (Invitrogen), 5x SSC, pH 5, 1% SDS, 50 ug / ml Heparin (Acros), 50 ug / ml Torula RNA (Sigma)), die auf 65 ° erwärmt worden ist, C (ohne Fühler). Lassen Embryonen zu regeln, dann entfernen Sie alle hybrization Puffer und ersetzen mit 1 ml frisch aufgewärmt Hybridisierungspuffer ohne Sonde.
- Prehybridize für 1 Stunde bei 65 ° C in einem Schüttel-Wasserbad oder in einem Ofen auf einem Schaukelstuhl Plattform (zum Beispiel die Boekle Shake 'n' Bake Oven, siehe Tabelle 1). Wenn Sie inkubieren die Ampullen in ein Wasserbad sicher sein Wasser kommt bis zu, aber nicht über die Spitzen der Ampullen. Die Hybridisierungstemperatur kann bis zu 70 ° C erhöht werden Hybridisierung Temperaturen über 65 ° C haben keinen Einfluss auf Signalstärke oder Hintergrund mit den meisten Sonden haben wir getestet. Die Embryonen werden durchsichtig, klebrig, und fragile in Formamid-Puffer (z. B. Hybridisierungspuffer) vorsichtig sein beim Umgang mit ihnen.
- Ersetzen Sie die Prähybridisierungspuffer mit 0,4 ml Hybridisierungspuffer mit Sonde in einer Konzentration von 0,25-1 g / ml. Verwenden Sie 0,5 ug / ml Sonde für Embryonen bis zu ca. E8.5. Für ältere Embryonen, sollten Sonden 1 bis 0,1 pg / ml titriert werden, um die optimale Konzentration für ein gutes Signal mit geringem Hintergrund (in der Regel 0,25-0,5 g / ml oder so) zu bestimmen. Beachten Sie, dass größere Mengen Hybridisierung kann notwendig sein, für die größeren Embryonen. Hybridisierung über Nacht bei 65 ° C.
- Wenn die Embryonen wurden über mit Proteinase K. behandelt sie erscheint sehr transparent. Sie können auch auf den Seiten der Flasche kleben, oder auseinander fallen. Embryonen, die auf diese Weise erscheinen soll an dieser Stelle in das Protokoll verworfen werden. Verarbeitung sie weiter nicht in interpretierbare Daten führen.
- Ersetzen Sie die Hybridisierungspuffer mit erwärmten Lösung I (50% Formamid, 5x SSC pH 5, 1% SDS) bei 70 ° C. Wash zweimal für 30 Minuten waschen E8.5 Embryonen dreimal 30 Minuten pro Waschgang für E9.5 und älter.
- Wash einmal mit vorgewärmten 50% Lösung I: 50% Lösung II (0,5 M NaCl, 10 mM Tris HCl pH 7,5, 0,1% Tween 20) bei 70 ° C für 10 Minuten.
- Dreimal für 5 Minuten pro Waschgang mit Lösung II bei Raumtemperatur.
- Ersetzen Puffer mit Lösung II mit RNase A 100 pg / ml, RNase T1 100 Einheiten / ml. Inkubieren auf einer Wippe in der warmen Stube bei 37 ° C zweimal für 30 Minuten pro Waschgang. Die RNase waschen ist wichtig für die Verringerung Hintergrund ergab sich aus unspezifisch hybridisierten Sonden.
- Wash in Lösung III (50% Formamid, 2x SSC pH5) bei 65 ° C zweimal für jeweils 30 Minuten waschen E8.5, 3-mal 30 Minuten pro Waschgang für E9.5 und älter.
- 3 x je 5 Minuten pro Waschgang mit 1X TBST (100 ml 10x TBS enthält 8g NaCl, 0,2 g KCl, 12,5 ml 2M Tris HCl pH 7,6 in Wasser. Um 1X TBST verdünnen 10X TBS in einem geeigneten Volumen Wasser und add Tween 20 bis 0,1% Endkonzentration)
- Sofern nicht anders angegeben, sind alle Wäschen in der Probenfläschchen mit Lösungen gefüllt, um die untere Ebene der durchgeführtenFläschchen Hals. Für alle Waschschritte sind Fläschchen unten auf ihrer Seite auf einer Wippe Plattform gelegt.
4. Antikörpernachweis der Digoxygenin markierten Sonde
- Die Embryonen werden in 0,5 ml 1X TBST + 10% wärmebehandelt Schafserum vor blockiert. Dann rocken die Fläschchen vertikal bei Raumtemperatur (~ 20 ° C) für mindestens 2,5 Stunden.
- Ersetzen Sie die Blocking-Lösung mit frischem 1X TBST + 10% Serum mit einer 1:2000 - 1:5000 Verdünnung der alkalischen Phosphatase-konjugierte Schaf-Anti-Digoxigenin-Fab-Fragmente (subtrahiert durch Inkubation mit Embryo Acetonpulver, siehe unten). Verwenden Sie höhere Verdünnungen für E9.5 und älteren Embryonen, bis 1:5000 in den Hintergrund zu minimieren. Inkubieren vertikal auf Wippe bei 4 ° C über Nacht.
Hinweis: Wir haben festgestellt, dass die Inkubation der Anti-Digoxigenin-Fab-Fragmente mit einem Embryo Acetonpulver Ebenen der Hintergrundfärbung in diesem Protokoll reduziert. Zur Vorbereitung des Embryos Acetonpulver gebündelt E12.5-14.5 Maus-Embryonen in einem Mindestvolumen von PBS zu homogenisieren. Add 4 Volumen eiskaltem Aceton, mischen und auf Eis inkubieren für 30 Minuten. Überstand entfernen, durch Drehen bei 10.000 xg für 10 Minuten. Waschen des Pellets mit eiskaltem Aceton und Spin wieder. Verbreiten Sie das Pellet aus und schleifen zu Pulver auf ein Blatt Filterpapier und lassen Sie es an der Luft trocknen. Das Pulver kann dann in einem luftdichten Röhrchen bei 4 ° C für Jahre gespeichert.
Für die Antikörper-Subtraktion, sollten die Antikörper gegen das Embryo Acetonpulver pre-absorbiert werden, um eine unspezifische Bindung während oder vor pre-blocking reduzieren. Inkubieren Antikörper bei 1 / 100 mit einer kleinen Menge von Embryo-Pulver (die Menge ist nicht kritisch) in 1xTBST/10% Serum für mindestens 1 Stunde bei 4 ° C unter Schütteln. Entfernen Sie Pulver, durch Drehen in einer Mikrozentrifuge für 10 Minuten bei 4 ° C. Subtrahiert Antikörper kann bei 4 ° C für mindestens 6 Monate gelagert werden. - Dreimal je 5 Minuten mit 1X TBST, dann 5-6 mal für 1 Stunde jeweils bei Raumtemperatur, um überschüssige Antikörper zu entfernen. Falls gewünscht, machen eine längere Nacht Waschen bei 4 ° C in den Hintergrund zu reduzieren. Die über Nacht waschen drastisch reduzieren können Hintergrundwerte wodurch das Signal-Rausch-Verhältnis.
- Zweimal waschen für 20 Minuten pro Waschgang mit frisch zubereiteten alkalische Phosphatase-Puffer (100 mM Tris, pH 9,5, 50 mM MgCl 2, 100 mM NaCl, 0,1% Tween 20. Add 0,5-Promillegrenze Levamisol, ein endogener Inhibitor AP, für E9.5 und älter Embryonen).
- Ersetzen Sie die letzte Waschung mit BM Lila AP-Substrat (Roche) vorgewärmt auf Raumtemperatur.
- Lassen Sie die Farbreaktion bei Raumtemperatur inkubieren, und achten Sie auf Signal in regelmäßigen Abständen über einem Binokular. Für längere Farbreaktionen (mehrere Stunden bis über Nacht) die Ampullen vor Licht geschützt werden. Signal für reichlich mRNAs sollte innerhalb von 15 Minuten sichtbar. Reaktionen sollte gestoppt, wenn Sie mit dem Signal zufrieden sind, oder wenn der Hintergrund ein Problem zu sein beginnt. Wenn die Lösung selbst zu drehen beginnt dunkel, kann die Farbreaktion durch Austausch der Substrat-Lösung zu erweitern.
- Die Reaktion durch zweimaliges Waschen für 5 Minuten pro Waschgang in 1X PBST/5mM EDTA.
- Transfer zum 4% Paraformaldehyd / PBST wieder zu beheben, von einigen Stunden bis über Nacht. Oder Embryonen in 4% paraformaldehyde/1X PBST für Jahre gelagert werden oder sofort zum Schneiden bearbeitet werden. Das endgültige Fixierung Schritt ist entscheidend für die langfristige Erhaltung der Färbungsmuster.
5. Paraffin eingebettet und Schnitte von gefärbten Embryonen
- Bevor die Embryonen nach der Farbreaktion (Schritt 4,8) Bilder von der ganze Berg gefärbten Embryonen sind fest genommen werden, wenn die Färbung Ebene ist in der gewünschten Intensität. Nach der Dokumentation der Expressionsmuster in der ganzen Embryonen sind sie in der BM purple Substrat über Nacht auf 24 Stunden bei Raumtemperatur inkubiert, bis die ganze Embryo wird dunkelblau. Dieser Schritt ist sicherzustellen, dass die Färbung wird ausreichend dunkel zu zeigen, bis in den Gewebeschnitten. Bitte beachten Sie, dass die blaue Farbe des Embryos durch die Abscheidung einer sehr dünnen Schicht von Pigment in der ganzen Embryo Oberfläche ist. Diese Hintergrundfärbung nicht nachweisbar innerhalb der Gewebe von Interesse nach Schneiden.
- Waschen Sie die gefärbten Embryonen mit PBS dreimal, um das überschüssige Substrat Niederschläge zu entfernen und zu beheben, in 4% PFA über Nacht bei 4 ° C.
- Waschen mit PBS dreimal, und dann trocknen die Embryonen in 100% Methanol (wie in 2.4 beschrieben).
- Ersetzen Sie die letzte Methanol mit Xylol. Wash in Xylol 2 bis 3-mal für je 2 min waschen, bis die Embryonen klar geworden. Nicht zu waschen in Xylol, da so die das Gewebe sehr spröde, nachdem es eingebettet ist, so dass es sehr schwierig, intakte Abschnitte zu erholen. Visuelle Überwachung jedes Xylol Inkubation um sicherzustellen, dass Embryonen nur zu dem Punkt, dass klare und sehr vorsichtig sein, nicht zuzulassen, das Xylol Inkubation, um über diesen Punkt hinaus zu verlängern.
- Transfer-Embryonen (E10.5 und älter) In Biopsie Sure-Tek-Kassetten (Fisherbrand) und inkubieren Sie die ganze Kassette in Wachs (Fisherbrand) für 15 ~ 30 min bei 65 ° C. Ändern Sie die frische Wachs für weitere 15 ~ 30min und dann wieder zu ändern. Kleinere Embryonen / Gewebe Stämme erfordern kürzere Wachs Inkubationszeit. E9.5 Embryonen können in das Gewebe Einbettformen für alle Wachs Veränderungen gestellt werden. In diesen Fällen können Sie entfernen das Wachs aus der Form zu übertragen, sondern die Embryonen.
- Nach drei Wachses,-Embryonen in das Gewebe Einbettschälchen (Polysciences) und betten mit frischem Wachs. Vorsichtig Position der Embryonen für die gewünschte Schnittebene. Lassen Sie das Wachs abkühlen lassen, bis verfestigt.
- § das Wachs-Blöcke in 8 ~ 14μm Scheiben mit Standard-Mikrotom (zB Leica RM2155). Sammeln Sie Schnitte auf Objektträgern (Fisherbrand).
- Lassen Sie die Dias flach und trocken auf einer slidewarmer. Die Folien können bei 4 ° C über Monate gelagert werden.
- Getrocknete Abschnitte sind durch zwei 5 min Xylol wäscht entwachst und rehydriert in Wasser durch serielle Methanol / Wasser wäscht (100% Methanol 2 min, 100% Methanol 2 min, 90% Methanol / Wasser-2min, 70% Methanol / Wasser-2min, deionisiertes Wasser 5 min).
- Stain die Abschnitte mit Kernechtrot (Sigma) Färbelösung für 1 Minute. Zur Erzeugung des Kernechtrot Färbelösung, verdünnte eine konzentrierte Bestandsaufnahme Kernechtrot (0,1% Kernechtrot in 5% Aluminiumsulfat in Wasser, mit Hitze und durch Whatman Filterpapier auflösen) 1:5 in einer wässrigen Lösung von 5 % Aluminiumsulfat. Nach der Färbung waschen die Folien mit fließendem Wasser, bis das Wasser klar wird. Die Kernechtrot fungiert als Gegenfärbung und zeigt insgesamt Gewebestruktur in den Sektionen. Seine Farbe kontrastiert gut mit dem lila-blauen Färbung, dass die Lokalisierung der RNA-Sonde markiert. Kernechtrot ist eine Sättigung Fleck und die Intensität der Färbung wird durch die Konzentration der Färbelösung gesteuert. Wenn die Intensität der Kernechtrot Färbung ist unzureichend, wenn die Nutzung dieser Färbelösung können Sie diesen Schritt mit einer stärker konzentrierten Kernechtrot Lösung zu wiederholen.
- Bereiten Sie die Folien für die Montage Deckgläser durch Waschungen in jeder der Lösungen in den folgenden Serien: 90% Methanol / Wasser von 2 Wäschen in 100% Methanol, gefolgt von 2 Wäschen in Xylole. Inkubieren Sie die Objektträger für 2 Minuten in jedem der Waschgänge.
- Montieren Sie die Folien mit Cytoseal 60 Eindeckmedium (Richard-Allan Scientific). Bitte beachten Sie, dass wässrige Eindeckmittel löst die Kernechtrot Fleck.
Hinweis: Wir haben auch einen Kunststoff eingebettet Harz (Immuno-Bed, Polysciences Inc) verwendet werden, um Teile des gefärbten Embryonen mit hervorragenden Ergebnissen 4 zu erzeugen.
6. Repräsentative Ergebnisse:
Abbildung 1 zeigt repräsentative Ergebnisse, die mit diesem Protokoll. Abbildung 1A zeigt das Expressionsmuster von GATA3 in E10.5 Maus-Embryonen. Dieses Panel zeigt ein gutes Ergebnis mit einem hohen Signal-Hintergrund-Verhältnis. Abbildung 1C, im Gegensatz dazu zeigt eine in-situ-Hybridisierung für GATA3 mRNA, die eine teilweise abgebaute RNA-Sonde fehlgeschlagen. Ein geringer spezifischer Aktivität Sonde wird in ähnliche Ergebnisse mit Low-Signal zu Hintergrund-Kontrast führen. Ein Vergleich der Platten A und C die Bedeutung zeigt der Durchführung einer sorgfältigen Qualitätskontrolle Einschätzungen der Sonde Qualität zu gewährleisten qualitativ hochwertige Ergebnisse. Das Ergebnis mit dem Foxg1 Sonde (Abbildung 1B) erhalten illustriert auch ein gutes Ergebnis mit spezifischen Färbung im Telencephalon mit geringem Hintergrund in anderen Teilen des Gehirns. Das Gehirn und das Herz sind häufig Orte der Hintergrundfärbung durch Sonde Trapping verursacht. Punktion des Gehirns und des Herzens mit einer Pinzette können Waschlösungen in den Embryo und minimiert Hintergrundfärbung (siehe Schritt 3.1). 1D zeigt den typischen hellblauen Hintergrund gefunden werden, wenn das Gehirn nicht durchbohrt ist. Dies zeigt eine suboptimale Ergebnis unter Verwendung eines Pax1 Sonde. Pax1 ist nicht in jedem Teil des Gehirns exprimiert und all der Färbung in das Gehirn dieses Embryos zu sehen ist aufgrund unspezifischer Hintergrund.
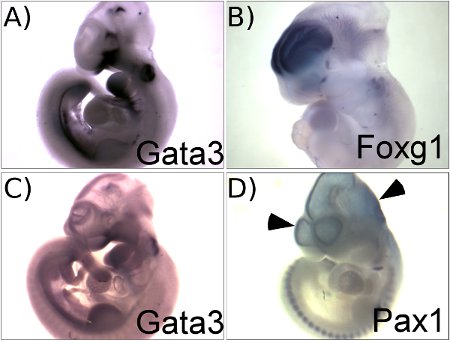
Abbildung 1. Repräsentative Ergebnisse zeigen Beispiele erfolgreicher (A, B) und der suboptimalen (C, D) Ergebnisse. A. E10.5 Embryos mit GATA3 Sonde hybridisiert. B. E11.5 Embryos mit Foxg1 Sonde hybridisiert. C. E10. 5 Embryo mit GATA3 Sonde, die teilweise abgebaut hybridisiert ist, zeigt sehr schwaches Signal, aber hohen Hintergrund durch den ganzen Embryo. D. E10.5 Embryos mit Pax1 Sonde ohne Punktion der Kopf hybridisiert, zeigt Hintergrundfärbung in den Herzkammern (Pfeilspitzen).
Diskussion
Die Methoden in diesem Protokoll beschriebenen aus einer Reihe von unterschiedlichen Quellen angepasst und optimiert für die ganze Berg E8.5-E11.5 Tage alten Maus-Embryonen. Methoden für die whole mount in situ Hybridisierung von Wirbeltierembryonen erschien erstmals in den frühen 1990er Jahren 2,5-12. Dieses Protokoll wurde in erster Linie von Methoden für Xenopus Embryonen 7,8 sowie Maus 2,11 entwickelt angepasst. In unserem Protokoll viel Wert auf eine sorgfältige Bewertung der Qualität der Sonde platziert. Vorsichtige Aufmerksamkeit zu generieren eine hohe Qualität und hoher spezifischer Aktivität RNA-Sonde durch die frühesten Schritte in diesem Protokoll bestimmt wird deutlich erhöhen die Qualität der Daten und sparen erhebliche Mengen an Zeit und Mühe in die lange Sicht. Unser Protokoll beinhaltet auch die Verwendung von PCR generierten DNA-Templates für Phagen-RNA-Polymerase-Transkription. Dieser Ansatz ist eine schnelle und sehr gut skalierbar. Es kann damit die Erzeugung einer großen Zahl von RNA-Sonden für in großem Maßstab in-situ-Hybridisierung Bildschirme 13-15.
Mehrere Schritte in diesem Protokoll sollte in Betracht gezogen, wenn der Hintergrund hoch ist, aber Signal niedrig sein. Eine Überlegung ist die Sonde. Wir hatten noch nie hohe Hintergrund mit RNA-Sonden, dass die Design-Kriterien in diesem Protokoll genannten Bedingungen erfüllt und das hatte alle drei Qualitätskontrollen (Ausbeute, spezifische Aktivität und Sondenlänge) übergeben. Ein weiterer Faktor, Hintergrund-Spiegel beeinflussen können, ist die Fähigkeit der Sonde in die embryonalen Gewebe eindringen. Es muss während der Präparation genommen werden, um die vollständige Entfernung des extraembryonalen Membranen, die den Embryo zu decken. Darüber hinaus ist die Proteinase K-Verdau Schritt absolut notwendig für Embryonen älter als E9.5. Längere Proteinase K-Verdau Zeiten geben unteren Hintergrund und stärkeres Signal, jedoch kann eine übermäßige Verdauung Mal erhöht Gewebeschäden führen und in der Auflösung des Embryos während des Verfahrens führen. Optimierung der Verdauung für jede Menge Proteinase K wird benötigt, um die besten Ergebnisse zu erzielen. Ein weiterer Schritt, die optimal auf High-Signal zu Rausch-Verhältnis zu erhalten kann, ist die RNase Inkubation (Schritt 3,14). Unser Protokoll verwendet eine Mischung aus RNase A und RNase T1. Beide Enzyme verdauen einsträngige RNA aber jedes Enzym hat eine andere Basis Spezifität. Die Kombination der beiden Enzyme führt zu einer weitgehenden Verdauung von einsträngige RNA zu kleinen Oligoribonukleotide. Dies ermöglicht den meisten nicht-spezifisch hybridisierte Sonde durch die post-Hybridisierung wäscht sich in einer starken Abnahme der Hintergrundfärbung entfernt werden. Verwendung von RNase A-oder T1-alone führt zu einem erhöhten Hintergrund und sollte vermieden werden. Wir haben auch festgestellt, dass die Qualität des Endprodukts Farbreaktion mit frischen AP-Puffer verbessert wird, hat unmittelbar vor dem Gebrauch.
Unser Protokoll kann für ältere Embryonen oder adulten Geweben mit zusätzlichen Verarbeitung angepasst werden. Zum Beispiel haben wir auch dieses Protokoll verwendet, um in situ Hybridisierungen auf dicke (100 um) Vibratom Abschnitte der erwachsenen Gehirn der Maus (Daten nicht gezeigt) durchzuführen. Wir haben auch dieses Protokoll verwendet, um die Genexpression in älteren Embryonen zu studieren. In einigen Fällen haben dieses Protokoll verwendet, um die Genexpression auf der Oberfläche der E13.5 und E14.5 Embryonen, wie GAD1 Ausdruck in den Follikeln der Tasthaare 4 visualisieren. In anderen Fällen haben wir genutzt einer Rasierklinge oder Skalpell, um E12.5-E14.5 Embryonen geschnitten Sonde den Zugang zu bestimmten Geweben zur Verfügung innerhalb des Embryos. Embryo-Fragmente auf diese Weise hergestellte verarbeitet dieses Protokoll verwenden, mit einigen Modifikationen werden. In diesen Fällen ist die Länge aller Behandlungen und Waschschritte muss nach Gewebe Größe angepasst und optimiert werden empirisch.
Unser Protokoll enthält auch Methoden für die Post-Hybridisierung Schneiden und Analyse der Expressionsmuster. Kombiniert mit der ganze Berg Visualisierung der Genexpression dieser zusätzliche Schritt eine große zusätzliche Informationen über die Expressionsmuster eines Gens hinzufügen können. Wir haben Embryonen nach Durchlaufen in situ Hybridisierung in Paraffin sowie in Kunstharz eingebettet 4,16. Abschnitte von gefärbten Embryonen verwendet werden, um eine dreidimensionale Rekonstruktion des Expressionsmusters von der ganzen mount in situ Hybridisierung Verfahren offenbart erzeugen. Wir haben den Wiederaufbau Software von SURFdriver Software für diesen Zweck verwendet.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Diese Arbeit wurde vom NIH gewährt R21MH082360 (BGC) und R01HD056315 (NRM) sowie der University of Georgia unterstützt.
Materialien
| Name | Company | Catalog Number | Comments | |
| Name | Typ | Firma | Katalog-Nummer | Kommentare |
|---|---|---|---|---|
| Digoxigenin-11-Uridin-5'-Triphosphat | Reagens | Roche | 11209256910 | |
| Ribonucleosid Triphosphat Set | Reagens | Roche | 11277057001 | |
| Quick Spin Columns für radioaktiv markierte RNA Aufreinigung | Versorgung | Roche | 11274015001 | |
| T7-RNA-Polymerase | Reagens | Roche | 10881767001 | |
| SP6-RNA-Polymerase | Reagens | Roche | 10810274001 | |
| T3-RNA-Polymerase | Reagens | Roche | 11031163001 | |
| CTP, [α-32P] - 800Ci/mmol 10mCi/ml, 250 Ci | Reagens | Perkin Elmer | BLU008X250UC | |
| DNase I rekombinante RNase-freie | Reagens | Roche | 4716728001 | |
| Urea, farblose bis weiße Kristalle oder kristallines Pulver | Reagens | Fischer | BP169-500 | |
| Gel-Ladepuffer | Reagens | Ambion | AM8547 | |
| Hybond-N +, Amersham | Versorgung | GE Healthcare | RPN82B | |
| UV Stratalinker 2400 | Ausstattung | Stratagene | ||
| Diethylpyrocarbonat | Reagens | Sigma | D5758-100ML | |
| Heparin-Natrium | Reagens | Acros | 41121-0010 | |
| Wasserstoffperoxid 30% in Wasser | Reagens | Fisher Scientific | BP2633-500 | |
| Glutaraldehyd, 8% EM grade | Reagens | Polysciences | 0 / 710 | Mit Vorsicht nach Herstellung der Anleitung |
| Blockierungsreagenz | Reagens | Roche | 11096176001 | Machen Sie sich in 5% Aktien und bei -20 ° C |
| Proteinase K | Reagens | Roche | 3115852001 | |
| RNase A | Reagens | Roche | 10109142001 | Machen Sie als 10 mg / ml Lager und bei -20 ° C. |
| RNase T1 | Reagens | Roche | 10109193001 | |
| Ultrapure Formamid | Reagens | Invitrogen | 15515-026 | |
| Levamisol Hydrochlorid | Reagens | ICN Biomedicals. Inc | 155228 | |
| Ribonukleinsäure aus Torula Hefe, Typ VI | Reagens | Sigma | R6625 | Phenol / Chloroform extrahiert mehrmals und ausgefällt, erneut in DEPC-dH2O und bei -20 ° C |
| Anti-Digoxigenin-AP Fab-Fragmente | Reagens | Roche | 11093274910 | |
| BM Lila AP Substrat, ausfällt. Ready-to-use Lösung. | Reagens | Roche | 11 442 074 001 | |
| 4ml, Clear, Closed Top, Lagervial Convenience Kit | Versorgung | Nationale wissenschaftliche | B7800-2 | |
| Shake n Bake Hybridisierungsofen | Ausstattung | Boekel Scientific | 136400 | |
| Biopsie Sure-Tek | Versorgung | Fisherbrand | 15 bis 200-402C | |
| Paraplast Plus-Gewebe Einbettmedium | Reagens | Fisherbrand | 23-021-400 | |
| Peel-A-Way Einweg-Kunststoff-Gewebe Einbettformen | Versorgung | Polysciences | 18646A | |
| Colorfrost Plus-Objektträger | Versorgung | Fisherbrand | 12-550-17 | |
| Nuclear Fast Red | Reagens | Sigma | N8002 | |
| Cytoseal 60 | Reagens | Richard-Allan Scientific | 8310-16 |
Referenzen
- Divjak, M., Glare, E. M., Walters, E. H. Improvement of non-radioactive in situ hybridization in human airway tissues: use of PCR-generated templates for synthesis of probes and an antibody sandwich technique for detection of hybridization. J. Histochem. Cytochem. 50, 541-548 (2002).
- Wilkinson, D. G., Nieto, M. A. Detection of messenger RNA by in situ hybridization to tissue sections and whole mounts. Methods Enzymol. 225, 361-373 (1993).
- Logel, J., Dill, D., Leonard, S. Synthesis of cRNA probes from PCR-generated DNA. Biotechniques. 13, 604-610 (1992).
- Maddox, D. M., Condie, B. G. Dynamic expression of a glutamate decarboxylase gene in multiple non-neural tissues during mouse development. BMC Dev Biol. 1, 1-1 (2001).
- Conlon, R. A., Rossant, J. Exogenous retinoic acid rapidly induces anterior ectopic expression of murine Hox-2 genes in vivo. Development. 116, 357-368 (1992).
- Krauss, S. Zebrafish pax[zf-a]: a paired box-containing gene expressed in the neural tube. EMBO J. 10, 3609-3619 (1991).
- Hemmati-Brivanlou, A. Localization of specific mRNAs in Xenopus embryos by whole-mount in situ hybridization. Development. 110, 325-330 (1990).
- Harland, R. M. In situ hybridization: an improved whole-mount method for Xenopus embryos. Methods Cell Biol. 36, 685-695 (1991).
- Herrmann, B. G. Expression pattern of the Brachyury gene in whole-mount TWis/TWis mutant embryos. Development. 113, 913-917 (1991).
- Conlon, R. A., Herrmann, B. G. Detection of messenger RNA by in situ hybridization to postimplantation embryo whole mounts. Methods Enzymol. 225, 373-383 (1993).
- Parr, B. A., Shea, M. J., Vassileva, G., McMahon, A. P. Mouse Wnt genes exhibit discrete domains of expression in the early embryonic CNS and limb buds. Development. 119, 247-261 (1993).
- Rosen, B., Beddington, R. S. Whole-mount in situ hybridization in the mouse embryo: gene expression in three dimensions. Trends Genet. 9, 162-167 (1993).
- Quiring, R. Large-scale expression screening by automated whole-mount in situ hybridization. Mech. Dev. 121, 971-976 (2004).
- Thut, C. J., Rountree, R. B., Hwa, M., Kingsley, D. M. A large-scale in situ screen provides molecular evidence for the induction of eye anterior segment structures by the developing lens. Dev. Biol. 231, 63-76 (2001).
- Neidhardt, L. Large-scale screen for genes controlling mammalian embryogenesis, using high-throughput gene expression analysis in mouse embryos. Mech. Dev. 98, 77-94 (2000).
- Gordon, J., Bennett, A. R., Blackburn, C. C., Manley, N. R. Gcm2 and Foxn1 mark early parathyroid- and thymus-specific domains in the developing third pharyngeal. 103, 141-143 (2001).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten