Method Article
Génération d'ADN / ARN hybrides d'ADN génomique par la transformation en utilisant l'ARN contenant Oligonucléotides
Dans cet article
Résumé
Ce travail montre comment former un hybride ARN / ADN au niveau chromosomique et de révéler le transfert de l'information génétique à partir d'ARN à l'ADN génomique dans les cellules de levure.
Résumé
Synthétique courte polymères d'acides nucléiques, les oligonucléotides (oligos), sont les outils les plus fonctionnels et généralisée de la biologie moléculaire. Oligos peuvent être produites pour contenir toute l'ADN ou l'ARN désiré séquence et peut être prêt à inclure une grande variété de base et des modifications de sucre. Par ailleurs, les oligos peuvent être conçus pour imiter altérations spécifiques des acides nucléiques et par conséquent, peuvent servir d'outils importants pour étudier les effets des dommages à l'ADN et les mécanismes de réparation. Nous avons constaté que Thermo Scientific Dharmacon contenant de l'ARN oligos avec une longueur entre 50 et 80 nucléotides peut être particulièrement adapté pour étudier, in vivo, les fonctions et les conséquences de l'ARN chromosomiques / hybrides ADN et de ribonucléotides intégré dans l'ADN. ADN / ARN hybrides peuvent facilement former lors de la réplication de l'ADN, la réparation et la transcription, cependant, très peu est connu au sujet de la stabilité des ARN / ADN dans les cellules hybrides et dans quelle mesure ces hybrides peuvent affecter l'intégrité génétique des cellules. Contenant de l'ARN oligos, par conséquent, représenter un vecteur idéal pour introduire ribonucléotides dans l'ADN chromosomique et de générer des hybrides ARN / ADN de longueur choisie et la composition de base. Nous présentons ici le protocole pour l'incorporation des ribonucléotides dans le génome de la levure système modèle eucaryote / Saccharomyces cerevisiae /. Pourtant, notre laboratoire a utilisé Thermo Scientific Dharmacon contenant de l'ARN oligos pour générer l'ARN / ADN hybrides au niveau chromosomique dans différents systèmes cellulaires, des bactéries aux cellules humaines.
Protocole
Justification
Exploiter l'utilisation de Thermo Scientific oligos Dharmacon, 50 à 80-mers, ici, nous présentons une procédure, basée sur un dosage de la correction de gène, par lequel oligos peuvent transférer l'information génétique de l'ADN génomique des cellules de levure, après recuit à l'ADN cible chromosomique. Le succès de ciblage par oligos est marqué par l'apparition de colonies de levures affichant le phénotype attendu. Si la modification génétique désirée est effectué dans un ou plusieurs ribonucléotides incorporé dans la séquence oligo, l'information génétique découle directement de l'appareil d'ARN à l'ADN cible chromosomique, donc, une forme d'ARN / ADN hybride au niveau chromosomique au cours du processus de ciblage. La ribonucléotide / s d'un Thermo Scientific Dharmacon ARN contenant des oligo peut servir de modèle pour le ciblage génique par synthèse ou de réparation de l'ADN peut être incorporé dans l'ADN et de servir de modèle lors de la réplication d'ADN. Dans ces expériences, nous utilisons la levure / Saccharomyces cerevisiae / système modèle eucaryote, cependant, une approche similaire peut également être appliquée dans d'autres organismes ou types cellulaires. Dans cette vidéo, nous utilisons un Thermo Scientific Dharmacon oligo conçu avec une homologie avec un locus génomique cible de la levure et contenant un ribonucléotide dans le milieu pour corriger une mutation dans le gène cible. Après la transformation avec les oligo-ARN contenant, on observe la correction du site ciblé à une certaine fréquence, ce qui indique que l'appareil de l'ARN oligo pourrait être incorporé dans l'ADN chromosomique et de servir de matrice pour la synthèse d'ADN lors de la réplication d'ADN. L'information génétique portée dans le tractus ARN est stable transmis aux générations suivantes de cellules. Nous décrivons ici les étapes de la transformation de cellules de levure par le Thermo Scientific Dharmacon ARN contenant des oligo et l'approche pour détecter les informations ARN transfert dans l'ADN chromosomique.
A. Conception de l'Oligo-ARN contenant utilisé dans cette expérience
Nous avons conçu un oligo-ARN contenant de corriger un défaut génétique dans la levure S. l'ADN chromosomique cerevisiae au niveau du locus trp5. La souche de levure utilisée dans le protocole contient un gène mutant trp5 avec une délétion de deux de base et une mutation non-sens (figure 1A). Telle est la souche de levure un mutant auxotrophe tryptophane et ne forme pas de colonies sur les médias sans tryptophane. La séquence d'ADN du gène TRP5 est disponible à la base de données du génome de Saccharomyces (http://www.yeastgenome.org/). Le Thermo Scientific Dharmacon ARN contenant des oligo utilisé dans ce protocole est un 65-mer avec une insertion d'ADN 2-base, destiné à corriger la mutation de suppression et d'un ribonucléotide conçu pour corriger la mutation non-sens de l'allèle trp5 (figure 1A). La séquence de l'oligo est conçu comme suit:
5'-AAAAGGGTTTTGATGAAGCTGTCG-CG-GATCCCACATTCTG-RG-GAAGACTTCAAATCCTTGTATTCT. Cet oligo est synthétisée par Dharmacon Thermo Scientific (Lafayette, CO) à 200 nM d'échelle, et est dessalé, déprotégé et utilisé sans purification PAGE.
B. Préparation de l'Oligo contenant de l'ARN pour la transformation de levure (modifié à partir Storici et al., 2007 1)
- Essuyez les matériaux qui seront utilisés pour l'expérience, y compris les tubes oligo, pipettes, vortex, racks, zone d'expérimentation et de gants portés par l'enquêteur avec une solution de décontamination RNase pour enlever la contamination potentielle de RNase avant le début de tout. Utilisez l'eau sans RNase, réactifs chimiques, tubes et embouts de pipette dans toutes les étapes. Chaque étape de cette expérimentation devrait être exempte de RNase.
- Reprendre le Thermo Scientific Dharmacon ARN contenant des oligo reçus de la société à 250 pmoles / ul solution stock d'eau exempte de RNase et vortexer vigoureusement pour dissoudre le culot. Conserver à -80 ° C.
- Juste avant la transformation, le dégel des oligo-ARN contenant de la glace et diluer à 50 pmoles / ul avec l'eau sans RNase dans les tubes sans RNase. Chaque transformation nécessite une nmole des oligos contenant de l'ARN.
- Dénaturer un montant choisi des oligos contenant de l'ARN sur le bloc de 100 ° C chaleur pendant 2 min pour éliminer les structures secondaires des oligos.
- Immédiatement après dénaturation placer le tube sur la glace. Restez sur la glace jusqu'à la transformation.
- Comme le contrôle dans l'expérience un ADN correspondant oligo-seule est utilisée. Cet oligo est décongelé de -20 ° C et préparées pour la transformation comme les oligo-ARN contenant, comme décrit ci-dessus.
Transformation des cellules de levure C. utilisant les oligo-ARN contenant
- Inoculer 5 ml de milieu liquide riche YPD avec les cellules de levure mutante trp5 et croître à 30 ° C pendant la nuit (voir matériaux).
- Transfert 1,5 ml de la culture de nuit dans 50 ml de milieu liquide YPD.
- Incuber les cellules dans un shaker à 30 ° C (225 rpm) pendant 4h.
- Préparer la solution 1et la solution 2 immédiatement avant la transformation de RNase tubes (voir matériaux).
- Transfert de la culture cellulaire à un 50 ml sans RNase tube et tournent à 3000 rpm, ce qui correspond à 1562 g, pendant 2 min. Le culot de la précipitation cellulaire est d'env. 0,3 cm 3.
- Retirer le surnageant et laver les cellules avec 50 ml d'eau exempte de RNase et de spin à 3000 rpm pendant 2 min.
- Répétez l'étape 6 pour 5 fois pour se débarrasser du milieu de culture et RNases qui pourraient être présents dans le milieu autant que possible.
- Retirer le surnageant et remettre les cellules dans 5 ml de solution 1 et tournent à 3000 rpm pendant 2 min.
- Retirer les cellules surnageant et remettre dans 250 ul de la solution 1. Cette quantité de cellules est suffisant pour 7-8 transformations.
- Aliquote de 50 pi de la suspension de cellules dans des microtubes sans RNase, ajouter 20 ul d'ARN contenant une solution d'oligo travail (1 nmole), ou 20 pi de l'ADN seule solution oligo travail (1 nmole), ou 20 pl d'eau stérile sans oligo pour le contrôle négatif. Puis ajouter 300 pi de solution de 2 pour chaque réaction de transformation. Il n'est pas nécessaire d'ajouter d'ADN de sperme de saumon dans le processus de transformation, comme l'acte oligos comme support eux-mêmes.
- Vortex vigoureusement pour mélanger les composants de manière homogène.
- Incuber réactions de transformation à 30 ° C pendant 30 min dans un shaker.
- Choc thermique à 42 ° C pendant 15 min.
- Isoler les cellules à 5000 rpm, correspondant à 2236 g, pendant 4 min.
- Retirer les cellules surnageant et remettre dans 100 pl d'eau stérile.
- Prélever une partie aliquote de cette suspension cellulaire et le diluer avec de l'eau stérile de 100.000 fois et cellules de la plaque sur une plaque YPD utilisant environ. 15 billes de verre stériles et incuber à 30 ° C pendant 2 jours.
- Plaque toutes les cellules en suspension de chaque réaction de transformation sur une boîte de Pétri d'un milieu solide synthèse complète sans tryptophane (SC-Trp) en utilisant environ. 15 billes de verre stériles et incuber à 30 ° C pendant 4-5 jours (figure 2).
D. Analyse de la correction de gène par l'ARN-Oligo contenant
- Comptez le nombre de colonies cultivées sur le milieu sélectif (figure 2) ainsi que sur le milieu YPD pour calculer la fréquence de correction génique pour les oligo-ARN contenant l'ADN oligo-seulement et pour le contrôle sans oligo. Comparez le nombre obtenu. Taux de réversion spontanée de la trp5 allèles avec les suppressions de deux de base et la mutation non-sens est moins à 10-9 dans la souche de levure utilisée, donc nous nous attendons à aucune formation des colonies sur le milieu sélectif quand aucun oligos sont ajoutés aux cellules.
- Streak à plusieurs choisis au hasard sur des colonies transformantes milieu YPD pour obtenir des isolats seule colonie. Attendez deux jours pour la croissance de colonies, puis prendre plusieurs (au moins 5) des colonies isolées et de faire des patchs sur le YPD et sur un milieu sélectif.
- Conception d'une paire d'amorces pour amplifier la région (250-1,000 pb) ciblées par les oligo-ARN contenant par PCR sur colonie (figure 1B). La procédure de PCR sur colonie est comme suit (modifié à partir Storici et Resnick, 2006 2).
- Resuspendre les cellules (environ 1 mm3) de la prise correctifs individuels dans 50 pi d'eau et ajouter 1 unité de lyticase. Incuber à température ambiante pendant 10 minutes, suivie d'une incubation dans un bloc chauffant à 100 ° C pendant 5 min pour briser les cellules et l'ADN génomique communiqué en solution.
- Les conditions de PCR: La réaction de PCR comprend 10 ul de la solution de resuspension cellulaire, 50 pmoles chacune des amorces avant et arrière, 1 pl de 10 mM de dNTPs, 1 unité de Taq polymérase, 5 pi de tampon 10x et est ajusté à l'eau stérile à une volume final de 50 ul. Le programme de PCR est de 3 min à 95 ° C, 30 cycles de 30 s à 95 ° C, 30 s à 55 ° C, et 1 min à 72 ° C, un temps d'extension finale de 7 min à 72 ° C, puis des échantillons sont conservés à 4 ° C. Une extension temps de 1 min / kb est supposé pour cette réaction.
- Après PCR, les échantillons sont exécutés sur un gel à 1% ou 2% (selon la taille attendue du produit de PCR) pour l'observation d'agarose des produits de PCR (figure 3).
- Si l'information génétique transmis par les oligo-ARN contenant génère un nouveau site de restriction dans la région cible de la levure génomique (figure 1B), il est possible de vérifier le transfert correct des informations en digérant le produit PCR avec les enzymes de restriction appropriées. Si aucun site de restriction est généré par les oligo-ARN contenant, passez à l'étape 6. Recueil des produits de PCR utilisant une enzyme de restriction spécifique. La réaction de digestion comprend 6 ul de produit PCR, tampon, BSA (peut ne pas être nécessaire pour certaines enzymes, voir les instructions pour l'enzyme utilisée), 0,5 pi de l'enzyme de restriction et de l'eau stérile à 15 pi. Les échantillons sont incubés pendant 1 h à la température spécifique de l'enzyme utilisée.
- Exécuter un échantillon non digérés avec les échantillons digérés sur la même ligne d'un gel d'agarose à 2%observer les modifications génétiques transférées par le tube de l'ARN oligo-ARN contenant (figure 3).
- Purifier les produits de PCR en utilisant un kit de purification PCR et les préparer pour le séquençage d'ADN. Soumettre les échantillons pour le séquençage avec les mêmes amorces utilisées pour amplifier le produit.
- Analyser les résultats de séquençage de l'ADN en utilisant un logiciel qui permet l'alignement des séquences multiples avec une séquence de référence choisie (figure 4).
E. traitement alcalin de l'oligo-ARN contenant (figure 5)
- Pour chaque réaction, ajouter 1 nmole (4 pi de 250 pmoles / ul solution stock) de l'oligo-ARN contenant, ou de l'ADN-oligo dans un tube de 1,5 ml.
- Ajouter 4 pi de NaOH 1 M pour l'hydrolyse, ou bien ajouter 4 ul H 2 O comme contrôle négatif, et incuber à 65 ° C dans un bain-marie pendant 1 h. Puis passer du bain d'eau à la glace.
- Neutraliser avec 2 pi de 1,2 M HCl, 4 pi de 1 pl M Tris-HCl, et 4 de H 2 O, ou bien 6 ul de H 2 O et 4 pi de 1 M Tris-HCl pour le contrôle négatif. Restez sur la glace jusqu'à la transformation.

Figure 1. Schéma de l'appareil défectueux trp5 gènes et de l'allèle TRP5 corrigé par les oligo-ARN contenant. A) Le gène mutant trp5 contient une délétion 2-base (triangles noirs) et une base de mutation non-sens (astérisque). Le seul brin d'ARN contenant des oligo avec 2 base d'insertion (bleu boucle) et la substitution de base 1-ARN (rectangle rouge) générant un Van91 je place l'enzyme de restriction (indiqué par la tranche) est transformé en cellules de levure à corriger les défauts génétiques du gène trp5. B) Après le gène est réparé TRP5 par les oligo-ARN contenant (bases corrigées sont indiquées par des rectangles bleus), le site Van91 j'ai est généré dans le gène TRP5. Un fragment de 278 pb dont un seul site de restriction Van91 I TRP5 gène est amplifié par PCR par une paire d'amorces (P1 et P2). Le pb 177 pb et 101 fragments générés après digestion de la P1 et P2 PCR produit par Van91 I sont également représentées.
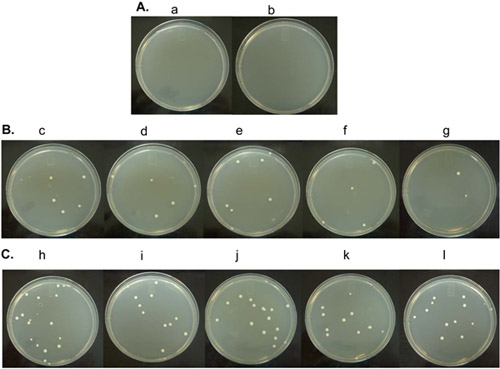
Figure 2. Résultat de la transformation avec les oligo-ARN contenant. A) Les cellules de levure transformées avec des oligo aucune ne forment pas une colonie sur le support SC-Trp, voir les plaques ab. B) Les plaques des colonies de levures cg montrent de plus en plus sur le SC-Trp après que les cellules sont transformées avec 1 nmole des oligo-ARN contenant. C) Plaques colonies hl de levures montrent de plus en plus sur le SC-Trp après que les cellules sont transformées avec 1 nmole de l'ADN uniquement correspondante oligo contrôle.

Figure 3. Détection de transfert d'information génétique de l'ARN contenant des oligo à l'ADN chromosomique de la levure par digestion de restriction du produit de PCR amplifiant la région génomique ciblée. 2% électrophorèse sur gel d'agarose des échantillons amplifiés par PCR par P1 et P2 et digéré par l'enzyme de restriction, je Van91. Les pistes 1, 8 et 15, échelle d'ADN avec des tailles de 100, 200, 300, 400 et 500 pb indiqué sur la gauche; piste 2, le produit de PCR de trp5 locus amplifié à partir de l'ADN génomique de la souche mutante trp5; piste 3, PCR produit amplifié de l'ADN génomique provenant d'une colonie de Trp + ciblés par les oligo-ADN seulement; voies 4-7, les produits de PCR amplifiés à partir de l'ADN génomique provenant Trp + colonies ciblées par les oligo-ARN contenant; piste 9 à 14 ans, Van91 digestion de restriction I des produits de PCR des voies de 2 à 7. La présence des bandes non coupées produit PCR dans les couloirs de 10 à 14 peut être expliquée par une digestion partielle par Van91 I au niveau du locus TRP5 (CCACATTCTGG). Considérant que le site de coupe pour Van91 I (CCANNNN NTGG) peut avoir plusieurs séquences, le site généré en TRP5 ne peut pas être la cible la plus optimale pour l'enzyme. En fait, l'ADN après séquençage de l'ensemble des produits de PCR ci-dessus nous détectons pas de changements supplémentaires à celles réalisées par les oligos (voir Figure 4). Les 278 pb amplifié par PCR la bande P1 et P2 amorces et les bandes de produit de digestion par Van91 I de 177 pb et 101 pb sont indiquées par les flèches sur la droite.
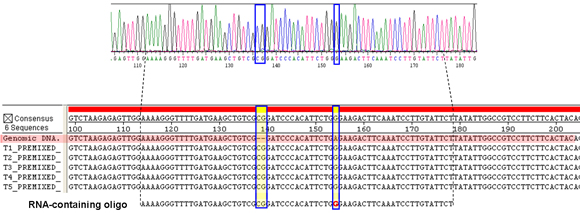
Figure 4. L'ADN montrant les résultats du séquençage du gène de correction par les oligo-ARN contenant. Un électrophérogramme ADN) de la région génomique ciblée par les oligo-ARN contenant. Le G dans la séquence d'ADN (bleu encadré) provient de la RG sur les oligo-ARN contenant. Aussi coffret est l'insertion des bases CG. Résultats du séquençage de tous les produits de PCR autres sont aussi en tant que clear celui-ci, avec des signaux fluorescents bien au-dessus du fond. B) Les séquences de la région TRP5 de l'Trp + transformants ciblés par les oligo ADN seulement (T1) et les oligo contenant de l'ARN (T2-T5) correspondent à la séquence consensus sur le dessus et sont comparés avec celui de la trp5 cellules mutantes avant que le ciblage par les oligos (ADN génomique, rouge ombré). La réparation d'ARN contenant des oligo sur le fond a insertion d'ADN de deux de base et une base (G) la substitution d'ARN marqués en rouge. Les régions encadrées en bleu avec nuance jaune montrent que l'ARN contenant des oligo ainsi que les oligo-ADN que justement corrigé la mutation et la suppression mutation non-sens dans tous les échantillons testés. Les lignes en pointillés marquent la position de la séquence d'ARN contenant des oligo.

Figure 5. Traitement alcalin empêche la correction de gène par l'ARN oligo-contenant. La fréquence de transformation par les oligo-ARN contenant (R) est indiqué dans la barre rouge et que par la oligo ADN uniquement (D) est indiqué dans les barres bleues. Les barres d'erreur représentent l'erreur standard de la moyenne pour les trois transformations indépendantes pour chaque oligo. L'ADN ne oligo affiche la fréquence de transformation similaires avec et sans traitement de NaOH. Différemment, la fréquence de transformation par les oligo-ARN contenant des gouttes à 0 après un traitement par NaOH. Par conséquent, la préparation avec les oligo-ARN contenant n'est pas contaminé par l'ADN ne oligo, par conséquent, la fréquence de transformation observée est spécifique à l'oligo-ARN contenant.
Discussion
Le fait que l'ARN peut transférer l'information génétique directement à l'ADN génomique dans les cellules a été découvert exploiter l'utilisation de l'ARN synthétique contenant oligos (oligos synthétisés Dharmacon) 1. Il était la preuve de principe que les cellules peuvent utiliser l'ARN contenant des molécules ou d'ARN-seulement des séquences en tant que modèles pour la synthèse de l'ADN. L'utilisation d'ARN contenant oligos non seulement conduit à la démonstration que l'information génétique peut être transféré directement à partir de l'ARN à l'ADN génomique sans avoir besoin d'une transcription inverse de copies d'ADN intermédiaires, mais aussi à la preuve que l'ARN peut être utilisé comme modèle dans homologue la réparation d'un dommage 1,3 ADN. Les oligos peuvent être faites de l'ARN-que ou qui contiennent juste un ribonucléotide simple intégré dans une séquence d'ADN, comme dans l'exemple, nous avons présenté ici. Les oligo-ARN contenant a un ribonucléotide embarqué destiné à corriger la mutation non-sens dans le génome défectueux trp5 allèle. Le transfert d'information génétique de la ribonucléotide à l'ADN génomique est révélée par un changement dans le phénotype (cellule en croissance sur le milieu dépourvu de tryptophane) des cellules ciblées. La fréquence de la correction de gène obtenu avec les oligo-ARN contenant est mesurée et comparée avec celle d'un ADN ne contrôlent oligo. En effectuant ce test de correction de gènes dans des cellules différentes origines, où nous muter les gènes de la levure différentes, nous pouvons détecter quels sont les facteurs / s touchent spécifiquement le ciblage par les oligos contenant de l'ARN. Ainsi, nous pouvons révéler les mécanismes de fonctionnement des cellules réguler la stabilité de l'ADN / ARN hybrides. Par une simple conception de différentes variantes de la oligos contenant de l'ARN, nous pouvons déterminer la probabilité pour un appareil particulier, l'ARN pour servir de modèle dans la modification de l'ADN et nous pouvons déterminer la stabilité des séquences d'ARN spécifiques intégrés dans l'ADN. Par ailleurs, nous pouvons identifier quelles sont les préférées des substrats in vivo de facteurs interagissant avec hybrides ARN / ADN.
Alors que plusieurs études in vitro, principalement en utilisant courts ARN contenant oligos, ont été menées pour caractériser la fonction des facteurs qui peuvent reconnaître l'ARN dans un hybride avec l'ADN, telles que les enzymes H RNase 4, les fonctions in vivo de RNases H ainsi que l'identité des autres protéines qui pourraient affecter ARN / ADN stabilité des hybrides demeurent inconnues. La possibilité d'utiliser l'ARN contenant oligos d'une durée significative (50 à 80-mères) et de qualité optimale (telles que Thermo Scientific Dharmacon contenant de l'ARN oligos) a ouvert la voie à l'examen d'un large éventail de processus moléculaires in vivo directement dans le cellules d'intérêt. Comme indiqué dans cette vidéo, la transformation en utilisant l'ARN contenant oligos nécessite essentiellement que quelques étapes supplémentaires par rapport à la transformation à l'aide de molécules d'ADN, afin de prévenir la dégradation par les RNases. Ainsi, la transformation en utilisant l'ARN contenant oligos ne se limite pas à la levure, mais peuvent être appliquées à tout organisme ou un type de cellules où la transformation par l'ADN oligos est compétent.
En conclusion, le ciblage des cellules par l'ARN contenant oligos offre la possibilité de générer des hybrides ARN / ADN et ribonucléotides intégré dans l'ADN in vivo dans les cellules. La stabilité, la fonction et les conséquences de ces in vivo généré hybrides ARN / ADN peuvent être analysés et caractérisés, éventuellement découvrir des mécanismes inconnus de la réparation de l'ADN et de révéler de nouvelles stratégies de ciblage génique.
Déclarations de divulgation
Remerciements
Ce travail a été soutenu par la Coalition contre le cancer en Géorgie accorde-R9028.
matériels
Réactifs Transformation A. et les médias (modifié à partir Storici et Resnick, 2006
- YPD (levure-peptone dextrose): Pour 1 L, 10 g extrait de levure, 20 g de soja peptone, 20 g de dextrose. Ajouter 15 g d'agar pour faire YPD milieu solide. Autoclave avant utilisation. Stocker à température ambiante.
- Solution 1: 0,1 M d'acétate de lithium. Préparer immédiatement avant la transformation. La solution 1 est une solution de travail, il est donc préparée directement à partir de la poudre. Pas de solution stock est faite. Conserver à température ambiante. LiAc augmente la perméabilité de la paroi des cellules de levure à l'ADN.
- Solution 2: 0,1 M d'acétate de lithium et 50% de polyéthylène glycol 4000. Aussi la solution 2 est une solution de travail et elle est faite directement à partir de la poudre. Pas de solution mère est préparée. Conserver à température ambiante. Dépôts PEG oligos sur des cellules de levure.
- Contenant de l'ARN oligos (Thermo Scientific Dharmacon), 50-80-Mers, dessalé, déprotégé et non purifiée. Resuspendre à 250 pmoles / ul. Conserver à -80 ° C.
- ADN ne oligos, 50-80-Mers, dessalé et non purifiée. Resuspendre à 50 pmol / ul. Conserver à -20 ° C.
- SC-Trp (Synthetic milieu complet dépourvu de tryptophane) des milieux solides.
- 0,5 cm de diamètre des perles de verre, stérilisé à l'autoclave.
- RNase-off: la solution de décontamination RNase.
- DNase / RNase, des tubes à centrifuger stériles.
- DNase / RNase, stériles tubes coniques.
- DNase / RNase, stérile en aérosol embouts de pipette avec ZAP: 1-200 ml, 100-1000 ml.
B. Colonie matériaux PCR.
- Amorces d'ADN, dessalé et non purifiée. Dissoudre dans l'eau stérile à 50 pmol / ul. Conserver à -20 ° C.
- ADN-polymérase Taq, tampon, des dNTP.
- Les tubes PCR.
C. PCR purification.
- Kit de purification de PCR.
D. gel d'électrophorèse.
- Agarose.
- 1 x tampon TBE de fonctionnement (45 tétracétate mM Tris-borate et 1 mM éthylènediamine) dilué de TBE 10x.
- Précolorés marqueur de poids moléculaire.
- Colorant de chargement d'ADN.
Digestion de restriction E..
- Les enzymes de restriction, les tampons 10x, BSA.
F. traitement alcalin pour les oligo-ARN contenant.
- 1 M de solution de NaOH.
- 1,2 M de solution de HCl.
- 1 M de Tris-HCl, pH 7,4 solution.
Références
- Storici, F., Bebenek, K., Kunkel, T. A., Gordenin, D. A., Resnick, M. A. RNA-templated DNA repair. Nature. 447, 338-3341 (2007).
- Storici, F., Resnick, M. The delitto perfetto approach to in vivo site-directed mutagenesis and chromosome rearrangements with synthetic oligonucleotides in yeast. Methods Enzymol. 409, 329-345 (2006).
- Storici, F. RNA-mediated DNA modifications and RNA-templated DNA repair. Curr Opin Mol Ther. 10, 224-230 (2008).
- Cerritelli, S. M., Crouch, R. J. Ribonuclease H: the enzymes in eukaryotes. FEBS J. 276, 1494-1505 (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon