Detección de microorganismos ambientales con la reacción en cadena de polimerasa y la electroforesis en Gel de
Visión general
Fuente: Laboratorios del Dr. Ian Pepper y el Dr. Charles Gerba - Universidad de Arizona
Demostrando autor: Bradley Schmitz
Reacción en cadena de polimerasa (PCR) es una técnica utilizada para detectar microorganismos que están presentes en entornos atmosféricos, agua y suelo. Amplificando la secciones específicas de la DNA, PCR puede facilitar la detección e identificación de microorganismos de la blanco a la especie, cepa y serovar/patovar nivel. La técnica también puede ser utilizada para caracterizar comunidades enteras de microorganismos en las muestras.
El cultivo de microorganismos en el laboratorio usando medios de cultivo especializados es una técnica larga y permanece en uso para la detección de microorganismos en muestras ambientales. Muchos microbios en el ambiente natural, mientras viva, mantienen bajos niveles de actividad metabólica y/o duplicación veces y así se refieren como viables pero no cultivables organismos (VBN). El uso de técnicas basadas en la cultura solo puede detectar estos microbios y, por lo tanto, no proporciona una evaluación exhaustiva de poblaciones microbianas en muestras. El uso de la PCR permite la detección de microbios cultivables, organismos VBN, y los que ya no están vivos o activos, como la amplificación de secuencias genéticas no requieren generalmente el enriquecimiento previo de microorganismos presentes en muestras ambientales. Sin embargo, la PCR no puede distinguir los Estados antes mencionados de la viabilidad y actividad. Cuando se combina con una o más técnicas basadas en la cultura, puede aún determinarse la viabilidad de ciertos subconjuntos de los microorganismos.
Principios
La premisa básica de PCR debe utilizar ciclos repetidos de cambio secuencial de temperatura para lograr la amplificación exponencial del ADN. La síntesis de ADN se lleva a cabo por las enzimas de polimerasa de la DNA que se obtienen de bacterias viven en aguas termales, como Thermus aquaticus (Taq). Estas polimerasas son estables, lo que les permite soportar las altas temperaturas utilizadas durante la PCR.
La secuencia de destino, conocida como el amplicón, se amplía de la plantilla de ADN mediante dos tramos cortos de nucleótidos conocidas como "cartillas". Debido a la alta especificidad de unión de cadenas complementarias de ácido nucleico, los iniciadores permiten la amplificación específica de secuencias muy específicas de interés. Mediante el diseño de primers que sólo amplificar una secuencia única (o una combinación de secuencias) de un organismo de interés, PCR puede utilizarse para detectar la presencia de ADN de ese organismo entre todos los materiales genéticos presentan en una muestra ambiental complejo diferencialmente.
Para realizar la PCR, se utiliza una máquina conocida como un termociclador ciclo automáticamente a través de las diferentes temperaturas requeridas para la reacción. Cada ciclo se divide en tres fases. El primero, conocido como "desnaturalización", generalmente se encuentra por encima de 92 ° C y dura unos 30 s. desnaturalización se utiliza para romper las moléculas de ADN en solos filamentos, para permitir la reacción de amplificación para proceder.
La segunda fase, "recocido", se encuentra 2-3 ° C por debajo de la parte inferior de la temperatura de fusión de los dos iniciadores, generalmente entre 50-65 ° C y también dura unos 30 s. temperatura de fusión es la temperatura a la que 50% de la DNA de doble hebra se han separado en solos filamentos, y así el paso de recocido permite que los cebadores enlazar a sus sitios de destino en la plantilla de la DNA.
La tercera fase de un ciclo PCR es el "alargamiento" o la "extensión", cuando la ADN polimerasa se une al duplex primer plantilla y cataliza la síntesis del producto. Situado a 72 ° C por la polimerasa de Taq, la duración de esta fase depende de la longitud del amplicón, generalmente 30 s / 500 bp. Después de cada ciclo, el DNA amplificado es una vez más desnaturalizado y sirve como una plantilla nueva, llevando a un aumento exponencial en la cantidad de productos de la PCR.
Una vez completada la reacción, los productos PCR se pueden resolver por el tamaño de un "gel" generalmente de la agarosa polímero, un proceso conocido como electroforesis. Se aplica un campo eléctrico a través del gel, y las cargas negativas en la columna vertebral de las moléculas de ADN hacer migrar hacia el extremo positivo del campo. En términos generales, moléculas de ADN lineales que son más grandes tomarán más tiempo para viajar a través de la matriz de gel.
Procedimiento
1. muestras
- Recoger el suelo con una barrena o pala a una profundidad determinada. Recogida de suelo de la rizosfera (región estrecha del suelo circundante e influenciado por raíces de las plantas y los microbios asociados), sólo recoger directamente alrededor de las raíces de la planta golpeando el suelo en un barril de la colección.
- Recoger muestras de agua sumergiendo una botella de plástico estéril en el agua sujetando el extremo del palillo que sumerge.
2. los ácidos nucleicos extracción y preparación
- Recoger organismos y virus de la muestra y extraer el ADN y el ARN de ellos. Para obtener más información, consulte la enseñanza de las Ciencias JoVE video de extracción de ácidos nucleicos de comunidad.
- Almacenar el ADN extraído en los tubos del microfuge etiquetados. Si el ADN debe almacenarse durante la noche o durante períodos prolongados de tiempo, congelarlo a-20 ° C y descongelar los tubos a temperatura ambiente cuando esté listo para su uso.
- Si el material genético a ensayarse RNA (sea el genoma de los virus ARN y el ARN transcrito de organismos celulares), realizar la transcripción reversa (RT) en la muestra para crear ADN complementario (cDNA) antes de proceder a la PCR. Para obtener más información, consulte la enseñanza de las Ciencias JoVE video de RT-PCR.
3. reacción en cadena de polimerasa
- Colocar la enzima de la polimerización en cadena (por ejemplo, Taq polimerasa) en el hielo y deshielo de los otros reactivos (tampón PCR, dNTPs, cebadores) dentro de una campana "limpia" designada a temperatura ambiente. La enzima se almacena a-20 ° C pero nunca se congela. Es de sensibles a la temperatura y así que debe estar fresco y su exposición a temperatura ambiente minimizado
- Calcular el volumen de cada reactivo que se necesita para hacer un "master mix" de todos los reactivos que son constantes entre todas las reacciones (tabla 1). Asegúrese de que cuenta para positivos (por ejemplo, una plantilla que contiene la región de destino) y controles negativos (p. ej., no plantilla) en los cálculos. Agregar un 10% adicional al volumen final para tener en cuenta errores de pipeteo. Volúmenes de cartilla dependen de ensayos para microorganismos específicos; Consulte la bibliografía para los valores apropiados.
- Usando una microcentrífuga de unión bajo tubo, que reduce al mínimo la adhesión de moléculas de reactivo a las paredes de plástico, agregar el volumen calculado de cada reactivo para montar la mezcla principal. Suavemente el vortex y centrifugar cada reactivo antes de agregar. Una vez que el amo de la mezcla es preparada, vortex para mezclar y recoge por centrifugación
- Preparar tiras de 8 tubos PCR. Designar un tubo para cada muestra, incluyendo controles positivos y negativos
- Distribuir el volumen apropiado de la mezcla PCR en cada tubo de la tira
- Añadir el volumen adecuado de la plantilla de la DNA de las muestras, así como 5 μL de la plantilla de positivo y 5 μL de agua grado molecular como control negativo, en los respectivos tubos PCR.
- Coloque la tapa firmemente en la franja de 8-tubo y centrifugar unos segundos con un minicentrifuge
- Coloque la tira de 8 tubos en un termociclador
- Establecer el programa PCR apropiado para funcionar en el termociclador. Un programa típico consta de los siguientes
- Desnaturalización a 94 ° C por 3 min.
- 30 – 40 ciclos de amplificación: desnaturalización a 94 ° C por 30 s, recocido a temperatura de cartilla específica (generalmente entre 50 – 60 ° C) durante 30 s y extensión a 72 ° C por 30 s / 500 bp.
- Extensión final a 72 ° C durante 7-10 minutos.
4. preparación del Gel de agarosa
- Basado en el volumen deseado de gel y gel de concentración (tabla 2), pesar la cantidad adecuada de agarosa en polvo en un erlenmeyer de 125 mL.
- Añadir el volumen adecuado de funcionamiento gel tampón en el matraz y agitar el matraz con la mano.
- Calentar la mezcla de buffer-agarosa en un microondas a potencia alta durante 1 minuto.
- Quitar el matraz del microondas y agitar a mano para asegurarse de que la agarosa se disuelva. Si la agarosa no ha disuelto completamente, repita el microondas en incrementos de 30 s.
- Después de asegurar bien la tapa en el frasco, enfriar a 50 ° C girando bajo el chorro de agua fría.
- Añadir 1 μL de bromuro de etidio (EtBr) a la mezcla de agarosa utilizando un micropippette designado para las EtBr. EtBr es un colorante que se une ácidos nucleicos de doble hebra y fluorescencia naranja cuando se ilumina con luz UV. Nota que EtBr es potencialmente cancerígeno, por lo que debe usarse equipo de protección personal (gafas, bata de laboratorio, guantes resistentes de EtBr).
- Verter el gel derretido en una bandeja de fundición del gel de electroforesis. Asegúrese de que no hay burbujas quedan atrapadas dentro de la agarosa. Coloque un peine en el gel y sujete firmemente. Esperar unos 20-30 min para el gel se solidifique.
- Una vez que el gel se ha solidificado, retirar el peine cuidadosamente sin causar cualquier lágrimas en el gel. El peine crea pocillos en el gel para la carga de las muestras.
5. electroforesis
- Coloque el gel de agarosa solidificada en la cámara de electroforesis.
- Añadir el buffer corriente adecuado en la cámara hasta que el gel esté apenas sumergido.
- Sobre un trozo de Parafilm, pipetee puntos de carga de tinte concentrado. También incluye una escalera de DNA de un rango adecuado para los tamaños esperados de los productos PCR. Como alternativa, utilice una nueva serie de tubos de microcentrífuga, uno para cada muestra.
- Una vez completada la polimerización en cadena, recuperar esa franja de tubo de 8 en el termociclador y centrifugar brevemente para recoger cualquier condensados. Añadir un volumen adecuado de producto ADN carga colorante y pipeta hacia arriba y hacia abajo para mezclar. Por ejemplo, 2 μl de 10 x carga colorante se mezcla con 8 μl de la muestra a un volumen final de 10 μl, con el tinte en 1 x.
- Pipetee cada mezcla de tinte-muestra en los pocillos designados en el gel de agarosa, teniendo cuidado de no perforar los pozos.
- Conecte los electrodos a la cámara de electroforesis. ADN está cargado negativamente, por lo que "corre" hacia el electrodo positivo. Por lo tanto, conectar el electrodo positivo hacia el lado opuesto de la cámara, donde fueron cargados a los pozos. Establecer la fuente de alimentación a una tensión apropiada para el sistema de buffer y el tamaño de la cámara de gel y ponerlo a funcionar. Pequeñas burbujas deben ser visibles en movimiento hacia los lados de la cámara si la electroforesis está procediendo correctamente.
- Una vez que el frente de tinte ha avanzado lo suficiente por el gel, apague la fuente de alimentación. Cuidadosamente el gel en el transiluminador o imágenes visuales de transporte y encienda la UV luz para visualizar las bandas de ADN en el gel.
- Analizar el tamaño de banda y las posiciones en el gel. Comparar las posiciones de la banda de las muestras para el control positivo para determinar si el ADN del organismo de interés está presente en la muestra (figura 1).
| Componente | Volumen por tubo (μL) | Volumen para 5 tubos (μL) | Concentración final |
| 10 x Ex buffer Taq | 5.0 | 25 | 1 x |
| dNTPs 2,5 mM | 4.0 | 20 | 0,2 mM |
| Primer avance * | 2.0 | 10 | 400 nM |
| Primer revés * | 2.0 | 10 | 400 nM |
| Molecular H2O | 31.75 | 158.75 | - |
| Ex Taq | 0.25 | 1.25 | 2.5 U |
| Mezcla PCR | 45 | 225 |
Tabla 1. Los volúmenes de reactivos para PCR master mix. * Volúmenes cartilla varían de ensayo del organismo. Ajustar el volumen de agua de grado molecular para que el volumen final 45 μL. Volúmenes de otros componentes no deben variar.
| Recomendada % de agarosa | Resolución óptima para Fragmentos de ADN lineal (pares de bases) |
| 0.5 | 1.000-30.000 |
| 0,7 | 800-12.000 |
| 1.0 | 500-10.000 |
| 1.2 | 400-7.000 |
| 1.5 | 200-3.000 |
| 2.0 | 50-2,00 |
Tabla 2. Gamas del tamaño del fragmento de ADN óptimamente resueltas por gel de agarosa diferentes porcentajes.
Resultados
En la figura 1, la escalera de ADN (carril 1) proporciona una referencia para el tamaño y la concentración aproximada para las bandas de los productos PCR. El control negativo (carril 2) no contiene ningún material genético, mientras que el control positivo (carril 3) es amplificado de plantillas que contienen el ADN para indicar el tamaño y la ubicación de las bandas de blanco Diana. Muestras de 4, 6, 8 y 9 muestran patrón de bandas similar como control positivo, por lo tanto, lo que indica que dichas muestras contienen el material genético de destino. Se puede inferir que el organismo está presente en los ambientes de que se obtuvieron las muestras.
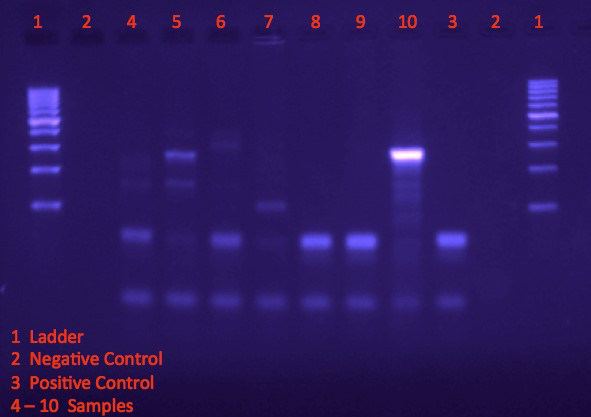
Figura 1. Visualización de bandas en gel de agarosa después de electroforesis.
Aplicación y resumen
PCR se puede emplear para determinar rápidamente la presencia o ausencia de patógenos en el ambiente. Por ejemplo, cartillas específicas para la ameba come cerebro, Naegleria fowleri, amplificar ADN y producir fuertes bandas en un gel si el organismo está presente en una muestra. Si un solo organismo no es el principal interés, sino más bien los genes asociados con la producción de la toxina de una variedad de organismos, PCR puede utilizarse para determinar la presencia o ausencia de estos materiales genéticos específicos.
PCR también puede utilizarse como un procedimiento de confirmación cuando se analizan los microbios ambientales en laboratorio. Si un método de cultivo no puede distinguir entre ciertos organismos que están presentes en una muestra ambiental, entonces PCR utilizado tal vez distinguir específicamente entre los microbios de candidato.
PCR convencional puede modificarse de varias maneras para fines experimentales particulares. PCR puede utilizarse para analizar plantillas de RNA monocatenario por acoplamiento a un paso de transcripción reversa (RT-PCR). Más allá de una determinación de la presencia versus ausencia, PCR cuantitativa (qPCR) puede medir la concentración de ADN específico de interés.
Saltar a...
Vídeos de esta colección:

Now Playing
Detección de microorganismos ambientales con la reacción en cadena de polimerasa y la electroforesis en Gel de
Environmental Microbiology
44.6K Vistas

Determinación del contenido de humedad en el suelo
Environmental Microbiology
359.5K Vistas

Técnica aséptica en ciencias ambientales
Environmental Microbiology
126.4K Vistas

Tinción de Gram de la Bacteria de fuentes ambientales
Environmental Microbiology
100.3K Vistas

Visualizarse los microorganismos del suelo mediante el contacto Deslice análisis y microscopia
Environmental Microbiology
42.3K Vistas

Hongos filamentosos
Environmental Microbiology
57.3K Vistas

Extracción de ADN de la comunidad de colonias bacterianas
Environmental Microbiology
28.8K Vistas

Análisis de muestras ambientales mediante RT-PCR del ARN
Environmental Microbiology
40.4K Vistas

Cuantificación de microorganismos ambientales y virus utilizando qPCR
Environmental Microbiology
47.8K Vistas

Análisis de la calidad del agua a través de organismos indicadores
Environmental Microbiology
29.5K Vistas

Aislamiento de bacterias fecales de muestras de agua por filtración
Environmental Microbiology
39.3K Vistas

Detección de bacteriófagos en muestras ambientales
Environmental Microbiology
40.7K Vistas

Cultivo y enumeración de bacterias a partir de muestras de suelo
Environmental Microbiology
184.5K Vistas

Análisis de la curva de crecimiento bacteriano y sus aplicaciones ambientales
Environmental Microbiology
296.0K Vistas

Enumeración de las algas mediante metodología cultivable
Environmental Microbiology
13.8K Vistas
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados