Method Article
Detección de campos de lentejas para detectar la presencia de marchitez por Fusarium y pudrición de la raíz en Türkiye en el clima terrestre
En este artículo
Resumen
Un estudio en la provincia de Yozgat encontró que los factores bióticos, como las enfermedades fúngicas como la marchitez y la pudrición de la raíz, limitan la producción de lentejas. Se encontraron aislados de Fusarium en el 95,4% de las muestras, lo que sugiere estudios locales periódicos y monitoreo regular para el desarrollo de tecnologías sostenibles y estrategias de control efectivas.
Resumen
La lenteja es una importante planta de cultivo de leguminosas autopolinizada. Su producción está limitada por varios factores bióticos, especialmente los agentes fúngicos causantes del complejo de marchitez y pudrición de la raíz. El estudio tuvo como objetivo comprender la epidemiología regional y la etiología de los agentes fúngicos fitopatógenos para desarrollar estrategias de control contra Fusarium spp. Este estudio investigó 83 localidades de siembra de lentejas en la provincia de Yozgat para enfermedades de marchitez, pudrición de la raíz y de la corona causadas por especies comunes de Fusarium durante 2022 y 2023. Se recolectaron plantas de lenteja sintomáticas para el aislamiento e identificación de hongos. Los aislados de Fusarium se agruparon de acuerdo con la morfología de la colonia y se cultivaron en medio PDA. Además, los ADN genómicos obtenidos de aislados de Fusarium se analizaron mediante PCR y se compararon con otros aislados de Fusarium registrados en el NCBI GenBank. Las relaciones genéticas entre los aislados de Fusarium se determinaron utilizando el método de Máxima Parsimonia (MP) en el programa Mega 11. Los resultados, la incidencia media y la tasa de gravedad de las enfermedades de marchitez y pudrición de la raíz en la provincia de Yozgat fueron de 16,9% y 38,6%, respectivamente. Se encontraron aislados de Fusarium en el 95,4% de las muestras. Hubo una homogeneidad de la secuencia de nucleótidos del 99,5% al 100% entre los aislados de F. oxysporum, F. culmorum, F. graminearum, F. acuminatum y F. solani, y la especie más aislada fue F. oxysporum. El dendrograma MP de los aislados de Fusarium se dividió en dos ramas principales, la primera rama incluyó todos los aislados de F. solani. La segunda rama principal incluyó otras especies de Fusarium aisladas en el presente estudio y en el GenBank del NCBI. El estudio sugiere realizar estudios locales periódicos para determinar la frecuencia de marchitez por Fusarium para su supresión en las lentejas. Se recomienda encarecidamente la supresión oportuna de los daños causados por Fusarium para controlar la enfermedad y conservar el sistema de producción de lentejas.
Introducción
La lenteja (Lens culinaris Medik.), una leguminosa de grano comestible pequeño que pertenece a la familia Fabaceae, es un cultivo de estación fría que se autopoliniza con hojas en forma de aguja y flores de color blanco a púrpura pálido o púrpura oscuro1. Fue domesticado por los humanos hace unos 10.000 años en la parte mesopotámica del Creciente Fértil y se extendió rápidamente al Nuevo Mundo, incluyendo la cuenca mediterránea y Asia Central, y más tarde se naturalizó en lasAméricas. La superficie mundial de cultivo de lentejas es de unos 5,5 millones de hectáreas con una producción de 6,6 millones de toneladas3. Türkiye ocupael 4º lugar en producción de lentejas después de Canadá, India y Australia. El cultivo de lentejas en Türkiye es muy importante y representa el 6,7% de la producción mundial. La producción total de lentejas de Türkiye es de 474.000 toneladas y se produce en al menos 40 provincias4. Alrededor del 89,5 por ciento de la producción de lentejas de Türkiye son lentejas rojas y verdes, que constituyen el 10,5 por ciento de la cosecha de invierno en la región de Anatolia sudoriental. El resto de la cosecha se cultiva como cultivos de verano. Las provincias de Yozgat (39,5 %), Konya (23,7 %), Kırşehir (16,3 %), Çorum (7,6 %) y Ankara (2,9 %) contribuyen en gran medida a la producción de lentejas verdes4. La producción de lentejas puede verse limitada por factores de estrés bióticos y abióticos. Las heladas y la sequía son los factores de estrés abiótico más comunes en la producción de lentejas verdes en verano5. Las enfermedades fúngicas como la marchitez, la raíz y el complejo de pudrición de la corona causadas por las especies Ascochyta lentis, Rhizoctonia solani, R. bataticola, Aphanomyces euteiche, Pythium y Fusarium son las enfermedades fúngicas más importantes, que causan una combinación de enfermedades que incluyen marchitamiento, tizón de las plántulas, marchitez y pudrición de la raíz, según el momento de la infección, la susceptibilidad del huésped y las condiciones meteorológicas 6,7, 8 de la Constitución.
Fusarium es un hongo filamentoso imperfecto que se encuentra en el suelo, las plantas y los sustratos orgánicos y es un género cosmopolita entre estos patógenos9. Causa diversas enfermedades como la marchitez por Fusarium, la pudrición de la raíz y el cuello de la raíz, así como el tizón de la espiga por Fusarium en el trigo, el marchitamiento por Fusarium en las cucurbitáceas y la pudrición de la raíz en la mayoría de las legumbres, incluidas las lentejas 10,11,12. La marchitez vascular, la pudrición de la raíz y del cuello de la raíz causada por Fusarium spp. es la enfermedad más importante de las lentejas en muchas áreas de cultivo de lentejas a nivel mundial10. Fusarium oxysporum es la especie de Fusarium más común asociada con la marchitez, la pudrición de la raíz y el cuello de la raíz en las lentejas. A nivel mundial, las enfermedades de marchitez, pudrición de la raíz y de la corona son causadas por F. graminearum, F. sporotrichioides, F. equity, F. acuminatum, F. redolent, F. avenaceum, F. culmorum, F. solani y F. verticillioides en áreas de plantación de lentejas7. Las enfermedades de marchitez, pudrición de la raíz y de la corona causadas por Fusarium spp. ocurren tanto en la etapa de plántula como en la adulta y causan marchitamiento repentino, secado y eventual muerte de las hojas. Los síntomas de la enfermedad incluyen pudrición de la semilla, pudrición de la raíz, marchitamiento de los folíolos superiores, retraso en el crecimiento, encogimiento y enroscamiento de las hojas. En las etapas intermedias y tardías de llenado de las vainas, las semillas generalmente se encogen y los síntomas de la raíz incluyen retraso en el crecimiento, decoloración marrón, puntas de raíz principal dañadas y proliferación de raíces secundarias. La decoloración del tejido vascular puede no observarse en todos los casos13.
En la región de Anatolia Central, se han realizado estudios sobre el estado de las enfermedades de marchitez y pudrición de la raíz en las lentejas en cantidades limitadas. Yozgat tiene un clima suave y moderado con abundantes precipitaciones en invierno en comparación con el verano y está clasificado como Dsb (Clima terrestre cálido y húmedo) por Köppen y Geiger14. La temperatura media es de 9,6 °C con una precipitación media de 512 mm. Yozgat se encuentra en el hemisferio norte. El verano ocurre en junio, julio, agosto y septiembre. Es muy importante contar con información sobre la epidemiología regional y la etiología de los agentes fúngicos fitopatógenos causantes de la enfermedad para desarrollar diferentes estrategias de control contra Fusarium spp. transmitido por el suelo, para el control de la enfermedad15. En este contexto, los objetivos del presente estudio son determinar e identificar los parámetros de la enfermedad (prevalencia de la enfermedad, incidencia y gravedad) de las enfermedades de marchitez, pudrición de la raíz y de la corona en lentejas mediante la realización de una encuesta en la provincia de Yozgat, donde aproximadamente el 40% de la producción total de lentejas verdes se realiza individualmente, el patógeno Fusarium especies que causan marchitez y pudrición de la raíz en las lentejas mediante análisis morfológicos y moleculares, y determinar los niveles de virulencia individuales de las especies de Fusarium mediante la realización de pruebas de patogenicidad.
Protocolo
NOTA: Los detalles de los reactivos y el equipo utilizado en el estudio se enumeran en la Tabla de Materiales.
1. Estudio de campo, muestreo y aislamiento de hongos
NOTA: En 2022 y 2023 se realizaron trabajos de relevamiento, según Endes16. Se observaron un total de 83 áreas de plantación de lentejas en nueve distritos de la provincia de Yozgat para la enfermedad de marchitez, pudrición de la raíz y del cuello de la raíz (Figura 1).
- Seleccione campos de lentejas con un área de más de 1000m2 como área de muestreo. Recoja cada muestra caminando aleatoriamente desde el borde hasta el centro o centro del campo en zigzags caminando a lo largo de las diagonales usando un marco de 1 m2 , colocándola aleatoriamente en un mínimo de tres puntos diferentes seleccionados al azar. Recoja las plantas de lentejas que muestren síntomas de enfermedad en cada punto, colóquelas en bolsas de papel y transfiera al laboratorio para su uso en estudios de aislamiento e identificación de hongos.
NOTA: Las raíces y los cuellos de las plantas de lentejas enfermas transferidas al laboratorio se lavaron primero con agua del grifo para eliminar los residuos gruesos; posteriormente, fueron sometidos a la desinfección de superficies para el aislamiento de los patógenos fúngicos según lo descrito por Endes16. - Remoje los tejidos vegetales enfermos en etanol al 70% durante 10-15 s, y luego enjuague 3 veces durante 3 minutos cada uno en agua estéril. Manténgalos todos en hipoclorito de sodio (NaOCl) al 1% durante 5 minutos y enjuague 3 veces durante 5 minutos cada uno nuevamente con agua estéril.
- Seque los tejidos vegetales húmedos en papeles de filtro en un gabinete estéril para finalizar el proceso de desinfección de superficies. A continuación, corte los tejidos vegetales desinfectados en trozos de 5-10 mm de largo y coloque 4-5 trozos en un medio PDA que contenga un 0,01% de estreptomicina dentro de placas de Petri (90 mm de diámetro). Coloque las placas de Petri en una incubadora en la oscuridad a 25 ± 1 °C durante 4-7 días y observe el crecimiento de hongos.
NOTA: Los aislados de hongos en desarrollo se purificaron mediante el método de aislamiento de esporas únicas. Para ello, se modificó y utilizó el estudio realizado por Choi et al.17 sobre la obtención de cultivos de esporas individuales de hongos pertenecientes a Ascomycetes, Basidiomycetes, Coelomycetes e Hyphomycetes como se describe a continuación. - Para purificar los aislados de hongos, mantenga los aislados de hongos obtenidos al final de los estudios de aislamiento en una incubadora a 25 ± 1 °C durante 12 h de luz fluorescente / 12 h de oscuridad durante 15 días para favorecer la formación de estructuras de reproducción anamórficas en PDA.
- Pese alrededor de 100 mg de micelio de hongos de cultivos de 15 días con una espátula, transfiéralo a un tubo de microcentrífuga estéril de 1,5 ml y luego muélalo completamente con morteros de plástico estériles para homogeneizarlo.
- Agregue 1 mL de agua estéril y vórtice durante 1 min para asegurar la transferencia de esporas al agua. Para ajustar el número de esporas que se transfieren al agua, extraiga 20 μL de esta mezcla con una pipeta y compruebe el número de esporas con un aumento de 10x del microscopio óptico.
- Cuando la cantidad de esporas sea mayor que la cantidad deseada, diluya la mezcla de esporas y agua en proporciones como 1/10, 1/100 y 1/1000. Proporcione una mezcla que contenga de 4 a 6 esporas en el campo de visión del microscopio.
- Tome 100 μL de la suspensión de esporas preparada y transfiérala a placas de Petri de 90 mm de diámetro que contengan medio PDA suplementado con estreptomicina al 0,1%. A continuación, extienda la suspensión transferida sobre el PDA con una espátula Drigalski.
- Incubar las placas de Petri preparadas en la oscuridad a 25 ± 1 °C durante 12-24 h. Al final de este período, transfiera pequeños trozos de hifas desarrollados a partir de un solo conidio con un asa de inoculación a una nueva placa de Petri que contenga medio PDA. Cada cultivo obtenido de cada espora es un cultivo de una sola espora. Almacenarlos para utilizarlos en pruebas de patogenicidad e identificación morfológica y molecular.
NOTA: El objetivo principal es mantener vivos los aislados de hongos durante mucho tiempo sin cambiar su morfología, genética y virulencia. El proceso de almacenamiento se llevó a cabo utilizando dos métodos diferentes18,19. Todos los métodos de almacenamiento utilizados en este estudio se explican en detalle a continuación.- El primer método para almacenar muestras es el siguiente. Cultivar aislados de hongos en medio PDA a 25 ± 1 °C con un 12:12 h oscuridad: luz durante 15 días para obtener discos de micelio de 4 mm de diámetro que podrían almacenarse en agua estéril.
- Cortar discos de micelio (4 mm de diámetro, 10 discos) con un barrenador del corcho de cultivos fúngicos que crecieron en las condiciones antes mencionadas. Transfiera los discos de micelio a tubos de microcentrífuga que contengan 1 mL de agua estéril. Mantener las muestras en tubos de microcentrífuga en el frigorífico a -20 °C durante 6 meses.
- El segundo método para almacenar muestras es el siguiente. En primer lugar, se toma una pizca del disco fúngico puro de los cultivos de esporas individuales obtenidos con un asa de inoculación y se transfiere a placas de Petri que contienen PDA suplementado con cloranfenicol, ácido láctico, ampicilina, rifampicina, tetraciclina, estreptomicina, etc.
- Cultivar durante 5-10 días antes del proceso de almacenamiento a 25 ± 1 °C con una condición de 12 h: 12 h oscuridad: luz. Corte y esterilice papeles de filtro de uso general de 1 cm x 1 cm en un autoclave a 121 °C, 15 psi durante 60 min.
- Coloque los papeles de filtro en placas de Petri nuevas con el mismo medio o selectivo. Corta colonias/esporas de cultivo fúngico puro y colócalas en la parte superior de cada pieza.
- Selle las placas de Petri y colóquelas en una incubadora en condiciones de crecimiento adecuadas (como se mencionó anteriormente). Los aislados de hongos crecen lentamente en el papel de filtro. Incubar durante aproximadamente 15 días para asegurar una colonización completa.
- Después de la esporulación o la formación completa de colonias en papeles de filtro, transfiera los trozos de papel individuales a una nueva placa de Petri sin un medio de cultivo. Posteriormente, coloque las placas de Petri en la incubadora hasta que el papel de filtro y los hongos estén completamente secos (aproximadamente 20-30 días).
- Después del secado, coloque 10 trozos de papel de filtro en cada sobre de papel estéril, etiquete cada sobre, coloque estos sobres en bolsas de plástico y guarde las bolsas de plástico que contienen los sobres en un recipiente de plástico y transparente a -20 °C.
- Calcule la tasa de prevalencia de la enfermedad de marchitez de las lentejas, la raíz y la pudrición de la corona en Yozgat de acuerdo con la fórmula que se da a continuación, considerando la prevalencia de la enfermedad en cada campo de lentejas, seguido del nombre del distrito de la provincia.
Tasa de prevalencia de la enfermedad (%) = (a / b) x 100
donde a indica el número de campos enfermos; b Indica el número total de campos topográficos en un distrito. - Calcular la incidencia de la enfermedad de acuerdo con el método de promedio ponderado reportado por Bora y Karaca20. Cuenta las plantas de cada cuadrado del campo. Sepárelas en plantas enfermas y no enfermas en cada cuadrado y calcule la prevalencia de enfermedades de acuerdo con la fórmula que se da a continuación.
Tasa de prevalencia de la enfermedad (%) = (x / y) x 100
donde x indica el número de plantas enfermas; y indica el número total de plantas encuestadas. - Calcule la gravedad de la enfermedad de acuerdo con la escala 0-4 de Öğüt21, donde 0 = no mostró síntomas, 1 = síntomas de nivel leve en el 25% de las hojas; 2 = síntomas de nivel moderado en el 26%-50% de las hojas; 3= clorosis severa y marchitez en el 51%-75% de las hojas; y 4 = clorosis muy severa y síntomas de marchitamiento o desecación, desprendimiento, retraso del crecimiento u hojas muertas en más del 75% de las plantas en el mismo orden (Figura 2).
- Calcule la gravedad de la enfermedad utilizando la siguiente fórmula con los valores de escala obtenidos16.
Gravedad de la enfermedad (%) = [∑ [ i (ni x vi) / (V) x N)] x 100
donde ni número de plantas en el valor de la escala; VI Valor de la escala; V valor de escala más alto; N número total de plantas observadas; i indica el número de clases.
2. Datos meteorológicos
- Realizar estudios de encuestas. Para este protocolo, se realizaron encuestas durante los periodos de mayo y junio de 2022 y 2023. Obtenga los valores de temperatura, humedad relativa y precipitación total para el período marzo-julio en 2022, 2023 y años largos de la Dirección de Meteorología Provincial en Yozgat (Tabla 1).
3. Identificación morfológica
- Comparar las características culturales (color de la colonia, micelio aéreo, tasa de crecimiento de micelios) y conidios (dimensiones de los conidios, forma, color y número de tabiques) de los aislados de Fusarium con las de estudios anteriores e identificar tentativamente las especies fúngicas.
- Seleccionar muestras representativas de los grupos que se utilizarán en la identificación morfológica. Purifique estas muestras de acuerdo con el método de aislamiento de una sola espora como se mencionó anteriormente. Durante este período, observe las características de pigmentación de las colonias de aislados de Fusarium , así como las estructuras de micro/macroconidios y clamidosporas.
- Para promover la formación de estructuras micromorfológicas como micro/macroconidios y clamidosporas en los cultivos de hongos puros obtenidos, incubar en PDA a 25 ± 1 °C y 12 h: 12 h oscuridad: luz durante 25-30 días en una incubadora. Mida la longitud y el ancho de los conidios para cada aislado de Fusarium mediante microscopía óptica. Además, documente la estructura, la forma, el color y los septos o sin septos de los conidios utilizando un microscopio óptico suministrado con una cámara digital.
- Con base en las observaciones anteriores, agrupe los aislados de Fusarium según el nivel de especie, como se describe en Leslie y Suerell22.
4. Identificación molecular
NOTA: El ADN genómico total de los aislados de Fusarium se extrajo utilizando el siguiente método, que fue ligeramente modificado del protocolo de Cenis23. Los análisis de PCR y electroforesis de los aislados de Fusarium se realizaron utilizando el protocolo descrito por Aras y Endes24.
- Extracción de ADN genómico fúngico
- A partir de un cultivo fresco (de 10 días de antigüedad) de aislados de Fusarium cultivados en PDA, raspe 100 mg de micelio con un bisturí estéril y luego transfiéralo a un tubo de microcentrífuga de 2 mL. Incubar los tubos a -20 °C durante la noche.
- Después de la incubación durante la noche, agregue 500 μL de tampón de extracción de ADN (200 mM de Tris-HCl pH: 8,5, 250 mM de NaCl, 25 mM de EDTA, 0,5% de dodecil sulfato de sodio) en los tubos y triture con un mortero de plástico estéril.
- A continuación, añadir 150 μL de acetato de sodio 3M (NaOAc) pH 5,2 a los tubos e incubar a -20 °C durante 30 min. Después de esta etapa, centrifugar los tubos a 4.000 x g durante 10 min.
- Después de la centrifugación, transfiera 400 μL del sobrenadante en la parte superior del tubo a tubos nuevos (1,5 mL) y agregue un volumen igual de isopropanol (2-propanol). Incubar los nuevos tubos a -20 °C durante 30 min. Durante este tiempo, mezcle suavemente los tubos 5 o 6 veces.
- Centrifugar a 4.000 x g durante 10 min para precipitar el ADN genómico y desechar todo el líquido que quede en los tubos.
- Agregue 1 mL de etanol al 70% a la bolita de ADN genómico como un sedimento blanco o de color crema en el fondo del tubo. Mezcle suavemente el tubo hacia arriba y hacia abajo 4x-5x durante aproximadamente 1 min, desechando todo el etanol en los tubos. Luego, abra los tubos en flujo de aire laminar durante 30 minutos para evaporar completamente el etanol en la pastilla de ADN.
- Para disolver el ADN genómico y almacenarlo durante mucho tiempo, añada 50 μL de solución tampón TE (1M Tris-HCl pH: 8,0; 0,5M EDTA pH 8,0) a los tubos e incubar los tubos en un baño de agua a 65 °C durante 1 h. Durante este tiempo, mezcle suavemente el tubo hacia arriba y hacia abajo de 8 a 10 veces. Almacene el ADN genómico disuelto en una solución tampón TE en un congelador a -20 °C para su uso en estudios moleculares.
- Para los estudios de PCR, utilice cebadores de oligonucleótidos ITS5 F (5'GGAAGTAAAGTCGTAACAAGG3') / ITS4 R (5'TCCTCCGCTTATTGATATGC3') para amplificar la región ITS del ADNr25. Realice cada reacción de PCR con 2,5 μL de tampón de PCR 10x, 2,5 μL de MgCl2 (25 mM), 2,5 μL de dNTP (2 mM), 0,5 μL de cada cebador (10 μM), 2 μL de ADN molde, 0,5 μL de ADN polimerasa Taq (1 U/μL, Fermantas) y 14 μL de sQH2O en un volumen total de 25 μL.
- Para la reacción de PCR de 25 μL, ejecute el siguiente programa de PCR a 95 °C durante 2 min (desnaturalización inicial), seguido de 40 ciclos; 95 °C durante 30 s (desnaturalización), 55 °C durante 45 s (recocido), 72 °C durante 90 s (extensión) y 72 °C durante 5 min (extensión final). Productos de PCR electroforesa durante 1,5 h a 90 V en gel de agarosa al 1,5% preparado en 1x solución tampón TAE (Tris Base - Ácido Acético Glacial - EDTA).
- Para preparar el tampón TAE para la electroforesis en gel, disuelva 242 g de base Tris en 700 ml de agua estéril; añadir los 57,1 mL de ácido acético glacial; añadir los 100 mL de solución de EDTA 0,5 M y ajustar el volumen a 1 L añadiendo agua estéril. Ajuste el pH final del tampón TAE de 1 L 50x a 8,5. Para hacer el tampón de trabajo 1x TAE, agregue 49 partes de agua estéril a 1 parte del tampón TAE 50x.
- Tiñir los geles con bromuro de etidio de 0,5 μg/mL e inspeccionarlos visualmente haciéndolos visibles en un transiluminador UV.
- Con el fin de examinar la relación filogenética entre los aislados de agentes de pudrición de la raíz y de la corona, se obtuvieron las secuencias de bases del gen ITS por PCR, que se sintetizaron bidireccionalmente (5'-3' y 3'-5') a través de un proveedor. Comparar las secuencias de bases con los datos genéticos del sitio web del NCBI (Centro Nacional de Información Biotecnológica) y las secuencias de bases del gen ITS de otros aislados de Fusarium en el mundo utilizando el programa Blast. Utilícelo para identificar los aislados a nivel de especie como se describe a continuación.
- Ve a https://www.ncbi.nlm.nih.gov sitio web. Haga clic en la pestaña BLAST en la sección Recursos populares.
- Haga clic en la pestaña Nucleotide BLAST en la nueva ventana. En la sección Ingresar secuencia de consulta en la nueva ventana, ingrese las secuencias base en formato Fasta y escriba el nombre del estudio en la sección Título del trabajo.
- A continuación, marque Bases de datos estándar (nr, etc.) en la pestaña Base de datos en la sección Elegir conjunto de búsqueda en la parte inferior.
- Marque Secuencias muy similares (megablast) en la pestaña Optimizar para en la sección Selección de programa y haga clic en la pestaña BLAST en la parte inferior de la página.
- Utilice el programa de análisis filogenético MEGA 11 para determinar la relación filogenética entre los aislados de Fusarium . Alinear las secuencias de bases utilizando el programa ClustalW y crear los árboles genealógicos genéticos de los aislados según la máxima parsimonia para el gen ITS26.
- Para descargar el software de Mega, vaya al sitio web https://www.megasoftware.net e instale el software de Mega. El software MEGA se proporciona GRATIS para su uso en investigación y educación.
- Primero, guarde las secuencias en el escritorio como un archivo de bloc de notas (.txt) en formato FASTA. Ejecute el programa de software Mega y haga clic en la pestaña ALIGN . Haga clic en Editar/Construir alineación en la ventana. A continuación, marque Crear una nueva alineación en la nueva ventana y haga clic en Aceptar para confirmar.
- Haga clic en la pestaña ADN . Suprímase el 1. Secuencia que aparece automáticamente en la ventana, y vaya a Editar archivo, luego haga clic en la pestaña Insertar secuencia desde archivo . Abra el archivo del Bloc de notas (.txt) que contiene las secuencias y que se encuentra en el escritorio.
- En esta etapa, todas las secuencias aparecen en la pantalla. Primero, haga clic en Cualquier secuencia que aparezca en la pantalla, luego marque todas las secuencias con CTRL + A. Abra el archivo de alineación y haga clic en Alinear por Clustal W desde allí, y haga clic en Aceptar para ejecutar el programa en la configuración predeterminada.
- Después de examinar las diferencias en la alineación de las secuencias, vaya al archivo de datos, haga clic en Análisis filogenético y luego haga clic en No en la casilla de verificación de la ventana para ver si las secuencias sintetizan proteínas o no. Nuestras secuencias no sintetizan proteínas porque pertenecen a la región ITS.
- Regrese a la ventana principal del software Mega. Haga clic en Filogenia y seleccione Construir/Probar árbol (s) de parsimonia máxima. En la nueva ventana, seleccione Método de arranque para la prueba de filogenia e introduzca los valores de arranque 1.000 para probar la fuerza de la bifurcación. En la pestaña Tratamiento de datos faltantes, seleccione Eliminación parcial, seleccione Subtree-prupuping-Regrafting (SPR) como método de búsqueda MP y haga clic en Aceptar para confirmar las operaciones.
- Espere a que el resultado del análisis muestre el árbol filogenético.
5. Prueba de patogenicidad
- Utilice cuatro aislados para estudios de patogenicidad para cada especie representativa de las especies de Fusarium que se identificaron por métodos moleculares. Realizar estudios de patogenicidad a 24 °C, 16 h de luz fluorescente/8 h de fotoperiodo oscuro, con 65% de humedad en una habitación climatizada.
- Siembre semillas de lentejas en viales de plástico negro con 45 agujeros de 5 cm de diámetro. Mantenga cada vial en hipoclorito de sodio al 1% durante 3 minutos y luego enjuague con agua destilada estéril 3 veces. Secar las semillas en un armario estéril durante 24 h y sembrar con una semilla en cada hoyo.
NOTA: Las semillas de lenteja de la variedad Kayı 91, sensibles a la enfermedad, se utilizaron en todas las pruebas de patogenicidad6. Como método de inoculación se utilizó la técnica de inmersión de plántulas27. - Incubar cultivos puros de cada aislado en PDA a 24 °C durante 7-10 días. Raspe las colonias cultivadas a partir del cultivo madre de la superficie del medio con una espátula y prepare la suspensión de esporas/micelio con agua destilada estéril.
- Elimine los residuos grandes de la suspensión mediante filtración a través de una gasa de 4 capas y ajuste la concentración de esporas/micelio a 1 x 106 esporas/mL con la ayuda de un hemocitómetro.
- Después de esta etapa, arranque las raíces de las plántulas previamente cultivadas en los viales cuando tengan 2-3 hojas verdaderas. Lavar con agua del grifo y herir ligeramente las raíces con una aguja estéril. Sumerja estas plántulas en la suspensión de esporas/micelio preparada durante 3 minutos y luego trasplántelas a viales de plástico que contengan una mezcla estéril de tierra/turba (2:1; v/v).
- Para las plántulas utilizadas como controles, arranque sus raíces, lastúrelas y luego plántelas sumergiéndolas solo en agua estéril. Realizar evaluaciones de las pruebas de patogenicidad 3 semanas después del proceso de inoculación según la escala 0-4.
- Después de que los valores de severidad de la enfermedad calculados se sometieron a transformación de ángulo, se sometió los valores obtenidos a un análisis de varianza y se evaluaron las diferencias entre las medias según la prueba HSD de Tukey (p = 0.05). Severidad de la enfermedad: Los aislados con 0%-15% se evaluaron con muy baja virulencia (LV), los aislados con 16%-35% se evaluaron como baja virulencia (LV), los aislados con 36%-50% se evaluaron como virulencia moderada (O), los aislados con 51%-70% se evaluaron como alta virulencia (VV), los aislados con 71%-100% se evaluaron como muy alta virulencia (VV) y los aislados sin síntomas de enfermedad se evaluaron como aislados saprófitos o epífitos.
Resultados
Determinación de los parámetros de la enfermedad
Se evaluaron un total de 83 áreas de siembra de lentejas que cubrían nueve regiones diferentes de Yozgat en términos de marchitez, raíz y síntomas de pudrición de la corona, extendiéndose sobre un área de 1.1984 x 106 m2 (Tabla 2). Se encontraron síntomas de marchitez o pudrición de la raíz en todos los campos. Sin embargo, la incidencia de la enfermedad de marchitez y pudrición de la raíz en Yozgat se determinó en 16,9%, con una gravedad de la enfermedad del 38,6% en los distritos de Sorgun y Sarıkaya. También se determinó una incidencia considerable de la enfermedad en los distritos de Şefaatli (26,4%), Boğazlayan (23,0%) y Sorgun (20,1%). Paradójicamente, el porcentaje más alto de gravedad de la enfermedad se determinó en el distrito de Sorgun, con un 45,2%, seguido de Boğazlayan, con un 36,0%, y Sarıkaya, con un 35,4%. Además, 679 plantas mostraron síntomas de enfermedad en muestras recogidas de los campos de lentejas de las áreas marcadas examinadas (Tabla 3). A continuación, se procedió a la identificación morfológica de los agentes fúngicos aislados distribuidos en dos grupos. Los aislados de Fusarium clasificados en el primer grupo incluyeron patógenos o saprófitos como Alternaria sp, Ascochyta sp. y Rhizoctonia sp. El porcentaje de tasa de aislamiento de aislados de Fusarium fue del 95,4%. Se determinó que F. oxysporum es la especie de hongo más aislada de las plantas de lentejas que muestra síntomas de enfermedad en Yozgat con un porcentaje de tasa de aislamiento de 59.5%. Esta especie patógena de Fusarium fue seguida por F. graminearum (15,8%) y F. culmorum (10,2%). Paradójicamente, F. solani (4,4%) y F. acuminatum (5,5%) se aislaron en niveles más bajos de plantas que mostraron síntomas de enfermedad. Se obtuvo F. oxysporum de todos los distritos donde se realizó el estudio de encuesta, y el porcentaje de tasa de aislamiento según los distritos se distribuyó entre 45.3% y 72.2%. F. oxysporum se aisló más en los distritos Central (72,2%), Şefaatli (71,9%), Akdağmadeni (68,3%) y Sorgun (62,2%) de Yozgat. En contraste, F. solani y F. acuminatum fueron las especies de Fusarium menos aisladas en Yozgat. F. solani no se limitó a los distritos de Çekerek y Central; F. acuminatum tampoco se aisló en los distritos de Akdağmadeni, Central y Şefaatli (Tabla 3).
Identificación morfológica
Los cultivos puros de aislados de Fusarium se identificaron morfológicamente de acuerdo con los caracteres de sus colonias, así como con las estructuras de microconidios, macroconidios y clamidosporas. Los estudios de identificación se realizaron a nivel de especie, según Leslie y Summerell22. Todos los aislados de hongos obtenidos de plantas que mostraron síntomas de marchitez, pudrición de la raíz y pudrición del cuello de la raíz en las lentejas se recolectaron en seis grupos de acuerdo con su colonia y micromorfología (Tabla 3). Mientras que los primeros cinco grupos incluían aislados pertenecientes a especies de Fusarium , el otro grupo incluía aislados de hongos patógenos distintos del género Fusarium , como Alternaria, Rhizoctonia y Ascochyta.
Los aislados de F. oxysporum con la tasa de aislamiento más alta tienen colonias de color blanco a amarillo y pigmentación de color lila púrpura. Los macroconios son de longitud corta a mediana, ligeramente curvados, y suelen tener de 3 a 5 septos. La estructura de las esporas de algunos aislados es ligeramente ganchuda, con unas dimensiones de macroconidios determinadas como 33,8 a 71,5 μm x 3,1 a 4,5 μm. Los microconidios son generalmente no septados, ovalados, elípticos o en forma de riñón. La formación de clamidosporas se produjo lentamente (4-6 semanas). Se observó en grupos dobles y una estructura de cadena corta16.
La segunda especie con la tasa más alta de aislamiento, los aislados de F. graminearum , tienen micelio aéreo de color blanco rosado y pigmentación de color rojo oscuro. Los macroconidios son delgados, rectos o ligeramente curvados, con cinco a siete septos. Los septos son bastante distintos. Tiene una célula apical puntiaguda y una célula basal claramente en forma de pie. Las dimensiones son de 25,7 a 97,3 μm x 3,5 a 5,5 μm. No se observaron microconidios ni formación de clamidosporas28.
Las colonias de aislados de F. culmorum en el tercer grupo fueron inicialmente blancas, pero con la edad se observó una estructura micelial de color rosa claro a rosa oscuro. Los macroconidios son cortos, 4-6 septados y ligeramente curvados. Dimensiones de las macrosporas; 15,8 - 60,0 μm x 3,2 - 5,1 μm. Son numerosos. No se observaron microsporas. La formación de clamidosporas es rápida en comparación con otras especies (3-5 semanas). Se encuentran solos o en grupos de dos29.
Las colonias de la cuarta especie, aisladas de F. acuminatum , son de color naranja pálido, naranja y burdeos claro. Es una especie de crecimiento relativamente lento. Sus macroconidios son delgados, tienen una curvatura distinta y tienen una estructura septada de 3 a 5. Las dimensiones de los macroconidios se determinaron como 31.0-65.5 × 4.3-6.6 μm. Los microconidios tienen 0 y 1 septados. Se observó raramente en imágenes microscópicas. La formación de clamidosporas es muy lenta (más de 6 semanas). Se forma en cadenas y racimos22.
El color de la colonia de la última especie patógena identificada, el aislado de F. solani , es blanco y de color crema. Sus macroconios son anchos, planos y ligeramente curvados. Las hifas son de 3 a 7 septadas y abundantes. Las dimensiones de las macrosporas se determinan de 20,2 a 50,6 μm x 3,1 a 6,2 μm. Los microconidios son indivisos o divididos en 1. Son de estructura ovalada y elipsoidal. Las clamidosporas se encuentran terminalmente en cadenas cortas en medio CLA dentro de 2-4 semanas30.
Identificación molecular
La PCR se realizó utilizando cebadores ITS4/ITS5 con ADN genómico total obtenido de aislados de Fusarium . Las secuencias de bases bidireccionales (5'-3' y 3'-5') se registraron y compararon con otros GenBank del NCBI utilizando el programa Blast (Tabla 4 y Tabla 5). Los aislados de F. oxysporum mostraron una homogeneidad de la secuencia de nucleótidos del 99,5% al 100% con aislados de India (MT740398), Lituania (KF646094) y Alemania (MT453296). Los aislados de F. culmorum mostraron una homogeneidad del 100% en la secuencia de nucleótidos con aislados de Canadá (AY147290), Francia (OW983123) y Chequia (MT453296). Los aislados de F. graminearum mostraron una homogeneidad de la secuencia de nucleótidos del 99,5% al 100% con aislados de Colombia (MT598163) y China (ON527490). Los aislados de F. acuminatum mostraron una homogeneidad de la secuencia de nucleótidos del 99,5% al 100% con aislados de Uzbekistán (OR975902) y China (MZ424810, PP336554). Los aislados de F. solani mostraron una homogeneidad del 100% de la secuencia de nucleótidos con aislados de Egipto (OR713084), China (PQ482231) e India (OP848138). Luego, se determinó la relación genética entre los aislados de Fusarium mediante el árbol filogenético obtenido según el método de Máxima Parsimonia (MP) utilizando el programa Mega 11 (Figura 3). En el árbol filogenético MP, se utilizaron 638 caracteres de nucleótidos, incluyendo huecos, y 145 de estos nucleótidos se determinaron como regiones informativas que proporcionan parsimonia. Los análisis de MP arrojaron uno de los árboles más parsimoniosos (Figura 3; Longitud del árbol: 172; Índice de consistencia (ConI): 0,974; Índice de retención (IR): 0,961; Índice compuesto (ComI): 0,7600). Cuando se examinó este dendrograma MP de aislados de Fusarium , el árbol genealógico se dividió primero en dos ramas principales. El primero de ellos correspondió a todos los aislados de F. solani soportados por el valor de bootstrap del 100%. La otra rama principal se reunió dentro de sí misma con un valor de bootstrap del 78% y cuatro subgrupos F. acuminatum, F. redolens, F. proliferatum y F. acutatum. La otra rama principal se dividió en cinco subgrupos F. oxysporum, F. culmorum, F. graminarum, F. pseudograminearum y F. equiseti, con un valor de bootstrap del 97%.
Determinación de los niveles de virulencia de aislados de Fusarium
En la Tabla 6 y Figura 4 se resumen los resultados de los estudios de patogenicidad de Fusarium spp. aislado de lentejas que muestran marchitez y pudrición de la raíz, para los cuales se completaron estudios de caracterización morfológica y molecular. En general, todos los aislados de Fusarium mostraron diferencias en los niveles de gravedad de las enfermedades que causaron en la variedad de lenteja Kayı 91 F(19-60; 0.05) = 43.06; p< 0,0001). Los aislados más virulentos fueron F. oxysporum (YBUFol4), F. culmorum (YBUFc1, YBUFc2) y F. graminearum (YBUFg1, YBUFg3). Se encontró que estos cinco aislados tenían un nivel de virulencia muy alto. Por otro lado, se encontró que un aislado de F. oxysporum (YBUFol2), F. solani (YBUFs1) y F. graminearum (YBUFg2) tenían virulencia moderada. Además, se encontró que los aislados de F. acuminatum (YBUFa1, YBUFa2, YBUFa3 e YBUFa4) tenían una virulencia débil o baja. Todos los aislados utilizados en los estudios de patogenicidad oscilaron entre 42,5% y 97,5% y se obtuvieron después de la inoculación de plántulas de lenteja.

Figura 1: Plantas de lentejas afectadas por especies de Fusarium. (A) Vista general de áreas enfermas localizadas en un campo de lentejas con alta gravedad de la enfermedad. (B) Vista de plantas con severidad de enfermedad 4. (C) Vista de plantas con gravedad de enfermedad 3. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Vista de plantas de lentejas en niveles de gravedad de la enfermedad 0 a 4. Nivel 0 = no mostró síntomas, 1 = los síntomas de las hojas en el 25%; 2 = 26%-50%; 3 = 51%-75% de las hojas; 4 = más del 75% en el mismo orden. Haga clic aquí para ver una versión más grande de esta figura.
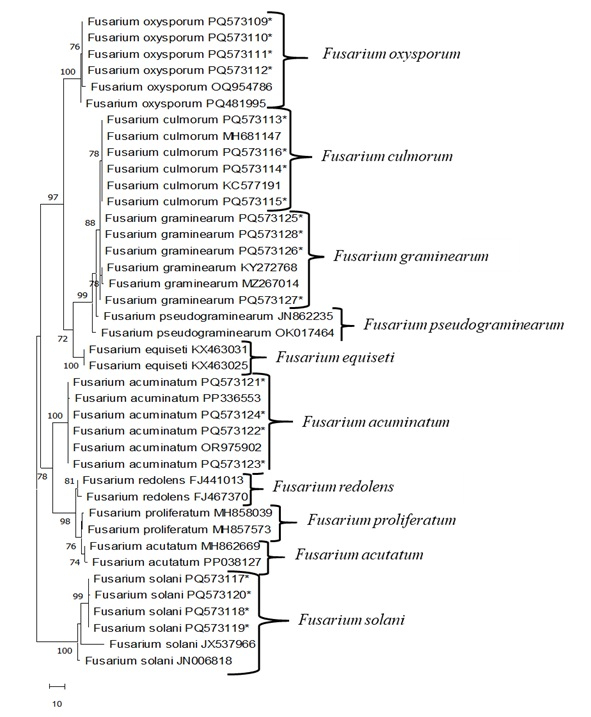
Figura 3: El árbol no enraizado más parsimonioso basado en la región ITS de Fusarium sp. usando MEGA 11. Los aislados están indicados con asteriscos. El resto se toman de GenBank. Haga clic aquí para ver una versión más grande de esta figura.
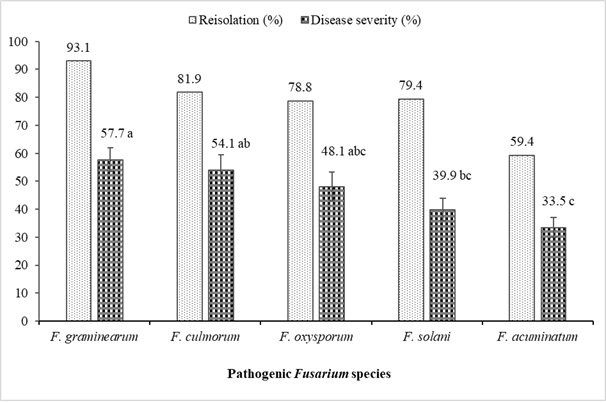
Figura 4: Reaislamiento (%) y tasas de severidad de la enfermedad (%) de Fusarium spp. aislado de plantas de lentejas. Las líneas verticales representan el error estándar en la barra (n = 16). Haga clic aquí para ver una versión más grande de esta figura.
| Temperatura (°C) | Humedad relativa (%) | Precipitación (mm) | |||||||
| Mes | 2022 | 2023 | A largo plazo | 2022 | 2023 | A largo plazo | 2022 | 2023 | A largo plazo |
| Akdağmadeni | |||||||||
| Marzo | 11.1 | 7.4 | 3.8 | 45.1 | 68.8 | 71.1 | 21.4 | 78.7 | 60.6 |
| Abril | 11.1 | 11.4 | 8.8 | 59.2 | 67 | 61.5 | 56.5 | 109.8 | 42.6 |
| Mayo | 16.3 | 15.1 | 12.9 | 64.3 | 73.9 | 64.8 | 48.5 | 83.6 | 74.1 |
| Junio | 16.5 | 18.4 | 16.2 | 63.4 | 53.2 | 69.1 | 0.3 | 20.1 | 57.2 |
| Julio | 22 | 22.2 | 18.7 | 48 | 40.5 | 60.5 | 1.4 | 0 | 7 |
| Boğazlıyan | |||||||||
| Marzo | 12.3 | 9.9 | 5.2 | 47.8 | 65.5 | 66 | 13 | 64 | 40.1 |
| Abril | 12.8 | 13.5 | 10.2 | 62.6 | 66.6 | 59.3 | 20.2 | 24.2 | 24.4 |
| Mayo | 18.7 | 17.7 | 14.6 | 62.5 | 67.4 | 61.2 | 65.4 | 20 | 35.1 |
| Junio | 19.7 | 20.6 | 18.6 | 52.9 | 49.9 | 59.9 | 0 | 2.4 | 35.5 |
| Julio | 24.3 | 24 | 21.7 | 42.5 | 39.5 | 50 | 0 | 0 | 3.9 |
| Çekerek | |||||||||
| Marzo | 13.5 | 10.8 | 6.6 | 52.8 | 70.4 | 69 | 24.3 | 111.3 | 48.7 |
| Abril | 14.1 | 13.9 | 11.5 | 63.9 | 72.3 | 61 | 48.1 | 73.4 | 32 |
| Mayo | 19.3 | 18.6 | 15.8 | 69.1 | 72.7 | 65.4 | 80.3 | 69 | 59.3 |
| Junio | 20.1 | 21.8 | 19.4 | 61.5 | 57.5 | 67.8 | 0 | 16.4 | 62.8 |
| Julio | 24.6 | 24.7 | 22 | 57.8 | 52.4 | 57.5 | 33.2 | 0 | 10.9 |
| Yozgat | |||||||||
| Marzo | 11.5 | 8.4 | 4 | 47.3 | 68.7 | 67.5 | 14.6 | 84.8 | 87.3 |
| Abril | 12.4 | 12.7 | 9.3 | 60.1 | 65.2 | 58.7 | 47.4 | 54.4 | 41.9 |
| Mayo | 17.5 | 16.8 | 13.7 | 64.2 | 68 | 60.2 | 92.6 | 61.6 | 72.2 |
| Junio | 18.3 | 20.3 | 17.4 | 57.9 | 50.7 | 61.2 | 1 | 4.8 | 63.6 |
| Julio | 22.9 | 23.6 | 20.5 | 50.9 | 44.3 | 52.7 | 7 | 0.2 | 8.8 |
| Saraykent | |||||||||
| Marzo | 14.8 | 9 | 6.2 | 50.9 | 71.1 | 71 | 11.1 | 77.3 | 55.3 |
| Abril | 12.5 | 12.3 | 10.5 | 64.6 | 73.2 | 60.7 | 35.3 | 36.1 | 29 |
| Mayo | 17.6 | 16.4 | 14.2 | 69.1 | 76.7 | 65.9 | 47.8 | 72.4 | 57 |
| Junio | 18.2 | 19.2 | 18.2 | 63.8 | 61 | 69.6 | 0 | 28.6 | 56.1 |
| Julio | 23.2 | 23.1 | 21.6 | 54.2 | 51.7 | 57.9 | 0 | 0 | 7.4 |
| Sarıkaya | |||||||||
| Marzo | 12.9 | 9.6 | 5.6 | 47 | 68.2 | 66.5 | 18.4 | 75.5 | 53.6 |
| Abril | 13.2 | 13.1 | 10.7 | 59 | 68.8 | 56.5 | 24.8 | 60.6 | 27.2 |
| Mayo | 18.5 | 17.3 | 14.7 | 63.2 | 71.6 | 60.6 | 57.6 | 64.5 | 47.2 |
| Junio | 18.9 | 20.5 | 18.4 | 59 | 53.9 | 62 | 0 | 16.1 | 50.6 |
| Julio | 24 | 24 | 21.3 | 48.6 | 45.4 | 51.5 | 0 | 0 | 6.4 |
| Şefaatli | |||||||||
| Marzo | 13.1 | 10.6 | 6.2 | 48.2 | 61.1 | 67 | 12.6 | 71.5 | 50.2 |
| Abril | 11.2 | 14.3 | 11.1 | 61.6 | 60.9 | 58.6 | 48.4 | 50.5 | 26 |
| Mayo | 19.9 | 18.6 | 15.4 | 66.7 | 61.9 | 62.3 | 65.2 | 95.4 | 53.7 |
| Junio | 20.4 | 22.2 | 19.7 | 55.8 | 45.5 | 61.2 | 1.2 | 2.5 | 46.8 |
| Julio | 25 | 25.3 | 23.2 | 47.9 | 39.5 | 47.1 | 0 | 0 | 4.1 |
| Sorgun | |||||||||
| Marzo | 12.2 | 9.4 | 4.8 | 48.4 | 68.2 | 67 | 21.6 | 104.2 | 49.8 |
| Abril | 13 | 13.3 | 10.1 | 59.7 | 66.1 | 58.9 | 53.2 | 33.8 | 31.7 |
| Mayo | 18.3 | 17.4 | 14.5 | 63.7 | 69.1 | 60.9 | 56.6 | 89.6 | 44.2 |
| Junio | 18.9 | 20.3 | 18.3 | 56.8 | 53.4 | 61.5 | 0 | 16.6 | 55.2 |
| Julio | 23.7 | 23.7 | 21.2 | 50 | 44.9 | 52.9 | 4 | 0 | 7.5 |
| Yerköy | |||||||||
| Marzo | 15 | 12.1 | 6 | 41.2 | 60.5 | 61.3 | 4.4 | 46.1 | 60.1 |
| Abril | 16.2 | 16.2 | 13.5 | 54.8 | 60.4 | 50.9 | 40.9 | 46.8 | 23 |
| Mayo | 21.5 | 20.6 | 17.4 | 55.4 | 59.6 | 52.2 | 57 | 23.6 | 35.1 |
| Junio | 23 | 24.5 | 20.9 | 45.2 | 41.4 | 56.4 | 0.2 | 0 | 39.1 |
| Julio | 27.5 | 27.7 | 24.4 | 38.2 | 35.5 | 42.2 | 0.2 | 0 | 3.5 |
Tabla 1: Datos meteorológicos de la localidad relevada durante la temporada de producción de lentejas por año.
| Condado | Número de campos | Área de siembra inspeccionada (decare) | Prevalencia de la enfermedad (%) | Incidencia de la enfermedad (%) | Gravedad de la enfermedad (%) |
| Akdağmadeni | 4 | 43.2 | 100 | 7 | 21.8 |
| Boğazlıyan | 4 | 86.4 | 100 | 23 | 36 |
| Çekerek | 1 | 2.7 | 100 | 1.6 | 14.4 |
| Merkez | 3 | 24.3 | 100 | 5.8 | 23 |
| Saraykent | 6 | 53.3 | 100 | 8.1 | 28.2 |
| Sarıkaya | 12 | 189.3 | 100 | 17 | 35.4 |
| Sorgun | 48 | 683.2 | 100 | 20.1 | 45.2 |
| Şefaatli | 2 | 73.7 | 100 | 26.4 | 23.4 |
| Yerköy | 3 | 42.3 | 100 | 9.1 | 32.9 |
| En general | 83 | 1198.4 | 100 | 16.9 | 38.6 |
Tabla 2: Parámetros de la enfermedad, prevalencia, incidencia y gravedad de la marchitez, la pudrición de la raíz y el cuello de la raíz en los campos de lentejas.
| Condado | Número de plantas utilizadas para el aislamiento | Frecuencia de aislamiento (%) | |||||
| F. oxysporum | F. culmorum | F. solani | F. acuminatum | F. graminearum | Otro | ||
| Akdağmadeni | 41 | 68.3 | 9.8 | 2.4 | 0 | 14.6 | 4.9 |
| Boğazlıyan | 57 | 54.4 | 1.8 | 12.3 | 7 | 15.8 | 8.8 |
| Çekerek | 21 | 52.4 | 4.8 | 0 | 23.8 | 9.5 | 9.5 |
| Merkez | 36 | 72.2 | 11.1 | 0 | 0 | 13.9 | 2.8 |
| Saraykent | 64 | 45.3 | 12.5 | 7.8 | 4.7 | 21.9 | 7.8 |
| Sarıkaya | 148 | 56.8 | 11.5 | 7.4 | 5.4 | 15.5 | 3.4 |
| Sorgun | 233 | 62.2 | 9.4 | 1.3 | 7.3 | 16.3 | 3.4 |
| Şefaatli | 32 | 71.9 | 15.6 | 3.1 | 0 | 9.4 | 0 |
| Yerköy | 47 | 57.4 | 14.9 | 4.3 | 2.1 | 14.9 | 6.4 |
| En general | 679 | 59.5 | 10.2 | 4.4 | 5.5 | 15.8 | 4.6 |
Tabla 3: Información sobre las lentejas muestreadas para determinar la frecuencia de aislamiento de hongos en la provincia de Yozgat de Türkiye.
| Especies de Fusarium | Aislar | Condado | Origen de aislamiento | Número de acceso al Banco de Germoplasma |
| F. oxysporum | YBUFol1 | Sorgun | Raíz | PQ573109 |
| YBUFol2 | Bogazlayan | Collar de raíz | PQ573110 | |
| YBUFol3 | Sarıkaya | Raíz | PQ573111 | |
| YBUFol4 | Akdagmadeni | Raíz | PQ573112 | |
| F. culmorum | YBUFc1 | Sorgun | Raíz | PQ573113 |
| YBUFc2 | Yerkoy | Raíz | PQ573114 | |
| YBUFc3 | Sarıkaya | Raíz | PQ573115 | |
| YBUFc4 | Saraykent | Collar de raíz | PQ573116 | |
| F. solani | YBUFs1 | Yozgat | Raíz | PQ573117 |
| YBUFs2 | Sorgun | Raíz | PQ573118 | |
| YBUFs3 | Sefaatli | Raíz | PQ573119 | |
| YBUFs4 | Sarıkaya | Raíz | PQ573120 | |
| F. acuminatum | YBUFa1 | Cekerek | Raíz | PQ573121 |
| YBUFa2 | Bogazlayan | Collar de raíz | PQ573122 | |
| YBUFa3 | Sarıkaya | Collar de raíz | PQ573123 | |
| YBUFa4 | Sorgun | Raíz | PQ573124 | |
| F. graminearum | YBUFg1 | Yozgat | Raíz | PQ573125 |
| YBUFg2 | Sorgun | Raíz | PQ573126 | |
| YBUFg3 | Saraykent | Raíz | PQ573127 | |
| YBUFg4 | Sarıkaya | Collar de raíz | PQ573128 |
Tabla 4: Aislados de Fusarium spp. de lenteja (Lens culinaris) de la provincia de Yozgat, en el centro de Turquía, utilizados en el estudio filogenético.
| Especies de Fusarium | Aislar | País | Origen de aislamiento | Número de acceso de GenBank |
| F. acuminatum | AAG4 | N/A | Prunus persica | OR975902 |
| WHWNSHJ1 | China | Malus domestica | PP336553 | |
| F. acutatum | CBS 739.97 | India | N/A | MH862669 |
| NSF1 | Egipto | Tetraena alba | PP038127 | |
| F. culmorum | 2090 | India | N/A | KC577191 |
| G49 | Polonia | Pisum sativum | MH681147 | |
| F. equiseti | Fusarium equiseti A577 | China | Pachulí | KX463031 |
| Fusarium equiseti A571 | China | Pachulí | KX463025 | |
| F. graminearum | WM-233 | China | N/A | MZ267014 |
| 16 bis | N/A | N/A | KY272768 | |
| F. oxysporum | YBUFoc4 | Turquía | Cicer arietinum | OQ954786 |
| LuC-8 | China | Crisantemo x morifolium | PQ481995 | |
| F. proliferatum | CBS 246.61 | Alemania | N/A | MH858039 |
| CBS 186.56 | N/A | N/A | MH857573 | |
| F. pseudograminearum | WZ-8A | China | Trigo | JN862235 |
| GAAET080 | China | Maíz | OK017464 | |
| F. redolens | M11 | N/A | Seta | FJ441013 |
| 2008 | N/A | Seta | FJ467370 | |
| F. solani | S2-27 | Francia | N/A | JX537966 |
| UENFCF279 | Brasil | Guayaba | JN006818 |
Tabla 5: Secuencias de especies de Fusarium utilizadas desde GenBank en el estudio filogenético.
| Especies de Fusarium | Aislar | Gravedad de la enfermedad (%) a | Nivel de virulencia del aislado | Reaislamiento (%) |
| (Error medio ± estándar) | ||||
| F. oxysporum | YBUFol1 | 20. 0 ± 1.1 i | Débil | 62.5 |
| YBUFol2 | 56,9 ± 1,2 CDE | Moderadamente | 85 | |
| YBUFol3 | 42.5 ± 1.0 EFG | Menos | 77.5 | |
| YBUFol4 | 73.1 ± 3.9 ab | Altamente | 90 | |
| F. culmorum | YBUFc1 | 72.5 ± 1.0 AB | Altamente | 95 |
| YBUFc2 | 72.5 ± 2.7 AB | Altamente | 95 | |
| YBUFc3 | 23.6 ± 1.6 hi | Débil | 60 | |
| YBUFc4 | 48.1 ± 2.6 def | Menos | 77.5 | |
| F. solani | YBUFs1 | 58,8 ± 1,6 bcd | Moderadamente | 97.5 |
| YBUFs2 | 46.9 ± 2.1 def | Menos | 75 | |
| YBUFs3 | 17,5 ± 2,3 i | Débil | 65 | |
| YBUFs4 | 36.3 ± 1.6 fgh | Menos | 80 | |
| F. acuminatum | YBUFa1 | 42.5 ± 2.3 EFG | Menos | 65 |
| YBUFa2 | 16,9 ± 2,8 i | Débil | 42.5 | |
| YBUFa3 | 30.6 ± 6.1 hi | Menos | 62.5 | |
| YBUFa4 | 44.1 ± 7.1 defg | Menos | 67.5 | |
| F. graminearum | YBUFg1 | 76,3 ± 2,2 A | Altamente | 97.5 |
| YBUFg2 | 50.6 ± 3.3 def | Moderadamente | 92.5 | |
| YBUFg3 | 68.1 ± 2.1 ABC | Moderadamente | 95 | |
| YBUFg4 | 35.6 ± 1.9 fgh | Menos | 87.5 |
Tabla 6: Pruebas de patogenicidad de especies de Fusarium en plantas de lentejas. Aislados rango medio <10% = no agresivo; 11%-25% = débil agresivo; 26%-50% = menos agresivo; 51%-70% = moderadamente agresivo; >70% = muy agresivo. un Diferencia estadística entre aislados según la prueba HSD de Tukey (p < 0,05).
Discusión
Se sabe que el marchitamiento por Fusarium causa graves pérdidas de rendimiento económico en algunas partes del mundo31. La enfermedad se notificó por primera vez en Hungría32 y posteriormente en muchos países como Egipto, India, Myanmar, Nepal, Pakistán, Turquía, Siria y Estados Unidos33. Kumar et al.34 informaron de una amplia distribución de marchitez de las lentejas, pudrición de la raíz y del cuello de la raíz, con informes de su aparición en al menos 26 países de todo el mundo. En un estudio reciente, se aislaron 12 especies de hongos a partir de plantas de lenteja enfermas colectadas en diferentes estados de la India, y se identificó a F. oxysporum f. sp. lentis como el patógeno más importante (30%), seguido de Rhizoctonia bataticola (17,5%) y Sclerotium rolfsii (15,7%)31. Del mismo modo, en el presente estudio, el género fúngico más frecuentemente aislado fue Fusarium (95,4%). De acuerdo con Zitnick-Anderson et al.7, Fusarium (50%) fue el género fúngico dominante que causó marchitamiento, pudrición de la raíz y del cuello de la raíz en las plantaciones de lentejas de Dakota del Norte. También informaron que F. oxysporum, F. solani, F. culmorum, F. equiseti, F. acuminatum, F. graminearum, F. redolens y F. avenaceum son patógenos dentro de este género.
Las características morfológicas de las cinco especies de Fusarium identificadas en este estudio fueron similares a las de estudios recientes35. De acuerdo con Rathod et al.36, los aislados de F. oxysporum reportaron que mostraron un desarrollo de colonias septadas, ramificadas, inicialmente blancas, y luego elevadas o hundidas en PDA, las cuales se colorearon en diferentes pigmentaciones. De manera similar, en el estudio actual, los aislados de F. oxysporum inicialmente formaron colonias blancas, luego amarillas, violetas o rosadas. Por otro lado, se observaron clamidosporas en forma de dos o tres cadenas, que se utilizaron como criterio morfológico de identificación de aislados de F. oxysporum , en colonias que tenían aproximadamente 30 días de edad. De hecho, Endes15 reportó que las clamidosporas de los aislados de F. oxysporum mantenidos durante >30 días obtenidos de tuvieron una mayor tendencia a infectar plantas en áreas de cultivo de garbanzo de la provincia de Yozgat fueron generalmente observadas en ambientes viejos.
La pudrición de la raíz por Fusarium en garbanzos y lentejas es causada por muchas especies de Fusarium, como F. solani, F. oxysporum y F. graminearum37. Según lo reportado por Dean et al.38, F. graminearum y F. oxysporum se encuentran entre los patógenos fúngicos de plantas más comúnmente aislados por los micólogos de plantas en todo el mundo. Sin embargo, Aydın et al.6 informaron que F. graminearum se encuentra entre los agentes causantes de la marchitez, la pudrición de la raíz y la corona en áreas de cultivo de lentejas en la región de Anatolia sudoriental de Turquía. Además, se sabe que F. graminearum causa la pudrición de la raíz y la corona en las lentejas en el estado de Dakota del Norte, EE. UU.7.
Fletcher et al.39 reportaron que F. culmorum rara vez puede ser aislado como patógeno en áreas de cultivo de lentejas. Además, en un estudio reciente de Zitnick-Anderson et al.7, se informó que F. culmorum está asociado con la marchitez, la pudrición de la raíz y el cuello de la raíz en las plantas de lentejas. Sin embargo, Aydın et al.6 identificaron muchas especies de Fusarium que causan marchitez y pudrición de la raíz en áreas de cultivo de lentejas, pero no reportaron a F. culmorum como patógeno. Esta situación, que difiere del presente estudio, puede deberse al diferente número de áreas estudiadas o al apoyo de estudios morfológicos con métodos moleculares en el presente estudio.
Zitnick-Anderson et al.7 informaron que F. acuminatum se encuentra entre las especies de Fusarium que causan marchitez y pudrición de la raíz y el cuello de la raíz en los campos de lentejas de Dakota del Norte. Por otro lado, Aydın et al.6 documentaron que F. solani se encuentra entre los agentes causantes de la marchitez y la pudrición de la raíz en las áreas de cultivo de lentejas. También se sabe que F. solani se aísla de las áreas de cultivo de lentejas como un patógeno débil7.
Las especies de Fusarium son parásitos facultativos. Las infecciones debidas al género Fusarium en las lentejas pueden causar la destrucción completa de las plantas, especialmente en temperaturas extremas a fines de primavera o principios de verano. Al Ahmad y Mouselli40 informaron que F. oxysporum y F. solani causaron síntomas de amarilleo, defoliación y desecación en las plantaciones de lentejas en el sur de Siria. F. solani causa pudrición de la raíz y enfermedades de marchitez en Anatolia Central, incluido Yozgat16. Además, también se ha encontrado que algunas otras especies de Fusarium son patógenas en ciertas tasas. Zitnick-Anderson et al.7 informaron que la enfermedad por marchitez por Fusarium es causada por Fusarium oxysporum f. sp lentis, pero las especies de F. culmorum, F. solani, F. graminearum también pueden causar la enfermedad por marchitez de acuerdo con las características morfológicas, fisiológicas y patológicas. Además, Fletcher et al.39 reportaron que F. acuminatum, que causa marchitez y pudrición de la raíz en las lentejas, tiene una virulencia débil o baja.
El estudio reveló que el 95,4% de los aislados de Fusarium obtenidos de los campos de lentejas examinados eran especies de Fusarium y tuvieron una incidencia del 1,6% al 26,4% y una gravedad de la enfermedad del 14,4% al 45,2% en las plantas de lentejas. Las condiciones climáticas adversas, como el calor cálido y seco de la primavera y el calor de principios del verano, debilitan el crecimiento de las plantas y las hacen susceptibles a las especies de Fusarium . La mayoría de las especies de Fusarium son patógenos débiles, y cuando las condiciones ambientales debilitan la planta huésped, causan un aumento en la marchitez por Fusarium , especialmente en condiciones de sequía.
Divulgaciones
Los autores declaran no tener conflictos de intereses.
Agradecimientos
Este estudio contó con el apoyo del Centro de Investigación y Aplicación de Coordinación de Proyectos de la Universidad de Bozok, unidad BAP con número de proyecto FÇD-2022-1096. Este estudio forma parte del estudio de doctorado de Sevim Atmaca.
Materiales
| Name | Company | Catalog Number | Comments |
| (S)-lactic acid | Merck | 100366 | Was used as an antibiotic in studies. |
| 2-propanol | Merck | 109634 | Used in molecular studies. |
| Adjustable micro automatic pipette (0.1-2.5 µL) | Eppendorf Research | LB.EP.3123000012 | Used to measure small volumes of liquids. |
| Adjustable micro automatic pipette set (2 – 20 µl, 20 – 200 µl, 100 – 1.000 µl) | Eppendorf Research | LB.EP.4924000916 | Used to measure small volumes of liquids. |
| Agar | Merck | 110453 | For use in making fungal media. |
| Agarose | Sigma-Aldrich | 18300012 | For use in gel preparation in electrophoresis. |
| Air conditioning room | ?klimlab | Was used to grow plants under controlled conditions. | |
| Ampisilin | Sigma-Aldrich | A9393 | Was used as an antibiotic in studies. |
| Analytical precision balance | Shimadzu ATX224 | Was used to weigh the solid materials used in the study. | |
| Autoclave sterilizer | Zealway | GF-120DR | It was used to sterilize solid and liquid materials at every stage of the study. |
| Binocular microscope | Leica DM750 | For use in morphological diagnosis. | |
| Biological safety cabinet | HFsafe Class II A2 | To ensure the safety of the work area, the user, the environment and the operation. | |
| Centrifugal | DLAB DM1424 | LB.DL.903001124 | Used to separate particles in a sample based on their shape, size and density |
| Chloramphenicol | Sigma-Aldrich | 220551 | Was used as an antibiotic in studies. |
| Cork-borer set | Sigma-Aldrich | Z165220 | It was used to take samples from fungus culture in petri dishes. |
| Cover glass and slide | ISOLAB | 075.01.006 / 075.02.005 | Was used in the preparation process for microscope studies. |
| D(+)-glucose monohydrate | Merck | 108342 | For use in making fungal media. |
| DFC450 with digital camera | Leica | Digital microscope camera with c-mount interface and with a 5 megapixel ccd sensor. | |
| Dm750 binocular microscope | Leica | MIC5246 | Was used for morphological identification of fungi. |
| DNA gel electrophoresis | thermo fisher scientific | B2-UVT | |
| Dna gel loading dye (6x) | Thermo Scientific | R0611 | For use in molecular diagnostics. |
| dNTP mix | Thermo Scientific | R0192 | Used in molecular studies. |
| Dreamtaq pcr master mixes (2x) | Thermo Scientific | K1082 | For use in molecular diagnostics. |
| Drigalski spatule | ISOLAB | 082.03.001 | It was used to scrape and spread fungal cultures grown in petri dishes. |
| Edta | Thermo Scientific | 17892 | For use in molecular diagnostics. |
| Ethanol | Merck | 100983 | Used in molecular studies and surface disinfection studies. |
| Ethidium bromide | Sigma-Aldrich | E7637 | Used to stain dna in gels during gel electrophoresis. |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | E6758 | Used in molecular studies. |
| Filter paper | ISOLAB | 107.58.158 | Used in stock culture studies. |
| Forced air drying cabinet | ZHICHENG ZXDS-A-1090 | For use in incubation processes. | |
| Fume hood | Elektromag EM1201 | LB.EM.EM1201 | It was used to control harmful chemical vapors, gases and dust. |
| Gel imaging system | Syngene G:BOX Chemi XX6 | For use in molecular diagnostics. | |
| Generuler 50 bp dna ladder | Thermo Scientific | SM0372 | For use in molecular diagnostics. |
| Glacial acetic acid | Merck | 1005706 | Used in molecular studies. |
| Glycerol | Merck | 104094 | For use in stock culture of fungi. |
| Lancet | ISOLAB | 048.50.002 | Used to remove diseased tissue from plant samples. |
| Magnesium chloride | Sigma-Aldrich | 814733 | Used in molecular studies. |
| Measuring tape | ISOLAB | 016.07.500 | Used to measure liquid volumes. |
| Microcentrifuge tubes | ISOLAB | 0778.03.001 / 0778.03.002 / 0778.03.003 | Used to store different volumes of liquids. |
| PCR tube | ISOLAB | 123.01.002 | It was used to put dna mix in pcr studies. |
| Petri dishes | ISOLAB | 120.13.090 | For use in growing fungus culture. |
| Pipette tips | ISOLAB | 005.01.001 / 005.01.002 / 005.01.003 / 005.01.004 | To transfer liquid volumes used in analyses. |
| Plastic bag | ISOLAB | 039.30.005 | Was used to transport samples to the laboratory. |
| Plastic pot | ToXA | Was used for growing plants. | |
| Pliers, clamps | ISOLAB | 048.08.130 | It was used to put filter papers into envelopes after the fungus grew in the petri dish. |
| Porcelain mortar | ISOLAB | 038.02.150 | Was used to crush fungal mycelia. |
| Potato dextrose agar | Condalab | 1022 | For the identification and cultivation and of fungi. |
| Pure water system | human CORPORATION | LT.HC.NHP009 | Was used in solution preparation and analysis throughout the studies. |
| Refrigerator (+4 °C / -20 °C) | Vestel | For use in the storage of stock materials. | |
| Rifampicin | Sigma-Aldrich | 557303 | Was used as an antibiotic in studies. |
| Sodium acetate | Merck | 106268 | Used in molecular studies. |
| Sodium chloride | Merck | 1064041000 | Used in molecular studies. |
| Sodium dodecyl sulfate | Sigma-Aldrich | 436143 | Used in molecular studies. |
| Sodium hypochlorite solution | Merck | 105614 | Used for surface disinfection. |
| Spatula | ISOLAB | 047.33.210 | It was used to scrape the fungus culture growing in petri dishes. |
| Streptomyc?n sulfate | BioShop Canada | STP101 | To prevent contamination in fungal culture cultivation. |
| Teksoll extra pure | Tekkim | TK.200650 | For use as a disinfectant in all stages of work. |
| Tetrasiklin | Sigma-Aldrich | T3258 | Was used as an antibiotic in studies. |
| Thermal cycler PCR | Bio?Rad T100 | For use in genomic analyses. | |
| Thoma lam | ISOLAB | 075.03.002 | For use in spore counting. |
| Tris HCL | Roche | 10812846001 | Used in molecular studies. |
| Trizma | Sigma-Aldrich | T1503 | Used in molecular studies. |
| Tween 80 | Merck | 822187 | For use in spore solution in pathogenicity testing. |
| Vortex mixer vorteks | Velp WIZARD | LB.VLP.F202A0175 | Used to mix substances in liquid volumes. |
| Water baths | Memmert WNB 22 | 1018-5702 | It was used during incubation in dna extraction studies. |
Referencias
- Skrzypkowski, W., Kiełkowska, A. Current status of haploidization in cool-season grain legume crop species. Agriculture. 14 (7), 1031 (2024).
- Liber, M., Duarte, I., Maia, A. T., Oliveira, H. R. The history of lentil (Lens culinaris subsp. culinaris) domestication and spread as revealed by genotyping-by-sequencing of wild and landrace accessions. Front Plant Sci. 12, 628439 (2021).
- . FAOSTAT Available from: https://www.fao.org/faostat/en/#data (2022)
- TÜİK. . Agricultural statistics report. , (2023).
- Baxevanos, D., et al. Lentil cultivar evaluation in diverse organic mediterranean environments. Agronomy. 14 (4), 790 (2024).
- Aydın, M., Koç, M., Sağır, A. Investigations on determination of soilborne fungal pathogens causing root rot, crown rot and wilt on lentil in Southeast Anatolia Region. Plant Protect Bulletin. 44 (1), 93-103 (2004).
- Zitnick-Anderson, K., et al. Fusarium species associated with root rot of lentil (Lens culinaris) in North Dakota. Plant Health Prog. 22 (4), 524-528 (2021).
- Kushwaha, D. A. . A research book of seed mycoflora of chickpea (Cicer arietinum). , (2020).
- Ekwomadu, T. I., Mwanza, M. Fusarium fungi pathogens, identification, adverse effects, disease management, and global food security: A review of the latest research. Agriculture. 13 (9), 1810 (2023).
- Jiskani, A. M., et al. A destructive disease of lentil: Fusarium wilt of lentil. Plant Arch. 21 (1), 2117-2127 (2021).
- Alisaac, E., Mahlein, A. K. Fusarium head blight on wheat: biology, modern detection and diagnosis and integrated disease management. Toxins. 15 (3), 192 (2023).
- Yang, F., et al. Effects of rhizosphere microbial communities on cucumber Fusarium wilt disease suppression. Microorganisms. 11 (6), 1576 (2023).
- Kumari, N., Katoch, S. Wilt and root rot complex of important pulse crops: their detection and integrated management. Management of Fungal Pathogens in Pulses. Fungal Biology. , (2020).
- Köppen, W., Geiger, R. . Handbuch der klimatologie. , (1936).
- Agrios, G. N. . Plant Pathology. , (2005).
- Endes, A. Occurrence and distribution of Chickpea root rot and wilt disease in Yozgat Kırşehir and Kırıkkale Provinces. Çukurova J Agri Food Sci. 38 (2), 284-298 (2023).
- Choi, Y. W., Hyde, K. D., Ho, W. H. Single spore isolation of fungi. Fungal Diversity. 3, 29-38 (1999).
- Baskarathevan, J., Jaspers, M. V., Jones, E. E., Ridgway, H. J. Evaluation of different storage methods for rapid and cost effective preservation of Botryosphaeria species. New Zealand Plant Protect. 62, 234-237 (2009).
- Dikilitaş, M., Katırcıoğlu, Z., Altınok, H. Latest developments and methods on long-term storage, protection, and recycle of fungi and fungal material. JAgric Fac HR U. 15 (1), 55-69 (2011).
- Bora, T., Karaca, &. #. 3. 0. 4. ;. . Measurement of disease and damage in cultivated plants. , (1970).
- Öğüt, E. . Pathogenic and molecular characterization of some Fusarium spp. causing root rot and wilt on lentil with determination of variety reactions in south eastern Anatolia. , (2015).
- Leslie, J. F., Summerell, B. A. . The Fusarium Laboratory manual. , (2006).
- Cenis, J. L. Rapid extraction of fungal DNA for PCR amplification. Nuc Acids Res. 20 (9), 2380 (1992).
- Aras, S., Endes, A. Effect of Fusarium oxysporum infection on strawberry under calcium, iron, and zinc deficiency conditions. Zemdirbyste-Agri. 110 (1), 71-78 (2023).
- White, T. J., Bruns, T., Lee, S., Taylor, J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. PCR Protoc A Guide Metho Appl. , 315-322 (1990).
- Endes, A. Characterization and pathogenicity of Botryosphaeriaceae species associated with Gummosis, Dieback, Trunk and Branch Cankers of almond trees in Türkiye. J Agri Sci. 30 (4), 698-711 (2024).
- Gordon, T. R., Okamoto, D., Jacobson, D. J. Colonization of muskmelon and non-susceptible crops by Fusarium oxysporum f. sp. melonis and other species of Fusarium. Phytopathology. 79 (10), 1095-1100 (1989).
- Xu, X., et al. Fusarium species associated with maize leaf blight in Heilongjiang Province, China. J Fungi. 8 (11), 1170 (2022).
- Fonseca-Guerra, I. R., Chiquillo-Pompeyo, J. C., Benavides Rozo, M. E., Díaz Ovalle, J. F. Fusarium spp. associated with Chenopodium quinoa crops in Colombia. Sci Rep. 12 (1), 20841 (2022).
- Khan, M. F., et al. First report of damping-off and seedling rot of Hemp (Cannabis sativa) caused by Fusarium solani in North Dakota, U.S.A. Plant Dis. 107 (1), 232 (2023).
- Chaudhary, R. G., Saxena, D. R., Dhar, V., Singh, R. K., Namdev, J. K. Prevalence of wilt-root rot and their associated pathogens at reproductive phase in lentil. Arch Phytopathol Plant Protect. 43 (10), 996-1000 (2010).
- Fleischmann, R. Some observations on Maize smut in Hungary. Pflanzenbau. 14 (5), 199-206 (1937).
- Bedasa, T. . Distribution and management of Fusarium wilt (Fusarium oxysporum f. sp. lentis) of lentil (Lens culinaris Medikus) in Central Highlands of Ethiopia. , (2018).
- Kumar, S., et al. Vascular wilt disease of lentil: A review. J Lentil Res. 4, 1-14 (2010).
- Chenari, S., Abbasi, S., Chehri, K. Phylogeny and host specificity of Fusarium solani species complex isolated from chickpea, lentil and common bean. Arch Phytopathol Plant Protect. , 1-15 (2024).
- Rathod, A., et al. Isolation of causal organism of wilt and collar rot of lentil and its pathogenicity tests. Int J Curr Microbiol Appl Sci. 10 (12), 276-282 (2021).
- Hayit, T., Endes, A., Hayit, F. The severity level classification of Fusarium wilt of chickpea by pre-trained deep learning models. J Plant Pathol. 106 (1), 93-105 (2024).
- Dean, R., et al. The Top 10 fungal pathogens in molecular plant pathology. Mol Plant Pathol. 13 (4), 414-430 (2012).
- Fletcher, J. D., Broadhurst, P. G., Bansal, R. K. F. avenaceum: A pathogen of lentil in New Zealand. New Zealand J Crop Horticultural Sci. 19 (2), 207-210 (1991).
- Al Ahmad, M., Mouselli, N. Wilt and root rot of lentis. Lens. 14 (1/2), 27-31 (1987).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados