Method Article
La levadura de fisión como plataforma para el cribado de fármacos antibacterianos dirigidos a las proteínas del citoesqueleto bacteriano
En este artículo
Resumen
La levadura de fisión se utiliza aquí como huésped heterólogo para expresar proteínas citoesqueléticas bacterianas como FtsZ y MreB como proteínas de fusión traduccional con GFP para visualizar su polimerización. Además, los compuestos que afectan a la polimerización se identifican mediante imágenes con un microscopio de fluorescencia.
Resumen
Las proteínas del citoesqueleto bacteriano, como FtsZ y MreB, realizan funciones esenciales como la división celular y el mantenimiento de la forma celular. Además, FtsZ y MreB se han convertido en objetivos importantes para el descubrimiento de nuevos antimicrobianos. Se han desarrollado varios ensayos para identificar compuestos dirigidos a la unión de nucleótidos y la polimerización de estas proteínas del citoesqueleto, centrándose principalmente en FtsZ. Además, muchos de los ensayos son laboriosos o costosos, y determinar si estas proteínas son el objetivo celular del fármaco a menudo requiere múltiples métodos. Por último, la toxicidad de los fármacos para las células eucariotas también plantea un problema. Aquí, describimos un ensayo basado en células de un solo paso para descubrir nuevas moléculas dirigidas al citoesqueleto bacteriano y minimizar los impactos que podrían ser potencialmente tóxicos para las células eucariotas. La levadura de fisión es susceptible a cribados de alto rendimiento basados en microscopía, y una criba visual puede identificar fácilmente cualquier molécula que altere la polimerización de FtsZ o MreB. Nuestro ensayo utiliza la placa estándar de 96 pocillos y se basa en la capacidad de las proteínas del citoesqueleto bacteriano para polimerizar en una célula eucariota como la levadura de fisión. Si bien los protocolos descritos aquí son para levaduras de fisión y utilizan FtsZ de Staphylococcus aureus y MreB de Escherichia coli, son fácilmente adaptables a otras proteínas del citoesqueleto bacteriano que se ensamblan fácilmente en polímeros en cualquier huésped de expresión eucariota. El método descrito aquí debería ayudar a facilitar el descubrimiento de nuevos antimicrobianos dirigidos a las proteínas del citoesqueleto bacteriano.
Introducción
La resistencia generalizada a casi todos los antibióticos que se emplean actualmente para combatir las infecciones bacterianas ha creado una necesidad inmediata de nuevas categorías de antibióticos. Un informe de 2019 indicó que las infecciones resistentes a los antibióticos provocaron la pérdida de 1,27 millones de vidas, lo que contribuyó a un recuento total de 4,95 millones de muertes si se consideran las complicaciones de las infecciones bacterianas resistentes1. Aunque sigue siendo eficaz en la práctica clínica, el arsenal actual de antibióticos se dirige principalmente a un espectro estrecho de procesos celulares, centrándose principalmente en la pared celular, el ADN y la síntesis de proteínas. En el último medio siglo, menos de 30 proteínas han sido explotadas comercialmente como dianas para el desarrollo de nuevos antibacterianos 2,3. Esta gama limitada de objetivos viables crea limitaciones significativas para descubrir nuevos antibióticos o sus derivados para combatir las bacterias resistentes a los antibióticos. Por lo tanto, para superar el problema emergente de la resistencia a los antibióticos, es necesario desarrollar nuevos antibióticos con nuevas dianas y mecanismos de acción.
Idealmente, un objetivo antibacteriano debería ser un componente esencial del crecimiento de las células bacterianas, conservarse en todas las especies filogenéticamente diversas, mostrar la menor homología eucariota y ser accesible a losantibióticos. Desde el descubrimiento de las proteínas del citoesqueleto bacteriano implicadas en la división celular y el mantenimiento de la forma celular, se han convertido en un prometedor punto focal para eldesarrollo de compuestos antibacterianos. Estas proteínas son esenciales para la viabilidad bacteriana y desempeñan un papel fundamental en el mantenimiento de la forma celular (MreB, CreS), la división (FtsZ, FtsA) y la segregación del ADN (ParM, TubZ, PhuZ, AlfA), similar al citoesqueleto de las células eucariotas. En particular, FtsZ exhibe un nivel notablemente alto de conservación en una amplia gama de organismos procariotas, mientras que MreB se encuentra en casi todas las bacterias en forma de bastoncillo. Esta amplia distribución y relevancia en la viabilidad celular hacen de estas proteínas un objetivo fascinante en la investigación de antibióticos 5,6,7.
Es crucial adoptar un enfoque múltiple que combine observaciones in vivo, interacciones in vitro y experimentos enzimáticos para validar exhaustivamente las proteínas del citoesqueleto bacteriano como el objetivo principal de un inhibidor potencial7. Los procedimientos laboriosos o las implicaciones de costos sustanciales obstaculizan muchos ensayos disponibles para este propósito. Estos son obstáculos notables para su utilización generalizada en el cribado de compuestos principales que podrían afectar al citoesqueleto bacteriano. Entre ellos, la microscopía destaca como un método excepcionalmente eficiente y rápido para evaluar la eficacia de los compuestos mediante el examen directo de los cambios en la morfología celular. Sin embargo, la heteroasociación de la proteína diana con otros complejos de citoesqueletos, los efectos indirectos debidos a la unión fuera del objetivo y los cambios en el potencial de membrana, la dificultad para penetrar en la célula de manera eficiente y la presencia de bombas de eflujo, especialmente en bacterias Gram-negativas, hacen que sea colectivamente complejo identificar la causa precisa de la deformación de las células bacterianas 8,9,10.
Schizosaccharomyces pombe, o levadura de fisión como se le conoce comúnmente, es un organismo eucariota unicelular en forma de bastón. La levadura de fisión es ampliamente utilizada como organismo modelo en biología celular y molecular debido a la extraordinaria conservación en procesos celulares como el ciclo celular y la división, la organización celular y la replicación cromosómica con eucariotas superiores, incluyendo humanos11,12. Además, Errington y sus colegas expresaron una proteína del citoesqueleto bacteriano DivIVA localizada en polos en levadura de fisión para demostrar que DivIVA se acumulabaen superficies curvadas negativamente. Una vez más, Balasubramanian y su grupo habían establecido por primera vez la levadura de fisión como un sistema modelo celular para aportar nuevos conocimientos sobre el mecanismo, el ensamblaje y la dinámica de la proteína del citoesqueleto de actina de E. coli como MreB14 y el homólogo de tubulina FtsZ15. También demuestran la capacidad de A22 para impedir de manera eficiente la polimerización de MreB a través de la microscopía de epifluorescencia cuando se expresa en levadura14. A continuación, otros grupos también han empleado con éxito la levadura de fisión para estudiar las propiedades de ensamblaje de las proteínas FtsZ1 y FtsZ2 del cloroplasto16. Más recientemente, hemos establecido una prueba de concepto de la viabilidad del uso de la levadura de fisión como plataforma celular para detectar específicamente inhibidores del citoesqueleto bacteriano mediante la realización de una evaluación exhaustiva del impacto de tres inhibidores conocidos de FtsZ (sanguinarina, berberina y PC190723) en las proteínas FtsZ derivadas de dos bacterias patógenas, a saber, Staphylococcus aureus y Helicobacter pylori17. Además, este ensayo basado en células de un solo paso resulta fundamental para minimizar el riesgo de identificar compuestos que pueden ser potencialmente tóxicos para las células eucariotas.
En este informe, utilizando el sistema de levadura de fisión, proponemos un flujo de trabajo sistemático utilizando la placa estándar de 96 pocillos para el cribado semiautomatizado y la cuantificación del efecto de los inhibidores de moléculas pequeñas dirigidos a FtsZ de Staphylococcus aureus y MreB de Escherichia coli. Aquí, configuramos y optimizamos el flujo de trabajo semiautomatizado utilizando los inhibidores establecidos PC190723 y A22 que se dirigen específicamente a FtsZ y MreB, respectivamente. Este flujo de trabajo utiliza un microscopio de epifluorescencia equipado con una platina motorizada de alta precisión y adquisición automatizada de imágenes en una placa estándar de 96 pocillos para mejorar la estandarización actual. Por lo tanto, se puede aplicar a pantallas de rendimiento medio y alto de bibliotecas de productos químicos sintéticos y evita algunos de los desafíos enumerados anteriormente.
Protocolo
1. Expresión de proteínas del citoesqueleto bacteriano marcadas con GFP en S. pombe
NOTA: Consulte la Tabla 1 para obtener información sobre todos los plásmidos y cepas utilizados aquí. Consulte la Tabla 2 para ver todas las composiciones de medios.
- Realizar la clonación de E. coli MreB con una fusión de GFP N-terminal (GFP-MreB) y FtsZ de S. aureus portadora de una GFP C-terminal (SaFtsZ-GFP) en el vector de expresión de S. pombe, pREP42 con un promotor represible de tiamina de potencia media, nmt4118,19 como se describió anteriormente14,15. Mantener, amplificar y aislar los plásmidos de las cepas de E. coli (DH10β) como se describe en20,21.
NOTA: También se pueden utilizar otras cepas de E. coli como DH5α, XL1Blue, TOP10, etcétera, u otras células competentes disponibles en el mercado que se utilizan habitualmente para la clonación molecular. - Transformación de los plásmidos pREP42-GFP-EcMreB y pREP42-SaFtsZ-GFP en S. pombe
- Transforme los plásmidos pCCD3 (pREP42-GFP-EcMreB) y pCCD713 (pREP42-SaFtsZ-GFP) en la cepa de S. pombe (h-leu1-32 ura4-D18) utilizando el método de acetato de litio22, como se menciona en los pasos a continuación.
- Día 1 - Cultivo primario: Inocular un bucle de cultivo de S. pombe recién veteado en 3 ml de extracto de levadura esterilizado en autoclave y caldo de suplementos (YES). Incubar en un agitador orbital a 30 °C durante la noche (O/N).
- Día 2:
- Cultivo secundario: Añadir unos 500 μL de cultivo primario a 30 mL de caldo YES esterilizado en autoclave. Incubar a 30 °C durante 3 - 4 h agitando hasta que el diámetro exterior de600 alcance 0,4 - 0,6.
NOTA: Para cada transformación, se utilizan 30 mL. - Granular el cultivo de 30 mL a 2.500 x g durante 6-8 min a temperatura ambiente. Deseche el sobrenadante y lave las células con 50 mL de agua destilada estéril (D/W). Vuelva a pellet y deseche el sobrenadante. Vuelva a suspender las células en 1 mL de D/W estéril y transfiéralo a un tubo de centrífuga de 2 mL. Centrifugar como se indicó anteriormente y desechar el sobrenadante.
- Agregue 1 mL de acetato de litio 0.1 M, solución de Tris-EDTA (LiAc-TE) y centrifugar como se indicó anteriormente. Deseche el sobrenadante y vuelva a suspender en 1 mL de LiAc-TE 0,1 M. Centrifugar y desechar el sobrenadante, dejando 100 μL de solución.
- Agregue de 10 a 20 μg de ADN portador (ADN de esperma de salmón; desnaturalizado y enfriado rápidamente en hielo) y 2 a 3 μg del ADN plásmido, que necesita ser transformado. Mezclar suavemente. Incubar a temperatura ambiente durante 10 min.
- Añadir 260 μL de 40 % de PEG/LiAc-TE; mezclar suavemente. Incubar durante 60 min en el agitador o en una termobatidora a 30 °C mezclando suavemente. Añadir 43 μL de DMSO; mezclar suavemente.
- Choque térmico a 42 °C durante 10 min en la termobatidora. Pellet a 2.500 x g durante 6-8 min y desechar el sobrenadante. Lave el pellet 1 vez con 1 mL de D/W estéril.
- Pellet, desechar el sobrenadante y resuspender el pellet en 200 μL de D/W. Placa estéril de 100 μL en placas de medio mínimo (EMM) de Edimburgo que contengan 5 μg/mL de tiamina (para reprimir el promotor nmt41 ) y suplementos de aminoácidos adenina (0,225 mg/mL), histidina (0,225 mg/mL) y leucina (0,225 mg/mL) pero que carezcan de uracilo (marcador de selección para el plásmido pREP42).
NOTA: Alternativamente, coloque 70 μL y 130 μL en dos placas diferentes. Se siembran dos volúmenes diferentes (70 μL y 130 μL) para obtener colonias aisladas en al menos una de las placas en función de las eficiencias de transformación, que pueden variar de un experimento a otro. - Incubar las placas a 30 °C durante 2-3 días hasta que aparezcan las colonias. Mezclar una sola colonia con 100 μL de D/W estéril y extenderla sobre una placa EMM fresca que contenga tiamina pero que carezca de uracilo como se ha descrito anteriormente.
- Incubar a 30 °C durante 2-3 días hasta que aparezca un césped completo. Deseche el césped crecido de células usando un asa de inoculación y vuelva a suspenderlo en 1 ml de medio YES que contenga 30% de glicerol en un criovial.
- Congele rápidamente el criovial en nitrógeno líquido y guárdelo a -80 °C para preservar las existencias de levadura congeladas.
- Cultivo secundario: Añadir unos 500 μL de cultivo primario a 30 mL de caldo YES esterilizado en autoclave. Incubar a 30 °C durante 3 - 4 h agitando hasta que el diámetro exterior de600 alcance 0,4 - 0,6.
- Expresión de proteínas del citoesqueleto bacteriano en levaduras de fisión
- Coloque asépticamente un parche de la reserva de glicerol en una placa específica de levadura fresca para obtener suficiente cultivo para experimentos posteriores.
NOTA: En el caso de pREP42, utilizamos una placa de agar EMM (medio mínimo) que contenía adenina, histidina y leucina (como se utilizó la cepa de S. pombe (h-leu1-32 ura4-D18)) sin uracilo (uracilo presente en pREP42 como marcador de selección). Añadimos 15 - 20 μM de tiamina a las placas de agar (ya que pREP42 tiene un promotor nmt41 represible de tiamina) para reprimir la expresión del gen de interés cuando crece en la placa. - Inocular un pequeño bucle del inóculo del parche veteado en 5 mL de medio EMM específico de levadura que carezca de tiamina e incubarlo durante 10 - 12 h a 30 °C.
NOTA: La expresión de FtsZ y MreB de diferentes especies bajo el promotor nmt41 en S. pombe puede variar, típicamente de 16 a 30 h. El tiempo óptimo para la expresión proteica de GFP-EcMreB y SaFtsZ-GFP en S. pombe es de 20 a 24 h y de 16 a 20 h a 30 °C, respectivamente, sin tiamina.
- Coloque asépticamente un parche de la reserva de glicerol en una placa específica de levadura fresca para obtener suficiente cultivo para experimentos posteriores.
2. Tratamiento de cultivos de S. pombe que expresan proteínas citoesqueléticas bacterianas marcadas con GFP
NOTA: Se prueban varias moléculas de fármacos diferentes en el cultivo de levadura cultivado durante la noche en una placa de 96 pocillos.
- Inocular un cultivo fresco transfiriendo 50 μL del cultivo durante la noche a cada pocillo de una placa de 96 pocillos que contiene 150 μL de medios EMM de levadura fresca utilizando el instrumento de pipeteo multicanal semiautomatizado de 96 pocillos.
- Mientras pipetea hacia adentro y hacia afuera para una mezcla adecuada con una pipeta multicanal semiautomática de 96 pocillos, introduzca lentamente diferentes concentraciones crecientes de fármacos en el cultivo en crecimiento, cada una realizada por triplicado como se representa en el esquema de la Figura 1.
- Como control positivo, utilice los fármacos ya conocidos, PC190723 para (S. aureus FtsZ) y A22 (para E. coli MreB) por duplicado.
NOTA: Como se informó anteriormente, PC190723 y A22 se utilizaron a una concentración de 56,2 μM17 y 72,6 μM 14,17, respectivamente. Mientras tanto, el tratamiento A22 de SaFtsZ y el tratamiento PC190723 de EcMreB sirvieron como controles negativos, respectivamente. - Utilice DMSO como control de solventes en tres concentraciones diferentes de acuerdo con las concentraciones más bajas y más altas de medicamentos.
NOTA: El disolvente en el que se disuelven los fármacos se utiliza como control de disolventes. - Incubar los cultivos control y tratados a 30 °C durante 6-10 h (hasta que las células muestren la expresión de proteínas fluorescentes bacterianas) y luego obtener imágenes mediante microscopía de epifluorescencia.
3. Visualización de los polímeros
- Aplique una capa de 20 μL de 1 mg/mL de concanavalina A a cada pocillo de la placa inferior ópticamente transparente de 96 pocillos (de paredes negras para imágenes de fluorescencia) e incube durante 20 min a temperatura ambiente. Aspira el líquido y déjalo secar al aire durante 10 min.
- Transfiera 20 μL de células de la placa de cultivo a cada pocillo respectivo y déjelo reposar durante 10 minutos. Lave la célula 3x-4x con los medios EMM estériles.
- Con la lente del objetivo que se va a utilizar en su lugar (consulte el paso 3.6), utilice el modo de navegador en el panel de adquisición del software de adquisición de imágenes para la alineación de placas de 96 pocillos y la cobertura de pocillos. Alinee los bordes del pozo en el navegador.
- A continuación, seleccione los parámetros de cobertura del pozo, que incluyen una selección de varias posiciones de la región de interés en el pocillo, y utilice el control de enfoque automático adaptativo con modo bajo demanda para mantener la muestra enfocada durante la obtención de imágenes.
- Adquiera imágenes utilizando contraste de interferencia diferencial (DIC) y fluorescencia. Ajuste filtros de excitación y emisión de 475/28 nm y 525/48 nm, respectivamente, para obtener imágenes de las proteínas bacterianas marcadas con GFP expresadas en la levadura. Obtenga pilas Z a un tamaño de paso de 0,2 μm a través del grosor de las células de levadura (5 μm).
- Capture imágenes utilizando un microscopio de epifluorescencia invertida equipado con un objetivo de inmersión en aceite de 100x 1.4-NA y una cámara sCMOS de 2.000 x 2.000 (tamaño de píxel de 6,5 μm x 6,5 μm). Utilice un sistema de iluminación LED para la excitación del fluoróforo.
NOTA: Cualquier sistema de microscopio de epifluorescencia que tenga una platina motorizada para una placa de 96 pocillos con posicionamiento multipunto preciso debe ser adecuado. Adquiera todas las imágenes celulares de control y tratadas con fármaco con un tiempo de exposición similar (generalmente en el rango de 0,3 a 0,5 s) a un binning de 1 x 1 y una iluminación del 15% al 20% para minimizar las variables del experimento y mantener la consistencia.
4. Cuantificación de las imágenes mediante ImageJ
- Procese imágenes de celdas y mida el número de puntos por celda y la densidad para FtsZ17. En el caso de MreB, medir la densidad y la anisotropía23 y comparar entre las células control y las tratadas utilizando Fiji (v2.0.0-rc-69/1.52p)24.
- Utilizando las imágenes del canal DIC, primero delinee las células de levadura individuales con la herramienta de dibujo a mano alzada y guárdelas como una región de interés (ROI) en el administrador de ROI. Este paso aún no está automatizado, pero las herramientas desarrolladas recientemente utilizando el aprendizaje automático25,26 podrían intentarse e incorporarse en un futuro próximo.
- Enmascare el área exterior de la celda y rellene de negro como se describe en27.
- Utilice la macro de análisis de umbral OPS IJ1, una función incorporada en Fiyi, para contar el número de puntos24.
- Utilice el método otsu para el umbral automático y enmascare las partículas segmentadas. Ejecute el complemento de análisis de partículas mediante una macro.
- Para medir la densidad del polímero (cantidad de citoesqueleto por unidad de área en una célula), procese las imágenes como se describe, incluidos los pasos de enmascaramiento y esqueletización27,28. Utilice Lpx 2Dfilter en el plug-in lpx para esqueletizar las imágenes.
- Para más detalles, consulte esta reciente publicación17. Realice la cuantificación de polímeros EcMreB como se mencionó anteriormente en23, use la anisotropía para cuantificar la organización espacial usando FibrilTool29 como se describió anteriormente23.
NOTA: Estos métodos de análisis se pueden utilizar para cuantificar cualquier otra proteína del citoesqueleto bacteriano que esté tratada o no con los fármacos.
Resultados
Instalación de la placa de 96 pocillos para el cribado de fármacos
Se ha establecido previamente el uso de S. pombe para expresar una FtsZ de S. aureus marcada con GFP terminal C a partir de un vector (pREP42) que contiene el promotor represible de tiamina de potencia media nmt4117 y, de manera similar, la MreB de E. coli marcada con GFP N-terminal también se expresó en S. pombe14. También hemos demostrado que PC190723, un inhibidor específico de SaFtsZ y A22, un inhibidor de MreB, pueden ejercer sus efectos sobre las respectivas proteínas del citoesqueleto bacteriano de manera específica cuando se expresan en levadura14,17.
Aquí, proponemos utilizar el sistema de expresión de levadura para desarrollar un cribado de rendimiento medio o alto de fármacos antibacterianos dirigidos a las proteínas del citoesqueleto bacteriano. Un microscopio de epifluorescencia con una platina motorizada puede automatizarse para obtener imágenes de una placa de 96 pocillos, y las bibliotecas comerciales de fármacos también están disponibles en formatos de placa de 96 pocillos. Por lo tanto, preferimos utilizar una placa de 96 pocillos para el cribado propuesto de fármacos. Las placas se colocan como se muestra en la Figura 1. La primera fila (Figura 1A) consta de SaFtsZ-GFP que expresa cultivo de levadura. La segunda fila (Figura 1B) consta de cultivos de levadura que expresan GFP-EcMreB. Las dos primeras columnas de las dos primeras filas (A1:B2) se establecen como medios en blanco. Las siguientes seis columnas de las dos primeras filas (A3:B12) se establecen como control DMSO con tres concentraciones diferentes según las diluciones de fármaco por duplicado. A continuación, se añaden PC190723 (56,2 μM) y A22 (72,6 μM) a las células de levadura que expresan SaFtsZ-GFP o GFP-EcMreB. PC190723 y A22 sirven como controles positivos para FtsZ y MreB, respectivamente. Todos los demás pocillos (C1:F12) se utilizan para añadir diversos fármacos (en tres concentraciones diferentes y por duplicado) utilizados en el cribado para identificar los efectores de las proteínas del citoesqueleto bacteriano, SaFtsZ o EcMreB. Los cultivos de levadura que expresan la SaFtsZ o MreB marcada con GFP se dispensan en estas placas de 96 pocillos y se cultivan hasta que se visualizan los efectos de los fármacos en el ensamblaje de polímeros.
Evaluación de los efectos del crecimiento en las células de levadura
Después de incubar las células subcultivadas de 96 pocillos a 30 °C durante 6 a 10 h, se mide la densidad óptica (DO) del cultivo de levadura utilizando un lector de microplacas. La medición de la OD ayuda a evaluar cualquier efecto inhibidor del crecimiento de los fármacos contra las células de levadura eucariotas y los posibles efectos deletéreos en las células humanas. Por lo tanto, este ensayo se puede utilizar para descartar múltiples medicamentos en función de su toxicidad en el sistema de levadura. Sin embargo, ni PC190723 ni A22 presentan efectos sobre el crecimiento ni toxicidad para las células de levadura, y OD600 de los cultivos que expresan SaFtsZ-GFP tratados con DMSO, PC190723 o A22 fueron de 0,38 ± 0,03, 0,44 ± 0,02 y 0,43 ± 0,06 (N ≥ 4), respectivamente. Asimismo, los OD600 de los cultivos que expresaron GFP-EcMreB y tratados con DMSO, PC190723 o A22 fueron 0,38 ± 0,04, 0,44 ± 0,07 y 0,41 ± 0,08 (N ≥ 4), respectivamente.
Visualización del efecto de los fármacos sobre las estructuras poliméricas ensambladas por SaFtsZ y EcMreB expresadas en S. pombe
Con el fin de evaluar el efecto de los fármacos en la polimerización de SaFtsZ o EcMreB, una alícuota de las células de levadura de la placa de 96 pocillos mencionada anteriormente que contiene fármacos se transfiere a otra placa de 96 pocillos con fondo de vidrio ópticamente transparente de 96 pocillos adecuado para la obtención de imágenes de fluorescencia. Esta placa de 96 pocillos también está recubierta con concanavalina A para permitir la adhesión de las células de levadura que expresan SaFtsZ o EcMreB marcadas con GFP. La placa de 96 pocillos se visualiza utilizando un microscopio de epifluorescencia que cubre todos los 96 pocillos de la placa en las posiciones predeterminadas controladas por el software de adquisición de imágenes (Figura 2). En los pocillos control (tratados con DMSO), las células de levadura que expresaban SaFtsZ-GFP mostraron estructuras poliméricas en forma de manchas o parches distribuidos por todas las células, después de 16 a 18 h de crecimiento en ausencia de tiamina. Sin embargo, a las 10 - 12 h después del crecimiento en ausencia de tiamina, las células solo exhibieron fluorescencia difusa o pocos parches, lo que sugiere que la expresión de FtsZ-GFP no había alcanzado la concentración crítica requerida para la polimerización (Figura 3A). Por el contrario, el fármaco estabilizador de FtsZ, PC190723, mostró un aumento considerable en las estructuras poliméricas (manchas o parches) de SaFtsZ-GFP en comparación con el control de DMSO después de 12 h de crecimiento (Figura 3B), sirviendo como control positivo. Por el contrario, las células tratadas con A22 no mostraron diferencias en las estructuras SaFtsZ-GFP ensambladas (Figura 3C). Sin embargo, a diferencia de los cultivos tratados con PC190723, el SaFtsZ-GFP se ensambló en parches en cultivos no tratados solo cuando se indujo durante períodos más largos, lo que demuestra que PC190723 actuó para reducir la concentración crítica de polimerización de SaFtsZ (Figura 3D). Del mismo modo, la GFP-EcMreB expresada en S. pombe formó matrices lineales de filamentos largos a lo largo del eje longitudinal de la levadura de fisión (Figura 4A). Si bien se encontró que PC190723 no tenía ningún efecto sobre la polimerización de EcMreB (Figura 4B), el tratamiento de las células con A22 resultó en fluorescencia difusa en todo el citoplasma de las células de levadura (Figura 4C). Las imágenes del resto de las placas de 96 pocillos se inspeccionan visualmente para detectar cualquier efecto estabilizador o inhibidor de los fármacos sobre el SaFtsZ o el EcMreB, según sea el caso.
El impacto de los fármacos en el ensamblaje de las proteínas del citoesqueleto bacteriano puede cuantificarse utilizando herramientas de análisis de imágenes y plug-ins en ImageJ o Fiji, como se informó anteriormente para SaFtsZ17 y EcMreB21. La adquisición automatizada de imágenes en formato de placa de 96 pocillos y la implementación de macros y scripts personalizados para el procesamiento de imágenes pueden acelerar el tiempo de obtención de imágenes y la cuantificación de los efectos de los fármacos. Por lo tanto, utilizando la levadura eucariota unicelular, S. pombe, como sistema huésped, proponemos que se puede llevar a cabo con éxito un cribado de rendimiento medio o alto para moléculas pequeñas dirigidas a las proteínas del citoesqueleto bacteriano, lo que finalmente conducirá al descubrimiento de nuevos antibióticos.
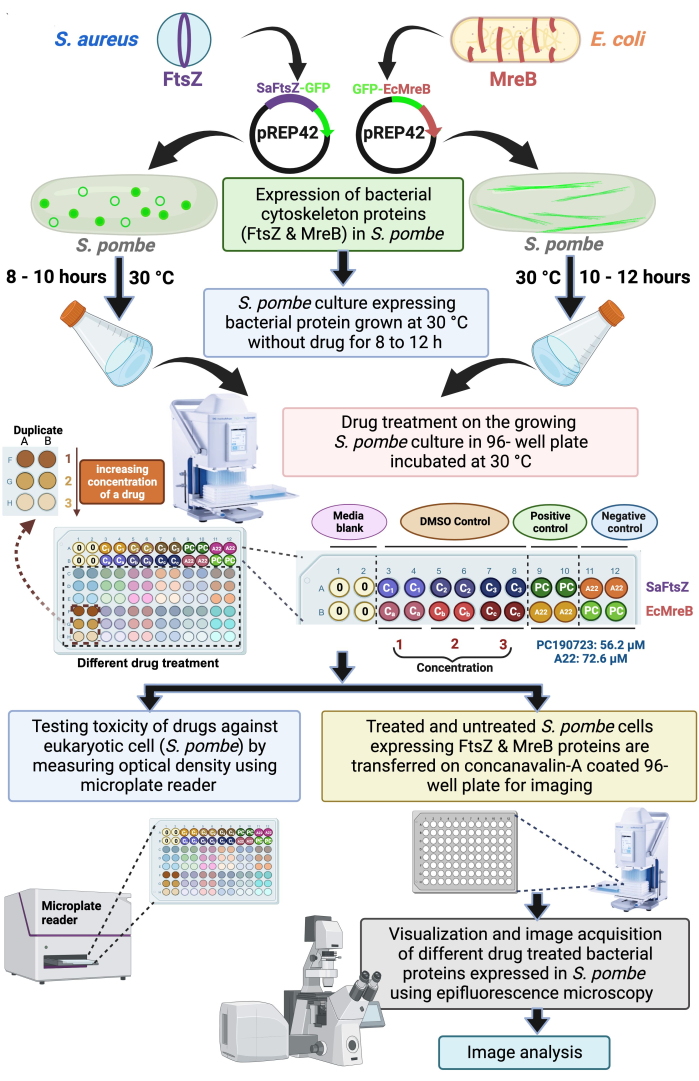
Figura 1: Esquema que muestra el uso de células de levadura de fisión para detectar fármacos dirigidos al ensamblaje de proteínas del citoesqueleto bacteriano SaFtsZ o EcMreB. FtsZ de S. aureus y MreB de E. coli se clonaron en el vector de expresión de levadura, pREP42 con etiqueta GFP en los extremos C y N-, respectivamente, y se transformaron en S. pombe para el estudio fenotípico y de fármacos. Los cultivos se cultivaron durante 8 - 12 h. Posteriormente, los cultivos se subcultivaron en placas de 96 pocillos con controles adecuados y se realizaron cribados los diferentes fármacos. Además, la placa se incubó a 30 °C durante 6 a 8 h hasta que el diámetro exterior de600 alcanzó 0,5-0,6, y la densidad óptica se mide utilizando un lector de microplacas para evaluar la toxicidad de los fármacos hacia las células de levadura. Otra placa de 96 pocillos con fondo de vidrio ópticamente transparente fue pre-recubierta con concanavalina A, para adherir las células de levadura. Esta placa de 96 pocillos se utilizó para la visualización y adquisición de imágenes con un microscopio de epifluorescencia. A continuación, se analizaron las imágenes obtenidas. Haga clic aquí para ver una versión más grande de esta figura.
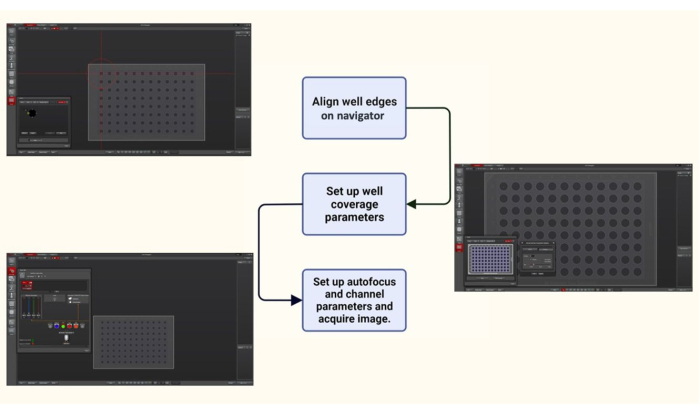
Figura 2: Esquema del software de adquisición de imágenes para la obtención automática de imágenes de una placa de 96 pocillos. Utilizando el navegador en el software de adquisición de imágenes, se alineó una placa de 96 pocillos marcando los bordes. Los parámetros de cobertura del pozo, como el número de imágenes que deben tomarse de cada pozo y su ubicación (aleatoria o desde el centro del pozo), se seleccionaron según lo deseado. Por último, los parámetros de la imagen, como el conjunto de filtros que se van a utilizar (DIC y filtro FITC; Ex 475/28 nm y Em 525/48 nm), se estableció el porcentaje de intensidad de la luz de iluminación, el enfoque automático y el tiempo de exposición para la captura de imágenes. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Visualización del efecto de los fármacos PC190723 y A22 sobre la SaFtsZ-GFP expresada en S. pombe. El cultivo de S. pombe que expresa SaFtsZ-GFP se cultivó en ausencia de tiamina durante 8 - 9 h a 30 °C antes del tratamiento con DMSO y fármaco. El cultivo se cultivó en una placa de 96 pocillos durante 7 a 9 h a 30 °C en presencia o ausencia de los fármacos. (A) En el control de DMSO, donde se agregó una cantidad equivalente de DMSO, las células exhibieron algunas estructuras poliméricas. (B) En presencia de PC190723 (56,2 μM), SaFtsZ expresa células de levadura que muestran un aumento considerable en las estructuras poliméricas que el control DMSO. (C) El inhibidor de MreB, A22 (72,6 μM), no afectó a las estructuras de SaFtsZ-GFP. (D) Se necesitaron más horas de expresión de proteínas para el ensamblaje de SaFtsZ-GFP en ausencia de PC190723 y, por lo tanto, los cultivos de control cultivados a 30 °C durante 12 a 15 h después del subcultivo en una placa de 96 pocillos que contiene DMSO también exhiben polímeros FtsZ. La barra de escala es de 5 μm. Haga clic aquí para ver una versión más grande de esta figura.
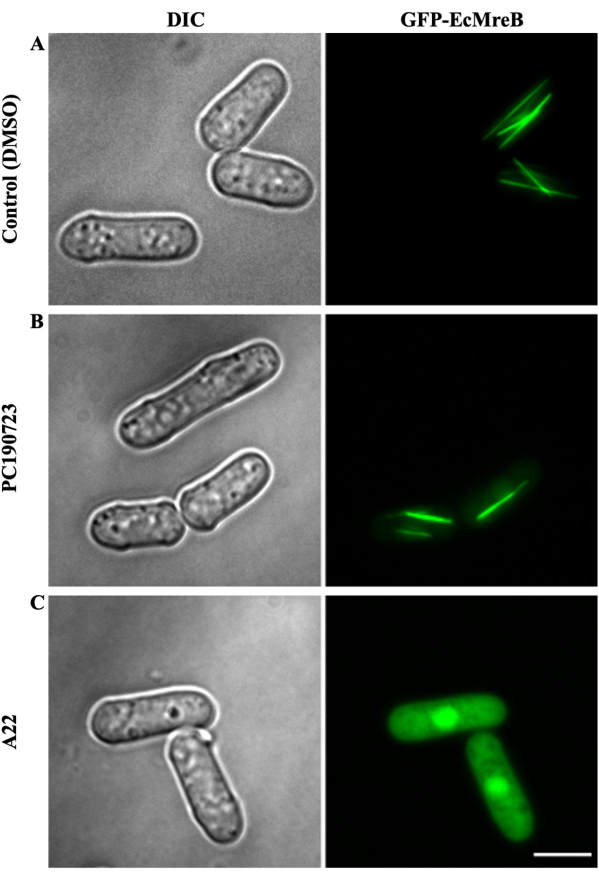
Figura 4: Visualización del efecto de los fármacos PC190723 y A22 sobre la GFP-EcMreB expresada en S. pombe. El cultivo de S. pombe que expresa GFP-EcMreB se cultivó en ausencia de tiamina durante 9 - 11 h a 30 °C antes de añadir DMSO o los fármacos (A22 o PC190723). El cultivo se cultivó en una placa de 96 pocillos durante 8 a 10 h a 30 °C con y sin fármacos como control. (A) En cultivos en los que se añadió una cantidad equivalente de DMSO como control, las células exhibieron filamentos lineales de EcMreB orientados a lo largo del eje longitudinal de las células de levadura. (B) El ensamblaje de GFP-EcMreB no se vio afectado en los cultivos tratados con PC190723 (56,2 μM). (C) La pequeña molécula A22 (72,6 μM), un inhibidor conocido de la polimerización de MreB, impidió el ensamblaje de GFP-EcMreB en células de levadura de fisión. La barra de escala es de 5 μm. Haga clic aquí para ver una versión más grande de esta figura.
Tabla 1: Cepas y plásmidos utilizados en este estudio. Haga clic aquí para descargar esta tabla.
Tabla 2: Composición de los medios y tampones utilizados en este estudio. Haga clic aquí para descargar esta tabla.
Discusión
La resistencia a los antimicrobianos (RAM) es una grave amenaza para la salud mundial, y existe una necesidad urgente de nuevos antibióticos con nuevos objetivos. El citoesqueleto bacteriano se ha convertido en una diana atractiva para el desarrollo de nuevos antibióticos, con inhibidores de moléculas pequeñas de la proteína de división celular FtsZ, como TXA709, que ya se encuentran en ensayos clínicos de faseI 30. Se han desarrollado varios métodos para identificar inhibidores de la polimerización de FtsZ 7,31. Recientemente hemos utilizado la levadura de fisión eucariota en un ensayo basado en células para demostrar que el fármaco estabilizador FtsZ, PC190723, puede dirigirse a HpFtsZ17. Además, también demostramos que, aunque fármacos como la berberina y la sanguinarina afectaban a la polimerización de la FtsZ, también afectaban a la morfología de las células de levadura17. A partir de estos estudios y observaciones, nos propusimos desarrollar un ensayo basado en células de un solo paso para detectar moduladores de moléculas pequeñas del citoesqueleto bacteriano utilizando la plataforma de expresión de levadura de fisión. Dado que el cribado se basa en la inhibición del ensamblaje de proteínas citoesqueléticas marcadas con GFP, las moléculas pequeñas que comparten propiedades de fluoróforo similares pueden dar lugar a falsos positivos. Evitar los filtros de paso largo y elegir filtros de paso de banda estrecha puede ayudar a reducir estos falsos positivos que surgen de las moléculas pequeñas fluorescentes. Aquí, mostramos los pasos involucrados en la configuración de una pantalla de rendimiento medio utilizando un microscopio de epifluorescencia equipado con un soporte de etapa de placa de 96 pocillos que se puede usar para detectar medicamentos dirigidos a SaFtsZ o EcMreB. El flujo de trabajo implica la adquisición y el análisis semiautomatizados de imágenes, que en el futuro pueden ampliarse fácilmente a pantallas de alto rendimiento mediante sistemas robóticos de manipulación de líquidos y tuberías de procesamiento de imágenes.
Además, el cribado debería ser útil para otras proteínas del citoesqueleto bacteriano, como la proteína de mantenimiento plásmida ParM32 que contiene pliegues de actina y contra cualquier proteína que se ensamble en estructuras poliméricas en la levadura de fisión. Sin embargo, los niveles óptimos de expresión para cada proteína de interés deberán estandarizarse antes de su uso en cualquier prueba de detección de fármacos. Mientras que para las FtsZ, hemos encontrado que nmt41 es un promotor óptimo, para otras proteínas de interés, una versión más fuerte o más débil del promotor nmt1 (Fuerza del promotor (y permeabilidad): nmt1 > nmt41 > nmt81) u otros promotores constitutivos como la levadura adh1 o act118,33 para obtener niveles óptimos de expresión y ensamblaje de estructuras poliméricas. Además, al utilizar plásmidos episomales, las variaciones en el número de copias de una célula a otra también plantean problemas para la expresión génica uniforme. Para eludir estos problemas, recientemente se han desarrollado varios vectores que pueden integrarse en el genoma de la levadura de fisión (SIV para vectores de integración estables) portadores de promotores de diversas fortalezas34. El uso de estos vectores puede ofrecer resultados más uniformes y cuantitativos en comparación con los vectores episomales utilizados en este estudio. El uso de SIV podría evitar la gran heterogeneidad en los niveles de expresión, observada como una variación en las intensidades de fluorescencia a través de las células, y debería ser más adecuado para cribados de alto rendimiento.
Además, fármacos como PC190723 actúan para estabilizar los polímeros FtsZ, lo que resulta en el ensamblaje temprano de filamentos FtsZ en comparación con las células no tratadas. Si bien el cribado es adecuado para el cribado de fármacos que inhiben la polimerización de las proteínas del citoesqueleto bacteriano, debido a la evaluación visual de la fluorescencia difusa en el punto final, el cribado de fármacos que estabilizan los polímeros FtsZ requiere una mayor optimización de los niveles de expresión de proteínas y el tiempo necesario para lograr el número máximo de células que tienen polímeros. Por lo tanto, es fundamental tener una estimación razonable del tiempo necesario para la expresión de proteínas para alcanzar la concentración crítica requerida para el ensamblaje y decidir el punto final del ensayo en consecuencia. En el caso de fármacos que podrían actuar para disminuir la concentración crítica requerida para la polimerización de la proteína, la elección del punto final sería típicamente un tiempo, en el que una población considerable de células no tratadas exhibe fluorescencia difusa, pero los cultivos tratados con fármacos mostrarían ensamblajes de polímeros.
Una de las limitaciones, que también es un paso del flujo de trabajo que requiere mucho tiempo, es el requisito de identificar y segmentar celdas individuales para el análisis cuantitativo de imágenes. Actualmente, en el esquema propuesto, este es un paso manual para identificar células de levadura individuales y asignar la región de interés (ROI) para la cuantificación del efecto de los medicamentos. Sin embargo, prevemos que los algoritmos de aprendizaje automático desarrollados recientemente utilizando conjuntos de entrenamiento como los que se pueden implementar en ilastik35 deberían minimizar la necesidad de intervención manual.
Si bien nuestro enfoque aquí utilizando la plataforma de levadura es útil para identificar impactos y posibles moléculas pequeñas dirigidas al citoesqueleto bacteriano, eventualmente será necesario probar su actividad antibacteriana utilizando las pruebas estándar de concentración inhibitoria mínima y eficacia en modelos animales. No obstante, el mayor desafío para el aumento global de la resistencia a los antimicrobianos (RAM) sigue radicando en la permeabilidad de la membrana externa de las bacterias gramnegativas y en las numerosas bombas de eflujo de muchas especies bacterianas patógenas. Además, las bombas de eflujo en la levadura de fisión también pueden plantear problemas similares y es posible que se requieran concentraciones más altas del fármaco para el proceso de detección. Alternativamente, las cepas de levadura de fisión que son hipersensibles a los fármacos (S. pombe MDR-supML), como resultado de la deleción de los siete genes de resistencia a múltiples fármacos, incluidos cuatro genes transportadores y un factor de transcripción, pueden utilizarse como huéspedes para el cribado32,33. Serán necesarios futuros desarrollos en tecnologías que combinen el descubrimiento de antibacterianos específicos y aborden la resistencia debida a fenómenos generales como la permeabilidad de la membrana para hacer frente a la creciente resistencia a los antimicrobianos.
Divulgaciones
Todos los autores declaran no tener conflictos de intereses.
Agradecimientos
SMP, SR y AKS reconocen las becas recibidas del Instituto Nacional de Educación e Investigación Científica, Departamento de Energía Atómica. RS reconoce el apoyo financiero interno del Departamento de Energía Atómica, y este trabajo está respaldado por una subvención de investigación para RS (BT/PR42977/MED/29/1603/2022) del Departamento de Biotecnología (DBT). Los autores también agradecen a V Badireenath Konkimalla por sus comentarios, sugerencias y discusiones a lo largo del desarrollo del protocolo.
Materiales
| Name | Company | Catalog Number | Comments |
| 96 Well CC2 Optical CVG Sterile, w/Lid. Black | Thermo Scientific™ | 160376 | |
| 96-well plate | Corning | CLS3370 | |
| A22 Hydrochloride | Sigma | SML0471 | Dissolved in DMSO |
| Adenine | FormediumTM | DOC0229 | 225 mg/L of media |
| Concanavalin A | Sigma | C5275-5MG | |
| DMSO | Sigma | 317275 | |
| Edinburg minimal medium (EMM Agar or EMM Broth) | FormediumTM | PMD0210 | See below for composition |
| EDTA | Sigma | EDS-500G | |
| epMotion® 96 with 2-position slider | Eppendorf | 5069000101 | |
| Histidine | FormediumTM | DOC0144 | 225 mg/L of media |
| Leica DMi8 inverted fluorescence microscope | Leica Microsystems | German company | |
| Leucine | FormediumTM | DOC0157 | 225 mg/L of media |
| Lithium acetate | Sigma | 517992-100G | |
| PC190723 | Merck | 344580 | Dissolved in DMSO |
| Polyethylene glycol (PEG) | Sigma | 202398 | |
| Thiamine | Sigma | T4625 | Filter sterilised |
| Tris-Hydrochloride | MP | 194855 | |
| Uracil | FormediumTM | DOC0214 | 225 mg/L of media, Store solution at 36°C |
| YES (Yeast extract + supplements) Agar | FormediumTM | PCM0410 | See below for composition |
| YES (Yeast extract + supplements) Broth | FormediumTM | PCM0310 | See below for composition |
| Yeast (S. pombe) media | |||
| Yeast extract + supplements (YES) | |||
| Composition | g/L | ||
| Yeast extract | 5 | ||
| Dextrose | 30 | ||
| Agar | 17 | ||
| Adenine | 0.05 | ||
| L-Histidine | 0.05 | ||
| L-Leucine | 0.05 | ||
| L-Lysine HCl | 0.05 | ||
| Uracil | 0.05 | ||
| Edinburg minimal medium (EMM) | |||
| Composition | g/L | concentration | |
| potassium hydrogen phthallate | 3 | 14.7mM | |
| Na2HPO4 | 2.2 | 15.5 mM | |
| NH4Cl | 5 | 93.5 mM | |
| glucose | 2% (w/v) or 20 g/L | 111 mM | |
| Salts (stock x 50) | 20 mL/L (v/v) | ||
| Vitamins (stock x 1000) | 1 mL/L (v/v) | ||
| Minerals (Stock x 10,000) | 0.1 mL/L (v/v) | ||
| Salts x 50 | 52.5 g/l MgCl2.6H20 (0.26 M) | 52.5 | 0.26 M |
| 0.735 mg/l CaCl2.2H20 (4.99 mM) | 0.000735 | 4.99 mM | |
| 50 g/l KCl (0.67 M) | 50 | 0.67 M | |
| 2 g/l Na2SO4 (14.l mM) | 2 | 14.1 mM | |
| Vitamins x 1000 | 1 g/l pantothenic acid | 1 | 4.20 mM |
| 10 g/l nicotinic acid | 10 | 81.2 mM | |
| 10 g/l inositol | 10 | 55.5 mM | |
| 10 mg/l biotin | 0.01 | 40.8 µM | |
| Minerals x 10,000 | boric acid | 5 | 80.9 mM |
| MnSO4 | 4 | 23.7 mM | |
| ZnSO4.7H2O | 4 | 13.9 mM | |
| FeCl2.6H2O | 2 | 7.40 mM | |
| molybdic acid | 0.4 | 2.47 mM | |
| KI | 1 | 6.02 mM | |
| CuSO4.5H2O | 0.4 | 1.60 mM | |
| citric acid | 10 | 47.6 mM | |
| Strains/ Plasmids | |||
| Strains | Description | Reference | |
| CCD190 | Escherichia coli DH10β | Invitrogen | |
| CCDY4 | MBY3532; CCDY346/pREP42- GFP-EcMreB | Srinivasan et al., 2007 | |
| CCDY340 | CCDY346/pREP42- SaFtsZ-GFP | Sharma et al., 2023 | |
| CCDY346 | MBY192; Schizosaccharomyces pombe [ura4-D18, leu1-32, h-] | Dr. Mithilesh Mishra (DBS, TIFR) | |
| Plasmids | |||
| pCCD3 | pREP42-GFP-EcMreB | Srinivasan et al., 2007 | |
| pCCD713 | pREP42-SaFtsZ-GFP | Sharma et al., 2023 |
Referencias
- Antimicrobial Resistance Collaborators. Global burden of bacterial antimicrobial resistance in 2019: a systematic analysis. Lancet. 399 (10325), 629-655 (2022).
- Haselbeck, R., et al. Comprehensive essential gene identification as a platform for novel anti-infective drug discovery. Curr Pharma Des. 8 (13), 1155-1172 (2002).
- Wang, J., et al. Discovery of a small molecule that inhibits cell division by blocking FtsZ, a novel therapeutic target of antibiotics. J Biol Chem. 278 (45), 44424-44428 (2003).
- Sanchez, S., Demain, A. L. Antibiotics: current innovations and future trends. , Caister Academic Press. (2015).
- Vollmer, W. The prokaryotic cytoskeleton: a putative target for inhibitors and antibiotics. Appl Microbiol Biotechnol. 73 (1), 37-47 (2006).
- Vollmer, W. Targeting the bacterial Z-ring. Chem Biol. 15 (2), 93-94 (2008).
- Kusuma, K. D., Payne, M., Ung, A. T., Bottomley, A. L., Harry, E. J. FtsZ as an antibacterial target: status and guidelines for progressing this avenue. ACS Infect Dis. 5 (8), 1279-1294 (2019).
- Kaul, M., Zhang, Y., Parhi, A. K., Lavoie, E. J., Pilch, D. S. Inhibition of RND-type efflux pumps confers the FtsZ-directed prodrug TXY436 with activity against Gram-negative bacteria. Biochem Pharmacol. 89 (3), 321-328 (2014).
- Casiraghi, A., Suigo, L., Valoti, E., Straniero, V. Targeting bacterial cell division: A binding site-centered approach to the most promising inhibitors of the essential protein FtsZ. Antibiotics. 9 (2), 69(2020).
- Piddock, L. J. V. Multidrug-resistance efflux pumps - not just for resistance. Nat Rev. Microbiol. 4 (8), 629-636 (2006).
- Käufer, N. F., Simanis, V., Nurse, P. Fission yeast Schizosaccharomyces pombe correctly excises a mammalian RNA transcript intervening sequence. Nature. 318 (6041), 78-80 (1985).
- Vyas, A., Freitas, A. V., Ralston, Z. A., Tang, Z. Fission yeast Schizosaccharomyces pombe: A unicellular micromammal model organism. Curr Prot. 1 (6), e151(2021).
- Edwards, D. H., Thomaides, H. B., Errington, J. Promiscuous targeting of Bacillus subtilis cell division protein DivIVA to division sites in Escherichia coli and fission yeast. EMBO J. 19 (11), 2719-2727 (2000).
- Srinivasan, R., Mishra, M., Murata-Hori, M., Balasubramanian, M. K. Filament formation of the Escherichia coli actin-related protein, MreB, in fission yeast. Curr Biol. 17 (3), 266-272 (2007).
- Srinivasan, R., Mishra, M., Wu, L., Yin, Z., Balasubramanian, M. K. The bacterial cell division protein FtsZ assembles into cytoplasmic rings in fission yeast. Gene Dev. 22 (13), 1741-1746 (2008).
- TerBush, A. D., Osteryoung, K. W. Distinct functions of chloroplast FtsZ1 and FtsZ2 in Z-ring structure and remodeling. J Cell Biol. 199 (4), 623-637 (2012).
- Sharma, A. K., et al. A mechanism of salt bridge-mediated resistance to FtsZ inhibitor PC190723 revealed by a cell-based screen. Mol Biol Cell. 34 (3), ar16(2023).
- Basi, G., Schmid, E., Maundrell, K. TATA box mutations in the Schizosaccharomyces pombe nmt1 promoter affect transcription efficiency but not the transcription start point or thiamine repressibility. Gene. 123 (1), 131-136 (1993).
- Moreno, M. B., Durán, A., Carlos Ribas, J. A family of multifunctional thiamine-repressible expression vectors for fission yeast. Yeast. 16 (9), 861-872 (2000).
- Green, M. R., Sambrook, J. Caring for Escherichia coli. Cold Spring Harb Protoc. , (2018).
- Green, M. R., Sambrook, J. Isolation and quantification of DNA. Cold Spring Harb Protoc. , (2018).
- Bähler, J., et al. Heterologous modules for efficient and versatile PCR-based gene targeting in Schizosaccharomyces pombe. Yeast. 14 (10), 943-951 (1998).
- Pande, V., Mitra, N., Bagde, S. R., Srinivasan, R., Gayathri, P. Filament organization of the bacterial actin MreB is dependent on the nucleotide state. J Cell Biol. 221 (5), e202106092(2022).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Meth. 9 (7), 676-682 (2012).
- Zhang, L. Machine learning for enumeration of cell colony forming units. Vis Comput Ind Biomed Art. 5 (1), 26(2022).
- Moen, E., et al. Deep learning for cellular image analysis. Nat Meth. 16 (12), 1233-1246 (2019).
- Higaki, T. Quantitative evaluation of cytoskeletal organizations by microscopic image analysis. Plant Morphol. 29 (1), 15-21 (2017).
- Henty-Ridilla, J. L., Li, J., Day, B., Staiger, C. J. ACTIN DEPOLYMERIZING FACTOR4 regulates actin dynamics during innate immune signaling in Arabidopsis. Plant Cell. 26 (1), 340-352 (2014).
- Boudaoud, A., et al. an ImageJ plug-in to quantify fibrillar structures in raw microscopy images. Nat Protoc. 9 (2), 457-463 (2014).
- Butler, M. S., Paterson, D. L. Antibiotics in the clinical pipeline in October 2019. J Antibio. 73 (6), 329-364 (2019).
- Andreu, J. M., Huecas, S., Araújo-Bazán, L., Vázquez-Villa, H., Martín-Fontecha, M. The search for antibacterial inhibitors targeting cell division protein ftsz at its nucleotide and allosteric binding sites. Biomedicines. 10 (8), 1825(2022).
- Vještica, A., et al. A toolbox of stable integration vectors in the fission yeast Schizosaccharomyces pombe. J Cell Sci. 133 (1), jcs240754(2020).
- Berg, S., et al. ilastik: interactive machine learning for (bio)image analysis. Nat Meth. 16 (12), 1226-1232 (2019).
- Shaevitz, J. W., Gitai, Z. The structure and function of bacterial actin homologs. Cold Spring Harb Pers Biol. 2 (9), a000364(2010).
- Hoffman, C. S., Wood, V., Fantes, P. A. An ancient yeast for young geneticists: A primer on the Schizosaccharomyces pombe model system. Genetics. 201 (2), 403-423 (2015).
- Aoi, Y., et al. Dissecting the first and the second meiotic divisions using a marker-less drug-hypersensitive fission yeast. Cell Cycle. 13 (8), 1327-1334 (2014).
- Zhang, J., et al. Improving drug sensitivity of HIV-1 protease inhibitors by restriction of cellular efflux system in a fission yeast model. Pathogens. 11 (7), 804(2022).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados