Method Article
Spalthefe als Plattform für antibakterielle Wirkstoffscreenings, die auf bakterielle Zytoskelettproteine abzielen
In diesem Artikel
Zusammenfassung
Spalthefe wird hier als heterologer Wirt verwendet, um bakterielle Zytoskelettproteine wie FtsZ und MreB als translationale Fusionsproteine mit GFP zu exprimieren, um deren Polymerisation sichtbar zu machen. Auch Verbindungen, die die Polymerisation beeinflussen, werden durch Bildgebung mit einem Fluoreszenzmikroskop identifiziert.
Zusammenfassung
Bakterielle Proteine des Zytoskeletts wie FtsZ und MreB erfüllen wesentliche Funktionen wie die Zellteilung und die Aufrechterhaltung der Zellform. Darüber hinaus haben sich FtsZ und MreB als wichtige Ziele für die Entdeckung neuartiger antimikrobieller Wirkstoffe herausgestellt. Es wurden mehrere Assays entwickelt, um Verbindungen zu identifizieren, die auf die Nukleotidbindung und Polymerisation dieser Zytoskelettproteine abzielen, die sich hauptsächlich auf FtsZ konzentrieren. Darüber hinaus sind viele der Assays entweder aufwendig oder kostenintensiv, und um festzustellen, ob diese Proteine das zelluläre Ziel des Medikaments sind, sind oft mehrere Methoden erforderlich. Schließlich stellt auch die Toxizität der Medikamente für eukaryotische Zellen ein Problem dar. Hier beschreiben wir einen einstufigen zellbasierten Assay, um neuartige Moleküle zu entdecken, die auf das bakterielle Zytoskelett abzielen, und um Treffer zu minimieren, die für eukaryotische Zellen potenziell toxisch sein könnten. Spalthefe ist für Hochdurchsatz-Screenings auf der Grundlage der Mikroskopie zugänglich, und ein visueller Screen kann jedes Molekül, das die Polymerisation von FtsZ oder MreB verändert, leicht identifizieren. Unser Assay verwendet die Standard-96-Well-Platte und stützt sich auf die Fähigkeit der bakteriellen Zytoskelettproteine, in einer eukaryotischen Zelle, wie z. B. der Spalthefe, zu polymerisieren. Während die hier beschriebenen Protokolle für Spalthefe gelten und FtsZ aus Staphylococcus aureus und MreB aus Escherichia coli verwenden, sind sie leicht an andere bakterielle Zytoskelettproteine anpassbar, die sich in allen eukaryotischen Expressionswirten leicht zu Polymeren zusammensetzen. Die hier beschriebene Methode sollte dazu beitragen, die weitere Entdeckung neuartiger antimikrobieller Wirkstoffe zu erleichtern, die auf bakterielle Zytoskelettproteine abzielen.
Einleitung
Die weit verbreitete Resistenz gegen fast alle Antibiotika, die derzeit zur Bekämpfung bakterieller Infektionen eingesetzt werden, hat einen unmittelbaren Bedarf an neuen Kategorien von Antibiotika geschaffen. Ein Bericht aus dem Jahr 2019 zeigte, dass antibiotikaresistente Infektionen zum Verlust von 1,27 Millionen Menschenleben führten, was zu einer Gesamtzahl von 4,95 Millionen Todesfällen beitrug, wenn man die Komplikationen durch resistente bakterielle Infektionen berücksichtigte1. Während das derzeitige Arsenal an Antibiotika in der klinischen Praxis immer noch wirksam ist, zielt es hauptsächlich auf ein schmales Spektrum zellulärer Prozesse ab, die sich hauptsächlich auf die Zellwand-, DNA- und Proteinsynthese konzentrieren. In den letzten 50 Jahren wurden weniger als 30 Proteine kommerziell als Ziele für die Entwicklung neuer antibakterieller Wirkstoffe genutzt 2,3. Diese begrenzte Auswahl an praktikablen Zielen schränkt die Entdeckung neuer Antibiotika oder ihrer Derivate zur Bekämpfung antibiotikaresistenter Bakterien erheblich ein. Um das sich abzeichnende Problem der Antibiotikaresistenz zu überwinden, besteht daher die Notwendigkeit der Entwicklung neuer Antibiotika mit neuartigen Zielen und Wirkmechanismen.
Ein antibakterielles Ziel sollte idealerweise ein wesentlicher Bestandteil des bakteriellen Zellwachstums sein, in allen phylogenetisch vielfältigen Spezies konserviert sein, die geringste eukaryotische Homologie aufweisen und für Antibiotika zugänglich sein4. Seit der Entdeckung bakterieller Zytoskelettproteine, die an der Zellteilung und der Aufrechterhaltung der Zellform beteiligt sind, haben sie sich zu einem vielversprechenden Schwerpunkt für die Entwicklung antibakterieller Wirkstoffe entwickelt5. Diese Proteine sind essentiell für die Lebensfähigkeit von Bakterien und spielen eine entscheidende Rolle bei der Aufrechterhaltung der Zellform (MreB, CreS), der Teilung (FtsZ, FtsA) und der DNA-Segregation (ParM, TubZ, PhuZ, AlfA), ähnlich dem Zytoskelett in eukaryotischen Zellen. Bemerkenswert ist, dass FtsZ ein bemerkenswert hohes Maß an Konservierung bei einer Vielzahl von prokaryotischen Organismen aufweist, während MreB in fast allen stäbchenförmigen Bakterien vorkommt. Diese weite Verbreitung und Relevanz für die Lebensfähigkeit von Zellen machen diese Proteine zu einem faszinierenden Ziel in der Antibiotikaforschung 5,6,7.
Es ist von entscheidender Bedeutung, einen mehrgleisigen Ansatz zu verfolgen, der In-vivo-Beobachtungen, In-vitro-Interaktionen und enzymatische Experimente kombiniert, um bakterielle Zytoskelettproteine als primäres Ziel eines potenziellen Inhibitors gründlich zu validieren7. Aufwändige Verfahren oder erhebliche Kostenauswirkungen belasten viele verfügbare Assays für diesen Zweck. Dies sind bemerkenswerte Hindernisse für ihre weit verbreitete Verwendung beim Screening von Leitverbindungen, die das bakterielle Zytoskelett beeinträchtigen könnten. Unter diesen sticht die Mikroskopie als eine außergewöhnlich effiziente und schnelle Methode zur Beurteilung der Wirksamkeit von Verbindungen hervor, indem Veränderungen in der Zellmorphologie direkt untersucht werden. Die Heteroassoziation des Zielproteins mit anderen Zytoskelettkomplexen, indirekte Effekte aufgrund von Off-Target-Bindung und Veränderungen des Membranpotentials, Schwierigkeiten bei der effizienten Durchdringung der Zelle und das Vorhandensein von Effluxpumpen, insbesondere in gramnegativen Bakterien, machen es jedoch komplex, die genaue Ursache der bakteriellen Zelldeformation zu bestimmen 8,9,10.
Schizosaccharomyces pombe, oder Spalthefe, wie sie allgemein genannt wird, ist ein stäbchenförmiger, einzelliger eukaryotischer Organismus. Die Spalthefe wird aufgrund der außergewöhnlichen Konservierung zellulärer Prozesse wie dem Zellzyklus und der Zellteilung, der zellulären Organisation und der Chromosomenreplikation mit höheren Eukaryoten, einschließlich des Menschen, häufig als Modellorganismus in der Zell- und Molekularbiologie verwendet11,12. Darüber hinaus exprimierten Errington und Kollegen ein pollokalisiertes bakterielles Zytoskelettprotein DivIVA in Spalthefe, um zu zeigen, dass sich DivIVA auf negativ gekrümmten Oberflächen ansammelte13. Auch hier hatten Balasubramanian und seine Gruppe zunächst die Spalthefe als zelluläres Modellsystem etabliert, um neue Einblicke in den Mechanismus, die Assemblierung und die Dynamik des Aktin-Zytoskelett-Proteins von E. coli wie MreB14 und dem Tubulin-Homolog FtsZ15 zu gewinnen. Sie zeigen auch die Fähigkeit von A22, die Polymerisation von MreB durch Epifluoreszenzmikroskopie effizient zu behindern, wenn es in Hefeexprimiert wird 14. Im Anschluss daran haben auch andere Gruppen erfolgreich Spalthefe eingesetzt, um die Assemblierungseigenschaften der Chloroplasten FtsZ1 und FtsZ2 Proteine zu untersuchen16. In jüngerer Zeit haben wir einen Proof-of-Concept für die Machbarkeit der Verwendung von Spalthefe als zelluläre Plattform für das spezifische Screening auf bakterielle Zytoskelett-Inhibitoren erstellt, indem wir eine umfassende Bewertung der Auswirkungen von drei bekannten FtsZ-Inhibitoren - Sanguinarin, Berberin und PC190723-on-FtsZ-Proteine - durchgeführt haben, die von zwei pathogenen Bakterien, nämlich Staphylococcus aureus und Helicobacter pylori17. Darüber hinaus erweist sich dieser einstufige zellbasierte Assay als entscheidend für die Minimierung des Risikos bei der Identifizierung von Verbindungen, die für eukaryotische Zellen potenziell toxisch sein können.
In diesem Bericht schlagen wir unter Verwendung des Spalthefesystems einen systematischen Arbeitsablauf vor, der die Standard-96-Well-Platte für das halbautomatische Screening und die Quantifizierung der Wirkung von niedermolekularen Inhibitoren verwendet, die auf FtsZ aus Staphylococcus aureus und MreB aus Escherichia coli abzielen. Hier haben wir den teilautomatisierten Workflow mit den etablierten Inhibitoren PC190723 und A22 aufgebaut und optimiert, die spezifisch auf FtsZ bzw. MreB abzielen. Bei diesem Arbeitsablauf wird ein Epifluoreszenzmikroskop verwendet, das mit einem motorisierten, hochpräzisen Tisch und einer automatisierten Bildaufnahme in einer Standard-96-Well-Platte ausgestattet ist, um die derzeitige Standardisierung zu verbessern. Daher kann es sowohl auf Mittel- als auch auf Hochdurchsatz-Screenings von synthetischen chemischen Bibliotheken angewendet werden und umgeht einige der oben genannten Herausforderungen.
Protokoll
1. Expression von GFP-markierten bakteriellen Zytoskelettproteinen in S. pombe
HINWEIS: In Tabelle 1 finden Sie Informationen zu allen hier verwendeten Plasmiden und Stämmen. In Tabelle 2 finden Sie alle Medienzusammensetzungen.
- Durchführung der Klonierung von E. coli MreB mit einer N-terminalen GFP-Fusion (GFP-MreB) und S. aureus FtsZ mit einem C-terminalen GFP (SaFtsZ-GFP) in den S. pombe-Expressionsvektor, pREP42 mit einem mittelstarken Thiamin-repressiblen Promotor, nmt4118,19, wie zuvor beschrieben14,15. Erhaltung, Amplifikation und Isolierung der Plasmide aus E. coli-Stämmen (DH10β), wie in20,21 beschrieben.
HINWEIS: Andere E. coli-Stämme wie DH5α, XL1Blue, TOP10 usw. oder andere im Handel erhältliche kompetente Zellen, die routinemäßig für die molekulare Klonierung verwendet werden, können ebenfalls verwendet werden. - Umwandlung der Plasmide pREP42-GFP-EcMreB und pREP42-SaFtsZ-GFP in S. pombe
- Transformieren Sie die Plasmide pCCD3 (pREP42-GFP-EcMreB) und pCCD713 (pREP42-SaFtsZ-GFP) in den S. pombe-Stamm (h-leu1-32 ura4-D18) unter Verwendung der Lithiumacetat-Methode22, wie in den folgenden Schritten erwähnt.
- Tag 1 - Primärkultur: Impfen Sie eine Schleife voll frisch ausgestreckter S. pombe-Kultur in 3 ml autoklaviertem Hefeextrakt und Nahrungsergänzungsmitteln (YES) Brühe. Inkubieren Sie es in einem Orbitalschüttler bei 30 °C über Nacht (O/N).
- Tag 2:
- Sekundärkultur: Fügen Sie etwa 500 μl Primärkultur zu 30 mL autoklavierter YES-Brühe hinzu. Bei 30 °C 3 - 4 h unter Schütteln inkubieren, bis der OD600 0,4 - 0,6 erreicht.
HINWEIS: Für jede Transformation werden 30 mL verwendet. - Pelletieren Sie die 30 mL Kultur bei 2.500 x g für 6-8 min bei Raumtemperatur. Entsorgen Sie den Überstand und waschen Sie die Zellen mit 50 ml sterilem destilliertem Wasser (D/W). Pelletieren Sie wieder und entsorgen Sie den Überstand. Resuspendieren Sie die Zellen in 1 mL sterilem D/W und überführen Sie sie in ein 2 mL Zentrifugenröhrchen. Wie oben zentrifugieren und Überstand verwerfen.
- 1 ml 0,1 M Lithiumacetat, Tris-EDTA (LiAc-TE) Lösung zugeben und wie oben zentrifugieren. Den Überstand verwerfen und in 1 ml 0,1 M LiAc-TE resuspendieren. Zentrifugieren und Überstand verwerfen, wobei 100 μl Lösung zurückbleiben.
- Fügen Sie 10 - 20 μg Träger-DNA (Lachsspermien-DNA; denaturiert und auf Eis schockgekühlt) und 2 - 3 μg der Plasmid-DNA hinzu, die transformiert werden muss. Vorsichtig mischen. 10 min bei Raumtemperatur inkubieren.
- 260 μl 40 % PEG/LiAc-TE zugeben; Vorsichtig mischen. 60 min im Shaker oder einem Thermomixer bei 30 °C unter schonendem Mischen inkubieren. Fügen Sie 43 μl DMSO hinzu; Vorsichtig mischen.
- Hitzeschock bei 42 °C für 10 min im Thermomixer. Bei 2.500 x g für 6-8 min pelletieren und Überstand entsorgen. Waschen Sie das Pellet 1x mit 1 mL sterilem D/W.
- Pelletieren, Überstand entsorgen und das Pellet in 200 μl sterilem D/W resuspendieren. Platte 100 μl auf Edinburgh-Minimalmediumplatten (EMM) mit 5 μg/ml Thiamin (zur Unterdrückung des nmt41-Promotors ) und den Aminosäurezusätzen Adenin (0,225 mg/ml), Histidin (0,225 mg/ml) und Leucin (0,225 mg/ml), aber ohne Uracil (Selektionsmarker für pREP42-Plasmid).
HINWEIS: Abwechselnd 70 μl und 130 μl in zwei verschiedenen Platten auffüllen. Zwei verschiedene Volumina (70 μl und 130 μl) werden plattiert, um isolierte Kolonien auf mindestens einer der Platten zu erhalten, abhängig von der Transformationseffizienz, die von Experiment zu Experiment variieren kann. - Inkubieren Sie die Platten bei 30 °C für 2-3 Tage, bis die Bienenvölker erscheinen. Mischen Sie eine einzelne Kolonie mit 100 μl sterilem D/W und verteilen Sie sie auf einer frischen EMM-Platte, die Thiamin enthält, aber kein Uracil enthält, wie oben beschrieben.
- Bei 30 °C 2-3 Tage inkubieren, bis ein kompletter Rasen entsteht. Den gewachsenen Rasen der Zellen mit einer Impfschlaufe verschrotten und in einem Kryofläschchen auf 1 ml YES-Medium mit 30 % Glycerin resuspendieren.
- Das Kryo-Fläschchen in flüssigem Stickstoff schockfrosten und bei -80 °C lagern, um die gefrorenen Hefebrühen zu konservieren.
- Sekundärkultur: Fügen Sie etwa 500 μl Primärkultur zu 30 mL autoklavierter YES-Brühe hinzu. Bei 30 °C 3 - 4 h unter Schütteln inkubieren, bis der OD600 0,4 - 0,6 erreicht.
- Expression von bakteriellen Zytoskelettproteinen in Spalthefe
- Streichen Sie ein Pflaster aus dem Glycerinvorrat aseptisch auf eine frische hefespezifische Platte, um genügend Kultur für weitere Experimente zu erhalten.
HINWEIS: Im Fall von pREP42 verwendeten wir eine EMM-Agarplatte (minimales Medium), die Adenin, Histidin und Leucin enthielt (wie bei Verwendung des S. pombe-Stammes (h-leu1-32 ura4-D18)) ohne Uracil (Uracil in pREP42 als Auswahlmarker). Wir fügen 15 - 20 μM Thiamin zu den Agarplatten hinzu (da pREP42 einen thiaminhaltigen repressiblen nmt41-Promotor hat), um die Expression des interessierenden Gens beim Wachstum auf der Platte zu unterdrücken. - Eine kleine Schleife des Inokulums aus dem gestreiften Fleck in 5 mL hefespezifisches EMM-Medium ohne Thiamin inokulieren und 10 - 12 h bei 30 °C inkubieren.
HINWEIS: Die Expression von FtsZ und MreB aus verschiedenen Spezies unter dem nmt41-Promotor in S. pombe kann variieren, typischerweise zwischen 16 und 30 Stunden. Die optimale Zeit für die Proteinexpression von GFP-EcMreB und SaFtsZ-GFP in S. pombe beträgt 20 - 24 h bzw. 16 - 20 h bei 30 °C ohne Thiamin.
- Streichen Sie ein Pflaster aus dem Glycerinvorrat aseptisch auf eine frische hefespezifische Platte, um genügend Kultur für weitere Experimente zu erhalten.
2. Behandlung von S. pombe-Kulturen , die GFP-markierte bakterielle Zytoskelettproteine exprimieren
HINWEIS: Eine Reihe verschiedener Wirkstoffmoleküle wird an der über Nacht gezüchteten Hefekultur in einer 96-Well-Platte getestet.
- Beimpfen Sie eine frische Kultur, indem Sie 50 μl der Übernachtkultur in jede Vertiefung einer 96-Well-Platte mit 150 μl frischem Hefe-EMM-Medium mit dem halbautomatischen 96-Well-Mehrkanal-Pipettiergerät übertragen.
- Während des Ein- und Auspipettierens für das richtige Mischen mit einer halbautomatischen 96-Well-Mehrkanalpipette führen Sie langsam unterschiedliche ansteigende Konzentrationen von Arzneimitteln in die wachsende Kultur ein, jeweils in dreifacher Ausfertigung, wie im Schema in Abbildung 1 dargestellt.
- Als Positivkontrolle sind die bereits bekannten Arzneimittel PC190723 für (S. aureus FtsZ) und A22 (für E. coli MreB) in doppelter Ausfertigung zu verwenden.
HINWEIS: Wie bereits berichtet, wurden PC190723 und A22 in einer Konzentration von 56,2 μM17 bzw. 72,6 μM 14,17 verwendet. Währenddessen dienten die A22-Behandlung von SaFtsZ bzw. die PC190723-Behandlung von EcMreB als Negativkontrollen. - Verwenden Sie DMSO als Lösungsmittelkontrolle in drei verschiedenen Konzentrationen, je nach der niedrigsten und der höchsten Konzentration von Arzneimitteln.
HINWEIS: Das Lösungsmittel, in dem Arzneimittel gelöst werden, wird als Lösungsmittelkontrolle verwendet. - Inkubieren Sie die kontrollierten und behandelten Kulturen bei 30 °C für 6-10 Stunden (bis die Zellen die Expression von bakteriellen fluoreszierenden Proteinen zeigen) und bilden Sie sie dann mit der Epifluoreszenzmikroskopie ab.
3. Visualisierung der Polymere
- Tragen Sie eine Beschichtung von 20 μl 1 mg/ml Concanavalin A auf jede Vertiefung der optisch transparenten unteren 96-Well-Platte auf (schwarzwandig für Fluoreszenzbildgebung) auf und inkubieren Sie sie 20 Minuten lang bei Raumtemperatur. Flüssigkeit ansaugen und 10 min an der Luft trocknen lassen.
- Übertragen Sie 20 μl Zellen aus der Kulturplatte in die jeweilige Vertiefung und lassen Sie sie 10 Minuten ruhen. Waschen Sie die Zelle 3x-4x mit dem sterilen EMM-Medium.
- Wenn die Objektivlinse an Ort und Stelle verwendet werden soll (siehe Schritt 3.6), verwenden Sie den Navigator-Modus im Aufnahmefenster der Bildaufnahmesoftware für die Ausrichtung der 96-Well-Platte und die Well-Abdeckung. Richten Sie die Well-Kanten im Navigator aus.
- Wählen Sie als Nächstes die Well-Abdeckungsparameter aus, zu denen eine Auswahl des interessierenden Bereichs im Well mit mehreren Positionen gehört, und verwenden Sie die adaptive Autofokussteuerung mit dem On-Demand-Modus, um die Probe während der Bildgebung im Fokus zu halten.
- Erfassen Sie Bilder mit differentiellem Interferenzkontrast (DIC) und Fluoreszenz. Setze Anregungs- und Emissionsfilter von 475/28 nm bzw. 525/48 nm, um die GFP-markierten bakteriellen Proteine abzubilden, die in Hefe exprimiert werden. Erhalten Sie Z-Stacks in einer Schrittweite von 0,2 μm durch die Dicke der Hefezellen (5 μm).
- Nehmen Sie Bilder mit einem inversen Epifluoreszenzmikroskop auf, das mit einem 100-fachen 1,4-NA-Ölimmersionsobjektiv und einer 2.000 x 2.000 sCMOS-Kamera (6,5 μm x 6,5 μm Pixelgröße) ausgestattet ist. Verwenden Sie ein LED-Beleuchtungssystem zur Anregung des Fluorophors.
HINWEIS: Jedes Epifluoreszenzmikroskopsystem, das über einen motorisierten Tisch für eine 96-Well-Platte mit präziser Mehrpunktpositionierung verfügt, sollte geeignet sein. Erfassen Sie alle Zellbilder der Kontroll- und Arzneimittelbehandlung mit einer ähnlichen Belichtungszeit (in der Regel im Bereich von 0,3 - 0,5 s) bei einem Binning von 1 x 1 und einer Beleuchtung von 15 % - 20 %, um die Versuchsvariablen zu minimieren und die Konsistenz zu wahren.
4. Quantifizierung der Bilder mit ImageJ
- Verarbeiten Sie Zellbilder und messen Sie die Anzahl der Spots pro Zelle und die Dichte für FtsZ17. Im Fall von MreB messen Sie die Dichte und Anisotropie23 und vergleichen Sie zwischen Kontrollzellen und behandelten Zellen mit Fiji (v2.0.0-rc-69/1.52p)24.
- Anhand der Bilder aus dem DIC-Kanal skizzieren Sie zunächst die einzelnen Hefezellen mit dem Freihand-Zeichenwerkzeug und speichern sie als Region of Interest (ROI) im ROI-Manager. Dieser Schritt ist noch nicht automatisiert, aber neu entwickelte Tools mit maschinellem Lernen25,26 könnten in naher Zukunft ausprobiert und integriert werden.
- Maskieren Sie den äußeren Bereich der Zelle und füllen Sie schwarz, wie in27 beschrieben.
- Verwenden Sie das Analysemakro IJ1 des OPS-Schwellenwerts, eine in Fidschi integrierte Funktion, um die Anzahl der Punkte24 zu zählen.
- Verwenden Sie die otsu-Methode für die automatische Schwellenwertbestimmung und maskieren Sie die segmentierten Partikel. Führen Sie das Plug-In aus, um Partikel mit einem Makro zu analysieren.
- Zur Messung der Polymerdichte (Menge des Zytoskeletts pro Flächeneinheit in einer Zelle) sind die Bilder wie beschrieben zu verarbeiten, einschließlich der Maskierungs- und Skelettierungsschritte27, 28. Verwenden Sie Lpx 2Dfilter im lpx-Plug-in, um die Bilder zu skelettieren.
- Weitere Informationen finden Sie in dieser aktuellen Veröffentlichung17. Führen Sie die EcMreB-Polymerquantifizierung durch, wie zuvor in23 erwähnt, und verwenden Sie die Anisotropie, um die räumliche Organisation mit FibrilTool29 zu quantifizieren, wie zuvorbeschrieben 23.
HINWEIS: Diese Analysemethoden können verwendet werden, um alle anderen bakteriellen Zytoskelettproteine zu quantifizieren, die mit den Arzneimitteln behandelt oder unbehandelt sind.
Ergebnisse
Aufbau der 96-Well-Platte für das Screening von Medikamenten
Die Verwendung von S. pombe zur Expression eines C-terminalen GFP-markierten S. aureus FtsZ aus einem Vektor (pREP42), der den mittelstarken thiaminhaltigen repressiblen Promotor nmt41enthält, wurde zuvor nachgewiesen17 und in ähnlicher Weise wurde das mit N- terminalem GFP markierte E. coli MreB auch in S. pombe14 exprimiert. Wir haben auch gezeigt, dass PC190723, ein spezifischer Inhibitor von SaFtsZ, und A22, ein MreB-Inhibitor, ihre Wirkung auf die jeweiligen bakteriellen Zytoskelettproteine in spezifischer Weise ausüben können, wenn sie in Hefeexprimiert werden 14,17.
Hier schlagen wir vor, das Hefeexpressionssystem zu nutzen, um ein mittleres oder hohes Durchsatz-Screening von antibakteriellen Wirkstoffen zu entwickeln, die auf die bakteriellen Zytoskelettproteine abzielen. Ein Epifluoreszenzmikroskop mit einem motorisierten Tisch kann automatisiert werden, um eine 96-Well-Platte abzubilden, und kommerzielle Arzneimittelbibliotheken sind auch in 96-Well-Plattenformaten erhältlich. Wir bevorzugen daher die Verwendung einer 96-Well-Platte für das vorgeschlagene Screening von Medikamenten. Die Platten werden wie in Abbildung 1 dargestellt eingestellt. Die erste Reihe (Abbildung 1A) besteht aus SaFtsZ-GFP, das die Hefekultur exprimiert. Die zweite Reihe (Abbildung 1B) besteht aus Hefekulturen, die GFP-EcMreB exprimieren. Die ersten beiden Spalten der ersten beiden Zeilen (A1:B2) werden als leeres Medium festgelegt. Die folgenden sechs Spalten der ersten beiden Zeilen (A3:B12) sind als DMSO-Kontrolle mit drei verschiedenen Konzentrationen entsprechend den Arzneimittelverdünnungen in doppelter Ausführung eingestellt. Als nächstes werden PC190723 (56,2 μM) und A22 (72,6 μM) zu Hefezellen hinzugefügt, die SaFtsZ-GFP oder GFP-EcMreB exprimieren. PC190723 und A22 dienen als Positivkontrollen für FtsZ bzw. MreB. Alle anderen Vertiefungen (C1:F12) werden für die Zugabe verschiedener Medikamente (in drei verschiedenen Konzentrationen und in doppelter Vielfalt) verwendet, die im Screening zur Identifizierung von Effektoren der bakteriellen Zytoskelettproteine, SaFtsZ oder EcMreB, verwendet werden. Hefekulturen, die das GFP-markierte SaFtsZ oder MreB exprimieren, werden in diese 96-Well-Platten abgegeben und gezüchtet, bis die Auswirkungen der Wirkstoffe auf den Zusammenbau von Polymeren sichtbar sind.
Abschätzung von Wachstumseffekten auf Hefezellen
Nach der Inkubation des 96-Wells mit subkultivierten Zellen bei 30 °C für 6 bis 10 Stunden wird die optische Dichte (OD) der Hefekultur mit einem Mikroplatten-Reader gemessen. Die Messung der OD hilft bei der Beurteilung einer wachstumshemmenden Wirkung der Arzneimittel gegen die eukaryotischen Hefezellen und möglicherweise schädlicher Auswirkungen auf menschliche Zellen. Somit kann dieser Assay verwendet werden, um mehrere Arzneimittel basierend auf ihrer Toxizität im Hefesystem auszusieben. Weder PC190723 noch A22 zeigen jedoch Wachstumseffekte oder Toxizität für Hefezellen, und die OD600 der Kulturen, die SaFtsZ-GFP exprimieren und mit DMSO, PC190723 oder A22 behandelt wurden, betrugen 0,38 ± 0,03, 0,44 ± 0,02 bzw. 0,43 ± 0,06 (N ≥ 4). Ebenso betrugen die OD600 der Kulturen, die GFP-EcMreB exprimieren und mit DMSO, PC190723 oder A22 behandelt wurden, 0,38 ± 0,04, 0,44 ± 0,07 bzw. 0,41 ± 0,08 (N ≥ 4).
Visualisierung der Wirkung der Wirkstoffe auf die polymeren Strukturen, die durch SaFtsZ und EcMreB in S. pombe exprimiert werden
Um die Wirkung der Wirkstoffe auf die Polymerisation von SaFtsZ oder EcMreB zu beurteilen, wird ein Aliquot der Hefezellen aus der oben erwähnten 96-Well-Platte, die Wirkstoffe enthält, auf eine andere 96-Well-Platte mit optisch klarem Glasboden 96-Well, die für die Fluoreszenzbildgebung geeignet ist, übertragen. Diese 96-Well-Platte ist ebenfalls mit Concanavalin A beschichtet, um die Adhäsion der Hefezellen zu ermöglichen, die das GFP-markierte SaFtsZ oder EcMreB exprimieren. Die 96-Well-Platte wird mit einem Epifluoreszenzmikroskop abgebildet, das alle 96-Wells der Platte an den vorgegebenen Positionen abdeckt, die von der Bildaufnahmesoftware gesteuert werden (Abbildung 2). In den Kontrollvertiefungen (DMSO-behandelt) zeigten Hefezellen, die SaFtsZ-GFP exprimierten, polymere Strukturen in Form von Flecken oder Flecken, die in den Zellen verteilt waren, nach 16 - 18 Stunden Wachstum in Abwesenheit von Thiamin. 10 - 12 h nach dem Wachstum des Thiaminmangels zeigten die Zellen jedoch nur diffuse Fluoreszenz oder wenige Flecken, was darauf hindeutet, dass die FtsZ-GFP-Expression nicht die für die Polymerisation erforderliche kritische Konzentration erreicht hatte (Abbildung 3A). Im Gegensatz dazu zeigte das FtsZ-stabilisierende Medikament, PC190723, eine signifikante Zunahme der polymeren Strukturen (Spots oder Patches) von SaFtsZ-GFP im Vergleich zur DMSO-Kontrolle nach 12 Stunden Wachstum (Abbildung 3B), die als Positivkontrolle diente. Im Gegensatz dazu zeigten Zellen, die mit A22 behandelt wurden, keinen Unterschied in den assemblierten SaFtsZ-GFP-Strukturen (Abbildung 3C). Anders als in Kulturen, die mit PC190723 behandelt wurden, assemblierte sich SaFtsZ-GFP in unbehandelten Kulturen jedoch nur dann zu Pflastern, wenn es über längere Zeiträume induziert wurde, was zeigt, dass PC190723 wirkte, um die kritische Polymerisationskonzentration von SaFtsZ zu reduzieren (Abbildung 3D). Ebenso bildete GFP-EcMreB, exprimiert in S. pombe , lineare Anordnungen langer Filamente entlang der Längsachse der Spalthefe (Abbildung 4A). Während PC190723 keinen Einfluss auf die EcMreB-Polymerisation hatte (Abbildung 4B), führte die Behandlung von Zellen mit A22 zu einer diffusen Fluoreszenz im gesamten Zytoplasma der Hefezellen (Abbildung 4C). Die Bilder der restlichen 96-Well-Platten werden visuell auf Stabilisierung oder inhibitorische Wirkungen der Medikamente auf das SaFtsZ bzw. EcMreB überprüft.
Der Einfluss der Medikamente auf die Assemblierung bakterieller Zytoskelettproteine kann dann mit Bildanalysewerkzeugen und Plug-ins in ImageJ oder Fidschi quantifiziert werden, wie bereits für SaFtsZ17 und EcMreB21 berichtet. Die automatisierte Bildaufnahme im 96-Well-Plattenformat und die Implementierung von benutzerdefinierten Makros und Skripten für die Bildverarbeitung können die Bildgebungszeit und die Quantifizierung der Wirkung der Medikamente beschleunigen. Unter Verwendung der einzelligen eukaryotischen Hefe S. pombe als Wirtssystem schlagen wir vor, dass ein Screening mit mittlerem oder hohem Durchsatz auf kleine Moleküle, die auf die bakteriellen Zytoskelettproteine abzielen, erfolgreich durchgeführt werden kann, was schließlich zur Entdeckung neuer Antibiotika führt.
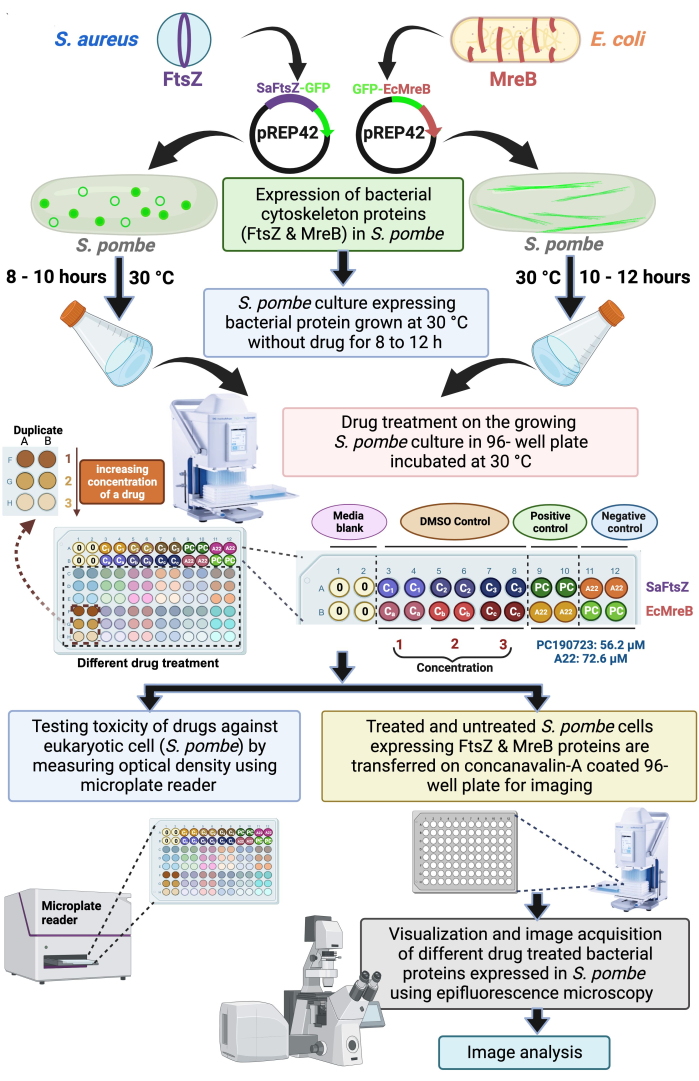
Abbildung 1: Schematische Darstellung der Verwendung von Spalthefezellen zum Screening nach Arzneimitteln, die auf den Zusammenbau der bakteriellen Zytoskelettproteine SaFtsZ oder EcMreB abzielen. FtsZ aus S. aureus und MreB aus E. coli wurden in den Hefeexpressionsvektor kloniert, wobei pREP42 einen GFP-Tag am C- bzw. N-Terminus aufweist und für die phänotypische und medikamentöse Studie in S. pombe transformiert. Die Kulturen wurden 8 - 12 h lang gezüchtet. Anschließend wurden die Kulturen in 96-Well-Platten mit geeigneten Kontrollen subkultiviert und die verschiedenen Medikamente gescreent. Des Weiteren wurde die Platte bei 30 °C für 6 - 8 Stunden inkubiert, bis der OD600 0,5-0,6 erreichte, und die optische Dichte wurde mit einem Mikroplatten-Reader gemessen, um die Toxizität der Arzneimittel gegenüber Hefezellen zu beurteilen. Eine weitere 96-Well-Platte mit optisch klarem Glasboden wurde mit Concanavalin A vorbeschichtet, um Hefezellen anhaften zu können. Diese 96-Well-Platte wurde für die Visualisierung und Bildaufnahme mit einem Epifluoreszenzmikroskop verwendet. Die erhaltenen Bilder wurden anschließend analysiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
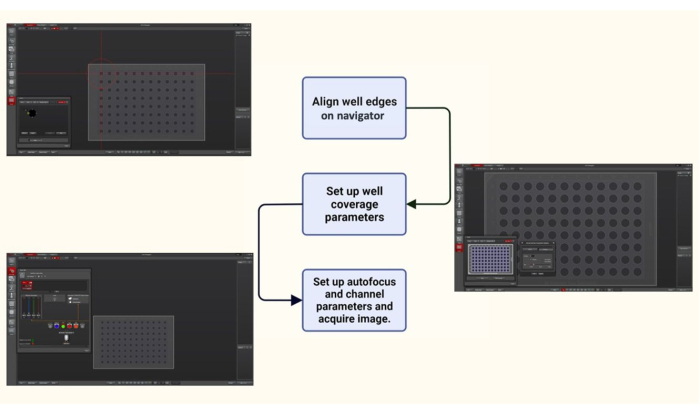
Abbildung 2: Schematische Darstellung der Bilderfassungssoftware für die automatisierte Bildgebung einer 96-Well-Platte. Mit dem Navigator in der Bilderfassungssoftware wurde eine 96-Well-Platte durch Markieren der Kanten ausgerichtet. Well-Coverage-Parameter wie die Anzahl der Bilder, die von jedem Well aufgenommen werden müssen, und seine Position (zufällig oder von der Mitte des Wells) wurden wie gewünscht ausgewählt. Schließlich werden Bildparameter wie der zu verwendende Filtersatz (DIC und FITC-Filter; Ex 475/28 nm und Em 525/48 nm) wurden die prozentuale Intensität des Beleuchtungslichts, der Autofokus und die Belichtungszeit für die Bildaufnahme eingestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Visualisierung der Wirkung von PC190723- und A22-Wirkstoffen auf SaFtsZ-GFP, exprimiert in S. pombe. S. pombe-Kultur , die SaFtsZ-GFP exprimiert, wurde in Abwesenheit von Thiamin für 8 - 9 h bei 30 °C vor DMSO und medikamentöser Behandlung gezüchtet. Die Kultur wurde in einer 96-Well-Platte für 7 - 9 h bei 30 °C in Gegenwart oder Abwesenheit der Medikamente weiter gezüchtet. (A) Bei der DMSO-Kontrolle, bei der eine äquivalente Menge DMSO zugesetzt wurde, wiesen die Zellen einige polymere Strukturen auf. (B) In Gegenwart von PC190723 (56,2 μM) exprimiert SaFtsZ Hefezellen, die eine beträchtliche Zunahme der polymeren Strukturen gegenüber der DMSO-Kontrolle aufweisen. (C) Der MreB-Inhibitor A22 (72,6 μM) hatte keinen Einfluss auf die SaFtsZ-GFP-Strukturen. (D) Für die Assemblierung von SaFtsZ-GFP waren längere Stunden der Proteinexpression in Abwesenheit von PC190723 erforderlich, und daher wiesen Kontrollkulturen, die 12 - 15 h nach der Subkultivierung in einer 96-Well-Platte, die DMSO enthielt, bei 30 °C gezüchtet wurden, ebenfalls FtsZ-Polymere auf. Der Maßstabsbalken beträgt 5 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
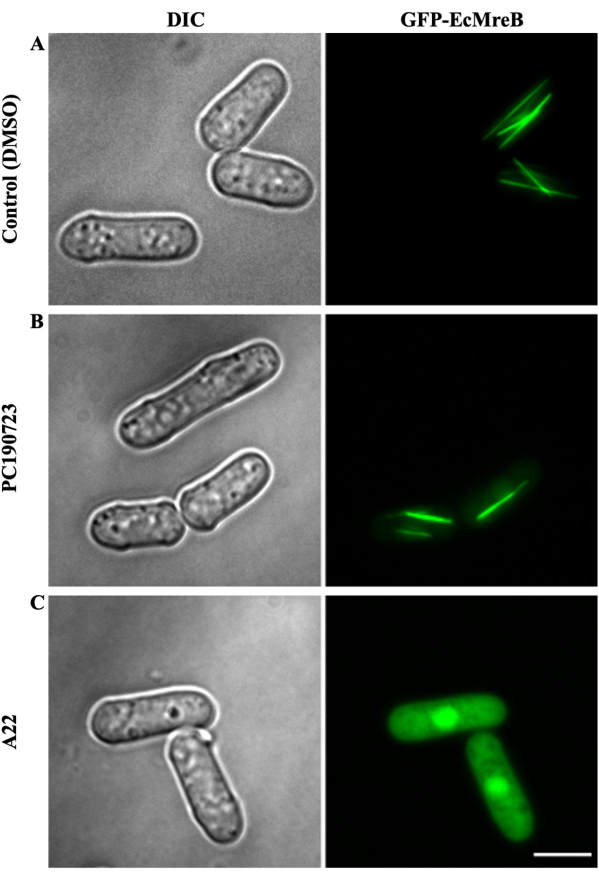
Abbildung 4: Visualisierung der Wirkung von PC190723- und A22-Wirkstoffen auf GFP-EcMreB, exprimiert in S. pombe. S. pombe-Kultur , die GFP-EcMreB exprimiert, wurde in Abwesenheit von Thiamin für 9 - 11 h bei 30 °C gezüchtet, bevor DMSO oder die Medikamente (A22 oder PC190723) hinzugefügt wurden. Die Kultur wurde in einer 96-Well-Platte für 8 - 10 h bei 30 °C mit und ohne Medikamente als Kontrolle weiter gezüchtet. (A) In Kulturen, in denen eine äquivalente Menge DMSO als Kontrolle zugesetzt wurde, wiesen die Zellen lineare Filamente von EcMreB auf, die entlang der Längsachse der Hefezellen ausgerichtet waren. (B) Die Assemblierung von GFP-EcMreB wurde in Kulturen, die mit PC190723 (56,2 μM) behandelt wurden, nicht beeinflusst. (C) Das kleine Molekül A22 (72,6 μM), ein bekannter Inhibitor der MreB-Polymerisation, verhinderte den Zusammenbau von GFP-EcMreB in Spalthefezellen. Der Maßstabsbalken beträgt 5 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tabelle 1: In dieser Studie verwendete Stämme und Plasmide. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 2: Zusammensetzung der in dieser Studie verwendeten Medien und Puffer. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
Antimikrobielle Resistenzen (AMR) sind eine ernsthafte globale Gesundheitsbedrohung, und es besteht ein dringender Bedarf an neuen Antibiotika mit neuen Zielen. Das bakterielle Zytoskelett hat sich als attraktives Ziel für die Entwicklung neuer Antibiotika erwiesen, wobei sich niedermolekulare Inhibitoren des Zellteilungsproteins FtsZ, wie TXA709, bereits in klinischen Phase-I-Studienbefinden 30. Es wurden mehrere Methoden entwickelt, um Inhibitoren der FtsZ-Polymerisationzu identifizieren 7,31. Wir haben kürzlich die eukaryotische Spalthefe in einem zellbasierten Assay verwendet, um zu zeigen, dass der FtsZ-stabilisierende Wirkstoff PC190723 auf HpFtsZ17 abzielen kann. Darüber hinaus zeigten wir, dass Medikamente wie Berberin und Sanguinarin zwar die Polymerisation von FtsZ beeinflussten, aber auch die Morphologie von Hefezellen beeinflussten17. Angeregt durch diese Studien und Beobachtungen machten wir uns daran, einen einstufigen zellbasierten Assay zu entwickeln, um unter Verwendung der Spalthefe-Expressionsplattform nach niedermolekularen Modulatoren des bakteriellen Zytoskeletts zu suchen. Da das Screening auf der Hemmung der Assemblierung von GFP-markierten Zytoskelettproteinen beruht, können kleine Moleküle mit ähnlichen Fluorophoreigenschaften zu falsch positiven Ergebnissen führen. Die Vermeidung von Langpassfiltern und die Wahl von Schmalbandfiltern kann dazu beitragen, diese Fehlalarme zu reduzieren, die durch fluoreszierende kleine Moleküle entstehen. Hier zeigen wir die Schritte, die bei der Einrichtung eines Screenings mit mittlerem Durchsatz unter Verwendung eines Epifluoreszenzmikroskops erforderlich sind, das mit einem 96-Well-Plattentischhalter ausgestattet ist, der zum Screening nach Medikamenten verwendet werden kann, die auf SaFtsZ oder EcMreB abzielen. Der Workflow umfasst eine halbautomatische Bilderfassung und -analyse, die in Zukunft mithilfe von robotischen Liquid-Handling-Systemen und Bildverarbeitungspipelines problemlos auf Hochdurchsatz-Bildschirme skaliert werden kann.
Des Weiteren sollte das Screening für andere bakterielle Zytoskelettproteine, wie das aktinfach enthaltende Plasmiderhaltungsprotein ParM32, und gegen jedes Protein, das sich in der Spalthefe zu polymeren Strukturen zusammensetzt, nützlich sein. Die optimalen Expressionsniveaus für jedes Protein von Interesse müssen jedoch standardisiert werden, bevor sie in Arzneimittel-Screenings verwendet werden. Während wir für FtsZs festgestellt haben, dass nmt41 ein optimaler Promotor ist, ist es für andere Proteine von Interesse eine stärkere oder schwächere Version des nmt1-Promotors (Promotorstärke (und Undichtigkeit): nmt1 > nmt41 > nmt81) oder anderer konstitutiver Promotoren wie der Hefe adh1 oder act118,33 Es kann versucht werden, ein optimales Maß an Expression und Assemblierung von Polymerstrukturen zu erreichen. Darüber hinaus stellen bei der Verwendung episomaler Plasmide auch Variationen der Kopienzahl von Zelle zu Zelle Probleme für eine einheitliche Genexpression dar. Um diese Probleme zu umgehen, wurden kürzlich mehrere Vektoren entwickelt, die in das Spalthefegenom integriert werden können (SIVs für stabile Integrationsvektoren), die Promotoren unterschiedlicher Stärke tragen34. Die Verwendung dieser Vektoren kann im Vergleich zu den episomalen Vektoren, die in dieser Studie verwendet wurden, zu einheitlicheren und quantitativeren Ergebnissen führen. Die Verwendung von SIVs könnte die große Heterogenität der Expressionsniveaus vermeiden, die als Variation der Fluoreszenzintensitäten zwischen den Zellen angesehen wird, und sollte besser für Hochdurchsatz-Screenings geeignet sein.
Darüber hinaus stabilisieren Medikamente wie PC190723 die FtsZ-Polymere, was zu einem frühen Zusammenbau von FtsZ-Filamenten im Vergleich zu unbehandelten Zellen führt. Während das Screening für das Screening von Arzneimitteln geeignet ist, die die Polymerisation von bakteriellen Zytoskelettproteinen hemmen, erfordert das Screening von Arzneimitteln, die FtsZ-Polymere stabilisieren, aufgrund der visuellen Beurteilung der diffusen Fluoreszenz am Endpunkt eine weitere Optimierung der Proteinexpressionsniveaus und der Zeit, die benötigt wird, um eine maximale Anzahl von Zellen mit Polymeren zu erreichen. Es ist daher von entscheidender Bedeutung, eine vernünftige Schätzung der Zeit zu haben, die für die Proteinexpression benötigt wird, um die für die Assemblierung erforderliche kritische Konzentration zu erreichen, und den Endpunkt für den Assay entsprechend zu bestimmen. Im Falle von Arzneimitteln, die wirken könnten, um die kritische Konzentration zu verringern, die für die Polymerisation des Proteins erforderlich ist, wäre die Wahl des Endpunkts typischerweise eine Zeit, in der eine beträchtliche Population unbehandelter Zellen diffuse Fluoreszenz aufweist, während Kulturen, die mit Arzneimitteln behandelt wurden, Polymeranordnungen aufweisen würden.
Eine der Einschränkungen, die auch ein zeitintensiver Schritt des Arbeitsablaufs ist, ist die Anforderung, einzelne Zellen für die quantitative Bildanalyse zu identifizieren und zu segmentieren. Derzeit handelt es sich in dem vorgeschlagenen Schema um einen manuellen Schritt, um einzelne Hefezellen zu identifizieren und die Region of Interest (ROIs) für die Quantifizierung der Wirkung der Medikamente zuzuweisen. Wir gehen jedoch davon aus, dass die kürzlich entwickelten Algorithmen des maschinellen Lernens unter Verwendung von Trainingssätzen, wie sie in ilastik35 implementiert werden können, den Bedarf an manuellen Eingriffen minimieren sollten.
Während unser Ansatz hier mit der Hefeplattform nützlich ist, um Treffer und potenzielle kleine Moleküle zu identifizieren, die auf das bakterielle Zytoskelett abzielen, muss man schließlich ihre antibakterielle Aktivität mit den Standardtests der minimalen Hemmkonzentration und die Wirksamkeit in Tiermodellen testen. Nichtsdestotrotz liegt die größte Herausforderung für die weltweite Zunahme von Antibiotikaresistenzen (AMR) nach wie vor in der äußeren Membranpermeabilität der gramnegativen Bakterien und den zahlreichen Effluxpumpen in vielen pathogenen Bakterienarten. Darüber hinaus können Effluxpumpen in Spalthefe ähnliche Probleme aufwerfen, und für den Screening-Prozess könnten höhere Wirkstoffkonzentrationen erforderlich sein. Alternativ können Spalthefestämme, die infolge der Deletion der sieben Multiresistenzgene, einschließlich vier Transportergenen und eines Transkriptionsfaktors, überempfindlich gegenüber Arzneimitteln sind (S. pombe MDR-supML), als Wirte für das Screening verwendet werden32,33. Zukünftige Entwicklungen bei Technologien, die die gezielte Entdeckung von Antibiotika kombinieren und Resistenzen aufgrund allgemeiner Phänomene wie der Membranpermeabilität angehen, werden notwendig sein, um die wachsende Antibiotikaresistenz zu bekämpfen.
Offenlegungen
Alle Autoren erklären, dass keine Interessenkonflikte bestehen.
Danksagungen
SMP, SR und AKS würdigen die Stipendien, die sie vom National Institute of Science Education and Research, Department of Atomic Energy, erhalten haben. RS bedankt sich für die interne finanzielle Unterstützung durch das Department of Atomic Energy, und diese Arbeit wird durch ein Forschungsstipendium des Department of Biotechnology (DBT) an RS (BT/PR42977/MED/29/1603/2022) unterstützt. Die Autoren danken auch V Badireenath Konkimalla für seine Kommentare, Vorschläge und Diskussionen während der Entwicklung des Protokolls.
Materialien
| Name | Company | Catalog Number | Comments |
| 96 Well CC2 Optical CVG Sterile, w/Lid. Black | Thermo Scientific™ | 160376 | |
| 96-well plate | Corning | CLS3370 | |
| A22 Hydrochloride | Sigma | SML0471 | Dissolved in DMSO |
| Adenine | FormediumTM | DOC0229 | 225 mg/L of media |
| Concanavalin A | Sigma | C5275-5MG | |
| DMSO | Sigma | 317275 | |
| Edinburg minimal medium (EMM Agar or EMM Broth) | FormediumTM | PMD0210 | See below for composition |
| EDTA | Sigma | EDS-500G | |
| epMotion® 96 with 2-position slider | Eppendorf | 5069000101 | |
| Histidine | FormediumTM | DOC0144 | 225 mg/L of media |
| Leica DMi8 inverted fluorescence microscope | Leica Microsystems | German company | |
| Leucine | FormediumTM | DOC0157 | 225 mg/L of media |
| Lithium acetate | Sigma | 517992-100G | |
| PC190723 | Merck | 344580 | Dissolved in DMSO |
| Polyethylene glycol (PEG) | Sigma | 202398 | |
| Thiamine | Sigma | T4625 | Filter sterilised |
| Tris-Hydrochloride | MP | 194855 | |
| Uracil | FormediumTM | DOC0214 | 225 mg/L of media, Store solution at 36°C |
| YES (Yeast extract + supplements) Agar | FormediumTM | PCM0410 | See below for composition |
| YES (Yeast extract + supplements) Broth | FormediumTM | PCM0310 | See below for composition |
| Yeast (S. pombe) media | |||
| Yeast extract + supplements (YES) | |||
| Composition | g/L | ||
| Yeast extract | 5 | ||
| Dextrose | 30 | ||
| Agar | 17 | ||
| Adenine | 0.05 | ||
| L-Histidine | 0.05 | ||
| L-Leucine | 0.05 | ||
| L-Lysine HCl | 0.05 | ||
| Uracil | 0.05 | ||
| Edinburg minimal medium (EMM) | |||
| Composition | g/L | concentration | |
| potassium hydrogen phthallate | 3 | 14.7mM | |
| Na2HPO4 | 2.2 | 15.5 mM | |
| NH4Cl | 5 | 93.5 mM | |
| glucose | 2% (w/v) or 20 g/L | 111 mM | |
| Salts (stock x 50) | 20 mL/L (v/v) | ||
| Vitamins (stock x 1000) | 1 mL/L (v/v) | ||
| Minerals (Stock x 10,000) | 0.1 mL/L (v/v) | ||
| Salts x 50 | 52.5 g/l MgCl2.6H20 (0.26 M) | 52.5 | 0.26 M |
| 0.735 mg/l CaCl2.2H20 (4.99 mM) | 0.000735 | 4.99 mM | |
| 50 g/l KCl (0.67 M) | 50 | 0.67 M | |
| 2 g/l Na2SO4 (14.l mM) | 2 | 14.1 mM | |
| Vitamins x 1000 | 1 g/l pantothenic acid | 1 | 4.20 mM |
| 10 g/l nicotinic acid | 10 | 81.2 mM | |
| 10 g/l inositol | 10 | 55.5 mM | |
| 10 mg/l biotin | 0.01 | 40.8 µM | |
| Minerals x 10,000 | boric acid | 5 | 80.9 mM |
| MnSO4 | 4 | 23.7 mM | |
| ZnSO4.7H2O | 4 | 13.9 mM | |
| FeCl2.6H2O | 2 | 7.40 mM | |
| molybdic acid | 0.4 | 2.47 mM | |
| KI | 1 | 6.02 mM | |
| CuSO4.5H2O | 0.4 | 1.60 mM | |
| citric acid | 10 | 47.6 mM | |
| Strains/ Plasmids | |||
| Strains | Description | Reference | |
| CCD190 | Escherichia coli DH10β | Invitrogen | |
| CCDY4 | MBY3532; CCDY346/pREP42- GFP-EcMreB | Srinivasan et al., 2007 | |
| CCDY340 | CCDY346/pREP42- SaFtsZ-GFP | Sharma et al., 2023 | |
| CCDY346 | MBY192; Schizosaccharomyces pombe [ura4-D18, leu1-32, h-] | Dr. Mithilesh Mishra (DBS, TIFR) | |
| Plasmids | |||
| pCCD3 | pREP42-GFP-EcMreB | Srinivasan et al., 2007 | |
| pCCD713 | pREP42-SaFtsZ-GFP | Sharma et al., 2023 |
Referenzen
- Antimicrobial Resistance Collaborators. Global burden of bacterial antimicrobial resistance in 2019: a systematic analysis. Lancet. 399 (10325), 629-655 (2022).
- Haselbeck, R., et al. Comprehensive essential gene identification as a platform for novel anti-infective drug discovery. Curr Pharma Des. 8 (13), 1155-1172 (2002).
- Wang, J., et al. Discovery of a small molecule that inhibits cell division by blocking FtsZ, a novel therapeutic target of antibiotics. J Biol Chem. 278 (45), 44424-44428 (2003).
- Sanchez, S., Demain, A. L. Antibiotics: current innovations and future trends. , Caister Academic Press. (2015).
- Vollmer, W. The prokaryotic cytoskeleton: a putative target for inhibitors and antibiotics. Appl Microbiol Biotechnol. 73 (1), 37-47 (2006).
- Vollmer, W. Targeting the bacterial Z-ring. Chem Biol. 15 (2), 93-94 (2008).
- Kusuma, K. D., Payne, M., Ung, A. T., Bottomley, A. L., Harry, E. J. FtsZ as an antibacterial target: status and guidelines for progressing this avenue. ACS Infect Dis. 5 (8), 1279-1294 (2019).
- Kaul, M., Zhang, Y., Parhi, A. K., Lavoie, E. J., Pilch, D. S. Inhibition of RND-type efflux pumps confers the FtsZ-directed prodrug TXY436 with activity against Gram-negative bacteria. Biochem Pharmacol. 89 (3), 321-328 (2014).
- Casiraghi, A., Suigo, L., Valoti, E., Straniero, V. Targeting bacterial cell division: A binding site-centered approach to the most promising inhibitors of the essential protein FtsZ. Antibiotics. 9 (2), 69(2020).
- Piddock, L. J. V. Multidrug-resistance efflux pumps - not just for resistance. Nat Rev. Microbiol. 4 (8), 629-636 (2006).
- Käufer, N. F., Simanis, V., Nurse, P. Fission yeast Schizosaccharomyces pombe correctly excises a mammalian RNA transcript intervening sequence. Nature. 318 (6041), 78-80 (1985).
- Vyas, A., Freitas, A. V., Ralston, Z. A., Tang, Z. Fission yeast Schizosaccharomyces pombe: A unicellular micromammal model organism. Curr Prot. 1 (6), e151(2021).
- Edwards, D. H., Thomaides, H. B., Errington, J. Promiscuous targeting of Bacillus subtilis cell division protein DivIVA to division sites in Escherichia coli and fission yeast. EMBO J. 19 (11), 2719-2727 (2000).
- Srinivasan, R., Mishra, M., Murata-Hori, M., Balasubramanian, M. K. Filament formation of the Escherichia coli actin-related protein, MreB, in fission yeast. Curr Biol. 17 (3), 266-272 (2007).
- Srinivasan, R., Mishra, M., Wu, L., Yin, Z., Balasubramanian, M. K. The bacterial cell division protein FtsZ assembles into cytoplasmic rings in fission yeast. Gene Dev. 22 (13), 1741-1746 (2008).
- TerBush, A. D., Osteryoung, K. W. Distinct functions of chloroplast FtsZ1 and FtsZ2 in Z-ring structure and remodeling. J Cell Biol. 199 (4), 623-637 (2012).
- Sharma, A. K., et al. A mechanism of salt bridge-mediated resistance to FtsZ inhibitor PC190723 revealed by a cell-based screen. Mol Biol Cell. 34 (3), ar16(2023).
- Basi, G., Schmid, E., Maundrell, K. TATA box mutations in the Schizosaccharomyces pombe nmt1 promoter affect transcription efficiency but not the transcription start point or thiamine repressibility. Gene. 123 (1), 131-136 (1993).
- Moreno, M. B., Durán, A., Carlos Ribas, J. A family of multifunctional thiamine-repressible expression vectors for fission yeast. Yeast. 16 (9), 861-872 (2000).
- Green, M. R., Sambrook, J. Caring for Escherichia coli. Cold Spring Harb Protoc. , (2018).
- Green, M. R., Sambrook, J. Isolation and quantification of DNA. Cold Spring Harb Protoc. , (2018).
- Bähler, J., et al. Heterologous modules for efficient and versatile PCR-based gene targeting in Schizosaccharomyces pombe. Yeast. 14 (10), 943-951 (1998).
- Pande, V., Mitra, N., Bagde, S. R., Srinivasan, R., Gayathri, P. Filament organization of the bacterial actin MreB is dependent on the nucleotide state. J Cell Biol. 221 (5), e202106092(2022).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Meth. 9 (7), 676-682 (2012).
- Zhang, L. Machine learning for enumeration of cell colony forming units. Vis Comput Ind Biomed Art. 5 (1), 26(2022).
- Moen, E., et al. Deep learning for cellular image analysis. Nat Meth. 16 (12), 1233-1246 (2019).
- Higaki, T. Quantitative evaluation of cytoskeletal organizations by microscopic image analysis. Plant Morphol. 29 (1), 15-21 (2017).
- Henty-Ridilla, J. L., Li, J., Day, B., Staiger, C. J. ACTIN DEPOLYMERIZING FACTOR4 regulates actin dynamics during innate immune signaling in Arabidopsis. Plant Cell. 26 (1), 340-352 (2014).
- Boudaoud, A., et al. an ImageJ plug-in to quantify fibrillar structures in raw microscopy images. Nat Protoc. 9 (2), 457-463 (2014).
- Butler, M. S., Paterson, D. L. Antibiotics in the clinical pipeline in October 2019. J Antibio. 73 (6), 329-364 (2019).
- Andreu, J. M., Huecas, S., Araújo-Bazán, L., Vázquez-Villa, H., Martín-Fontecha, M. The search for antibacterial inhibitors targeting cell division protein ftsz at its nucleotide and allosteric binding sites. Biomedicines. 10 (8), 1825(2022).
- Vještica, A., et al. A toolbox of stable integration vectors in the fission yeast Schizosaccharomyces pombe. J Cell Sci. 133 (1), jcs240754(2020).
- Berg, S., et al. ilastik: interactive machine learning for (bio)image analysis. Nat Meth. 16 (12), 1226-1232 (2019).
- Shaevitz, J. W., Gitai, Z. The structure and function of bacterial actin homologs. Cold Spring Harb Pers Biol. 2 (9), a000364(2010).
- Hoffman, C. S., Wood, V., Fantes, P. A. An ancient yeast for young geneticists: A primer on the Schizosaccharomyces pombe model system. Genetics. 201 (2), 403-423 (2015).
- Aoi, Y., et al. Dissecting the first and the second meiotic divisions using a marker-less drug-hypersensitive fission yeast. Cell Cycle. 13 (8), 1327-1334 (2014).
- Zhang, J., et al. Improving drug sensitivity of HIV-1 protease inhibitors by restriction of cellular efflux system in a fission yeast model. Pathogens. 11 (7), 804(2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten