Method Article
Interacción planta-microorganismo: Respuesta transcripcional de Bacillus Mycoides a exudados de raíces de papa
En este artículo
Resumen
El objetivo del protocolo presentado aquí es estudiar la respuesta de transcriptómicos de endosphere aislado de Bacillus mycoides a exudados de raíces de papa. Este método facilita la identificación de genes bacterianos importantes implicados en interacciones planta-microorganismo y es en principio aplicable a las plantas, con ajustes menores y otros endófitos.
Resumen
Bacterias beneficiosas planta juegan un papel importante en promover el crecimiento y prevención de enfermedades en plantas. La aplicación de planta rizobacterias promotoras del crecimiento (PGPR) como agentes biofertilizantes o control biológico se ha convertido en una alternativa eficaz al uso de fertilizantes convencionales y puede aumentar la productividad de los cultivos a bajo costo. Interacciones planta-microorganismo dependen de señales secretadas por la planta anfitrión y una reacción de aquí en adelante por sus bacterias asociadas. Sin embargo, los mecanismos moleculares de bacterias beneficiosas como responden a sus asociados de origen vegetal las señales no se entienden completamente. Valoración de la respuesta de transcriptómicos de bacterias en los exudados de la raíz es un poderoso enfoque para determinar la expresión génica bacteriana y Reglamento condiciones rizosféricos. Tal conocimiento es necesario entender los mecanismos subyacentes involucrados en interacciones planta-microorganismo. Este papel describe un protocolo detallado para estudiar la respuesta de transcriptómicos de B. mycoides EC18, una cepa aislada de la endosphere de la patata, a exudados de raíces de papa. Con la ayuda de la reciente tecnología de secuenciación de alto rendimiento, este protocolo puede realizarse en varias semanas y produce enormes conjuntos de datos. En primer lugar, recogemos los exudados de la raíz en condiciones estériles, después de lo cual se agregan a las culturas B. mycoides . El ARN de estas culturas se aísla método de fenol/cloroformo, combinada con un kit comercial y sujeto a control de calidad por un instrumento de electroforesis automatizada. Después de la secuenciación, análisis de datos se realizaron con la tubería T-REx en la web y se identifica un grupo de genes diferencialmente expresados. Este método es una herramienta útil para facilitar nuevos descubrimientos sobre los genes bacterianos implicados en interacciones planta-microorganismo.
Introducción
Las plantas pueden exudado hasta un 20% del carbono fijado durante la fotosíntesis a través de las raíces en la rizosfera1, es decir, la zona estrecha de tierra cerca de las raíces. Debido a la mayor disponibilidad de nutrientes, la rizosfera es un habitat conveniente para diversos microorganismos, incluyendo bacterias promotoras de crecimiento vegetal. Los exudados de la raíz contienen una gama de compuestos inorgánicos como agua, iones, ácidos inorgánicos y oxígeno. Sin embargo, la mayoría de los exudados de la raíz está formada por materiales orgánicos, que pueden dividirse en compuestos de bajo peso molecular y alto peso molecular compuestos. Los compuestos de bajo peso molecular son aminoácidos, ácidos orgánicos, azúcares, compuestos fenólicos, ácidos grasos y una gran variedad de metabolitos secundarios. Los compuestos de alto peso molecular consisten en mucílagos y proteínas2,3. Microorganismos de la rizosfera pueden utilizar algunos de estos compuestos como fuente de energía para el crecimiento y desarrollo. Los exudados de la raíz juegan un papel importante en la conformación de la comunidad de rizobacterias puesto que los compuestos producidos por la planta en los exudados pueden influir en el comportamiento de las bacterias de rizósfera asociados que afectan a la expresión de genes específicos.
Comprensión de la respuesta bacteriana a los exudados de la raíz es un paso clave en el desciframiento de los mecanismos de interacción planta-microorganismo. Como la respuesta bacteriana a interacciones planta-microorganismo es el producto de la expresión génica diferencial, puede ser estudiada mediante el análisis de transcriptoma. Usando este método, estudios previos identificaron varios genes importantes implicados en interacciones planta-microorganismo. En Pseudomonas aeruginosa, genes implicados en metabolismo, quimiotaxis y tipo de secreción de II han demostrado responder a remolacha raíz exudados4. Fan et al. 5 estudia el perfil transcriptómico de B. amyloliquefaciens FZB42 en respuesta a los exudados de raíces de maíz. Sus resultados muestran que, de los genes fuertemente inducidos por los exudados de la raíz, varios grupos están involucrados en rutas metabólicas relacionadas con la utilización de nutrientes, quimiotaxis, motilidad y síntesis no ribosomal de péptidos antimicrobianos y Policétidos.
La exactitud de estos estudios se basa en la colección de exudados de la raíz. Aunque varios métodos han descrito la colección de exudados de la raíz para diversos propósitos, se requieren instrumentos sofisticados o no se hacen en condiciones bien controladas6,7,8. Además, inhibir la rizosfera microorganismos pueden influir en composición del exudado de raíz afectando la permeabilidad de la membrana de la célula vegetal y daño de los tejidos de la raíz, particularmente en el caso de consorcios de microorganismos9. Al investigar la respuesta microbiana a los exudados de la raíz, es importante utilizar condiciones bien definidas para evitar la alteración de los compuestos por otros microorganismos10. Además, RNA de alta calidad se requiere para RNA-seq basados en estudios de transcriptoma. Sin embargo, cuando se trata con cepas bacterianas de modelo no, los protocolos estándar o kits comerciales suelen tienen un bajo rendimiento debido a factores desconocidos o propiedades de crecimiento especial.
El protocolo descrito aquí se verificó utilizando B. mycoides, que es una bacteria gram-positiva, formadora de esporas del filo Firmicute. Es ubicuo en la rizosfera de diferentes especies de plantas. Varias propiedades promotoras de crecimiento de planta han sido reportadas para esta especie, incluida la inducción de resistencia sistemática (ISR) en remolacha azucarera11, inhibición del damping-off patógeno Pythium pepino12, así como nitrógeno fijación en la rizósfera de girasol13. Sin embargo, los mecanismos moleculares de la interacción con una planta de host no están bien estudiados.
El objetivo de los experimentos aquí presentados es estudiar la respuesta de transcriptómico del endosphere aislado B. mycoides a exudados de raíces de papa. En Resumen, el protocolo consta de los siguientes pasos: en primer lugar, recoger exudados de raíces de papa bajo condiciones estériles. Luego, extraer RNA de alta calidad de las células bacterianas tratadas con exudados de la raíz. El último paso es el análisis de datos uso de tubería de T-REx en la web14. Este protocolo se utiliza para identificar los genes B. mycoides que muestran un cambio en niveles de expresión al entrar en contacto con exudados de la raíz y así podrían desempeñar un papel importante en las interacciones planta-microorganismo.
Protocolo
1. germinación de patatas en condiciones estériles
- Enjuague la superficie de la patata con agua estéril. Bañar la patata en etanol al 70% y luego en hipoclorito de sodio 3%, durante 5 minutos. Enjuague nuevamente con agua destilada para eliminar cualquier resto de hipoclorito sódico.
- Preparar los materiales necesarios para la germinación y crecimiento de tubérculos de papa; esterilizar las macetas de plástico, cestas de engraftment, vermiculita y agua en autoclave a 121 ° C por 20 min.
Nota: Asegúrese de que todos los materiales utilizados son esterilizables en autoclave. De lo contrario, utilice otros métodos de esterilización. - Ponga la patata superficie esterilizada en la cesta de engraftment y colóquelo en un recipiente esterilizado que contiene vermiculita húmeda.
- Mantenga el recipiente en una cámara climática a 24 ° C durante tres semanas. Establece en ciclos de 16 h de luz (120 μmol m-2s-1) y 8 h de oscuridad. Para evitar contaminaciones microbianas, use una caja de fibra de vidrio grande para cubrir la olla.
2. recogida de exudados de raíces de papa
- Cuando el brote de los brotes, la transferencia la cesta con la patata entera en un vaso esterilizado con 150 mL de agua desionizada en autoclave, con la patata tubérculos colocados justo encima de la superficie del agua y las raíces sumergieron en el agua (ver figura 1). Mantener la planta en la cámara climática y utilizar la misma configuración que antes [24 ° C; ciclos de 16 horas de luz (120 μmol m-2s-1), 8 h de oscuridad].
- Desde el segundo día, recoger el agua que contiene el exudado y rellenar el vaso con agua desionizada estéril. Realizar el muestreo cada día hasta el séptimo día después de la transferencia de la plántula.
- Almacenar cada muestra por separado a 4 ° C. Para cada muestra, extensión 100 μL en una Luria-Bertani (LB; 1% triptona, extracto de levadura 0.5%, 0.5% NaCl) placa de agar para verificar la contaminación microbiana. Deseche las muestras contaminadas.
- Combine las muestras recolectadas de una planta de semillero y concentrado por liofilización a-40 ° C hasta un volumen final de 150 mL.
3. bacterias
- Una cepa B. mycoides la raya de un stock de glicerol de-80 ° C sobre una placa de agar LB e incubar la placa a 30 ° C durante la noche.
- Inocular un medio líquido LB con una sola Colonia de la placa y hacer crecer la cultura en un incubador de agitación a 200 rpm durante la noche a 30 ° C.
4. tratamiento y muestreo de bacterias
- Para comparar la curva de crecimiento de las bacterias tratadas con exudados de la raíz a un control negativo, preparar una serie de matraces de 50 ml de medio líquido LB. Añadir 0,5 mL del cultivo bacteriano durante la noche a todos los matraces y agregar el 0%, 5%, 10% o 15% (v/v) exudado de raíz o agua desionizada estéril al medio, respectivamente.
- Utilizar una cultura con agua desionizada en lugar de exudados de la raíz como un control. Medir la densidad óptica a 600 nm (OD600) cada 1 h después. Generar una curva de crecimiento mediante la representación de los valores de OD600 versus tiempo.
Nota: Hemos visto que el 10% de los exudados de la raíz no afectó el crecimiento de B. mycoides (ver figura 2); Esta relación fue utilizada para experimentos de RNA-seq. - Diluir una noche cultura B. mycoides con 90 mL de medio LB con el OD inicial600 de ~ 0.05 en un matraz de 300 mL. Añadir 10 mL de exudados de la raíz a la cultura e incubar a 30 ° C por 1 h.
- Utilizar un tratamiento de agua desionizada estéril de 10 mL como control. Recoger las células de la cultura por centrifugación a 9.000 x g durante 2 min a 4 ° C. Eliminar el sobrenadante, inmediatamente congelar la pelotilla en nitrógeno líquido y almacenarlo a-80 ° C hasta su uso.
5. aislamiento de ARN
Nota: Antes de iniciar el aislamiento, preparar la mesa de trabajo, racks y pipetas por limpiarlos con una solución de descontaminación de Rnasa (véase Tabla de materiales). Use guantes en todo momento y asegúrese de que todos los tubos, consejos y soluciones son libres de Rnasa. Siempre mantenga las muestras en hielo siempre que sea posible.
- Añadir 0,1% (v/v) de dietil pirocarbonato (DEPC) a agua ultrapura (100 μl de DEPC por 100 mL de solución), mezclar bien y mantener a temperatura ambiente durante la noche. Autoclave de la mezcla para inactivar la DEPC después del tratamiento durante la noche. Utilizar esta agua ultrapura tratada con DEPC para preparar el tampón TE [10 mM Tris-HCl (pH = 8), 1 mM EDTA] y una solución de SDS (p/v) de 10%.
- Descongelar los pellets de células en el hielo y suspender a cada uno en 400 μL de tampón TE (DEPC). Transferir la suspensión a tubos de tapón de rosca de 2 mL.
- Mezcle 300 μL de cloroformo-isoamyl alcohol (24:1 v/v) y 300 μL de fenol (fenol ácido, grado de RNA) y permite la mezcla en reposo durante 5 minutos. Tomar 500 μl de la fase superior orgánica para agregar a las células resuspendidas. Luego agregar 50 μl de SDS 10% y 0,5 g de perlas de vidrio (0,5 μm) en el tubo.
- Cierre firmemente la tapa y coloque el tubo en un homogeneizador de molino de grano (véase Tabla de materiales). Realizar una homogeneización de pulso s 3 × 45 con un intervalo de 1 min en hielo.
- Centrifugar las muestras durante 10 min a 11.000 x g (4 ° C) y transferir la fase superior a un tubo nuevo.
- Añadir 500 μl de cloroformo-isoamyl alcohol (24:1) a la fase superior del paso 5.5 y Centrifugue la mezcla durante 5 minutos a 11.000 x g (4 ° C).
Nota: Los pasos siguientes se modificaron de las instrucciones del fabricante de un aislamiento de RNA base de filtro fibra de vidrio comercial kit (véase Tabla de materiales). - Transferir 500 μl de la fase superior a un tubo nuevo, añadir 2 volúmenes (1 mL) de un tampón de lisis/encuadernación y mezclar mediante pipeteo arriba y abajo.
- Combinar los tubos de filtro y colección (desde el kit de aislamiento de RNA) y pipetear la mezcla del paso 5.7 para el tubo del filtro. Centrifugar los tubos por 15 s en 8.000 x g y descartar el flujo a través.
- Preparar 1,5 mL en tubos de microcentrífuga con 100 μl de tampón de ADNasa, 10 μl de DNasa I y 5 μl de inhibidor de Rnasa (véase Tabla de materiales). Añadir la mezcla de la solución preparada en el filtro del tubo del filtro e incube durante 20-30 min a 15-25 ° C.
- Realice los pasos de lavado según las instrucciones del fabricante. Utilizar 50 μl de un buffer de elución para eluir el RNA.
- Guardar el ARN eluído a-80 ° C y poner una película de parafina plástico alrededor de él. Al mismo tiempo, transferir unos pocos microlitros de la muestra a un tubo de microcentrífuga de 1,5 mL para llevar a cabo una verificación de calidad.
6. secuenciación y control de calidad ARN
- Compruebe el ARN con un espectrofotómetro de microvolume.
Nota: Coeficientes de absorbancia a 260/280 deben estar por encima de 1,8 y cocientes en 260/230 deben estar por encima de al menos 1.8, preferiblemente por encima de 2.0. - Ejecutar el ARN purificado de un instrumento de electroforesis automatizada usando el análisis de RNA recomienda kit (véase Tabla de materiales); el número de integridad del RNA (RIN) debe ser al menos por encima de 7 para proceder.
- Utilizar un total de 3 μg de ARN por muestra para generar bibliotecas con una preparación de biblioteca de RNA-Seq kit (véase Tabla de materiales) siguiendo las recomendaciones del fabricante.
Nota: Las preparaciones de la biblioteca fueron ordenadas en una plataforma de secuenciación de alto rendimiento y se generaron extremo apareado Lee.
7. datos análisis utilizando el tubería en la Web de T-REx
- Realizar una filtración de calidad utilizando las herramientas de FASTX versión 0.0.13 (Phred puntuaciones de calidad de > 20). Recortar el raw Lee RNA-Seq de las secuencias del adaptador con la herramienta Trimmomatic.
Nota: Todos los análisis posteriores se basaron en datos limpios con alta calidad. - Descargar la base de datos del genoma B. mycoides ATCC 6462 NCBI (adhesión de NCBI n °: CP009692.1) y usarlo como un genoma de referencia para el mapeo de la Lee limpia usando Bowtie2/2.2.3.
- Uso HTSeq v0.6.1 para contar el número de lecturas asignadas para cada gen y la Lee por kilobase de transcripción por millón lecturas asignadas (RPKM) de cada gen depende de la gene y Lee cuenta asignada a este gen.
- Para el análisis de los datos de la tubería del T-REx, utilice la tabla RPKM entrada, así como tres archivos descriptivos: (i) un archivo de factores para describir el experimento en factores, (ii) un archivo de contrastes para definir los factores que se compararán para expresión génica diferencial y (iii) un archivo de clase para describir a grupos de genes de interés.
Nota: Los archivos descriptivos están en formato in.txt. Pueden encontrarse ejemplos en la Página Web de T-REx (http://genome2d.molgenrug.nl/). Una instrucción detallada para el análisis de datos con T-REx se puede encontrar en una publicación anterior por de Jong et al. 14.
Resultados
Microorganismos asociados a plantas pueden influir positivamente en crecimiento de las plantas y la salud. Sin embargo, los mecanismos de las interacciones complejas entre las plantas y sus simbiontes microbianos no se entienden completamente. Exudados de la raíz juegan un papel importante en la regulación de la actividad de rizobacterias y comportamiento, y generalmente se postula que la colonización microbiana de raíces inicia con la atracción de los microbios a los exudados de la raíz. El objetivo de este trabajo fue investigar la respuesta transcriptómicos de rizobacterias B. mycoides a exudados de raíces de papa. Para cumplir esto, tubérculos de Papa fueron superficie esterilizada y germinaron en vermiculita esterilizada. Luego se recolectaron los exudados de la raíz como se muestra en la figura 1. Con el fin de descartar la posibilidad de los exudados de la raíz que afectan el crecimiento bacteriano, hasta 15% de los exudados de la raíz fueron agregado a la cultura B. mycoides , y ningún cambio en el crecimiento fue detectada durante el tiempo de medición (figura 2). Así, los cambios de expresión génica observados en este estudio no fueron causados probablemente por los efectos relacionados con el crecimiento.
Después de la recolección, los exudados de la raíz fueron agregados a la cultura B. mycoides a una proporción de 10% (v/v) y el ARN total bacteriano fue aislado como se describió anteriormente. El RNA entonces fue sometido a una verificación de calidad por un instrumento de electroforesis automatizada de la cual los resultados se muestran en la figura 3. Figura 3A y 3B representan el ARN aislado de B. mycoides tratados con los exudados de la raíz, y figura 3 y 3D representan el ARN aislado del grupo de control. Todas las muestras anotaron un valor RIN por encima de 9 con dos bandas claras corresponden a las subunidades de RNA 16S y 23S, demostrando que se obtuvo RNA de alta calidad por este protocolo.
Después de la preparación de la biblioteca, Lee par final se obtuvieron con una plataforma de secuenciación de alto rendimiento. El crudo Lee RNA-Seq fueron recortado de las secuencias de adaptador y asignado contra la secuencia del genoma de referencia. De los datos obtenidos, se generó la tabla RPKM. El análisis del transcriptoma se realizó con la tubería T-REx. La trama de intensidad la relación de todos los genes expresados diferencialmente se muestra en la figura 4. En comparación con un control, la adición de exudados de la raíz de papa inducido 715 genes se expresen diferencialmente. De los, 408 genes fueron alza y 307 genes fueron dando15. El cambio relativo de algunos de los genes con expresión alterada se enumera en la tabla 1.
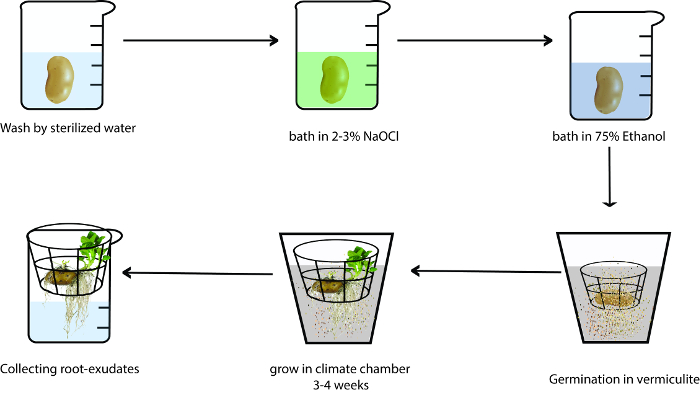
Figura 1: esquema del proceso de la colección de exudados de raíces de patata. Los materiales utilizados son autoclave y la germinación se realiza en una cámara climática. Lava el tubérculo de la patata con agua esterilizada y baño en 2-3% NaOCl 5 min baño en etanol al 75% por 5 min otro lugar la patata en una canasta de autoclave y ponerlo en un recipiente con vermiculita húmeda. Cultivar la patata en una cámara climática para 3-4 semanas y luego transferir la cesta con la planta de semillero de papa en un vaso. Recoger los exudados de la raíz de todos los días y llenar el vaso con agua esterilizada. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: La curva de crecimiento de B. mycoides con diferentes concentraciones de exudados de la raíz de papa. La cepa EC18 fue cultivada en un medio LB líquido con la adición de exudados de raíces de patata o estéril de H2O. OD600 se midió cada 1 h y se representan gráficamente frente al tiempo. Todos los grupos no muestran un patrón de crecimiento similar, indicando que hasta un 15% de la adición de exudados de la raíz tiene ningún efecto significativo sobre el crecimiento de B. mycoides . Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: control de calidad de RNA por el instrumento de electroforesis automatizada. A y B representan el ARN aislado de B. mycoides tratados con exudados de la raíz y C y D representan el ARN aislado del grupo de control. Todas las muestras de RNA muestran dos bandas claras corresponden a 23S y 16S rRNA y una banda de rRNA de 5S débil. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Diagrama de intensidad de relación para la visualización de la expresión génica diferencial de las muestras de RNA-seq de B. mycoides en respuesta a los exudados de la raíz de papa. La figura se genera automáticamente por el T-REx. El eje x representa el nivel de expresión del gene, y el eje y representa el cambio de doble log2 transformado. Los genes ser para arriba - y o tienen valores de relación positivos y negativos log2. Los puntos en el área rayado indican genes que no son significativamente sobre - o bajo-expresó. Haga clic aquí para ver una versión más grande de esta figura.
| Etiqueta de gene | Strand | Doble cambio | Anotación |
| BG05_RS09165 | + | 3.3 | proteína de multidrug efflux |
| BG05_RS20930 | + | 25.3 | proteína de la membrana |
| BG05_RS10935 | + | 23.3 | etapa proteína de esporulación III AD |
| BG05_RS08990 | - | 11.8 | proteína de esporulación |
| BG05_RS16405 | + | 3.9 | Regulador transcripcional familia IclR |
| BG05_RS24905 | - | 3 | alfa de la subunidad de triptófano sintasa |
| BG05_RS24920 | - | 2.3 | indol-3-glicerol fosfato sintasa |
| BG05_RS22255 | - | 27 | Acetohidroxiácido sintasa |
| BG05_RS22250 | - | 6.5 | ketol-acid reductoisomerase |
| BG05_RS22265 | - | 26.4 | aminoácidos de cadena ramificada aminotransferasa |
| BG05_RS18715 | - | 2.8 | pululanasa |
| BG05_RS18040 | + | -9.1 | proteína de germinación Jie |
| BG05_RS16930 | + | -4.2 | proteína de unión a ATP transportador ABC de azúcar |
| BG05_RS27345 | + | -2.9 | Transportador MFS |
| BG05_RS19555 | - | -3.3 | Subunidad de transportador de celobiosa PTS IIB |
| BG05_RS24345 | - | -2,7 | importador de putrescina |
| BG05_RS22525 | - | -12.4 | cardiolipina sintasa |
| BG05_RS15225 | - | -3.3 | proteína de la membrana |
| BG05_RS18475 | + | -5.7 | proteína de la membrana |
| BG05_RS19095 | + | -2.1 | proteína de germinación |
Tabla 1: Lista de genes diferencialmente expresados de un grupo de tratados con exudados de la raíz en comparación con un control.
Discusión
Han planteado la hipótesis de interacciones planta-microorganismo para ser determinado por un equilibrio finamente sintonizado entre bacterias y plantas. Estas interacciones son muy complejas y difíciles de estudiar en un sistema natural, que comprende diversas especies microbianas, potencialmente actuar como consorcios. Este papel describe un protocolo simplificado para estudiar la respuesta bacteriana a los exudados de la raíz en condiciones bien controladas. El perfil del transcriptoma de rizobacterias, sobre exposición a los exudados de la raíz, proporciona información detallada sobre adaptación bacteriana a la hornacina de la rizosfera. Este protocolo de extracción de exudados de raíz no requiere procedimientos complicados y equipo especializado. Sin embargo, todos los procedimientos deben llevarse a cabo bajo condiciones estrictamente estériles y un control de esterilidad debe incluirse. Recomendamos el crecimiento de varios tubérculos de papa en paralelo y descartar los más contaminados. Modificaciones pueden hacerse en el presente Protocolo si se están estudiando otras especies vegetales. Es importante utilizar un método adecuado para esterilizar cualquier semillas/tubérculos ya que pueden variar en la tolerancia a los desinfectantes aplicados.
Una vez obtenidos los exudados de la raíz, RNA de alta calidad debe ser aislado de las células bacterianas. Diferentes reactivos y protocolos que se basan principalmente en el método de TRIzol o fenol/cloroformo son desperdiciadoras de tiempo16. Los kits comerciales están principalmente diseñados para organismos modelo pero son menos aplicables a los demás. Este protocolo de aislamiento de RNA que combina el método de fenol/cloroformo y un kit de aislamiento de RNA supera las desventajas de estos métodos. Por otra parte, un paso de talón-golpes es para homogeneizar las células B. mycoides que normalmente se agregan en la cultura planctónica. Posible contaminación de ADN se elimina mediante una incubación adicional con DNasa antes de la elución. El alto número RIN implica el aislamiento de ARN intacto. Así, este protocolo es especialmente eficiente y ahorro de tiempo para las cepas bacterianas ambientales.
Después de la secuencia de RNA, análisis de datos se realizaron con T-REx, una tubería de análisis estadístico basado en la web de RNA-seq gene expresión datos14. Esta tubería es fácil de usar, especialmente para los biólogos sin conocimiento extenso de la bioinformática. El archivo de entrada es que el RNA expresión nivel datos brutos, como RPKM, fragmentos por kilobase por millón lecturas asignadas (FPKM), cuenta por millones lecturas asignadas (CPM), u otras unidades de expresión de gene. Tales archivos de valor de expresión génica pueden ser generadas por herramientas disponibles incluyendo SAMtools17, BEDtools18y19de NGS-Trex. Para ejecutar el análisis de RNA-seq, son necesarios otros tres archivos de entrada. Estos archivos se utilizan para definir los factores que describen los experimentos y las repeticiones, las comparaciones entre las diferentes condiciones experimentales y los grupos de genes de interés. Cuando se cargan los archivos de entrada, el proceso de análisis llevará sólo un par de minutos.
Genes diferencialmente expresados varios de B. mycoides EC18, con exudados de la raíz, se enumeran en la tabla 1. Entre ellos, se altera la expresión de varios genes que codifican proteínas de membrana. Genes relacionados con la esporulación o proceso de germinación se expresan diferencialmente. También cambia la expresión de genes implicados en la esporulación en B. subtilis cuando cultivadas conjuntamente con las plántulas de arroz, porque los exudados de la raíz suministran la energía necesaria para el crecimiento dinámico de bacterias células18. La expresión de la IclR regulador transcripcional, que está relacionada con la resistencia del multidrug y la degradación de compuestos aromáticos en bacterias del suelo, es upregulated. La cepa de la canceladura de IclR de rizobacterias Klebsiella pneumoniae ha disminuido la solubilización de fosfato mineral, en comparación con la cepa de tipo salvaje19. Varios genes relacionados con la síntesis y metabolismo de los aminoácidos son estimulados, mientras que los genes implicados en el transporte de azúcar como un transportador de celobiosa PTS son reguladas. Esto sugiere eso tensión EC18 tenga una preferencia metabólica de aminoácidos en azúcares en la rizosfera. La función de los genes alteradas puede ser más estudiado en situ haciendo mutantes knockout o sobreexpresión. En Resumen, el protocolo aquí descrito permite una rápida identificación de un gran número de genes bacterianos potencialmente importantes implicados en interacciones planta-microorganismo.
Divulgaciones
Los autores declaran que no tienen intereses financieros que compiten.
Agradecimientos
Agradecemos a Jakob Viel por sus valiosos comentarios y sugerencias. También agradecemos a Anne de Jong por su ayuda en el análisis de la bioinformática. Yanglei Yi y Zhibo Li son apoyados por el Consejo de becas de China (CSC). Agradecemos a NWO-TTW Perspectief Programma Back2Roots (TKI-AF-15510) por su apoyo financiero a OPK.
Materiales
| Name | Company | Catalog Number | Comments |
| sodium hypochlorite | Sigma | CAS: 7681-52-9 | 10-15% active chlorine |
| Luria-Bertani (LB) broth | |||
| incubater | New Brunswick Scientific | Innova 4000 | |
| spectrophotometer | Thermo Fisher Scientific | Genesys 20 | |
| liquid nitrogen | |||
| glass beads | Sigma | G8893 | 0.5 µm |
| 2.0 ml tube with screw cap | RNase free | ||
| 1.5 ml and 2.0 ml eppendorf tube | RNase free | ||
| Bead mill homogenizer | BioSpec | 607 | Mini_beadbeater |
| centrifuge | Eppendorf | 5430 | |
| Diethyl pyrocarbonate (DEPC) | sigma | CAS: 1609-47-8 | |
| Sodium Dodecyl Sulfate (SDS) | sigma | CAS: 151-21-3 | 10% solution prepared with DEPC treated MQ water |
| TE buffer | 10 mM Tris-HCl; 1 mM EDTA, pH=8 | ||
| phenol | Sigma | RNA grade | |
| chloroform-isoamyl alcohol | prepare 24:1 of chloroform:isoamyl alcohol, store at room temperature | ||
| High pure RNA isolation kit | Roche | 11828665001 | |
| RNase Decontamination Solution | Invitrogen | AM9780 | RNase-Zap |
| Automated electrophoresis instrument | Agilent | 2100 | Bioanalyzer |
| Microvolume spectrophotometer | Thermo Fisher Scientific | Nanodrop ND-1000 | |
| RNA quality analysis kit | Agilent | RNA 6000 Nano kit | |
| RNase inhibitor | Thermo Fisher Scientific | RiboLock | |
| Directional RNA library Prep kit | NEB | Ultra | For Illumina |
Referencias
- Haichar, F. e. Z., et al. Plant host habitat and root exudates shape soil bacterial community structure. The ISME Journal: Multidisciplinary Journal of Microbial Ecology. 2 (12), 1221-1230 (2008).
- Badri, D. V., Vivanco, J. M. Regulation and function of root exudates. Plant, Cell & Environment. 32 (6), 666-681 (2009).
- Rohrbacher, F., St-Arnaud, M. Root exudation: the ecological driver of hydrocarbon rhizoremediation. Agronomy Journal. 6 (1), 19 (2016).
- Mark, G. L., et al. Transcriptome profiling of bacterial responses to root exudates identifies genes involved in microbe-plant interactions. Proceedings of the National Academy of Sciences of the United States of America. 102 (48), 17454-17459 (2005).
- Fan, B., et al. Transcriptomic profiling of Bacillus amyloliquefaciens FZB42 in response to maize root exudates. BMC Microbiology. 12 (1), 116 (2012).
- Yoshitomi, K. J., Shann, J. R. Corn (Zea mays L.) root exudates and their impact on 14C-pyrene mineralization. Soil Biology and Biochemistry. 33 (12), 1769-1776 (2001).
- Lambert, M. R. Clover root exudate produces male-biased sex ratios and accelerates male metamorphic timing in wood frogs. Royal Society Open Science. 2 (12), (2015).
- Tuason, M. M. S., Arocena, J. M. Root organic acid exudates and properties of rhizosphere soils of white spruce (Picea glauca) and subalpine fir (Abies lasiocarpa). Canadian Journal of Soil Science. 89 (3), 287-300 (2009).
- Grayston, S. J., Vaughan, D., Jones, D. Rhizosphere carbon flow in trees, in comparison with annual plants: the importance of root exudation and its impact on microbial activity and nutrient availability. Applied Soil Ecology. 5 (1), 29-56 (1997).
- Rovira, A. D. Plant root exudates. Botanical Review. 35 (1), 35-57 (1969).
- Bargabus, R. L., Zidack, N. K., Sherwood, J. E., Jacobsen, B. J. Characterisation of systemic resistance in sugar beet elicited by a non-pathogenic, phyllosphere-colonizing Bacillus mycoides, biological control agent. Physiological and Molecular Plant Pathology. 61 (5), 289-298 (2002).
- Peng, Y. -. H., et al. Inhibition of cucumber Pythium damping-off pathogen with zoosporicidal biosurfactants produced by Bacillus mycoides. Journal of Plant Diseases and Protection. 124 (5), 481-491 (2017).
- Ambrosini, A., et al. Diazotrophic bacilli isolated from the sunflower rhizosphere and the potential of Bacillus mycoides B38V as biofertiliser. Annals of Applied Biology. 168 (1), 93-110 (2016).
- de Jong, A., van der Meulen, S., Kuipers, O. P., Kok, J. T-REx: transcriptome analysis webserver for RNA-seq expression data. BMC Genomics. 16 (1), 663 (2015).
- Yi, Y., de Jong, A., Frenzel, E., Kuipers, O. P. Comparative transcriptomics of Bacillus mycoides strains in response to potato-root exudates reveals different genetic adaptation of endophytic and soil isolates. Frontiers in Microbiology. 8, 1487 (2017).
- Nwokeoji, A. O., Kilby, P. M., Portwood, D. E., Dickman, M. J. RNASwift: a rapid, versatile RNA extraction method free from phenol and chloroform. Analytical Biochemistry. 512, 36-46 (2016).
- Ramirez-Gonzalez, R. H., Bonnal, R., Caccamo, M., MacLean, D. Bio-samtools: Ruby bindings for SAMtools, a library for accessing BAM files containing high-throughput sequence alignments. Source Code for Biology and Medicine. 7 (1), 6 (2012).
- Quinlan, A. R. BEDTools: the Swiss-army tool for genome feature analysis. Current Protocols in Bioinformatics. , (2014).
- Boria, I., Boatti, L., Pesole, G., Mignone, F. NGS-trex: next generation sequencing transcriptome profile explorer. BMC Bioinformatics. 14 (7), S10 (2013).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados