Method Article
Pflanze-Mikroben-Interaktion: Transcriptional Antwort des Bacillus Mycoides auf Kartoffel Wurzelausscheidungen
In diesem Artikel
Zusammenfassung
Das Ziel des hier vorgestellten Protokolls soll die transkriptomischen Reaktion der Endosphere isoliert Bacillus Mycoides auf Kartoffel Wurzelausscheidungen zu studieren. Diese Methode erleichtert die Identifizierung von wichtigen bakteriellen Gene Pflanze-Mikroben-Interaktionen beteiligt und gilt grundsätzlich für andere Endophyten und Pflanzen, mit geringfügigen Anpassungen.
Zusammenfassung
Pflanze-assoziierte Bakterien spielen eine wichtige Rolle bei der Förderung von Wachstum und Verhütung von Krankheiten bei Pflanzen. Die Anwendung der Pflanze wachstumsfördernde standarddüngung (PGPR) als biodüngemittel oder Biocontrol Mittel geworden eine effektive Alternative zu der Verwendung von herkömmlichen Düngemitteln und Ernte-Produktivität bei geringen Kosten zu erhöhen. Pflanze-Mikroben-Interaktionen abhängig Host Pflanze abgesondert Signale und eine Reaktion hereon durch ihre assoziierten Bakterien. Die molekularen Mechanismen der wie nützlichen Bakterien reagieren jedoch auf die damit verbundenen pflanzliche, dass Signale nicht vollständig verstanden sind. Beurteilung der transkriptomischen auf der Bakterien Wurzelausscheidungen ist ein leistungsfähiger Ansatz um die bakterielle Genexpression und Verordnung unter rhizospheric Bedingungen zu bestimmen. Solches Wissen ist notwendig, die zugrunde liegenden Mechanismen Pflanze-Mikroben-Interaktionen zu verstehen. Dieses Whitepaper beschreibt ein detailliertes Protokoll um die transkriptomischen Antwort von B. Mycoides EC18, ein Stamm von der Kartoffel Endosphere, Kartoffel Wurzelausscheidungen isoliert zu studieren. Mit Hilfe der jüngsten Hochdurchsatz-Sequenzierung-Technologie kann dieses Protokoll in mehreren Wochen und produzieren massive Datasets durchgeführt werden. Zunächst sammeln wir die Wurzelausscheidungen unter sterilen Bedingungen, nach denen sie B. Mycoides Kulturen hinzugefügt werden. Die RNA aus diesen Kulturen ist isoliert mit einem Phenol/Chloroform-Methode kombiniert mit einem kommerziellen Kit und durch automatisierte Elektrophorese Instrument einer Qualitätskontrolle unterzogen. Nach der Sequenzierung, Datenanalyse erfolgt mit der Web-basierten T-REx-Pipeline und eine Gruppe von differentiell exprimierten Genen identifiziert. Diese Methode ist ein nützliches Werkzeug, um neue Entdeckungen auf die bakterielle Gene in Pflanzen-Mikroben-Interaktionen zu ermöglichen.
Einleitung
Pflanzen können Exsudat bis zu 20 % des Kohlenstoffs in der Rhizosphäre1, d. h., die schmale Zone des Bodens in der Nähe der Wurzeln während der Photosynthese durch Wurzeln fixiert. Durch die höhere nährstoffverfügbarkeit ist die Rhizosphäre einen geeigneten Lebensraum für verschiedene Mikroorganismen, einschließlich der Förderung des Pflanzenwachstums Bakterien. Die Wurzelausscheidungen enthalten eine Reihe von anorganischen Verbindungen wie Ionen, anorganische Säuren, Sauerstoff und Wasser. Jedoch wird der Großteil der Wurzelausscheidungen von organischen Materialien, gebildet, in niedermolekulare Verbindungen und hochmolekularen Verbindungen unterteilt werden kann. Die niedermolekularen Verbindungen enthalten Aminosäuren, organische Säuren, Zucker, Phenolverbindungen, Fettsäuren und eine Reihe von sekundären Pflanzenstoffen. Die hochmolekularen Verbindungen bestehen aus Schleim und Proteine,2,3. Rhizosphäre Mikroorganismen können einige dieser Verbindungen als Energiequelle für Wachstum und Entwicklung. Die Wurzelausscheidungen spielen eine wichtige Rolle bei der Gestaltung der Wurzelbakterien Gemeinschaft, da die Pflanze produziert Verbindungen in die Exsudate durch Einwirkung auf die Expression bestimmter Gene das Verhalten der Rhizosphäre-assoziierte Bakterien beeinflussen können.
Verständnis der bakteriellen Antwort auf Wurzelausscheidungen ist ein wichtiger Schritt bei der Entschlüsselung wechselwirkungsmechanismen Pflanze-Mikroben. Da die bakterielle Reaktion auf Pflanze-Mikroben-Interaktionen das Produkt der differentiellen Genexpression, kann es durch die Transkriptom-Analyse untersucht werden. Mit dieser Methode, identifiziert frühere Studien mehrere wichtige Gene in Pflanzen-Mikroben-Interaktionen. In Pseudomonas Aeruginosawurden Gene, die im Stoffwechsel, Chemotaxis und Typ II-Sekretion zur Beantwortung von Zuckerrüben Wurzel Exsudate4gezeigt. Ventilator Et Al. 5 studierte die transkriptomischen Profilierung der B. Amyloliquefaciens FZB42 als Reaktion auf Mais Wurzelausscheidungen. Ihre Ergebnisse zeigen, dass die Gene, die stark durch die Wurzelausscheidungen induziert, Stoffwechselwege in Bezug auf Nährstoff Auslastung, Chemotaxis, Motilität und nicht-ribosomale Synthese von antimikrobiellen Peptiden und Polyketides mehrere Gruppen beteiligt sind.
Die Genauigkeit dieser Studien stützt sich auf die Sammlung von Wurzelausscheidungen. Obwohl mehrere Methoden der Sammlung von Wurzelausscheidungen für verschiedene Zwecke beschrieben haben, entweder verlangen ausgeklügelte Instrumente oder sind nicht in kontrollierten Bedingungen6,7,8durchgeführt. Darüber hinaus können Hemmung der Rhizosphäre Mikroorganismen Wurzel Exsudat Zusammensetzung beeinflussen, indem Pflanze Zellmembran Durchlässigkeit beeinträchtigen und schädigen die Wurzel Gewebe, insbesondere im Falle von Konsortien von Mikroorganismen9. Bei der Untersuchung der mikrobiellen auf Wurzelausscheidungen ist es wichtig, genau definierte Bedingungen zu verwenden, um Veränderung der Verbindungen durch andere Mikroorganismen10zu vermeiden. Darüber hinaus ist qualitativ hochwertige RNA erforderlich für RNA-Seq Transkriptom-Studien. Jedoch haben beim Umgang mit nicht-Modell-bakterielle Belastungen, die standard-Protokolle oder kommerziellen Kits in der Regel eine geringe Effizienz durch unbekannte Faktoren oder spezielle Wachstumseigenschaften.
Das hier beschriebene Protokoll wurde mit B. Mycoides, das ist ein grampositives, Spore bildet Bakterium Firmicute Phylum überprüft. Es ist allgegenwärtig in der Rhizosphäre der verschiedenen Pflanzenarten. Für diese Spezies, einschließlich Induktion des systematischen Widerstandes (ISR) in Zuckerrüben11, Hemmung der Dämpfung-off-Erregers Pythium Gurke12sowie Stickstoff wurden mehrere Pflanze Förderung Wachstumseigenschaften gemeldet. Fixierung in der Sonnenblume Rhizosphäre13. Allerdings sind die molekularen Mechanismen der Interaktion mit einer Wirtspflanze nicht gut untersucht.
Das Ziel der hier vorgestellten Experimente ist es, die transkriptomischen Reaktion der Endosphere isoliert B. Mycoides auf Kartoffel Wurzelausscheidungen zu studieren. Kurzum, das Protokoll besteht aus den folgenden Schritten: zunächst sammeln Kartoffeln Wurzelausscheidungen unter sterilen Bedingungen. Ziehen Sie dann, qualitativ hochwertige RNA aus Bakterienzellen mit Wurzelausscheidungen behandelt. Der letzte Schritt ist die Analyse der Daten mithilfe der webbasierten T-REx Pipeline14. Dieses Protokoll wurde verwendet, um B. Mycoides Gene zu identifizieren, die zeigen eine Verschiebung im Ausdruck Niveaus beim Kontakt mit Wurzelausscheidungen und damit möglicherweise eine wichtige Rolle in Pflanze-Mikroben-Interaktionen.
Protokoll
(1) keimenden Kartoffel unter sterilen Bedingungen
- Spülen Sie die Kartoffel-Oberfläche mit sterilem Wasser. Baden Sie die Kartoffel in 70 % igem Ethanol und dann in 3 % Natriumhypochlorit für 5 min. Spülen Sie ihn wieder mit sterilem Wasser, alle übrigen Natriumhypochlorit zu entfernen.
- Bereiten Sie die Materialien für das Keimen und wachsen Kartoffelknollen benötigt; die Kunststoff-Töpfe, Engraftment Körbe, Vermiculit, sterilisieren und wässern durch Autoklavieren bei 121 ° C für 20 Minuten.
Hinweis: Stellen Sie sicher, dass alle verwendeten Materialien autoklavierbar sind. Andernfalls verwenden Sie andere Sterilisation Methoden. - Setzen Sie die Oberfläche sterilisiert Kartoffel in den Engraftment Korb, und platzieren Sie diese in einem Autoklaven Pot mit nassen Vermiculit.
- Halten Sie den Topf in einer Klimakammer bei 24 ° C für drei Wochen. Setzen Sie ihn auf Zyklen von 16 h Licht (120 µmol m-2s-1) und 8 Stunden der Dunkelheit. Um mikrobielle Kontaminationen zu vermeiden, verwenden Sie ein großes Glasfaser Feld um den Pot zu decken.
2. Sammeln von Kartoffel Wurzelausscheidungen
- Wenn die Triebe sprießen, Transfer der Korb mit die ganze Kartoffel in einen sterilen Becher mit 150 mL autoklaviert deionisiertes Wasser gefüllt, mit der Kartoffel Knollen platziert oberhalb der Wasseroberfläche und die Wurzeln in das Wasser getaucht (siehe Abbildung 1). Halten Sie den Keimling in der Klimakammer, und verwenden Sie dieselben Einstellungen wie zuvor [24 ° C; Zyklen von 16 h Licht (120 µmol m-2s-1), 8 h der Finsternis].
- Ab dem zweiten Tag auf sammeln Sie das Wasser mit den Ausscheidungen und Nachfüllen Sie den Becher mit sterilem deionisiertes Wasser. Führen Sie die Probenahme jeden Tag bis zum siebten Tag nach der Übertragung des Keimlings.
- Jede Probe separat bei 4 ° c Lagern Für jede Probe verteilt 100 µL auf ein Luria-Bertani (LB; 1 % Tryptone, 0,5 % Hefeextrakt, 0,5 % NaCl) nährbodenplatte für mikrobielle Kontaminationen zu überprüfen. Entsorgen Sie die kontaminierten Proben.
- Kombinieren Sie die gesammelten Proben von einem Sämling, und durch Gefriertrocknung sie bei-40 ° C zu einem Endvolumen von 150 mL zu konzentrieren.
(3) wachsenden Bakterien
- Streifen Sie eine B. Mycoides Belastung aus einem-80 ° C Glycerin bestand auf eine LB-Agar-Platte, und inkubieren Sie die Platte bei 30 ° C über Nacht.
- Impfen Sie eine LB-Flüssigmedium mit eine einzige Kolonie von der Platte zu, und wachsen Sie die Kultur in einem schütteln Inkubator bei 200 u/min über Nacht bei 30 ° C.
4. Behandlung und Beprobung von Bakterien
- Um die Wachstumskurve der Bakterien mit Wurzelausscheidungen behandelt, um eine Negativkontrolle zu vergleichen, bereiten Sie eine Reihe von Flaschen mit 50 mL LB-Flüssigmedium. Alle Flaschen 0,5 mL die Übernachtung Bakterienkultur hinzu, und fügen Sie 0 %, 5 %, 10 % oder 15 % (V/V) Wurzel Exsudat oder sterilen deionisiertes Wasser bzw. auf das Medium.
- Verwenden Sie eine Kultur mit entionisiertem Wasser anstelle von Wurzelausscheidungen als Kontrolle. Messung die optische Dichte bei 600 nm (OD600) jeweils 1 h danach. Generieren einer Wachstumskurs von Plotten die OD600 Werte im Vergleich zur Zeit.
Hinweis: Wir haben gesehen, dass 10 % der die Wurzelausscheidungen keinen Einfluss auf das Wachstum von B. Mycoides (siehe Abbildung 2). Dieses Verhältnis wurde für RNA-Seq Experimente verwendet. - Verdünnen Sie eine Übernachtung B. Mycoides -Kultur mit 90 mL vorgewärmten LB-Medium auf die anfängliche OD600 von ~ 0,05 in eine 300 mL Flasche. Die Kultur 10 mL Wurzelausscheidungen hinzu und bei 30 ° C für 1 h inkubieren.
- Verwenden Sie eine 10 mL sterilem deionisiertes Wasserbehandlung als Kontrolle. Sammeln Sie Zellen aus der Kultur durch Zentrifugation bei 9.000 x g für 2 min bei 4 ° C. Den überstand verwerfen und sofort das Pellet in flüssigem Stickstoff eingefroren, dann bis zur Verwendung bei-80 ° C lagern.
(5) RNA-Isolation
Hinweis: Bevor Sie beginnen die Isolation, bereiten Sie die Werkbank, Racks und Pipetten mit einer RNase Dekontaminationslösung reinigt (siehe Tabelle der Materialien). Tragen Sie Handschuhe bei aller Zeiten, und sicherstellen, dass alle Rohre, Tipps und Lösungen sind RNase-freie. Halten Sie die Proben auf Eis wenn möglich.
- Reinstwasser (100 µL DEPC pro 100 mL Lösung) 0,1 % (V/V) von Diethyl-Pyrocarbonate (DEPC) hinzufügen, gut vermischen und über Nacht bei Zimmertemperatur aufbewahren. Autoklaven die Mischung, die DEPC nach Behandlung über Nacht zu inaktivieren. Diese DEPC-behandeltem Reinstwasser zur Vorbereitung der TE-Puffer verwenden [10 mM Tris-HCl (pH = 8), 1 mM EDTA] und einer 10 % igen SDS (w/V) Lösung.
- Die Zelle-Pellets auf dem Eis Auftauen und jeweils in 400 µL TE (DEPC) Puffer auszusetzen. Übertragen Sie die Suspensionen in 2 mL Schraubverschluss Rohre.
- Premix 300 µL Chloroform Isoamyl Alkohol (24:1 V/V) und 300 µL Phenol (saure Phenol, RNA-Klasse), und die Mischung für 5 min stehen lassen. Nehmen Sie 500 µL der oberen organischen Phase die resuspendierte Zellen hinzu. Dann fügen Sie 50 µL 10 % SDS und 0,5 g Glasperlen (0,5 µm) in die Röhre.
- Schließen Sie den Deckel fest und legen Sie das Rohr in eine Rührwerksmühle Homogenisator (siehe Tabelle der Materialien). Führen Sie eine 3 × 45 s Puls Homogenisierung mit einem Intervall von 1 min auf Eis.
- Zentrifugieren Sie Proben für 10 min bei 11.000 x g (4 ° C) und übertragen Sie die obere Phase auf einen neuen Schlauch.
- Die obere Phase ab Schritt 5.5 500 µL Chloroform Isoamyl Alkohol (24:1) hinzu und Zentrifugieren Sie die Mischung für 5 min bei 11.000 x g (4 ° C).
Hinweis: Die folgenden Schritte wurden geändert von Anweisungen des Herstellers eine kommerzielle Glasfaser filterbasierte RNA Isolation kit (siehe Tabelle der Materialien). - Übertragen Sie 500 µL der oberen Phase auf einem frischen Rohr, fügen Sie 2 Bände (1 mL) eines Puffers Lyse/Bindung hinzu und mischen Sie es durch pipettieren rauf und runter.
- Kombinieren Sie die Filter und Sammlung Röhrchen (aus dem RNA-Isolierung-Kit) und pipette die Mischung aus Schritt 5.7 mit dem Filter-Rohr. Zentrifugieren Sie die Rohre für 15 s bei 8.000 x g und entsorgen der durchströmten.
- Bereiten Sie 1,5 mL Mikrozentrifugenröhrchen mit 100 µL DNase Puffer, 10 µL der DNase I und 5 µL RNase-Inhibitor (siehe Tabelle der Materialien). Fügen Sie die fertige Lösung mischen auf dem Filter der Filter-Röhre, und 20-30 Minuten bei 15-25 ° c inkubieren
- Der Wasch-Schritte nach Anweisungen des Herstellers. Verwenden Sie 50 µL eines Puffers Elution, um die RNA zu eluieren.
- Speichern der eluierten RNS bei-80 ° C und einen Kunststoff Paraffin Film um ihn herum. Zum gleichen Zeitpunkt übertragen Sie wenige Mikroliter der Probe zu einem 1,5 mL Microcentrifuge Schlauch einen Qualitäts-Check durchführen.
(6) RNA-Qualitäts-Check und Sequenzierung
- Überprüfen Sie die RNA mit einem Microvolume Spektrophotometer.
Hinweis: Extinktion Verhältnisse bei 260/280 sollte über 1,8 und Kennzahlen auf 260/230 sollte über mindestens 1,8, vorzugsweise über 2.0. - Lauf die gereinigte RNA in automatisierte Elektrophorese Instrument mit der empfehlen-RNA-Analyse kit (siehe Tabelle der Materialien); die RNA-Integrität-Anzahl (RIN) muss mindestens über 7 um fortzufahren.
- Verwenden Sie einen Gesamtbetrag von 3 µg RNA pro Probe, um Bibliotheken mit einer RNA-Seq-Bibliothek-Vorbereitung zu generieren kit (siehe Tabelle der Materialien) nach den Empfehlungen des Herstellers.
Hinweis: Die Bibliothek Vorbereitungen wurden auf einer Plattform von Hochdurchsatz-Sequenzierung sequenziert und gepaart Ende liest generiert wurden.
(7) Datenanalyse mit Hilfe den Web-basierten Pipeline T-REx
- Führen Sie eine Qualität Filterung mit dem FASTX-Toolkit Version 0.0.13 (Phred Qualitätsfaktoren von > 20). Schneiden Sie die rohen RNA-Seq liest aus dem Adapter-Sequenzen mit dem Trimmomatic-Werkzeug.
Hinweis: Die nachfolgenden Analysen beruhten auf saubere Daten mit hoher Qualität. - Laden Sie die B. Mycoides ATCC 6462 NCBI-Genom-Datenbank (NCBI Beitritt Nr.: CP009692.1) und verwenden Sie es als eine Referenz-Genom für die Zuordnung der sauberen liest mit Bowtie2/2.2.3.
- Verwendung HTSeq v0.6.1 liest Zahlen zählen, jedes Gen zugeordnet und die Lesevorgänge pro Kilobase Abschrift pro million zugeordneten liest (RPKM) jedes Gens basierend auf der Länge der Anzahl der Gene und liest dieses Gen zugeordnet.
- Für die Analyse der Daten von der T-REx-Pipeline, verwenden Sie die RPKM-Tabelle als Eingabe sowie drei beschreibenden Dateien: (i) eine Datei Faktoren zu beschreiben, das Experiment in Faktoren, (Ii) eine Kontraste-Datei zu definieren, welche Faktoren für differenzielle Genexpression verglichen werden , und (Iii) eine Klassendatei um Gruppen von Genen von Interesse zu beschreiben.
Hinweis: Die beschreibenden Dateien sind in.txt Format. Beispiele finden Sie auf der Webseite von T-REx (http://genome2d.molgenrug.nl/). Eine detaillierte Anleitung für die Datenanalyse mit T-REx finden Sie in einer früheren Publikation von de Jong Et al. 14.
Ergebnisse
Pflanze-assoziierten Mikroorganismen können Wachstum und die Gesundheit positiv beeinflussen. Allerdings sind die Mechanismen die komplexen Wechselwirkungen zwischen Pflanzen und ihre mikrobiellen Symbionten nicht vollständig geklärt. Wurzelausscheidungen spielen eine wichtige Rolle bei der Regulierung der Wurzelbakterien Aktivität und Verhalten, und es wird in der Regel postuliert, dass die mikrobielle Besiedlung der Wurzeln mit dem Reiz der Mikroben zu Wurzelausscheidungen initiiert. Das Ziel dieser Arbeit war die transkriptomischen Antwort von Wurzelbakterien B. Mycoides , Kartoffel Wurzelausscheidungen untersuchen. Um dies zu erfüllen, wurden Kartoffelknollen Oberfläche sterilisiert und in autoklaviert Vermiculit gekeimt. Dann wurden die Wurzelausscheidungen gesammelt, wie in Abbildung 1dargestellt. Um ausschließen die Wurzelausscheidungen, die Auswirkungen auf das Wachstum von Bakterien, bis zu 15 % der die Wurzelausscheidungen wurden hinzugefügt, um die B. Mycoides -Kultur und keine Veränderung im Wachstum während der Messzeit (Abbildung 2) erkannt wurde. So wurden die Veränderungen der Genexpression beobachtet in dieser Studie nicht wahrscheinlich durch Wachstum verbundenen Effekte verursacht.
Nach der Entnahme die Wurzelausscheidungen wurden hinzugefügt, um die B. Mycoides -Kultur bei einem Verhältnis von 10 % (V/V), und die bakterielle total RNA isoliert wie zuvor beschrieben. Die RNA wurde dann durch automatisierte Elektrophorese Instrument einer Qualitätsprüfung unterzogen, die Ergebnisse in Abbildung 3dargestellt sind. Abbildung 3A und 3 b die RNA isoliert von B. Mycoides behandelt die Wurzelausscheidungen und Abbildung 3 und 3D darstellen der RNS isoliert von der Kontrollgruppe. Alle Proben erzielte RIN Werten über 9 mit zwei klare Streifen entsprechend der 16 s und 23 s RNA-Untereinheiten, zeigen, dass qualitativ hochwertige RNA durch dieses Protokoll erhalten wurde.
Nach Vorbereitung der Bibliothek wurden paar Ende liest mit einer Hochdurchsatz-Sequenzierung-Plattform erhalten. Der rohen RNA-Seq liest wurden aus dem Adapter Sequenzen getrimmt und gegen die Referenz Genomsequenz zugeordnet. Die daraus resultierenden Daten wurde die RPKM-Tabelle generiert. Mit der T-REx-Pipeline wurde die Transkriptom-Analyse durchgeführt. Das Verhältnis Intensität Grundstück der differentiell exprimierten Gene ist in Abbildung 4dargestellt. Im Vergleich zu einem Steuerelement induziert die Zugabe von Kartoffeln Wurzelausscheidungen 715 Gene differenziell ausgedrückt werden. Von denen 408 Gene wurden hochreguliert und 307 Gene wurden herunterreguliert15. Die relative Veränderung der Gene mit veränderten Ausdruck ist in Tabelle 1aufgeführt.
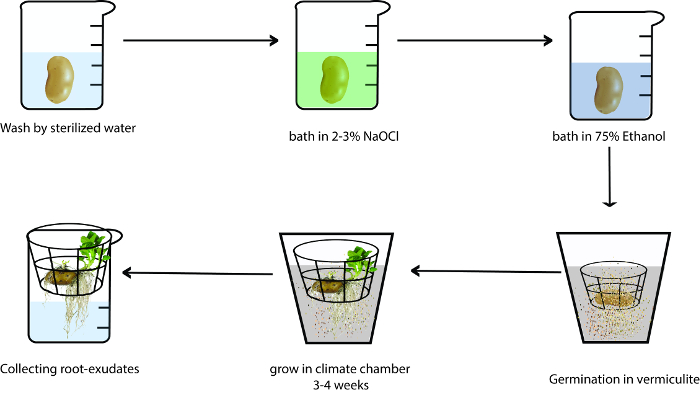
Abbildung 1: Verfahrensschema der Sammlung von Kartoffel Wurzelausscheidungen. Die verwendeten Materialien sind autoklaviert und die Keimung erfolgt in einer Klimakammer. Die Kartoffelknolle mit sterilisierten Wasser waschen und in ca. 2-3 % NaOCl Bad, für 5 min. Bad es in 75 % Ethanol für eine weitere 5 min. Platz die Kartoffel in einem Autoklaven Korb und steckte es in einen Topf mit nassen Vermiculit. Wachsen Sie die Kartoffel in einer Klimakammer für 3-4 Wochen, und übertragen Sie dann den Korb mit der Kartoffel-Keimling in ein Becherglas. Sammeln Sie die Wurzelausscheidungen jeden Tag und füllen Sie den Becher mit Wasser sterilisiert. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 2: Die Wachstumskurve von B. Mycoides mit verschiedenen Konzentrationen der Kartoffel Wurzelausscheidungen. Der Sorte EC18 war angebaut in einem flüssigen LB-Medium mit dem Zusatz von Kartoffel Wurzelausscheidungen oder sterilen H2O. OD600 wurde jeweils 1 h gemessen und über der Zeit aufgetragen. Alle Gruppen zeigen ein ähnliches Wachstumsmuster hat darauf hinweist, dass bis zu 15 % der Wurzel Exsudate Addition keine wesentlichen Auswirkungen auf B. Mycoides Wachstum. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 3: RNA-Qualitäts-Check durch das automatisierte Elektrophorese Instrument. A und B stellen die RNA isoliert von B. Mycoides mit Wurzelausscheidungen und C und D sind die RNA isoliert von der Kontrollgruppe behandelt. Die RNA-Proben zeigen zwei klare Bänder entsprechend 23 s und 16 s rRNA und eine schwache 5 s rRNA Band. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 4: Verhältnis Intensität Plot zur Visualisierung von differenziellen Genexpression von RNA-Seq-Proben von B. Mycoides in Reaktion auf Kartoffel Wurzelausscheidungen. Die Figur wird automatisch vom T-REx generiert. Die X-Achse repräsentiert die Gen-Expression, und die Y-Achse stellt die log2-transformierten Falte Änderung dar. Die Gene werden up- und herunterreguliert haben positive und negative log2 Verhältniswerte. Die Punkte im Bereich gestreiften kennzeichnen Gene, die nicht deutlich über - oder unter-zum Ausdruck gebracht werden. Bitte klicken Sie hier für eine größere Version dieser Figur.
| Gen tag | Strang | Falten Sie ändern | Annotation |
| BG05_RS09165 | + | 3.3 | Multidrug Efflux protein |
| BG05_RS20930 | + | 25,3 | Membranprotein |
| BG05_RS10935 | + | 23.3 | Stadium III Sporenbildung Protein AD |
| BG05_RS08990 | - | 11.8 | Sporenbildung protein |
| BG05_RS16405 | + | 3.9 | IclR Familie transcriptional Regler |
| BG05_RS24905 | - | 3 | Tryptophan-Synthase Untereinheit alpha |
| BG05_RS24920 | - | 2.3 | Indol-3-Glycerin-Phosphat-synthase |
| BG05_RS22255 | - | 27 | Acetolactate-synthase |
| BG05_RS22250 | - | 6.5 | Ketol-Säure reductoisomerase |
| BG05_RS22265 | - | 26.4 | verzweigtkettigen Aminosäuren-aminotransferase |
| BG05_RS18715 | - | 2.8 | Pullulanase |
| BG05_RS18040 | + | -9.1 | Keimung Protein YpeB |
| BG05_RS16930 | + | -4,2 | ABC-Transporter-ATP-bindeprotein Zucker |
| BG05_RS27345 | + | -2,9 | MFS-transporter |
| BG05_RS19555 | - | -3,3 | PTS Zellobiose Transporter Untereinheit IIB |
| BG05_RS24345 | - | -2,7 | Putrescin Importeur |
| BG05_RS22525 | - | -12,4 | Cardiolipin-synthase |
| BG05_RS15225 | - | -3,3 | Membranprotein |
| BG05_RS18475 | + | -5,7 | Membranprotein |
| BG05_RS19095 | + | -2,1 | Keimung protein |
Tabelle 1: Liste der differentiell exprimierten Gene einer Wurzel Exsudate-behandelten Gruppe im Vergleich zu einem Steuerelement.
Diskussion
Pflanze-Mikroben-Interaktionen haben vermutet, um durch ein fein abgestimmtes Gleichgewicht zwischen Bakterien und Pflanzen bestimmt werden. Solche Interaktionen sind sehr komplex und schwierig zu studieren in ein natürliches System, umfasst verschiedene Mikrobenarten, potenziell als Konsortien. Dieses Whitepaper beschreibt ein vereinfachtes Protokoll, um die bakterielle Reaktion auf Wurzelausscheidungen unter kontrollierten Bedingungen zu studieren. Das Transkriptom-Profil des standarddüngung, bei Kontakt mit Wurzelausscheidungen, liefert detaillierte Informationen über bakterielle Anpassung an die Rhizosphäre-Nische. Diese Wurzel Exsudate Sammlung Protokoll erfordert keine komplizierte Verfahren und Spezialausrüstung. Allerdings müssen alle Verfahren unter streng sterilen Bedingungen durchgeführt werden und eine Sterilität Kontrolle einbezogen werden sollten. Wir empfehlen mehrere Kartoffelknollen parallel wachsen und verwerfen die kontaminierten. Dieses Protokolls können Änderungen vorgenommen werden, wenn andere Pflanzenarten untersucht werden. Es ist wichtig, eine geeignete Methode zu verwenden, um alle Samen/Knollen zu sterilisieren, weil sie Toleranz gegenüber den Desinfektionsmitteln angewendet variieren.
Sobald die Wurzelausscheidungen erhalten sind, muss qualitativ hochwertige RNA aus den bakteriellen Zellen isoliert werden. Verschiedene Reagenzien und Standardprotokolle, die in erster Linie auf die Phenol/Chloroform oder TRIzol Methode basieren sind zeitraubende16. Die kommerziellen Kits eignen sich vor allem für Modellorganismen aber weniger auf andere anwendbar sind. Diese RNA-Isolierung-Protokoll, das Phenol/Chloroform-Methode und ein RNA-Isolierung-Kit verbindet überwindet die Nachteile dieser Methoden. Darüber hinaus gehört ein Wulst-schlagende Schritt B. Mycoides Zellen zu homogenisieren, die in der Regel in der planktonischen Kultur zu aggregieren. Potenzielle DNA-Kontamination wird durch eine zusätzliche Inkubation mit DNase vor der Elution entfernt. Die hohe RIN Zahl bedeutet die Isolation intakt RNA. Somit ist dieses Protokoll besonders effizient und zeitsparend für ökologische Bakterienstämme.
Nach RNA Sequenzierung, Datenanalyse erfolgt mit T-REx, eine Web-basierte statistische Analyse Pipeline für RNA-Seq Gen Ausdruck Daten14. Diese Pipeline ist benutzerfreundlich, besonders für Biologen ohne umfangreiche Bioinformatik wissen. Die Eingabedatei ist, dass die RNA Ausdruck Ebene Rohdaten, wie RPKM, Fragmente pro Kilobase pro million zugeordneten liest (FPKM), zählt pro million zugeordneten liest (CPM) oder andere Gen-Ausdruck-Einheiten. Solche Gene Expression Wert Dateien können durch verfügbaren Werkzeuge einschließlich SAMtools17, BEDtools18und NGS-Trex19erzeugt werden. Um die RNA-Seq-Analyse durchführen, sind drei andere input-Dateien erforderlich. Diese Dateien werden verwendet, um die Faktoren zu definieren, die beschreiben, die Experimente und die Wiederholungen, die Vergleiche zwischen den verschiedenen experimentellen Bedingungen und die Gruppen von Genen von Interesse. Wenn die input-Dateien hochgeladen werden, dauert die Analyseprozess nur ein paar Minuten.
Mehreren differentiell exprimierten Genen von B. Mycoides EC18, Behandlung mit Wurzelausscheidungen, sind in Tabelle 1aufgeführt. Unter ihnen ist der Ausdruck von mehreren Genen Codierung Membranproteine verändert. Gene im Zusammenhang mit der Sporenbildung oder Keimprozess werden differenziell ausgedrückt. Die Expression von Genen beteiligt Sporenbildung ändert sich auch in B. Subtilis als Co mit Reis Sämlinge kultiviert, weil Wurzelausscheidungen die Energie liefern für das dynamische Wachstum der bakteriellen18 Zellen. Der Ausdruck der IclR transcriptional Regler, bezogen auf multidrug-Resistenz und der Abbau von aromatischen Verbindungen in Bodenbakterien ist hochreguliert. Die IclR Löschung Stamm von standarddüngung Klebsiella Pneumoniae ist die mineralische Phosphat Solubilisierung, verglichen mit der Wildtyp Stamm19zurückgegangen. Mehrere Gene, die im Zusammenhang mit Aminosäuren-Stoffwechsel und Synthese werden stimuliert, während Gene, die in Zucker Verkehr einschließlich Zellobiose PTS Transporter herunterreguliert werden. Dies deutet darauf hin, dass Belastung EC18 möglicherweise eine metabolische Vorliebe für Aminosäuren über Zucker in der Rhizosphäre. Die Funktion der veränderten Gene kann weitere studierte in Situ werden, indem ko oder Überexpression Mutanten. Zusammenfassend lässt sich sagen ermöglicht das hier beschriebene Protokoll eine schnelle Identifizierung einer Vielzahl potenziell wichtigen bakteriellen Gene in Pflanzen-Mikroben-Interaktionen.
Offenlegungen
Die Autoren erklären, dass sie keine finanziellen Interessenkonflikte.
Danksagungen
Wir danken Jakob Viel für seine hilfreichen Kommentare und Vorschläge. Wir danken auch Anne de Jong für seine Hilfe bei der Analyse der Bioinformatik. Yanglei Yi und Zhibo Li werden von der China Scholarship Council (CSC) unterstützt. Wir bedanken uns bei NWO-TTW Perspectief Programma Back2Roots (TKI-AF-15510) für ihre finanzielle Unterstützung auf OPK.
Materialien
| Name | Company | Catalog Number | Comments |
| sodium hypochlorite | Sigma | CAS: 7681-52-9 | 10-15% active chlorine |
| Luria-Bertani (LB) broth | |||
| incubater | New Brunswick Scientific | Innova 4000 | |
| spectrophotometer | Thermo Fisher Scientific | Genesys 20 | |
| liquid nitrogen | |||
| glass beads | Sigma | G8893 | 0.5 µm |
| 2.0 ml tube with screw cap | RNase free | ||
| 1.5 ml and 2.0 ml eppendorf tube | RNase free | ||
| Bead mill homogenizer | BioSpec | 607 | Mini_beadbeater |
| centrifuge | Eppendorf | 5430 | |
| Diethyl pyrocarbonate (DEPC) | sigma | CAS: 1609-47-8 | |
| Sodium Dodecyl Sulfate (SDS) | sigma | CAS: 151-21-3 | 10% solution prepared with DEPC treated MQ water |
| TE buffer | 10 mM Tris-HCl; 1 mM EDTA, pH=8 | ||
| phenol | Sigma | RNA grade | |
| chloroform-isoamyl alcohol | prepare 24:1 of chloroform:isoamyl alcohol, store at room temperature | ||
| High pure RNA isolation kit | Roche | 11828665001 | |
| RNase Decontamination Solution | Invitrogen | AM9780 | RNase-Zap |
| Automated electrophoresis instrument | Agilent | 2100 | Bioanalyzer |
| Microvolume spectrophotometer | Thermo Fisher Scientific | Nanodrop ND-1000 | |
| RNA quality analysis kit | Agilent | RNA 6000 Nano kit | |
| RNase inhibitor | Thermo Fisher Scientific | RiboLock | |
| Directional RNA library Prep kit | NEB | Ultra | For Illumina |
Referenzen
- Haichar, F. e. Z., et al. Plant host habitat and root exudates shape soil bacterial community structure. The ISME Journal: Multidisciplinary Journal of Microbial Ecology. 2 (12), 1221-1230 (2008).
- Badri, D. V., Vivanco, J. M. Regulation and function of root exudates. Plant, Cell & Environment. 32 (6), 666-681 (2009).
- Rohrbacher, F., St-Arnaud, M. Root exudation: the ecological driver of hydrocarbon rhizoremediation. Agronomy Journal. 6 (1), 19 (2016).
- Mark, G. L., et al. Transcriptome profiling of bacterial responses to root exudates identifies genes involved in microbe-plant interactions. Proceedings of the National Academy of Sciences of the United States of America. 102 (48), 17454-17459 (2005).
- Fan, B., et al. Transcriptomic profiling of Bacillus amyloliquefaciens FZB42 in response to maize root exudates. BMC Microbiology. 12 (1), 116 (2012).
- Yoshitomi, K. J., Shann, J. R. Corn (Zea mays L.) root exudates and their impact on 14C-pyrene mineralization. Soil Biology and Biochemistry. 33 (12), 1769-1776 (2001).
- Lambert, M. R. Clover root exudate produces male-biased sex ratios and accelerates male metamorphic timing in wood frogs. Royal Society Open Science. 2 (12), (2015).
- Tuason, M. M. S., Arocena, J. M. Root organic acid exudates and properties of rhizosphere soils of white spruce (Picea glauca) and subalpine fir (Abies lasiocarpa). Canadian Journal of Soil Science. 89 (3), 287-300 (2009).
- Grayston, S. J., Vaughan, D., Jones, D. Rhizosphere carbon flow in trees, in comparison with annual plants: the importance of root exudation and its impact on microbial activity and nutrient availability. Applied Soil Ecology. 5 (1), 29-56 (1997).
- Rovira, A. D. Plant root exudates. Botanical Review. 35 (1), 35-57 (1969).
- Bargabus, R. L., Zidack, N. K., Sherwood, J. E., Jacobsen, B. J. Characterisation of systemic resistance in sugar beet elicited by a non-pathogenic, phyllosphere-colonizing Bacillus mycoides, biological control agent. Physiological and Molecular Plant Pathology. 61 (5), 289-298 (2002).
- Peng, Y. -. H., et al. Inhibition of cucumber Pythium damping-off pathogen with zoosporicidal biosurfactants produced by Bacillus mycoides. Journal of Plant Diseases and Protection. 124 (5), 481-491 (2017).
- Ambrosini, A., et al. Diazotrophic bacilli isolated from the sunflower rhizosphere and the potential of Bacillus mycoides B38V as biofertiliser. Annals of Applied Biology. 168 (1), 93-110 (2016).
- de Jong, A., van der Meulen, S., Kuipers, O. P., Kok, J. T-REx: transcriptome analysis webserver for RNA-seq expression data. BMC Genomics. 16 (1), 663 (2015).
- Yi, Y., de Jong, A., Frenzel, E., Kuipers, O. P. Comparative transcriptomics of Bacillus mycoides strains in response to potato-root exudates reveals different genetic adaptation of endophytic and soil isolates. Frontiers in Microbiology. 8, 1487 (2017).
- Nwokeoji, A. O., Kilby, P. M., Portwood, D. E., Dickman, M. J. RNASwift: a rapid, versatile RNA extraction method free from phenol and chloroform. Analytical Biochemistry. 512, 36-46 (2016).
- Ramirez-Gonzalez, R. H., Bonnal, R., Caccamo, M., MacLean, D. Bio-samtools: Ruby bindings for SAMtools, a library for accessing BAM files containing high-throughput sequence alignments. Source Code for Biology and Medicine. 7 (1), 6 (2012).
- Quinlan, A. R. BEDTools: the Swiss-army tool for genome feature analysis. Current Protocols in Bioinformatics. , (2014).
- Boria, I., Boatti, L., Pesole, G., Mignone, F. NGS-trex: next generation sequencing transcriptome profile explorer. BMC Bioinformatics. 14 (7), S10 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten