Method Article
Células T Captura bacterias por transinfection de células dendríticas
En este artículo
Resumen
Aquí se presenta un protocolo para medir la captura bacteriana por las células T CD4 + que se produce durante la presentación de antígenos a través de transinfection de células dendríticas infectadas previamente (DC). Mostramos cómo realizar los pasos necesarios: el aislamiento de pilas, la infección de la formación de conjugado de células DC, DC / T, y la medición de bacterias transfección de células T.
Resumen
Recently, we have shown, contrary to what is described, that CD4+ T cells, the paradigm of adaptive immune cells, capture bacteria from infected dendritic cells (DCs) by a process called transinfection. Here, we describe the analysis of the transinfection process, which occurs during the course of antigen presentation. This process was unveiled by using CD4+ T cells from transgenic OTII mice, which bear a T cell receptor (TCR) specific for a peptide of ovoalbumin (OVAp), which therefore can form stable immune complexes with infected dendritic cells loaded with this specific OVAp. The dynamics of green fluorescent protein (GFP)-expressing bacteria during DC-T cell transmission can be monitored by live-cell imaging and the quantification of bacterial transinfection can be performed by flow cytometry. In addition, transinfection can be quantified by a more sensitive method based in the use of gentamicin, a non-permeable aminoglycoside antibiotic killing extracellular bacteria but not intracellular ones. This classical method has been used previously in microbiology to study the efficiency of bacterial infections. We hereby explain the protocol of the complete process, from the isolation of the primary cells to the quantification of transinfection.
Introducción
Cuando un patógeno infecta a su huésped, por lo general hay una activación de la respuesta inmune innata y adaptativa, necesarios para la eliminación de bacterias. La inmunidad innata es la primera línea de defensa que impide que la mayoría de las infecciones. La inmunidad innata distinguir en unos elementos precisos manera que se conservan entre grandes grupos de microorganismos (patrones moleculares asociados a patógenos, PAMP) 1. Los mecanismos de la inmunidad innata incluyen barreras físicas tales como la piel, las barreras químicos (péptidos antimicrobianos, la lisozima) y los leucocitos innatos, que incluyen los fagocitos (macrófagos, neutrófilos y células dendríticas), mastocitos, eosinófilos, basófilos y células asesinas naturales 2. Estas células identificar y eliminar los agentes patógenos, ya sea atacando a través de contacto o mediante fagocitosis, que incluye patógenos que envuelve y matando. Este sistema no permite la defensa de toda la vida, en contraste con la inmunidad adaptativa, que confieren memoria inmunológica contra pathogens. El sistema inmune adaptativo es la segunda línea de defensa y es capaz de reconocer y reaccionar a los antígenos específicos de microbios múltiple y sustancias no microbianas 3. Los principales componentes del sistema inmune adaptativo son los linfocitos, que incluyen células B y T. Las células B están involucrados en la respuesta humoral, secretoras de anticuerpos contra agentes patógenos o proteínas exógenas. Sin embargo, las células T representan la inmunidad mediada por células, la modulación de la respuesta inmune con la secreción de citoquinas o matar las células infectadas por patógenos 4.
Células presentadoras de antígenos (APC), incluyendo células dendríticas o macrófagos, componentes del sistema inmune innato, pueden reconocer patógenos fagocitan y componentes bacterianos proceso en antígenos, que se presentan en la superficie celular por el complejo mayor de histocompatibilidad (MHC) 5.7. Después de APC han fagocitado patógenos, por lo general migran a los ganglios linfáticos de drenaje, donde interactúan con Tlas células. Linfocitos T pueden reconocer los complejos de péptido-MHC específicos por sus receptores de células T. La sinapsis inmune (IS) se produce en la interfaz entre una APC cargadas con antígeno y un linfocito durante la presentación de antígenos 8,9. Algunas bacterias pueden sobrevivir fagocitosis y difundir sistemáticamente dentro APC. En este punto de vista, las CPA infectadas sirven como reservorios de bacterias o "caballos de Troya" que facilitan la propagación de bacterias 10. El contacto íntimo entre APC y los linfocitos que tienen lugar durante el curso de la formación es también funcionan como una plataforma para el intercambio de parte de las membranas, material genético y los exosomas y puede ser secuestrado por algunos virus para infectar las células T; este proceso se llama transinfection 11-13.
Algunas bacterias patógenas (Listeria monocytogenes, Salmonella enterica y Shigella flexneri) son capaces de invadir los linfocitos T in vivo y modificar su comportamiento 14-16 de. Tenemosdescriben recientemente que los linfocitos T también son capaces de capturar las bacterias por transinfection a partir de células dendríticas infectadas previamente (DCs) durante el curso de la presentación del antígeno 16. Células T de captura bacteriana por transinfection excesivamente más eficaz (1,000-4,000x) que las infecciones directos. T patógeno captura de las células y las bacterias no patógenas que indica que transinfection es un proceso impulsado por las células T. Sorprendentemente, transinfected T (TIT) células murieron rápidamente las bacterias capturadas y lo hicieron más eficiente que los fagocitos profesionales 16. Estos resultados, que se rompen un dogma de la inmunología, muestran que las células de la inmunidad adaptativa pueden realizar funciones que eran supuestamente exclusiva de la inmunidad innata. Además, se demostró que las células Tit secretan grandes cantidades de citoquinas pro-inflamatorias y protegen de las infecciones bacterianas en vivo.
Aquí se presentan los diferentes protocolos utilizados para estudiar el proceso transinfection bacteriana enun modelo de ratón. Este modelo se basa en el uso de células T CD4 + procedentes de ratones transgénicos otii, que llevan un TCR específico para el péptido 323 a 339 de OVA (OVAp) en el contexto de la I-Ab 17 que interactúan específicamente con el hueso bacteriana infectada marrow- DCs derivadas (18,19) BMDCs cargados con OVAp, formando sinapsis inmunes estables.
Transinfection células T puede ser visualizado y rastreado mediante microscopía de fluorescencia. Además, la citometría de flujo se puede utilizar para la detección de células infectadas mediante el aprovechamiento de la fluorescencia emitida por las bacterias que expresan la proteína verde fluorescente (GFP) 16,20. Por otra parte, transinfection de células T se puede cuantificar mediante un enfoque más sensible, el ensayo de supervivencia de gentamicina que permite la medición de un gran número de eventos. La gentamicina es un antibiótico que no puede penetrar las células eucariotas. Por lo tanto, el uso de este antibiótico permite la diferenciación de bacterias intracelulares que sobrevivió a la fr Además antibióticolos extracelulares om que fueron asesinados 21.
Protocolo
Nota: Los procedimientos experimentales fueron aprobados por el Comité de Ética de Investigación de la Universidad Autónoma de Madrid y llevaron a cabo bajo la supervisión de la Universidad Autónoma de Madrid, Jefe de Protección de los Animales y la Salud, de acuerdo con las directrices españolas y europeas. Los ratones fueron criados en el libre (SPF) vivienda patógeno específico y fueron sacrificados por personal entrenado y calificado utilizando dióxido de carbono (CO 2) método de inhalación.
1. Ratón médula ósea derivada de los DC La diferenciación y la infección
Nota: La Figura 1 resume este primer paso. Todos los procedimientos deben llevarse a cabo en la capilla de ahora en adelante, utilizando sólo estériles medios, instrumentos, puntas de pipetas y placas de cultivo.
- Derivadas de la médula ósea de ratón DC Diferenciación
- Diseccionar tibias y fémures de un C57 / BL6 ratón 22 y transferir en un plato de plástico con 10 ml de medio RPMI.
- Cortar cada epífisis fuera, con tijeras estériles y exponer la médula ósea, que tiene un color rojo brillante característico.
- Infundir el interior del hueso con 10 ml de solución salina tamponada con fosfato (PBS) conteniendo 0,5% de albúmina de suero bovino (BSA) y 5 mM de ácido etilendiaminotetraacético (EDTA) (tampón / BSA / EDTA PBS) usando una jeringa estéril en un Petri estéril plato.
- Recoger la suspensión de células y se centrifuga en un tubo a 600 xg en una centrífuga refrigerada (4 ° C).
- Resuspender las células en 1 ml de cloruro de amonio--potasio (ACK) de tampón de lisis (0,15 M NH 4 Cl, 10 mM KHCO3 y EDTA 0,1 mM) y se incuba durante 30 segundos a RT con el fin de eliminar las células rojas de la sangre.
- Añadir 10 ml de medio RPMI 1640 con suero bovino fetal al 10% (FBS) y filtrar a través de un filtro de células (70 micras) para eliminar los fragmentos de hueso y grupos de células antes de centrifugar a la misma velocidad (600 xg) porque grumos se eliminaron por filtración.
- Contar las células de und ajustar a 5 x 10 5 células / ml con RPMI 10% FBS (que contiene 50 mM B-mercaptoetanol, piruvato de sodio 1 mM) y se añade 20 ng / ml de factor recombinante murino de granulocitos y macrófagos estimulante de colonias (rm-GM-CSF) como una concentración final.
- Añadir esta suspensión a una, la calidad microbiológica estéril, 15 cm placa de Petri, y la cultura en un incubador de CO2 (37 ° C, 5% de CO 2).
- Cada tres días, giran hacia abajo tanto las células no adherentes y células independientes con EDTA 5 mM en PBS y resuspender en 5 x 10 5 células / ml de densidad celular, con un nuevo medio que contiene 20 ng / ml rm-GM-CSF.
- La maduración de las DC y Antigen Cargando
- Al día 9, resuspender las células en 5 x 10 5 células / ml de densidad en medio que contiene 20 ng / ml rm-GM-CSF. A continuación, añadir 20 ng / ml de lipopolisacárido (LPS) para permitir una alta expresión de MHC-II en las células y se incuba en un no-tejido de cultivo estéril tratada con plato de 15 cm Petri durante 24 horas.
- Después de unadía de LPS-estimulación, manchar algunas células DC con anticuerpos contra CD11c ratón, MHCII y Gr-1 para comprobar los DC diferenciación mediante citometría de flujo. Un ejemplo de análisis de citometría de flujo de DCs se muestra en la Figura 3A y 3B.
- Incubar las DC (5 x 10 5 células / muestra) con un anticuerpo monoclonal anti-ratón-CD16 / CD32 a 2,5 g / ml de concentración final en 50 l de PBS / BSA / tampón EDTA por muestra. Después, se añaden 50 l de los anticuerpos contra MHCII (IA / IE), CD11c y Gr-1 conjugado con diferentes fluorocromos a 1: 100, 1: 200 y 1: 500 en tampón de EDTA BSA PBS / /, respectivamente.
- Finalmente, lavar con DCs BSA tampón de EDTA PBS / / y analizar por citometría de flujo de acuerdo con el protocolo del fabricante.
Nota: Si las células están bien diferenciadas, las células están listas para el antígeno de carga.
- Wash DC con RPMI 10% FBS (sin antibióticos) y se incuba con 10 mg / ml otii OVAp (OVA 323-339; ISQAVHAAHAEINEAGR) en un tubo de plástico en 1 ml de RPMI 10% FBS medio por cada 5 x 10 6 países en desarrollo para un mínimo de 1 hora a 37 ºC. Como control negativo, deje las DC sin incubación con OVAp.
- La infección de las DC
- Infectar a los países en desarrollo a una multiplicidad de infección (MOI) de 10 bacterias (10 bacterias por DC) durante 1 hora en un incubador de CO2 (37 ° C, 5% de CO 2).
- Lavar las células DC 3 veces en PBS 1x y en RMPI 10% de FBS y se centrifuga a 450 xg para lavar la mayoría de las bacterias extracelulares. Finalmente, Resuspender las células en RPMI 10% FBS medio (sin antibióticos) en 20 x 10 6 células / ml.
2. Aislamiento de las células CD4 + linfocitos T de ratones transgénicos otii
Nota: La figura 1 resume este segundo paso. Los ganglios linfáticos se deben utilizar en lugar de bazo para aislar células T CD4 +, porque la proporción de CD4 + linfocitos en los ganglios linfáticoss (~ 50%) es mayor que en el bazo (~ 25%) y por lo tanto la purificación sería más eficaz.
- Hacer suspensión de células única de los ganglios linfáticos
- Eliminar inguinal, axilar, braquial, cervical y nódulos linfáticos mesentéricos de ratones transgénicos 23 otii y transferir en un plato de plástico con 10 ml de RPMI 10% FBS medio.
- Ganglios linfáticos Grind bajo una campana estéril utilizando dos portaobjetos esmerilado. Coloca los ganglios linfáticos en un lado esmerilado de una diapositiva de microscopio, y frote con el lado mate de la segunda diapositiva hasta órganos han sido terreno.
- Lavar suspensión de células individuales en tampón PBS / BSA / EDTA.
- Filtre las células a través de un filtro de células (70 micras) para eliminar el tejido conectivo y lavar con BSA EDTA buffer PBS / /.
- Resuspender las células en 1 ml de tampón de lisis ACK y se incuba durante 1 min a TA con el fin de eliminar las células rojas de la sangre.
- Aislamiento de células T CD4 +
- Lave de nuevo como en el paso 2.1.3 y el recuentocélulas para resuspender en 100 x 10 6 células / ml en tampón de EDTA BSA PBS / /. Añadir anticuerpos biotinilados contra CD8, IgM, B220, CD19, MHC de clase II (I-Ab), CD11b, CD11c y DX5 a 1: 250 durante 30 min en hielo durante posterior selección negativa de las células T CD4 +.
- Lavar las células en tampón de EDTA BSA PBS / / e incubar las células (100 x 10 6 células / ml) con microperlas de estreptavidina a la concentración recomendada en el protocolo del fabricante durante 15 min en hielo.
- Lavar las células en tampón de EDTA BSA PBS / / y filtrarlas a través de un filtro de células (30 micras) antes del aislamiento de células T CD4 +.
- Aislar células T CD4 + por selección negativa usando una máquina de separación celular magnética de acuerdo con el protocolo del fabricante.
- Contar las células y ajustar a 4 x 10 6 células / ml con RPMI 10% FBS medio (sin antibióticos).
- Fijar y mantener en hielo aislado células T CD4 + (3 x 10 5 células) para comprobar la pureza de flujocitómetro de 24 como se describe. Un ejemplo de T CD4 + de purificación de las células se muestra en la Figura 3C.
3. Células T transinfection Medición por ensayo de protección Gentamicina
Nota: La Figura 2 resume esta tercera etapa del protocolo.
- Transinfection células T
- Se incuban las células T CD4 + aisladas con células infectadas DC (1: 1) (bacterias libres los países en desarrollo fueron infectadas durante 1 hora y se lavan para eliminar) durante 30 minutos para permitir la formación de sinapsis inmunológica en un incubador de CO2 (37 ° C, 5% CO 2).
- Añadir 0,5 ml de las células T CD4 + aisladas (2 x 10 6 células) y 0,1 ml de DCs infectadas (2 x 10 6 células) en una placa de cultivo de 24 pocillos. Como control negativo, infectar directamente 0,5 ml de las células T CD4 + a una MOI de 10 bacterias por 30 min.
- Incluir controles adicionales que separan las DC infectadas de las células T utilizandouna barrera transwell policarbonato (con 3 micras de tamaño de poro), impidiendo DC-T contacto físico. Añadir 0,1 ml de DCs dentro de la transwell y 0,5 ml de las células T CD4 + en el compartimiento inferior del pozo (placa de 24 pocillos).
- Por último, completa con medio RMPI hasta que es de 0,6 ml en cada pocillo para hacer volúmenes iguales en todas las muestras.
- Después de 30 minutos de la formación de sinapsis inmunológica, añadir 100 mg / ml de gentamicina y se incuba durante 1 hora antes de recoger las células en un incubador de CO2 (37 ° C, 5% de CO 2).
- Re-aislamiento de células T CD4 +
- Recoger las células no adherentes y volver a suspender en tampón PBS / BSA / EDTA. La mayor parte de los países en desarrollo de permanecer unidos a la placa de cultivo de plástico. Tubos Vortex suavemente para asegurar la desagregación celular. Mantenga 500 l de sobrenadante celular como control para demostrar que la gentamicina ha funcionado bien.
- Re-aislar células T CD4 + a partir de muestras que contienen DCs y las células T juntos como en el paso 2.2, Aunque la mayoría de los PED deberían haber fijado en el plato. Mantenga las muestras de control en el hielo. Tome en cuenta sólo los experimentos con la contaminación de menos de 2%. Un ejemplo de T CD4 + de purificación de células después de transinfection se muestra en la figura 3D.
- Lyse células T y de Semillas ellos en bacterias placas de medio de agar.
- Lavar las células T CD4 + dos veces con PBS.
- Contar las células y resuspender ellos en 2 x 10 6 células / ml en PBS.
- Añadir 500 l de 0,1% de Triton X-100 en 500 l de las células T para lisar ellos en 1 x 10 6 células / ml, la liberación de las bacterias intracelulares fuera de las células T.
- Haga 3 diluciones decimales seriadas de células lisadas y sembrar 50 l de cada dilución en bacterias placas de agar medianas. Divida placas en 4 porciones y sembrar las diferentes diluciones de células lisadas en cada parte. Los resultados representativos se muestran en la Figura 4.
- Como control, también la placasobrenadante de células almacenada de los conjugados en agar, para asegurar que la gentamicina ha funcionado bien. También la placa DCs infectadas como un control adicional. En caso de baja contaminación DC se produce (<2%), restar las unidades formadoras de colonias (UFC) correspondientes a la contaminación de bajo DC.
4. Cuantificación de T transinfection celular por citometría de flujo
Nota: La figura 2 resume este paso.
- T transinfection celular
- Infectar BMDCs como en el paso 1.3, pero el uso de bacterias que expresan GFP en este caso.
- Células T CD4 + (las manchas aisladas de los ganglios linfáticos en el paso 2.2) con un aminocumarina clorometil rastreador celular (CMAC) durante 30 min a 37 ºC de acuerdo con el protocolo del fabricante.
- Lavar las células T CD4 + marcadas dos veces con RMPI 10% de FBS (sin antibióticos).
- Incubar DCs infectadas (después de 1 h de incubación con bacterias) con la etiqueta CD4 +Células T durante 30 min para permitir la formación de sinapsis inmunitaria a 37 ºC incluyendo todos los controles que explican en el paso 3.1.1.
- La tinción de las células infectadas T CD4 +
- Después de 30 minutos de DC-T la formación de sinapsis inmunológica, recoger las células y lávelas en PBS / BSA / EDTA.
- Después de dos lavados con tampón PBS / BSA / EDTA, las células agregados separados mediante agitación con vórtex suavemente los tubos.
- Fijar las muestras en 0,2 ml / tubo de PBS que contenía 3% PFA durante 15 min a RT. A continuación, lavar en PBS.
- Incubar con el anticuerpo monoclonal anti-ratón-CD16 / CD32 (2,5 g / ml) en 50 l / muestra de PBS / BSA / EDTA durante 15 min en hielo para bloquear los receptores Fc de las células dendríticas.
- Añadir un conejo anti-bacterias anticuerpo (10 mg / ml como concentración final) en 50 l / muestra de PBS / BSA / EDTA durante 30 min en hielo para teñir las bacterias extracelulares.
- Lavar las células con PBS / BSA / EDTA y se incuba con un anticuerpo contra CD11c ratón conjugado con ficoeritrina (PE) y un secundaria anticuerpo contra conejo conjugado con otro fluorocromo como Alexa Fluor-647 a 1: 200 en 100 l / muestra de PBS / BSA / EDTA durante 30 min en hielo.
- Lavar las células y analizar muestras por citometría de flujo. No te olvides de incluir una muestra de las células T transinfected utilizando bacterias no fluorescentes como control negativo y muestras etiquetadas con sólo uno de cada fluorocromo utilizado en el experimento para compensar las muestras y ajustar la configuración de citometría de flujo 25. Análisis representativo se muestra en la Figura 5.
Nota: Si las bacterias no expresan GFP, para distinguir intracelular y bacterias extracelulares, las bacterias extracelulares de la etiqueta, fijar y permeabilizar las muestras con Triton X-100 al 0,5% en PBS durante 5 min. Después manchar bacterias totales (intracelulares + extracelular) con un fluorocromo diferente.
Resultados
Aquí describimos cómo realizar transinfection bacteriana de células T murino desde la médula ósea infectada derivado de DCs y cómo medir transinfection bacteriana a través de dos enfoques diferentes: citometría de flujo y gentamicina supervivencia ensayo de la Figura 1 resume el procedimiento para obtener las células.. DC se generan mediante la incubación de células de médula ósea con GM-CSF durante 9 días. Entonces, los países en desarrollo son madurados con LPS para aumentar MHCII en su membrana para cargarlos más tarde con un péptido específico de OVA (OVAp). Como control, algunos países en desarrollo no se cargan con OVAp. Ambos DCs (OVAp-cargado y no cargado) se infectaron a una multiplicidad de infección (MOI) de 10 bacterias. Por otro lado, las células T CD4 + se aislaron de los ganglios linfáticos de ratones transgénicos otii, que reconocen la OVAp específico. La Figura 2 representa el procedimiento para llevar a cabo y cuantificar transinfection células T incluyendo los controles negativos apropiados. Como descricama antes, algunos países en desarrollo no se cargan con OVAp. En este caso, hay una interacción entre los DC y células T, pero no la formación de sinapsis inmune debido a la ausencia de OVAp. Un control adicional se incluye, separando los DC y células T con una barrera transwell policarbonato (con 3 micras de tamaño de poro), lo que impide DC-T contacto físico. DC se incluyen dentro de la transwell; como resultado, no son capaces de pasar a través de este tamaño de poro. Otro control negativo también se incorpora, la infección directa de las células T, mediante la incubación de las células T directamente a una MOI de 10 bacterias. Transinfection de células T se puede cuantificar mediante dos enfoques: citometría de flujo o ensayo de supervivencia de gentamicina. Para medir mediante citometría de flujo, las DCs debería haber sido infectado previamente con células bacterias-GFP y T CD4 + mancha con un rastreador de células (células azules-T). Después de la formación de conjugado, las células deben ser teñidas con anticuerpos contra DC (CD11c-PE). Las bacterias extracelulares también deben teñirse con el fin de quantify el porcentaje de células T CD4 + infectados por citometría de flujo más adelante. Para medir transinfection por ensayo de supervivencia de gentamicina, las células se incuban con gentamicina durante 1 hr para matar las bacterias extracelulares, a continuación, las células T se vuelven a aislar a sembrar en placa de agar. Diluciones en serie decimales se hacen para sembrar 50 l de cada dilución en placa de agar y facilitar el recuento de UFC.
Como se muestra en la Figura 3, la diferenciación DC tiene que ser comprobado por citometría de flujo (Figura 3A y 3B). DCs debe expresar CD11c y MHCII (Figura 3A) y no Gr-1, que es un marcador de neutrófilos. Tal como se presenta en la Figura 3B, algunos neutrófilos (3,27% de Gr-1 +, MHC -) se presentaron en la cultura, pero la cultura DCs con más de 5% de neutrófilos contaminación no deben ser utilizados. T CD4 + células pureza debe ser revisado después del aislamiento de Lynodos mph (Figura 3C) y después de la separación de los DC conjuga la formación (Figura 3D). Tener en cuenta los experimentos con menos de 2% de contaminantes después de la formación de conjugados.
La Figura 4 muestra un ejemplo de T experimento transinfection usando Salmonella. Como se ilustra en la figura 4A, las placas se dividieron en cuatro porciones a las semillas de las diluciones en serie en cada parte. Salmonella han crecido en placas de agar LB y unidades formadoras de colonias se pueden contar. Como se muestra en la figura 4A, Salmonella fue capturado por las células T de las DC infectadas y este proceso fue claramente mejorado con reconocimiento OVAp (placas a la izquierda de la Figura 4A y Figura 4B). Sin embargo, cuando había una barrera física que impide el contacto entre las células DCs y T, prácticamente no hubo transinfection, como en el caso de hacer una camiseta directo la infección de células (placas a la derecha de la Figura 4A y Figura 4B).
Como se muestra en la Figura 5, transinfection de células T se puede cuantificar mediante citometría de flujo utilizando bacterias que expresan una GFP brillante. Células DCS y T pueden ser diferenciados por tamaño y complejidad en un diagrama de dispersión hacia adelante y lateral, pero diferentes marcadores se pueden utilizar para así distinguirlos. Células T CD4 + se marcaron con un rastreador celular (CMAC) y DCS con un anticuerpo contra CD11c. Bacterias extracelulares se tiñeron con un anticuerpo de conejo contra Salmonella y el anticuerpo secundario anti-conejo conjugado con Alexa Fluor 647. Por lo tanto, el porcentaje de células T CD4 + infectadas se puede cuantificar porque están etiquetados con CMAC (no con CD11c-PE) y expresaron GFP (bacterias-GFP), pero no se tiñeron con el anticuerpo contra las bacterias extracelulares (Alexa Fluor 647).
ntent "fo: keep-together.within-page =" 1 ">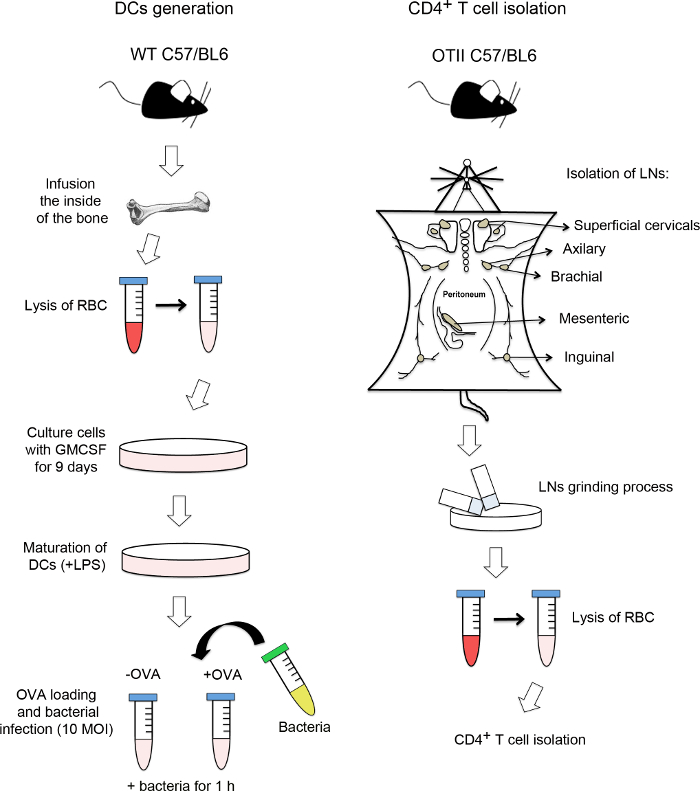
Figura gráfico 1. Flujo de la generación de DC y T CD4 + células aislamiento. En el lado izquierdo de la figura, el proceso para diferenciar las células dendríticas (DC) a partir de células de médula ósea es detallada. Después de la disección de fémur y la tibia, células de médula ósea se extraen desde el interior de los huesos. Después, las células rojas de la sangre (RBC) y se lisan las células de la médula ósea se cultivan con el factor recombinante murino de granulocitos y macrófagos estimulante de colonias (GM-CSF) durante 9 días. Una vez que están bien diferenciado, las DC se incuban con lipopolisacárido (LPS) para aumentar Complejo Mayor de Histocompatibilidad II (MHCII) expresión. Entonces, las DCs se cargan con un péptido ovoalbúmina específica (OVAp) y se infectaron con una multiplicidad de infección (MOI) de 10 bacterias. Algunos de ellos no están cargados de OVAp como control. En el lado derecho de la figura, la forma deaislar células T CD4 + de ratones transgénicos se representa otii. Los ganglios linfáticos (LNS) se quitan y se muele, obteniéndose una suspensión de una sola célula. Por último, las células T CD4 + se aislaron por selección negativa usando una máquina de separación celular magnética. Por favor haga clic aquí para ver una versión más grande de esta figura.

Figura 2. Diagrama de flujo del procedimiento de T transinfection celular. Infectada (bacterias se representan en verde) y OVAp cargan-DCS (glóbulos rojos) se incuban con las células T CD4 + (células azules) para permitir la formación de sinapsis inmune. DCs infectadas (pero no cargado con OVAp) también se incuban con células T, que permite la interacción entre ambas células pero sin la formación de sinapsis inmune. Un control adicional se incluye, separando las células CD4 + células T y los países en desarrollo con una barrera transwell policarbonato (+ TW), lo que impide el contacto de células DC-T. La infección directa de las células T también se incorpora como control negativo. Finalmente, transinfection de células T se puede cuantificar mediante dos métodos, por citometría de flujo (lado izquierdo de la figura), que indica el número de células T captura de bacterias, y por el ensayo de gentamicina supervivencia (lado derecho de la figura) un método muy sensible ( una bacteria que infecta puede ser detectado) que indica la tasa de infección bacteriana. Haga clic aquí para ver una versión más grande de esta figura.
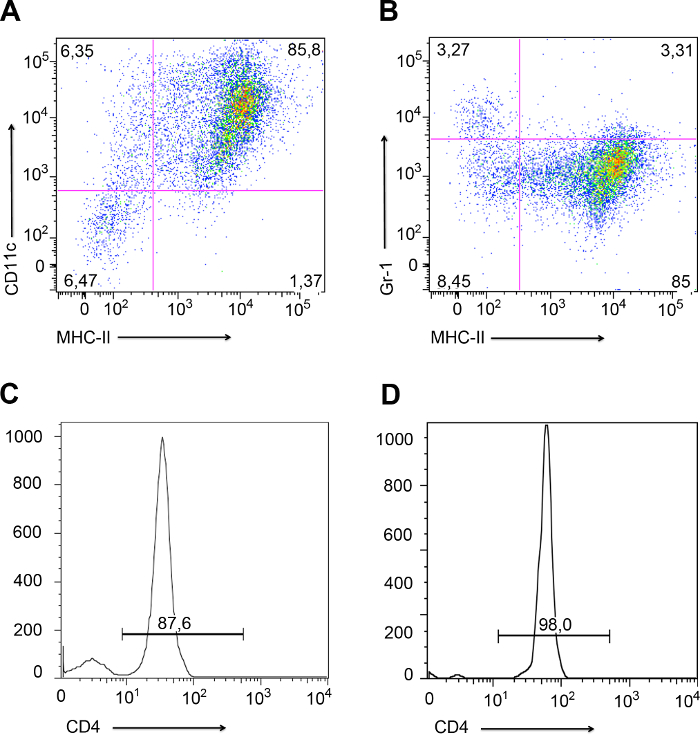
Figura 3. citómetro de flujo análisis de la médula ósea derivada de DCs células T CD4 + aisladas y. Gráficos de puntos representativos de la médula ósea derivados-DCS analizados porcitometría de flujo. (A) DC expresó MHCII y CD11c (85,8%; cuadrante superior derecho) (B) DC expresaron MHCII pero no Gr-1 (85%; cuadrante inferior derecho). Sin embargo, había un 3.27% de las células que expresaban las células MHCII GR-1 pero no (cuadrante superior izquierdo). Estas células fueron neutrófilos contaminados ligeras. (C y D) Los histogramas que representan la pureza de las células T CD4 + aisladas de los ganglios linfáticos (C), y después de la formación de conjugado con las células dendríticas (D). Haga clic aquí para ver una versión más grande de esta figura.
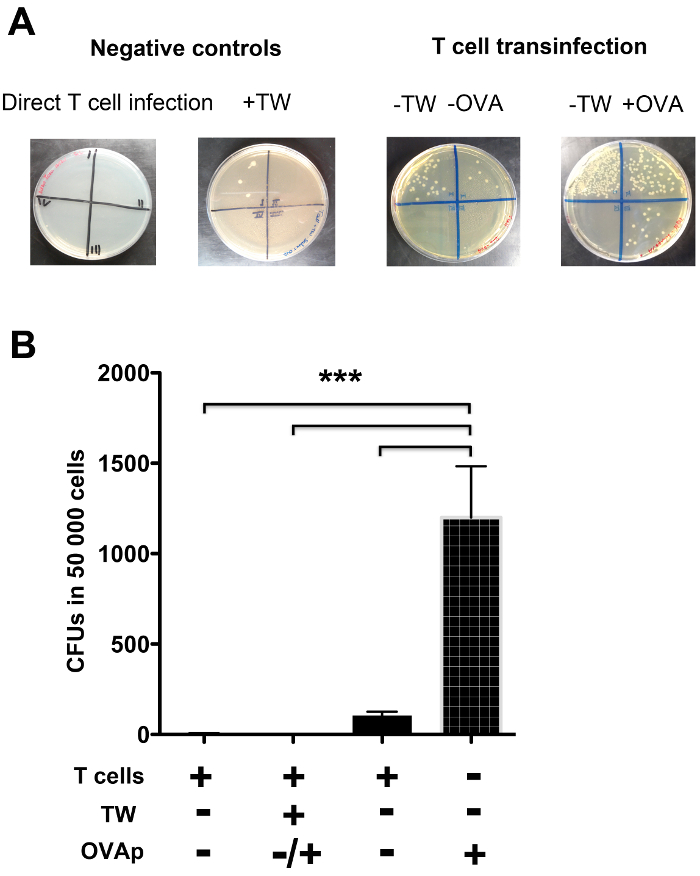
Figura 4. T transinfection celular cuantificado por ensayo de supervivencia de gentamicina. Placas de agar LB se dividieron en 4 partes correspondientes a Decimal diluciones en serie de las células T CD4 + lisadas. (A) unidades formadoras de colonias de Salmonella enterica crecieron en placas de agar LB que corresponde a transinfection de células T en presencia (+) o ausencia (-) de OVAp en la superficie de DC infectadas, y, en condiciones que permitan el contacto DC / células T (- TW) o en presencia de una barrera física que impide tales contactos (+ TW). También se muestra un plato vacío correspondiente a la infección directa de las células T (control negativo). Casi no había transinfection cuando se impidió el contacto de células DC / T (+ TW). (B) Cuantificación de unidades formadoras de colonias (UFC) a partir de varios experimentos que muestran la tasa de bacteriano capturado por las células T de las DC infectadas. La infección de células T directa, y las condiciones que impiden el contacto de células DC / T producen poco o nada de los niveles de infección de células T. Cuando se les permitió contactos de células DC / T, hubo transinfection células T, y se ve reforzada por el reconocimiento de antígenos (+ OVAp). Bares columna representan la media de 3 iexperimentos ndependent. Las barras de error indican la SD. Las diferencias significativas están representadas por ***, correspondiente a p <0,0001. Por favor haga clic aquí para ver una versión más grande de esta figura.

Figura 5. T transinfection celular cuantificado por citometría de flujo. Dot Representante gráfico que muestra los países en desarrollo y los conjugados de células T después de proceso transinfection. Células T CD4 + son pequeños y redondos como hacia adelante y dispersión lateral (FCS - SSC) shows Dot Blot, y con el fin de ser bien diferenciados, que se tiñeron con un rastreador celular (CMAC). Los DC que expresan CD11c no están etiquetados con CMAC y son más grandes que las células T CD4 + T con forma irregular. Células T CD4 + infectadas que contienen bacterias-GFP intracelular pero negativo para extracelbacterias lular (6,19%) se muestran en el cuadrante inferior derecho. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Células T o linfocitos T son un tipo de leucocitos que juegan un papel central en la inmunidad mediada por células y que pertenecen a la respuesta inmune adaptativa 26. Las células T son refractarios a ser infectados in vitro pero algunos informes indican que pueden ser infectados in vivo 14,15. Los contactos íntimos de células APC y T durante la sinapsis inmune sirven como plataformas para el intercambio de material biológico 13, incluyendo algunos virus como el VIH 11. Se ha demostrado recientemente que, contrariamente al dogma, las células T, el paradigma de las células de la inmunidad adaptativa, también son capaces de capturar eficientemente las bacterias por transinfection de los DC infectadas 11,16. Aquí mostramos detalló protocolos de cuantificar transinfection bacteriana de células T in vitro a partir BMDCs infectados.
T transinfection bacteriana celular se ve reforzada por el reconocimiento del antígeno 16. Hemos demostrado que mediante el uso de células T CD4 + aisladas deRatones transgénicos otii reconociendo un OVAp específica, BMDCs cargan con OVAp y ratones infectados con diferentes bacterias (aquí los datos se presenta de Salmonella enterica). T CD4 + de células aisladas de ratones otii se incuban con DCs infectadas durante 30 min para permitir la formación de sinapsis inmune. Los datos aquí presentados corresponden a las bacterias que células T pueden capturar de los países en desarrollo en este corto período de tiempo. Sabemos que exposiciones más largas de las células T a las DC infectadas resultaron en capturas bacterianas más grandes (que no se muestra), pero como las células T también destruyen las bacterias especialmente captado muy rápidamente, la cuantificación de transinfection bacteriana que ocurre durante largos DC / T contactos de tiempo celular resultó ser difícil . Ensayo de supervivencia de gentamicina es un método muy sensible para cuantificar transinfection de células T debido a que es capaz de detectar una sola bacteria infectante 21. Después de la incubación con gentamicina que matar sólo las bacterias extracelulares, las células T se aíslan y se lisan para sembrar en medio de agar bacterias plates. El aislamiento de las células T después de formar conjugados con países en desarrollo es un paso crítico en el protocolo. Solamente los experimentos con menos de 2% de los contaminantes deben tenerse en cuenta. Contaminación DC puede ser medida por citometría de flujo y las UFC correspondientes a la contaminación bajo DC deben ser restados. Alternativamente, las células T CD4 + de pureza se puede mejorar mediante el uso de clasificación de células.
Otro enfoque para cuantificar transinfection de células T es por citometría de flujo, utilizando bacterias que expresan GFP y etiquetado de las bacterias extracelulares con un fluoróforo compatible. Una limitación de este enfoque es que las bacterias tienen que expresar una GFP que es suficientemente brillante para la detección de células infectadas contra el fondo de autofluorescencia. Alternativamente, las bacterias pueden ser teñidas antes de infectar las DC con un rastreador celular. Otro enfoque sería la tinción de las bacterias extracelulares, permeabilización de las células T, a continuación, el etiquetado de todas las bacterias (intracelular + extracelular)con un fluorocromo diferente.
En cuanto a la orientación futura de este protocolo, que estamos tratando de establecer un protocolo para cuantificar el número de células T que capturaron bacterias después de la exposición más larga a los DC infectadas, mediante el uso de bacterias decoradas con partículas magnéticas. Las células T captura de bacterias se retienen las partículas magnéticas y por lo tanto deben ser fáciles de aislarlos mediante el uso de imanes.
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
This work was supported by grants BFU2011-29450, BFU2008-04342/BMC from the Spanish Ministry of Science and Innovation and PIES201020I046 from Consejo Superior de Investigaciones Cientìficas (CSIC).
Materiales
| Name | Company | Catalog Number | Comments |
| RPMI | Fisher Scientific | SH3025501 | |
| r-GMCSF | Peprotech | 315-03 | |
| LPS | SIGMA | L2630-10mg | |
| Na Pyruvate | Thermo Scientific | SH3023901 | |
| 2-ME | Gibco | 31350-010 | |
| OVAp OTII (323–339) | GenScript | --- | |
| Cell Strainer 70uM | BD | 352350 | |
| 30uM Syringe Filcons Sterile | BD | 340598 | |
| AutoMacs Classic | Miltenyi Biotec | 130-088-887 | |
| Gentamicin | Normon | 624601.6 | |
| Transwell | Costar | 3415 | |
| LB | Pronadisa | 1231 | |
| Agar | Pronadisa | 1800 | |
| Paraformaldehyde 16% | Electron Microscopy Sciences | 15710 | |
| Triton X-100 | |||
| CD8 biot | BD Biosciences | 553029 | |
| IgM Biot | ImmunoStep | Clone RMM-1 | |
| B220 Biot | BD Biosciences | 553086 | |
| CD19 biot | BD Biosciences | 553784 | |
| MHC-II Biot (I-A/I-E) | BD Biosciences | 553622 | |
| CD11b biot | Immunostep | 11BB-01mg | |
| CD11c biot | Immunostep | 11CB3-01mg | |
| DX5 biot | BD Biosciences | 553856 | |
| Gr-1 biot | BD Biosciences | 553125 | |
| CD16/CD32 | ImmunoStep | M16PU-05MG | |
| anti Salmonella | ABD Serotec | 8209-4006 | |
| CD11cPE | BD Biosciences | 553802 | |
| CD4-APC | Tonbo Biosciences | 20-0041-U100 | |
| Gr-1 APC | BD Biosciences | 553129 | |
| MHC-II (I-A/I-E) FITC | BD Biosciences | 553623 | |
| Alexa-Fluor 647 Goat Anti-Rabbit IgG (H+L) Antibody, highly cross-adsorbed | Invitrogen | A-21245 | |
| CMAC (7-amino-4-chloromethylcoumarin) | Life technologies | C2110 | |
| BSA | SIGMA | A7030-100G | |
| Streptavidin MicroBeads | Miltenyi Biotec | 130-048-101 | |
| BD FACSCanto II | BD Biosciences | --- |
Referencias
- Medzhitov, R. Recognition of microorganisms and activation of the immune response. Nature. 449 (7164), 819-826 (2007).
- Alberts, B., Johnson, A., Lewis, J., Raff, M., Roberts, K., Walter, P. . Molecular biology of the cell. , (1989).
- Pancer, Z., Cooper, M. D. The evolution of adaptive immunity. Annual Review of Immunology. 24, 497-518 (2006).
- Rhoades, R. A., Bell, D. R. . Medical Phisiology. , (2012).
- Cossart, P. Bacterial Invasion: The Paradigms of Enteroinvasive Pathogens. Science. 304 (5668), 242-248 (2004).
- Kaufmann, S. H., Schaible, U. E. Antigen presentation and recognition in bacterial infections. Current Opinion in Immunology. 17 (1), 79-87 (2005).
- Pizarro-Cerdá, J., Cossart, P. Bacterial Adhesion and Entry into Host Cells. Cell. 124 (4), 715-727 (2006).
- Dustin, M. L. T-cell activation through immunological synapses and kinapses. Immunological Reviews. 221, 77-89 (2008).
- Calabia-Linares, C., Robles-Valero, J., et al. Endosomal clathrin drives actin accumulation at the immunological synapse. Journal of Cell Science. 124 (5), 820-830 (2011).
- Westcott, M. M., Henry, C. J., Cook, A. S., Grant, K. W., Hiltbold, E. M. Differential susceptibility of bone marrow-derived dendritic cells and macrophages to productive infection with Listeria monocytogenes. Cellular Microbiology. 9 (6), 1397-1411 (2007).
- Geijtenbeek, T. B., Kwon, D. S., et al. DC-SIGN, a dendritic cell-specific HIV-1-binding protein that enhances trans-infection of T cells. Cell. 100 (5), 587-597 (2000).
- Izquierdo-Useros, N., Naranjo-Gòmez, M., et al. HIV and mature dendritic cells: Trojan exosomes riding the Trojan horse. PLoS Pathogens. 6 (3), e1000740 (2010).
- Mittelbrunn, M., Sanchez-Madrid, F. Intercellular communication: diverse structures for exchange of genetic information. Nature Reviews. Molecular cell biology. 13 (5), 328-335 (2012).
- McElroy, D. S., Ashley, T. J., D'Orazio, S. E. Lymphocytes serve as a reservoir for Listeria monocytogenes growth during infection of mice. Microbial Pathogenesis. 46 (4), 214-221 (2009).
- Salgado-Pabon, W., Celli, S., et al. Shigella impairs T lymphocyte dynamics in vivo. Proceedings of the National Academy of Sciences. 110 (12), 4458-4463 (2013).
- Cruz Adalia, A., Ramirez-Santiago, G., et al. T cells kill bacteria captured by transinfection from dendritic cells and confer protection in mice. Cell Host and Microbe. 15 (5), 611-622 (2014).
- Barnden, M. J., Allison, J., Heath, W. R., Carbone, F. R. Defective TCR expression in transgenic mice constructed using cDNA-based alpha- and beta-chain genes under the control of heterologous regulatory elements. Immunology and Cell Biology. 76 (1), 34-40 (1998).
- Matheu, M. P., Sen, D., Cahalan, M. D., Parker, I. Generation of bone marrow derived murine dendritic cells for use in 2-photon imaging. Journal of Visualized Experiments: JoVE. (17), (2008).
- Inaba, K., Inaba, M., et al. Generation of large numbers of dendritic cells from mouse bone marrow cultures supplemented with granulocyte/macrophage colony-stimulating factor. Journal of Experimental Medicine. 176 (6), 1693-1702 (1992).
- Thöne, F., Schwanhäusser, B., Becker, D., Ballmaier, M., Bumann, D. FACS-isolation of Salmonella-infected cells with defined bacterial load from mouse spleen. Journal of Microbiological Methods. 71 (3), 220-224 (2007).
- Vaudaux, P., Waldvogel, F. A. Gentamicin antibacterial activity in the presence of human polymorphonuclear leukocytes. Antimicrobial Agents and Chemotherapy. 16 (6), 743-749 (1979).
- Zhang, X., Goncalves, R., Mosser, D. M., Coligan, J. E. Chapter 14, The isolation and characterization of murine macrophages. Current protocols in immunology. , Unit 14.1 (2008).
- Bedoya, S. K., Wilson, T. D., Collins, E. L., Lau, K., Larkin, J. Isolation and th17 differentiation of naïve CD4 T lymphocytes. Journal of Visualized Experiments: JoVE. (79), e50765 (2013).
- Basu, S., Campbell, H. M., Dittel, B. N., Ray, A. Purification of specific cell population by fluorescence activated cell sorting (FACS). Journal of Visualized Experiments: JoVE. (41), (2010).
- Foucar, K., Chen, I. M., Crago, S. Organization and operation of a flow cytometric immunophenotyping laboratory. Seminars in diagnostic pathology. 6 (1), 13-36 (1989).
- Itano, A. A., Jenkins, M. K. Antigen presentation to naive CD4 T cells in the lymph node. Nature Immunology. 4 (8), 733-739 (2003).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados