Erkennen von ökologischen Mikroorganismen mit der Polymerase-Kettenreaktion und Gelelektrophorese
Überblick
Quelle: Labors von Dr. Ian Pfeffer und Dr. Charles Gerba - Arizona University
Demonstrierende Autor: Bradley Schmitz
Polymerase-Kettenreaktion (PCR) ist eine Technik verwendet, um Mikroorganismen zu erkennen, die in Boden, Wasser und atmosphärischen Umgebungen vorhanden sind. Durch die Verstärkung bestimmte Abschnitte der DNA, kann PCR Detektion und Identifizierung von Ziel Mikroorganismen bis zu den Arten, Dehnung und Serovar/Pathovar Ebene erleichtern. Die Technik kann auch verwendet werden, um ganze Gemeinden von Mikroorganismen in Proben zu charakterisieren.
Die Kultivierung von Mikroorganismen im Labor mit speziellen Wachstumsmedien ist eine seit langem etablierte Technik und bleibt im Einsatz für die Detektion von Mikroorganismen in Umweltproben. Viele Mikroben in der natürlichen Umwelt zu Lebzeiten, geringe Stoffwechselaktivität und/oder Verdoppelung Mal erhalten und sind somit als lebensfähig aber nicht kultivierbarer Organismen (VBNC) bezeichnet. Kultur-basierte Techniken allein kann diese Mikroben nicht erkennen und daher nicht zur Verfügung, stellen einer gründlichen Bewertung der mikrobiellen Populationen in Proben. Die Verwendung der PCR ermöglicht die Erkennung von kultivierbaren Mikroorganismen, VBNC Organismen, und diejenigen, die nicht mehr am Leben oder aktiv, wie die Verstärkung der genetischen Sequenzen nicht in der Regel die Voranreicherung von Mikroorganismen erfordern in Umweltproben. PCR kann nicht jedoch die genannten Staaten der Lebensfähigkeit und Aktivität unterscheiden. In Kombination mit einem oder mehreren Kultur-basierte Techniken kann die Lebensfähigkeit der bestimmte Teilmengen von Mikroorganismen noch bestimmt werden.
Grundsätze
Die grundlegende Prämisse der PCR ist, wiederholte Zyklen der sequentiellen Temperaturschwankungen um exponentielle Amplifikation von DNA zu erreichen. Die DNA-Synthese erfolgt durch DNA-Polymerase Enzyme, die von Bakterien leben in heißen Quellen, wie z. B. Thermus Aquaticus (Taq) bezogen werden. Diese Polymerasen sind hitzestabil, so dass sie die hohen Temperaturen während PCR verwendet.
Die Zielsequenz, bekannt als der Amplifikate wird aus der DNA-Vorlage mit zwei kurzen Abschnitten von Nukleotiden, bekannt als "Primer" verstärkt. Aufgrund der hohen Spezifität der komplementäre Nukleinsäure-Bindung erlauben die Primer für die gezielte Verstärkung der sehr spezifischen Reihenfolgen von Interesse. Durch die Gestaltung Zündkapseln, die nur eine eindeutige Sequenz (oder eine einzigartige Kombination von Sequenzen) aus dem Organismus des Interesses verstärken werden, kann PCR verwendet werden, differentiell auf das Vorhandensein des Organismus DNA unter allen präsentieren die genetischen Materials in einer komplexen Umwelt Probe zu erkennen.
Um PCR durchzuführen, wird eine Maschine bekannt als ein Thermocycler verwendet, um automatisch durchlaufen die unterschiedlichen Temperaturen, die für die Reaktion benötigt. Jeder Zyklus gliedert sich in drei Phasen. Die zuerst bekannt als "Denaturierung", befindet sich in der Regel über 92 ° C und dauert ca. 30 S. Denaturierung wird verwendet, um DNA-Moleküle in Einzelstränge, erlauben die Verstärkung Reaktion auf gehen zu brechen.
Die zweite Phase, "Glühen", ist 2-3 ° C unterhalb der unteren der Schmelztemperatur der beiden Primer, in der Regel zwischen 50-65 ° C, und auch hält, was ca. 30 S. schmelzende Temperatur ist die Temperatur, bei der 50 % der doppelsträngigen DNA in Einzelstränge getrennt haben, und so glühen Schritt ermöglicht die Primer binden an ihre Ziel-Sites in der DNA-Vorlage, festgelegt.
Die dritte Phase eines PCR-Zyklus ist "Dehnung" oder "Erweiterung", wenn die DNA-Polymerase an den Primer-Vorlage-Duplex bindet und katalysiert die Synthese des Produkts. Bei 72 ° C für die Taq-Polymerase, die Dauer dieser Phase hängt die Länge der Amplifikate, in der Regel 30 s / 500 bp. Nach jedem Zyklus die amplifizierte DNA ist wieder einmal denaturiert und dient als eine neue Vorlage führt zu einem exponentiellen Anstieg in Höhe von PCR-Produkte.
Sobald die Reaktion abgeschlossen ist, können die PCR-Produkte nach Größe auf einem "Gel" Polymer Agarose, auch als Elektrophorese bezeichnet in der Regel aus gelöst werden. Ein elektrisches Feld wird über das Gel angewendet, und die negativen Ladungen in das Rückgrat der DNA-Moleküle veranlassen, gegen positive Ende des Feldes zu migrieren. Im Allgemeinen werden lineare DNA-Moleküle, die größer sind, durch die Gelmatrix Reisen länger dauern.
Verfahren
1. die Probenahme
- Boden mit einer Schnecke zu sammeln oder Schaufel bis zu einer bestimmten Tiefe. Wenn Boden von der Rhizosphäre (die schmale Region des Bodens rund um und von Pflanzenwurzeln und ihre damit verbundenen Mikroben beeinflusst) sammeln, nur sammeln Sie direkt von rund um die Wurzeln der Pflanzen durch schlagen der Erde aus in ein Fass Sammlung.
- Wasserprobe durch eine sterile Plastikflasche ins Wasser eintauchen, halten Sie das Ende des DIP Stick zu sammeln.
(2) Nukleinsäuren Gewinnung und Vorbereitung
- Organismen und Viren aus der Probe zu sammeln, und DNA und RNA extrahieren. Einzelheiten hierzu finden Sie in der Jupiter Wissenschaftsbildung video-on-Gemeinschaft Nukleinsäure-Extraktion.
- Speichern Sie die extrahierte DNA in beschrifteten Microfuge Röhren. Wenn die DNA über Nacht oder für längere Zeit gelagert werden muss, bei-20 ° C frieren Sie ein und tauen Sie der Rohre bei Raumtemperatur, wenn Sie bereit sind für den Einsatz auf.
- Wenn das genetische Material untersucht werden ist RNA (ob es das Genom von RNA-Viren oder transkribierten RNA des zellulären Organismen ist), führen reversen Transkription (RT) auf die Probe um komplementäre DNA (cDNA) vor der PCR zu erstellen. Einzelheiten hierzu finden Sie in der Jupiter Wissenschaftsbildung video auf RT-PCR.
(3) Polymerase-Kettenreaktion
- Das PCR-Enzym (z.B. Taq Polymerase) auf Eis legen und andere Reagenzien (PCR-Puffer, dNTPs, Primer) in einer dafür vorgesehenen "sauber" Kapuze bei Raumtemperatur auftauen. Das Enzym ist bei-20 ° C gelagert, aber nie friert. Es ist temperaturempfindlich, und muss so gehalten werden, Cool und seine Exposition auf Umgebungstemperatur minimiert
- Berechnen Sie das Volumen jedes Reagens benötigt, um ein "master-Mix" alle Reagenzien, die konstant unter allen Reaktionen (Tabelle 1) sind zu machen. Stellen Sie sicher, positiv (z.B. eine Vorlage bekannt, dass die Zielregion enthalten) entfallen und negativ (z.B. keine Vorlage) Kontrollen in den Berechnungen. Das Endvolumen zu pipettieren Fehler entfallen fügen Sie zusätzlichen 10 % hinzu. Grundierung Volumen abhängig von Assays für die spezifische Organismen; beziehen sich auf Fachliteratur für die entsprechenden Werte.
- Mit einer Low-Bindung Microfuge Schlauch, der das Anhaften von Reagenz-Moleküle die Kunststoffwände minimiert wird, fügen Sie die berechneten Mengen jedes Reagens zu den master-Mix zu montieren. Sanft Wirbel und Zentrifugieren jedes Reagenz vor dem hinzufügen. Sobald der Master mix ist bereit, Vortex mischen und sammeln durch Zentrifugation
- 8-Röhre PCR-Streifen vorzubereiten. Benennen einer Röhre für jede Probe, positive und negative Kontrollen einschließlich
- Das entsprechende Volumen der PCR Mischung in jedem Röhrchen des Streifens zu verzichten
- Fügen Sie das entsprechende Volumen der DNA Schablone aus der Proben, sowie 5 μL der positiven Vorlage und 5 μL der molekularen Grade Wasser als Negativkontrolle, in die jeweiligen PCR-Röhrchen.
- Setzen Sie die Kappe fest auf dem 8-Röhre Streifen und Zentrifuge für ein paar Sekunden mit einem minicentrifuge
- Platz 8-Röhre Streifen in einem thermocycler
- Legen Sie das entsprechende PCR-Programm auf den Thermocycler ausgeführt. Ein typisches Programm besteht aus den folgenden
- Denaturierung bei 94 ° C für 3 Minuten.
- 30-40 Zyklen der Verstärkung: Denaturierung bei 94 ° C für 30 s, Glühen bei Grundierung-spezifische Temperatur (in der Regel zwischen 50 – 60 ° C) für 30 s und Erweiterung bei 72 ° C für 30 s / 500 bp.
- Letzte Verlängerung bei 72 ° C für ca. 7-10 min.
(4) Agarose-Gel-Vorbereitung
- Basierend auf der gewünschten Gel Volumen und Gel-Konzentration (Tabelle 2), wiegen Sie die entsprechende Menge Agarose-Pulver in einer 125-mL-Erlenmeyerkolben.
- Fügen Sie das entsprechende Volumen der Gel-laufen-Puffer in den Kolben, und wirbeln Sie den Kolben mit der hand.
- Erhitzen Sie die Puffer-Agarose-Mischung in der Mikrowelle bei hoher Leistung für 1 min.
- Entfernen Sie Kolben aus der Mikrowelle und von hand schwenken Sie, um sicherzustellen, dass die Agarose aufgelöst hat. Wenn die Agarose nicht vollständig aufgelöst hat, wiederholen Sie die Mikrowelle in Schritten von 30 s.
- Kühlen Sie nach der Sicherung fest der Kappe auf der Flasche es auf 50 ° C durch Drehen unter fließendem kalten Wasser ab.
- Die Agarose-Mischung mit einer Micropippette EtBr vorgesehenen fügen Sie 1 μL der Interkalation Bromid (EtBr hinzu). EtBr ist ein Farbstoff, der bindet doppelsträngiger Nukleinsäuren und fluoresziert Orange mit UV-Licht beleuchtet. Beachten Sie, dass EtBr potenziell krebserregend ist, so dass persönlicher Schutzausrüstung (Schutzbrille, Kittel, beständige EtBr Handschuhe) getragen werden muss.
- Gießen Sie die geschmolzene Gel in einer Elektrophorese Gel Casting Fach. Stellen Sie sicher, dass keine Luftblasen in der Agarose gefangen sind. Legen Sie einen Kamm in das Gel und sicher spannen. Warten Sie ca. 20-30 min für das Gel zu verfestigen.
- Sobald das Gel verfestigt hat, entfernen Sie den Kamm vorsichtig, ohne dass Risse in das Gel. Der Kamm schafft Brunnen in das Gel für die Verladung der Proben.
5. die Gelelektrophorese
- Legen Sie die erstarrten Agarosegel in der Elektrophorese-Kammer.
- Fügen Sie den entsprechenden laufenden Puffer in die Kammer, bis das Gel kaum eingetaucht ist.
- Auf ein Stück Parafilm pipette Flecken Farbstoff Konzentrat zu laden. Auch eine DNA-Leiter einer geeigneten Palette für die erwarteten Größen der PCR-Produkte. Alternativ verwenden Sie einen neuen Satz Microfuge Röhren, eine für jede Probe.
- Nach Abschluss die PCR rufen Sie ab, dass 8-Röhre Streifen aus der Thermocycler und Zentrifugieren Sie kurz um Kondensate zu sammeln. Fügen Sie einem entsprechenden Volumen des DNA-Produkt laden Farbstoff und Pipette rauf und runter zu mischen hinzu. Zum Beispiel ist 8 µL der Probe zu einem Endvolumen von 10 µL, mit dem Farbstoff mit 1 X 2 µL 10 x laden Farbstoff beigemischt.
- Pipette jeder Farbstoff-Sample-Mischung in die dafür vorgesehenen Vertiefungen im Agarosegel, darauf achten, nicht zu die Brunnen zu durchstechen.
- Schließen Sie die Elektroden an die Elektrophorese-Kammer. DNA ist negativ geladen, so dass es "" in Richtung der positiven Elektrode läuft. Daher verbinden Sie die positive Elektrode auf die gegenüberliegende Seite der Kammer, wo die Brunnen geladen wurden. Legen Sie die Stromversorgung auf eine Spannung für das Puffersystem und Größe der Kammer Gel geeignet, und es laufen. Kleine Bläschen soll an den Seiten der Kammer bewegen, wenn die Elektrophorese ordnungsgemäß verläuft sichtbar sein.
- Sobald Farbstoff vorne unten das Gel weit genug fortgeschritten ist, schalten Sie die Stromversorgung. Vorsichtig das Gel in die Transilluminator oder visuelle Imager zu transportieren und drehen auf die UV-Licht die DNA-Bänder auf dem Gel zu visualisieren.
- Analysieren Sie die Bandgröße und Positionen auf dem Gel. Vergleichen Sie die Band-Positionen der Proben, die positive Kontrolle, ob DNA aus dem Organismus des Interesses in der Probe (Abbildung 1) vorhanden ist.
| Komponente | Volumen pro Röhre (μL) | Volumen für 5 Tuben (μL) | Endkonzentration |
| 10 X Ex Taq Puffer | 5.0 | 25 | 1 x |
| 2,5 mM dNTPs | 4.0 | 20 | 0,2 mM |
| Forward Primer * | 2.0 | 10 | 400 nM |
| Reverse Primer * | 2.0 | 10 | 400 nM |
| Molekulare H2O | 31.75 | 158.75 | - |
| Ex- Taq | 0,25 | 1.25 | 2.5 U |
| PCR Mischung | 45 | 225 |
Tabelle 1. Reagenz Bände für die PCR master Mix. * Primer Volumen variieren je nach Organismus Assay. Die Lautstärke der molekularen Grade Wasser um die 45 μL Endvolumen zu machen. Mengen von anderen Komponenten sollten nicht variieren.
| Empfohlene % Agarose | Optimale Auflösung für Lineare DNA-Fragmente (Basenpaare) |
| 0,5 | 1.000-30.000 |
| 0,7 | 800-12.000 |
| 1.0 | 500-10.000 |
| 1.2 | 400-7.000 |
| 1.5 | 200-3.000 |
| 2.0 | 50-2,00 |
Tabelle 2. DNA-Fragment Größenbereiche optimal gelöst, indem verschiedene Agarose-Gel-Prozentsätze.
Ergebnisse
In Abbildung 1dient die DNA-Leiter (Spur 1) als Referenz für die Größe und die ungefähre Konzentration für Bands die PCR-Produkte. Die negative-Kontrolle (Spur 2) enthält kein genetischen Materials, während die positive-Kontrolle (Spur 3) bekannt, dass das Ziel DNA an Größe und Position des Ziels Bänder enthalten Vorlagen verstärkt wird. Muster 4, 6, 8 und 9 zeigen Ähnliches Band Muster als die positive Kontrolle, daher darauf hinweist, dass diese Proben das Ziel genetische Material enthalten. Dabei ist zu entnehmen, dass der Organismus in die Umgebungen vorhanden ist diese Proben entnommen wurden.
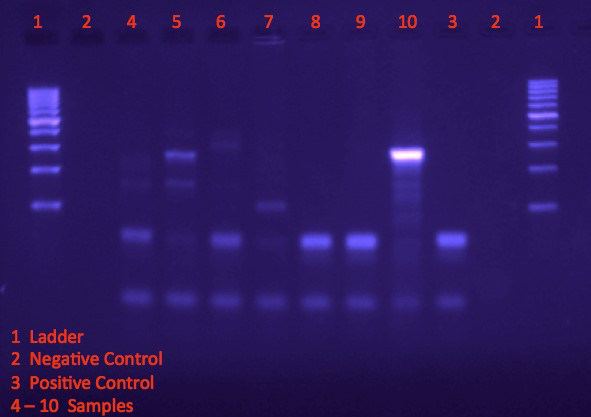
Abbildung 1. Bands auf Agarosegel-Elektrophorese nach zu visualisieren.
Anwendung und Zusammenfassung
PCR kann eingesetzt werden, um das Vorhandensein oder Fehlen von Krankheitserregern in der Umwelt schnell zu ermitteln. Zum Beispiel werden Primer spezifisch für die Gehirn-Essen Amöbe Naegleria Fowleri, DNA zu verstärken und starken Bands auf einem Gel zu produzieren, wenn der Organismus in einer Probe vorhanden ist. Wenn ein einziger Organismus nicht das Hauptinteresse, sondern eher die Gene, die Toxinproduktion aus einer Vielzahl von Organismen zugeordnet ist, kann auch PCR verwendet werden, um das Vorhandensein oder fehlen diese spezifischen genetischen Materials festzustellen.
PCR kann auch als Bestätigungsverfahren verwendet werden, beim Umweltschutz Mikroben im Labor zu analysieren. Wenn eine Kultur-Methode zwischen bestimmten Organismen, die in einer ökologischen Probe vorhanden sind unterscheiden kann, verwendet PCR vielleicht speziell zwischen den Kandidaten Mikroben zu unterscheiden.
Konventionelle PCR kann auf verschiedene Weise zu bestimmten experimentellen Zwecken geändert werden. PCR kann verwendet werden um einsträngige RNA Vorlagen zu analysieren durch Kopplung mit einer reversen Transkription Schritt (RT-PCR). Darüber hinaus eine Bestimmung der Anwesenheit und Abwesenheit kann quantitative PCR (qPCR) Messung der Konzentration für spezifische DNA von Interesse.
pringen zu...
Videos aus dieser Sammlung:

Now Playing
Erkennen von ökologischen Mikroorganismen mit der Polymerase-Kettenreaktion und Gelelektrophorese
Environmental Microbiology
44.5K Ansichten

Bestimmung des Feuchtigkeitsgehalts im Boden
Environmental Microbiology
359.0K Ansichten

Aseptische Techniken in den Umweltwissenschaften
Environmental Microbiology
126.3K Ansichten

Gramfärbung von Bakterien aus Umweltquellen
Environmental Microbiology
100.0K Ansichten

Visualisierung von Bodenmikroorganismen mit dem Contact Slide Assay und Mikroskopie
Environmental Microbiology
42.1K Ansichten

Filamentöse Pilze
Environmental Microbiology
57.2K Ansichten

Extraktion von DNA aus Bakterienkolonien
Environmental Microbiology
28.8K Ansichten

RNA-Analyse von Umweltproben mittels RT-PCR
Environmental Microbiology
40.3K Ansichten

Quantifizierung der ökologischen Mikroorganismen und Viren mit qPCR
Environmental Microbiology
47.8K Ansichten

Wasserqualitätsanalyse über Indikatororganismen
Environmental Microbiology
29.5K Ansichten

Isolierung von fäkalen Bakterien aus Wasserproben durch Filtration
Environmental Microbiology
39.2K Ansichten

Erkennung von Bakteriophagen in Umweltproben
Environmental Microbiology
40.6K Ansichten

Kultivierung und Auflisten von Bakterien aus Bodenproben
Environmental Microbiology
183.9K Ansichten

Analyse der bakteriellen Wachstumskurve und ihre Umweltanwendungen
Environmental Microbiology
295.6K Ansichten

Algenzählung mittels kultivierbarer Methodik
Environmental Microbiology
13.7K Ansichten
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten