Method Article
Im lebenden Organismus Überwachung der transkriptionellen Aktivität während des metabolischen Übergangs mit einem biolumineszierenden Reporter in Hefe
In diesem Artikel
Zusammenfassung
Dieses Protokoll verwendet einen biolumineszierenden Reporter, der Messungen der transkriptionellen Aktivität in Saccharomyces eubayanus ermöglicht, um den Übergang von Glukose zu Maltose zu überwachen, eine Echtzeitanalyse metabolischer Anpassungen zu ermöglichen und die Stammoptimierung für die industrielle Fermentation unter verschiedenen Bedingungen zu unterstützen.
Zusammenfassung
Der sequentielle Zuckerkonsum, von einer bevorzugten Zuckerquelle zu einer weniger bevorzugten Zuckerquelle, stellt eine kritische metabolische Anpassung der Hefe dar, die besonders relevant für das Überleben in schwankenden Umgebungen ist, wie sie bei der Bierfermentation zu finden sind. Zuckerübergänge sind jedoch eine Umweltvariable, die schwer vorherzusagen und zu erkennen ist und sich auf das Ergebnis der Biergärung auswirkt. Dieses Protokoll beschreibt ein in vivo System zur Überwachung der transkriptionellen Aktivierung, die mit der metabolischen Verschiebung von Glukose zu Maltose in Saccharomyces eubayanus verbunden ist, die für verschiedene wilde Saccharomyces-Hefestämme gilt.
Das System verwendet einen episomalen biolumineszenten Transkriptionsreporter für den Maltosestoffwechsel, wobei der Schwerpunkt auf MAL32 liegt, da er eine gute Auslesung für metabolische Verschiebungen liefert, wie sie in S. cerevisiae untersucht wurden. Zu diesem Zweck wurden Hefestämme mit Plasmiden transformiert, die die MAL32-regulatorische Region von S. eubayanus enthielten, wobei die Expression eines Gens kontrolliert wurde, das für eine destabilisierte Version der Glühwürmchen-Luciferase1 kodiert, und eines Hygromycin-Resistenzgens, das ausschließlich während der Transformation verwendet wurde, um den Plasmiderwerb sicherzustellen. Nach der Selektion können transformierte Hefezellen unter nicht-selektiven Bedingungen kultiviert werden, da das episomale Plasmid unter Kulturbedingungen bis zu 7 Tage stabil bleibt.
Dieses System wurde in Mikrofermentationsassays unter einer komplexen Zuckerumgebung validiert und bestätigte die Wirksamkeit des Luciferase-Reporters bei der Information über metabolische Übergänge. Die Proben wurden regelmäßig entnommen und mit einem Luminometer analysiert, was einen kontinuierlichen Einblick in die Reaktionen der Hefen ermöglichte. Obwohl dieses Protokoll breit anwendbar ist, ist es besonders wertvoll für die Beurteilung der Hefeleistung unter Fermentationsbedingungen, bei denen metabolische Veränderungen eine erhebliche Herausforderung darstellen. Darüber hinaus kann diese Methodik durch die Auswahl alternativer Promotoren angepasst werden, um ein breiteres Spektrum von Reaktionen auf Umweltveränderungen zu untersuchen und so die Charakterisierung und Optimierung von Wildhefestämmen für verschiedene industrielle Anwendungen zu ermöglichen.
Einleitung
Mikroorganismen wie Hefen müssen sich ständig an dynamische Umweltbedingungen anpassen, um fit zu bleiben und zu überleben1. Diese Anpassungen beinhalten oft komplexe genregulatorische Schaltkreise, die mehrere extrazelluläre Signale integrieren, um präzise metabolische Reaktionen zu orchestrieren 2,3. In industriellen Umgebungen ist die Effizienz dieser Stoffwechselübergänge von entscheidender Bedeutung, insbesondere bei Fermentationsprozessen, bei denen Störungen zu suboptimalen Ausbeuten oder unvollständigen Fermentationen führen können3. Eine zentrale metabolische Herausforderung, die es zu bewältigen gilt, besteht darin, dass Zellen von einer bevorzugten zu einer sekundären Kohlenstoffquelle übergehen, wie z. B. die Verschiebung von Glukose zu Maltose. Dieser Prozess leitet eine Verzögerungsphase ein, in der Gene, die für den Stoffwechsel der sekundären Kohlenstoffquellen benötigt werden, unterdrückt werden, was eine Wiederaufnahme des Wachstums ermöglicht 4,5.
Beim Brauen müssen Saccharomyces-Hefen effizient vom Glukose- in den Maltosestoffwechsel übergehen. Insbesondere S. eubayanus, die kältetolerante Elternart der Lagerhefen, weist eine erhebliche phänotypische Variabilität in ihrer Fähigkeit auf, sich an solche Übergänge anzupassen6. Wildisolate, wie z. B. aus Patagonien, weisen im Vergleich zu domestizierten Stämmen, die aufgrund ihrer optimierten Fermentationskapazitäten ausgewählt wurden, häufig verlängerte Verzögerungsphasen und einen langsameren Maltoseverbrauch auf 7,8. Während domestizierte Stämme sich an die effiziente Fermentation von Mischzuckerumgebungen angepasst haben, weisen wilde Stämme oft einen langsameren metabolischen Übergang auf, was möglicherweise auf eine stärkere Glukoseunterdrückung und eine variable Regulation des MAL-Locuszurückzuführen ist 6,9.
Diese Studie nutzt die natürliche Variabilität von S. eubayanus als Modell, um metabolische Anpassungen unter Glukose-zu-Maltose-Bedingungen zu untersuchen, indem ein episomaler destabilisierter Luciferase-Reporter genutzt wird, um die Genexpression in vivo zu überwachen, indem Lumineszenzverfolgt wird 1. Der ausgewählte Reporter MAL32 kodiert für ein Maltase-Protein, ein zentrales Enzym für den Maltose-Katabolismus während des Übergangs von Glukose zu Maltose10,11. Bemerkenswert ist, dass der MAL32-Promotor einen erfolgreichen Marker für die Beurteilung der Induktion des Maltosestoffwechsels nach Glukosemangel darstellt12. Durch die Einbeziehung dieses Reportersystems wollten wir stammspezifische adaptive Mechanismen aufklären und potenzielle Ziele für die Optimierung der Fermentationsleistung identifizieren. Darüber hinaus kann dieses Protokoll über das Brauen hinaus erweitert werden und bietet Anwendungen in der Biotechnologie und in Umweltstudien, bei denen komplexe Zuckerumgebungen eine wichtige Rolle spielen. Das Verständnis der genetischen und regulatorischen Determinanten fluktuanter Umweltreaktionen in S. eubayanus erweitert unser Wissen über die Hefephysiologie und unterstützt die Entwicklung robuster Stämme für verschiedene Industrie- und Forschungsanwendungen.
Protokoll
1. Konstruktion episomaler Reporter
HINWEIS: Wir wählten eine Reporter-Regulationsregion auf der Grundlage der Hefeliteratur aus, um das episomale Plasmid zur Überwachung des Maltoseverbrauchszu konstruieren 6,11,12. Der Promotor des Kandidaten-Reportergens wurde definiert als die regulatorische Sequenz unmittelbar stromaufwärts vom ORF-Kandidaten-Kandidaten bis zu dem Nukleotid, das den angrenzenden stromaufwärts gelegenen ORF flankiert. Diese Region wurde aus der genomischen DNA von S. eubayanus CBS12357T Referenzstamm10 amplifiziert. Dieser Ansatz gewährleistet die High-Fidelity-Konstruktion von episomalen Plasmiden, die für nachgelagerte Anwendungen geeignet sind, einschließlich der Untersuchung anderer interessanter Bedingungen in Hefe.
- Entwerfen Sie die DNA-Fragmente und überlappenden Primer so, dass sie 30 Nukleotidpaarungssequenzen zwischen den Fragmenten enthalten, um einen nahtlosen Zusammenbau durch rekombinatorische Klonierung von Hefenzu ermöglichen 13 (Abbildung 1). Siehe Ergänzende Tabelle S1 für Primersequenzen.
- Um Mutationen zu vermeiden, amplifizieren Sie die Promotorregion (siehe oben) sowie die destabilisierte Luciferase (LUC-PEST) und die hphMX-Kassetten 1 mit einer High-Fidelity-DNA-Polymerase (Einzelheiten zum Protokoll finden Sie beim Polymerasehersteller). Stellen Sie den Thermocycler für 10 Minuten auf 98 °C für die erste Denaturierung, gefolgt von 30 Zyklen mit 98 °C Denaturierung, 60 °C Glühen und 72 °C Verlängerung; Vervollständigen Sie das Protokoll mit einer abschließenden Verlängerung bei 72 °C für 10 Minuten.
HINWEIS: Bei der Verwendung von Hefe-Shuttle-Plasmiden als Templates für die PCR wird empfohlen, Amplikonreaktionen einem DpnI-Verdau zu unterziehen (siehe Protokoll des Enzymherstellers), um alle Spuren von Template-Plasmid zu beseitigen, die die Hefetransformation beeinträchtigen könnten. - Verdauen Sie das Rückgrat des pRS426-Plasmids14 mit EcoRI- und XhoI-Restriktionsenzymen, um den Vektor zu linearisieren, ihn an der multiplen Klonierungsstelle zu schneiden und für die Rekombination vorzubereiten (siehe Protokoll des Enzymherstellers).
- Mischen Sie das linearisierte pRS426-Rückgrat mit den amplifizierten DNA-Fragmenten und transformieren Sie das Gemisch in Saccharomyces cerevisiae BY4741 unter Verwendung der Hefe-Rekombinationskolonierung13,15, damit die homologe Rekombinationsmaschinerie der Hefe das Plasmidkonstrukt in vivo zusammensetzen kann.
- Die transformierte Hefe wird auf ein synthetisches vollständiges (SC) Medium ohne Uracil (SC-URA) aufgebracht, um auxotrophe Transformanten auszuwählen, die das neuartige konstruierte Vektor pRS426-MAL32-LUC-hphMX-Plasmid enthalten.
- Extrahieren Sie Plasmide aus Hefe mit einem Hefe-Mini-Prep-Kit gemäß den Anweisungen des Herstellers und wandeln Sie das extrahierte Plasmid in Escherichia coli DH5α um, um das Konstrukt für weitere Analysen zu amplifizieren.
- Screening von Bakterienkolonien mittels PCR, um den korrekten Zusammenbau der Konstrukte zu bestätigen.
- Reinigen Sie die Plasmid-DNA mit einem Standard-Plasmid-Aufreinigungskit und führen Sie eine Sanger-Sequenzierung durch, um die Integrität und Sequenz der assemblierten Plasmide zu überprüfen.
2. Transformation von Hefestämmen
HINWEIS: Das Hefeumwandlungsprotokoll wurde von einer zuvor etablierten Methode für S. eubayanus16 angepasst und erfolgreich auf andere Saccharomyces-Spezies und fermentative Hefestämme angewendet. Dieses Protokoll wurde von der traditionellen Hefetransformationsmethode aus dem Gietz Lab15 abgeleitet. Dieser Ansatz ermöglicht eine effiziente Plasmidintegration und -selektion unter verschiedenen experimentellen Bedingungen und bietet eine robuste und flexible Methode zur Transformation verschiedener Hefestämme. Es gewährleistet eine zuverlässige Selektion und Aufrechterhaltung des episomalen Plasmids unter Hygromycindruck.
- Eine einzelne Kolonie frisch gewachsener Hefe in 5 mL YPD (1 % w/v Hefeextrakt, 2 % w/v Pepton, 2 % w/v Glukose) bei 20 °C bei 200 U/min Rührwerk über Nacht vorkultivieren.
- Die Kultur wird auf einen OD620 von 0,2 in frischem YPD-Medium verdünnt und bei 20 °C unter Rühren bei 200 U/min bis zur Mitte der logarithmischen Phase (OD620 0,4-0,6) inkubiert. Je nach Hefestamm dauert dies typischerweise 3-4 h.
- Ernten Sie die Zellen durch Zentrifugation bei 21.380 × g für 1 min bei Raumtemperatur, entsorgen Sie den Überstand und waschen Sie das Pellet 3x mit sterilem Wasser.
- Die gewaschenen Zellen werden in 100 μl 0,1 M Lithiumacetat resuspendiert, wie in Schritt 2.3 beschrieben zentrifugiert und der Vorgang 2x wiederholt.
- Fügen Sie den Zellen in dieser Reihenfolge die folgenden Komponenten hinzu: 240 μl 50 % w/v PEG-4000, 35 μl 1 M LiAc, 5 μl Plasmid-DNA (100 ng/μl), 20 μl einzelsträngige Träger-DNA (ssDNA), die zuvor 10 Minuten lang auf 98 °C erhitzt wurde. Vorsichtig mischen, um die Transformationsmischung zu homogenisieren.
- Die Mischung wird 30 Minuten lang bei 20 °C inkubiert, gefolgt von einem Hitzeschock bei 34 °C für 55 Minuten. Nach dem Hitzeschock 38 μl 100 % Ethanol auf eine Endkonzentration von fast 0,1 % zugeben und weitere 5 Minuten bei 34 °C inkubieren.
HINWEIS: Die in diesem Protokoll verwendeten Temperaturen sind für Saccharomyces eubayanus optimiert, einen kryotoleranten Stamm, der aus gemäßigten Wäldern isoliert wurde.
Die Verwendung von 100% Ethanol ist erforderlich, um zellulären Stress zu induzieren und die Effizienz des Transformationsprotokolls zu erhöhen. - Geben Sie 600 μl YPD zu den transformierten Zellen, zentrifugieren Sie wie in Schritt 2.3 und verwerfen Sie den Überstand.
- Das Zellpellet wird in 600 μl frischem YPD resuspendiert und die Suspension ohne Rühren über Nacht bei 4 °C inkubiert, um eine Wiederherstellung zu ermöglichen.
- Die transformierten Zellen werden auf YPD-Agar mit 200 μg/ml Hygromycin gefüllt und bei 20 °C-30 °C für 48-72 h inkubiert.
- Wählen Sie 10-12 Kolonien aus den Transformationsplatten aus und streichen Sie sie erneut auf YPD-Agar mit 200 μg/ml Hygromycin, um das Vorhandensein des Plasmids sicherzustellen und ein gemischtes Pflaster der ausgewählten Kolonien für die weitere Verwendung zu erzeugen.
3. Validierung der Lumineszenz
HINWEIS: Um die Funktionalität der lumineszierenden Reporter zu validieren, wurden transformierte Stämme unter Bedingungen getestet, die eine differentielle Expression des Luciferase-Reporters induzieren. Diese schrittweise Validierung ermöglicht die Bewertung der Reporterfunktionalität unter schwankenden Zuckerbedingungen, wobei die Robustheit von Lumineszenz-Assays genutzt wird, um metabolische Reaktionen in Echtzeit zu erfassen.
- Entnehmen Sie eine Probe aus einem transformierten Hefestammpflaster und inokulieren Sie sie in YP-Medium (1 % w/v Hefeextrakt, 2 % w/v Pepton) mit 5 % w/v Glucose und 200 μM Hygromycin. 24 h lang bei 25 °C ohne Schütteln inkubieren. Die Kulturen werden in frischem Medium in einer Verdünnung von 1:10 aufgefrischt und unter den gleichen Bedingungen weitere 24 Stunden inkubiert.
- Waschen Sie die Zellen mit YP-Medium ohne Zucker und inokulieren Sie sie in einer Verdünnung von 1:10 in das Testmedium. Das Testmedium wird mit Luciferin bis zu einer Endkonzentration von 3 mM ergänzt und enthält 200 μM Hygromycin, um die Plasmidstabilität zu erhalten.
- Verwenden Sie für Wachstumstests mit Mischzucker eine Glukose-Maltose-Matrix mit den folgenden Konzentrationen (% w/v): Glukose (0 %, 0,5 %, 1 %, 2 %) und Maltose (0 %, 2 %, 5 %, 10 %, 20 %, 30 %) in YP-Medium. Jede Bedingung wird mit Luciferin (3 mM Endkonzentration) und 200 μM Hygromycin ergänzt. Geben Sie 200 μl Kultur in jede Vertiefung einer 96-Well-Platte (Abbildung 2).
- Messen Sie die Lumineszenz und OD620 alle 30 Minuten für 72 Stunden mit einem Luminometer-Plattenlesegerät, ohne Dämpfung und mit einer Integrationszeit von 1 s, um eine kontinuierliche Überwachung der Reporteraktivität und der Zelldichte zu gewährleisten.
4. Fermentationsprobenahme und Lumineszenzüberwachung
HINWEIS: Transformierte Stämme wurden kontrollierten Mikrofermentationsbedingungen unterzogen, um die Lumineszenzaktivierung während der Fermentation zu bewerten. Dies ermöglicht den Vergleich der Lumineszenzaktivierung unter verschiedenen Fermentationsbedingungen und gibt Einblicke in die Stoffwechselreaktionen der Hefe während längerer Fermentationsperioden.
- Malzextraktmedium (12 °Plato) herstellen, indem Malzextrakt in Wasser aufgelöst und bei 100 °C für 20 min sterilisiert wird. Lassen Sie das Medium vor der Inokulation auf Raumtemperatur abkühlen.
- Vorkultivierte transformierte Hefestämme in 5 mL YPD (1 % w/v Hefeextrakt, 2 % w/v Pepton, 2 % w/v Glucose) bei 20 °C mit Rühren bei 200 U/min für 24 h. Die Vorkultur wird auf 50 ml Malzextraktmedium umgefüllt. Inkubieren Sie die Kultur bei 20 °C und rühren Sie sie bei 200 U/min für 24 h.
- Ernten Sie die Zellen durch Zentrifugation bei 21.380 × g für 1 min bei Raumtemperatur und resuspendieren Sie sie, um dreifache 50 mL Mikrofermentationen in 12 °Plato Malzextrakt Medium9 vorzubereiten. Um die Fermentationsleistung zu verbessern, ergänzen Sie das Medium mit 0,3 mg/L ZnCl2.
- Um eine konsistente Zelldichte über alle Replikate hinweg zu gewährleisten, berechnen Sie das Inokulumvolumen mit der folgenden Formel:
Inokulumvolumen = (1,5 × 106) × Volumen (ml) × Grad Plato - Das vorbereitete Medium wird beimpft und die Kulturen werden 14 Tage lang bei 20 °C ohne Schütteln inkubiert. Überwachen Sie den Fermentationsfortschritt, indem Sie täglich den CO2 - Verlust aufzeichnen. Wiegen Sie die Gefäße jeden Tag gleichzeitig, um den kumulativen Gewichtsverlust als Indikator für die CO2 -Produktion zu messen.
- Für die Lumineszenzüberwachung nehmen Sie regelmäßig eine Probe von 200 μl von jeder Mikrofermentation. Geben Sie Luciferin zu den Proben, um eine Endkonzentration von 3 mM zu erreichen, und messen Sie die Lumineszenz und OD620 mit einem Luminometer-Plattenlesegerät ohne Dämpfung und 1 s Integrationszeit. Führen Sie Messungen in definierten Intervallen während der gesamten Fermentationszeit durch (Abbildung 3).
Ergebnisse
Die folgenden Ergebnisse zeigen die Eignung des neu konstruierten lumineszierenden Reporters zur Überwachung des Glukose-zu-Maltose-Übergangs in Hefezellen in einem fermentativen Prozess. Die Reporterplasmide werden zunächst unter Verwendung der Hefe-Rekombinationskolonierung13 assembliert, um episomale Reporterkonstrukte zu erzeugen. Dieser Prozess erfordert eine Überlappung der Nukleotidsequenz von mindestens 30 Nukleotiden zwischen den verschiedenen Amplikons, die alle in Abbildung 1 dargestellt sind. Die regulatorischen Regionen, die 858 bp stromaufwärts der ATG-Genstartstelle für MAL32 umfassen, wurden aus genomischer DNA mit dem S. eubayanus-Stamm CBS12357T7 amplifiziert. Das Genom von S. eubayanus enthält zwei funktionelle MAL32-Gene, die in vollständigen MAL-Loci auf den Chromosomen V und XVI11 kodiert sind. Basierend auf der Literatur11 wählten wir den Promotor des letzteren für unseren episomalen Reporter. Die destabilisierten Luciferase-ORF- und hphMX-Kassetten wurden aus den zuvor berichteten Plasmiden pRS426-P TDH3-Luc und pRS426-mCherry 1 mit einer Amplikongröße von 1.901 bzw. 1.951 bp amplifiziert. Diese Amplikons erforderten einen zusätzlichen Schritt, bei dem die DpnI-Enzymrestriktion verwendet wurde, um jede Spur von Template-Plasmid zu verdauen. Alle diese Fragmente wurden mit dem offenen pRS426-Plasmid kombiniert, um einen episomalen Reporter für MAL32 zu erhalten, der als pRS426-PMAL32-LUC-hphMX bekannt ist (auf Anfrage erhältlich). Anschließend wurden drei verschiedene S. eubayanus-Stämme, CBS12357T, CL467.1 und QC18, die unter Maltosebedingungen6 ein differentielles Wachstum aufweisen, mit diesem Reporterkonstrukt transformiert. Anschließend selektierten wir erfolgreiche Transformanten über Hygromycin-Antibiotikaresistenz, um den Einbau des Plasmidreporters sicherzustellen.
Wir testeten jeden transformierten Stamm unter Glukose-zu-Maltose-Bedingungen, um die transkriptionelle Aktivität des Reporters zu validieren. Wir maßen die Aktivität des MAL32-Promotors unter einer Reihe von Glukose- und Maltosekonzentrationen (Abbildung 2). Die Luciferase-Aktivität variierte in der Signalintensität und der Timing-Reaktion zwischen den drei Stämmen (p < 0,05, ANOVA), was die unterschiedliche Aktivierung in jedem genetischen Hintergrund belegt. Zum Beispiel trat die Aktivierung des Reporters im QC18-Hintergrund nur oberhalb von 1% Glukosekonzentrationen auf. Im Gegensatz dazu zeigten die beiden phänotypisch ähnlichen Stämme, CBS12357T und CL467.1, hohe transkriptionelle Aktivierungswerte, wenn zunächst keine Glukose in den Medien vorhanden war6. Interessanterweise spiegelte das Reportersystem die differentielle transkriptionelle Aktivierung in diesen beiden genetischenHintergründen wider, wobei wir die transkriptionelle Aktivierung des Reportergens nach 6 h bzw. 24 h Wachstum unter Bedingungen ohne Glukose in CBS12357 T bzw. CL467.1 nachweisen konnten. Diese Ergebnisse zeigen, dass sich phänotypisch ähnliche Ergebnisse in der transkriptionellen Regulation zwischen den Stämmen unterscheiden können.
Um die Wirksamkeit der Reportersysteme unter Braubedingungen zu bewerten, führten wir einen Mikrofermentationsassay unter Würzebedingungen mit den drei transformierten Stämmen durch. Dazu wurden aus jeder Mikrofermentation 7 Tage lang täglich 100 μL Proben entnommen. Anschließend wurden jeweils die Luciferase-Aktivität und OD620 gemessen, um die transkriptionelle Aktivierung der metabolischen Übergangsreaktionen während der Fermentation zu bewerten (Abbildung 3). Alle drei Stämme zeigten eine robuste Lumineszenz des pRS426-PMAL32-LUC-hphMX, die in den Wildtyp-Stämmen nicht vorhanden war. Der Stamm CL467.1 zeigte die höchsten Werte und erreichte nach vier Tagen seinen Höhepunkt (p < 0,05, ANOVA). Im Gegensatz dazu erreichten sowohl CBS12357T als auch QC18 nach 24 h ihren Höhepunkt, wobei CBS12357T signifikant höhere Werte als QC18 aufwies (p < 0,05, ANOVA). Diese Ergebnisse sind vergleichbar mit denen, die unter Mikrokultivierungsbedingungen erzielt wurden (Abbildung 2), und zeigen eine höhere transkriptionelle Aktivierung in CL467.1 im Vergleich zu den beiden anderen Stämmen und eine verzögerte Promotoraktivierung in QC18. Diese Ergebnisse unterstreichen die Fähigkeit von lumineszierenden Reportern, die Genexpression als Reaktion auf metabolische Veränderungen in Hefe dynamisch zu überwachen. Dieser Ansatz stimmt gut mit früheren Studien überein, wie z. B. der Verwendung von Hefe als Chassis für die Entwicklung funktioneller Assays.
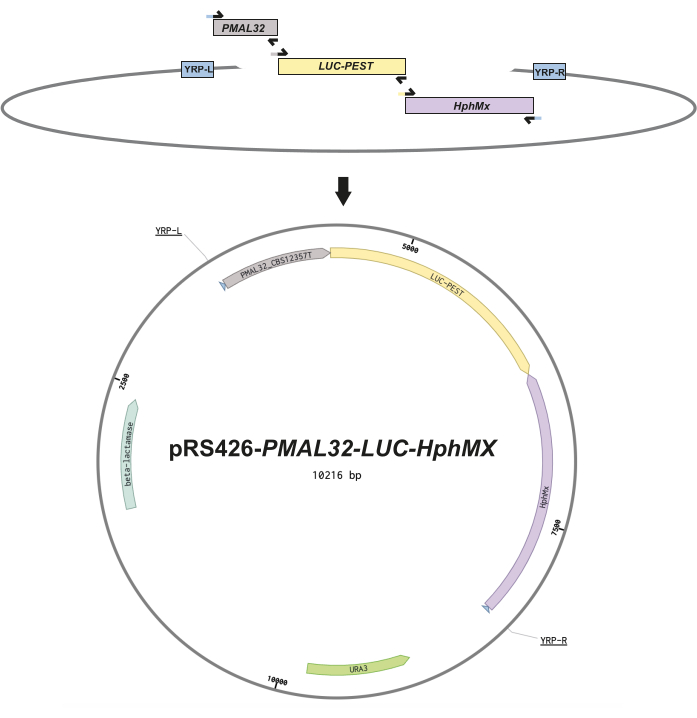
Abbildung 1: Konstruktionsdesign von episomalen Reportern. Diese schematische Abbildung veranschaulicht den Aufbau jedes Plasmids. (A) DNA-Fragmente, die die überlappenden Primer darstellen (schwarze Pfeile), die 30 homologe Nukleotidsequenzen zwischen den Fragmenten enthalten, um den nahtlosen Zusammenbau durch homologe Rekombination der Hefe zu erleichtern. (B) Vektorkarte für den episomalen Reporter unter Verwendung der MAL32-Promotorregion (PMAL32) aus dem CBS12357 T-Stamm, der destabilisierten Luciferase (LUC-PEST) und der Hygromycinkassette (hphMX) stromabwärts des LUC-PEST in umgekehrter Ausrichtung. Diese Fragmente werden in die multiplen pRS426-Klonierungsstellen eingebaut, die dem Hefe-Rekombinationsprimer links und rechts (YPR-L und YRP-R) folgen, der in der Vektorkarte angegeben ist. Abschließend werden die offenen Leserahmen für bakterielle Antibiotikaresistenz (Beta-Lactamase) und Uracil-Auxotrophie (URA3) gezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
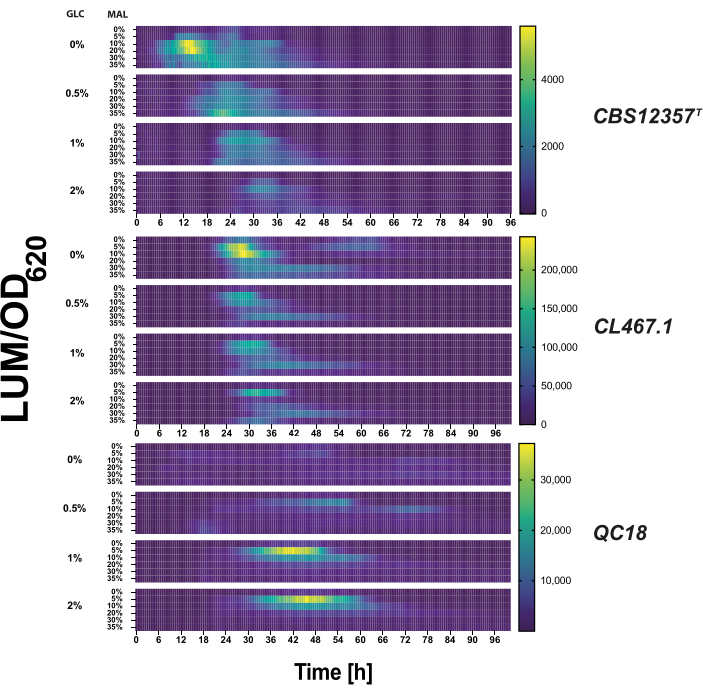
Abbildung 2: Reportertest des metabolischen Übergangs unter Mikrokultivierungsbedingungen. Unter Verwendung einer Matrix unterschiedlicher Glukose- (0%, 0,5%, 1%, 2%) und Maltose- (0%, 2%, 5%, 10%, 20%, 30%) Konzentrationen im YP-Medium untersuchten wir die pMAL32-Transkriptionsaktivierung in den Saccharomyces eubayanus-Stämmen CBS12357T (oben), CL467.1 (Mitte) und QC18 (unten). Die Luciferase-Aktivität wird in einer Heatmap als durchschnittliche Lumineszenz normalisiert durch die optische Dichte bei 620 nm (LUM/OD620) in beliebigen Einheiten für jeden transformierten Stamm in der Glukose-Maltose-Matrix für 100 h dargestellt. Die höchste Lumineszenz (oder Promotoraktivierung) wird in der Heatmap in hellem Gelb und die Inaktivierung in dunkelblau dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
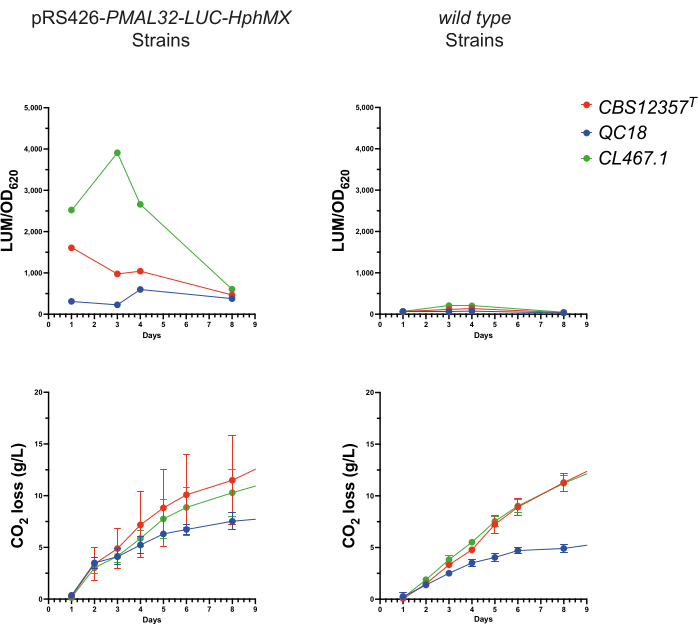
Abbildung 3: Lumineszenzüberwachung während der Fermentation. Bewertung der Leistung des pRS426-PMAL32-LUC-hphMX Reporters während eines Fermentationsprozesses. In den oberen Panels ist das Lumineszenzsignal (LUM/OD620) über eine 7-tägige Fermentationsperiode in den transformierten und den Wildtyp-Stämmen dargestellt. Die unteren Felder zeigen Diagramme des CO2 - Verlusts (g/L) während der Fermentation, die eine ähnliche Leistung zwischen beiden Gruppen zeigen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Tabelle S1: Grundierungen für die Reporterkonstruktion. Diese Tabelle enthält detaillierte Informationen zu den in dieser Studie verwendeten Primern, einschließlich ihrer Namen, Sequenzen (5' bis 3') und Längen. Die Primer wurden entwickelt, um Zielregionen bei der Konstruktion episomaler Reporter zu amplifizieren und ihre Funktionalität in Saccharomyces eubayanus zu validieren. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Diese Studie zeigt die Wirksamkeit eines episomalen biolumineszierenden Reporters zur Überwachung der transkriptionellen Aktivierung in S. eubayanus unter metabolischen Übergängen. Durch die Verwendung von MAL32 als transkriptioneller Reporter11 konnten wir wichtige metabolische Übergänge in Echtzeit verfolgen und so einen robusten Rahmen für das Verständnis stammspezifischer Anpassungen bieten. Dieser Reporter, der aufgrund seiner Rolle im Maltosestoffwechsel ausgewählt wurde, bietet deutliche Vorteile bei der Bewertung der metabolischen Flexibilität in Hefe, wie z.B. in vivo Monitoring, genaue Identifizierung von metabolischen Verschiebungen und detaillierte phänotypische Charakterisierung verschiedener genetischer Hintergründe. Die Assays unter Mischzuckerbedingungen zeigten eine signifikante Variabilität in der Reporteraktivierung zwischen den getesteten S. eubayanus-Stämmen, was die phänotypische Diversität innerhalb dieser Art unterstreicht. Diese Variabilität, die wahrscheinlich auf genetische Unterschiede in der MAL-Transkriptionsaktivierung zurückzuführen ist, unterstreicht das Potenzial wilder S. eubayanus-Stämme als Ressource zur Identifizierung von Merkmalen, die die industrielle Fermentationsleistung verbessern könnten. Bemerkenswert ist, dass S. eubayanus-Isolate aus Patagonien die größte genetische Vielfalt aufweisen, die mehrere Abstammungslinien innerhalb einer einzigen geographischen Region umfasst 6,17, was zu der beobachteten phänotypischen Variabilität beitragen könnte. Die anhaltenden metabolischen Verschiebungen, die bei einigen Stämmen beobachtet wurden, deuten auf Möglichkeiten zur Stammoptimierung hin, hauptsächlich durch genetische oder adaptive Laborevolutionsansätze. Das Verständnis dieser Reaktion ist entscheidend für die Optimierung industrieller Fermentationsprozesse.
Ein bemerkenswerter Vorteil des beschriebenen Protokolls ist seine Anpassungsfähigkeit. Das System kann so angepasst werden, dass es ein breiteres Spektrum von Transkriptionsreaktionen oder Stoffwechselwegen untersucht, indem alternative Promotoren substituiert werden, wodurch seine Anwendbarkeit über das Brauen hinaus auf andere biotechnologische Prozesse erweitert wird. Darüber hinaus gewährleistet die episomale Natur des Plasmidsystems eine stabile Expression, ohne dass eine genomische Integration erforderlich ist, was die Stammkonstruktion und die experimentellen Arbeitsabläufe vereinfacht. Die Entdeckung von S. eubayanus als kältetoleranter Elternteil der Lagerbierhefen hat Einblicke in die Domestizierung und Evolution dieser industriellen Hybriden gegeben 7,9,16. Unsere Ergebnisse unterstreichen die Nützlichkeit von S. eubayanus als Modellorganismus für die Untersuchung komplexer metabolischer Anpassungen. Seine natürliche Variabilität und seine Bedeutung für das Brauen von Lagerbier machen es zu einem idealen Kandidaten für die Untersuchung der genetischen und regulatorischen Mechanismen, die der Fermentationsleistung zugrunde liegen. Zukünftige Arbeiten könnten sich auf die Erweiterung des Spektrums der getesteten Stämme konzentrieren, indem domestizierte Stämme oder neuartige Wildstämme mit Braupotenzial einbezogen werden, um ihre metabolische Effizienz unter ähnlichen Bedingungen direkt zu vergleichen.
Obwohl dieses Protokoll zahlreiche Vorteile bietet, sollten einige Einschränkungen beachtet werden. Episomale Reporter können beispielsweise eine Variabilität in der Plasmidkopienzahl aufweisen, was sich auf die Konsistenz der Genexpressionsmessungen über Stämme hinweg auswirken kann18. Darüber hinaus können transkriptionelle Reaktionen, die in S. eubayanus beobachtet wurden, aufgrund von Unterschieden in der Promotorkompatibilität oder der zellulären Maschinerie möglicherweise nicht direkt auf andere Organismen übertragenwerden 19. Die Anpassung dieses Protokolls erfordert eine sorgfältige Kalibrierung der Wachstums- und Selektionsbedingungen, um die Plasmidstabilität zu gewährleisten und den Selektionsdruck aufrechtzuerhalten, was für das Erzielen reproduzierbarer Ergebnisse entscheidend ist18. Darüber hinaus ist die Optimierung der Transformationseffizienz und die Anpassung der Wachstumsparameter an bestimmte Stämme oder Reporter für eine erfolgreiche Implementierung unerlässlich20. Ähnliche Ansätze bei S. cerevisiae, wie z. B. die Verwendung des MAL32-Promotorsystems in Verbindung mit fluoreszierenden Markern zur Untersuchung der Transkriptionsspiegel unter Glukose- und Maltosebedingungen, unterstreichen die Relevanz und den Nutzen von Reporterwerkzeugen für die Untersuchung der transkriptionellen Aktivität und der Stoffwechselwege12. Insgesamt konzentrieren sich diese Überlegungen auf die sorgfältige Anpassung des Protokolls an verschiedene biologische Systeme unter Betonung seiner Vielseitigkeit und weitreichenden Anwendbarkeit.
Zusammenfassend lässt sich sagen, dass die Verwendung von biolumineszierenden Reportern ein leistungsfähiges Werkzeug darstellt, um die Reaktionen von Brauhefe auf Umweltveränderungen zu analysieren. Dieser Ansatz verbessert nicht nur unser Verständnis der Physiologie von S. eubayanus , sondern unterstützt auch die Entwicklung robuster Hefestämme, die auf industrielle Bedürfnisse zugeschnitten sind. Die Flexibilität und Effektivität dieses Protokolls positionieren es sowohl für die Forschung als auch für biotechnologische Anwendungen.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Diese Forschung wurde von der Agencia Nacional de Investigación y Desarrollo (ANID), der FONDECYT (1220026) und der ANID-Programa Iniciativa Científica Milenio ICN17_022 und NCN2024_040 finanziert. FM wurde unterstützt durch das ANID FONDECYT Postdoctorado Stipendium N°3220597. PQ wurde durch den ANID-Zuschuss Nr. 21201057 unterstützt. Finanzielle Unterstützung wird auch dem Centro Ciencia & Vida, FB210008, Financiamiento Basal para Centros Científicos y Tecnológicos de Excelencia de ANID zuerkannt.
Materialien
| Name | Company | Catalog Number | Comments |
| Ampicillin, sodium salt | ThermoFisher Scientific | 11593027 | |
| D-Glucose | Sigma-Aldrich | G8270 | |
| DpnI | New England Biolabs | R0176S | |
| EcoRI | New England Biolabs | R0101S | |
| Hygromycine B | Gold Biotechnology | H-270-1 | |
| L-Luciferine | Gold Biotechnology | L-127-10 | |
| Maltose monohydrate | Sigma-Aldrich | 47288 | |
| Phusion Plus PCR Master Mix | ThermoFisher Scientific | F631S | |
| Tecan Infinite 200 PRO M | Tecan | ||
| Wizard Plus SV Minipreps DNA Purirfication System | Promega | A1330 | |
| XhoI | New England Biolabs | R0146S | |
| Zymoprep Yeast Plasmid Miniprep I | Zymo Research | D2001 |
Referenzen
- Salinas, F., Rojas, V., Delgado, V., López, J., Agosin, E., Larrondo, L. F. Fungal light-oxygen-voltage domains for optogenetic control of gene expression and flocculation in yeast. mBio. 9 (4), e00626-e00718 (2018).
- Jacob, F., Monod, J. Genetic regulatory mechanisms in the synthesis of proteins. J Mol Biol. 3, 318-356 (1961).
- Perez-Samper, G., et al. The Crabtree Effect shapes the Saccharomyces cerevisiae lag phase during the switch between different carbon sources. mBio. 9 (5), e01331-e01418 (2018).
- Vermeersch, L., et al. On the duration of the microbial lag phase. Curr Genet. 65 (3), 721-727 (2019).
- New, A. M., et al. Different levels of catabolite repression optimize growth in stable and variable environments. PLoS Biol. 12 (1), e1001764 (2014).
- Molinet, J., et al. Natural variation in diauxic shift between Patagonian Saccharomyces eubayanus Strains. mSystems. 7 (6), e0064022 (2022).
- Libkind, D., et al. Microbe domestication and the identification of the wild genetic stock of lager-brewing yeast. Proc Natl Acad Sci USA. 108 (35), 14539-14544 (2011).
- Peris, D., et al. Complex ancestries of lager-brewing hybrids were shaped by standing variation in the wild yeast Saccharomyces eubayanus. PLoS Genet. 12 (7), e1006155 (2016).
- Mardones, W., et al. Molecular profiling of beer wort fermentation diversity across natural Saccharomyces eubayanus isolates. Microb Biotechnol. 13 (4), 1012-1025 (2020).
- Brickwedde, A., et al. physiological and regulatory analysis of maltose transporter genes in Saccharomyces eubayanus CBS 12357T. Front Microbiol. 9, 1786 (2018).
- Brouwers, N., et al. In vivo recombination of Saccharomyces eubayanus maltose-transporter genes yields a chimeric transporter that enables maltotriose fermentation. PLoS Genet. 15 (4), e1007853 (2019).
- Meurer, M., Chevyreva, V., Cerulus, B., Knop, M. The regulatable MAL32 promoter in Saccharomyces cerevisiae: characteristics and tools to facilitate its use. Yeast. 34 (1), 39-49 (2017).
- Mashruwala, A. A., Boyd, J. M. De novo assembly of plasmids using yeast recombinational cloning. Methods Mol Biol. 1373, 33-41 (2016).
- Sikorski, R. S., Hieter, P. A system of shuttle vectors and yeast host strains designed for efficient manipulation of DNA in Saccharomyces cerevisiae. Genetics. 122 (1), 19-27 (1989).
- Gietz, R. D., Schiestl, R. H. High-efficiency yeast transformation using the LiAc/SS carrier DNA/PEG method. Nat Protoc. 2 (1), 31-34 (2007).
- Baker, E. P., Hittinger, C. T. Evolution of a novel chimeric maltotriose transporter in Saccharomyces eubayanus from parent proteins unable to perform this function. PLoS Genet. 15 (4), e1007786 (2019).
- Nespolo, R. F., et al. An Out-of-Patagonia migration explains the worldwide diversity and distribution of Saccharomyces eubayanus lineages. PLoS Genet. 16 (5), e1008777 (2020).
- Hohnholz, R., Pohlmann, K. J., Achstetter, T. Impact of plasmid architecture on stability and yEGFP3 reporter gene expression in a set of isomeric multicopy vectors in yeast. Appl Microbiol Biotechnol. 101 (23-24), 8455-8463 (2017).
- Wu, Y., et al. Engineering an efficient expression using heterologous GAL promoters and transcriptional activators in Saccharomyces cerevisiae. ACS Synth Biol. 12 (6), 1859-1867 (2023).
- Calvey, C. H., Willis, L. B., Jeffries, T. W. An optimized transformation protocol for Lipomyces starkeyi. Curr Genet. 60 (3), 223-230 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten