Method Article
Effiziente Erzeugung Menschen induzierten pluripotenten Stammzellen aus menschlichen Körperzellen mit Sendai-Virus
In diesem Artikel
Zusammenfassung
Hier präsentieren wir unsere etablierte Methode zur menschlichen Körperzellen in gentechnikfreien menschlichen iPS-Zellen mit Sendai-Virus, das konsequente Ergebnis-und Effizienzsteigerung zeigt neu zu programmieren.
Zusammenfassung
Vor ein paar Jahren, die Einrichtung des menschlichen induzierten pluripotenten Stammzellen (iPS-Zellen) in eine neue Ära in der Biomedizin eingeläutet. Mögliche Verwendung von menschlichen iPS-Zellen umfassen Modellierung Pathogenese der menschlichen genetischen Erkrankungen, autologe Zelltherapie nach der Gen-Korrektur und personalisierte Wirkstoff-Screening durch eine Quelle von Patienten-spezifischen und Symptom relevanten Zellen. Allerdings gibt es einige Hürden zu überwinden, wie die Beseitigung der verbleibenden Umprogrammierung Faktor Transgen-Expression nach menschlichen iPS-Zellen-Produktion. Noch wichtiger ist, könnte Rest Transgen-Expression in undifferenzierten menschlichen iPS richtige Differenzierungen behindern und verleiten die Auslegung von krankheitsrelevanten In-vitro-Phänotypen. Mit diesem Grund haben die Integration frei und / oder Transgen-freien menschlichen iPS-Zellen mit verschiedenen Methoden, wie z. B. Adenovirus, der piggyBac System, Minicircle-Vektor, episomaler Vektoren, direkte Lieferung Protein entwickelt und synthetisiert mRNA. Jedoch Effizienz reprogrammierung mit Integration freie Verfahren ist in den meisten Fällen sehr gering.
Hier präsentieren wir eine Methode, um menschliche iPS-Zellen unter Verwendung von Sendai-Virus (RNA-Virus) auf der Basis Umprogrammierung System zu isolieren. Diese Umprogrammierung Methode zeigt, konsistente Ergebnisse und eine hohe Effizienz in kostengünstiger Weise.
Einleitung
Menschliche embryonale Stammzellen (hES-Zellen) haben eine Kapazität zur Selbsterneuerung in vitro und haben Pluripotenz, die potenziell nützlich für Krankheit Modellierung sein könnte, für Wirkstoff-Screening, und zellbasierte Therapien zu entwickeln, um Krankheiten und Gewebeverletzungen zu behandeln. Allerdings haben hESCs eine Beschränkung für die Zell-Ersatztherapie wegen der immunologischen, onkologischen und ethischen Schranken, und im Zusammenhang mit Krankheit Gene studieren, konnte krankheitsspezifische hESCs durch Präimplantations-Diagnostik (PID) Ansätze isoliert werden, aber es ist immer noch eine technische Herausforderung und die Embryo-Spenden sind ziemlich selten. Diese Fragen werden an die Fortschritte in der Stammzellbiologie, die zur Entwicklung von induzierten pluripotenten Stammzellen (hiPSCs) geführt hat, zusammen.
Menschliche iPS sind genetisch aus menschlichen adulten somatischen Zellen umprogrammiert und Hafen pluripotente Stammzelle-ähnliche Funktionen ähnlich hESCs, der sie macht eine nützliche Quelle für die regenerative Medizin wie dTeppich Entdeckung, Krankheit Modellierung und Zelltherapie in Patienten-spezifischen Weise 1,2.
Bis jetzt gibt es mehrere Methoden, um menschliche iPS-Zellen zu generieren, einschließlich Virus-vermittelte (Retrovirus und Adenovirus) 3, nicht-Virus-vermittelte (BAC-System und Vektoren Transfektion) 4-Gen Transduktionen und Protein-Liefersystem 7.5.
Obwohl eine Lieferung von Virus-vermittelte Gene können ein gewisses Maß an Effizienz zu gewährleisten, konnte virale Vektoren genetischen Fußabdruck zu verlassen, weil sie in Wirts Chromosomen zu integrieren, um eine Anpassung Gene unkontrolliert auszudrücken. Selbst wenn virale Integration Transkriptionsfaktoren aktivieren oder inaktivieren Wirtsgene 8 kann eine unerwartete genetische Aberration und das Risiko von Tumorgenese 5,9 verursachen. Auf der anderen Seite wurden die direkte Einführung von Proteinen oder RNA in somatische Zellen berichtet, haben aber einige Nachteile, wie arbeitsintensiv, wiederholt transfbschnitt, und niedrige Niveau der Umprogrammierung 7,10. Auch episomal und nicht-integrierenden Adenovirus, Adeno-assoziiertes Virus und Plasmid-Vektoren sind noch relativ weniger effizient 11. Aus diesen Gründen ist es plausibel, Nicht-Integration Umprogrammierung Verfahren mit hoher Wirksamkeit iPSC Erzeugung und weniger genetische Anomalien zu wählen. In dieser Studie verwenden wir ein Sendai-Virus-basierten Neuprogrammierung. Diese Methode ist bekannt, dass in das Wirtsgenom nicht integrierten und konsequent produziert menschlichen iPS-Zellen ohne Transgen-Integrationen.
Protokoll
1. Herstellung von Zell und Medien (Tag 1)
- Kultur und erweitern menschlichen Fibroblasten mit DMEM-Medium mit 10% FBS.
- Teller menschlichen Fibroblasten (Abbildung 1) auf eine 24-Well-Platte auf der entsprechenden Dichte pro Well am Tag vor der Transduktion.
HINWEIS: Die folgenden serielle Verdünnungen werden empfohlen (200K, 100K, 50K, 25K, 12.5K und 6.25K), weil verschiedene Zelltypen haben unterschiedliche Befestigungs Fähigkeit. - Die Zellen für einen Tag Inkubation in einem 37 ° C, 5% CO 2-Inkubator, wodurch die Zellen vollflächig verklebt und erweitert.
2. Führen Transduction (Tag 2)
- Am Tag der Transduktion, überprüfen Sie die Zelldichte und wählte die effizientesten Dichte Brunnen (Abbildung 2). Es ist besser, drei verschiedenen Dichten wählen: hoch (80 ~ 90%), Mitte (50 ~ 70%) und niedriger (20 ~ 40%).
- Mindestens 1 Stunde vor Übertragungs absaugen fibroblast Medien aus den Zellen und verändern neue 300 ul von Fibroblasten-Medium.
- Entfernen Sie einen Satz von 4 verschiedenen Sendai-Virus-Rohre von -80 ° C-Lagerung. Auftauen jedes Rohr in der gleichen Zeit in einem 37 ° C Wasserbad für einige Sekunden und dann die Rohre aus dem Wasserbad. Danach auftauen sie auf Raumtemperatur; Zentrifugenröhrchen bei 6000 g für 10 sec und legen sie auf Eis, bis Gebrauch. Wieder einfrieren und auftauen Sie nicht das Virus, da die Titer nicht aufrecht zu erhalten.
- Hinzufügen der angegebenen Mengen von jedem der vier Röhrchen Sendai-Virus (Oct-4, Klf-4, c-Myc und Sox-2) auf den Mikrozentrifugenröhrchen. Stellen Sie sicher, dass die Lösung auch durch Pipettieren vorsichtig gemischt.
Zum Beispiel, wenn 50K Zellen / ein gut 24-Well-Platte für die Transduktion Lookwell:
(Titer auf der Grundlage der Analysezertifikat von Life Technologies, kann von Charge zu Charge sein)
Rohr hOct-4 = 6,0 x 10 7 CIU, 3 MOI = 5 ul
Rohr hSox-2 = 6,5 x 10 7 CIU, 3 MOI = 4,6 μ; L
Rohr hKlf-4 = 6,3 x 10 7 CIU, 3 MOI = 4,8 ul
U-hc-Myc = 7,8 x 10 7 CIU, 3 MOI = 3,8 ul
Total = 18,2 ul von vier Faktoren Virus Mischung / ein gut (50K-Zellen) von 24-Well-Platte - Je nach Zelldichte; hinzufügen 2X, 1X, 0,5 X und Volumen der Virusgemisch in den Brunnen. Schütteln Sie die Platte von vorne nach hinten, links und rechts (2X: 36,4 ul Virus Mischung, 1X: 18,2 ul 0,5 X und 9,1 ul).
- Platzieren der Zellen in ein 37 º C, 5% CO 2-Inkubator über Nacht inkubieren.
3. Austausch von Kultur-Medium (Tag 3 und Tag 5)
- 24 Stunden nach der Transduktion, ersetzen Sie das Medium mit 500 ul frisch Fibroblasten-Medium.
- An Tag 5, verändern Medium mit frischen Fibroblasten-Medien.
4. Bereiten MEF Geschirr für die Co-Kultur (Tag 8)
- Bereiten MEF-Zellen in 60 mm-Kulturschalen mit 10% FBS enthielt DMEM-Medium, da einy vor Passagieren des transduzierten Fibroblasten auf MEF Feeder-Zellen.
Hinweis: Wir sind mit 5 x 10 5 von MEF in jedem 60 mm-Schale.
5. Starten Co-Kultur Transduzierte MEF-Zellen mit Feeder-Zellen (Tag 9)
- Vor der Beschichtung transduzierten Zellen absaugen 10% FBS enthielt DMEM-Medium von MEF Feeder-Gerichte und fügen Sie frisches 10% FBS DMEM-Medium enthalten ist.
- Entfernen des Mediums von den transduzierten Fibroblasten, die in der 24-Well-Platte und wasche die Zellen einmal mit D-PBS. Nach Zugabe von 200 ul 0,25% Trypsin / EDTA in die Fibroblastenzellen inder 24-Well-Platte. Nach weniger als 5 min Inkubation mit Trypsin / EDTA, wenn die Zellen beginnen, von der Platte zu lösen, sammeln Sie die Zellen mit Wachstumsmedium in 15 ml konischen Röhrchen. Dann zentrifugieren in 6000 g für 4 min.
HINWEIS: Die Bündelung 2X, 1X und 0,5 X Virus transduzierten Fibroblasten wird empfohlen. - Entfernen Sie das überstehende Medium und zu waschen, um erneut zu suspendierendas Zellpellet mit 4-5 ml frischem Fibroblasten-Medium für die Neutralisation von Trypsin. Dann zentrifugieren bei 135 × g für 4 min.
- Platte Zellen als serielle Verdünnung Dichte und beginnen zu Kokultur mit Fibroblasten-Medium auf MEF: z. B. 1/2, 1/4, 1/8, 1/16, 1/32, 1/64 und 1/128 pro 60 mm-Schale. Je nach Zellsterberate während der ersten Woche der Transduktion kann man nicht zu auf 1/2 oder 1/128 Dichte zu verdünnen. Beiseite verbleibenden Zellen Gesamt-RNA als Positivkontrolle für Transgene RT-PCR zu extrahieren.
- Platz 60 mm Schalen in 37 ° C, 5% CO 2-Inkubator über Nacht.
6. Feed-menschlichen embryonalen Stammzellen Medium und Überwachung der Zellen (Tag 10)
- Am nächsten Tag ändern Fibroblasten-Medien auf menschliche ES-Medien mit 10 uM Y-27632. Danach ändern Medium täglich mit menschlichen ES-Medium: 800 ml + 200 ml DMEM/F12 Knockout Serumersatz + 10 ml L-Glutamin + 10 ml 10 mM MEM Mindest nicht-essentiellen Aminosäuren+ 1 ml 2-Mercaptoethanol + 10 ng / ml basischen Fibroblastenwachstumsfaktor.
- Führen Sie eine tägliche Medienwechsel mit frischen menschlichen ES-Medien. Nach einer Woche, prüfen die Gerichte einmal pro 2 ~ 3 Tage unter einem Mikroskop für die Bildung von ES-Kolonie-wie Zellklumpen. Je nach Zelltyp, wird die Kultur mehr oder weniger Zeit in Anspruch nehmen, bevor Kolonien gefunden.
7. Picking die induzierten pluripotenten Stammzellen Kolonien und erweitern die Zellen (Tag 20 ~)
- Drei Wochen nach Transduktion, sollte Kolonien richtig für die Kommissionierung gewachsen erscheinen.
- Am Tag vor der Kommissionierung die Kolonien, bereiten MEF Feeder im 24-Well-Platte.
HINWEIS: Wir sind mit 12,5 x 10 5 von MEF in einer 24-Well-Platte. - Am nächsten Tag ändern Medium der MEF Gerichte aus Fibroblasten-Medien auf menschliche ES-Medien mit 10 uM Y-27632. Manuell holen eine Kolonie zu jeder Zeit unter dem Mikroskop in der Kommissionierung-Haube und machen kleinere Klumpen durch Pipettieren und übertragen Sie sieauf neu erstellten MEF dishes.Try mehrere Klone holen.
- Passage und erweitern die jeweils auch von einer 24-Well-Platte mit einer 6-Well-Platte zunächst. Dann aus einer 6-Well-Platte auf eine 60-mm-Schalen, bevor die weitere Vermehrung.
8. Charakterisierung humaner iPS-Zellen (nach 10 Passagen)
- Immunfluoreszenz
- Teller menschlichen iPS-Zellen in 24-Well-Platte und Kultur während 4-5 Tage mit täglichen Medienwechsel.
- Zum Starten Immunfluoreszenz-Test, waschen Sie die Platte mit D-PBS einmal. Dann fixieren Sie die Zellen sofort in 0,4% Paraformaldehyd für 30 min. Danach waschen dreimal in D-PBS für 5 min.
- Behandlung von fixierten Zellen mit 0,1% Triton X-100-Lösung in PBS für 15 min bei Raumtemperatur.
- Absaugen Permeabilisierung Lösung (0,1% Triton X-100-Lösung), dann mit 0,5% BSA-Blockierungslösung für 1 Stunde bei Raumtemperatur.
- Führen Sie eine Erkennung von Nanog, Oct-4, SSEA (Stufe spezifischen embryonalen Antigde) und TRA-1 bis 81 (Tumorantigen) mit einem Anti-Human-Antikörper in 0,5% BSA-Lösung (überprüfen Sie die Materialtabelle für die Antikörperverdünnungsrate). Inkubieren des primären Antikörpers für 2 h bei Raumtemperatur. Dann waschen dreimal in D-PBS für jeweils 5 min.
- Überprüfen mittels Fluoreszenzmikroskopie nach 1 h Inkubation mit Alexa Fluor 488 Ziege anti-Maus-IgG-Antikörper als 2. Antikörper, der 1:2000 in 0,5% BSA-Lösung bei Raumtemperatur verdünnt. Stain alle Kerne von je MEF Feeder-Zellen und hiPSCs mit DAPI.
- RT-PCR von Transgene Bestätigung
- Auszug Gesamt-mRNA aus Zellpellet durch RNA-Extraktion mit Reagenzien.
- Ein Reverse Transkription von Aliquoten (1 ug) von Gesamt-RNA und die resultierende cDNA für die PCR-Amplifikation durch reverse Transkriptase.
- Die PCR-Gemische zu 2 ul cDNA-Matrize wurden 10 ul 2X PCR-Master-Mix, 2 &mgr; l von 2 pmol Primer enthalten(Vorwärts: 1 ul-und Rückwärts: 1 ul) und 6 ul der DW. Um die Genexpression zu erfassen, führen die RT-PCR mit den in Tabelle 1 angegebenen Primer.
- Führen die RT-PCR mit GAPDH-Gen als Kontrolle für die Effizienz der Amplifikationsreaktionen. Visualisieren und PCR-Produkt analysieren, um 1% Agarose-Gel-Elektrophorese.
Ergebnisse
Normalerweise infizierten Fibroblasten keine morphologischen Veränderungen in mehrere Tage nach Sendai-Virus-Transduktion zu zeigen, aber fünf Tage später, beginnen sie zu verschiedenen Formen (Abbildung 1) zu haben. Wie im rechten Teil der Abbildung 1 beschrieben, funktionieren Zellen nicht typisch Fibroblastenmorphologie mehr. Sie haben eine runde Form und größeren Kern als Zytoplasma. Auch bei Übertragungs wird in 80% zellulären Konfluenz durchgeführt wird, sieht es aus wie sie sind weniger in der konfluenten gut, nachdem sie beginnen, neu zu programmieren. Wenn diese Art von morphologischen Veränderungen beginnen, in der die meisten der gut zu erkennen ist, sind transduzierten Fibroblasten bereit, auf MEF-Feeder-Kulturschalen plattiert werden.
In unserem Protokoll, minimiert wir Zellkultur Skala und gab auch verschiedene Möglichkeiten Reprogrammierung, wie Fibroblasten, die unterschiedliche Zelldichte und Viren-Titer und die anschließende Re-Beschichtung mehrere Verhältnis MEF, die hiPSCs Erzeugungseffizienz erhöhen wird ( Abbildung 2). Vor Transduktion, die Streifen mit verschiedenen Konfluenz (niedrig, mittel und hoch) für weitere Sendai-Virus-Transduktion gewählt werden; als rote, grüne und gelbe Farbe in der Platte vertreten.
Zu Beginn der Umprogrammierung auf MEF Feeder, ist es schwer zu transduzierten Zelltypen zu unterscheiden, aber nach über einer Woche Co-Kultur, erscheinen teilweise neu programmiert Fibroblasten und sehen aus wie gelockert ES-ähnliche Form (Abbildung 3). Aus diesem Grund, wenn menschliche iPS-Kolonien sind bereit für die Passage in neue MEF Feeder-Gericht, ist die manuelle Kommissionierung effizienter Weg für die Übertragung statt Spaltung mit dem Enzym.
In Abbildung 3 und Abbildung 4, unter dem Mikroskop nur menschliche iPS-Zellen haben klare Kanten, die eine klare Unterscheidung zwischen menschlichen iPS-Zellen (als 'völlig neu programmiert Zellen "betrachtet) und" unvollständig Zellen umprogrammiert' von Morphologie zu machen. Komplett neu programmiert Zellen look, wie kompakt und dicht verpackt. Vor allem die Kolonien haben klare Grenze, während teilweise neu programmiert Zellen aussehen versammeln, um Kolonie zu machen, aber sehr zerbrechlich zu verlieren und zu brechen. Selbst wenn wir eine Pipettieren vorsichtig tun, es ist leicht gelöst. Auch nach dem Ausschluss teilweise umprogrammiert menschlichen iPS-Zellen Kolonien (während sie erweitern und Passagieren), müssen vollständig neu programmiert menschlichen iPS-Zellen, die von der manuellen Kommissionierung gepflegt werden.
Nach der Kommissionierung Kolonien und den Ausbau hiPSC Klone in mehreren Wochen, Ausdruck stemness Marker bestimmt werden muss. Nach dem Ausbau der hiPSC Klone, haben wir sie überprüfen mit verschiedenen Gruppen von Tests, einschließlich Antikörperfärbung (SSEA4, Oct-4, TRA-81 und Nanog) und RT-PCR von pluripotenten Marker (Daten nicht gezeigt) als eine Qualitätskontrolle.
Im Vergleich zu H9, sind zwei verschiedene hiPSCs positiv SSEA4, Oct-4, TRA-81, und Nanog. Dieses Ergebnis zeigt, dass isolierte menschliche pluripotente iPS erworben Qualität ( Abbildung 5).
Schließlich Abbildung 6 zeigt, dass wir keine Expression des Transgens in hiPSC Klone nicht erkennen, durch RT-PCR. Zumindest über 10 Passagen hiPSCs, spezifischen Primern (Tabelle 1) für exogene Oct-4, Sox-2, Klf-4 und c-Myc mit positiven Kontrollproben mit starker Expression des Transgens Ebene verwendet werden.
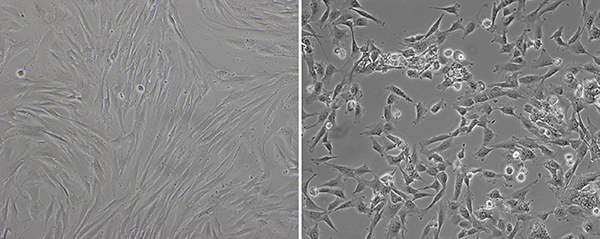
Abbildung 1. Morphologische Veränderung nach Sendai-Virus-Transduktion. Vor der Transduktion, Fibroblasten zeigen typische spindelförmig (links). Etwa acht Tage nach Transduktion, Morphologie der transduzierten Zellwechsel wie viel mehr runde Form (rechts). Bitte klicken Sie hehe um eine größere Version dieser Figur zu sehen.

Abbildung 2. Diagramm der Einarbeitungsplan, um maximale Effizienz zu gewährleisten. Über vier verschiedene Zelltypen können in einer 24-Well-Platte überzogen werden (von 1 bis 4). Da ihre Befestigung und Zellüberlebensfähigkeit könnte abhängig von den verschiedenen Zelltypen sein, wird vorgeschlagen, unterschiedliche Zelldichte (von 200 K bis 6.25K Zellen pro Vertiefung) zu plattieren. Darüber hinaus kann Viruskonzentration eingestellt werden (0,5 X, 1X und 2X).

Abbildung 3. Bildung von menschlichen induzierten pluripotenten Stammzellen Zellkolonie aus transduzierten fibroblast. In etwa zwei Wochen nach der erneuten Plattierung sie in MEFs, sollte umgehen Kolonien erscheinen und sehen aus wie menschliche embryonale Stammzellkolonien. Normalerweise werden die vollständig neu programmiert menschlichen iPS-Kolonien haben sehr klare Grenzen. (Pfeil: vollständig neu programmiert menschlichen iPS Kolonien, Pfeilspitze: teilweise neu programmiert menschlichen iPS-Zellen). Klicken Sie hier für eine größere Ansicht .

Abbildung 4. Picking up menschlichen induzierten pluripotenten Stammzellen Zellkolonien. Menschliche iPS Kolonien werden oft von teilweise neu programmiert Zellen (links) umgeben ist. Unter dem Mikroskop, Kommissionierung der ES-Kolonie nur wie, brechen in kleine Klumpen und dann Übertragung (rechts). Nach 2 bis 3 Passagen, es sind undifferenzierte hiPSC Kolonien (unten und links). (Pfeil: vollständig neu programmiert menschlichen iPS Kolonien, Pfeilspitze: teilweise neu programmiert menschlichen iPS-Zellen) Klicken Sie hier für eine größere Ansicht .
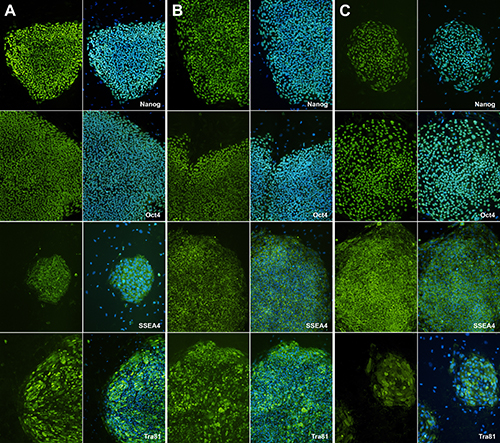
Abbildung 5. Immunfluoreszenzfärbung mit pluripotenten Stammzellmarker. Immunfluoreszenz-Test zeigt menschliche iPS-Zellen haben stemness features.SSEA-4, TRA-81, Nanog, Oct-4as pluripotenten Marker und DAPI ist eine Steuerung für Kernfärbung. (A) stellt H9-Färbung als eine Kontrollprobe. (B) und (C) zeigen menschlichen iPS-Klone.et = "_blank"> Klicken Sie hier für eine größere Ansicht.

6. Bestätigung der Transgenexpression Ebene. Spätestens nach 10 Gang Kultur menschlicher iPSCs, Silencing von exogenen Sox2, Klf-4, c-Myc und Oct-4-Genexpression wird durch reverse Transkriptase-Polymerase-Kettenreaktion bestätigt. hGAPDH ist eine interne Kontrolle. Für eine positive Kontrolle wird RNA aus Rest transduzierten Fibroblasten (Tag 7 nach der Sendai-Virus-Infektion) extrahiert. (Spur 1: Marker, Spuren 2-6: GAPDH, Klf4, c-Myc, Oct-4, Sox-2 und in Positiv-Kontrolle, Bahnen 7-11: GAPDH, Klf4, c-Myc, Oct-4 und Sox -2 in hiPSCs).
| Primer Namen | Sequenz |
| hOCT4 (F) | CCC GAA GAA AGA AGC GAA CCA G |
| hOCT4 (R) | AAT GTA TCG AAG GTG CTC AA |
| hMYC (F) | TAA CTG ACT AGC AGG CTT GTC G |
| hMYC (R) | TCC ACA TAC AGT CCT GGA TGA TGA TG |
| hSOX2 (F) | ACA AGA GAA AAA ACA TGT ATG G |
| hSOX2 (R) | ATG CGC TGG TTC ACG CCC GCG CCC AGG |
| hKLF4 (F) | ACA AGA GAA AAA ACA TGT ATG G |
| hKLF4 (R) | CGC GGC GCT AGG GCC GCT GCT CGA C |
| hGAPDH (F) | AGG TCG GAG TCA ACG TTG GAT |
| hGAPDH (R) | ATG GTG GCA TGG ACT GTG GT |
Tabelle 1. RT-PCR-Primer für Transgen Bestätigung. Sind die Primer-Sequenzen, die für tra verwendet werden,NsGene Bestätigung. T m 50,6 ° C in allen Primer-Sets.
Diskussion
Reprogrammierung menschlicher Körperzellen zu hiPSCs hält beispiellosen Versprechungen in Grundlagen der Biologie, persönliche Medizin, Transplantation und 12. Zuvor benötigte menschlichen iPS Generationen DNA-Virus, das Integrationsrisiko in das Wirtsgenom, die unerwünschte genetische Mutationen, die weitere klinische Anwendungen wie Arzneimittelentwicklung und Transplantationstherapien 13 begrenzen erstellen hat. Mit aus diesem Grund haben viele Studien wurde berichtet, Vektor-und gentechnikfreien System menschlichen iPS-Zellen von mehreren alternativen Methoden zu generieren, aber in der gleichen Zeit, die Effizienz der Isolierung "Fuß-print frei" menschlichen iPS-Zellen berücksichtigt werden. So sollte beispielsweise das RNA-Virus ein minimales Maß an genetischer Aberration im Vergleich zu anderen Methoden 14 haben, obwohl es keine Veröffentlichung Sequenzierung ganzer Genome, die "genomische Sicherheit" des Sendai-Virus-vermittelte iPSC Generation noch zeigen. Hier präsentieren wir ein Verfahren zur Erzeugung hiPSCs mit derSendai-Virus, die eine hohe Effizienz bei der Umprogrammierung kostengünstige Möglichkeit hat. Die daraus resultierenden menschlichen iPS-Zellen sind frei von Transgens mit der Erhaltung der zellulären und molekularen Ähnlichkeiten zu hESCs in proliferative und Entwicklungspotenzial.
Wir können verschiedene Ursprünge Fibroblasten (> 4) gleichzeitig mit nur einem Satz von Sendai-Virus umzuprogrammieren. Und in unserer Methode, wenden wir verschiedene Zellzusammenflüsse und Viren Dosierung Unterschiede. Dieser "Mix and Match" Kombinationstechnik können die Umprogrammierung Wirksamkeit zu maximieren. Fibroblasten (von einem einzigen Patienten) in jedem gut eine andere Virusinfektion Ebene, sondern durch die Bündelung zusammen, konnte die Möglichkeit, hiPSCs Kolonien auf der Grundlage unserer Erfahrung zu erhöhen. Andere Ausgaben der klonalen Variation unter hiPSC Linien Proliferationsrate, die Zellanheftung / Überleben, Status der X-Chromosom Inaktivierung 15. Differenzierungspotentiale 16 usw. In der Tat wurde beobachtet, dass jeder Klon eine unterschiedlichent neuronalen Neigung, die durch Messen des Expressionsspiegels einer miR-371-Cluster vorhergesagt werden konnte. Darüber hinaus haben wir bei der Erzeugung menschlicher iPSCs aus menschlichen Myoblasten mit dieser Methode, die zeigt, dass das Sendai-Virus kann für viele verschiedene Zelltypen in Umprogrammierung verwendet werden gelungen. Außerdem wir mit unserer Methode haben wir hiPSCs in mehr als 10 verschiedene Krankheits Zusammenhang Fibroblasten erzeugt. Im Durchschnitt könnte zwischen 5 und 10 hiPSC Klone aus einer Fibroblasten, erhalten werden.
Obwohl die Ableitung von gentechnikfreien menschlichen iPS-Zellen mit dem Sendai-Virus ist die effizienteste Methode, die Umprogrammierung die praktischsten Wege sein könnte, gibt es ein paar Einschränkungen in diesem Protokoll berücksichtigt werden;
- Je nach jedem Sendai-Virus-Charge, muss der Virustiter um konsistente MOI berechnet werden (wie in 2.4 beschrieben).
- Wir beobachteten die Änderung der Infektiosität unter verschiedenen somatischen Zellen, die auch benötigt zusätzliche Titration Robust Virusinfektion.
- Die Sendai-Virus-vermittelte Umprogrammierung kann als eine der besten Entscheidungen, die für Forschungszwecke in Betracht gezogen werden. Aber für die klinische Studie, ist es vielleicht nicht die erste Wahl sein, da gibt es ein Problem mit der Lizenz-Unternehmen, die ursprünglich die Sendai-Virus-System entwickelt.
- Es dauert etwa zwei Monate, bis die neu programmiert Körperzellen sind frei von Transgenen, deshalb müssen wir die Expression des Transgens nach mindestens 10 Passagen zu überprüfen.
- Es scheint so, dass der Durchgang Nummer des primären kultivierten Fibroblasten kann die Effizienz der Reprogrammierung mit dem Sendai-Virus beeinflussen, obwohl wir nicht über einen direkten Vergleich. Die Proliferationsrate muss auch bestimmt werden. Weitere Studien sind erforderlich, um die Wirkung von Fibroblasten Gang-Nummern und ihre Verbreitung auf der Umprogrammierung Effizienz zu bestimmen.
- In dieser Studie verwenden wir eine Maus Feeder-Schicht für hiPSC Erstellung und Pflege, aber ein Feeder-freien System kann ein alternativ seinE Weise in einer zukünftigen Studie.
Unsere aktuellen Protokoll ist ein wichtiger Schritt in Richtung Studium patientenspezifische hiPSCs für Krankheit Modellierung, regenerative Medizin, und andere Anwendungen.
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Wir möchten den Mitgliedern des Lee-Labor für wertvolle Diskussionen zum Manuskript danken. Die Arbeit im Labor Lee wurde durch Zuschüsse von Robertson Investigator Award der New York Stem Cell Foundation und von Maryland Stem Cell Research Fund (TEDCO) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| CytoTune-iPS Reprogramming Kit | Invitrogen | A1378002 | |
| CF-6,MEFs, neomycin-resistant, mitomycin C treated | Global stem | GSC-6105M | 5 x 105/6 cm or 12.5 x 105/24-well plate |
| Trypsin EDTA 0.25% Trypsin with EDTA 4Na 1X | Invitrogen | 25200114 | |
| DMEM/F-12 medium | Invitrogen | 11330-032 | |
| 24-well Cell Culture Plate, flat-bottom with lid | BD | 353935 | |
| Y-27632 | TOCRIS | 1254 | 10 μM (Stock: 10 mM) |
| basic fibroblast growth factor | LIFE TECHNOLOGIES | PHG0263 | 10 ng (Stock : 100 ug) |
| Knock-out serum replacement | Gibco | 10828028 | |
| Dulbecco's Modified Eagle Medium (D-MEM, DMEM) (1X), liquid (high glucose) | Invitrogen | 11965118 | |
| Fetal bovine serum | Thermo Scientific Fermentas | SH30071.03 | |
| L-Glutamine-200 mM (100X), liquid | GIBCO | 25030-081 | 1/100 |
| MEM Non-Essential Amino Acids Solution, 100X | LIFE TECHNOLOGIES | 11140050 | 1/100 |
| 2-Mercaptoethanol (1,000X), liquid | GIBCO | 21985023 | 1/1,000 |
| Hausser Phase Contrast Hemacytometers | Hausser Scientific | 02-671-54 | |
| EmbryoMax 0.1% Gelatin Solution | Millipore | ES-006-B | |
| SSEA-4 | DSHB | MC-813-70 | 1/200 |
| anti-Tra-1-81 | Cell Signaling | 4745S | 1/200 |
| mouse monoclonal Oct4 antibody | Santa Cruz | SC-5279 | 1/1,000 |
| Nanog | R&D | AF1997 | 1/1,000 |
| Alexa Flouor 488 goat anti-mouse | Invitrogen | 948492 | 1/2,000 |
| DPBS (Dulbecco's Phosphate-Buffered Saline), 1X without calcium & magnesium | Cellgro | 21-031-CV | |
| QuantiTect Reverse Transcription Kit | QIAGEN | 205313 | |
| PCR Master Mix [2X] | Thermo Scientific Fermentas | K0171 | |
| Trizol | Invitrogen | 15596018 | |
| picking hood | NuAire | NU-301 | |
| dissecting scope | Nikon | SMZ745 |
Referenzen
- Yu, J., et al. Induced pluripotent stem cell lines derived from human somatic cells. Science. 318, 1917-1920 (2007).
- Okita, K., Nakagawa, M., Hyenjong, H., Ichisaka, T., Yamanaka, S. Generation of mouse induced pluripotent stem cells without viral vectors. Science. 322, 949-953 (2008).
- Zhou, W., Freed, C. R. Adenoviral gene delivery can reprogram human fibroblasts to induced pluripotent stem cells. Stem Cells. 27, 2667-2674 (2009).
- Woltjen, K., et al. piggyBac transposition reprograms fibroblasts to induced pluripotent stem cells. Nature. 458, 766-770 (2009).
- Okita, K., et al. A more efficient method to generate integration-free human iPS cells. Nature Methods. 8, 409-412 (2011).
- Yusa, K., Rad, R., Takeda, J., Bradley, A. Generation of transgene-free induced pluripotent mouse stem cells by the piggyBac transposon. Nature Methods. 6, 363-369 (2009).
- Kim, D., et al. Generation of human induced pluripotent stem cells by direct delivery of reprogramming proteins. Cell Stem Cell. 4, 472-476 (2009).
- Yu, J., et al. Human induced pluripotent stem cells free of vector and transgene sequences. Science. 324, 797-801 (2009).
- Okita, K., Ichisaka, T., Yamanaka, S. Generation of germline-competent induced pluripotent stem cells. Nature. 448, 313-317 (2007).
- Yoshioka, N., et al. Efficient Generation of Human iPSCs by a Synthetic Self-Replicative RNA. Cell Stem Cell. 13, 246-254 (2013).
- Dowey, S. N., Huang, X., Chou, B. K., Ye, Z., Cheng, L. Generation of integration-free human induced pluripotent stem cells from postnatal blood mononuclear cells by plasmid vector expression. Nature Protocols. 7, 2013-2021 (2012).
- Fluri, D. A., et al. Derivation, expansion and differentiation of induced pluripotent stem cells in continuous suspension cultures. Nature Methods. 9, 509-516 (2012).
- Nakagawa, M., et al. Generation of induced pluripotent stem cells without Myc from mouse and human fibroblasts. Nature Biotechnology. 26, 101-106 (2008).
- Fusaki, N., Ban, H., Nishiyama, A., Saeki, K., Hasegawa, M. Efficient induction of transgene-free human pluripotent stem cells using a vector based on Sendai virus, an RNA virus that does not integrate into the host genome. P Jpn Acad B-Phys. 85, 348-362 Forthcoming.
- Silva, S. S., Rowntree, R. K., Mekhoubad, S., Lee, J. T. X-chromosome inactivation and epigenetic fluidity in human embryonic stem cells. P Natl Acad Sci USA. 105, 4820-4825 (2008).
- Kim, H., et al. miR-371-3 expression predicts neural differentiation propensity in human pluripotent stem cells. Cell Stem Cell. 8, 695-706 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten