Method Article
Die Beurteilung Somatische Hypermutation in Ramos B-Zellen nach Überexpression oder Knockdown von spezifischen Genen
In diesem Artikel
Zusammenfassung
Wir beschreiben, wie retroviralen oder lentiviralen Infektionen der Überexpression oder shRNA-Konstrukte, die in das menschliche Ramos B-Zell-Linie und wie somatische Hypermutation in diesen Zellen zu messen. Durchführen
Zusammenfassung
B-Zellen beginnen ihr Leben mit niedriger Affinität Antikörper durch V (D) J-Rekombination erzeugt. Doch beim Erkennen eines Krankheitserregers, wird die Variable (V) Region eines Immunglobulins (Ig)-Gen etwa 100.000-fach stärker als der Rest des Genoms durch somatische Hypermutation (SHM), mutiert wodurch Antikörper mit hoher Affinität 1,2. Darüber hinaus produziert der Klasse Switch-Rekombination (CSR)-Antikörper mit verschiedenen Effektor-Funktionen, je nach Art der Immunantwort, die für einen bestimmten Erreger benötigt wird. Beide CSR und SHM durch activation-induced Cytidindeaminase (AID), die Cytosin-Reste in DNA um Uracile produzieren desaminiert eingeleitet. Diese Uracile sind fehleranfällig Formen der Reparaturwege verarbeitet, was schließlich zu Mutationen und Rekombination 1-3.
Unser gegenwärtiges Verständnis der molekularen Details der SHM und CSR komme aus einer Kombination von Studien an Mäusen, primäre Zellen, Zelllinien und Zell-free Experimente. Mausmodelle bleiben den Goldstandard mit genetischen Knockouts zeigt eine entscheidende Rolle für viele Reparatur-Faktoren (zB Ung, MSH2, MSH6 Exo1 und Polymerase η) 4-10. Allerdings sind nicht alle Gene zugänglich für Knockout-Studien. Zum Beispiel sind Knockouts von mehreren Doppelstrangbruch Reparatur-Proteine embryonal letalen oder beeinträchtigen B-Zell-Entwicklung 11-14. Darüber hinaus manchmal die spezifische Funktion eines Proteins in SHM-oder CSR kann durch weitere globale Defekte, die durch die Ko verursacht maskiert werden. Darüber hinaus können seit Experimente in Mäusen als langwierig, zu verändern Expression einzelner Gene in Zelllinien ist ein zunehmend beliebtes erste Schritt zur Identifizierung und Charakterisierung Kandidatengene 15-18.
Ramos - ein Burkitt-Lymphom-Zelllinie, die konstitutiv erfährt SHM - ein beliebtes Zell-Linie Modell SHM 18-24 Studie. Ein Vorteil der Ramos-Zellen ist, dass sie einen eingebauten günstigen, halb haben-Quantitatives Maß für SHM. Wildtyp-Zellen exprimieren IgM und, wie sie abholen Mutationen, einige der Mutationen knock out IgM Ausdruck. Deshalb Testen IgM Verlust durch Fluoreszenz-activated cell Scanning (FACS) bietet einen schnellen Auslesen für die Höhe der SHM. Eine quantitative Messung der SHM kann durch direkte Sequenzierung der Antikörper-Gene erreicht werden.
Seit Ramos-Zellen schwer zu transfizieren sind, produzieren wir stabile Derivate, die erhöht oder gesenkt haben Ausdruck eines einzelnen Gens durch Infektion der Zellen mit retroviralen oder lentiviralen Konstrukte, die entweder eine Überexpression Kassette oder eine kurze Haarnadel-RNA (shRNA), jeweils enthalten. Hier beschreiben wir, wie wir Ramos Zellen zu infizieren und dann diese Zellen die Rolle bestimmter Gene auf SHM (Abbildung 1) zu untersuchen.
Protokoll
1. Probenvorbereitung und Zellen
- Führen Sie eine mittlere Vorbereitung von DNA (Midiprep) der Überexpression oder shRNA-haltigen Konstrukte und die retroviralen Vektors Verpackung pKat2 oder lentiviralen Verpackung Vektoren pVSV-G, pMDLg / pRRE und pRSV-Rev 25. Verwenden Sie 6 ug DNA für jedes Konstrukt und entweder 4 pg pKat2 (für retrovirale Infektionen) oder 1,5 ug DNA für jedes der pVSV-g, pMDLg / pRRE und pRSV-Rev Verpackung Vektoren (für lentiviralen Infektionen).
- Bereiten Sie BOSC 23 Medien. Für die Transfektion mit beiden unverändert Dulbecco modifiziertem Eagle-Medium (DMEM) sowie komplette DMEM. Um komplette DMEM, fügen Sie 1% HEPES-Puffer, 1% Penicillin-Streptomycin-Glutamin (PGS), und 10% Neugeborenen Kälberserum (NCS), um eine Flasche DMEM.
- Bereiten Ramos Medien. Für die Infektion Schritt machen kompletten RPMI-1640 Medium (RPMI) durch Zugabe von 1% HEPES, 1% PGS, und 10% fötalem Rinderserum (FBS), um eine Flasche RPMI. Für einzelne Zelle Aussaat Schritt make "konditioniert" Medien durch die wachsende Ramos-Zellen in komplettem RPMI für 2 Tage, Abzentrifugieren der Zellen nach unten bei 400 X g für 4 Minuten und Filterung der Überstand mit einer 0,2 um Filter in eine sterile Flasche. Wie andere komplette Medien ist konditionierten Medien bei 4 ° C gelagert, bis sie benötigt.
- Split 3 x 10 6 BOSC 23-Zellen in einen 100-mm-Platte in 10 mL kompletten DMEM Tag vor der Transfektion so dass die Zellen bei 50-60% Konfluenz Tag der Transfektion werden. Machen Sie eine Platte von Zellen für jeden Vektor Sie planen Transfektion. Es ist wichtig, dass die Zellen nicht in Kultur zu lange gehalten, die Nutzung niedrigen Durchgang Anzahl Zellen oder kürzlich aufgetauten Zellen sowohl für die Transfektion und Infektion vor, um einen guten Infektion zu versichern.
- Fügen Sie Steuerelemente für Ihre Experimente. Für Transfektionen und Infektionen, erstellen Sie eine Negativ-Kontrolle (nicht transfizierte oder infizierte Zellen) und eine positive Kontrolle (Virus, das GFP und Puromycin Resistenzgen-Ausdruck). Für shRNA knock-down experiments, infizieren auch mit einem Rühr-oder irrelevant shRNA Kontrolle.
2. Transfektion BOSC 23-Zellen
- Wärmen Sie die Flaschen unverändert DMEM und vollständige DMEM bei 37 ° C.
- Entfernen Sie das Medium aus dem BOSC 23-Zellen und ersetzen mit 5 ml komplettem DMEM. Bringen Sie die Zellen auf eine 37 ° C Inkubator bis zur Verwendung in Schritt 2.6.
- Wärmen Sie die Flasche FuGENE 6 bei Raumtemperatur für 5 Minuten dann kurz vortexen (<1 Sekunde).
- Aliquot 600 ul DMEM unverändert in ein 1,5 ml Reaktionsgefäß.
- Verdünnen Sie 30 ul FuGENE 6 in die 600 ul DMEM unverändert, kurz vortexen (<1 Sekunde) und dann bei Raumtemperatur inkubieren für fünf Minuten. Achten Sie darauf, direkt Pipette in den Medien. Lassen Sie sich nicht die FuGENE 6 Berühren Sie die Seiten des Rohres.
- Add 6 ug konstruieren und entweder 4 ug pKat2 (retrovirale Infektionen) oder 1,5 ug jedes pVSV-G, pMDLg / pRRE und pRSV-Rev (lentiviralen Infektionen), die DMEM / FuGENE 6-Mix, Wirbel bri eFly (<1 Sekunde) und dann bei Raumtemperatur inkubieren für 25 Minuten.
- Fügen Sie die DNA / DMEM / FuGENE 6 mischen, um die BOSC 23-Zellen und zu einer 37 ° C Inkubator für 24 Stunden.
- Nach 24 Stunden, 5 ml komplettem DMEM auf die transfizierten Zellen und Rückkehr zu einer 37 ° C Inkubator für weitere 24 Stunden.
3. Harvesting Viruspartikel
- Ernten Sie die 10 ml Medium aus dem transfizierten BOSC 23 Zellen mit einer 10 ml-Spritze.
- Bringen Sie ein 0,45 um-Filter bis zum Ende der Spritze und Filter der Medien in ein sauberes 15 ml Polypropylen-Röhrchen.
- Verwenden Sie die viralen Medien sofort oder einfrieren als 1 mL Aliquots in 2 ml Kryoröhrchen bei -80 ° C.
- Nach der Ernte der viralen Medien, bestätigen, dass effiziente Transfektion wurde in der positiven Kontrolle Zellen durch FACS aufgetreten. Dies kann auch für alle Proben durchgeführt werden, wenn das Konstrukt enthält entweder ein fluoreszierendes (zB GFP) oder ein Zelloberflächen-Marker (zB Thy1).
- Seed 2 mL von 2 x 10 5 Zellen / ml Ramos-Zellen in komplettem RPMI in einer 6-Well-Platte 24 Stunden vor der Infektion. Auch hier achten Sie darauf, niedrigen Durchgang Anzahl Zellen oder kürzlich aufgetauten Zellen zu verwenden, um gute Infektion zu versichern.
- Bei Verwendung von eingefrorenen Aliquots von Virus, tauen die virale Medien schnell bei 37 ° C.
- Mix 1 mL viralen Medien mit 3 ul von 10 mg / mL Polybren. Die Endkonzentration von Polybren sollten 10 ug / ml in einem Gesamtvolumen von 3 mL werden.
- Fügen Sie den Virus / Polybren Mix zu den Zellen und gut mischen durch Pipettieren. Wie bereits erwähnt, ein gut sollte einem nicht infizierten Kontrolle verwendet werden, fügen Sie 1 mL komplettem RPMI und 3 ul Polybren diese auch anstelle von 1 ml Virus. Achten Sie darauf, ein gut infizieren mit einem abgestimmten negative Kontrolle. Wir haben auch infizieren man gut mit einem GFP-enthaltenden Vektor, um Infektionen Effizienz durch FACS zu bestimmen.
- Zentrifugieren Sie die 6-Well-Platte bei 3000 X g für 90 Minuten bei Raumtemperatur.
- Bringen Sie die Platte37 ° C Inkubator für 48 Stunden.
- Nach 48 Stunden, Spin-down der infizierten Zellen in einer 15 ml Polypropylen-Röhrchen bei 400 X g für 4 Minuten bei Raumtemperatur. Entfernen Sie die Medien durch Aspiration, dass auf jeden Fall nicht stören das Zellpellet. Resuspendieren der Zellen in frischem RPMI.
- Bestätigen Sie die Effizienz der Infektion in die positive Kontrollprobe durch FACS. Nach wie vor können Sie diese für Ihre Proben tun, wenn das Konstrukt enthält entweder ein fluoreszierendes (zB GFP) oder ein Zelloberflächen-Marker (zB Thy1).
- Starten Sie eine Selektion von Zellen (wenn zutreffend). Unsere shRNA-haltigen Konstrukte enthalten alle eine Puromycin-Resistenz-Kassette. Wir wählen shRNA-infizierten Ramos-Zellen mit 0,25 pg / ml Puromycin. Wir finden, dass Derivate von Ramos-Zellen zeigen manchmal die Anfälligkeit für Puromycin verändert, und deshalb führen wir töten Kurven, um die Konzentration von Puromycin für jede Ableitung zu optimieren. Auch behandeln infizierten Kontroll-Zellen mit Puromycin, um zu bestätigen, dass die Auswahl wirksam ist.
5. Einzel-Zell-Aussaat
Einzel Aussaat kann in einem von zwei Wegen durchgeführt werden: durch FACS-Sortierung oder manuell.
- FACS: Mit einem FACS-Sorter mit einem Adapter für Saatgut 1 Zelle / well in einer 96-Well-Platte mit 100 ul 40% RPMI/40% konditioniert media/20% FBS vorgefüllt. Diese Methode gibt eine Menge mehr einzelne Zellklone pro Platte.
- Manuell: Zählen Sie die Zellen. Einen aliquoten von Zellen bis 10 Zellen / ml in 40% RPMI/40% konditioniert media/20% FBS. Verwenden Sie 10 mL der verdünnten Zellen für jedes gesetzte Platte. Verschiedene Klone variable Erholung in diesem Stadium zeigen daher sinnvoll, mehrere Platten in unterschiedlichen Konzentrationen (im Bereich von 3 bis 20 Zellen / ml). Verwenden Sie die eine, die weniger Auswüchse, um Klone, die aus mehreren Zellen zu vermeiden) produziert. Verwenden Sie eine Mehrkanalpipette, Saatgut 96-well-Platten mit 100 ul der verdünnten Zellen.
- Die Platte wird in einem 37 ° C Inkubator für 2 Wochen.
- Prüfen Sie die einzelnen Vertiefungen under das Mikroskop auf das Vorhandensein von Zellen einen Tag nach Aussaat zu bestätigen. Einzelne Zellen können schwierig zu finden, durch die Notwendigkeit, auf die richtige Tiefe zu konzentrieren. Die Konzentration auf das Wohl Zahlen am unteren Rand der 96-Well-Platte eingeprägt sollte helfen. Die Zellen sind leichter zu finden, wenn durch die FACS-Sorter ausgesät, da die meisten Brunnen einer Zelle haben.
- Nach 2 Wochen kann wachsende Kolonien kaum mit bloßem Auge werden, indem man die Platten von unten gesehen. Sie sind viel leichter zu unter dem Mikroskop zu sehen. Kolonien neigen dazu, an den Rändern des Brunnens und in der Regel die meisten Kolonien in einer Platte wachsen entlang der gleichen Kante in verschiedenen Vertiefungen wachsen. Mark Wells mit robusten Zelle Wachstum und die Übertragung der Zellen auf 24-Well-Platten in 1 ml RPMI und Rückkehr zu einer 37 ° C Inkubator.
- Pflegen Sie Zellen mit 1 x 05 bis 1 Oktober x 10 6 Zellen / ml. Ändern Sie die Medien, durch Drehen der Zellen bei 400 X g für 4 Minuten bei Raumtemperatur und Entfernen von den Medien als notwendig. Ersetzen Sie mit 1 ml frisches RPMI.
- Nach mehreren days des Wachstums, der Transfer der Zellen auf einer 6-Well-Platte und weiter wachsen in einem 37 ° C Inkubator.
- Nach 3 Wochen, analysieren die Klone für IgM Verlust (wie unten beschrieben).
- Weiter zu den Zellen für weitere 2 Wochen wachsen und dann analysieren die Klone wieder an der 5 Wochen Zeitpunkt.
6. Analyse: IgM Verlust (3 und 5 Wochen)
- Aliquot 5 x 10 5 Zellen aus jeder Vertiefung in ein 1,5 mL Mikrozentrifugenröhrchen und drehen Sie die Zellen nach unten bei 400 X g für 4 Minuten bei Raumtemperatur. Verwenden Sie Zellen, die aus jedem der Steuerung und shRNA-knock-down Brunnen, sowie eine Probe für eine ungefärbte Kontrolle.
- Waschen Sie die Zellen in 1 ml PBS und dann wieder drehen sich mit 400 X g für 4 Minuten bei Raumtemperatur.
- Resuspendieren in 50 ul PBS (ungefärbte Kontrolle) oder PBS + 1:20 Verdünnung von PE-markiertem α-human-IgM-Antikörper. Inkubieren auf Eis für 1 Stunde.
- Drehen Sie die Zellen nach unten bei 400 X g für 4 Minuten bei Raumtemperatur.
- Waschen Sie die cEllen zweimal mit 1 ml PBS und Spin für 400 X g für 4 Minuten bei Raumtemperatur.
- Entfernen Sie den Überstand und resuspendieren in 100 ul PBS. Übertragen Sie die resuspendiert gefärbten Zellen auf eine FACS-Röhrchen.
- Analysieren Sie die Zellen mit Hilfe eines FACS-Maschine.
- Vergleichen Sie den Prozentsatz der IgM - Zellen aus den Kontrollen im Vergleich zu den shRNA Boden geworfen Zellen. Wie bereits erwähnt, ist IgM Verlust durch FACS gemessen eine zuverlässige semi-quantitatives Maß für SHM in Ramos-Zellen.
7. Analyse: Bestätigen knock-down durch quantitative RT-PCR (5 Wochen)
- Aliquot 3 x 10 6 Zellen aus jeder Vertiefung und Spin-down bei 400 X g für 4 Minuten bei Raumtemperatur.
- Bereiten RNA aus den aliquoten Teilen von Zellen. Wir verwenden TRIzol nach den Empfehlungen des Herstellers.
- Bereiten cDNA aus der RNA prep. Wir verwenden 2 pg RNA und Superscript II nach den Empfehlungen des Herstellers.
- Führen Sie eine quantitative PCR mit der cDNA zur Bestätigungm knock-down. Wir verwenden GAPDH als Normalisierung Kontrolle, und die SYBR FAST qPCR Kit in einem 10 ul Reaktionsvolumen.
8. Analyse: Direkte Sequenzierung (5 Wochen)
- Aliquot 5 x 10 6 Zellen aus jeder Vertiefung und Spin-down bei 400 X g für 4 Minuten bei Raumtemperatur.
- Isolieren von genomischer DNA aus diesen Zellen. Wir verwenden den Assistenten SV genomische DNA Purification Kit gemäß den Empfehlungen des Herstellers.
- PCR zu amplifizieren der genomischen DNA für die Ig V-Gene 19 (Tabelle 1) und alle anderen Gene von Interesse mit dem KAPA HiFi-DNA-Polymerase. Wir verwenden diese Polymerase aufgrund ihrer geringen Fehler-Raten, aber keine High-Fidelity-PCR-Polymerase sollte akzeptabel sein.
- Gel reinigt die PCR-Produkte auf einem 1% Agarosegel mit dem QIAquick Gel Extraction Kit.
- Einige Polymerasen einen einzelnen Desoxyadenosin (A) Rest der 3 'Enden der PCR-Produkte, während andere dies nicht tun. Diese 3 'A Schwänze sind für den TOPO TA notwendigKlonierungsschritt. Die KAPA HiFi-Polymerase nicht verlassen 3 'A Schwänze. A-Schwanz der PCR-Produkte durch Inkubation 25 ul Gel-gereinigtes PCR-Produkt mit 3 ul 10x Taq Reaktionspuffer, 1 ul 10 mM dNTPs und 1 Einheit Taq-Polymerase bei 72 ° C für 30 Minuten.
- Clone dem Gel-gereinigten PCR-Produkte mit dem TOPO TA Cloning-Kit. Transformieren Sie die Produkte aus dem TOPO Reaktion in die dafür vorgesehenen E. coli DH5-T1 R kompetenten Zellen und Teller auf LB / Agarplatten mit 100 ug / ml Ampicillin und ergänzt mit 1,6 mg X-gal nach den Empfehlungen des Herstellers.
- Send einzelnen Kolonien für die Sequenzierung.
- Führen Sie die Sequenzanalyse, um Typen und Standorte von Mutationen zu bestimmen.
9. Repräsentative Ergebnisse:
Wie bereits erwähnt, verwenden wir eine positive Kontrolle viraler Vektor, die sowohl GFP sowie eine Puromycin-Resistenz-Gen exprimiert. Wir sehen in der Regel 50-75% GFP +-Zellen 2 Tagenach der Transfektion. In unserem Infektionen, wir in der Regel 50-70% sehen Zellen sind GFP + 2 Tage nach der Infektion, aber vor der Auswahl. Nach der Auswahl abgeschlossen ist, sind> 95% der Zellen GFP +.
Als Vertreter Experimenten zeigen wir die Auswirkungen der Überexpression von AID oder umzuwerfen eine Reparatur Faktor in Ramos-Zellen. Insbesondere transfizierten wir eine retrovirale Überexpression Vektor für AID über ein IRES Site Thy1.1 zusammen mit dem retroviralen Vektors Verpackung pKat2 in BOSC 23-Zellen verknüpft. Virus-haltigen Medien wurde filtriert und anschließend verwendet, um Ramos-Zellen zu infizieren. Sowohl Wildtyp (WT) und AID-überexprimierenden (AID hallo)-Zellen wurden einzelne Zelle ausgesät und angebaut für 3 Wochen (Abbildung 3, an welcher Stelle sie für die Genexpression wurden durch qRT-PCR (Abbildung 2) und für IgM Verlust durch FACS analysiert ). Für die FACS-Färbung wurden die Zellen mit den beiden 1:20 verdünnt PE-markiertem α-human-IgM-Antikörper sowie 1:400 verdünnt FITC-markiertem α-Ratte CD90/mouse inkubiertCD90.1 (Thy1.1) Antikörper. In einem separaten Experiment wurden Lentiviren, die entweder eine irrelevante shRNA (Kontrolle) oder eine shRNA gegen einen High-Fidelity DNA-Reparatur-Faktor erwartet, dass gegen SHM schützen BOSC 23-Zellen gemacht, und dann infiziert zu einem AID hallo Klon von Ramos-Zellen. Wieder waren die Genexpression (Abbildung 4) und IgM-Verlust (Abbildung 5) in einzelne Zellklone nach 3 Wochen analysiert. Da die Reparatur Faktor ist gedacht zum Schutz vor Mutationen während SHM geben, sehen wir eine Erhöhung der IgM Verlust und SHM im Fehlen des Faktors. Wenn wir ein Faktor bei der Erzeugung von SHM-assoziierte Mutationen beteiligt war, zu Boden geworfen, hätten wir einen Rückgang der IgM Verlust gesehen, statt zu haben.
Wie erwartet, zeigen unsere qRT-PCR-Ergebnisse eine deutliche Erhöhung der Entwicklungshilfe Ausdruck nach der Infektion von Zellen mit einem AID-Überexpression Vektor (Abbildung 2), während das Niveau der DNA-Reparatur-Faktor Drop in das Vorhandensein einer gezielten shRNA gegen diesen Faktor (Abbildung 4 ). Da in den einzelnen clones kann variieren, sehen Sie möglicherweise einige Variationen in der Genexpression Ebenen. Da Genexpression kann variieren, ist es notwendig, den Ausdruck in jedem Klon Ihnen bei der Planung auf die Analyse weiter zu bestätigen. Verschiedene shRNAs gegen das gleiche Gen können unterschiedliche Auswirkungen auf die Genexpression als auch, so sollten Sie mehrere shRNAs Bildschirm, um festzustellen, welche den größten Einfluss. Knockdown kann auch auf Protein-Ebene durch Immunoblot bestätigt werden. Ebenso können einzelne Klone stark variieren in Stufen von IgM-Verlust. Einige Klone könnten zeigen, ein "Jackpot"-Effekt, wo ein IgM-Mutation in einem der frühesten Zellteilungen eingetreten - zum Beispiel, werden Sie> 50% IgM Verlust zu sehen, nur weil einer der Zellen IgM wurde - im Zwei-Zell-Stadium. Der Einfluss dieser Klone wird durch die Ermittlung des Medians für mehrere einzelne Zellklone minimiert.

Abbildung 1. Schematic für die transfection, Infektion und Analyse von IgM Verlust. Siehe Text für Details.

Abbildung 2. AID-Überexpression in Ramos-Zellen. WT und AID hallo Ramos-Zellen wurden einzelne Zelle geklont gewachsen und für 3 Wochen, an welcher Stelle AID Expression in einzelnen Klone durch qRT-PCR gemessen wurde. GAPDH-Expression wurde zur Normalisierung verwendet. WT wurde auf 1 gesetzt. Der Medianwert angegeben. * Zeigt einen p-Wert von <0,01, als One-tailed Student t-Test berechnet.

Abbildung 3. Erhöhte IgM Verlust in AID hallo Zellen zu WT-Zellen verglichen. Oberflächen-IgM wurde durch FACS an der 3 Wochen Zeitpunkt gemessen, und der Prozentsatz der IgM Verlust war für WT und AID hallo Zellen berechnet. (A) Repräsentative FACS Handlung eines WT-Klon und ein AID hallo zu klonen. Die AID-Überexpression vektor hat Beihilfen in Verbindung mit Thy1.1 über ein IRES site, so Thy1.1 Ausdruck ist als Ersatz für AID Ausdruck verwendet. (B) Quantifizierung der IgM Verlust in mehreren WT und AID hallo Klone. Der Medianwert angegeben. * Zeigt einen p-Wert von <0,01, als One-tailed Student t-Test berechnet.

Abbildung 4. Knockdown einer Reparatur Faktor in AID hallo Zellen. Die Zellen wurden entweder mit einem irrelevanten shRNA (Steuerung) oder unsere shRNA von Interesse (shRNA), und einzelne Zelle seeded infiziert. Nach 3 Wochen wurde die Genexpression in einzelnen Klone durch qRT-PCR gemessen. GAPDH-Expression wurde zur Normalisierung verwendet. WT wurde auf 1 gesetzt. Der Medianwert angegeben. * Zeigt einen p-Wert von <0,01, als One-tailed Student t-Test berechnet.
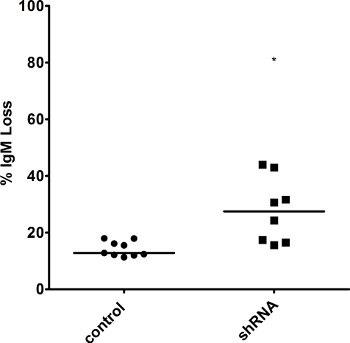
Abbildung 5. Erhöhte IgM Verlustin AID hallo Zellen mit Boden geworfen Ebenen einer Reparatur Faktor. Oberflächen-IgM in mehrere Klone wurde durch FACS an der 3 Wochen Zeitpunkt gemessen, und der Prozentsatz der IgM Verlust war in Zellen noch überexprimierenden AID berechnet. Der Medianwert angegeben. * Zeigt einen p-Wert von <0,01, als One-tailed Student t-Test berechnet.

. Tabelle 1 Primer-Sequenzen für die Ig-Gene 19: die konstante Region Cμ, die schwere Kette V-Region (V H) und die leichte Kette V-Region (V L).
Diskussion
Wie bereits erläutert, haben Zelllinie Modelle für die Antikörper-Diversifizierung zu einem beliebten Ausgangspunkt, um neuartige Proteine, die verschiedene Schritte Einfluss während Antikörper Diversifizierung zu identifizieren. Wir stellen hier ein Verfahren zur Verwendung Virusinfektion entweder knock-down oder überexprimieren Proteine in der Ramos B-Zell-Linie und dann die Auswirkungen auf SHM.
Für diese Studien nutzen wir sowohl WT Ramos und AID hallo Ramos-Zellen. WT-Zellen können verwendet werden, um Faktoren bei der Ausrichtung oder Expression von AID als auch die Reparatur von AID-generierten Läsionen beteiligt zu studieren, während HILFE hallo Zellen Faktoren, die AID Expression beeinflussen auszuschließen.
Es sollte auch darauf hingewiesen, dass SHM ist hier durch die Bestimmung für den Verlust von IgM aus einer Population von Zellen, die zunächst IgM + gemessen werden. Alternativ ist es möglich, mit IgM Start -. Zellen und suchen Sie nach einem Gewinn von IgM +-Zellen in ein Rückfall Assay 23
Durch die Verwendung unterschiedlicher Selektionsmarker (zB Puromycin, Hygromycin, GFP, oder Thy1.1), können wir auch mehrere Infektionen gleichzeitig mit dem gleichen Protokoll. Allerdings haben wir festgestellt, dass bei der Verwendung von zwei verschiedenen Antibiotika-Selektionsmarker, die Empfindlichkeiten der Zellen zu ändern. Es wird notwendig sein, zusätzliche töten Kurven durchzuführen, um die Bedingungen für die Auswahl der mit zwei Antibiotika auf einmal zu optimieren. Wir können leicht erzeugen Doppel knock-downs von zwei unterschiedlichen Faktoren oder ein Knock-down eines Faktors sowie eine Überexpression eines anderen, etc.
Wir haben auch dieses Protokoll für andere B-Zell-Linie Modelle angepasst sowie. Zum Beispiel mit CH12F3-2-Zellen (eine Maus B-Zell-Linie verwendet, um CSR-Studie) 26,27, kann die gleiche Infektion Schritte zum 1 x 10 6 CH12F3-2-Zellen am Tag der Infektion ausgesät (Variante zu infizieren Schritt 4.1). Diese Zellen werden mit 1 ug / ml Puromycin selektiert. Ebenso kann das gleiche Protokoll aucheiner Zelllinie Modell für AID-initiierte Genkonversion - für Hähnchen DT40 Zellen verwendet werden.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Die pMSCV-AID-I-Thy1.1 und pKat2 Vektoren wurden freundlicherweise von DG Schatz und die pVSV-G, pRSV-Rev und pMDLg / pRRE Vektoren wurden freundlicherweise von BR Cullen.
Materialien
Empfohlene Reagenzien - die meisten von ihnen kann mit ähnlichen Produkten anderer Hersteller ersetzt werden.
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | Kommentare |
|---|---|---|---|
| 6-well klaren TC-behandelten Platten | Corning | 3516 | |
| 10 mL BD Luer-Loksyringes | BD Medical | 309604 | |
| 24-well klaren TC-behandelten Platten | Corning | 3526 | |
| 96-well deutlich flacher Boden Polystyrol TC-behandelten Mikrotiterplatten | Corning | 3596 | |
| 100 mm TC-behandelten Kulturschalen | Corning | 430167 | |
| Acrodisc Spritzenfilter, 0,45 um | Pall Life Sciences | 4604 | |
| Agar | Teknova | A7777 | |
| Agarose | GeneMate | E-3120-500 | |
| Ampicillin | Sigma | A0166 | 100 mg / mL in Wasser |
| BD FACSCanto II Durchflusszytometer | BD Biosciences | oder ähnliche | |
| BD Falcon Rundboden Polystyrol-Röhrchen | BD Biosciences | 352054 | für FACS |
| BOSC 23-Zellen | ATCC | CRL-11270 | |
| CO 2-Inkubator in der Lage 37 ° C | |||
| DMEM (Dulbecco modifiziertem Eagle-Medium) | Sigma | D6429 | |
| FBS (fötales Rinderserum) | Gemini-Bio-Produkte | 100-106 | |
| FITC α-Ratte CD90/mouse CD90.1 Antikörper | BioLegend | 202503 | FITCα-Thy1.1 |
| FuGENE 6 Transfektionsreagenz | Roche | 11814443001 | |
| HEPES-Pufferlösung | Invitrogen | 15630-080 | |
| KAPA HiFi-DNA-Polymerase | KAPA Biosystems | KK2101 | |
| LB Broth (Lysogenie Brühe - Luria) Pulver | Difco | 240230 | |
| MISSION TRC shRNA bakterielle Glycerin Lager | Sigma | shRNA-Vektoren | |
| NCS (Neugeborenen Kälberserum) | Gemini-Bio-Produkte | 100-504 | |
| PBS (Phosphat-gepufferte Lösung) | Invitrogen | 70011 | verdünnt auf 1x im Wasser |
| PE α-human-IgM-Antikörper | BioLegend | 314508 | |
| PGS (Penicillin-Streptomycin-Glutamin-Lösung) | Gemini-Bio-Produkte | 400-110 | |
| Polybrene (Hexadimethrinbromid) | Sigma | 107689 | 10 mg / mL in Wasser |
| PureYield Plasmid Midiprep-System | Promega | A2495 | |
| Puromycin | Sigma | P8833 | 250 ug / mL in Wasser |
| QIAquick Gel Extraction Kit | QIAGEN | 28706 | |
| Ramos (RA 1) Zellen | ATCC | CRL-1596 | |
| RPMI-1640 Medium | Sigma | R8758 | |
| SuperScript II | Invitrogen | 18064-022 | |
| SYBR FAST qPCR Kit | KAPA Biosystems | KK4601 | |
| Taq DNA Polymerase | Invitrogen | 18038-042 | |
| TOPO TA Cloning Kit | Invitrogen | K4520-01 | |
| TRIzol | Invitrogen | 15596-026 | |
| Wizard SV Genomic DNA Reinigungssystem | Promega | A2361 | |
| X-Gal [5-Brom-4-Chlor-3-indoyl-β-D-galatopyranoside] | Growcells | C-5687 | 40 mg / mL in DMSO |
Referenzen
- Di Noia, J. M., Neuberger, M. S. Molecular mechanisms of antibody somatic hypermutation. Annu. Rev. Biochem. 76, 1-22 (2007).
- Peled, J. U. The biochemistry of somatic hypermutation. Annu. Rev. Immunol. 26, 481-511 (2008).
- Longerich, S., Basu, U., Alt, F., Storb, U. AID in somatic hypermutation and class switch recombination. Curr. Opin. Immunol. 18, 164-174 (2006).
- Bardwell, P. D. Altered somatic hypermutation and reduced class-switch recombination in exonuclease 1-mutant mice. Nat. Immunol. 5, 224-229 (2004).
- Martomo, S. A., Yang, W. W., Gearhart, P. J. A role for Msh6 but not Msh3 in somatic hypermutation and class switch recombination. J. Exp. Med. 200, 61-68 (2004).
- Phung, Q. H. Increased hypermutation at G and C nucleotides in immunoglobulin variable genes from mice deficient in the MSH2 mismatch repair protein. J. Exp. Med. 187, 1745-1751 (1998).
- Rada, C., Ehrenstein, M. R., Neuberger, M. S., Milstein, C. Hot spot focusing of somatic hypermutation in MSH2-deficient mice suggests two stages of mutational targeting. Immunity. 9, 135-141 (1998).
- Rada, C. Immunoglobulin isotype switching is inhibited and somatic hypermutation perturbed in UNG-deficient mice. Curr. Biol. 12, 1748-1755 (2002).
- Delbos, F., Aoufouchi, S., Faili, A., Weill, J. C., Reynaud, C. A. DNA polymerase eta is the sole contributor of A/T modifications during immunoglobulin gene hypermutation in the mouse. J. Exp. Med. 204, 17-23 (2007).
- Masuda, K. DNA polymerase eta is a limiting factor for A:T mutations in Ig genes and contributes to antibody affinity maturation. Eur. J. Immunol. 38, 2796-2805 (2008).
- Lim, D. S., Hasty, P. A mutation in mouse rad51 results in an early embryonic lethal that is suppressed by a mutation in p53. Mol. Cell. Biol. 16, 7133-7143 (1996).
- Xiao, Y., Weaver, D. T. Conditional gene targeted deletion by Cre recombinase demonstrates the requirement for the double-strand break repair Mre11 protein in murine embryonic stem cells. Nucleic. Acids. Res. 25, 2985-2991 (1997).
- Zhu, J., Petersen, S., Tessarollo, L., Nussenzweig, A. Targeted disruption of the Nijmegen breakage syndrome gene NBS1 leads to early embryonic lethality in mice. Curr. Biol. 11, 105-109 (2001).
- Luo, G. Disruption of mRad50 causes embryonic stem cell lethality, abnormal embryonic development, and sensitivity to ionizing radiation. Proc. Natl. Acad. Sci. U. S. A. 96, 7376-7381 (1999).
- Pavri, R. Activation-induced cytidine deaminase targets DNA at sites of RNA polymerase II stalling by interaction with Spt5. Cell. 143, 122-133 (2010).
- Lee-Theilen, M., Matthews, A. J., Kelly, D., Zheng, S., Chaudhuri, J. CtIP promotes microhomology-mediated alternative end joining during class-switch recombination. Nat. Struct. Mol. Biol. 18, 75-79 (2011).
- Basu, U. The RNA exosome targets the AID cytidine deaminase to both strands of transcribed duplex DNA substrates. Cell. 144, 353-363 (2011).
- Yabuki, M., Fujii, M. M., Maizels, N. The MRE11-RAD50-NBS1 complex accelerates somatic hypermutation and gene conversion of immunoglobulin variable regions. Nat. Immunol. 6, 730-736 (2005).
- Sale, J. E., Neuberger, M. S. TdT-accessible breaks are scattered over the immunoglobulin V domain in a constitutively hypermutating B cell line. Immunity. 9, 859-869 (1998).
- Cumbers, S. J. Generation and iterative affinity maturation of antibodies in vitro using hypermutating B-cell lines. Nat. Biotechnol. 20, 1129-1134 (2002).
- Papavasiliou, F. N., Schatz, D. G. Cell-cycle-regulated DNA double-stranded breaks in somatic hypermutation of immunoglobulin genes. Nature. 408, 216-221 (2000).
- Parsa, J. Y. AID mutates a non-immunoglobulin transgene independent of chromosomal position. Mol. Immunol. 44, 567-575 (2007).
- Zhang, W. Clonal instability of V region hypermutation in the Ramos Burkitt's lymphoma cell line. Int. Immunol. 13, 1175-1184 (2001).
- Ukai, A. Induction of a:T mutations is dependent on cellular environment but independent of mutation frequency and target gene location. J. Immunol. 181, 7835-7842 (2008).
- Dull, T. A third-generation lentivirus vector with a conditional packaging system. J. Virol. 72, 8463-8471 (1998).
- Nakamura, M. High frequency class switching of an IgM+ B lymphoma clone CH12F3 to IgA+ cells. Int. Immunol. 8, 193-201 (1996).
- Muramatsu, M. Specific expression of activation-induced cytidine deaminase (AID), a novel member of the RNA-editing deaminase family in germinal center B cells. J. Biol. Chem. 274, 18470-18476 (1999).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten