Method Article
5-Etil-2'-Deoksiroridin/Fosfo-Histon H3 Drosophila Nöral Kök Hücrelerde Hücre Döngüsü İlerleme Analizi için Çift Etiketleme Protokolü
Bu Makalede
Özet
5-etil-2'-deoksiyridin (EdU) ve fosfo-histon H3 (pH3) etiketlemeli hücre döngüsü analizi, kapsamlı optimizasyon gerektirebilecek çok adımlı bir prosedürdür. Burada, farklı hücre döngüsü aşamalarındaki hücreleri ayırt etmek için görüntü analizi ve niceleme dahil olmak üzere bu prosedür için tüm adımları açıklayan ayrıntılı bir protokol sunuyoruz.
Özet
In vivo hücre döngüsü ilerleme analizi, mitoz ve DNA replikasyonunu düzenleyen genler üzerinde yapılan çalışmalarda rutin olarak yapılmaktadır. 5-Etil-2'-deoksiyridin (EdU) replikatif/S faz ilerlemesini araştırmak için kullanılırken, fosfo-histon H3'e karşı antikorlar mitotik çekirdekleri ve hücreleri işaretlemek için kullanılmıştır. Her iki etiketin birleşimi, G0/G1 (Boşluk fazı), S (çoğaltıcı) ve M (mitotik) fazlarının sınıflandırılmasını sağlar ve mitotik gen devirmelerinin veya boş mutantların hücre döngüsü ilerlemesi üzerindeki etkilerini değerlendirmek için önemli bir araç olarak hizmet eder. Bununla birlikte, EdU etiketli hücreleri işaretlemek için kullanılan reaktifler birkaç ikincil antikor floresan etiketiyle uyumsuzdur. Bu, birincil ve etiketli ikincil antikorların pH3-pozitif mitotik hücreleri işaretlemek için kullanıldığı immünostainingi zorlaştırır. Bu makalede, mitotik faktörleri incelemek için yoğun olarak kullanılan bir sistem olan Drosophila larva nöral kök hücrelerinde EdU ve pH3'ün çift etiketli olarak etiketlenerek kullanılması için adım adım bir protokol açıklanmaktadır. Ayrıca, etiketli hücreleri G0/G1, S, S>G2/M (S'den G2/M'ye ilerleme) ve M aşamaları olmak üzere 3 ayrı kategoride ayırmak için görüntü analizi ve niceleme için bir protokol sağlanır.
Giriş
Hücre bölünme döngüsü bir G1 fazı (ilk boşluk fazı), bir çoğaltıcı/S fazı, bir G2 (ikinci boşluk aşaması) ve bir M (mitotik) fazını içerir. Bu aşamalardan geçen hücre, sitoskeletal makinelerin hücresel transkripsiyonunda, çevirisinde ve yeniden düzenlenmesinde dramatik değişikliklere uğrar1,2. Gelişimsel ve çevresel ipuçlarına yanıt olarak, hücreler geçici olarak bölünmeyi ve sessiz hale gelebilir (G0) veya farklılaşabilir ve kalıcı olarak3bölmeyi durdurabilir. DNA hasarı gibi diğer senaryolar erken farklılaşmaya veya apoptoz3,4'eneden olabilir. Bu tür ipuçlarına yanıt, hücre bölünme döngüsünün bir sonraki aşamasına geçmeden önce temel hücresel süreçlerin bütünlüğünü sağlamak için bir gözetim sistemi görevi gören hücre döngüsü kontrol noktaları tarafından aracılık edilir5. Bu nedenle, DNA replikasyonunu düzenleyen genler, kontrol noktaları ve mitotik makineler üzerinde yapılan çalışmaların, mutant hücrelerde veya bu genlerin siRNA nakavtında oluşabilecek olası hücre döngüsü ilerleme kusurlarını analiz etmesi gerekir. Ayrıca, bu tür analizler genel hücre sağlığını ve ilaç tedavisine verilen hücresel yanıtları test etmek için kullanılabilir.
5-bromo-2'-deoksiyridin (BrdU), replikasyon sırasında DNA'ya dahil edilen bir timidin analogudur6. Bu yöntem, S fazındaki hücreleri tanımlamak için yoğun olarak kullanılmıştır. Bununla birlikte, hücreler daha sonra anti-BrdU antikorları kullanılarak BrdU'nun tespit edilmesine izin vermek için sert DNA denatürasyon prosedürlerine tabi tutulur6. Bu sert tedavi hücresel epitoplara zarar verebilir ve immünostainleme yoluyla numunenin daha fazla karakterizesini önleyebilir. EdU dahil ve daha sonra bakır katalize, 'tıklama reaksiyonu' ile küçük, hücre geçirgen, floresan etiketli azide boyaları ile tespit sert denatürasyon prosedürleri ihtiyacını ortadan kaldırır7. Bu nedenle, bu yöntem BrdU şirketleşme için daha pratik bir alternatif olarak ortaya çıkmıştır.
Ayrıca, pH3 mitotik / M faz hücreleri8için güvenilir bir işaretleyici olarak tanımlanmıştır. Histone H3, geç G2 fazında erken M fazına kadar fosforilize olan ve anafaz8'insonuna doğru fosforilasyondan arındırılan DNA ilişkili bir çekirdek histone proteinidir. Standart immünostaining protokolleri kullanılarak pH3'ün tespit edilmesi için çeşitli ticari antikorlar kullanılabilir. Bu nedenle EdU ve pH3'ün çift boyanması, S fazındaki ve M fazlı hücrelerin tespitini sağlayacaktır. Ek olarak, G1 ve erken G2 fazındaki hücreler her iki belirteç için de olumlu leke olmaz.
Drosophila nural kök hücreler veya nöroblastlar (NB'ler), hücrelerin asimetrik olarak bölünerek aynı kendini yenileyen NB ve farklılaşma için yazılmış bir ganglion ana hücre (GMC) ürettiği iyi karakterize bir kök hücre modeli sunar9. Ek olarak, çeşitli genetik araçlar ve NB'ye özgü antikorlar bu sistemi genetik manipülasyon ve canlı hücre görüntüleme için uygun hale getirir. Sonuç olarak, çeşitli çalışmalar asimetrik bölünmeleri ve hücre kaderini belirlemeyi düzenleyen genleri incelemek içinNB'lerdenyararlanmıştır 9 . Larva beyninin merkezi beyninde (CB) ve optik lobunda (OL) farklı NB popülasyonlarıvardır 9; Mevcut çalışmada CB NB'ler kullanılmıştır. Bu üçüncü instar larva CB NB'ler, mitotik mil montajlarını düzenleyen faktörleri incelemek için de uygun olan büyük hücrelerdir. Hücre döngüsü ilerleme kusurlarını analiz etmek için bir protokol bu tür çalışmalarda hayati bir araç olacaktır.
Daha önce yayınlanan protokoller, EdU dahil ve algılama10için çeşitli Alexa Fluor boyaları ile etiketlenmiş çeşitli reaksiyon bileşenleri ve azide boyaları sağlayan Click-iT EdU Alexa Fluor Hücre Çoğalma Kiti gibi ticari kitler kullandı. Bununla birlikte, bu tür kitlerle birlikte verilen reaktifler, genellikle ikincil antikorlarla kullanılan bazı floresan etiketlerle uyumlu değildir. Bu EdU tespit kiti (Alexa Fluor 647-konjuge azide boyası ile birlikte verilen Click-iT EdU Alexa Fluor Hücre Çoğalma Kiti) Drosophila üçüncü instar larva NB'lerinde test edildi ve NB'ler için bir belirteç olan pH3 ve Miranda'ya karşı antikorlarla birlikte boyama denendi. Ayrıca, NBs11'inplazma zarında Miranda etiketlemesinin tespiti için Alexa Fluor 568 veya Cy3 etiketli ikincil antikorlar kullanılmıştır. Ancak EdU saptanması sonrasında immün yetinme yapılırken bu sekonder antikorlarla beklenen sinyal yoğunluğu ve lekelenme paterni (yayınlanmamış sonuçlar) gözlenmedi.
EdU kuruluşu için, Daul ve meslektaşları tarafından açıklanan protokol, larvaların EdU ve bromofenol mavisi (BPB)10ile karıştırılmış Kankel-Beyaz ortamı ile beslenmesini gerektiriyor. Larvalar, larva bağırsağı yutulması üzerine mavi rengiyle görülebilen EdU ve BPB çivili yiyeceklerle beslenir. Bu yöntem Mms19 fonksiyon kaybı (Mms19 P)üçüncü instar larvalarında EdU dahil için kullanılmış olsa da, Mms19P larvaları görünüşe göre larva bağırsağinde neredeyse hiç mavi renk tespit edildiği için beslenmedi (yayınlanmamış sonuçlar). Mms19P larvaları sert gelişimsel deformiteler gösterir ve sonunda üçüncü başlangıç aşamasında tutuklanır. Bu bir şekilde üçüncü instar larvaların beslenme davranışını etkileyebilir ve EdU besleme protokolünü bu gibi durumlar için uygun hale getirebilir.
Mevcut literatürü inceledikten ve temel adımların standartlaştırılması üzerinde kapsamlı bir şekilde çalıştıktan sonra, Drosophila NBs'de EdU/ pH3 çift etiketleme için alternatif bir yaklaşım önerildi, bu da EdU'yu larvalara beslemeyi gerektirmez. Önceki bir çalışmada, NB'lerdeki hücre döngüsünü analiz etmek için çift EdU / pH3 boyama kullanıldı, ancak ayrıntılı bir protokol sunmadı4. Bu, bu yöntemi uygulamaya çalışan laboratuvarlar için gereksiz bir engel sunar. Ayrıca, çeşitli reaktiflerin EdU kiti ile uyumluluğunu değerlendirmek ve daha fazla optimizasyon yapmak zaman alıcı bir süreç olabilir. Bu makale, parçalanmış larva beyinlerinde EdU birleşmesini ve anti-pH3 antikorları ile immünostainingi kapsayan adım adım bir protokol sunar, ardından NB'leri dört ayrı kategoriye ayırmak için konfokal mikroskopi ve görüntü analizi: G0/G1 fazı, S fazı, S>G2/M (S'den G2/M'ye ilerleme) ve M fazı. En iyi duruma getirilmesi gereken adımlar özetlenir ve büyük veri kümelerinin görüntü analizi için ipuçları sağlanır. Ek olarak, vahşi tip NB'lerdeki EdU/pH3 okuması analiz edilir ve yakın zamanda hücre döngüsü gecikmesi gösterdiği bildirilen Mms19P NB'lerle karşılaştırılır11.
Protokol
1. Click-it EdU tahlilleri için reaktiflerin ve stokların hazırlanması
NOT: Kit ve kit ile birlikte verilen reaktifler hakkında ayrıntılı bilgi için Malzeme Masası ve Tablo 1'e bakın.
- Çözümleri hazırlamadan önce şişeleri oda sıcaklığına getirin.
- 2 mL dimetilsüllfoksit (DMSO, bileşen C) ekleyerek 10 mM EdU (bileşen A) stok çözeltisi hazırlayın. İyice karıştırın ve -20 °C'de saklayın.
- 70μL DMSO (bileşen C) ekleyerek Alexa Fluor 647-azide (bileşen B) çalışma çözeltisi hazırlayın. İyice karıştırın ve -20 °C'de saklayın.
- Bu çözeltinin 4 mL'lik kısmını 36 mL deiyonize su ile karıştırarak 1x Click-iT EdU reaksiyon tamponu (bileşen D) hazırlayın. Kalan çözeltiyi 2-6 °C'de saklayın.
- 2 mL deiyonize su ekleyerek Click-iT EdU tampon katkı maddesinin (bileşen F) 10x stokunu (200 mg/mL) yapın. İyice karıştırın ve -20 °C'de saklayın.
- Hoechst 33342'i (G bileşeni) 2-6 °C'de saklayın. DMSO'yu (bileşen C) -20 °C'de bir kurutucuda saklayın.
NOT: Eldivenler DMSO ve Hoechst'i idare etmek için kullanılmalıdır.
2. Üçüncü instar larva beyinlerinin diseksiyonu ve EdU şirketleşmesi
NOT: Beyin diseksiyonları protokolü daha önce tanımlanmıştır12. Diseksiyonlara başlamadan önce, 2.6 ve 3.1'de açıklandığı gibi yeterli miktarda EdU ve PFA çözeltisinin hazırlandığından ve çözüldüğünden emin olun.
- 25,6 g Na 2 HPO 4,7H 2 0, 80 g NaCl,2g KCl ve2gKH2 PO4 ila 1 L deiyonize su ekleyerek 10x Fosfat tamponlu salin (PBS) hazırlayın. pH'ı 7.4'e ayarlayın ve daha sonra deiyonize suda 1x'lik bir çözelti hazırlayın.
- Schneiders diseksiyon ortamını (SM) bir camın ardışık iki çökemine (3 veya 9) çöken cam spot plaka ekleyin. Bir sonraki ardışık depresyona 1x PBS ekleyin.
- Bir çift toparlama kullanarak, dolaşan üçüncü instar larvalarını seçin ve sinek yiyecek kalıntılarını temizlemek için PBS ile ıslanmış bir dokuya yerleştirin. Larvaları PBS ile depresyona ve daha sonra SM ile ardışık depresyona yerleştirin.
- Bir çift ince kümes gücü kullanarak, larvaların alt gövdesinin3/4'lerini çıkarın.
- Larva ağız kancalarını bir çift ön uçla hafifçe tutun ve diğer ucundaki manikürleri diğer çift asalarla tutun.
- Ağız kancalarını içe doğru iterek ve aynı anda diğer ucundaki dokuyu soyarak larva başını ters çevirin.
- Ekli hayal diskleri ile larva beynini gözlemleyin. Beyne bağlı diğer dokuları çıkarın ve beyni SM ile bir sonraki ardışık depresyona aktarın.
- 10 mM EdU stok çözümünü çözün. Bu 10 mM stoğu SM'de seyrelterek 100 μM'lik bir çözelti hazırlayın.
- Pyrex plakasındaki başka bir çökemde bu 100 μM EdU+SM çözeltisinin 100 μL'sini ekleyin. 25 °C'de 2 saat boyunca 100 μL 100 μM EdU+SM çözeltisinde ~5-10 parçalanmış beyni kuluçkaya yatırın.
NOT: NB'ler ~2 saat içinde bir kez bölündükçe, bu protokol bir tam hücre döngüsünü analiz etmeyi amaçlamaktadır. Sadece daha ileri adımlar için sağlam beyinler kullanın. Diseksiyon sırasında hasar gören beyinleri atın.
3. Fiksasyon ve immünostaining
- Duman kaputunda %4 paraformaldehit (PFA) çözeltisi hazırlayın.
- Manyetik bir karıştırıcıya yerleştirilen bir behere 80 mL 1x PBS'ye 4 g paraformaldehit tozu ekleyin ve 60 °C'ye ısıtın. PFA'yı çözmek için birkaç damla 1 N NaOH ekleyin. pH'ı kontrol edin ve gerekirse birkaç damla 1 M HCl ile 7.0'a ayarlayın.
- 1x PBS ile ses seviyesini 100 mL'ye ayarlayın. PFA çözeltisini aliquot ve 2-6 ° C'de 1 aya kadar saklayın. Larva beyni gibi büyük dokuların verimli bir şekilde sabitlenmesi için PFA çözümüne% 0.3 iyonik olmayan deterjan ekleyin (Malzeme Tablosunabakın).
- EdU dahil edildikten sonra, beyinleri% 4 PFA içeren 0.6 mL mikrosantrifüj tüplerine aktarın. 15 dakika boyunca bir besleyici üzerinde oda sıcaklığında kuluçkaya yatırın. Laboratuvar tezgahındaki tüpe dokunun, böylece beyinler tüpün altına yerleşir.
- PFA'yı çıkarın ve oda sıcaklığında PBS + %0,3 iyonik olmayan deterjan (PBST) ile 3 kez yıkayın. Her yıkamanın bir besleyicide en az 10 dakika sürdüğüne emin olun. Son yıkamadan sonra PBST'yi çıkarın, blokaj çözeltisi ekleyin (PBST + % 5 sığır serum albümini) ve oda sıcaklığında 30 dakika kuluçkaya yatırın.
- PBST'de Anti-Miranda antikor (1:250) ve anti-pH3 antikorlarını (1:200) seyreltin (her ikisi de aynı tüpteki antikorlar). Engelleme tamponu çıkarın ve birincil antikor çözeltisini ekleyin. Bir besleyici üzerinde 2-6 ° C'de gece boyunca kuluçkaya yatırın.
NOT: Miranda, CB NB'lerinde bulunan bir membran proteinidir. CB bölgesindeki bu büyük yuvarlak hücreleri tanımlamaya yarar13. - Birincil antikor çözeltisini çıkarın ve PBST ile 3 kez yıkayın. Her yıkamanın oda sıcaklığında bir somun makinesinde 10 dakika sürdüğüne emin olun.
- PBST'ye 1:500 seyreltilmiş Alexa Fluor 488 anti-Rabbit ve Alexa Fluor 568 anti-Rat ekleyerek ikincil antikor çözeltisi hazırlayın. PBST'yi çıkarın ve incelenmiş beyinlere ikincil antikor çözeltisini ekleyin. Karanlıkta 2-6 °C'de, bir besleyicide gece boyunca kuluçkaya yatırın ve PBST ile 3 kez yıkayın (adım 3.5).
4. EdU tespiti, DNA boyama ve montaj
- EdU algılama kokteylini hazırlamak için, bileşenleri 1,5 mL'lik bir tüpte karıştırın (bkz. Tablo 2).
- Son yıkamadan sonra (adım 3.6), PBST'yi çıkarın ve EdU algılama kokteylini parçalanmış beyinlere ekleyin. Bir fındık makinesinde 30 dakika karanlıkta oda sıcaklığında kuluçkaya yatırın. PBST ile her biri 10 dakika boyunca karanlıkta 2 kez yıkayın.
- DNA boyama için, 5 μg/mL çözeltisi hazırlamak için PBST'de Hoechst 33342 (G bileşeni) 1:2.000'i seyreltin.
- İkinci yıkamadan sonra PBST'yi çıkarın (adım 4.2) ve parçalanmış beyinlere 500 μL 5 μg/ mL Hoechst çözeltisi ekleyin. Karanlıkta 10 dakika kuluçkaya yaslanın. Hoechst'i çıkarın ve PBST ile bir kez 10 dakika yıkayın.
- Tüpü laboratuvar tezgahına dokunun ve beyinlerin sakinleşmesine izin verin. PBST'yi tüpten çıkarın, ancak geride sadece 50-100 μL bırakın.
- 200 μL'lik bir mikropipette ucun ucunu kesin ve beyinleri dikkatlice temiz bir cam kaydırağa aktarın. Filtre kağıdı şeritleriyle şişirerek slayttan fazla PBST'yi çıkarın. Filtre kağıdının beyinlere dokunmasına izin vermemeye dikkat edin.
- Bir damla suda çözünür, floresan olmayan montaj ortamını beyinlere koyun ve beyinleri, ventral sinir kordonu kaydırağa, loblar yukarı bakacak şekilde yönlendirin. Beyinleri tek bir dosyada düzenleyin, böylece beyinleri seri olarak görüntüleyebilme daha kolaydır.
- Beyinlerin üzerine hafifçe bir kapak kılıfı yerleştirin. Slaydı gece boyunca 2-6 °C'ye yerleştirin.
NOT: Montaj ortamı gece depolamada sertleşir, böylece kapak kapağını oje ile kapatmaya gerek yoktur.
5. Görüntüleme
NOT: Bu protokolde kullanılan lazer tarama mikroskobu ve yağ daldırma hedefi hakkında ayrıntılı bilgi için Malzeme Tablosuna bakın.
- Edinme yazılımından 63x hedefiniseçin.
- Dokuyu göz merceğinden bulmayı kolaylaştırmak için monte edilmiş beyinlerin hemen üstündeki kapak kapağına bir damla daldırma yağı koyun.
- DAPI/Hoechst 33342 kanalını kullanarak beyni göz merceğinden bulun ve ardından yazılımdaki alım moduna geçin.
- Hoechst 33342 (DNA), Alexa Fluor 488 (pH3), Alexa Fluor 568 (Miranda) ve Alexa Fluor 647 (EdU) görüntü için 4 kanal ayarlayın. Seçilen boyalar için heyecan lazerlerini ve emisyon filtrelerini otomatik olarak ayarlayan boya asistanı aracını kullanın.
- Görüş alanını tüm beyin lobını kapsayacak şekilde ayarlayın. 0,8 μm aralıklı z yığınları elde ederek beyin lobunun tüm hacmini görüntüleyin. Görüntüleme oturumundaki tüm görüntüleri *.lif kitaplığı biçiminde depolayın.
6. Görüntü analizi
NOT: Aşağıdaki adımda, elde edilen görüntülerin analizi ve hücrelerin G0/G1 fazı, S fazı, S>G2/M (S'den G2/M'ye ilerleme) ve ImageJ yazılımını kullanarak M fazına nasıl sıralanılacağı açıklanmaktadır.
- Fiji'yi (Fiji, ImageJ) aşağıdaki URL'den indirin: https://fiji.sc/. Fiji'yi açın, sonra .lif dosyalarını sürükleyip Fiji'ye bırakın.
NOT: Fiji, imagej'ın önceden yüklenmiş olarak gelen bir sürümüdür14. .lif dosyalarını Fiji'ye aktarmak, .lif dosyalarını işlemek için gereken Bio-formatları eklentisini açacaktır. Nikon mikroskobundan oluşturulan nd2 dosyaları gibi diğer bazı mikroskop markalarından oluşturulan görüntü dosyalarını açmak için biyo-formatlar eklentisi de gereklidir. Bu eklenti Fiji ile önceden yüklenmiş olarak gelir. - Yığın görüntüleme sekmesinden Veri tarayıcısı'nı seçin ve biyo-formatlar eklentislerindeki bellek yönetimi sekmesinden sanal yığını kullanın.
NOT: 8-10 beyinden z-yığınları olan Lif dosyaları genellikle 300-400 megabayt boyutundadır. Düşük RAM'li bilgisayarlarda, bu tür birkaç dosyayı açmak ImageJ'deki mevcut RAM'i hızla düşürebilir ve daha fazla görüntü işlenmesini önleyebilir. Sanal yığın 'salt okunur', işleme için yüksek RAM'e ihtiyaç duymaz ve fiji'ye büyük veri kümeleri yüklemek için ideal bir seçenektir. - ImageJ'de görüntülenen çok kanallı görüntüyü gözlemleyin. Görüntü | renk | kanalları aracını kullanarak menü çubuğundan kanalların renginideğiştirin. Miranda etiketli NB'leri CB bölgesindeki büyük yuvarlak hücreler olarak gözlemleyin.
- NB'yi iki kez saymaktan kaçınmak için her NB'nin üzerine elips aracını kullanarak bir ilgi alanı (ROI) çizin.
- ImageJ menü çubuğundan | araçları çözümle | Yatırım getirisi yöneticisi. Geçerli z bölümündeki tüm NB'leri işaretleyin ve her hücreyi işaretledikten sonra t tuşuna basın.
- Geçerli z bölümündeki tüm NB'ler işaretlendikten sonra, kanalları pH3 ve EdU olarak değiştirin ve EdU pozitif NB'lerin, pH3 pozitif NB'lerin, hem EdU hem de pH3 için pozitif boyama NB'lerin sayısını ve her iki işaretleyici için de NB'lerin lekelenmediğini manuel olarak sayın.
- Sonraki z bölümlerinde NB'leri arayın, eski ROI'leri silin ve NB'leri farklı yığınlarda saymak için yeni ROI'ler ekleyin.
- Aşağıdaki sütunlarla bir elektronik tablo hazırlayın: 1. EdU- pH3-: Bu bölüm, hücre döngüsünün G0/G1 aşamasında bulunan çift negatif hücreleri temsil eder; 2. EdU+: bu kategorideki hücreler EdU'yu bünyesine katmıştır ve DNA replikasyonu (S fazı) geçirmektedir; 3. EdU+ pH3+: bu çift pozitif hücreler S fazını tamamlamış ve G2 veya M fazı aşamasına ilerlemiş; 4. pH3+: Bu NB'ler mitoz geçiriyor.
- Her lob için yukarıdaki 4 kategorinin tümlerinde bulunan NB'lerin yüzdelerini hesaplayın. Her kategorideki NB yüzdeleri için havuza alınan verileri gösteren elektronik tablo yazılımını kullanarak bir çubuk grafik hazırlayın.
Sonuçlar
İki loblu Drosophila üçüncü instar larva beyni, temel hücresel ve gelişimsel süreçleri incelemek için bir model sistemi olarak kullanılmıştır9. Mevcut çalışmanın odak noktası, CB bölgesinin EdU ve pH3 etiketli NB'lerinde hücre döngüsü ilerlemesinin analizi için bir protokol sunmaktı (Şekil 1). CB NB'leri tip I ve tip II'ye alt olarak ayrılmıştır ve karakteristik asimetrik bölme deseni9'u görüntülerler. Her tip I NB bölümü, kendini yenileyebilen bir NB ve farklılaşma için kaderi olan başka bir GMC oluşturur9. Buna karşılık, tip II NB'ler, kendi kendini yenileyen ve 2 GMC9üreten transit yükseltici, olgunlaşmamış nöral progenitör hücreler (INP'ler) oluşturur. OL bölgesinde belirgin bir NB popülasyonu olmasına rağmen, bu çalışma NB'ye özgü belirteçlerle büyük ve kolayca tanımlanabilen CB NB'lere odaklanmıştır.
Bu çalışma için Miranda CB NB'leri tanımlamak ve analiz etmek için kullanılmıştır (Şekil 2). Miranda ol diğer hücre popülasyonlarını da lekelese de, CB NB'leri büyük boyutları ve CB konumları nedeniyle Miranda ile tanımlanabilir13. Diğer daha spesifik belirteçler, NB kendini yenilemeyi düzenleyen bir transkripsiyon faktörü olan Deadpan gibi CB NB'leri etiketlemek için de kullanılabilir. Bununla birlikte, hem pH3 hem de EdU kromatin işaretledikçe, pH3 ve EdU ile işaretlenmiş NB'lerin görselleştirilmesini ve analizini kolaylaştırmak için miranda memb işaretleyicisi kullanılmıştır. EdU şirketleşmesi, sonraki tespit ve immünostaining ve görüntü analizi ile daha fazla karakterizasyon karmaşık bir prosedürdür. Optimum boyama ve görüntüleme koşullarını belirlemek için her adımı standartlaştırmak önemlidir. Yayınlanan bazı raporlar EdU/pH3 etiketleme stratejileri sunsa da, bu raporlar optimizasyon ve görüntü analizi adımları hakkında ayrıntılı bilgi vermiyor. Şekil 1'de özetlenen iş akışı, EdU birleştirilmesinden görüntü analizine kadar olan adımları özetler.
Son zamanlarda normal mitotik ilerleme için NB'lerin gerektirdiği Mms19 geni karakterize ettik11. Canlı hücre görüntüleme analizleri sayesinde, fonksiyonel Mms19'dan yoksun NB'lerin mitozun bitmesi için vahşi tip NB'lerin iki kat daha uzun sürdüğü gösterilmiştir. Bu, EdU / pH3 analizine açıkça yansımıştır ve burada mms19P NB'lerin önemli ölçüde daha yüksek bir oranının M aşamasında olduğu bulunmuştur vahşi tip NB'lere kıyasla ( Şekil3A). Mms19::eGFP füzyon proteininin Mms19P arka planında ifadesi fenotipik kusurları kurtardığıgösterilmiştir 11,15. Bu, M fazındaki hücrelerin oranının Mms19::eGFP ifadesinde Mms19 P arka planında vahşi tip seviyelere kurtarıldığı hücre döngüsü ilerleme analizinin sonuçlarıyla iyi ilişkiliydi (Şekil 3A,B).

Şekil 1: EdU/pH3 çift etiketleme için basitleştirilmiş bir iş akışı. Üçüncü instar larva beyinleri 2 saat boyunca 100 μM EdU ile parçalandı ve inkübe edildi. Daha sonra, beyin dokusu PFA ile sabitlendi ve Miranda ve pH3'e karşı antikorlarla immünostain edildi. Lekeli beyinlerin görüntüleri konfokal mikroskopta elde edildi ve hücreleri farklı hücre döngüsü aşamalarına tahsis etmek için ImageJ / Fiji ile daha fazla işlendi. Kısaltmalar: EdU = 5-etil-2'-deoksiyridin ile; pH3 = fosfo-histon H3; PFA = paraformaldehit; SM = Schneiders diseksiyon ortamı; PBS = fosfat tamponlu salin; PBST = PBS + %0,3 iyonik olmayan deterjan; BSA = sığır serum albümin. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
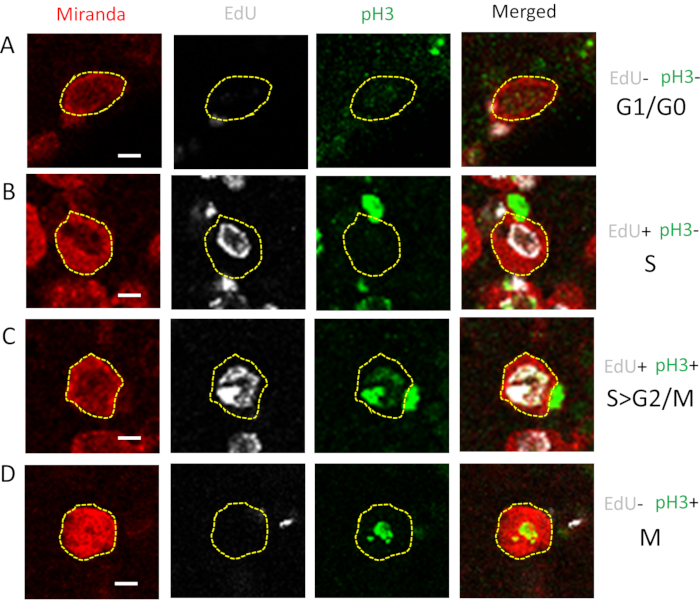
Şekil 2: Hücrelerin G1/G0'a Tahsisi; S; S>G2/M ve M fazları. Hücreler tam hücre döngüsünü tamamlamak için G1, S, G2 ve M aşamalarında ilerler. Üçüncü başlangıç larva beyinlerinden CB NB'leri Miranda (kırmızı), EdU (gri), pH3 (yeşil) ve DNA Hoechst 33342 (mavi) ile etiketlendi. Etiketli NB'ler ImageJ'de analiz edildi ve(A) ne EdU ne de pH3 (G1/G0), (B) sadece EdU (S fazı), (C) hem EdU hem de pH3 (S>G2/M) ve (D) sadece pH3 (M fazı) için pozitif lekelenip lekelenmediklerine bağlı olarak 4 ayrı faza ayrıldı. Tek tek NB'lerin yalnızca vahşi tip beyinlerden örnekleri burada gösterilmiştir. Ölçek çubukları = 5 μm. Kısaltmalar: CB = merkezi beyin; NB'ler = nöroblastlar; EdU = 5-etil-2'-deoksiyridin ile; pH3 = fosfo-histon H3. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
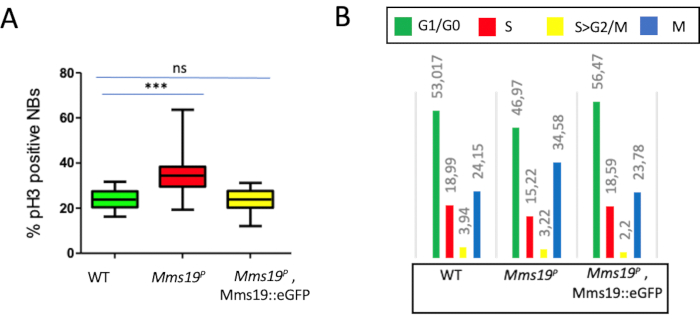
Şekil 3: Mms19P'nin NB hücre döngüsü faz dağılımına etkisi. (A) Yabani tip (w;+;+) NB'lerin analizi üzerine~ %25 NB'nin M fazında olduğu bulunmuştur. Bununla birlikte, MMS19P NB'lerde bu oran neredeyse% 40'a yükseldi. Mms19::eGFP ifadesi Mms19P arka planında Mms19P fenotiplerini kurtardığı bilinmektedir ve bu arka plandaki M faz NB'lerin oranı vahşi tiple karşılaştırılabilirdi. (B) G1/G0, S, S>G2/M ve M hücre döngüsü aşamalarına karşılık gelen 4 farklı kategorideki NB'lerin yüzdeleri vahşi tipte karşılaştırıldı; Mms19Pve Mms19::eGFP, Mms19P genotipleri. M fazı ve G1/G0 fazlarının oranı, mms19P NB'lerde vahşi tip NB'lere kıyasla önemli ölçüde farklılık gösterir. Buna karşılık, NB'lerin Mms19::eGFP, Mms19P beyinlerindeki hücre döngüsü dağılımı vahşi tipte bulunanla karşılaştırılabilir. İstatistiksel önem, Kruskal-Wallis testi ve Dunn'ın test sonrası ile karşılaştırıldığında birden fazla sütun kullanılarak hesaplandı; (S<0.001). Bu rakam 11'den değiştirilmiştir. Kısaltmalar: NB'ler = nöroblastlar; eGFP = gelişmiş yeşil floresan protein; WT = vahşi tip. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
| Reaktif | Tutar/hacim |
| EdU (bileşen A) | 5 mg |
| Alexa Fluor 647 – azit (bileşen B) | 1 şişe |
| Dimetilsüllfoksit (DMSO, bileşen C) | 4 mL |
| Click-iT EdU reaksiyon arabelleği (bileşen D) | 4 mL (10x çözelti) |
| Bakır sülfat (CuSO4, bileşen E) | 100 mM; 1 şişe |
| Click-iT EdU arabellek katkı maddesi (bileşen F) | 400 mg |
| Hoechst 33342 (G bileşeni) | Suda 10 mg/mL, 35 μL |
Tablo 1: EdU kit bileşenleri. Click-iT EdU Alexa Flor Hücre Çoğalma Kiti ve ilgili miktarlar/hacimler ile sağlanan bileşenler. Kısaltma: EdU = 5-etil-2'-deoksiyridin ile.
| Reaksiyon bileşenleri | Birim (mL) |
| 1x Click-iT reaksiyon tamponu (adım 1.4'te hazırlanmıştır) | 430 |
| CuSO4 (bileşen E) | 20 |
| Alexa Fluor 647 – azide (B bileşeni, adım 1.3'te hazırlanmıştır) | 1.2 |
| Reaksiyon tampon katkı maddesi (F bileşeni, adım 1.5'te hazırlanmıştır) | 50 |
| Toplam birim | ~500 |
Tablo 2: EdU algılama kokteylinin hazırlanması. Kit bileşenlerinin belirtilen hacimleri karıştırılarak EDU algılama kokteyli hazırlanmıştır (bölüm 2'de hazırlanmıştır). Kısaltma: EdU = 5-etil-2'-deoksiyridin ile.
Tartışmalar
EdU şirketleşme ve hücre geçirgen azide ile sonraki 'tıklama' reaksiyonu, bu tekniğin daha önce kullanılan BrdU yöntemine göre pratik avantajlarını sunar7. Bununla birlikte, bu reaksiyon Cu(I) iyonları tarafından katalizöre edilir ve Click-it EdU kit üreticisi tarafından açıkça tavsiye edildiği gibi, bu bakır katalizörün varlığında birkaç boya kararsız olabilir. EdU algılama adımının gerçekleştirilmesinden sonra immünostaining deneyleri yapıldığı zaman, kırmızı kanal boyaları, Alexa Fluor 568 ve Cy3 ile beklenen sinyal yoğunluğu gözlenmedi. Ancak protokol, EdU tespit adımından önce immün yetinme reaksiyonları tamamlandığında işe yaradı. Bu protokol ile Hoechst, pH3, Miranda ve EdU'yu görselleştirmek için dört kanal kullanıldı.
Bu protokolde optimize edilmiş bir diğer adım, EdU çivili sinek yiyeceklerini larvalara beslemenin aksine, EdU'nun parçalanmış beyinler tarafından alınmasıydı. Tam Drosophila NB hücre döngüsü yaklaşık 2-2.5 h16için sürdüğünden, EdU bu protokolde olduğu gibi 2.5 h beyinler tarafından alımı, NB'lerin bir tam hücre döngüsünün izlenmesini sağlar. NB'lerin aktif olarak bölündüğü üçüncü bir instar larva durumunda, bu dinlenme hücreleri büyük olasılıkla hücre döngüsünün G1 aşamasında olacaktır. Bununla birlikte, geç embriyonikten erken ikinci başlangıç aşamalarına kadar, NB'ler geçici olarak sessiz hale gelir17. Bu erken gelişim evrelerinde, çift negatif lekeleme büyük olasılıkla bir G0/ sessiz fazı temsil edecektir.
DNA replikasyonundan geçen NB'ler EdU için pozitif lekelenmeli, S fazından (EdU inklüzyonu) geç G2 veya M fazı için ilerleyen hücreler ise hem EdU hem de pH3 için pozitif lekelenmelidir. Bununla birlikte, erken G2 hücreleri EdU veya pH3 için olumlu lekelenmediği için G2 fazının karakterizasyonu bu protokolle zor olacaktır. Bununla birlikte, EdU'yu birleştiren ve erken G2'ye ilerleyen hücreler de EdU için olumlu lekeler ve G2 fazının analizini zorlaştırabilir. DNA replikasyonını engelleyen birkaç ilaç S faz18 , 19,20'dehücre döngüsüdurmasınaneden olur. Bu protokol, S faz bloke edilmiş hücrelerin M fazı ilerlemesi ve EdU, pH3 çift pozitif hücrelerin fraksiyonu bu durumda azalacağı ve hatta bulunmadığı için bu tür ilaç tedavilerinin etkisini analiz etmek ve tanımlamak için uygun bir araç olabilir.
Yakın zamanda yayınlanan bir çalışmada, Mms19 geninin NB'lerde mitotik ilerleme üzerindeki etkisini değerlendirdik11. Canlı hücre görüntüleme sayesinde, M fazlı ilerlemede Mms19P NB'lerin dramatik bir gecikmesini gösterdik. Vahşi tip NB'ler M fazını ~10 dakikada tamamlarken, Mms19P NB'lerin Mfazı 11'ibitirmesi yaklaşık iki kat zaman aldı. Buna göre, bu EdU/pH3 çift etiketleme protokolü, mms19P beyinlerinde pH3 için vahşi tip beyinlere kıyasla pozitif olarak lekelenen NB'lerin neredeyse 1,5 kat kadar olduğunu gözlemlediğimiz için mitotik bir gecikmeyi de yansıttı. Bu EdU protokolünden elde edilen veriler bu nedenle doğrudan canlı hücre görüntüleme sonuçlarıyla karşılaştırılabilir. Bu tür doğrudan, canlı hücre yaklaşımları zaman alıcıdır ve kapsamlı optimizasyona ihtiyaç duyar, bu protokol, belirli aşamalardaki kusurları anlamak için bu tür hücre döngüsü mutantlarının hızlı bir ilk taramasını gerçekleştirmek için kullanılabilir. İlginin bir aşaması belirlendikten sonra, bu daha sonra doğrudan hücre görselleştirme tahlilleri ile takip edilebilir.
Hücre döngüsü evrelerinin süresi farklı hücre tipleri ve gelişim evreleri arasında önemli ölçüde değişebilir. Örneğin, Drosophila embriyolarının geliştirilmesinde, mitotik kinazların küçültülmesi S fazının uzun bir süresiyle sonuçlanırken, gelincik (Mustela putorius furo) beyin nöral progenitörleri gibi farklılaşma için yağlanmış hücrelerde, S faz süresi belirgin şekilde kısadır21,22. EdU darbe etiketleme süresi, verilen hücre tipindeki S ve G fazlarının uzunluğuna bağlı olarak optimize edilmelidir. EdU darbesinin süresi deneysel hedeflere de bağlı olacaktır, örneğin, kısa bir darbe şu anda S fazında bulunan hücrelerin fraksiyonunu ölçmek için yeterlidir, daha uzun bir darbe ise hücrelerin S fazı boyunca ilerlemesinin analizini sağlar.
EdU darbesinin kapsamlı optimizasyonu, Pereira ve meslektaşları tarafından zarif bir şekilde gösterildiği gibi S fazının tahmin edilmesine de izin verebilir23. Bu araştırmacılar, HCT116 hücrelerinin artımlı zaman dilimleri için EdU ile nabız etiketli olduğu yeni, akış sitometrisi tabanlı bir yöntem göstermiştir. Yazarlar, EdU'nun maksimum floresan yoğunluğunun, titreşim süresi S faz23'ünuzunluğuyla eşleştiğinde elde edildiğini göstermiştir. Ayrıca, EdU etiketli hücrelerin zamansal ilerlemesinin analizi, G1 ve G2/M fazlarının nicelleştirilmesini de sağladı. EdU/pH3 çift etiketleme için mevcut protokol S ve M faz kusurlarının analizini mümkün olsa da, bu protokol ile aşamaların kesin süresini ölçmek mümkün değildir. Pereira ve meslektaşları tarafından açıklanan protokol, hücre döngüsü faz uzunluklarının belirlenmesini gerektiren tahliller için uygun bir alternatif olabilir. Bu protokolün ek bir pH3 etiketlemesi ile uyarlanmıştır, bu da M faz hücrelerini algılamada daha yüksek hassasiyete neden olabilir.
EdU/pH3 yaklaşımının yanı sıra, memelihücrelerinde ve Drosophila dokularında hücre döngüsü ilerlemesini incelemek için f luorescent ubiquitin tabanlı hücre döngüsü göstergesi (FUCCI) yöntemi de kullanılmıştır24,25. Bu sistem, sırasıyla APC/C ve SCFSkp2tarafından spesifik her yerde bulunma ve proteazomal bozulma motifleri içeren geminin ve Cdt1 olmak üzere floresan etiketli iki protein kullanır. APC/C sadece mitozun sonundan G1'e kadar aktif olduğundan ve SCFSkp2 S ve G2 aşamalarında aktif olduğundan, floresan etiketli geminin ve Cdt1'in hücre döngüsü aşamasına özgü bozulması hücre döngüsüfazı 24'ünbelirlenmesini sağlar. Drosophila dokuları için biraz değiştirilmiş bir 'Fly-FUCCI' yöntemi, bunun yerine, sırasıyla APC / C (mitoz sırasında) ve CRL4Cdt2 (S fazı başlangıcında) tarafından bozulan floresan etiketli Cyclin B veE2F1'edayanır. Drosophila larva NB'lerin anöploidiye yanıt olarak erken farklılaştırılmasını karakterize eden önceki bir çalışma, hem Fly-FUCCI hem de EdU / pH3 yöntemleri ile hücre döngüsü kusurlarını analiz etti4. Aneuploidi NB'lerin erken farklılaşmasına neden olur ve bu nedenle NB'lerin yüksek bir kısmı hücre döngüsünden çıkar.
Bu hem Fly-FUCCI hem de EdU / pH3 verilerine doğru bir şekilde yansıdı4. Bu nedenle, EdU/pH3 yönteminden elde edilen veriler, Fly-FUCCI gibi diğer hücre döngüsü izleme yöntemleriyle de karşılaştırılabilir. Fly-FUCCI güçlü bir araçtır ve dokuya özgü organizatörlere kaynaşmış her yerde sürülen floresan işaretleyicileri veya işaretleyicileri olan tüm sinek stokları stok merkezlerinden temin edilebilir. Bununla birlikte, bu yapıları boş mutantlardaki hücre döngüsü kusurlarını analiz etmek için veya siRNA nakavt ile kullanmak, FUCCI elementlerini, dokuya özgü sürücüleri, belirli bir mutasyonu veya ilgi çekici bir siRNA'yı taşıyan bir sinek stoğu oluşturmak için genetik rekombinasyon içerecektir. Bu işlem gerçek deneyi birkaç hafta geciktirirken, EdU/pH3 yöntemi genetik boş arka plana sahip uçucu hatlara kolayca uygulanabilir. Alternatif olarak, siRNA nakavtları için, EdU / pH3 analizi için bir sürücü stoğu ile siRNA stoğu arasında tek adımlı bir haç kullanılabilir.
Bu yaklaşımın bir dezavantajı, birkaç beyin örneğinden çok sayıda hücrenin manuel olarak ölçülmesini içeren görüntüleme ve görüntü analizi işlem hattıdır. Son zamanlarda, akış sitometrisi yaklaşımı, hücre döngüsü analizi için geleneksel mikroskopi tabanlı bir teste yüksek verimli bir alternatif olaraksunulmuştur 26. Bu yaklaşımla binlerce hücrenin aynı anda hızlı analizi avantajlıdır ve birden fazla belirteci tespit etme yeteneği belirli hücre türlerinin nicelleştirilmesini sağlar. Kültürlü hücre hatlarıyla kolayca kullanılabilir olmasına rağmen, bu protokolün Drosophila larva beyni gibi büyük doku örneklerine uygulanması zor olabilir. Bununla birlikte, beyin dokusu ayrışmasını ve izole larva NB'lerin akış sitometrisi27 , 28ile analizini açıklayan birkaç protokol yayınlanmıştır. Bu yöndeki daha yenilikçi yaklaşımlar, Drosophila dokularında yüksek verimli hücre döngüsü analizi için yeni fırsatlar sunabilir.
Açıklamalar
Yazarlar rakip çıkarların olmadığını açıkladılar.
Teşekkürler
Bu çalışma, İsviçre Ulusal Bilim Vakfı (proje hibe 31003A_173188; www.snf.ch) ve Bern Üniversitesi'nden (www.unibe.ch) BS'ye fon sağlanmasıyla desteklendi. Fon sağlayıcılar çalışma tasarımı, veri toplama ve analiz, yayınlama kararı veya makalenin hazırlanmasında hiçbir rol oynamamışlardır.
Malzemeler
| Name | Company | Catalog Number | Comments |
| fly stocks | |||
| P{EPgy2}Mms19EY00797/TM3, Sb1 Ser1 (Mms19p) | Bloomington stock center | #15477 | P-element insertion in the third exon of Mms19 |
| +; Mms19::eGFP, Mms19p | Generated in house | eGFP-tagged Mms19 protein expressed in Mms19p background | |
| w1118 | Bloomington stock center | #3605 | wild-type stock (w;+;+) |
| Primary antibodies | Dilution | ||
| Rat anti-Miranda | Abcam | Ab197788 | 1/250 |
| Rabbit anti-pH3 | Cell Signaling | 9701 | 1/200 |
| Secondary antibodies | |||
| Goat anti-Rat Cy3 | Jackson Immuno | 112-165-167 | 1/150 |
| Goat anti-Rat Alexa Fluor 568 | Invitrogen | A11077 | 1/500 |
| Goat anti-Rabbit Alexa Fluor 488 | Invitrogen | A27034 | 1/500 |
| Reagent/Kit | |||
| Aqua Poly/Mount mounting medium | Polysciences Inc | 18606-20 | |
| Click-it EdU incorporation kit, Alexa Flour 647 | Thermo Fischer Scientific | C10340 | |
| Schneider’s Drosophila medium | Thermo Fischer Scientific | 21720-024 | |
| Bovine serum albumin (BSA) fraction V | Merck | 10735078001 | |
| Triton X-100 | Fischer Scientific | 9002-93-1 | non-ionic detergent |
| Software | |||
| Fiji (Imagej) | https://imagej.net/Fiji | ||
| Leica Application Suite (LAS X) | Leica microsystems | ||
| PRISM | Graph pad software | Version 5 | |
| Microsoft Excel | Microsoft office | 2016 | |
| Equipment | |||
| Leica TCS SP8 laser scanning confocal microscope with 63x oil-immersion, 1.4 NA Plan-apochromat objective | |||
| Materials | |||
| Aqua Poly/Mount mounting medium | water-soluble, non-fluorescing mounting medium | ||
| Pyrex 3 or 9 depression glass spot plate | |||
| Whatman filter paper |
Referanslar
- Harashima, H., Dissmeyer, N., Schnittger, A. Cell cycle control across the eukaryotic kingdom. Trends in Cell Biology. 23 (7), 345-356 (2013).
- Vermeulen, K., Van Bockstaele, D. R., Berneman, Z. N. The cell cycle: a review of regulation, deregulation and therapeutic targets in cancer. Cell Proliferation. 36 (3), 131-149 (2003).
- Pardee, A. B. A restriction point for control of normal animal cell proliferation. Proceedings of the National Academy of the Sciences of the United States of America. 71 (4), 1286-1290 (1974).
- Gogendeau, D., et al. Aneuploidy causes premature differentiation of neural and intestinal stem cells. Nature Communications. 6, 8894 (2015).
- Barnum, K. J., O'Connell, M. J. Cell cycle regulation by checkpoints. Methods in Molecular Biology. 1170, 29-40 (2014).
- Gratzner, H. G. Monoclonal antibody to 5-bromo- and 5-iododeoxyuridine: A new reagent for detection of DNA replication. Science. 218 (4571), 474-475 (1982).
- Salic, A., Mitchison, T. J. A chemical method for fast and sensitive detection of DNA synthesis in vivo. Proceedings of the National Academy of the Sciences of the United States of America. 105 (7), 2415-2420 (2008).
- Kim, J. Y., et al. The value of phosphohistone H3 as a proliferation marker for evaluating invasive breast cancers: A comparative study with Ki67. Oncotarget. 8 (39), 65064-65076 (2017).
- Homem, C. C., Knoblich, J. A. Drosophila neuroblasts: a model for stem cell biology. Development. 139 (23), 4297-4310 (2012).
- Daul, A. L., Komori, H., Lee, C. Y. EdU (5-ethynyl-2'-deoxyuridine) labeling of Drosophila mitotic neuroblasts. Cold Spring Harbor Protocols. 2010 (7), 5461 (2010).
- Chippalkatti, R., Egger, B., Suter, B. Mms19 promotes spindle microtubule assembly in Drosophila neural stem cells. PLoS Genetics. 16, 1008913 (2020).
- Daul, A. L., Komori, H., Lee, C. Y. Immunofluorescent staining of Drosophila larval brain tissue. Cold Spring Harbor Protocols. (7), (2010).
- Mollinari, C., Lange, B., Gonzalez, C. Miranda, a protein involved in neuroblast asymmetric division, is associated with embryonic centrosomes of Drosophila melanogaster. Biology of the Cell. 94 (1), 1-13 (2002).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Nag, R. N., Niggli, S., Sousa-Guimaraes, S., Vazquez-Pianzola, P., Suter, B. Mms19 is a mitotic gene that permits Cdk7 to be fully active as a Cdk-activating kinase. Development. 145 (2), (2018).
- Cabernard, C., Doe, C. Q. Live imaging of neuroblast lineages within intact larval brains in Drosophila. Cold Spring Harbor Protocols. 2013 (10), (2013).
- Hartenstein, V., Spindler, S., Pereanu, W., Fung, S. The development of the Drosophila larval brain. Advances in Experimental Medicine and Biology. 628, 1-31 (2008).
- Hebar, A., Rutgen, B. C., Selzer, E. NVX-412, a new oncology drug candidate, induces S-phase arrest and DNA damage in cancer cells in a p53-independent manner. PLoS One. 7, 45015 (2012).
- Wyllie, F. S., et al. S phase cell-cycle arrest following DNA damage is independent of the p53/p21(WAF1) signalling pathway. Oncogene. 12 (5), 1077-1082 (1996).
- Xu, X., et al. Inhibition of DNA replication and induction of S phase cell cycle arrest by G-rich oligonucleotides. Journal of Biological Chemistry. 276 (46), 43221-43230 (2001).
- Duronio, R. J. Developing S-phase control. Genes & Development. 26 (8), 746-750 (2012).
- Turrero Garcia, M., Chang, Y., Arai, Y., Huttner, W. B. S-phase duration is the main target of cell cycle regulation in neural progenitors of developing ferret neocortex. Journal of Comparative Neurology. 524 (3), 456-470 (2016).
- Pereira, P. D., et al. Quantification of cell cycle kinetics by EdU (5-ethynyl-2'-deoxyuridine)-coupled-fluorescence-intensity analysis. Oncotarget. 8 (25), 40514-40532 (2017).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132 (3), 487-498 (2008).
- Zielke, N., et al. Fly-FUCCI: A versatile tool for studying cell proliferation in complex tissues. Cell Reports. 7 (2), 588-598 (2014).
- Shen, Y., Vignali, P., Wang, R. Rapid profiling cell cycle by flow cytometry using concurrent staining of DNA and mitotic markers. Bio-protocol. 7 (16), 2517 (2017).
- Berger, C., et al. FACS purification and transcriptome analysis of drosophila neural stem cells reveals a role for Klumpfuss in self-renewal. Cell Reports. 2 (2), 407-418 (2012).
- Harzer, H., Berger, C., Conder, R., Schmauss, G., Knoblich, J. A. FACS purification of Drosophila larval neuroblasts for next-generation sequencing. Nature Protocols. 8 (6), 1088-1099 (2013).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır