Method Article
Protocollo a doppia marcatura 5-etinil-2'-deossiuridina/fosfo-istone H3 per l'analisi della progressione del ciclo cellulare nelle cellule staminali neurali della drosofila
In questo articolo
Riepilogo
L'analisi del ciclo cellulare con etichettatura 5-etinil-2'-deossiuridina (EdU) e fosfo-istone H3 (pH3) è una procedura in più fasi che può richiedere un'ampia ottimizzazione. Qui, presentiamo un protocollo dettagliato che descrive tutti i passaggi per questa procedura, compresa l'analisi e la quantificazione delle immagini per distinguere le cellule in diverse fasi del ciclo cellulare.
Abstract
L'analisi della progressione del ciclo cellulare in vivo viene eseguita di routine in studi sui geni che regolano la mitosi e la replicazione del DNA. La 5-etinil-2'-deossiuridina (EdU) è stata utilizzata per studiare la progressione replicativa / fase S, mentre gli anticorpi contro il fosfo-istone H3 sono stati utilizzati per marcare nuclei e cellule mitotiche. Una combinazione di entrambe le etichette consentirebbe la classificazione delle fasi G0/G1 (fase Gap), S (replicativa) e M (mitotica) e servirebbe come strumento importante per valutare gli effetti dei knockdown dei geni mitotici o dei mutanti nulli sulla progressione del ciclo cellulare. Tuttavia, i reagenti utilizzati per contrassegnare le cellule marcate con EdU sono incompatibili con diversi tag fluorescenti anticorpali secondari. Ciò complica l'immunocolorazione, in cui gli anticorpi primari e secondari marcati vengono utilizzati per contrassegnare le cellule mitotiche pH3-positive. Questo documento descrive un protocollo passo-passo per la doppia etichettatura di EdU e pH3 nelle cellule staminali neurali larvali di Drosophila, un sistema ampiamente utilizzato per studiare i fattori mitotici. Inoltre, viene fornito un protocollo per l'analisi e la quantificazione delle immagini per allocare le cellule etichettate in 3 categorie distinte, G0 / G1, S, S >G2 / M (progressione da S a G2 / M) e M fasi.
Introduzione
Il ciclo di divisione cellulare comprende una fase G1 (prima fase gap), una fase replicativa/S, una G2 (seconda fase gap) e una fase M (mitotica). Passando attraverso queste fasi, la cellula subisce cambiamenti drammatici nella trascrizione cellulare, nella traduzione e nella riorganizzazione del macchinario citoscheletrico1,2. In risposta a segnali di sviluppo e ambientali, le cellule possono temporaneamente cessare di dividersi e diventare quiescenti (G0) o differenziarsi e cessare permanentemente di dividersi3. Altri scenari, come il danno al DNA, possono causare differenziazione prematura o apoptosi3,4. La risposta a tali segnali è mediata da checkpoint del ciclo cellulare, che fungono da sistema di sorveglianza per garantire l'integrità dei processi cellulari essenziali prima che la cellula si impegni nella fase successiva del ciclo di divisione5. Pertanto, gli studi sui geni che regolano la replicazione del DNA, i checkpoint e i macchinari mitotici devono analizzare possibili difetti di progressione del ciclo cellulare che possono verificarsi nelle cellule mutanti o dopo l'abbattimento del siRNA di questi geni. Inoltre, tali analisi possono essere utilizzate per testare la salute generale delle cellule e le risposte cellulari al trattamento farmacologico.
La 5-bromo-2'-deossiuridina (BrdU) è un analogo della timidina che viene incorporato nel DNA durante la replicazione6. Questo metodo è stato ampiamente utilizzato per identificare le cellule in fase S. Tuttavia, le cellule vengono poi sottoposte a dure procedure di denaturazione del DNA per consentire il rilevamento di BrdU attraverso l'uso di anticorpi anti-BrdU6. Questo duro trattamento può danneggiare gli epitopi cellulari e impedire un'ulteriore caratterizzazione del campione attraverso l'immunocolorazione. L'incorporazione di EdU e il successivo rilevamento da parte di una "reazione a clic" catalizzata dal rame con coloranti azide piccoli, permeabili alle cellule e marcati fluorescentemente, elimina la necessità di procedure di denaturazione severe7. Questo metodo, quindi, è emerso come un'alternativa più pratica all'incorporazione di BrdU.
Inoltre, pH3 è stato descritto come un marcatore affidabile per le cellule mitotiche/in fase M8. L'istone H3 è una proteina istonica del nucleo associata al DNA che viene fosforilata intorno alla fase G2 tardiva alla fase M iniziale e viene defosforilata verso la fine dell'anafase8. Diversi anticorpi commerciali possono essere utilizzati per rilevare il pH3 utilizzando protocolli di immunocolorazione standard. La doppia colorazione di EdU e pH3 consentirebbe quindi il rilevamento di cellule in fase S e in fase M. Inoltre, le cellule nella fase G1 e nella fase G2 iniziale non si macchierebbero positivamente per nessuno dei marcatori.
Le cellule staminali neurali o neuroblasti (NB) di Drosophila offrono un modello di cellule staminali ben caratterizzato in cui le cellule si dividono in modo asimmetrico per produrre un NB auto-rinnovante identico e una cellula madre gangliare (GMC), che è destinata alla differenziazione9. Inoltre, diversi strumenti genetici e anticorpi nb-specifici rendono questo sistema adatto per la manipolazione genetica e l'imaging di cellule vive. Di conseguenza, diversi studi hanno utilizzato NB per studiare i geni che regolano le divisioni asimmetriche e la determinazione del destino cellulare9. Popolazioni distinte di NB esistono nel cervello centrale (CB) e nel lobo ottico (OL) del cervello larvale9; Per il presente studio sono stati utilizzati CB NB. Questi CB NB larvali instar sono cellule di grandi dimensioni che sono adatte anche per studiare i fattori che regolano l'assemblaggio del fuso mitotico. Un protocollo per analizzare i difetti di progressione del ciclo cellulare sarebbe uno strumento vitale in tali studi.
I protocolli pubblicati in precedenza impiegavano kit commerciali, come Click-iT EdU Alexa Fluor Cell Proliferation Kit, che forniscono diversi componenti di reazione e coloranti azide etichettati con una varietà di coloranti Alexa Fluor per l'incorporazione e il rilevamento di EdU10. Tuttavia, i reagenti forniti con tali kit non sono compatibili con alcuni tag fluorescenti spesso utilizzati con anticorpi secondari. Questo kit di rilevamento EdU (Click-iT EdU Alexa Fluor Cell Proliferation Kit fornito con colorante azide coniugato Alexa Fluor 647) è stato testato in Drosophila terzo NB larvale instar e la co-colorazione è stata tentata con anticorpi contro pH3 e Miranda, un marcatore per NB. Inoltre, gli anticorpi secondari alexa Fluor 568 o Cy3 sono stati utilizzati per il rilevamento dell'etichettatura Miranda sulla membrana plasmatica degli NB11. Tuttavia, l'intensità del segnale atteso e il modello di colorazione (risultati non pubblicati) non sono stati osservati con questi anticorpi secondari quando l'immunocolorazione è stata eseguita dopo il rilevamento di EdU.
Per l'incorporazione di EdU, il protocollo descritto da Daul e colleghi richiedeva l'alimentazione delle larve con kankel-white medio mescolato con EdU e bromofenolo blu (BPB)10. Le larve si nutrivano con il cibo a spillo EdU e BPB, che poteva essere visto dal suo colore blu all'ingestione nell'intestino larvale. Sebbene questo metodo sia stato utilizzato per l'incorporazione di EdU nella perdita di funzione di Mms19 (Mms19P) terza larva instar, le larve di Mms19P apparentemente non si sono nutrite poiché quasi nessun colore blu è stato rilevato nell'intestino larvale (risultati non pubblicati). Le larve di Mms19P mostrano drastiche deformità dello sviluppo e alla fine si arrestano nel terzo stadio instar. Ciò può in qualche modo influenzare il comportamento alimentare delle larve della terza stella e rendere il protocollo di alimentazione EdU inadatto a tali casi.
Dopo aver studiato la letteratura disponibile e aver lavorato ampiamente sulla standardizzazione dei passaggi essenziali, è stato proposto un approccio alternativo per la doppia etichettatura EdU/ pH3 nei NB Di Drosophila, che non richiede l'alimentazione di EdU alle larve. Uno studio precedente ha impiegato la doppia colorazione EdU / pH3 per analizzare il ciclo cellulare nei NB, ma non ha presentato un protocollo dettagliato4. Questo rappresenta un ostacolo inutile per i laboratori che cercano di implementare questo metodo. Inoltre, valutare la compatibilità di vari reagenti con il kit EdU ed eseguire un'ulteriore ottimizzazione può essere un processo che richiede molto tempo. Questo documento presenta un protocollo passo-passo che copre l'incorporazione di EdU nel cervello larvale sezionato e l'immunocolorazione con anticorpi anti-pH3, seguita dalla microscopia confocale e dall'analisi delle immagini per assegnare NB a quattro categorie distinte: fase G0 / G1, fase S, S >G2 / M (progressione da S a G2 / M) e fase M. I passaggi che richiedono l'ottimizzazione sono delineati e vengono forniti suggerimenti per l'analisi delle immagini di set di dati di grandi dimensioni. Inoltre, la lettura EdU/pH3 nei NB wild-type viene analizzata e confrontata con i NB P Mms19, che sono stati recentemente segnalati per mostrare un ritardo del ciclo cellulare11.
Protocollo
1. Preparazione dei reagenti e delle scorte per il test Click-it EdU
NOTA: fare riferimento alla Tabella dei materiali e alla Tabella 1 per i dettagli sul kit e sui reagenti forniti con il kit.
- Portare i flaconcini a temperatura ambiente prima di preparare le soluzioni.
- Preparare 10 mM di soluzione madre EdU (componente A) aggiungendo 2 mL di dimetilsolfossido (DMSO, componente C). Mescolare bene e conservare a -20 °C.
- Preparare una soluzione di lavoro di Alexa Fluor 647-azide (componente B) aggiungendo 70μL di DMSO (componente C). Mescolare bene e conservare a -20 °C.
- Preparare una soluzione 1x di tampone di reazione Click-iT EdU (componente D) mescolando 4 mL di questa soluzione con 36 mL di acqua deionizzata. Conservare la soluzione rimanente a 2-6 °C.
- Fare un 10x stock (200 mg/mL) dell'additivo tampone Click-iT EdU (componente F) aggiungendo 2 mL di acqua deionizzata. Mescolare bene e conservare a -20 °C.
- Conservare Hoechst 33342 (componente G) a 2-6 °C. Conservare DMSO (componente C) in un essiccatore a -20 °C.
NOTA: i guanti devono essere utilizzati per maneggiare DMSO e Hoechst.
2. Dissezione del cervello larvale del terzo instar e incorporazione di EdU
NOTA: Il protocollo per le dissezioni cerebrali è stato descritto in precedenza12. Prima di iniziare le dissezioni, assicurarsi che quantità sufficienti di soluzioni edU e PFA siano preparate e scongelate come descritto ai punti 2.6 e 3.1.
- Preparare 10x soluzione salina tamponata con fosfato (PBS) aggiungendo 25,6 g na2HPO4,7H20, 80 g NaCl, 2 g KCl e 2 g KH2PO4 a 1 L di acqua deionizzata. Regolare il pH a 7,4 e successivamente preparare una soluzione 1x in acqua deionizzata.
- Aggiungere il mezzo di dissezione (SM) di Schneider a due depressioni consecutive di una lastra di vetro (3 o 9) di vetro. Aggiungi 1x PBS alla successiva depressione consecutiva.
- Usando un paio di pinze, scegli le larve di terza stella vaganti e posizionale su un tessuto bagnato con PBS per pulire i residui di cibo della mosca. Posizionare la larva nella depressione con PBS e poi nella depressione consecutiva con SM.
- Usando un paio di pinze fini, rimuovere3/4 della parte inferiore del corpo della larva.
- Afferrare delicatamente i ganci della bocca larvale con un paio di pinze e tenere la cuticola all'altra estremità con l'altra coppia di pinze.
- Ruotare la testa larvale dall'interno verso l'esterno spingendo verso l'interno i ganci della bocca e contemporaneamente staccando il tessuto all'altra estremità.
- Osserva il cervello larvale con dischi immaginali attaccati. Rimuovere altri tessuti attaccati al cervello e trasferire il cervello alla successiva depressione consecutiva con SM.
- Scongelare la soluzione stock EdU da 10 mM. Preparare una soluzione da 100 μM diluendo questo stock da 10 mM in SM.
- Aggiungere 100 μL di questa soluzione EdU+SM da 100 μM in un'altra depressione sulla piastra Pyrex. Incubare ~5-10 cervelli sezionati in 100 μL di soluzione EdU+SM da 100 μM per 2 ore a 25 °C.
NOTA: poiché i NB si dividono una volta in ~ 2 h, questo protocollo mira ad analizzare un ciclo cellulare completo. Usa solo cervelli intatti per ulteriori passaggi. Scartare i cervelli danneggiati durante la dissezione.
3. Fissazione e immunocolorazione
- Preparare la soluzione di paraformaldeide al 4% (PFA) in una cappa aspirante.
- Aggiungere 4 g di polvere di paraformaldeide a 80 mL di 1x PBS in un becher posto su un agitatore magnetico e riscaldare a 60 °C. Per sciogliere il PFA, aggiungere alcune gocce di 1 N NaOH. Controllare il pH e regolare a 7,0 con alcune gocce di 1 M HCl, se necessario.
- Regolare il volume a 100 ml con 1x PBS. Aliquotare la soluzione di PFA e conservare a 2-6 °C per un massimo di 1 mese. Aggiungere lo 0,3% di detergente non ionico (vedere la Tabella dei materiali)alla soluzione di PFA per una fissazione efficiente di tessuti di grandi dimensioni come il cervello larvale.
- Dopo l'incorporazione di EdU, trasferire il cervello in tubi microcentrifuga da 0,6 ml contenenti il 4% di PFA. Incubare a temperatura ambiente su un nutator per 15 min. Toccare il tubo sul banco di laboratorio in modo che il cervello si stabilizzi nella parte inferiore del tubo.
- Rimuovere il PFA e lavare 3 volte con PBS + 0,3% detergente non ionico (PBST) a temperatura ambiente. Assicurarsi che ogni lavaggio duri almeno 10 minuti su un nutatore. Dopo l'ultimo lavaggio, rimuovere PBST, aggiungere la soluzione bloccante (PBST + 5% di albumina sierica bovina) e incubare per 30 minuti a temperatura ambiente.
- Anticorpo anti-Miranda diluito (1:250) e anticorpo anti pH3 (1:200) in PBST (entrambi gli anticorpi nello stesso tubo). Rimuovere il tampone di blocco e aggiungere la soluzione anticorpale primaria. Incubare durante la notte a 2-6 °C su un nutatore.
NOTA: Miranda è una proteina di membrana presente sui CB NB. Serve a identificare queste grandi cellule rotonde nella regione CB13. - Rimuovere la soluzione anticorpale primaria e lavare 3 volte con PBST. Assicurarsi che ogni lavaggio duri 10 minuti su un nutatore a temperatura ambiente.
- Preparare la soluzione anticorpale secondaria aggiungendo 1:500 Alexa Fluor 488 anti-Rabbit diluito e Alexa Fluor 568 anti-Rat in PBST. Rimuovere il PBST e aggiungere la soluzione anticorpale secondaria al cervello sezionato. Incubare durante la notte a 2-6 °C al buio, su un nutatore, e lavare 3 volte con PBST (passaggio 3.5).
4. Rilevamento EdU, colorazione del DNA e montaggio
- Per preparare il cocktail di rilevamento EdU, mescolare i componenti (vedere Tabella 2) in un tubo da 1,5 ml.
- Dopo l'ultimo lavaggio (passaggio 3.6), rimuovere PBST e aggiungere il cocktail di rilevamento EdU al cervello sezionato. Incubare a temperatura ambiente al buio per 30 minuti su un nutator. Lavare 2 volte con PBST per 10 minuti ciascuno, al buio.
- Per la colorazione del DNA, diluire Hoechst 33342 (componente G) 1:2.000 in PBST per preparare una soluzione da 5 μg/mL.
- Rimuovere PBST dopo il secondo lavaggio (fase 4.2) e aggiungere 500 μL di 5 μg/mL di soluzione di Hoechst al cervello sezionato. Incubare al buio per 10 minuti. Rimuovere Hoechst e lavare una volta con PBST per 10 minuti.
- Tocca il tubo sul banco di laboratorio e lascia che il cervello si stabilizzi. Rimuovere il PBST dal tubo, ma lasciare solo 50-100 μL.
- Tagliare l'estremità di una punta di micropipetta da 200 μL e trasferire con cura il cervello su un vetrino pulito. Rimuovere il PBST in eccesso dalla diapositiva tamponando con strisce di carta da filtro. Fai attenzione a non lasciare che la carta da filtro tocchi il cervello.
- Metti una goccia di mezzo di montaggio solubile in acqua e non fluorescente sul cervello e orienta il cervello in modo tale che il cordone nervoso ventrale sia rivolto verso lo scivolo e i lobi rivolti verso l'alto. Disporre i cervelli in un unico file in modo che sia più facile visualizzare il cervello in serie.
- Posiziona delicatamente una copertina sopra il cervello. Posizionare il vetrino a 2-6 °C durante la notte.
NOTA: il supporto di montaggio si indurisce durante la conservazione durante la notte in modo che non sia necessario sigillare il coperchio con lo smalto per unghie.
5. Imaging
NOTA: Vedere la Tabella dei materiali per i dettagli sul microscopio a scansione laser e sull'obiettivo ad immersione in olio utilizzati in questo protocollo.
- Dal software di acquisizione, selezionare l'obiettivo 63x.
- Metti una goccia di olio per immersione sulla copertina appena sopra il cervello montato per rendere più facile individuare il tessuto attraverso l'oculare.
- Utilizzando il canale DAPI/Hoechst 33342, trovare il cervello attraverso l'oculare, quindi passare alla modalità di acquisizione nel software.
- Imposta 4 canali per visualizzare Le immagini di Hoechst 33342 (DNA), Alexa Fluor 488 (pH3), Alexa Fluor 568 (Miranda) e Alexa Fluor 647 (EdU). Utilizzare lo strumento dye-assistant, che imposta automaticamente i laser di eccitazione e i filtri di emissione per i coloranti selezionati.
- Imposta il campo visivo in modo tale che comprenda l'intero lobo cerebrale. Immagina l'intero volume del lobo cerebrale acquisendo z-stack distanziati di 0,8 μm l'uno dall'altro. Memorizza tutte le immagini di una sessione di imaging in un formato di libreria *.lif.
6. Analisi delle immagini
NOTA: i seguenti passaggi descrivono l'analisi delle immagini acquisite e come ordinare le celle in fase G0/G1, fase S, S>G2/M (progressione da S a G2/M) e fase M utilizzando il software ImageJ.
- Scarica Fiji (Fiji is ImageJ) dal seguente URL: https://fiji.sc/. Apri Fiji, quindi trascina e rilascia i file .lif in Fiji.
NOTA: Fiji è una versione di ImageJ che viene fornita preinstallata con diversi plugin14. Il trasferimento di file .lif nelle Figi aprirà il plug-in Bio-formats, necessario per elaborare i file .lif. Il plug-in Bio-formats è necessario anche per aprire file di immagini generati da altre marche di microscopi, ad esempio file nd2 generati da un microscopio Nikon. Questo plugin viene fornito preinstallato con Fiji. - Selezionare Browser dati dalla scheda di visualizzazione dello stack e utilizzare lo stack virtuale dalla scheda di gestione della memoria nel plug-in bioformati.
NOTA: i file Lif con z-stack da 8-10 cervelli hanno spesso una dimensione di 300-400 megabyte. Sui computer con POCA RAM, l'apertura di alcuni di questi file può esaurire rapidamente la RAM disponibile su ImageJ e impedire qualsiasi ulteriore elaborazione delle immagini. Lo stack virtuale è di "sola lettura", non richiede RAM elevata per l'elaborazione ed è un'opzione ideale per caricare set di dati di grandi dimensioni sulle Figi. - Osservare l'immagine multicanale visualizzata in ImageJ. Modificare il colore dei canali dalla barra dei menu utilizzando lo strumento Immagine | colore | canali. Osserva i NB etichettati con Miranda come grandi cellule rotonde nella regione CB.
- Disegna una regione di interesse (ROI) utilizzando lo strumento ellisse su ogni NB per evitare di contare l'NB due volte.
- Dalla barra dei menu ImageJ, selezionare analizza | strumenti | ROI manager. Contrassegnare tutti i NB nella sezione z corrente e premere t dopo aver contrassegnato ogni cella.
- Una volta che tutti gli NB nella sezione z corrente sono stati contrassegnati, cambiare i canali in pH3 ed EdU e contare manualmente il numero di NB EdU-positivi, NB pH3-positivi, NB che si colorano positivamente sia per EdU che per pH3 e NB non macchiati per entrambi i marcatori.
- Cerca i NB nelle sezioni z successive, elimina i vecchi ROI e aggiungi nuovi ROI per contare i NB in stack diversi.
- Preparare un foglio di calcolo con le seguenti colonne: 1. EdU- pH3-: questa sezione rappresenta le celle a doppio negativo che si trovano nella fase G0/G1 del ciclo cellulare; 2. EdU+: le cellule di questa categoria hanno incorporato EdU e sono in fase di replicazione del DNA (fase S); 3. EdU+ pH3+: queste cellule dual-positive hanno completato la fase S e sono passate alla fase G2 o M; 4. pH3+: questi NB sono sottoposti a mitosi.
- Calcola le percentuali di NB presenti in tutte le 4 categorie precedenti per ciascun lobo. Prepara un grafico a barre utilizzando il software del foglio di calcolo che mostra i dati in pool per le percentuali di NB in ogni categoria.
Risultati
Il cervello larvale bilobato di Drosophila terzo instar è stato utilizzato come sistema modello per studiare i processi cellulari e di sviluppo fondamentali9. L'obiettivo del presente studio è stato quello di presentare un protocollo per l'analisi della progressione del ciclo cellulare nei NB marcati con EdU e pH3 della regione CB (Figura 1). I CB NB sono suddivisi in tipo I e tipo II e presentano il caratteristico modello di divisione asimmetrica9. Ogni divisione NB di tipo I genera un NB, che è in grado di auto-rinnovarsi, e un altro GMC che è destinato alla differenziazione9. Al contrario, gli NB di tipo II generano cellule progenitrici neurali immature (INP) che si auto-rinnovano e generano 2 GMC9. Sebbene esista una popolazione NB distinta nella regione OL, questo studio si è concentrato sui CB NB, che sono grandi e facilmente identificabili con marcatori NB specifici.
Per questo studio, Miranda è stato utilizzato per identificare e analizzare i CB NB (Figura 2). Sebbene Miranda macchi anche altre popolazioni cellulari nell'OL, i CB NB possono essere identificati con Miranda a causa delle loro grandi dimensioni e della posizione CB13. Altri marcatori più specifici possono anche essere utilizzati per etichettare i CB NB, come Deadpan, che è un fattore di trascrizione che regola l'auto-rinnovamento NB. Tuttavia, poiché sia pH3 che EdU marcano la cromatina, il marcatore a membrana Miranda è stato utilizzato per facilitare la visualizzazione e l'analisi dei NB contrassegnati con pH3 ed EdU. L'incorporazione di EdU, il successivo rilevamento e l'ulteriore caratterizzazione mediante immunocolorazione e analisi delle immagini è una procedura complessa. È importante standardizzare ogni fase per determinare le condizioni ottimali di colorazione e imaging. Sebbene alcuni report pubblicati presentino strategie di etichettatura EdU/pH3, questi report non approfondiscono le fasi di ottimizzazione e analisi delle immagini. Il flusso di lavoro descritto nella Figura 1 riepiloga i passaggi dall'incorporazione di EdU fino all'analisi delle immagini.
Recentemente abbiamo caratterizzato il gene Mms19, che è richiesto dai NB per la normale progressione mitotica11. Attraverso le analisi di imaging delle cellule vive, gli NB privi di Mms19 funzionale hanno dimostrato di impiegare il doppio del tempo degli NB wild-type per terminare la mitosi. Ciò si è chiaramente riflesso nell'analisi EdU/pH3 in cui una percentuale significativamente più elevata di NBP Mms19 è risultata essere in fase M rispetto agli NB wild-type ( Figura3A). L'espressione della proteina di fusione Mms19::eGFP sullo sfondo di Mms19P aveva dimostrato di salvare i difetti fenotipici11,15. Ciò è correlato bene con i risultati dell'analisi della progressione del ciclo cellulare in cui la proporzione di cellule in fase M è stata salvata a livelli wild-type su espressione di Mms19::eGFP nello sfondo di Mms19P (Figura 3A,B).

Figura 1: Un flusso di lavoro semplificato per la doppia etichettatura EdU/pH3. Il terzo cervello larvale instar è stato sezionato e incubato con 100 μM EdU per 2 ore. Successivamente, il tessuto cerebrale è stato fissato con PFA e immunocolorato con anticorpi contro Miranda e pH3. Le immagini dei cervelli macchiati sono state ottenute su un microscopio confocale e ulteriormente elaborate con ImageJ / Fiji per allocare le cellule a diverse fasi del ciclo cellulare. Abbreviazioni: EdU = con 5-etinil-2'-deossiuridina; pH3 = fosfo-istone H3; PFA = paraformaldeide; SM = mezzo di dissezione Schneiders; PBS = soluzione salina tamponata con fosfato; PBST = PBS + 0,3% detergente non ionico; BSA = albumina sierica bovina. Fare clic qui per visualizzare una versione più grande di questa figura.
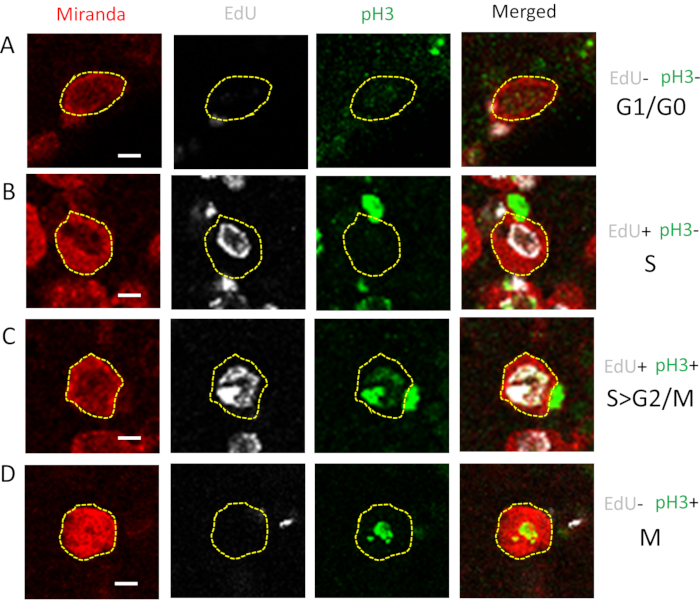
Figura 2: Allocazione delle celle a G1/G0; S; Fasi S>G2/M e M. Le cellule progrediscono attraverso le fasi G1, S, G2 e M per completare un ciclo cellulare completo. I CB NB provenienti dal cervello larvale vagante della terza stella sono stati contrassegnati con Miranda (rosso), EdU (grigio), pH3 (verde) e il DNA è stato etichettato con Hoechst 33342 (blu). Gli NB etichettati sono stati analizzati in ImageJ e assegnati a 4 fasi distinte in base al fatto che si siano colorati positivamente per (A) né EdU né pH3 (G1 / G0), (B) solo EdU (fase S), (C) sia EdU che pH3 (S>G2 / M) e (D) solo pH3 (fase M). Esempi di singoli NB solo da cervelli wild-type sono mostrati qui. Barre di scala = 5 μm. Abbreviazioni: CB = cervello centrale; NB = neuroblasti; EdU = con 5-etinil-2'-deossiuridina; pH3 = fosfo-istone H3. Fare clic qui per visualizzare una versione più grande di questa figura.
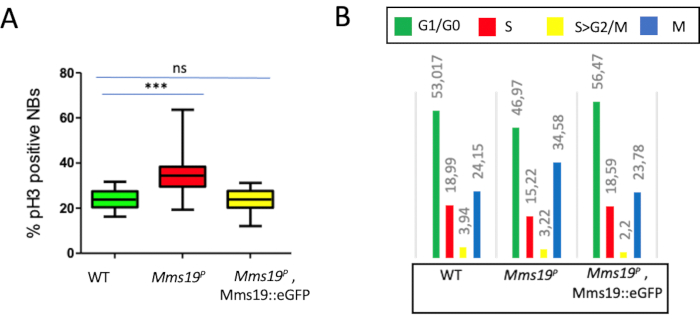
Figura 3: Effetto di Mms19P sulla distribuzione della fase del ciclo cellulare NB. (A) Dopo l'analisi dei NB wild-type (w;+;+), circa il 25% degli NB è risultato essere in fase M. Negli NB Mms19P, tuttavia, questa percentuale è aumentata a quasi il 40%. L'espressione di Mms19::eGFP nello sfondo di Mms19P è nota per salvare i fenotipi di Mms19P e la proporzione di NB di fase M in questo background era paragonabile a quella del tipo wild. (B) Le percentuali di NB in 4 diverse categorie corrispondenti alle fasi del ciclo cellulare G1/G0, S, S>G2/M e M sono state confrontate tra wild type; Mms19P e Mms19::eGFP, mms19P genotipi. La proporzione di fasi M e G1/G0 differisce considerevolmente nei NB Mms19P rispetto ai NB wild-type. Al contrario, la distribuzione del ciclo cellulare dei NB nei cervelli Mms19::eGFP, Mms19P è paragonabile a quella che si trova nel tipo selvatico. La significatività statistica è stata calcolata utilizzando il test di Kruskal-Wallis e più colonne confrontate utilizzando il post-test di Dunn; (P<0,001). Questa cifra è stata modificata da 11. Abbreviazioni: NB = neuroblasti; eGFP = proteina fluorescente verde potenziata; WT = wild-type. Fare clic qui per visualizzare una versione più grande di questa figura.
| Reagente | Quantità/volume |
| EdU (componente A) | 5 mg |
| Alexa Fluor 647 – azide (componente B) | 1 flaconcino |
| Dimetilsolfossido (DMSO, componente C) | 4 ml |
| Buffer di reazione Click-iT EdU (componente D) | 4 mL (soluzione 10x) |
| Solfato di rame (CuSO4, componente E) | 100 mM; 1 flaconcino |
| Additivo tampone Click-iT EdU (componente F) | 400 mg |
| Hoechst 33342 (componente G) | 10 mg/mL in acqua, 35 μL |
Tabella 1: Componenti del kit EdU. Componenti forniti con il Click-iT EdU Alexa Fluor Cell Proliferation Kit e le rispettive quantità/volumi. Abbreviazione: EdU = con 5-etinil-2'-deossiuridina.
| Componenti di reazione | Volume (mL) |
| 1x buffer di reazione Click-iT (preparato nel passaggio 1.4) | 430 |
| CuSO4 (componente E) | 20 |
| Alexa Fluor 647 – azide (componente B, preparato nella fase 1.3) | 1.2 |
| Additivo tampone di reazione (componente F, preparato nella fase 1.5) | 50 |
| Volume totale | ~500 |
Tabella 2: Preparazione del cocktail di rilevamento EdU. Il cocktail di rilevamento EdU è stato preparato mescolando i volumi indicati dei componenti del kit (preparati nella sezione 2). Abbreviazione: EdU = con 5-etinil-2'-deossiuridina.
Discussione
L'incorporazione di EdU e la sua successiva reazione "click" con azide permeabile alle cellule presenta vantaggi pratici di questa tecnica rispetto al metodo BrdU utilizzato in precedenza7. Tuttavia, questa reazione è catalizzata dagli ioni Cu(I) e diversi coloranti possono essere instabili in presenza di questo catalizzatore di rame, come è chiaramente consigliato dal produttore del kit Click-it EdU. Quando sono stati eseguiti esperimenti di immunocolorazione dopo aver eseguito la fase di rilevamento EdU, l'intensità del segnale prevista non è stata osservata con i coloranti a canale rosso, Alexa Fluor 568 e Cy3. Tuttavia, il protocollo ha funzionato quando le reazioni immunocoloranti sono state completate prima della fase di rilevamento edU. Con questo protocollo, sono stati utilizzati quattro canali per visualizzare Hoechst, pH3, Miranda ed EdU.
Un altro passo ottimizzato in questo protocollo è stato l'assorbimento di EdU da parte di cervelli sezionati invece di nutrire il cibo di mosca a spillo edU alle larve. Poiché l'intero ciclo cellulare di Drosophila NB dura circa 2-2,5 ore16,l'assorbimento di EdU da parte del cervello per 2,5 ore come in questo protocollo, consente il monitoraggio di un ciclo cellulare completo di NB. Le cellule che non si macchiano positivamente né per EdU né per pH3 sono cellule a riposo. Nel caso di una terza larva instar in cui i NB si dividono attivamente, queste cellule a riposo sarebbero molto probabilmente nella fase G1 del ciclo cellulare. Tuttavia, dal tardo embrione ai primi secondi stadi instar, i NB diventano temporaneamente quiescenti17. In queste prime fasi di sviluppo, la colorazione a doppia negatività rappresenterebbe molto probabilmente una fase G0 / quiescente.
Gli NB sottoposti a replicazione del DNA dovrebbero macchiarsi positivamente per EdU, mentre le cellule che sono progredite dalla fase S (incorporazione edU) alla fase G2 o M tardiva dovrebbero macchiarsi positivamente sia per EdU che per pH3. Tuttavia, la caratterizzazione della fase G2 sarebbe difficile con questo protocollo poiché le prime cellule G2 non si macchiano positivamente per EdU o pH3. Tuttavia, le cellule che hanno incorporato EdU e sono progredite fino all'inizio di G2 si macchierebbero positivamente anche per EdU e potrebbero complicare l'analisi della fase G2. Diversi farmaci che inibiscono la replicazione del DNA causano l'arresto del ciclo cellulare alla fase S18,19,20. Questo protocollo potrebbe essere uno strumento conveniente per analizzare e identificare l'effetto di tali trattamenti farmacologici in quanto le cellule bloccate nella fase S non progredirebbero nella fase M e la frazione di EdU, le cellule doppie positive pH3 sarebbero diminuite o addirittura assenti in questo caso.
In uno studio pubblicato di recente, abbiamo valutato l'effetto del gene Mms19 sulla progressione mitotica negli NB11. Attraverso l'imaging di cellule vive, abbiamo dimostrato un drammatico ritardo dei NB P mms19 nella progressione della fase M. Mentre i NB wild-type completano la fase M in ~ 10 minuti, gli NBP Mms19 hanno impiegato circa il doppio del tempo per completare la fase M11. Di conseguenza, questo protocollo di doppia etichettatura EdU / pH3 rifletteva anche un ritardo mitotico poiché abbiamo osservato quasi 1,5 volte più NB che si coloravano positivamente per pH3 nei cervelli mms19P rispetto ai cervelli wild-type. I dati di questo protocollo EdU sono quindi comparabili con i risultati diretti dell'imaging delle cellule vive. Poiché tali approcci diretti e vivi richiedono molto tempo e necessitano di un'ampia ottimizzazione, questo protocollo potrebbe essere utilizzato per eseguire un rapido screening iniziale di tali mutanti del ciclo cellulare per comprendere i difetti in fasi specifiche. Una volta identificata una fase di interesse, questa potrebbe essere seguita da saggi di visualizzazione cellulare diretta.
La durata delle fasi del ciclo cellulare può variare considerevolmente tra i diversi tipi di cellule e le fasi di sviluppo. Ad esempio, nello sviluppo di embrioni di Drosophila, la downregulation delle chinasi mitotiche si traduce in una durata prolungata della fase S, mentre nelle cellule destinate alla differenziazione, come i progenitori neurali cerebrali del furetto (Mustela putorius furo), la durata della fase S è marcatamente breve21,22. Il tempo di etichettatura degli impulsi EdU dovrebbe quindi essere ottimizzato in base alla lunghezza delle fasi S e G nel tipo di cella specificato. La durata dell'impulso EdU dipenderebbe anche dagli obiettivi sperimentali, ad esempio, un impulso breve è sufficiente per misurare la frazione di cellule attualmente in fase S, mentre un impulso più lungo consente l'analisi della progressione delle cellule attraverso la fase S.
Un'ampia ottimizzazione dell'impulso EdU può anche consentire la stima della fase S, come è stato elegantemente dimostrato da Pereira e colleghi23. Questi ricercatori hanno dimostrato un nuovo metodo basato sulla citometria a flusso in cui le cellule HCT116 sono state marcate con EdU per periodi di tempo incrementali. Gli autori hanno dimostrato che l'intensità fluorescente massima di EdU si ottiene quando il tempo di pulsazione corrisponde alla lunghezza della fase S23. Inoltre, l'analisi della progressione temporale delle cellule marcate con EdU ha anche permesso la quantificazione delle fasi G1 e G2/M. Sebbene l'attuale protocollo per la doppia etichettatura EdU/pH3 consenta l'analisi dei difetti di fase S e M, non è possibile misurare la durata precisa delle fasi con questo protocollo. Il protocollo descritto da Pereira e colleghi potrebbe essere un'alternativa adatta per i saggi che richiedono la determinazione delle lunghezze delle fasi del ciclo cellulare. L'adattamento di questo protocollo con un'ulteriore etichettatura pH3 può anche comportare una maggiore sensibilità nel rilevamento delle cellule in fase M.
Oltre all'approccio EdU/pH3, ilmetodo FUCCI (f luorescent ubiquitin-based cell cycle indicator) è stato utilizzato anche per studiare la progressione del ciclo cellulare nelle cellule di mammifero e nei tessuti di Drosophila24,25. Questo sistema utilizza due proteine marcate fluorescentemente, geminina e Cdt1, che contengono motivi per l'ubiquitinazione specifica e la degradazione proteasomiale da APC/ C e SCFSkp2, rispettivamente. Poiché APC/C è attivo solo dalla fine della mitosi attraverso G1 e SCFSkp2 è attivo nelle fasi S e G2, la degradazione specifica dello stadio del ciclo cellulare della geminina marcata fluorescentmente e del Cdt1 consente la determinazione della fase24del ciclo cellulare. Un metodo "Fly-FUCCI" leggermente modificato per i tessuti di Drosophila si basa, invece, su ciclina B ed E2F1 marcate fluorescentemente, che sono degradate da APC / C (durante la mitosi) e CRL4Cdt2 (durante l'insorgenza della fase S), rispettivamente25. Uno studio precedente che caratterizzava la differenziazione prematura di NB larvali di Drosophila in risposta all'aneuploidia ha analizzato i difetti del ciclo cellulare con metodi Fly-FUCCI e EdU/ pH34. L'aneuploidia induce una differenziazione prematura dei NB e, pertanto, un'alta frazione di NB esce dal ciclo cellulare.
Ciò è stato accuratamente riflesso sia nei dati Fly-FUCCI che EdU/pH34. I dati ottenuti dal metodo EdU/pH3 sono, quindi, anche comparabili con altri metodi di tracciamento del ciclo cellulare come il Fly-FUCCI. Fly-FUCCI è uno strumento potente e tutti gli stock di mosche con marcatori fluorescenti o marcatori onnipresenti fusi a promotori specifici del tessuto sono disponibili presso i centri di stoccaggio. Tuttavia, l'uso di questi costrutti per analizzare i difetti del ciclo cellulare in mutanti nulli o con knockdown di siRNA comporterebbe la ricombinazione genetica per creare un fly stock che trasporta gli elementi FUCCI, i driver specifici del tessuto, una mutazione specifica o un siRNA di interesse. Questo processo ritarderebbe l'esperimento vero e proprio per diverse settimane, mentre il metodo EdU/pH3 può essere facilmente applicato alle linee di volo con uno sfondo genetico nullo. In alternativa, per gli abbattimenti di siRNA, è possibile utilizzare un incrocio in un passaggio tra uno stock driver e uno stock di siRNA per l'analisi EdU / pH3.
Uno svantaggio di questo approccio è la pipeline di imaging e analisi delle immagini, che comporta la quantificazione manuale di un gran numero di cellule da diversi campioni di cervello. Recentemente, un approccio di citometria a flusso è stato presentato come un'alternativa ad alto rendimento a un saggio convenzionale basato sulla microscopia per l'analisi del ciclo cellulare26. La rapida analisi di migliaia di cellule contemporaneamente con questo approccio è vantaggiosa e la capacità di rilevare più marcatori consente la quantificazione di specifici tipi di cellule. Sebbene facilmente utilizzabile con linee cellulari coltivate, questo protocollo potrebbe essere difficile da applicare a grandi campioni di tessuto come il cervello larvale di Drosophila. Tuttavia, sono stati pubblicati alcuni protocolli che descrivono la dissociazione del tessuto cerebrale e l'analisi di NB larvali isolati mediante citometria a flusso27,28. Ulteriori approcci innovativi in questa direzione possono presentare nuove opportunità per l'analisi del ciclo cellulare ad alto rendimento nei tessuti della Drosophila.
Divulgazioni
Gli autori hanno dichiarato che non esistono interessi concorrenti.
Riconoscimenti
Questo lavoro è stato sostenuto da finanziamenti del Fondo nazionale svizzero per la ricerca scientifica (sovvenzione del progetto 31003A_173188; www.snf.ch) e dell'Università di Berna (www.unibe.ch) a BS. I finanziatori non avevano alcun ruolo nella progettazione dello studio, nella raccolta e nell'analisi dei dati, nella decisione di pubblicare o nella preparazione del manoscritto.
Materiali
| Name | Company | Catalog Number | Comments |
| fly stocks | |||
| P{EPgy2}Mms19EY00797/TM3, Sb1 Ser1 (Mms19p) | Bloomington stock center | #15477 | P-element insertion in the third exon of Mms19 |
| +; Mms19::eGFP, Mms19p | Generated in house | eGFP-tagged Mms19 protein expressed in Mms19p background | |
| w1118 | Bloomington stock center | #3605 | wild-type stock (w;+;+) |
| Primary antibodies | Dilution | ||
| Rat anti-Miranda | Abcam | Ab197788 | 1/250 |
| Rabbit anti-pH3 | Cell Signaling | 9701 | 1/200 |
| Secondary antibodies | |||
| Goat anti-Rat Cy3 | Jackson Immuno | 112-165-167 | 1/150 |
| Goat anti-Rat Alexa Fluor 568 | Invitrogen | A11077 | 1/500 |
| Goat anti-Rabbit Alexa Fluor 488 | Invitrogen | A27034 | 1/500 |
| Reagent/Kit | |||
| Aqua Poly/Mount mounting medium | Polysciences Inc | 18606-20 | |
| Click-it EdU incorporation kit, Alexa Flour 647 | Thermo Fischer Scientific | C10340 | |
| Schneider’s Drosophila medium | Thermo Fischer Scientific | 21720-024 | |
| Bovine serum albumin (BSA) fraction V | Merck | 10735078001 | |
| Triton X-100 | Fischer Scientific | 9002-93-1 | non-ionic detergent |
| Software | |||
| Fiji (Imagej) | https://imagej.net/Fiji | ||
| Leica Application Suite (LAS X) | Leica microsystems | ||
| PRISM | Graph pad software | Version 5 | |
| Microsoft Excel | Microsoft office | 2016 | |
| Equipment | |||
| Leica TCS SP8 laser scanning confocal microscope with 63x oil-immersion, 1.4 NA Plan-apochromat objective | |||
| Materials | |||
| Aqua Poly/Mount mounting medium | water-soluble, non-fluorescing mounting medium | ||
| Pyrex 3 or 9 depression glass spot plate | |||
| Whatman filter paper |
Riferimenti
- Harashima, H., Dissmeyer, N., Schnittger, A. Cell cycle control across the eukaryotic kingdom. Trends in Cell Biology. 23 (7), 345-356 (2013).
- Vermeulen, K., Van Bockstaele, D. R., Berneman, Z. N. The cell cycle: a review of regulation, deregulation and therapeutic targets in cancer. Cell Proliferation. 36 (3), 131-149 (2003).
- Pardee, A. B. A restriction point for control of normal animal cell proliferation. Proceedings of the National Academy of the Sciences of the United States of America. 71 (4), 1286-1290 (1974).
- Gogendeau, D., et al. Aneuploidy causes premature differentiation of neural and intestinal stem cells. Nature Communications. 6, 8894 (2015).
- Barnum, K. J., O'Connell, M. J. Cell cycle regulation by checkpoints. Methods in Molecular Biology. 1170, 29-40 (2014).
- Gratzner, H. G. Monoclonal antibody to 5-bromo- and 5-iododeoxyuridine: A new reagent for detection of DNA replication. Science. 218 (4571), 474-475 (1982).
- Salic, A., Mitchison, T. J. A chemical method for fast and sensitive detection of DNA synthesis in vivo. Proceedings of the National Academy of the Sciences of the United States of America. 105 (7), 2415-2420 (2008).
- Kim, J. Y., et al. The value of phosphohistone H3 as a proliferation marker for evaluating invasive breast cancers: A comparative study with Ki67. Oncotarget. 8 (39), 65064-65076 (2017).
- Homem, C. C., Knoblich, J. A. Drosophila neuroblasts: a model for stem cell biology. Development. 139 (23), 4297-4310 (2012).
- Daul, A. L., Komori, H., Lee, C. Y. EdU (5-ethynyl-2'-deoxyuridine) labeling of Drosophila mitotic neuroblasts. Cold Spring Harbor Protocols. 2010 (7), 5461 (2010).
- Chippalkatti, R., Egger, B., Suter, B. Mms19 promotes spindle microtubule assembly in Drosophila neural stem cells. PLoS Genetics. 16, 1008913 (2020).
- Daul, A. L., Komori, H., Lee, C. Y. Immunofluorescent staining of Drosophila larval brain tissue. Cold Spring Harbor Protocols. (7), (2010).
- Mollinari, C., Lange, B., Gonzalez, C. Miranda, a protein involved in neuroblast asymmetric division, is associated with embryonic centrosomes of Drosophila melanogaster. Biology of the Cell. 94 (1), 1-13 (2002).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Nag, R. N., Niggli, S., Sousa-Guimaraes, S., Vazquez-Pianzola, P., Suter, B. Mms19 is a mitotic gene that permits Cdk7 to be fully active as a Cdk-activating kinase. Development. 145 (2), (2018).
- Cabernard, C., Doe, C. Q. Live imaging of neuroblast lineages within intact larval brains in Drosophila. Cold Spring Harbor Protocols. 2013 (10), (2013).
- Hartenstein, V., Spindler, S., Pereanu, W., Fung, S. The development of the Drosophila larval brain. Advances in Experimental Medicine and Biology. 628, 1-31 (2008).
- Hebar, A., Rutgen, B. C., Selzer, E. NVX-412, a new oncology drug candidate, induces S-phase arrest and DNA damage in cancer cells in a p53-independent manner. PLoS One. 7, 45015 (2012).
- Wyllie, F. S., et al. S phase cell-cycle arrest following DNA damage is independent of the p53/p21(WAF1) signalling pathway. Oncogene. 12 (5), 1077-1082 (1996).
- Xu, X., et al. Inhibition of DNA replication and induction of S phase cell cycle arrest by G-rich oligonucleotides. Journal of Biological Chemistry. 276 (46), 43221-43230 (2001).
- Duronio, R. J. Developing S-phase control. Genes & Development. 26 (8), 746-750 (2012).
- Turrero Garcia, M., Chang, Y., Arai, Y., Huttner, W. B. S-phase duration is the main target of cell cycle regulation in neural progenitors of developing ferret neocortex. Journal of Comparative Neurology. 524 (3), 456-470 (2016).
- Pereira, P. D., et al. Quantification of cell cycle kinetics by EdU (5-ethynyl-2'-deoxyuridine)-coupled-fluorescence-intensity analysis. Oncotarget. 8 (25), 40514-40532 (2017).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132 (3), 487-498 (2008).
- Zielke, N., et al. Fly-FUCCI: A versatile tool for studying cell proliferation in complex tissues. Cell Reports. 7 (2), 588-598 (2014).
- Shen, Y., Vignali, P., Wang, R. Rapid profiling cell cycle by flow cytometry using concurrent staining of DNA and mitotic markers. Bio-protocol. 7 (16), 2517 (2017).
- Berger, C., et al. FACS purification and transcriptome analysis of drosophila neural stem cells reveals a role for Klumpfuss in self-renewal. Cell Reports. 2 (2), 407-418 (2012).
- Harzer, H., Berger, C., Conder, R., Schmauss, G., Knoblich, J. A. FACS purification of Drosophila larval neuroblasts for next-generation sequencing. Nature Protocols. 8 (6), 1088-1099 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon