Method Article
MS2-Benzeşim Saflaştırma gram-pozitif bakterilerde RNA Dizilimi ile birleştiğinde
Bu Makalede
Özet
MAPS teknolojisi, belirli bir düzenleyici RNA in vivo'nun targetom'ını incelemek için geliştirilmiştir. İlgi çekici sRNA, RNA ortaklarının birlikte arındırılmasını ve RNA dizilimi ile tanımlanmasını sağlayan bir MS2 aptamer ile etiketlenmiştir. Bu modifiye protokol özellikle Gram-pozitif bakteriler için uygundur.
Özet
Yaşamın bakteriyel etki alanı arasında küçük düzenleyici RNA'lar (sRNA'lar) yaygın olsa da, birçoğunun işlevleri özellikle mRNA hedeflerini belirlemenin zorluğu nedeniyle kötü karakterize edilmeye devam etmektedir. Burada, ms2-benzeşim saflaştırma RNA Dizileme (MAPS) teknolojisi ile birleştiğinde, belirli bir sRNA in vivo tüm RNA ortaklarını ortaya çıkarmayı amaçlayan değiştirilmiş bir protokol açıkladık. Genel olarak, MS2 aptamer, ilgi çekici sRNA'nın 5' ekstremitesine kaynaşmıştır. Bu yapı daha sonra in vivo olarak ifade edilir ve MS2-sRNA'nın hücresel ortaklarıyla etkileşime girmesini sağlar. Bakteri hasadından sonra hücreler mekanik olarak yutmuş. Ham ekstrakt, daha önce maltoz bağlayıcı proteine kaynaşmış MS2 proteini ile kaplanmış amiloz bazlı bir kromatografi sütununa yüklenir. Bu, MS2-sRNA'nın ve etkileşimli RNA'ların özel olarak yakalanmasını sağlar. Efüzyondan sonra, birlikte saflaştırılmış RNA'lar yüksek verimli RNA dizilimi ve daha sonra biyoinformatik analiz ile tanımlanır. Aşağıdaki protokol Gram-pozitif insan patojeni Staphylococcus aureus'ta uygulanmıştır ve prensip olarak herhangi bir Gram-pozitif bakteriye transpoze edilebilir. Özetlemek gerekirse, MAPS teknolojisi, belirli bir sRNA'nın düzenleyici ağını derinlemesine keşfetmek için etkili bir yöntem oluşturur ve tüm targetome'unun bir anlık görüntüsünü sunar. Bununla birlikte, MAPS tarafından tanımlanan putatif hedeflerin hala tamamlayıcı deneysel yaklaşımlarla doğrulanmış olması gerektiğini akılda tutmak önemlidir.
Giriş
Çoğu bakteri genomunda yüzlerce, belki de binlerce küçük düzenleyici RNA (sRNA) tanımlanmıştır, ancak bunların büyük çoğunluğunun işlevleri tanımlanmamıştır. Genel olarak, sRNA'lar kısa kodlama dışı moleküllerdir, bakteriyel fizyolojide önemli roller oynarlar ve dalgalanan ortamlara adaptasyon1,2,3. Gerçekten de, bu makromoleküller metabolik yolları, stres yanıtlarını ve aynı zamanda virülans ve antibiyotik direncini etkileyen çok sayıda karmaşık düzenleyici ağın merkezinde yer almaktadır. Mantıksal olarak, sentezleri belirli ortam uyaranları (örneğin, besin açlığı, oksidatif veya membran gerilmeleri) tarafından tetiklenir. Çoğu sRNA, kısa ve bitişik olmayan baz eşleştirme yoluyla transkripsiyon sonrası düzeyde birden fazla hedef mRNA düzenler. Genellikle çeviri başlatma bölgeleri için ribozomlarla rekabet ederek çeviri başlatmayı önlerler4. sRNA:mRNA dublekslerinin oluşumu da genellikle belirli RNa'ların işe alınmasıyla hedef mRNA'nın aktif olarak bozulmasına neden olur.
Bir sRNA targetome'nin karakterizasyonu (yani, hedef RNA'larının tüm kümesi), müdahale ettiği metabolik yolların ve cevap verdiği potansiyel sinyalin tanımlanmasını sağlar. Sonuç olarak, belirli bir sRNA'nın işlevleri genellikle targetomundan çıkarılabilir. Bu amaçla, IntaRNA ve CopraRNA5,6,7gibi siliko tahmin araçlarında birkaç tane geliştirilmiştir. Özellikle putatif sRNA ortaklarını belirlemek için sıra tamamlayıcılığına, eşleştirme enerjisine ve potansiyel etkileşim sitesinin erişilebilirliğine güvenirler. Bununla birlikte, tahmin algoritmaları, RNA refakatçileri8'in alt optimal etkileşimleri veya her iki ortağın birlikte ifadesini tercih etme gibi temel eşleştirmeyi etkileyen tüm faktörleri entegre etmez. Doğal sınırlamaları nedeniyle, tahmin araçlarının yanlış pozitif oranı yüksek kalır. Çoğu deneysel büyük ölçekli yaklaşım, etiketli bir RNA bağlayıcı protein (RBP)6,9ile etkileşime giren sRNA:mRNA çiftlerinin birlikte saflaştırılmasına dayanmaktadır. Örneğin, Ligation ve dizileme ile RNA Etkileşimi (RIL-seq) yöntemi, Escherichia coli10 , 11'dekiHfq ve ProQ gibi RNA refakatçileriyle birlikte saflaştırılmış RNA dublekslerinitanımladı. UV-Crosslinking, Ligation And Sequencing of Hybrids (CLASH) adı verilen benzer bir teknoloji, E. coli12,13'teki RNase E- ve Hfq ilişkili sRNA'lara uygulandı. Srna aracılı regülasyonda SRNA aracılı düzenlemede Hfq ve ProQ'nun iyi tanımlanmış rollerine rağmen 8,14,15, sRNA tabanlı regülasyon S. aureus16 , 17,18gibi çeşitli organizmalarda RNA refakatçisiz gibi görünmektedir. RNases ile ilişkili RNA dublekslerinin saflaştırılması Waters ve iş arkadaşları13tarafından gösterildiği gibi mümkün olsa bile, RNases hızlı bozulmalarını tetikledikçe bu zor olmaya devam etmektedir. Bu nedenle, MS2 Benzeşim Saflaştırması RNA Dizilimi (MAPS) yaklaşımı19,20 ile birleştiğindebutür organizmalarda sağlam bir alternatif oluşturur.
Yukarıda belirtilen yöntemlerin aksine, MAPS tüm etkileşime giren RNA'ları yakalamak için yem olarak belirli bir sRNA kullanır ve bu nedenle bir RBP'nin katılımına dayanmaz. Tüm süreç Şekil 1'de tasvir edilir. Kısacası, sRNA 5' ile ms2 kat proteini tarafından özel olarak tanınan MS2 RNA aptamer ile etiketlenmiştir. Bu protein, amiloz reçinesi üzerinde hareketsiz hale getirmek için maltoz bağlayıcı protein (MBP) ile kaynaştırılır. Bu nedenle, MS2-sRNA ve RNA ortakları benzeşim kromatografi sütununda tutulur. Maltoz ile elüsyondan sonra, birlikte saflaştırılmış RNA'lar yüksek verimli RNA dizilimi ve ardından biyoinformatik analiz kullanılarak tanımlanır (Şekil 2). MAPS teknolojisi sonuçta in vivo meydana gelen tüm potansiyel etkileşimlerin etkileşimli bir haritasını çizer.
MAPS teknolojisi başlangıçta patojenik olmayan Gram-negatif bakteri E. coli21'deuygulanmıştır. Dikkat çekici bir şekilde MAPS, hem RyhB hem de RybB sRNA'ları ile özellikle etkileşime giren ve teşvik edici olmayan koşullarda mRNA hedeflerini düzenlemek için herhangi bir sRNA transkripsiyonal gürültüyü önleyen tRNA türevi bir parçanın tanımlanmasına yardımcı oldu. Bundan sonra MAPS, DsrA 22 , RprA 23 , CyaR 23ve GcvB24( Tablo1)gibi diğer E. coli sRNA'larına başarıyla uygulanmıştır. DAHA ÖNCE BILINEN HEDEFLerİ ONAYLADI MAPS, bu iyi bilinen sRNA'ların targetome'larını genişletti. Son zamanlarda, MAPS Salmonella Typhimurium'da gerçekleştirildi ve SraL sRNA'nın rho mRNA'ya bağlandığını, transkripsiyon sonlandırma faktörü25için kodlama yaptığını ortaya koydu. Bu eşleştirme sayesinde SraL, rho mRNA'yı Rho'nun kendisi tarafından tetiklenen erken transkripsiyon sonlandırmasından korur. İlginçtir ki, bu teknoloji sRNA'larla sınırlı değildir ve tRNA türevi bir parça 26 ve mRNA22'nin 5'çevrilmemiş bir bölgesinin kullanılmasıyla örneklenen her türlü hücresel RNA'ya uygulanabilir (Tablo 1).
MAPS yöntemi patojenik Gram-pozitif bakteri S. aureus19'ada uyarlanmıştır. Özellikle, liziz protokolü, Gram negatif bakterilerden daha kalın bir hücre duvarı nedeniyle hücreleri verimli bir şekilde kırmak ve RNA bütünlüğünü korumak için yaygın olarak değiştirilmiştir. Bu uyarlanmış protokol zaten RsaA27, RsaI28 ve RsaC29'un interactom'ınıçözdü. Bu yaklaşım, bu SRNA'ların hücre yüzeyi özelliklerinin düzenleyici mekanizmaları, glikoz alımı ve oksidatif stres yanıtlarındaki önemli rolü hakkında fikir verdi.
2015 yılında E. coli'de geliştirilen ve uygulanan protokol son zamanlarda ayrıntılı olarak açıklanmıştır30. Burada, patojenik olmayan veya patojenik (güvenlik önlemleri) olsun, Gram-pozitif (daha kalın hücre duvarı) bakterilerdeki sRNA düzenleyici ağları incelemek için özellikle uygun olan değiştirilmiş MAPS protokolünü sunuyoruz.
Protokol
1. Tamponlar ve ortamlar
- MAPS denemeleri için aşağıdaki arabellekleri ve ortamları hazırlayın:
- Tampon A (150 mM KCl, 20 mM Tris-HCl pH 8, 1 mM MgCl2 ve 1 mM DTT)
- Tampon E (250 mM KCl, 20 mM Tris-HCl pH 8, 12 mM maltoz, %0,1 Triton, 1 mM MgCl2 ve 1 mM DTT)
- RNA yükleme tamponu (8 M ürede %0,025 ksilen siyanol ve %0,025 bromofenol mavisi)
- Beyin Kalbi İnfüzyonu (BHI) ortamı (12,5 g baldır beyni, 10 g pepton, 5 g sığır kalbi, 5 g NaCl, 2,5 g Na2HPO4 ve 1 L için 2 glikoz)
- Lysogeny Et Suyu (LB) ortamı (10 g pepton, 5 g maya özü ve 1 L için 10 g NaCl) - Kuzey blot tahlilleri için aşağıdaki arabellekleri hazırlayın:
- Blokaj çözeltisi (1x maleik asit ve% 1 bloke reaktif)
- Hibridizasyon çözeltisi (%50 formamid, 5x SSC, %7 SDS, %1 bloke çözeltisi ve %0,2 N-lauryl sarkozin, 50 mM sodyum fosfat). Çözünecek ajitasyon ile ısıtın.
DİkKAT: Her ürünle ilgili güvenlik önlemlerine dikkat edin.
- 1 M sodyum fosfat (58 mM sodyum fosfat dibasik ve 42 mM sodyum fosfat monobasik)
- Salin, sodyum sitrat (SSC) tampon, 20x konsantre (3 M NaCl ve 300 mM trisyot sitrat)
2. Güvenlik sorunları
- Seviye 2 muhafaza laboratuvarında uygulanabilir patojenik bakterileri içeren tüm adımları uygulayın.
NOT: Lizizden sonra dışarıya sadece hücre özleri alınabilir (adım 5). - Laboratuvar önlüğü ve eldiven giy.
- Bileklerin kapalı olduğundan emin olun.
- Biyolojik güvenlik kabinini (Sınıf II) bir dezenfektan çözeltisi ile temizleyin.
- Bakterilere maruz kalan katı atıkları uygun biyomedikal depo gözüne atın.
- Kontamine sıvı içeren şişeleri bir dezenfektan çözeltisi ile tedavi edin. Sonra, bir lavaboya atın.
- Ellerinizi ve bileklerinizi sabunla dikkatlice yıkayın ve seviye 2 muhafaza laboratuvarından ayrılmadan önce laboratuvar önlüğü çıkarın.
3. Plazmid yapımı
NOT: Klonlama amacıyla, öncelikle endojen sRNA'nın sınırlarını belirlemek çok önemlidir. pCN51-P3 ve pCN51-P3-MS2 plazmidleri Tomasini ve ark. P3 promotörü, sRNA'nın hücre yoğunluğuna bağlı bir şekilde yüksek şekilde ifadesini sağlar (yani, bakteriler büyümenin sabit aşamasına girdiğinde). Bu büyüme aşamasında birçok stafilokok sRNA'sı birikir.
- Yüksek kaliteli bir DNA polimeraz ve bir PCR makinesi kullanarak sRNA dizisini PCR ile güçlendirin. Üreticinin talimatlarını dikkatlice izleyin ve daha fazla bilgi için Garibyan ve Avashia (2013)31'i okuyun.
- Belirli astarları tasarlamak için aşağıdaki şablonları kullanın:

ve ileri ve ters astarlar için sırasıyla 5'-CGCGGATCC(N)-3'.
NOT: Bu oligonükleotidler, MS2 sırasını (kalın olarak) ilgi çekici sRNA'nın 5' ucuna birleştirmeyi sağlar. Pst I ve BamHI kısıtlama siteleri (altı çizili) amfiloni pCN51-P3 plazmid27'yeklonlamak için MS2-sRNA yapının 5' ve 3' ekstremitelerinde eklenir. (N) gene özgü diziye (15-20 nükleotit) karşılık gelir. - Üreticinin önerilerine göre uygun tamponda 2 U Pst I ve 1 U BamHI ile MS2-sRNA PCR ürününün 1 μg pCN51-P3 plazmidini ve 1 μg'sını sindirin.
- 37 °C'de 1 saat kuluçkaya yatırın ve PCR saflaştırma kiti kullanarak DNA'yı arındırın (bkz. Malzeme Tablosu).
- Sindirilen pCN51-P3 plazmid (300 ng) ve MS2-sRNA amplicon'u (vektör için molar oran:insert = 1:3) 1,5 mL'lik bir tüpte karıştırın. Ligasyon verimliliğini en üst düzeye çıkarmak için Revie ve ark. (1988)32'ye bakın. Her tüpe 1 μL Ligase Tamponu ve 10 U T4 Ligase ekleyin. Ultra saf su ile hacmi 10 μL'ye ayarlayın.
- En az 2 saat boyunca 22 °C'de kuluçkaya yatırın.
- Donmuş DH5α kimyasal olarak yetkin E. coli hücrelerine 50 μL ligasyon karışımı ekleyin. Plazmid dönüşümü ve kimyasal olarak yetkin hücreler hakkında daha fazla bilgi edinmekiçin Seidman ve ark.
- Buzda 30 dakika kuluçkaya yaslanın.
- Isı bloğu veya su banyosu kullanarak dönüşüm tüpünün ısı şoku (42 °C'de 45 sn).
- 900 μL LB orta ekleyin ve 37 °C'de 30 dakika kuluçkaya yatırın.
- Ampisilin (100 μg/μL) ile desteklenmiş bir LB agar plakasındaki bakteriyel süspansiyonun plakası 100 μL.
NOT: pCN51-P3 vektörü, yalnızca pCN51-P3-MS2-sRNA plazmidini taşıyan E. coli klonlarını seçmeyi sağlayan bir ampisilin direnç genini kodlar. - Bir plazmid DNA miniprep kiti kullanarak ampisilin (100 μg/μL) varlığında yetişen bir gecede bakteri kültüründen (5 mL) pCN51-P3-MS2-sRNA plazmidini çıkarın (bkz. Malzeme Tablosu).
- Sanger'ın34'ü sıralayarak aşağıdaki astar olan 5'-TCTCGAAAATAATAGAGGG-3' ile yapıyı doğrulayın.
- pCN51-P3-MS2-sRNA plazmidini DC10B kimyasal olarak yetkin E. coli hücrelerine dönüştürün. 3.7 ile 3.11 arasında olan adımları yineleyin.
- pCN51-P3-MS2-sRNA plazmidini çıkarın (bkz. adım 3.12) ve 1-5 μg plazmid DNA'yı bir elektroporasyon aparatı kullanarak HG001 ΔsRNA elektro-uyumlu S. aureus hücrelerine dönüştürün. Üreticinin talimatlarını dikkatlice izleyin. Grosser ve Richardson (2016)35'i okuyarak elektro-rekabetçi S. aureushazırlama yöntemleri hakkında daha fazla bilgiedinin.
DİkKAT: Bu adım patojenik bakterilerin işlenmesini içerir (bkz. adım 2). - 900 μL BHI orta ekleyin ve 3 saat boyunca 37 °C'de kuluçkaya yatırın.
- Santrifüj 1 dakika 16.000 x g. Üstnatant atın.
- Peletin 100 μL BHI'de yeniden süzülür ve eritromisin (10 μg/μL) ile desteklenmiş BHI agar plakalarındaki bakteri süspansiyonu plakalayın.
NOT: pCN51-P3 vektörü ayrıca, yalnızca pCN51-P3-MS2-sRNA plazmidini taşıyan S. aureus klonlarını seçmeyi sağlayan bir eritromisinin direnç genini kodlar.
4. Bakteri hasadı
DİkKAT: Bu adım patojenik bakterilerin işlenmesini içerir (bkz. adım 2).
- PCN51-P3-MS2-sRNA veya pCN51-P3-MS227 plazmidlerini taşıyan bir suş kolonisini, mükerrer olarak eritromisinin (10 μg/μL) takviyeli 3 mL BHI ortamında büyütün.
- Her bir gece kültürünü 50 mL (≈1/100) taze BHI ortamında seyreltin ve 0,05'lik bir OD600nm'ye ulaşmak için eritromisinin (10 μg/μL) takviyesini yaptırın. 250 mL sterilize şişe kullanın (5:1 şişe-orta oranı).
NOT: Orta ve büyüme koşulları çalışılan sRNA'nın ifade modeline göre ayarlanmalıdır. - 37 °C'de kültürleri 6 saat boyunca 180 rpm'de sallayarak büyütün.
- Her kültürü 50 mL santrifüj tüpüne aktarın.
- 4 °C'de 15 dakika boyunca 2.900 x g'da santrifüj. Üstnatant atın.
- Peletleri buzda tutun ve doğrudan mekanik hücre lizisi (adım 5) yapın veya peletleri -80 °C'de dondurup saklayın.
5. Mekanik hücre lizisi
DİkKAT: Aşağıdaki adımlar buz üzerinde yapılmalı ve tamponlar 4 °C'de olmalıdır. Eldiven kullanın ve örnekleri RNases'ten korumak için tüm önlemleri alın.
- 5 mL Tampon A'da peletleri (adım 4,6) yeniden dirsintir.
- Yeniden depolanan hücreleri 3,5 g silika boncuk (0,1 mm) ile 15 mL santrifüj tüplerine aktarın.
- Tüpleri mekanik hücre liziz cihazına yerleştirin(bkz. Malzeme Tablosu). 40 sn'lik bir döngüyü 4,0 m/s'de çalıştırın.
NOT: Hücreleri kırmak için bir döngü yeterli değilse, numuneleri buzda tutarken cihazın 5 dakika soğumasını bekleyin. Ardından, 40 sn'lik bir döngüyü 4.0 m/ s'de tekrarlayın. Hücre lizizinin verimliliği, üst tabak BHI-agar plakasına kaplanarak test edilebilir. - 15 dakika boyunca 15.700 x g'da santrifüj. Üst saltantayı kurtarın ve buzda tutun.
6. Sütun hazırlama
DİkKAT: Amiloz reçinesinin kurumasına izin vermemeye dikkat edin. Gerekirse, sütunu bir uç kapakla kapatın. Benzeşim saflaştırmasına başlamadan önce tüm çözümleri hazırlayın.
- Sütun rafı içine kromatografi sütunu koyun (bkz. Malzeme Tablosu).
- Sütun ucunu çıkarın ve kolonu ultra saf suyla yıkayın.
- 300 μL amiloz reçinesi ekleyin.
- Sütunu 10 mL Tampon A ile yıkayın.
- 1.200 pmol MBP-MS2 proteinini 6 mL Tampon A'da seyreltin ve sütuna yükleyin.
- Sütunu 10 mL Tampon A ile yıkayın.
7. MS2 benzeşim saflaştırması (Şekil 1)
- Hücre lisatını sütuna yükleyin.
NOT: Toplam RNA 'yı (adım 8) çıkarmak ve Kuzey lekesi (adım 9) ve transkriptomik (adım 10) analizini yapmak için hücre lisatının 1 mL'lik kısmını (Ham özü, CE) tutun. - Akış fraksiyonunu (FT) temiz bir toplama tüpünde toplayın.
- Sütunu 10 mL Tampon A ile 3 kez yıkayın.
- Sütunu 1 mL Tampon E ile yağlayın ve elution fraksiyonunu (E) 2 mL mikrotüpte toplayın.
- Toplanan tüm fraksiyonları RNA ekstraksiyonu (adım 8) kadar buzda tutun veya daha sonra kullanmak üzere -20 °C'de dondurun.
8. Toplanan fraksiyonların RNA ekstraksiyonu (CE, FT, W ve E)
- RNA ekstraksiyonu için her kesirden 1 mL (FT ve W dahil) kullanın.
- 1 hacim fenol ekleyin. Kuvvetlice karıştırın.
DİkKAT: Fenol uçucu ve aşındırıcıdır, dikkat edin ve duman kaputunun altında güvenli bir şekilde çalışın. - 20 °C'de 10 dakika boyunca 16.000 x g'da santrifüj.
- Üst fazı temiz bir 2 mL mikrotüple aktarın.
- 1 hacim kloroform/izoamil alkol (24:1) ekleyin ve 8,3 ila 8,4 adımlarını tekrarlayın.
DİkKAT: Duman kaputunun altında güvenle çalışın. - 2,5 hacim soğuk etanol% 100 ve 1/10 hacim 3 M sodyum asetat (NaOAc) pH 5,2 ekleyin.
- -20 °C'de gece çökelt.
NOT: Yağış, etanol/kuru buz banyosunda 20 dakika veya -80 °C'de 2 saat boyunca da yapılabilir. - 4 °C'de 15 dakika boyunca 16.000 x g'da santrifüj. Peletin rahatsız etmemeye dikkat ederken bir pipetle etanol'u yavaşça çıkarın.
DİkKAT: RNA peleti her zaman görünmez ve bazen etanol varlığında gevşektir. - 500 μL% 80 soğuk etanol ekleyin.
- 4 °C'de 5 dakika boyunca 16.000 x g'da santrifüj.
- Etanol yavaşça pipetleme ile atın. Peletin vakum konsantratör kullanarak kurulayın, çalışma modunda 5 dk.
- Peletin uygun hacimde (15-50 μL) ultra saf suda yeniden sulansın. Peletin daha sonra kullanılmak üzere -20 °C'de dondurulması.
- Spektrofotometre/florometre kullanarak RNA miktarını (260 nm) ve kaliteyi (260/280 ve 260/230 dalga boyu oranları) değerlendirin (bkz. Malzeme Tablosu). Üreticinin talimatlarını dikkatlice izleyin.
NOT: 3-4 μg genellikle elüsyon fraksiyonunda (E) elde edilir. Bu esas olarak test edilen koşullara bağlıdır.
9. Ms2 benzeşim saflaştırmasının Kuzey blot36 ile analizi
- 10 μL ultra saf suda 5 μg RNA CE, FT, W fraksiyonları ve 500 ng E fraksiyonunu seyreltin ve 10 μL RNA yükleme tamponu ile karıştırın.
- 90 °C'de 3 dk kuluçkaya yaslanın.
- Numuneleri 20 mM guanidiyum tiyosiyanat ile desteklenmiş% 1 agarose jel kuyusuna yükleyin ve jeli 4 °C'de TBE 1x tamponda 100-150 V'da çalıştırın. Daha fazla bilgi için Koontz (2013)37'ye bakın.
- RNA'ları bir nitroselüloz membran üzerinde vakum transferi ile 1 saat veya bir gecede kapillarite transferi için aktarın.
NOT: Kapillarite yöntemi büyük RNA'lar için daha etkilidir. - Membrandaki UV çapraz bağlantı RNA'ları (254 nm'de 120 mJ) ultraviyole çapraz bağlantı kullanarak.
- Membranı, RNA tarafı yukarı bakacak şekilde bir melezleme şişesi içine yerleştirin.
- Önceden ısıtılmış 10-20 mL hibridizasyon çözümü ekleyin. 68 °C'de 30 dk kuluçkaya yatır.
- Çözeltiyi atın ve sRNA'ya özgü probun 1 μL'si ile desteklenmiş 10-20 mL taze hibridizasyon çözeltisi ekleyin. 68 °C'de bir gecede kuluçkaya yaslanın.
NOT: DIG etiketli RNA probu, DIG RNA etiketleme kiti kullanılarak ve üreticinin yönergelerine uyularak sentezlenmiştir. Alternatif olarak, radyolabelled prob kullanılabilir. - Membranı 10-20 mL yıkama çözeltisi 1 (2x SSC ve% 0.1 SDS) ile 20 ° C'de 5 dakika boyunca yıkayın. Bir kez tekrarlayın.
- Membranı 68 °C'de 15 dakika boyunca 10-20 mL yıkama çözeltisi 2 (0,2x SSC ve% 0,1 SDS) ile yıkayın. Bir kez tekrarlayın.
- 20 °C'de en az 30 dakika boyunca 10-20 mL blokaj çözeltisi ile kuluçkaya yaslanın.
- Çözeltiyi atın ve alkali fosfataz için konjuge edilmiş poliklonal anti-digoksigenin antikoru (1/1000) ile desteklenmiş blokaj çözeltisinin 10-20 mL'lik kısmını ekleyin. 20 °C'de 30 dk kuluçkaya yaslanın.
- Membranı 10-20 mL yıkama çözeltisi 3 (1x maleik asit ve% 0.3 Ara 20) ile 20 ° C'de 15 dakika boyunca yıkayın. Bir kez tekrarlayın.
- Membranı algılama çözeltisinin 10-20 mL'si (0,1 M Tris HCl ve 0,1 M NaCl pH 9,5) ile 20 °C'de 5 dk ile kuluçkaya yaslanın.
- Membranı plastik bir filme koyun ve substratla ıslatın (bkz. Malzeme Tablosu). Karanlıkta 5 dakika kuluçkaya yaslanın.
- Zarı plastik bir filme kapatın. Zarı otoradyografi kasetine koy.
- Membranı özel karanlık odada bir otoradyografi filmine maruz bırak.
NOT: Sergi süresi, birkaç saniyeden dakikaya kadar sinyal gücüne bağlıdır. - Açığa çıkan filmi otomatik olarak gelişen bir cihazda ortaya çıkarmak.
10. Numunelerin RNA dizilimi için hazırlanması
NOT: Bu adım yalnızca E ve CE kesirlerinden çıkarılan RNA'larla ilgilidir.
- Her numuneye 10 μL 10x DNase tamponu ve DNase I (1 U/μg işlenmiş RNA) ekleyin. 100 μL'lik son hacim için su ekleyin.
- 37 °C'de 1 saat kuluçkaya yaslanın.
- RNA'ları daha önce açıklandığı gibi ayıklayın ve arındıryın (adım 8.2 ila 8.11).
- RNA peletini 20 μL ultra saf suda yeniden hissedi.
NOT: Kalan DNA'nın varlığı PCR ve spesifik astarlar (örneğin, 16S geni) kullanılarak kontrol edilebilir. - Mikroakışkan bazlı elektroforez analiz sistemi kullanarak RNA miktarını ve kalitesini değerlendirin (bkz. Malzeme Tablosu).
NOT: 1 μg genellikle DNase tedavisinden sonra elüasyon fraksiyonunda (E) elde edilir. - Ribozomal RNA'ları bakteriyel bir rRNA tükenme kiti ile çıkarın.
NOT: Büyük ve bol miktarda RNA (yani rRNA'lar) benzeşim sütunuyla özel olarak etkileşime girme eğilimindedir. Bu adımı gerçekleştirmek için 500 ng çıkarılmış RNA gereklidir. - Yine mikroakışkan bazlı elektroforez analiz sistemi kullanarak RNA miktarını ve kalitesini değerlendirin.
- CDNA kütüphane hazırlık kiti kullanarak ve üreticinin talimatlarını izleyerek 10-20 ng ribodepleted RNA ile cDNA kütüphaneleri hazırlayın.
- Elde edilen kitaplıkları bir sıralama aleti kullanarak sıralayın (örneğin, tek uçlu, 150 bp; bkz. Malzeme Tablosu).
NOT: Örnek başına 5-10 milyon okuma genellikle yeterlidir.
11. RNAseq veri analizi (Şekil 2)
- FastQ sıralama dosyalarını sıralama platformundan indirin.
- Roscoff biyolojik istasyonunun(https://galaxy.sb-roscoff.fr/)Galaxy örneğine erişim ve giriş yapın.
NOT: Bahsedilen her algoritma arama çubuğu kullanılarak kolayca bulunabilir. Her araç için bir kullanım kılavuzu sağlanır.
DİkKAT: Gerekli araçların sürümü genel Galaxy server38. - Veri Al simgesine tıklayın ve ardından Bilgisayarınızdan Dosya Yükle 'yetıklayın. Her MS2 denetiminin ve MS2-sRNA örneklerinin FastQ sıralama dosyasını karşıya yükleyin. Ayrıca FASTA referans genom dosyasını ve GFF ek açıklama dosyasını yükleyin.
- FastQC Okuma Kalitesi raporlarını çalıştırın (Galaxy Sürüm 0.69).
NOT: Bu araç ham dizilerin kalite değerlendirmesini sağlar (örneğin, kalite puanı, adaptör dizilerinin varlığı). - Adaptör dizilerini ve düşük kaliteli okumaları önemli ölçüde kaldırmak için Trimmomatic esnek okuma kırpma aracını (Galaxy Sürüm 0.36.6) çalıştırın. Kitaplık hazırlama için kullanılan bağdaştırıcı sıralarını belirtin (örneğin, TruSeq 3, tek uçlu). Aşağıdaki Trimmomatic işlemlerini ekleyin: SLIDINGWINDOW (Baz sayısı=4; Ortalama kalite=20) ve MINLEN (En az okuma uzunluğu=20).
- FastQC Okuma Kalitesi raporlarını (Galaxy Sürüm 0.69) tekrar çalıştırın.
- Bowtie2'yi çalıştırın - harita referans genoma karşı okur (Galaxy Sürüm 2.3.2.2). Varsayılan ayarlarla (çok hassas yerel) okumaları eşlemek için geçmişteki Genom Başvurusu FASTA dosyasını kullanın.
NOT: Bowtie2 aracı tarafından oluşturulan BAM dosyası Bütünleştirici Genomik Görüntüleyici (IGV) kullanılarak görselleştirilebilir. İlişkili BAI dosyası da gerekli olacaktır. - İsteğe bağlı olarak, BAM veri kümesi (Galaxy Sürüm 2.0) için istatistikleri derleyen Flagstat'ı çalıştırın.
- GFF ek açıklama dosyasındaki okuma çakışan özellikleri hizalayan htseq-count - Count (Galaxy Version 0.6.1) çalıştırın. Kesişim (boş olmayan) modunu kullanın.
- Htseq-count çözümlemesinden tüm ham sayım dosyalarını tek bir Zip dosyasına arşivle.
- Verileri karşılaştırmak için SARTools DESeq2'yi çalıştırın (Galaxy Sürüm 1.6.3.0). Ham sayım dosyalarını içeren Zip dosyasını ve denemeyi açıklayan sekmeyle ayrılmış bir dosya olan tasarım dosyasını sağlayın. Tasarım dosyasını oluşturmak için sağlanan yönergeleri dikkatle izleyin.
Sonuçlar
Temsili sonuçlar, RsaC targetome'nin S. aureus29 'daki çalışmasındankaynaklanmaktadır. RsaC alışılmadık bir 1.116 nt uzunluğunda sRNA'dır. 5' ucu birkaç yinelenen bölge içerirken, 3' ucu (544 nt) yapısal olarak bağımsızdır ve mRNA hedefleriyle tahmin edilen tüm etkileşim alanlarını içerir. Bu sRNA'nın ekspresyişi manganez (Mn) az olduğunda indüklenmiştir, bu da sıklıkla konak immün yanıtı bağlamında karşılaşılan bir durumdur. MAPS teknolojisini kullanarak, RsaC ile doğrudan etkileşime giren birkaç mRNA belirledik ve oksidatif stres (sodA, ldh1 ve sarA) ve metalle ilgili (znuBC-zur ve sufCDSUB) yanıtlarındaki önemli rolünü ortaya koyduk.
MS2-sRNA yapısı ve deneysel koşulların doğrulanması
MAPS deneylerini yapmadan önce, çalışılan sRNA'nın optimum ifade koşullarını belirlemek önemlidir. Yerel olmayan bir promotör kullanılırsa, hedefleri mevcut olduğunda MS2-sRNA yapısını üretmeye kesin olarak yardımcı olur. Ayrıca, MS2-sRNA yapısı boyut, kararlılık, ifade ve işlev açısından dikkatlice doğrulanmalıdır. MS2 aptamer, RsaC'nin 3' kısmına karşılık gelen tam uzunlukta RsaC (MS2-RsaC1116)veya daha kısa formun (MS2-RsaC544) 5'ucuna kaynaştırıldı. Her iki yapı da S. aureus HG001 ΔrsaC'deki bağımlı P3 promotörü algılayan çekirdek kontrolü altında in vivo olarak ifade edilmiştir. RsaC geninin silinmesi endojen RsaC ve MS2-RsaC arasındaki rekabeti önler. Kontrol olarak sadece MS2 etiketiyle aynı vektörü içeren vahşi tip gerinim kullanıldı. Bu denetim, MS2 etiketiyle oluşan belirsiz etkileşimlerin çıkarılmasına izin verir.
Yapıları doğrulamak ve ifade şekillerini görselleştirmek için, bakteri hücreleri BHI ortamında 37 °C'de 2 saat, 4 saat ve 6 saat büyümeden sonra hasat edildi. RNA ekstraksiyonundan sonra, RsaC'ye özgü DIG probu kullanılarak Kuzey leke analizi yapıldı (Şekil 3A). Endojen RsaC (şerit 1-3) seviyesi, 6 saatlik büyümeden sonra önemli ölçüde artmış ve MAPS deneyleri için bu zaman noktasının seçilmesini haklı çıkarmıştır. Daha da önemlisi, MS2-RsaC544 (şerit 7-9) ve MS2-RsaC1,116 (şerit 10-12) seviyeleri 6 saat'te endojen RsaC ile karşılaştırılabilirdi. Bu nedenle, RsaC'nin endojen ifade desenini taklit etmelidirler. RsaC'nin daha büyük ama küçük bir formu ayırt edilebilirdi ve verimsiz bir transkripsiyonun sona eki olabilir. Bu fenomen, bir MS2-sRNA plazmid21'dengüçlü bir promotörün kontrolü altında ifade edildiğinde sıklıkla gözlenir. Sapkın transkripsiyon sonlandırması veya bozulmasından kaynaklanan daha kısa formlar gözlenmedi.
MS2 aptamerinin 5' sRNA'lara eklenmesi de uygun katlamalarını bozabilir ve işlevlerini etkileyebilir. Bu adım, RsaC olarak son derece yapılandırılmış sRNA'lar için kritik öneme sahiptir. Bu nedenle MS2-sRNA aktivitesi test edilmeli ve mümkün olduğunda endojen sRNA ile karşılaştırılmalıdır. Daha önce bilinen bir hedef veya gözlemlenebilir bir fenotip onu izlemeye yardımcı olabilir. Örneğin, RsaC'nin hücre içi ROS birikimi üzerindeki etkisi MS2-RsaC544 ve MS2-RsaC1.116 yapı29'udoğrulamak için kullanılmıştır.
Benzeşim saflaştırması sırasında toplanan kesirlerin analizi
RNA'lar WT geriniminde sadece MS2 etiketini ifade eden CE, FT ve E fraksiyonlarından veMS2-RsaC 544 yapısını ifade eden Δ rsaC geriniminden çıkarılmıştır. Kuzey blot analizini kullanarak 1.116 nt uzunluğunda endojen RsaC'nin elution fraksiyonunda zenginleştiğini gösterdik, ancak özellikle benzeşim sütunuyla etkileşime girdiği ortaya çıktı(Şekil 3B, şeritler 2-3). Aynı fenomeni MS2-RsaC1,116 ile gözlemledik (veriler gösterilmedi). Bu kesinlikle uzunluğu ve karmaşık ikincil yapısından kaynaklanmaktadır. Bu nedenle, MAPS denemelerini gerçekleştirmek için yalnızca 3' kısmına karşılık gelen daha az yapılandırılmış ve daha kısa bir RsaC formu (544 nt) kullanılmıştır. Şekil 3B'de,MS2-RsaC544, MS2-MBP füzyon proteini (şerit 6) tarafından başarıyla korunduğunu gösteren elution fraksiyonunda oldukça zenginleştirilmiştir. Şekil 3A'daolduğu gibi MS2-RsaC544'ün daha büyük ama küçük bir formu gözlenmiştir. Burada, yıkamaların katılığı ve sayısı, spesifik olmayan bağlamayı azaltacak veya aksine gerçek etkileşim ortaklarının kaybını sınırlamak için ayarlanabilir.
MAPS analizinden sonra putatif mRNA hedeflerinin doğrulanması
Biyoinformatik analizden sonra, putatif mRNA hedefleri, DeSeq2 (Şekil 2)kullanılarak elde edilen MS2-sRNA ve MS2 kontrolü arasındaki Kat değişimine göre listelenir. Örneğin, MS2-RsaC544 MAPS veri29, S. aureus'tabir süperoksit dismutaz için kodlayan sodA mRNA'nın ana hedef olduğunu (en iyi isabet, daha yüksek Katlama değişimi) önerdi. MS2 benzeşim saflaştırmasından sonra sodA'yaözgü dig probu ile gerçekleştirilen Kuzey blot analizi, SODA'nın MS2 kontrolüne kıyasla MS2-RsaC544 ile verimli bir şekilde birlikte zenginleştiğini göstermektedir (Şekil 3C).
CE fraksiyonunda sistematik olarak küresel bir transkriptomik analiz yapılır. MAPS verilerinin ve bu transkriptomik analizin karşılaştırılması, zenginleştirme oranının ayarlanmasına yardımcı olur ve olası bir hedef hiyerarşisini ortaya koymaktadır. Gerçekten de, MS2 benzeşimi saflaştırmasından sonra oldukça zenginleştirilmiş kötü ifade edilmiş bir mRNA, kesinlikle son derece zenginleştirilmiş ve yüksek oranda ifade edilen bir mRNA'dan daha büyük bir bağlayıcılığa sahiptir.
MAPS tarafından tanımlanan tüm adayların, elektroforezi hareketlilik kayması tahlilleri (EMSA) veya muhabir gen tahlilleri gibi in vitro ve/veya in vivo deneyler kullanılarak ayrı ayrı doğrulanmış olması gerektiğini unutmayın (daha fazla bilgi için bkz. Jagodnik vd. (2017)39).

Şekil 1. S. aureus'auyarlanmış MAPS protokolünün şematik çizimi. Plazmid yapımından veri analizine kadar. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
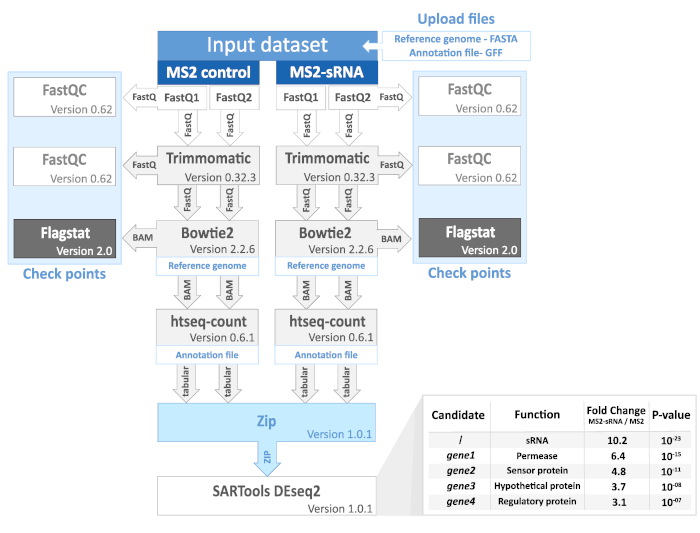
Şekil 2. MAPS analiz iş akışı ve işlenmiş veriler. Her adım, onay noktası ve dosya biçimi temsil edilir (ayrıca bkz. adım 11). FastQ formatı, DNA dizileri ve ilgili kalite puanlarından oluşan bir metin dosyasıdır. BAM biçimi, hizalanmış diziler içeren sıkıştırılmış bir dosyadır. Tablo biçimi, her gen için sayımları olan sekmeyle ayrılmış bir metin dosyasıdır. Sonuç grafiği biyoinformatik analizden sonra elde edilen veri türünü göstermektedir. Sunulan sonuçlar hayalidir ve herhangi bir çalışmadan kaynaklanmıyor. Daha fazla ayrıntı için, temel öğreticiler Galaxy Project web sitesinde (https://galaxyproject.org/) mevcuttur. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 3: Doğrulama ve MAPS denetimlerini oluşturur. A. Endojen RsaC sRNA ve ilgili MS2 yapılarının kuzey leke analizi. WT suşu pCN51-P3-MS2 (kontrol) ve ΔrsaC mutant suşunu taşır pCN51-P3-MS2, pCN51-P3-MS2-RsaC544 veya pCN51-P3-MS2-RsaC1,116. BHI'de 37 °C'de 2 saat, 4 saat ve 6 saat büyümeden sonra örnekler alındı. Kuzey blot tahlilleri RsaC'ye özgü bir DIG probu kullanılarak gerçekleştirildi. B. RsaC'ye özgü dig probu kullanılarak MS2 benzeşimli saflaştırma fraksiyonlarının kuzey leke analizi. Ortak saflaştırma WT suşu + pCN51-P3-MS2 (kontrol) ve ΔrsaC mutant suşu + pCN51-P3-MS2-RsaC544kullanılarak gerçekleştirildi. Hücreler BHI'de 37 °C'de 6 saat büyümeden sonra hasat edildi. Ham ekstrakt (CE), akış (FT), elution (E). C. MS2 benzeşimli saflaştırma fraksiyonlarının (CE ve E) sodA'yaözgü dig probu kullanılarak kuzey leke analizi. Ayrıntılar için (B) bakın. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
| Rna | tür | referans |
| Escherichia coli | ||
| RyhB | sRNA | Lalaouna vd. (2015)21 |
| RybB | sRNA | Lalaouna ve ark. (2015) 21 |
| 3'ETSleuZ | tRNA türevi parça | Lalaouna ve Massé (2015)26 |
| DsrA | sRNA | Lalaouna ve ark. (2015) 22 |
| Hns | mRNA (5'UTR) | Lalaouna ve ark. (2015) 22 |
| Cyar | sRNA | Lalaouna ve ark. (2018) 23 |
| RprA | sRNA | Lalaouna ve ark. (2018) 23 |
| GcvB | sRNA | Lalaouna ve ark. (2019) 24 |
| Salmonella Typhimurium | ||
| SraL | sRNA | Silva ve ark. (2019) 25 |
| Staphylococcus aureus | ||
| RsaA | sRNA | Tomasini ve ark. (2017) 27 |
| RsaC | sRNA | Lalaouna ve ark. (2019) 29 |
| RsaI | sRNA | Bronesky ve ark. (2019) 28 |
Tablo 1. MAPS teknolojisi, çeşitli organizmalarda birkaç RNA'nın hedeflemini ortaya çıkardı.
Tartışmalar
Gram-pozitif bakteriler için değiştirilmiş bir protokol
MAPS'in ilk protokolü, E. coli20 , 30model organizmasında sRNA interactom'u incelemek için geliştirilmiştir. Burada, fırsatçı insan patojeni S. aureus'ta sRNA'ya bağımlı düzenleyici ağların karakterizasyonu için uygun olan ve patojenik olsun olmasın diğer Gram-pozitif bakterilere kesinlikle aktarılabilir olan değiştirilmiş bir protokolü açıklıyoruz.
Hücre liziz adımına özellikle dikkat edildi. Fransız basınının yerini mekanik hücre liziz aleti aldı. Bu yöntem Gram-pozitif hücreleri kırmak ve patojenik suşların işlenmesiyle ilgili riskleri sınırlamak için etkilidir. MS2 benzeşim saflaştırmasının verimini artırmak için, amiloz reçinesinde hareketsiz hale getirilen MS2-MBP miktarı büyük ölçüde artmıştır. Bu, yıkamaların sıkılığını ve sayısını ayarlamak için gereklidir.
İlk protokolün aksine, MAPS burada iki biyolojik kopyadan kopya olarak gerçekleştirilir. Elde edilen verilerin sağlamlığını kesin olarak artıran istatistiksel analiz (Şekil 2) uygulamak için bir iş akışı geliştirilmiştir.
MS2 yapısı ve ifadesi
Bu MAPS protokolü stafilokok sRNA'larının targetomlarını tanımlamak için kurulmuştur. MS2-sRNA yapısı, düşük kopyalı bir plazmid numarasından (pCN51, 20 ila 25 kopya/hücre) ve çekirdek algılama bağımlı P3 promotörünün kontrolü altında, esas olarak sabit faz sırasında indüklenen ifade edilir. Bu ifade deseni, S. aureus'ta (rsaa, RsaI ve RsaC) çalışılan sRNA'lara karşılık gelir ve oldukça güçlü bir MS2-sRNA sentezi sağlar. Bununla birlikte, diğer durumlarda, P3 promotörünün uygun olmayabileceğini ve sRNA indüklendiğinde bakterilerin doğal fizyolojik durumunu yansıtmadığını dışlayamayız. MS2-sRNA üretimini kontrol etmenin bir başka yolu da kimyasal olarak indükleyici promotörler (örneğin, tetrasiklin indükleyiciler) kullanmaktır. Bu araçlar MS2-sRNA yapının darbe ifadesine izin verir, ancak bu ayar RNA hedeflerinin birlikte ifade edileceğini garanti etmez. Başka bir dezavantajı, bir plazmidden gelen ifadenin, yüksek kopya numarası vektörü ile daha da vurgulanan, çalışılan sRNA'nın aşırı üretimine yol açabileceğidir. Bu, potansiyel olarak artefactual etkileşimlere veya ilişkili RNA refakatçi işlevlerinin bozulmasına neden olabilir. En uygun alternatif, endojen sRNA geninin 5' ucuna bir MS2 etiketi eklemektir. Böylece, MS2-sRNA kromozomal olarak kodlanacak ve yerel organizatörünün kontrolü altında olacaktır. Bu, çalışılan sRNA'nın endojen ekspresyonunun daha iyi taklit edilmesi ve çalışılan sRNA'nın aşırı üretilmesi nedeniyle önyargıdan kaçınmalıdır. Her durumda, önemli adım MS2-sRNA yapının uzunluğunu, kararlılığını ve işlevselliğini dikkatlice doğrulamaktır, özellikle endojen sRNA üretimini ve işlevini tetikleyen koşullar altında yetiştirilen kültürlerden çıkarılan toplam RNA ile Kuzey blot tahlillerini kullanmaktır. İnkar edilemez bir şekilde, yanlış/işlevsiz bir MS2-sRNA yapının kullanılması, yukarıda açıklanan denetimlerin önemini destekleyerek oluşturulan sonuçları büyük ölçüde etkileyebilir. Son olarak, MS2-sRNA her zaman mRNA hedeflerinin zenginleştirilmesini en üst düzeye çıkarmak için ΔsRNA arka planda üretilir. Ayrıca, sRNA bağımlı RNases alımı ile mRNA hedeflerinin bozulmasını önlemek için RNazlarda (örneğin, silme mutantları veya sıcaklığa duyarlı mutantlar) belirli mutasyonlara sahip konak suşlarında deneyler yapılabilir.
MAPS tarafından tanımlanan adayların deneysel olarak doğrulanması hala gereklidir
Dikkate alınması gereken bir nokta, MAPS'in putatif RNA hedeflerinin bir listesini sağlamasıdır. Bununla birlikte, bazı RNA'lar paylaşılan bir mRNA hedefi veya RNA bağlayıcı protein ile etkileşim yoluyla dolaylı olarak zenginleştirilebilir. Sonuç olarak, ortaya çıkan adayların diğer deneysel yaklaşımlarla onaylanması gerekir. EMSA ve ayak izi deneyleri genellikle bir sRNA'nın putatif RNA hedeflerine doğrudan bağlanmasını görselleştirmek için kullanılır. Daha sonra, in vitro toeprinting tahlilleri, bir sRNA'nın mRNA hedefinin çeviri başlatma üzerindeki etkisini izlemeye yardımcı eder. Son olarak, Kuzey blot ve/veya gen muhabiri tahlilleri (lacZ veya gfp) bu deneysel doğrulama in vivo'yu tamamlar. Tüm bu yaklaşımlar Jagodnik ve ark. (2017)39.
RIL-seq ve CLASH teknolojilerinin aksine MAPS, etkileşim siteleri hakkında doğrudan bilgi sağlamaz. Bununla birlikte, eşlenen okumaların zirvesi, RNA hedefinin kısıtlı bir bölgesinde (örneğin, glyW-cysT-leuZ operon21'in 3'ucu) sıklıkla görülür ve eşleştirme bölgesinin tanımlanmasını kolaylaştırır. Alternatif olarak, intaRNA40gibi tanımlanan hedefteki putatif bağlama sitesini tahmin etmek için çeşitli tahmin araçları kullanılabilir.
MAPS, belirli bir sRNA'nın targetome'sını inceler
MAPS teknolojisi, E. coli ve S. Typhimurium gibi Gram-negatif bakterilerde sRNA targetomunun incelenmesi için uygundur, ancak şimdi S. aureusgibi Gram-pozitif bakterilerde bu değiştirilmiş protokol ile . Temel olarak MAPS, belirli bir zamanda ve belirli büyüme koşullarında oluşturulan RNA:sRNA dublekslerinin anlık görüntüsünü sunar. Ne yazık ki, mRNA hedeflerinin listesi eksik olabilir. Örneğin, bir konyak mRNA hedefi uygunsuz deneysel koşullar nedeniyle kaçırılabilir ve yukarıda belirtilen önlemleri haklı çıkarır.
RIL-seq veya CLASH gibi yaygın olarak kullanılan diğer yöntemlerle karşılaştırıldığında MAPS, etkileşimde bulunan tüm RNA hedeflerini birlikte arındırmak için belirli bir sRNA kullanır. Burada, sRNA:RNA etkileşimi diğer RBP ilişkili dubleksler arasında seyreltilmez ve teorik olarak hedef beni daha derinden karakterize etmesine izin verir. Bununla birlikte, RIL-seq/CLASH ve MAPS yöntemlerinin farklı putatif sRNA hedefleri12,41, bu deneysel yaklaşımların tamamlayıcı olduğunu ortaya koyduğu görülüyor. Bu tutarsızlık, büyüme koşullarındaki farklılıklar ve/veya her yöntemin yarattığı önyargı ile açıklanabilir.
Buna göre, çalışmanın amacı yöntemin seçimini etkilemelidir. SRNA targetomlarının küresel analizi için ve bir RBP sRNA aracılı düzenlemeye dahil olduğunda, RIL-seq ve CLASH yöntemleri tercih edilmelidir. Her iki yaklaşım da belirli bir RBP'ye dayanan düzenleyici ağlara genel bir bakış sağlar ve sonuç olarak çok çeşitli sRNA'ları ve ilişkili hedefleri ortaya çıkarır. Aksine, belirli bir sRNA'nın incelenmesi için veya RBP bilinmedi/dahil olmadığında, MAPS uygun bir seçeneği temsil eder.
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Bu çalışma "Agence Nationale de la Recherche" (ANR, Grant ANR-16-CE11-0007-01, RIBOSTAPH ve ANR-18-CE12- 0025-04, CoNoCo, PR) tarafından desteklendi. Ayrıca labEx NetRNA ANR-10-LABX-0036 ve ANR-17-EURE- 0023 (PR) çerçevesinde, gelecekteki program için yapılan yatırımların bir parçası olarak ANR tarafından yönetilen devletten fon olarak yayınlanmıştır. DL, 753137-SaRNAReg sayılı Marie Skłodowska-Curie Hibe Anlaşması kapsamında Avrupa Birliği'nin Horizon 2020 araştırma ve yenilik programı tarafından desteklendi. E. Massé Lab'deki çalışmalar, Kanada Sağlık Araştırmaları Enstitüleri (CIHR), Kanada Doğa Bilimleri ve Mühendisliği Araştırma Konseyi (NSERC) ve Ulusal Sağlık Enstitüleri NIH Ekibi Grant R01 GM092830-06A1'den verilen hibelerle desteklenmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tube | Sarstedt | 72.690.001 | |
| 15 mL centrifuge tubes | Falcon | 352070 | |
| 2 mL microcentrifuge tube | Starstedt | 72.691 | |
| 2100 Bioanalyzer Instrument | Agilent | G2939BA | RNA quantity and quality |
| 250 mL culture flask | Dominique Dutscher | 2515074 | Bacterial cultures |
| 50 mL centrifuge tubes | Falcon | 352051 | Culture centrifugation |
| Absolute ethanol | VWR Chemicals | 20821.321 | RNA extraction and purification |
| Allegra X-12R Centrifuge | Beckman Coulter | Bacterial pelleting | |
| Ampicilin (amp) | Sigma-Aldrich | A9518-5G | Growth medium |
| Amylose resin | New England BioLabs | E8021S | MS2-affinity purification |
| Anti-dioxigenin AP Fab fragment | Sigma Aldrich | 11093274910 | Northern blot assays |
| Autoradiography cassette | ThermoFisher Scientific | 50-212-726 | Northern blot assays |
| BamHI | ThermoFisher Scientific | ER0051 | Plasmid construction |
| BHI (Brain Heart Infusion) Broth | Sigma-Aldrich | 53286 | Growth medium |
| Blocking reagent | Sigma Aldrich | 11096176001 | Northern blot assays |
| CDP-Star | Sigma Aldrich | 11759051001 | Northern blot assays (substrate) |
| Centrifuge 5415 R | Eppendorf | RNA extraction and purification | |
| Chloroform | Dominique Dutscher | 508320-CER | RNA extraction and purification |
| DIG-RNA labelling mix | Sigma-Aldrich | 11277073910 | Northern blot assays |
| DNase I | Roche | 4716728001 | DNase treatment |
| Erythromycin (ery) | Sigma-Aldrich | Fluka 45673 | Growth medium |
| FastPrep device | MP Biomedicals | 116004500 | Mechanical lysis |
| Guanidium Thiocyanate | Sigma-Aldrich | G9277-250G | Northern blot assays |
| Hybridization Hoven Hybrigene | Techne | FHB4DD | Northern blot assays |
| Hybridization tubes | Techne | FHB16 | Northern blot assays |
| Isoamyl alcohol | Fisher Scientific | A/6960/08 | RNA extraction and purification |
| LB (Lysogeny Broth) | Sigma-Aldrich | L3022 | Growth medium |
| Lysing Matrix B Bulk | MP Biomedicals | 6540-428 | Mechanical lysis |
| MicroPulser Electroporator | BioRad | 1652100 | Plasmid construction |
| Milli-Q water device | Millipore | Z00QSV0WW | Ultrapure water |
| NanoDrop spectrophotometer | ThermoFisher Scientific | RNA/DNA quantity and quality | |
| Nitrocellulose membrane | Dominique Dutsher | 10600002 | Northern blot assays |
| Phembact Neutre | PHEM Technologies | BAC03-5-11205 | Cleaning and decontamination |
| Phenol | Carl Roth | 38.2 | RNA extraction and purification |
| Phusion High-Fidelity DNA Polymerase | New England Biolabs | M0530 | Plasmid construction |
| pMBP-MS2 | Addgene | 65104 | MS2-MBP production |
| Poly-Prep chromatography column | BioRad | 7311550 | MS2-affinity purification |
| PstI | ThermoFisher Scientific | ER0615 | Plasmid construction |
| Qubit 3 Fluorometer | Invitrogen | 15387293 | RNA quantity |
| RNAPro Solution | MP Biomedicals | 6055050 | Mechanical lysis |
| ScriptSeq Complete Kit | Illumina | BB1224 | Preparation of cDNA librairies |
| Spectrophotometer Genesys 20 | ThermoFisher Scientific | 11972278 | Bacterial cultures |
| SpeedVac Savant vacuum device | ThermoFisher Scientific | DNA120 | RNA extraction and purification |
| Stratalinker UV Crosslinker 1800 | Stratagene | 400672 | Northern blot assays |
| T4 DNA ligase | ThermoFisher Scientific | EL0014 | Plasmid construction |
| TBE (Tris-Borate-EDTA) | Euromedex | ET020-C | Northern blot assays |
| ThermalCycler T100 | BioRad | 1861096 | Plasmid construction |
| Tween 20 | Sigma Aldrich | P9416-100ML | Northern blot assays |
| X-ray film processor | hu.q | HQ-350XT | Northern blot assays |
| X-ray films Super RX-N | FujiFilm | 4741019318 | Northern blot assays |
Referanslar
- Carrier, M. C., Lalaouna, D., Masse, E. Broadening the Definition of Bacterial Small RNAs: Characteristics and Mechanisms of Action. Annual Review of Microbiology. 72, 141-161 (2018).
- Hör, J., Matera, G., Vogel, J., Gottesman, S., Storz, G. Trans-Acting Small RNAs and Their Effects on Gene Expression in Escherichia coli and Salmonella enterica. EcoSal Plus. 9 (1), (2020).
- Desgranges, E., Marzi, S., Moreau, K., Romby, P., Caldelari, I. Noncoding RNA. Microbiology Spectrum. 7 (2), (2019).
- Adams, P. P., Storz, G. Prevalence of small base-pairing RNAs derived from diverse genomic loci. Biochimica et Biophysica Acta - Gene Regulatory Mechanisms. 1863 (7), 194524 (2020).
- Pain, A., et al. An assessment of bacterial small RNA target prediction programs. RNA Biology. 12 (5), 509-513 (2015).
- Desgranges, E., Caldelari, I., Marzi, S., Lalaouna, D. Navigation through the twists and turns of RNA sequencing technologies: Application to bacterial regulatory RNAs. Biochimica et Biophysica Acta - Gene Regulatory Mechanisms. , 194506 (2020).
- Wright, P. R., et al. CopraRNA and IntaRNA: predicting small RNA targets, networks and interaction domains. Nucleic Acids Research. 42, 119-123 (2014).
- Smirnov, A., Schneider, C., Hor, J., Vogel, J. Discovery of new RNA classes and global RNA-binding proteins. Current Opinion in Microbiology. 39, 152-160 (2017).
- Saliba, A. E., Santos, S., Vogel, J. New RNA-seq approaches for the study of bacterial pathogens. Current Opinion in Microbiology. 35, 78-87 (2017).
- Melamed, S., Adams, P. P., Zhang, A., Zhang, H., Storz, G. RNA-RNA Interactomes of ProQ and Hfq Reveal Overlapping and Competing Roles. Molecular Cell. 77 (2), 411-425 (2020).
- Melamed, S., et al. Global Mapping of Small RNA-Target Interactions in Bacteria. Molecular Cell. 63 (5), 884-897 (2016).
- Iosub, I. A., et al. Hfq CLASH uncovers sRNA-target interaction networks linked to nutrient availability adaptation. Elife. 9, (2020).
- Waters, S. A., et al. Small RNA interactome of pathogenic E. revealed through crosslinking of RNase E. The EMBO Journal. 36 (3), 374-387 (2017).
- Dos Santos, R. F., Arraiano, C. M., Andrade, J. M. New molecular interactions broaden the functions of the RNA chaperone Hfq. Current Genetics. , (2019).
- Kavita, K., de Mets, F., Gottesman, S. New aspects of RNA-based regulation by Hfq and its partner sRNAs. Current Opinion in Microbiology. 42, 53-61 (2018).
- Bohn, C., Rigoulay, C., Bouloc, P. No detectable effect of RNA-binding protein Hfq absence in Staphylococcus aureus. BMC Microbiology. 7, 10 (2007).
- Jousselin, A., Metzinger, L., Felden, B. On the facultative requirement of the bacterial RNA chaperone, Hfq. Trends in Microbiology. 17 (9), 399-405 (2009).
- Olejniczak, M., Storz, G. ProQ/FinO-domain proteins: another ubiquitous family of RNA matchmakers. Molecular Microbiology. 104 (6), 905-915 (2017).
- Lalaouna, D., Desgranges, E., Caldelari, I., Marzi, S. Chapter Sixteen - MS2-Affinity Purification Coupled With RNA Sequencing Approach in the Human Pathogen Staphylococcus aureus. Methods in Enzymology. 612, 393-411 (2018).
- Lalaouna, D., Prevost, K., Eyraud, A., Masse, E. Identification of unknown RNA partners using MAPS. Methods. 117, 28-34 (2017).
- Lalaouna, D., et al. A 3' external transcribed spacer in a tRNA transcript acts as a sponge for small RNAs to prevent transcriptional noise. Molecular Cell. 58 (3), 393-405 (2015).
- Lalaouna, D., Morissette, A., Carrier, M. C., Masse, E. DsrA regulatory RNA represses both hns and rbsD mRNAs through distinct mechanisms in Escherichia coli. Molecular Microbiology. 98 (2), 357-369 (2015).
- Lalaouna, D., Prevost, K., Laliberte, G., Houe, V., Masse, E. Contrasting silencing mechanisms of the same target mRNA by two regulatory RNAs in Escherichia coli. Nucleic Acids Research. 46 (5), 2600-2612 (2018).
- Lalaouna, D., Eyraud, A., Devinck, A., Prevost, K., Masse, E. GcvB small RNA uses two distinct seed regions to regulate an extensive targetome. Molecular Microbiology. 111 (2), 473-486 (2019).
- Silva, I. J., et al. SraL sRNA interaction regulates the terminator by preventing premature transcription termination of rho mRNA. Proceedings of the National Academy of Sciences. 116 (8), 3042-3051 (2019).
- Lalaouna, D., Masse, E. Identification of sRNA interacting with a transcript of interest using MS2-affinity purification coupled with RNA sequencing (MAPS) technology. Genomics Data. 5, 136-138 (2015).
- Tomasini, A., et al. The RNA targetome of Staphylococcus aureus non-coding RNA RsaA: impact on cell surface properties and defense mechanisms. Nucleic Acids Research. 45 (11), 6746-6760 (2017).
- Bronesky, D., et al. A multifaceted small RNA modulates gene expression upon glucose limitation in Staphylococcus aureus. The EMBO Journal. 38 (6), (2019).
- Lalaouna, D., et al. RsaC sRNA modulates the oxidative stress response of Staphylococcus aureus during manganese starvation. Nucleic Acids Research. 47 (1), 9871-9887 (2019).
- Carrier, M. C., Laliberte, G., Masse, E. Identification of New Bacterial Small RNA Targets Using MS2 Affinity Purification Coupled to RNA Sequencing. Methods in Molecular Biology. 1737, 77-88 (2018).
- Garibyan, L., Avashia, N. Polymerase chain reaction. Journal of Investigative Dermatology. 133 (3), 1-4 (2013).
- Revie, D., Smith, D. W., Yee, T. W. Kinetic analysis for optimization of DNA ligation reactions. Nucleic Acids Research. 16 (21), 10301-10321 (1988).
- Seidman, C. E., Struhl, K., Sheen, J., Jessen, T. Introduction of plasmid DNA into cells. Current Protocols in Molecular Biology. , (2001).
- Sanger, F., Coulson, A. R., Barrell, B. G., Smith, A. J. H., Roe, B. A. Cloning in single-stranded bacteriophage as an aid to rapid DNA sequencing. Journal of Molecular Biology. 143 (2), 161-178 (1980).
- Grosser, M. R., Richardson, A. R. Method for Preparation and Electroporation of S. aureus and S. epidermidis. Methods in Molecular Biology. 1373, 51-57 (2016).
- Krumlauf, R. Northern blot analysis. Methods in Molecular Biology. 58, 113-128 (1996).
- Koontz, L. Agarose gel electrophoresis. Methods in Enzymology. 529, 35-45 (2013).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2016 update. Nucleic Acids Reseaerch. 44, 3-10 (2016).
- Jagodnik, J., Brosse, A., Le Lam, T. N., Chiaruttini, C., Guillier, M. Mechanistic study of base-pairing small regulatory RNAs in bacteria. Methods. 117, 67-76 (2017).
- Mann, M., Wright, P. R., Backofen, R. IntaRNA 2.0: enhanced and customizable prediction of RNA-RNA interactions. Nucleic Acids Res. 45, 435-439 (2017).
- Georg, J., et al. The power of cooperation: Experimental and computational approaches in the functional characterization of bacterial sRNAs. Molecular Microbiology. 113 (3), 603-612 (2020).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır