Method Article
טיהור MS2-זיקה בשילוב עם רצף RNA בחיידקים גראם חיוביים
In This Article
Summary
טכנולוגיית MAPS פותחה כדי לבחון את היעד של RNA רגולטורי ספציפי ב vivo. sRNA של עניין מתויג עם APTAMER MS2 המאפשר טיהור משותף של שותפי RNA שלה וזיהוי שלהם על ידי רצף RNA. פרוטוקול שונה זה מתאים במיוחד לחיידקים גראם חיוביים.
Abstract
למרות RNAs רגולטוריים קטנים (sRNAs) נפוצים בקרב התחום החיידקי של החיים, הפונקציות של רבים מהם נשארים מאופיינים בצורה גרועה בעיקר בשל הקושי לזהות את מטרות mRNA שלהם. כאן, תיארנו פרוטוקול שונה של טיהור MS2-Affinity יחד עם טכנולוגיית רצף RNA (MAPS), במטרה לחשוף את כל שותפי ה- RNA של sRNA ספציפי ב- vivo. באופן כללי, APTAMER MS2 הוא התמזגו הקיצוניות 5 'של sRNA של עניין. מבנה זה בא לידי ביטוי לאחר מכן vivo, המאפשר MS2-sRNA לקיים אינטראקציה עם השותפים הסלולריים שלה. לאחר קצירת חיידקים, התאים הם lysed מכנית. התמצית הגולמית נטענת לתוך עמוד כרומטוגרפיה המבוסס על אמילוס, שהיה מצופה בעבר בחלבון MS2 המותך לחלבון הכריכה המלטוזי. פעולה זו מאפשרת לכידה ספציפית של MS2-sRNA ו- RNAs אינטראקציה. לאחר התרוממות רוח, RNAs מטוהרים מזוהים על ידי רצף RNA בעל תפוקה גבוהה וניתוח ביואינפורמטי עוקב. הפרוטוקול הבא יושם בפתוגן האנושי גראם חיובי סטפילוקוקוס aureus והוא, באופן עקרוני, transposable לכל חיידקים גראם חיובי. לסיכום, טכנולוגיית MAPS מהווה שיטה יעילה לחקור לעומק את הרשת הרגולטורית של sRNA מסוים, ומציעה תמונת מצב של כל היעד שלה. עם זאת, חשוב לזכור כי מטרות putative שזוהו על ידי MAPS עדיין צריך להיות מאומת על ידי גישות ניסיוניות משלימות.
Introduction
מאות, אולי אפילו אלפי RNAs רגולטוריים קטנים (sRNAs) זוהו ברוב הגנומים החיידקיים, אך הפונקציות של רובם המכריע נותרו לא אופייניות. בסך הכל, sRNAs הם מולקולות קצרות ללא קידוד, ממלא תפקידים מרכזיים בפיזיולוגיה חיידקית והסתגלות לסביבותמשתנות 1,2,3. אכן, מקרומולקולות אלה נמצאות במרכזן של רשתות רגולטוריות מורכבות רבות, המשפיעות על מסלולים מטבוליים, תגובות מתח אך גם ארסיות ועמידות לאנטיביוטיקה. מבחינה הגיונית, הסינתזה שלהם מופעלת על ידי גירויים סביבתיים ספציפיים (למשל, רעב לחומרים מזינים, לחצים חמצוניים או קרום). רוב sRNAs לווסת mRNAs יעד מרובים ברמה שלאחר שעתוק באמצעות זיווג בסיס קצר ולא רציף. הם בדרך כלל למנוע ייזום תרגום על ידי מתחרה עם ריבוזומים עבור אזורי ייזום תרגום4. היווצרות של sRNA:mRNA דופלקסים גם לעתים קרובות תוצאות השפלה פעילה של mRNA היעד על ידי גיוס של RNases ספציפיים.
האפיון של יעד sRNA (כלומר, כל הסט של RNAs היעד שלה) מאפשר זיהוי של המסלולים המטבוליים שבהם הוא מתערב ואת האות הפוטנציאלי שהוא עונה. כתוצאה מכך, הפונקציות של sRNA מסוים בדרך כלל ניתן להסיק מן היעד שלה. לשםכךפותחו מספר כלי חיזוי סיליקו כגון IntaRNA ו- CopraRNA 5,6,7. הם מסתמכים בעיקר על השלמות רצף, זיווג אנרגיה ונגישות של אתר האינטראקציה הפוטנציאלי כדי לקבוע שותפי sRNA putative. עם זאת, אלגוריתמי חיזוי אינם משלבים את כל הגורמים המשפיעים על זיווג בסיס ב vivo כגון מעורבות של מלווים RNA8 לטובת אינטראקציות תת אופטימליות או ביטוי משותף של שני בני הזוג. בשל המגבלות הטבועות בהם, שיעור החיזוי החיובי הכוזב נותר גבוה. רוב הגישות הניסיוניות בקנה מידה גדול מבוססות על טיהור משותף של זוגות sRNA:mRNA אינטראקציה עם חלבון מחייב RNA מתויג (RBP)6,9. לדוגמה, שיטת האינטראקציה RNA על ידי קשירה ורצף (RIL-seq) זיהתה דופלקסים RNA מטוהרים במשותף עם מלווים RNA כגון Hfq ו ProQ ב Escherichia coli10,11. טכנולוגיה דומה הנקראת UV-Crosslinking, קשירה ורצף של כלאיים (CLASH) הוחלה על RNase E- ו- Hfq הקשורים sRNAs ב E. coli12,13. למרות התפקידים המתוארים היטב של Hfq ו- ProQ ברגולציה בתיווך sRNA בחיידקים מרובים8,14,15, רגולציה מבוססת sRNA נראה RNA מלווה עצמאי בכמה אורגניזמים כמו S. aureus16,17,18. גם אם הטיהור של דופלקסים RNA בשיתוף עם RNases הוא ריאלי כפי שהוכח על ידי ווטרס ועמיתים לעבודה13, זה נשאר מסובך כמו RNases לעורר השפלה מהירה שלהם. לפיכך, טיהור MS2-Affinity בשילוב עם רצף RNA (MAPS)גישה 19,20 מהווה חלופה מוצקה אורגניזמים כאלה.
שלא כמו שיטות שהוזכרו לעיל, MAPS משתמש sRNA ספציפי כפיתיון כדי ללכוד את כל RNAs אינטראקציה ולכן אינו מסתמך על מעורבות של RBP. התהליך כולו מתואר באיור 1. בקצרה, sRNA מתויג ב 5 'עם MS2 RNA aptamer כי הוא מוכר במיוחד על ידי חלבון מעיל MS2. חלבון זה הוא התמזגו עם חלבון מחייב מלטוז (MBP) להיות משותק על שף amylose. לכן, MS2-sRNA ושותפיו RNA נשמרים בעמודה כרומטוגרפיה זיקה. לאחר התרוממות רוח עם RNAs מלטוזיים ומטוהרים מזוהים באמצעות רצף RNA בעל תפוקה גבוהה ואחריו ניתוח ביואינפורמטי (איור 2). טכנולוגיית MAPS מציירת בסופו של דבר מפה של כל האינטראקציות הפוטנציאליות המתרחשות ב- vivo.
טכנולוגיית MAPS יושמה במקור בחיידק הגרם-שלילי הלא פתוגניים E. coli21. למרבה הפלא, MAPS סייעה לזהות שבר שמקורו ב-tRNA במיוחד באינטראקציה עם RyhB ו-RybB sRNAs ולמנוע רעש שעתוק של sRNA כדי לווסת מטרות mRNA בתנאים שאינם מעוררים. לאחר מכן, MAPS הוחל בהצלחה על SRNAs אחרים E. coli כמו DsrA22, RprA23, CyaR23 ו GcvB24 (טבלה 1). בנוסף לאישור מטרות ידועות בעבר, MAPS הרחיבה את היעד של sRNAs ידועים אלה. לאחרונה, MAPS בוצע סלמונלה Typhimurium וחשף כי SRNA SraL נקשר rho mRNA, קידוד עבור גורם סיום שעתוק25. באמצעות זיווג זה, SraL מגן rho mRNA מפני סיום שעתוק מוקדם מופעלות על ידי רו עצמה. מעניין, טכנולוגיה זו אינה מוגבלת sRNAs והוא יכול להיות מיושם על כל סוג של RNAs הסלולר כפי שהודגם על ידי שימוש שבר נגזר tRNA26 ואזור 5'-untranslated של mRNA22 (טבלה 1).
שיטת MAPS הותאמה גם לחיידק גרם חיובי פתוגניים S. aureus19. באופן ספציפי, פרוטוקול התמוגה שונה באופן נרחב כדי לשבור תאים ביעילות עקב דופן תאים עבה יותר מאשר חיידקים גראם שליליים ולשמור על שלמות RNA. פרוטוקול מותאם זה כבר חשף את האינטראקציה של RsaA27, RsaI28 ו RsaC29. גישה זו נתנה תובנות על התפקיד המכריע של sRNAs אלה במנגנונים רגולטוריים של תכונות פני התא, ספיגת גלוקוז, ותגובות מתח חמצוני.
הפרוטוקול שפותח ומיושם ב- E. coli בשנת 2015 תואר לאחרונה בפירוט רב30. כאן, אנו מספקים את פרוטוקול MAPS שונה, אשר מתאים במיוחד לחקר רשתות רגולטוריות sRNA בחיידקים גראם חיובי (קיר תא עבה) אם לא פתוגניים או פתוגניים (אמצעי זהירות).
Protocol
1. מאגרים ומדיה
- עבור ניסויי MAPS, הכן את המאגרים והמדיה הבאים:
- מאגר A (150 mM KCl, 20 מ"מ Tris-HCl pH 8, 1 mM MgCl2 ו 1 mM DTT)
- חוצץ E (250 mM KCl, 20 mM Tris-HCl pH 8, 12 מ"מ מלטוז, 0.1% טריטון, 1 mM MgCl2 ו 1 mM DTT)
- מאגר טעינה RNA (0.025% קסילן ציאנול ו 0.025% כחול ברומופנול ב 8 M אוריאה)
- עירוי לב המוח (BHI) בינוני (12.5 גרם של מוח עגל, 10 גרם של פפטון, 5 גרם של לב בקר, 5 גרם של NaCl, 2.5 גרם שלNa 2HPO4 ו 2 גרם של גלוקוז עבור 1 L)
- מרק ליסוגניים (LB) בינוני (10 גרם של פפטון, 5 גרם של תמצית שמרים ו 10 גרם של NaCl עבור 1 L) - עבור מבחני כתמים צפוניים, הכן את המאגרים הבאים:
- פתרון חסימה (1x חומצה מלמית ו 1% חסימת מגיב)
- פתרון הכלאה (50% formamide, 5x SSC, 7% SDS, 1% פתרון חסימה ו 0.2% N-לאוריל סרקוסין, 50 מ"מ נתרן פוספט). מחממים בתסיסה כדי להתמוסס.
התראה: פעל בקפידה בהתאם לאמצעי הבטיחות הקשורים לכל מוצר.
- 1 M נתרן פוספט (58 מ"מ נתרן פוספט דיבאסי ו 42 מ"מ נתרן פוספט מונובאסי)
- מלח, מאגר נתרן ציטראט (SSC), רכז 20x (3 M NaCl ו 300 מ"מ טריסודיום ציטראט)
2. בעיות בטיחות
- בצע את כל השלבים הכרוכים בחיידקים פתוגניים בני קיימא במעבדת בלימה ברמה 2.
הערה: רק תמציות תאים ניתן לקחת בחוץ לאחר תמוגה (שלב 5). - תלבש חלוק מעבדה וכפפות.
- ודא כי פרקי הידיים מכוסים.
- נקו את ארון הבטיחות הביולוגי (מחלקה II) בתמיסת חיטוי.
- יש להשליך פסולת מוצקה החשופה לחיידקים בפח הביו-רפואי המתאים.
- לטפל בקבוקונים המכילים נוזלים מזוהמים עם תמיסת חיטוי. לאחר מכן, להשליך אותו בכיור.
- יש לשטוף בזהירות את הידיים ופרקי כף היד בסבון ולהסיר את חלוק המעבדה לפני היציאה ממעבדת הבלימה ברמה 2.
3. בניית פלסמיד
הערה: למטרות שיבוט, חיוני לזהות תחילה את גבולות ה- sRNA האנדוגני. pCN51-P3 ו pCN51-P3-MS2 פלסמידים מתוארים תומסיני ואח '(2017)27. מקדם P3 מאפשר ביטוי גבוה של sRNA באופן תלוי צפיפות התא (כלומר, כאשר חיידקים נכנסים לשלב הנייח של הצמיחה). SRNAs סטפילוקוקליים רבים מצטברים במהלך שלב צמיחה זה.
- להגביר את רצף sRNA על ידי PCR באמצעות פולימראז DNA נאמנות גבוהה ומכונת PCR. פעל בקפידה בהתאם להוראות היצרן וקרא את Garibyan ו- Avashia (2013)31 לקבלת פרטים נוספים.
- השתמש בתבניות הבאות כדי לעצב את פריימנים ספציפיים:

ו-GGATCC GGATCC(N)-3' של 5'-CGC עבור פרייומרים קדימה והפוך, בהתאמה.
הערה: אוליגונוקלאוטידים אלה מאפשרים להתיך את רצף MS2 (מודגש) לסוף 5 ' של sRNA של עניין. מספר Pst אתרי ההגבלה I ו- BamHI (מסומנים בקו תחתון) מתווספים בגפיים 5 ' ו -3 'של מבנה MS2-sRNA כדי לשכפל את האמפליקון לתוך pCN51-P3פלסמיד 27. (N) מתאים לרצף הספציפי לגנים (15-20 נוקלאוטידים). - לעכל 1 מיקרוגרם של pCN51-P3 פלסמיד ו 1 מיקרוגרם של מוצר PCR MS2-sRNA עם 2 U של pstI ו 1 U של באםHI במאגר המתאים על פי המלצות היצרן.
- דגירה 1 שעה ב 37 מעלות צלזיוס לטהר DNA באמצעות ערכת טיהור PCR (ראה טבלה של חומרים).
- מערבבים את פלסמיד pCN51-P3 המתעכל (300 ng) ואמפליקון MS2-sRNA (יחס טוחן לווקטור:הכנס = 1:3) בצינור של 1.5 מ"ל. ראה Revie et al. (1988)32 כדי למקסם את יעילות הקשירה. הוסף 1 μL של מאגר ליגאז ו 10 U של T4 ליגאז בכל צינור. כוונן את עוצמת הקול ל- 10 μL עם מים דקים במיוחד.
- דגירה ב 22 מעלות צלזיוס לפחות 2 שעות.
- הוסף 5 μL של תערובת קשירה ל 50 μL של DH5α קפוא כימית מוסמך E. coli תאים. קרא את Seidman et al. (2001)33 כדי ללמוד עוד על טרנספורמציה פלסמיד ותאים בעלי יכולת כימית.
- דגירה 30 דקות על קרח.
- הלם חום (45 s ב 42 מעלות צלזיוס) צינור השינוי באמצעות בלוק חום או אמבט מים.
- הוסף 900 μL של LB בינוני דגירה ב 37 °C (69 °F) במשך 30 דקות.
- צלחת 100 μL של ההשעיה החיידקית על צלחת אגר LB בתוספת אמפילין (100 מיקרוגרם / μL).
הערה: הווקטור pCN51-P3 מקודד גן עמידות לאמפצילין, המאפשר לבחור רק שיבוטי E. coli הנושאים את פלסמיד pCN51-P3-MS2-sRNA. - לחלץ את pCN51-P3-MS2-sRNA פלסמיד מתרבות חיידקית לילה (5 מ"ל) גדל בנוכחות אמפיקילין (100 מיקרוגרם / μL) באמצעות ערכת miniprep DNA פלסמיד (ראה טבלה של חומרים).
- אמת את המבנה על-ידי רצף סנגר34 באמצעות פריימר הבא, 5'-TCTCGAAAATAATAGAGGG-3'.
- להפוך את pCN51-P3-MS2-sRNA פלסמיד לתוך DC10B כימית מוסמך E. coli תאים. חזור על שלבים 3.7 עד 3.11.
- לחלץ את pCN51-P3-MS2-sRNA פלסמיד (ראה שלב 3.12) ולהפוך 1-5 מיקרוגרם של DNA פלסמיד לתוך HG001 ΔsRNA אלקטרו-כשיר S. aureus תאים באמצעות מנגנון electroporation. פעל בזהירות בהתאם להוראות היצרן. קרא גרוסר ריצ'רדסון (2016)35 כדי ללמוד עוד על שיטות להכנת אלקטרו-כשיר S. aureus.
התראה: שלב זה כרוך בטיפול בחיידקים פתוגניים (ראה שלב 2). - הוסף 900 μL של בינוני BHI ודגר ב 37 °C (69 °F) עבור 3 שעות.
- צנטריפוגה 1 דקות ב 16,000 x g. השלך את הסופרנט.
- Resuspend גלולה ב 100 μL של BHI צלחת ההשעיה חיידקי על לוחות אגר BHI בתוספת אריתרומיצין (10 מיקרוגרם / μL).
הערה: וקטור pCN51-P3 גם מקודד גן התנגדות אריתרומיצין, המאפשר לבחור רק S. שיבוטים אוראוס נושאת את pCN51-P3-MS2-sRNA פלסמיד.
4. קצירת חיידקים
התראה: שלב זה כרוך בטיפול בחיידקים פתוגניים (ראה שלב 2).
- לגדל מושבה אחת של זנים הנושאים או pCN51-P3-MS2-sRNA או pCN51-P3-MS227 פלסמידים ב 3 מ"ל של בינוני BHI בתוספת אריתרומיצין (10 מיקרוגרם / μL) בכפילויות.
- לדלל כל תרבות לילה ב 50 מ"ל (≈1/100) של בינוני BHI טרי בתוספת אריתרומיצין (10 מיקרוגרם / μL) כדי להגיע OD600nm של 0.05. השתמש 250 מ"ל צלוחי מעוקרים (5:1 צלף לבינוני יחס).
הערה: יש להגדיר תנאים בינוניים ותנאי צמיחה בהתאם לתבנית הביטוי של ה- sRNA הנחקר. - לגדל תרבויות ב 37 מעלות צלזיוס עם רועד ב 180 סל"ד עבור 6 שעות.
- מעבירים כל תרבות לצינור צנטריפוגות של 50 מ"ל.
- צנטריפוגה ב 2,900 x גרם במהלך 15 דקות ב 4 מעלות צלזיוס. השלך את הסופרנט.
- שמור כדורי על קרח ישירות לבצע תמוגה תא מכני (שלב 5) או להקפיא ולאחסן כדורי ב -80 מעלות צלזיוס.
5. תמוגה תאית מכנית
התראה: השלבים הבאים חייבים להתבצע על קרח ומאגרים חייבים להיות ב 4 מעלות צלזיוס. השתמש בכפפות ולנקוט בכל אמצעי הזהירות כדי להגן על דגימות מפני RNases.
- גלולות resuspend (שלב 4.6) ב 5 מ"ל של חוצץ A.
- מעבירים את התאים שעברו שימוש חוזר בצינורות צנטריפוגה של 15 מ"ל עם 3.5 גרם חרוזי סיליקה (0.1 מ"מ).
- הכנס צינורות למכשיר תמוגה תא מכני (ראה טבלה של חומרים). הפעל מחזור של 40 s ב 4.0 m/s.
הערה: אם מחזור אחד אינו מספיק כדי לשבור תאים, תן למכשיר להתקרר במשך 5 דקות תוך שמירה על דגימות על הקרח. לאחר מכן, חזור על מחזור נוסף של 40 שניות ב 4.0 m/s. היעילות של תמוגה התא ניתן לבדוק על ידי ציפוי supernatant על צלחת BHI-אגר. - צנטריפוגה ב 15,700 x גרם במשך 15 דקות. לשחזר את supernatant ולשמור אותו על קרח.
6. הכנת טורים
התראה: היזהר לא לאפשר את שף אמילוס להתייבש. במידת הצורך, אטמו את העמודה עם כובע קצה. הכינו את כל הפתרונות לפני תחילת טיהור הזיקה.
- שים עמודת כרומטוגרפיה בארון תקשורת בעמודה (ראה טבלת חומרים).
- הסר את קצה העמודה ושטוף את העמודה במים דקים במיוחד.
- הוסף 300 μL של שרף אמילוס.
- לשטוף את העמודה עם 10 מ"ל של חוצץ A.
- לדלל 1,200 pmol של חלבון MBP-MS2 ב 6 מ"ל של חוצץ A ולטעון אותו לתוך העמודה.
- לשטוף את העמודה עם 10 מ"ל של חוצץ A.
7. טיהור הזיקה ל-MS2 (איור 1)
- טען את lysate התא לתוך העמודה.
הערה: שמור 1 מ"ל של lysate התא (תמצית גולמית, CE) כדי לחלץ RNA הכולל (שלב 8) ולבצע כתם צפון (שלב 9) ו transcriptomic (שלב 10) ניתוח. - אסוף את שבר הזרימה (FT) בצינור איסוף נקי.
- לשטוף את הטור 3 פעמים עם 10 מ"ל של חוצץ A. לאסוף את שבר לשטוף (W).
- אלוט את העמודה עם 1 מ"ל של Buffer E ולאסוף את שבר elution (E) ב microtube 2 מ"ל.
- שמור את כל השברים שנאספו על הקרח עד מיצוי RNA (שלב 8) או להקפיא אותם ב -20 מעלות צלזיוס לשימוש מאוחר יותר.
8. מיצוי RNA של שברים שנאספו (CE, FT, W ו- E)
- השתמש 1 מ"ל של כל שבר (כולל FT ו- W) עבור מיצוי RNA.
- הוסף 1 נפח של פנול. מערבבים במרץ.
התראה: פנול הוא הפכפך וקורוזיבי, לשים לב ולעבוד בבטחה מתחת למכסה המנוע אדים. - צנטריפוגה ב 16,000 x גרם במשך 10 דקות ב 20 מעלות צלזיוס.
- העבר את השלב העליון במיקרו-צינור נקי של 2 מ"ל.
- הוסף נפח אחד של אלכוהול כלורופורם/איזואמיל (24:1) וחזור על שלבים 8.3 עד 8.4.
התראה: עבוד בבטחה מתחת למכסה המנוע. - הוסף 2.5 כרכים של אתנול קר 100% ו 1/10 נפח של 3 M נתרן אצטט (NaOAc) pH 5,2.
- לזרז לילה ב -20 מעלות צלזיוס.
הערה: משקעים יכולים להתבצע גם באמבט אתנול / קרח יבש במהלך 20 דקות או ב -80 מעלות צלזיוס במהלך 2 שעות. - צנטריפוגה ב 16,000 x גרם במשך 15 דקות ב 4 מעלות צלזיוס. לאט להסיר אתנול עם פיפטה תוך נזהר לא להפריע גלולה.
התראה: גלולת RNA לא תמיד גלוי והוא לפעמים רופף בנוכחות אתנול. - הוסף 500 μL של 80% אתנול קר.
- צנטריפוגה ב 16,000 x גרם במשך 5 דקות ב 4 מעלות צלזיוס.
- השלך אתנול על ידי pipetting אותו לאט. יבש את גלולה באמצעות רכז ואקום, 5 דקות על מצב ריצה.
- Resuspend את גלולה בנפח המתאים (15-50 μL) של מים אולטרה סגולים. להקפיא את גלולה ב -20 מעלות צלזיוס לשימוש מאוחר יותר.
- להעריך את כמות ה-RNA (260 ננומטר) ואת האיכות (260/280 ו-260/230 יחסי אורך גל) באמצעות ספקטרופוטומטר/פלואורומטר (ראו טבלת חומרים). פעל בזהירות בהתאם להוראות היצרן.
הערה: 3-4 מיקרוגרם מתקבלים בדרך כלל בשבר elution (E). זה תלוי בעיקר בתנאים שנבדקו.
9. ניתוח של MS2-זיקה טיהור על ידי כתם צפון36
- לדלל 5 מיקרוגרם של RNA של CE, FT, שברי W ו 500 ng של שבר E ב 10 μL של מים אולטרה סגולים ומערבבים עם 10 μL של מאגר טעינת RNA.
- דגירה 3 דקות ב 90 מעלות צלזיוס.
- לטעון דגימות לתוך בארות של 1% ג'ל agarose בתוספת 20 מ"מ של guanidium thiocyanate ולהפעיל את הג'ל ב 100-150 V ב TBE 1x חיץ ב 4 מעלות צלזיוס. קרא את קונץ (2013)37 לפרטים נוספים.
- העבר RNAs על קרום nitrocellulose על ידי העברת ואקום במשך 1h או העברת נימי לילה.
הערה: שיטת נימי יעילה יותר עבור RNAs גדולים. - RNAs מקושרים UV על הממברנה (120 mJ ב 254 ננומטר) באמצעות crosslinker אולטרה סגול.
- הכנס את הממברנה לבקבוק הכלאה כשצד ה- RNA פונה כלפי מעלה.
- הוסף 10-20 מ"ל של פתרון הכלאה שחומם מראש. דגירה 30 דקות ב 68 מעלות צלזיוס.
- השלך את הפתרון והוסף 10-20 מ"ל של פתרון הכלאה טרי בתוספת 1 μL של בדיקה ספציפית sRNA. דגירה לילה ב 68 °C (68 °F).
הערה: גשוש ה-RNA בעל התווית DIG מסונתז באמצעות ערכת תיוג DIG RNA והוראות היצרן הבאות. לחלופין, ניתן להשתמש בגשוש רדיו-פעמון. - לשטוף את הממברנה עם 10-20 מ"ל של פתרון לשטוף 1 (2x SSC ו 0.1% SDS) במשך 5 דקות ב 20 °C (69 °F). חזור פעם אחת.
- לשטוף את הממברנה עם 10-20 מ"ל של פתרון לשטוף 2 (0.2x SSC ו 0.1% SDS) במשך 15 דקות ב 68 °C (68 °F). חזור פעם אחת.
- דגירה עם 10-20 מ"ל של פתרון חסימה לפחות 30 דקות ב 20 מעלות צלזיוס.
- השלך את הפתרון והוסף 10-20 מ"ל של פתרון החסימה בתוספת נוגדן אנטי דיגוקסיגנין פוליקלונלי (1/1000), מצומד phosphatase אלקליין. דגירה 30 דקות ב 20 מעלות צלזיוס.
- לשטוף את הממברנה עם 10-20 מ"ל של פתרון לשטוף 3 (1x חומצה גברית ו 0.3% Tween 20) במשך 15 דקות ב 20 מעלות צלזיוס. חזור פעם אחת.
- דגירה הממברנה עם 10-20 מ"ל של פתרון הזיהוי (0.1 M טריס HCl ו 0.1 M NaCl pH 9.5) 5 דקות ב 20 °C (69 °F).
- שים את הממברנה על סרט פלסטיק להשרות אותו עם המצע (ראה טבלה של חומרים). דגירה 5 דקות בחושך.
- לאטום את הממברנה בסרט פלסטיק. שים את הממברנה בקלטת אוטורדיוגרפיה.
- לחשוף את הממברנה לסרט אוטורדיוגרפיה בחדר החושך הייעודי.
הערה: זמן התערוכה תלוי בחוזק האות, מכמה שניות לדקות. - לחשוף את הסרט החשוף במכשיר פיתוח אוטומטי.
10. הכנת הדגימות לריצוף RNA
הערה: שלב זה נוגע רק ל-RNAs שחולצו משברי E ו-CE.
- הוסף לכל דגימה 10 μL של מאגר DNase 10x ו- DNase I (1 U / מיקרוגרם של RNAs שטופלו). מוסיפים מים לנפח סופי של 100 μL.
- דגירה 1 שעה ב 37 °C (69 °F).
- לחלץ ולטהר RNAs כפי שתואר קודם לכן (שלבים 8.2 עד 8.11).
- Resuspend גלולת RNA ב 20 μL של מים אולטרה סגולים.
הערה: ניתן לבדוק את נוכחותם של הדנ"א הנותר באמצעות PCR ופרימרים ספציפיים (למשל, גן 16S). - להעריך את כמות ואיכות ה-RNA באמצעות מערכת ניתוח אלקטרופורזה מבוססת מיקרופלואידיקה (ראה טבלת חומרים).
הערה: 1 מיקרוגרם מתקבל בדרך כלל בשבר elution (E) לאחר טיפול DNase. - הסר RNAs ריבוזומלי עם ערכת דלדול rRNA חיידקי.
הערה: RNAs גדולים ושופעים (כלומר, rRNAs) נוטים לקיים אינטראקציה לא ספציפית עם עמודת הזיקה. 500 ng של RNA שחולצו נדרשים לבצע שלב זה. - שוב להעריך את כמות ואיכות RNA באמצעות מערכת ניתוח אלקטרופורזה מבוססת microfluidics.
- הכינו ספריות cDNA עם 10-20 ng של RNA ריבודפולד באמצעות ערכת הכנה לספריית cDNA וציות להוראות היצרן.
- רצף הספריות שהושגו באמצעות כלי רצף (למשל, חד-פעמי, 150 bp; ראה טבלת חומרים).
הערה: 5-10 מיליון קריאות לכל מדגם הן בדרך כלל מספיקות.
11. ניתוח נתונים של RNAseq (איור 2)
- הורד את קבצי הרצף FastQ מפלטפורמת הרצף.
- גישה למופע הגלקסיה של התחנה הביולוגית Roscoff (https://galaxy.sb-roscoff.fr/) ולהתחבר.
הערה: ניתן למצוא בקלות כל אלגוריתם שהוזכר באמצעות סרגל החיפוש. מדריך למשתמש מסופק עבור כל כלי.
התראה: הגירסה של הכלים הדרושים עשויה להיות שונה משרת Galaxy38הציבורי . - לחץ על סמל קבל נתונים ולאחר מכן העלה קובץ מהמחשב . העלה קובץ רצף FastQ של כל דוגמאות בקרת MS2 ו- MS2 sRNA. העלה גם קובץ גנום הפניה FASTA וקובץ ביאור GFF.
- הפעל דוחות איכות קריאה FastQC (גרסת גלקסי 0.69).
הערה: כלי זה מספק הערכת איכות של רצפים גולמיים (למשל, ציון איכות, נוכחות של רצפי מתאמים). - הפעל את כלי חיתוך הקריאה הגמיש של Trimmomatic (גרסת Galaxy 0.36.6) כדי להסיר רצפי מתאמים וקריאות באיכות ירודה. ציין רצפי מתאמים המשמשים להכנת ספריה (לדוגמה, TruSeq 3, חד-פעמי). הוסף את הפעולות הבאות של Trimmomatic: הזזהWINDOW (מספר בסיסים =4; איכות ממוצעת =20) ו MINLEN (אורך מינימלי של קריאות = 20).
- הפעל שוב דוחות איכות קריאה FastQC (גרסת גלקסי 0.69).
- הפעל Bowtie2 - המפה קוראת נגד הגנום הפניה (גרסת גלקסי 2.3.2.2). השתמש בקובץ FASTA של חומר עזר לגנום מההיסטוריה כדי למפות קריאות עם הגדרות ברירת מחדל (מקומיות רגישות מאוד).
הערה: ניתן לדמיין קובץ BAM שנוצר על-ידי הכלי Bowtie2 באמצעות מציג הגנומיקה האינטגרטיבית (IGV). יהיה צורך גם בקובץ BAI משויך. - לחלופין, הפעל את Flagstat האוסף נתונים סטטיסטיים עבור ערכת הנתונים של BAM (גרסה גלקסי 2.0).
- הפעל ספירת htseq - ספירה (גרסת גלקסי 0.6.1) אשר מיישר קורא תכונות חופפות בקובץ ביאור GFF. השתמש במצב חיתוך (לא ריק).
- אחסן בארכיון את כל קבצי הספירה הגולמית מניתוח ספירת htseq לקובץ Zip יחיד.
- הפעל SARTools DESeq2 כדי להשוות נתונים (גרסת גלקסי 1.6.3.0). ספק את קובץ Zip המכיל קבצי ספירה גולמית ואת קובץ העיצוב, קובץ מופרד באמצעות טאב המתאר את הניסוי. פעל בקפידה בהתאם להוראות שסופקו כדי ליצור את קובץ העיצוב.
תוצאות
התוצאות הייצוגיות מקורן במחקר של יעד RsaC ב S. aureus29. RsaC הוא SRNA לא קונבנציונלי באורך 1,116 nt. הקצה של 5' מכיל מספר אזורים חוזרים ונשנים בעוד הקצה 3 ' שלה (544 nt) הוא עצמאי מבחינה מבנית ומכיל את כל אתרי האינטראקציה החזויים עם מטרות mRNA שלה. הביטוי של sRNA זה הוא המושרה כאשר מנגן (Mn) הוא נדיר, אשר נתקל לעתים קרובות בהקשר של התגובה החיסונית המארחת. באמצעות טכנולוגיית MAPS, זיהינו מספר mRNAs אינטראקציה ישירה עם RsaC, חושף את תפקידה המכריע בלחץ חמצוני (sodA, ldh1 ו sarA) ו הקשורות מתכת (znuBC-zur ו sufCDSUB) תגובות.
אימות מבנה MS2-sRNA ותנאי ניסוי
לפני ביצוע ניסויים MAPS, חשוב לקבוע את התנאים האופטימליים של ביטוי של sRNA למד. אם נעשה שימוש באמרגן שאינו יליד, הוא בהחלט יעזור לייצר את מבנה ה- MS2-sRNA כאשר מטרותיו קיימות. בנוסף, מבנה MS2-sRNA צריך להיות מאומת בקפידה לגבי גודל, יציבות, ביטוי ותפקוד. ה- MS2 aptamer הותך לקצה 5' של RsaC באורך מלא (MS2-RsaC1116)או הצורה הקצרה יותר (MS2-RsaC544) המתאימה לחלק 3 ' של RsaC. שני המבנים באו לידי ביטוי vivo תחת שליטתו של מניין חישת מקדם P3 תלוי S. aureus HG001 ΔrsaC. מחיקת הגן rsaC מונעת תחרות בין RsaC אנדוגני MS2-RsaC. הזן הפראי המכיל את אותו וקטור עם תג MS2 בלבד שימש כפקד. פקד זה מאפשר הפחתה של אינטראקציות לא ספציפיות המתרחשות עם התג MS2.
כדי לאשר את המבנים לדמיין את דפוס הביטוי שלהם, תאים חיידקיים נקצרו לאחר 2 שעות, 4 שעות ו 6 שעות של צמיחה במדיום BHI ב 37 °C (69 °F). לאחר חילוץ RNA, ניתוח כתם צפון בוצע באמצעות בדיקה DIG ספציפית RsaC (איור 3A). רמת RsaC אנדוגני (נתיבים 1-3) גדל באופן משמעותי לאחר 6 שעות של צמיחה, המצדיק את הבחירה של נקודת זמן זו עבור ניסויים MAPS. חשוב לציין, רמות MS2-RsaC544 (נתיבים 7-9) ו MS2-RsaC1,116 (נתיבים 10-12) היו דומים RsaC אנדוגני ב 6 שעות. לפיכך, הם צריכים לחקות את דפוס הביטוי אנדוגני של RsaC. צורה גדולה יותר אך מינורית של RsaC הייתה ניתנת להבחנה ועשויה לנפרד מסיום לא יעיל של שעתוק. תופעה זו נצפית לעתים קרובות כאשר MS2-sRNA מתבטא תחת שליטה של מקדם חזק מ plasmid21. לא נצפו צורות קצרות יותר הנובעות מסיום שעתוק חריג או השפלה.
התוספת של APTAMER MS2 ב 5 ' של sRNAs יכול גם לשבש את הקיפול הנכון שלהם להשפיע על הפונקציות שלהם. שלב זה הוא קריטי עבור sRNAs מובנים מאוד כמו RsaC. לפיכך פעילות MS2-sRNA צריך להיבדק בהשוואה sRNA אנדוגני כאשר הדבר אפשרי. יעד ידוע בעבר או פנוטיפ ניתן לצפייה יכול לעזור לפקח עליו. לדוגמה, ההשפעה של RsaC על הצטברות ROS תאיים שימשה לאימות MS2-RsaC544 ו MS2-RsaC1,116 מבנים29.
ניתוח שברים שנאספו במהלך טיהור זיקה
RNAs חולצו שברי CE, FT ו- E בזן WT המבטא תג MS2 לבד זןrsaC Δ המבטא מבנה MS2-RsaC544. הראינו באמצעות ניתוח כתם צפון כי RsaC אנדוגני 1,116 nt ארוך היה מועשר בשבר elution אבל התברר אינטראקציה לא ספציפית עם עמודת הזיקה(איור 3B,נתיבים 2-3). ראינו את אותה תופעה עם MS2-RsaC1,116 (נתונים לא מוצגים). זאת בוודאי בשל אורכו ומבנהו המשני המורכב. לכן, רק צורה פחות מובנית וקצרה יותר (544 nt) של RsaC המתאימה לחלק 3' שלה שימש לביצוע ניסויים MAPS. באיור 3B, MS2-RsaC544 היה מועשר מאוד בשבר האלויון והוכיח שהוא נשמר בהצלחה על ידי חלבון ההיתוך MS2-MBP (נתיב 6). צורה גדולה יותר אך מינורית של MS2-RsaC544 נצפתה כמו באיור 3A. כאן, ניתן להתאים את ההקפדה ואת מספר השטיפות לכריכה לא ספציפית מופחתת או להיפך, כדי להגביל אובדן של שותפים אינטראקציה אמיתית.
אימות יעדי mRNA putative לאחר ניתוח MAPS
בעקבות ניתוח ביואינפורמטי, יעדי mRNA putative מפורטים על פי שינוי הקיפול בין בקרת MS2-sRNA ו- MS2, המתקבל באמצעות DeSeq2 (איור 2). לדוגמה, MS2-RsaC544 MAPS נתונים29 הציע כי sodA mRNA, קידוד עבור סופראוקסיד דיסמוטאז ב S. aureus, הוא היעד העיקרי (הלהיט הטוב ביותר, גבוה יותר לקפל שינוי). ניתוח כתם צפוני, שבוצע עם גשוש DIG ספציפי לסודהלאחר טיהור MS2-affinity, מראה כי SODA היה מועשר ביעילות עם MS2-RsaC544 בהשוואה לבקרת MS2 (איור 3C).
ניתוח תמלול גלובלי מבוצע באופן שיטתי על שבר CE. ההשוואה בין נתוני MAPS לניתוח תמלולומי זה מסייעת להתאים את יחס ההעשרה וחושפת היררכיית יעד פוטנציאלית. אכן, mRNA מבוטא היטב, אשר מועשר מאוד לאחר טיהור MS2-זיקה יש בהחלט זיקה מחייבת גדולה יותר מאשר mRNA מועשר מאוד הביע מאוד.
חשוב לציין כי כל המועמדים שזוהו על ידי MAPS חייבים להיות מאומתים בנפרד באמצעות ניסויים במבחנה ו /או ב vivo כגון אלקטרופורזה ניידות Shift Assays (EMSA) או כתב גנים assays (ראה Jagodnik ואח ' (2017)39 לפרטים נוספים).

איור 1. איור סכמטי של פרוטוקול MAPS המותאם ל- S. aureus. מבניית פלסמיד לניתוח נתונים. לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
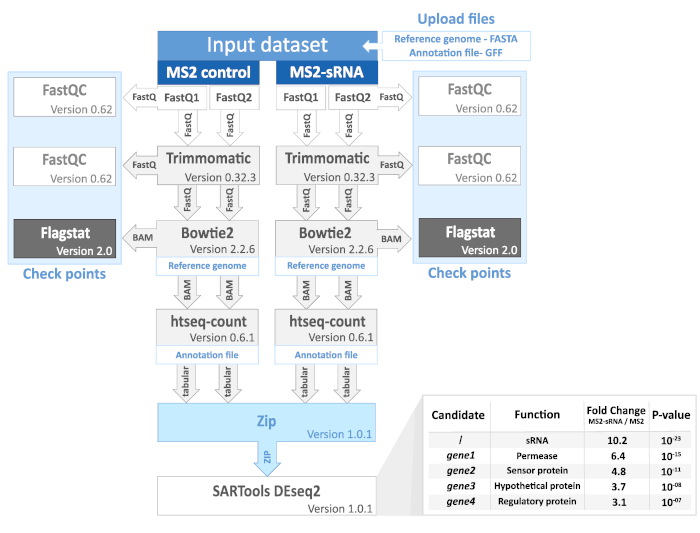
איור 2. זרימת עבודה של ניתוח MAPS ונתונים מעובדים. כל שלב, נקודת ביקורת ותבנית קובץ מיוצגים (ראה גם שלב 11). FastQ פורמט הוא קובץ טקסט המורכב רצפי DNA ציונים איכות המתאימים. תבנית BAM היא קובץ דחוס המכיל רצפים מיושרים. תבנית טבלאית היא קובץ טקסט המופרד באמצעות טאבים עם ספירות עבור כל גן. תרשים התוצאות ממחיש את סוג הנתונים שהושגו לאחר ניתוח ביואינפורמטי. התוצאות המוצגות הן פיקטיביות ואינן נובעות ממחקר כלשהו. לפרטים נוספים, ערכות לימוד בסיסיות זמינות באתר האינטרנט של Galaxy Project (https://galaxyproject.org/). לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 3: בניית פקדי אימות ומפות. A. ניתוח כתם צפון של SRNA RsaC אנדוגני ומבנים הקשורים MS2. זן WT נושא את pCN51-P3-MS2 (שליטה) ואת Δ זןמוטציה rsaC לשאת גם pCN51-P3-MS2, pCN51-P3-MS2-RsaC544 או pCN51-P3-MS2-RsaC1,116. דגימות נלקחו לאחר 2 שעות, 4 שעות ו 6 שעות של צמיחה BHI ב 37 מעלות צלזיוס. מבחני כתם צפוניים בוצעו באמצעות גשוש DIG ספציפי ל- RsaC. B.ניתוח כתמים צפון של שברי טיהור MS2-זיקה באמצעות בדיקה DIG ספציפי RsaC. הטיהור המשותף בוצע באמצעות זן WT + pCN51-P3-MS2 (בקרה) ו זן מוטציהrsaC Δ + pCN51-P3-MS2-RsaC544. תאים נקצרו לאחר 6 שעות של צמיחה BHI ב 37 °C (60 °F). תמצית גולמית (CE), זרימה דרך (FT), elution (E). ג. ניתוח כתמים צפוני של שברי טיהור זיקה MS2 (CE ו- E) באמצעות בדיקה DIG ספציפית לסודה. לפרטים ראו (B). לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
| חומצה ריבונוקלאית | סוג | הפניה |
| אשריצ'יה קולי | ||
| RyhB | sRNA | לאלאונה ואח' (2015)21 |
| RybB | sRNA | Lalaouna ואח '. (2015) 21 ,21 |
| 3'ETSלוז | קטע שמקורו ב- tRNA | לאלונה ומסה (2015)26 |
| DsrA | sRNA | Lalaouna ואח '. (2015) 22 (22) |
| הנס (לא) | mRNA (5'UTR) | Lalaouna ואח '. (2015) 22 (22) |
| CyaR | sRNA | Lalaouna ואח '. (2018) 23 , 23 |
| RprA | sRNA | Lalaouna ואח '. (2018) 23 , 23 |
| GcvB | sRNA | Lalaouna ואח '. (2019) 24 (24) |
| סלמונלה טיפימוריום | ||
| סרל (2012) | sRNA | סילבה ואח '. (2019) 25 |
| סטפילוקוקוס אוראוס | ||
| RsaA | sRNA | טומאסיני ואח '. (2017) 27 , 27 |
| RsaC | sRNA | Lalaouna ואח '. (2019) 29 ,29 |
| רסא"י (RsaI) | sRNA | ברונסקי ואח '. (2019) 28 (28) |
שולחן 1. טכנולוגיית MAPS חשפה את המטרה של מספר RNAs באורגניזמים שונים.
Discussion
פרוטוקול שונה עבור חיידקים גראם חיובי
הפרוטוקול הראשוני של MAPS פותח כדי לחקור אינטראקציה sRNA באורגניזם המודל E. coli20,30. כאן, אנו מתארים פרוטוקול שונה אשר מתאים לאפיון של רשתות רגולטוריות תלויות sRNA בפתוגן האנושי האופורטוניסטי S. aureus והוא בהחלט משתנה לחיידקים גראם חיוביים אחרים, פתוגניים או לא.
תשומת לב מיוחדת הוקדשה לשלב התמוגה של התא. העיתונות הצרפתית הוחלפה בכלי תמוגה מכני לתאים. שיטה זו יעילה כדי לשבור תאים גראם חיובי להגביל את הסיכונים הקשורים לטיפול זנים פתוגניים. כדי לשפר את התשואה של טיהור MS2-זיקה, כמות MS2-MBP משותק על שרף amylose הוגדלה באופן דרסטי. פעולה זו דרשה כדי להתאים את ההקפדה ואת מספר השטיפות.
שלא כמו הפרוטוקול הראשוני, MAPS מבוצע כאן בכפילות, משני עותקים משוכפלים ביולוגיים. זרימת עבודה פותחה כדי ליישם ניתוח סטטיסטי (איור 2), אשר מגדיל באופן סופי את החוסן של נתונים שהתקבלו.
מבנה MS2 והביטוי שלו
פרוטוקול MAPS זה הוקם כדי לזהות את היעד של sRNAs סטפילוקוקלי. מבנה MS2-sRNA מבוטא מפלסמיד מספר עותק נמוך (pCN51, 20 עד 25 עותקים / תא) ותחת שליטתו של מקדם P3 תלוי חישת מניין, בעיקר המושרה במהלך שלב נייח. תבנית ביטוי זו תואמת ל- sRNAs שנחקרו ב- S. aureus (כלומר RsaA, RsaI ו- RsaC) ומבטיחה סינתזת MS2-sRNA חזקה למדי. עם זאת, אנחנו לא יכולים לשלול כי במקרים אחרים, מקדם P3 לא יכול להיות מתאים ואינו משקף את המצב הפיזיולוגי הטבעי של חיידקים כאשר sRNA הוא המושרה. דרך נוספת לשלוט בייצור MS2-sRNA היא להשתמש מקדמים בלתי ניתנים להזנה כימית (למשל, מקדמי טטרציקלין בלתי ניתנים להערכה). כלים אלה מאפשרים ביטוי דופק של מבנה MS2-sRNA, אך הגדרה זו אינה מבטיחה שיעדי RNA יתבטאו בו-זמנית. חיסרון נוסף הוא כי ביטוי של פלסמיד יכול להוביל ייצור יתר של sRNA למד, מודגש עוד יותר עם וקטור מספר עותק גבוה. הדבר עלול לגרום לאינטראקציות אמנותיות או לשיבוש פונקציות נלוות של מלווים של RNA. החלופה המתאימה ביותר היא להכניס תג MS2 בקצה 5 ' של גן sRNA אנדוגני. לכן, MS2-sRNA יהיה מקודד כרומוזומלית תחת שליטתו של האמרגן המקורי שלה. זה צריך לחקות טוב יותר את הביטוי האנדוגני של sRNA למד ולהימנע הטיה עקב ייצור יתר של sRNA למד. בכל מקרה, הצעד המכריע הוא לאמת בזהירות את האורך, היציבות והפונקציונליות של מבנה MS2-sRNA, במיוחד באמצעות מבחני כתם צפון עם RNA הכולל שחולצו מתרבויות הגדלות בתנאים המפעילים את הייצור והתפקוד של sRNA אנדוגני. ללא ספק, השימוש במבנה MS2-sRNA לא תקין/לא מתפקד יכול להשפיע באופן דרסטי על התוצאות שנוצרו, ולתמוך בחשיבות הפקדים שתוארו לעיל. לבסוף, MS2-sRNA מיוצר תמיד ברקעsRNA Δ כדי למקסם את העשרת מטרות mRNA. יתר על כן, ניסויים יכולים להתבצע זנים מארחים עם מוטציות ספציפיות RNases (למשל, מוטציות מחיקה או מוטציות רגישות לטמפרטורה) כדי למנוע השפלה מטרות mRNA על ידי גיוס תלוי SRNA של RNases.
האימות הניסיוני של מועמדים שזוהו על ידי MAPS עדיין נדרש
נקודה אחת שיש לקחת בחשבון היא כי MAPS מספק רשימה של מטרות RNA putative. עם זאת, RNAs מסוימים עשויים להיות מועשרים בעקיפין באמצעות אינטראקציה עם יעד mRNA משותף או חלבון מחייב RNA. כתוצאה מכך, מועמדים שנחשפו חייבים להיות מאושרים על ידי גישות ניסיוניות אחרות. EMSA וניסויים טביעת רגל משמשים בדרך כלל כדי לדמיין את הכריכה הישירה של sRNA למטרות RNA putative במבחנה. לאחר מכן, במבחנה toeprinting מבחנים לעזור לפקח על ההשפעה של sRNA על ייזום תרגום של יעד mRNA שלה. לבסוף, כתמים צפון ו / או כתב גנים מבחנים (lacZ או gfp) להשלים אימות ניסיוני זה vivo. כל הגישות הללו מתוארות Jagodnik ואח '(2017)39.
שלא כמו טכנולוגיות RIL-seq ו- CLASH, MAPS אינה מספקת ישירות מידע באתרי האינטראקציה. עם זאת, שיא של קריאות ממופה נצפית לעתים קרובות באזור מוגבל של יעד RNA (למשל, הקצה 3 ' של glyW-cysT-leuZ operon21), להקל על זיהוי של אתר הזיווג. לחלופין, ניתן להשתמש במספר כלי חיזוי כדי לחזות את אתר האיגוד המכניס ביעד המזוהה כגון IntaRNA40.
MAPS בוחן את המטרה של sRNA מסוים
טכנולוגיית MAPS מתאימה לחקר יעד SRNA בחיידקים גראם שליליים כגון E. coli ו- S. Typhimurium, אך גם כעת עם פרוטוקול זה שונה בחיידקים גראם חיוביים כמו S. aureus. בעיקרון, MAPS מציע תמונת מצב של דופלקסים RNA:sRNA נוצר בזמן נתון ובתנאי צמיחה ספציפיים. למרבה הצער, רשימת מטרות mRNA יכול להיות שלם. לדוגמה, יעד mRNA קוגנט יכול להחמיץ בשל תנאים ניסיוניים לא הולמים, המצדיקים את אמצעי הזהירות שהוזכרו לעיל.
בהשוואה לשיטות נפוצות אחרות כגון RIL-seq או CLASH, MAPS משתמשת ב- sRNA ספציפי כדי לטהר במשותף את כל יעדי ה- RNA המקיימים אינטראקציה. כאן, אינטראקציה sRNA:RNA אינם מדוללים בין דופלקסים אחרים הקשורים RBP, המאפשר תיאורטית לאפיין את היעד שלה עמוק יותר. עם זאת, נראה כי שיטות RIL-seq / CLASH ו- MAPS חשפו סטים שונים של מטרות sRNA putative12,41, מה שמרמז כי גישות ניסיוניות אלה משלימות. פער זה יכול להיות מוסבר על ידי וריאציות בתנאי צמיחה ו / או הטיה שנוצר על ידי כל שיטה.
לפיכך, מטרת המחקר צריכה להשפיע על בחירת השיטה. לניתוח גלובלי של מטרות sRNA וכאשר RBP מעורב ברגולציה בתיווך sRNA, יש להעדיף שיטות RIL-seq ו- CLASH. שתי הגישות מספקות סקירה כללית של רשתות רגולטוריות המסתמכות על RBP מסוים, וכתוצאה מכך חושפות מגוון רחב של sRNAs ויעדים נלווים. להיפך, עבור המחקר של sRNA מסוים או כאשר אין RBP ידוע / מעורב, MAPS מייצג אפשרות מתאימה.
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
עבודה זו נתמכה על ידי "סוכנות לאומית דה לה Recherche" (ANR, גרנט ANR-16-CE11-0007-01, ריבוסטף, ו ANR-18-CE12- 0025-04, CoNoCo, ליחסי ציבור). הוא פורסם גם במסגרת labEx NetRNA ANR-10-LABX-0036 ושל ANR-17-EURE-0023 (ליחסי ציבור), כמימון מהמדינה המנוהלת על ידי ANR כחלק מההשקעות בתוכנית העתידית. DL נתמכה על ידי תוכנית המחקר והחדשנות Horizon 2020 של האיחוד האירופי במסגרת הסכם המענק מארי סקלודובסקי-קירי מס' 753137-SaRNAReg. העבודה במעבדת E. Massé נתמכה על ידי מענקי הפעלה של המכונים הקנדיים לחקר הבריאות (CIHR), המועצה לחקר מדעי הטבע וההנדסה של קנדה (NSERC), והמכונים הלאומיים לבריאות NIH צוות גרנט R01 GM092830-06A1.
Materials
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tube | Sarstedt | 72.690.001 | |
| 15 mL centrifuge tubes | Falcon | 352070 | |
| 2 mL microcentrifuge tube | Starstedt | 72.691 | |
| 2100 Bioanalyzer Instrument | Agilent | G2939BA | RNA quantity and quality |
| 250 mL culture flask | Dominique Dutscher | 2515074 | Bacterial cultures |
| 50 mL centrifuge tubes | Falcon | 352051 | Culture centrifugation |
| Absolute ethanol | VWR Chemicals | 20821.321 | RNA extraction and purification |
| Allegra X-12R Centrifuge | Beckman Coulter | Bacterial pelleting | |
| Ampicilin (amp) | Sigma-Aldrich | A9518-5G | Growth medium |
| Amylose resin | New England BioLabs | E8021S | MS2-affinity purification |
| Anti-dioxigenin AP Fab fragment | Sigma Aldrich | 11093274910 | Northern blot assays |
| Autoradiography cassette | ThermoFisher Scientific | 50-212-726 | Northern blot assays |
| BamHI | ThermoFisher Scientific | ER0051 | Plasmid construction |
| BHI (Brain Heart Infusion) Broth | Sigma-Aldrich | 53286 | Growth medium |
| Blocking reagent | Sigma Aldrich | 11096176001 | Northern blot assays |
| CDP-Star | Sigma Aldrich | 11759051001 | Northern blot assays (substrate) |
| Centrifuge 5415 R | Eppendorf | RNA extraction and purification | |
| Chloroform | Dominique Dutscher | 508320-CER | RNA extraction and purification |
| DIG-RNA labelling mix | Sigma-Aldrich | 11277073910 | Northern blot assays |
| DNase I | Roche | 4716728001 | DNase treatment |
| Erythromycin (ery) | Sigma-Aldrich | Fluka 45673 | Growth medium |
| FastPrep device | MP Biomedicals | 116004500 | Mechanical lysis |
| Guanidium Thiocyanate | Sigma-Aldrich | G9277-250G | Northern blot assays |
| Hybridization Hoven Hybrigene | Techne | FHB4DD | Northern blot assays |
| Hybridization tubes | Techne | FHB16 | Northern blot assays |
| Isoamyl alcohol | Fisher Scientific | A/6960/08 | RNA extraction and purification |
| LB (Lysogeny Broth) | Sigma-Aldrich | L3022 | Growth medium |
| Lysing Matrix B Bulk | MP Biomedicals | 6540-428 | Mechanical lysis |
| MicroPulser Electroporator | BioRad | 1652100 | Plasmid construction |
| Milli-Q water device | Millipore | Z00QSV0WW | Ultrapure water |
| NanoDrop spectrophotometer | ThermoFisher Scientific | RNA/DNA quantity and quality | |
| Nitrocellulose membrane | Dominique Dutsher | 10600002 | Northern blot assays |
| Phembact Neutre | PHEM Technologies | BAC03-5-11205 | Cleaning and decontamination |
| Phenol | Carl Roth | 38.2 | RNA extraction and purification |
| Phusion High-Fidelity DNA Polymerase | New England Biolabs | M0530 | Plasmid construction |
| pMBP-MS2 | Addgene | 65104 | MS2-MBP production |
| Poly-Prep chromatography column | BioRad | 7311550 | MS2-affinity purification |
| PstI | ThermoFisher Scientific | ER0615 | Plasmid construction |
| Qubit 3 Fluorometer | Invitrogen | 15387293 | RNA quantity |
| RNAPro Solution | MP Biomedicals | 6055050 | Mechanical lysis |
| ScriptSeq Complete Kit | Illumina | BB1224 | Preparation of cDNA librairies |
| Spectrophotometer Genesys 20 | ThermoFisher Scientific | 11972278 | Bacterial cultures |
| SpeedVac Savant vacuum device | ThermoFisher Scientific | DNA120 | RNA extraction and purification |
| Stratalinker UV Crosslinker 1800 | Stratagene | 400672 | Northern blot assays |
| T4 DNA ligase | ThermoFisher Scientific | EL0014 | Plasmid construction |
| TBE (Tris-Borate-EDTA) | Euromedex | ET020-C | Northern blot assays |
| ThermalCycler T100 | BioRad | 1861096 | Plasmid construction |
| Tween 20 | Sigma Aldrich | P9416-100ML | Northern blot assays |
| X-ray film processor | hu.q | HQ-350XT | Northern blot assays |
| X-ray films Super RX-N | FujiFilm | 4741019318 | Northern blot assays |
References
- Carrier, M. C., Lalaouna, D., Masse, E. Broadening the Definition of Bacterial Small RNAs: Characteristics and Mechanisms of Action. Annual Review of Microbiology. 72, 141-161 (2018).
- Hör, J., Matera, G., Vogel, J., Gottesman, S., Storz, G. Trans-Acting Small RNAs and Their Effects on Gene Expression in Escherichia coli and Salmonella enterica. EcoSal Plus. 9 (1), (2020).
- Desgranges, E., Marzi, S., Moreau, K., Romby, P., Caldelari, I. Noncoding RNA. Microbiology Spectrum. 7 (2), (2019).
- Adams, P. P., Storz, G. Prevalence of small base-pairing RNAs derived from diverse genomic loci. Biochimica et Biophysica Acta - Gene Regulatory Mechanisms. 1863 (7), 194524 (2020).
- Pain, A., et al. An assessment of bacterial small RNA target prediction programs. RNA Biology. 12 (5), 509-513 (2015).
- Desgranges, E., Caldelari, I., Marzi, S., Lalaouna, D. Navigation through the twists and turns of RNA sequencing technologies: Application to bacterial regulatory RNAs. Biochimica et Biophysica Acta - Gene Regulatory Mechanisms. , 194506 (2020).
- Wright, P. R., et al. CopraRNA and IntaRNA: predicting small RNA targets, networks and interaction domains. Nucleic Acids Research. 42, 119-123 (2014).
- Smirnov, A., Schneider, C., Hor, J., Vogel, J. Discovery of new RNA classes and global RNA-binding proteins. Current Opinion in Microbiology. 39, 152-160 (2017).
- Saliba, A. E., Santos, S., Vogel, J. New RNA-seq approaches for the study of bacterial pathogens. Current Opinion in Microbiology. 35, 78-87 (2017).
- Melamed, S., Adams, P. P., Zhang, A., Zhang, H., Storz, G. RNA-RNA Interactomes of ProQ and Hfq Reveal Overlapping and Competing Roles. Molecular Cell. 77 (2), 411-425 (2020).
- Melamed, S., et al. Global Mapping of Small RNA-Target Interactions in Bacteria. Molecular Cell. 63 (5), 884-897 (2016).
- Iosub, I. A., et al. Hfq CLASH uncovers sRNA-target interaction networks linked to nutrient availability adaptation. Elife. 9, (2020).
- Waters, S. A., et al. Small RNA interactome of pathogenic E. revealed through crosslinking of RNase E. The EMBO Journal. 36 (3), 374-387 (2017).
- Dos Santos, R. F., Arraiano, C. M., Andrade, J. M. New molecular interactions broaden the functions of the RNA chaperone Hfq. Current Genetics. , (2019).
- Kavita, K., de Mets, F., Gottesman, S. New aspects of RNA-based regulation by Hfq and its partner sRNAs. Current Opinion in Microbiology. 42, 53-61 (2018).
- Bohn, C., Rigoulay, C., Bouloc, P. No detectable effect of RNA-binding protein Hfq absence in Staphylococcus aureus. BMC Microbiology. 7, 10 (2007).
- Jousselin, A., Metzinger, L., Felden, B. On the facultative requirement of the bacterial RNA chaperone, Hfq. Trends in Microbiology. 17 (9), 399-405 (2009).
- Olejniczak, M., Storz, G. ProQ/FinO-domain proteins: another ubiquitous family of RNA matchmakers. Molecular Microbiology. 104 (6), 905-915 (2017).
- Lalaouna, D., Desgranges, E., Caldelari, I., Marzi, S. Chapter Sixteen - MS2-Affinity Purification Coupled With RNA Sequencing Approach in the Human Pathogen Staphylococcus aureus. Methods in Enzymology. 612, 393-411 (2018).
- Lalaouna, D., Prevost, K., Eyraud, A., Masse, E. Identification of unknown RNA partners using MAPS. Methods. 117, 28-34 (2017).
- Lalaouna, D., et al. A 3' external transcribed spacer in a tRNA transcript acts as a sponge for small RNAs to prevent transcriptional noise. Molecular Cell. 58 (3), 393-405 (2015).
- Lalaouna, D., Morissette, A., Carrier, M. C., Masse, E. DsrA regulatory RNA represses both hns and rbsD mRNAs through distinct mechanisms in Escherichia coli. Molecular Microbiology. 98 (2), 357-369 (2015).
- Lalaouna, D., Prevost, K., Laliberte, G., Houe, V., Masse, E. Contrasting silencing mechanisms of the same target mRNA by two regulatory RNAs in Escherichia coli. Nucleic Acids Research. 46 (5), 2600-2612 (2018).
- Lalaouna, D., Eyraud, A., Devinck, A., Prevost, K., Masse, E. GcvB small RNA uses two distinct seed regions to regulate an extensive targetome. Molecular Microbiology. 111 (2), 473-486 (2019).
- Silva, I. J., et al. SraL sRNA interaction regulates the terminator by preventing premature transcription termination of rho mRNA. Proceedings of the National Academy of Sciences. 116 (8), 3042-3051 (2019).
- Lalaouna, D., Masse, E. Identification of sRNA interacting with a transcript of interest using MS2-affinity purification coupled with RNA sequencing (MAPS) technology. Genomics Data. 5, 136-138 (2015).
- Tomasini, A., et al. The RNA targetome of Staphylococcus aureus non-coding RNA RsaA: impact on cell surface properties and defense mechanisms. Nucleic Acids Research. 45 (11), 6746-6760 (2017).
- Bronesky, D., et al. A multifaceted small RNA modulates gene expression upon glucose limitation in Staphylococcus aureus. The EMBO Journal. 38 (6), (2019).
- Lalaouna, D., et al. RsaC sRNA modulates the oxidative stress response of Staphylococcus aureus during manganese starvation. Nucleic Acids Research. 47 (1), 9871-9887 (2019).
- Carrier, M. C., Laliberte, G., Masse, E. Identification of New Bacterial Small RNA Targets Using MS2 Affinity Purification Coupled to RNA Sequencing. Methods in Molecular Biology. 1737, 77-88 (2018).
- Garibyan, L., Avashia, N. Polymerase chain reaction. Journal of Investigative Dermatology. 133 (3), 1-4 (2013).
- Revie, D., Smith, D. W., Yee, T. W. Kinetic analysis for optimization of DNA ligation reactions. Nucleic Acids Research. 16 (21), 10301-10321 (1988).
- Seidman, C. E., Struhl, K., Sheen, J., Jessen, T. Introduction of plasmid DNA into cells. Current Protocols in Molecular Biology. , (2001).
- Sanger, F., Coulson, A. R., Barrell, B. G., Smith, A. J. H., Roe, B. A. Cloning in single-stranded bacteriophage as an aid to rapid DNA sequencing. Journal of Molecular Biology. 143 (2), 161-178 (1980).
- Grosser, M. R., Richardson, A. R. Method for Preparation and Electroporation of S. aureus and S. epidermidis. Methods in Molecular Biology. 1373, 51-57 (2016).
- Krumlauf, R. Northern blot analysis. Methods in Molecular Biology. 58, 113-128 (1996).
- Koontz, L. Agarose gel electrophoresis. Methods in Enzymology. 529, 35-45 (2013).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2016 update. Nucleic Acids Reseaerch. 44, 3-10 (2016).
- Jagodnik, J., Brosse, A., Le Lam, T. N., Chiaruttini, C., Guillier, M. Mechanistic study of base-pairing small regulatory RNAs in bacteria. Methods. 117, 67-76 (2017).
- Mann, M., Wright, P. R., Backofen, R. IntaRNA 2.0: enhanced and customizable prediction of RNA-RNA interactions. Nucleic Acids Res. 45, 435-439 (2017).
- Georg, J., et al. The power of cooperation: Experimental and computational approaches in the functional characterization of bacterial sRNAs. Molecular Microbiology. 113 (3), 603-612 (2020).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved