Method Article
グラム陽性菌におけるRNAシーケンシングと結合したMS2-アフィニティ精製
要約
MAPS技術は、生体内の特定の調節RNAの標的を精査するために開発されました。目的のsRNAは、そのRNAパートナーの共精製とRNAシーケンシングによるそれらの同定を可能にするMS2アプタマーでタグ付けされます。この改変プロトコルは、特にグラム陽性菌に適しています。
要約
小さな調節RNA(sRNA)は、生命の細菌領域の間で広く普及しているが、それらの多くの機能は、彼らのmRNA標的を同定するのが難しいため、特に十分に特徴付けられていない。ここでは、RNAシーケンシング(MAPS)技術と組み合わせたMS2-アフィニティ精製の改変プロトコルについて説明し、インビボで特定のsRNAのすべてのRNAパートナーを明らかにすることを目指した。大まかに言えば、MS2アプタマーは、関心のあるsRNAの5'の端部に融合する。この構築物は、次いで生体内で発現され、MS2-sRNAがその細胞パートナーと相互作用することを可能にする。細菌の収穫後、細胞は機械的に取り付けられる。粗抽出物は、以前にマルトース結合タンパク質に融合したMS2タンパク質でコーティングされたアミロースベースのクロマトグラフィーカラムにロードされます。これにより、MS2-sRNAおよび相互作用RNAの特異的な捕獲が可能になります。溶出後、共精製RNAは、ハイスループットRNAシーケンシングとその後のバイオインフォマティクス解析によって同定されます。以下のプロトコルは、ヒトの植物体の黄色 ブドウ球菌 のグラム陽性で実施されており、原則として、任意のグラム陽性菌にトランスポーザブルである。要するに、MAPS 技術は、特定の sRNA の規制ネットワークを深く探索する効率的な手法を構成し、そのターゲットのスナップショットを提供します。しかし、MAPSによって特定された推定ターゲットは、補完的な実験的アプローチによって検証される必要があることを覚えておいてください。
概要
ほとんどの細菌ゲノムでは、何百もの、おそらく何千もの小さな調節RNA(sRNA)が同定されていますが、その大部分の機能は依然として特徴付けられません。全体として、sRNAは短い非コード分子であり、細菌生理学および変動環境への適応において主要な役割を果たしている1、2、3である。確かに、これらの高分子は、代謝経路、ストレス応答だけでなく、毒性および抗生物質耐性にも影響を与え、多数の複雑な調節ネットワークの中心にあります。論理的には、その合成は、特定の環境刺激(例えば、栄養飢餓、酸化または膜ストレス)によって引き起こされる。ほとんどの sRNA は、短いベースペアと非連続的な基本ペアを使用して、転写後レベルで複数のターゲット mRNA を調節します。彼らは通常、翻訳開始領域4のためにリボソームと競合することによって翻訳開始を防ぎます。sRNA:mRNA二重鎖の形成はまた、しばしば特定のRNasesのリクルートによって標的mRNAの活性分解をもたらす。
sRNA標的の特徴付け(すなわち、標的RNAのセット全体)は、それが介入する代謝経路およびそれが応答する潜在的なシグナルを同定することを可能にする。その結果、特定のsRNAの機能は、一般にその標的から推測することができる。この目的のために、インタRNAおよびCopraRNA5、6、7のようなインシリコ予測ツールの幾つかが開発されている。彼らは特に、推定sRNAパートナーを決定するために、潜在的な相互作用部位のエネルギーとアクセシビリティを組み合わせた、シーケンスの相補性に依存しています。しかし、予測アルゴリズムは、RNAシャペロン8の関与が最適でない相互作用または両パートナーの共発現を支持するなど、生体内での塩基対に影響を与えるすべての因子を統合するわけではない。その固有の制限により、予測ツールの誤検出率は依然として高いままです。大部分の実験的大規模アプローチは、sRNA:mRNAカップルがタグ付きRNA結合タンパク質(RBP)6,9と相互作用する共精製に基づいている。例えば、ライゲーションとシーケンシングによるRNA相互作用(RIL-seq)法により、エシェリヒア・コリ10,11におけるHfqおよびProQなどのRNAシャペロンと共精製されたRNA二重鎖を同定した。ハイブリッドのUV架橋、ライゲーションおよびシーケンシング(CLASH)と呼ばれる同様の技術が、大腸菌12,13のRNase E-およびHfq関連sRNAに適用された。複数の細菌におけるsRNA媒介性調節におけるHfqおよびProQの十分に記述された役割にもかかわらず、sRNAベースの調節は、S.アウレウス16、17、18のようないくつかの生物においてRNAシャペロン非依存性であるようである。RNasesに関連するRNA二重鎖の精製がウォーターズと同僚13によって示されるように実現可能であっても、これはRNasesが急速な劣化を引き起こすので難しいままです。したがって、RNAシーケンシング(MAPS)アプローチ19,20と結合したMS2-アフィニティ精製は、このような生物における固体代替を構成する。
上記の方法とは異なり、MAPSは、すべての相互作用RNAを捕捉するために餌として特定のsRNAを使用するため、RBPの関与に依存しません。プロセス全体を 図 1に示します。簡単に言えば、sRNAはMS2コートタンパク質によって特異的に認識されるMS2 RNAアプタマーで5'でタグ付けされます。このタンパク質は、アミロース樹脂上に固定化されるマルトース結合タンパク質(MBP)と融合している。したがって、MS2-sRNAおよびそのRNAパートナーは、アフィニティークロマトグラフィーカラム上に保持されます。マルトースとの溶出後、共精製RNAを、高スループットRNAシーケンシングを用いて、バイオインフォマティクス解析を行って同定する(図2)。MAPS テクノロジーは、最終的に、インビボで発生するすべての潜在的な相互作用の相互作用マップを描画します。
MAPS技術は、もともと非病原性グラム陰性細菌大腸菌21に実装されました。驚くべきことに、MAPSは、RyhBおよびRybB sRNAの両方と特異的に相互作用するtRNA由来の断片を同定し、非誘導条件でmRNA標的を調節するためのsRNA転写ノイズを防止するのに役立った。その後、MAPSはDsrA 22、RprA 23、CyaR23およびGcvB24のような他の大腸菌sRNAに正常に適用された(表1)。MAPS は、以前に知られていた標的を確認した上で、これらの既知の sRNA のターゲットを拡張しました。最近、MAPSはサルモネラ・チフィムリウムで行われ、SraL sRNAがrho mRNAに結合することを明らかにし、転写終結因子25に対するコード化を行う。この組み合わせを通じて、SraLはRho自体によって引き起こされる早期転写終結からrho mRNAを保護する。興味深いことに、この技術はsRNAに限定されず、tRNA由来フラグメント26およびmRNA2の5'未翻訳領域の使用によって例示されるあらゆるタイプの細胞RNAに適用することができる(表1)。
MAPS法は、病原性グラム陽性菌S.アウレウス19にも適応されている。具体的には、リシスプロトコルは、グラム陰性細菌よりも細胞壁が厚いため、細胞を効率的に破壊し、RNAの完全性を維持するために広く改変されている。この適応プロトコルは、すでにRsaA 27、RsaI28とRsaC29の相互作用を解明しました。このアプローチは、細胞表面特性、グルコース取り込み、および酸化ストレス応答の調節機構におけるこれらのsRNAの重要な役割に関する洞察を与えた。
2015年に 大腸菌 で開発され実装されたプロトコルは、最近では非常に詳細に説明されています30.ここでは、非病原性または病原性(安全上の注意)にかかわらず、グラム陽性(より厚い細胞壁)細菌におけるsRNA調節ネットワークの研究に特に適した改変MAPSプロトコルを提供する。
プロトコル
1. バッファとメディア
- MAPS の実験では、次のバッファーとメディアを準備します。
バッファ A (150 mM KCl、20 mM トリス-HCl pH 8、1 mM MgCl2 および 1 mM DTT)
バッファE(250 mM KCl、20 mMトリス-HCl pH 8、12 mMマルトース、0.1%トリトン、1 mM MgCl2 および1 mM DTT)
- RNAローディングバッファー(0.025%キシレンシアノール、8 M尿素中の0.025%ブロモフェノールブルー)
脳心注入(BHI)培地(ふくらはぎ脳12.5g、ペプトン10g、牛肉心臓5g、NaCl5g、NaClの2.5g、1Lのグルコース2g)
- リソゲニーブロス(LB)培地(ペプトン10g、酵母エキス5g、NaCl1G1G) - ノーザンブロットアッセイの場合は、次のバッファーを準備します。
- ブロッキング溶液(1xマレイン酸および1%ブロッキング試薬)
- ハイブリダイゼーション溶液(50%ホルムアミド、5x SSC、7%SDS、1%ブロッキング溶液および0.2%N-ラウリルサルコシン、50 mMリン酸ナトリウム)。溶解する攪拌で熱します。
注意: 各製品に関する安全上の注意に注意してください。
- 1 M リン酸ナトリウム (58 mM リン酸ナトリウム二分性ナトリウムおよび 42 mM リン酸ナトリウム一塩基)
- 生理塩水、クエン酸ナトリウム(SSC)バッファー、20x濃縮物(3 M NaClおよび300 mMクエン酸三ナトリウム)
2. 安全上の問題
- レベル2の封じ込めラボで生じうる病原性細菌を含むすべてのステップを実行します。
注:細胞抽出物のみ、リシス後に外に取り出すことができます(ステップ5)。 - ラボコートと手袋を着用してください。
- 手首が覆われていることを確認します。
- 消毒液で生物安全キャビネット(クラスII)を洗浄します。
- 適切な生物医学のビンに細菌にさらされた固形廃棄物を処分します。
- 汚染された液体を含むフラスコを消毒液で処理します。その後、シンクに捨てます。
- 慎重に石鹸で手と手首を洗い、レベル2の封じ込めラボを離れる前にラボコートを取り外します。
3. プラスミド建設
注: クローニングの目的では、まず内因性 sRNA の境界を特定することが重要です。pCN51-P3およびpCN51-P3-MS2プラスミドはトマシニら(2017)27に記載されている。P3プロモーターは、細胞密度依存的な方法(すなわち、細菌が成長の静止期に入るとき)でsRNAの高い発現を可能にする。多くのブドウ球菌は、この成長期に蓄積します。
- 高忠実度DNAポリメラーゼおよびPCRマシンを用いてPCRによりsRNA配列を増幅します。メーカーの指示に慎重に従い、詳細については、ガリビアンとアバシア(2013)31を読んでください。
- 次のテンプレートを使用して、特定のプライマーを設計します。

それぞれフォワードプライマーとリバースプライマー用の 5'-CGCGGATCC(N)-3'
注:これらのオリゴヌクレオチドは、目的のsRNAの5'末端までMS2配列(太字)を融合することを可能にする。 PstIおよび BamHI制限部位(下線部)は、PCN51-P3プラスミド27にアンプリコンをクローン化するためにMS2-sRNA構築物の5'および3'の四肢に追加される。(N)は、遺伝子特異的配列(15〜20ヌクレオチド)に対応する。 - メーカーの推奨に従って、適切なバッファに2 Uの PstIと1 Uの BamHIを備えたpCN51-P3プラスミド1μgとMS2-sRNA PCR産物1μgを消化します。
- 37°Cで1時間インキュベートし、PCR精製キットを使用してDNAを精製します( 材料表を参照)。
- 消化したpCN51-P3プラスミド(300 ng)とMS2-sRNAアンプリコン(ベクターのモル比:挿入=1:3)を1.5 mLチューブに混ぜます。ライゲーション効率を最大化する Revie ら (1988)32 を参照してください。各チューブにリガーゼバッファー1μL、T4リガーゼ10 Uを加えます。超純水で10μLに音量を調整します。
- 22°Cで2時間以上インキュベートする。
- 5 μL のライゲーション混合物を 50 μL の凍結 DH5α 化学的に有能な大腸菌細胞に加えます。プラスミド変換と化学的に有能な細胞の詳細については、Seidmanら(2001)33を読んでください。
- 氷の上で30分インキュベート。
- ヒートショック(42°Cで45s)をヒートブロックまたはウォーターバスを用いた変換チューブ。
- LB培地900μLを加え、37°Cで30分間インキュベートします。
- アンピシリン(100 μg/μL)を補充したLB寒天プレート上の細菌懸濁液のプレート100 μL。
注:pCN51-P3ベクターはアンピシリン耐性遺伝子をコードし、pCN51-P3-MS2-sRNAプラスミドを運ぶ 大腸菌 クローンのみを選択することができます。 - プラスミドDNAミニプレップキットを使用して、アンピシリン(100 μg/μL)の存在下で増殖した一晩の細菌培養物(5 mL)からpCN51-P3-MS2-sRNAプラスミドを抽出する( 材料表を参照)。
- サンガーシーケンシング34 で構築を確認するには、次のプライマーである5'-TCTCGAAAATAATAGAGGG-3'を使用します。
- pCN51-P3-MS2-sRNA プラスミドを DC10B 化学的に有能 な大腸菌 細胞に変換します。ステップ 3.7 ~ 3.11 を繰り返します。
- pCN51-P3-MS2-sRNAプラスミド(ステップ3.12を参照)を抽出し、エレクトロポレーション装置を用いてプラスミドDNAの1〜5μgをHG001 ΔsRNA電管性S.アウレウス細胞に変換します。製造元の指示に慎重に従ってください。エレクトロコンピテントS.アウレウスを準備する方法の詳細については、グロッサーとリチャードソン(2016)35を読んでください。
注意:このステップは、病原性細菌の取り扱いを含みます(ステップ2を参照)。 - BHI培地を900μL加え、37°Cで3時間インキュベートします。
- 遠心分離機 1 分で 16,000 x g.上清を捨てます。
- 100 μL の BHI にペレットを再懸濁し、エリスロマイシン(10 μg/μL)を加えたBHI寒天プレートに細菌懸濁液をプレートします。
注:pCN51-P3ベクターはまた、エリスロマイシン耐性遺伝子をコードし、pCN51-P3-MS2-sRNAプラスミドを運ぶ S.アウレウス クローンのみを選択することを可能にする。
4. バクテリアの収穫
注意:このステップは、病原性細菌の取り扱いを含みます(ステップ2を参照)。
- pCN51-P3-MS2-sRNAまたはpCN51-P3-MS227 プラスミドのいずれかを持つ株の1コロニーを、エリスロマイシン(10μg/μL)を添加したBHI培地の3mLで増殖させます。
- エリスロマイシン(10μg/μL)を添加した新鮮なBHI培地の1晩培養液を50mL(≈1/100)で希釈し、0.05のOD600nm に達します。250 mL滅菌フラスコ(フラスコ対ミディアム比5:1)を使用してください。
注:中および成長条件は、研究されたsRNAの発現パターンに従って設定する必要があります。 - 180 rpmで6時間振ると37°Cで培養を成長させます。
- 各培養物を50 mL遠心チューブに移します。
- 4°Cで15分の間に2,900 x g で遠心分離機。 上清を捨てます。
- 氷の上にペレットを保持し、直接機械的な細胞のリシスを行う(ステップ5)または凍結し、-80 °Cでペレットを保存します。
5. 機械的細胞のライシス
注意: 氷上で以下の手順を実行する必要があり、バッファーは 4 °C でなければなりません。 手袋を使用し、RNasesからサンプルを保護するためにすべての予防措置を取ります。
- ペレットをリサスペンド(ステップ 4.6)、バッファー A の 5 mL にします。
- リサスペンドセルを3.5 gのシリカビーズ(0.1 mm)で15 mL遠心分離管に移します。
- 機械的な細胞のライシスの器械に管を挿入する( 材料のテーブルを参照して)。4.0 m/s で 40 s のサイクルを実行します。
注:1サイクルで細胞を壊すのに十分でない場合は、サンプルを氷の上に置いたまま、デバイスを5分間冷却してください。次に、4.0 m/s で 40 s のサイクルを繰り返します。細胞のライシスの効率は、BHI寒天板上に上清をめっきすることにより試験することができる。 - 15,700 x g で遠心分離機を15分間使用します。上清を回収し、氷の上に保管してください。
6. カラムの準備
注意:アミロース樹脂を乾燥させないように注意してください。必要に応じて、エンドキャップで柱を密封します。アフィニティー精製を開始する前に、すべてのソリューションを準備してください。
- クロマトグラフィー列を列ラックに配置します ( 資料表を参照)。
- 柱の先端を取り出し、超純水で洗います。
- アミロース樹脂を300μL加えます。
- 10 mLのバッファー A でカラムを洗います。
- 6 mLのバッファAでMBP-MS2タンパク質の1,200 pmolを希釈し、カラムにロードします。
- 10 mLのバッファー A でカラムを洗います。
7. MS2-アフィニティ精製 (図 1)
- セルのライセートをカラムにロードします。
注:細胞ライセートの1mL(粗抽出物、CE)を保持し、総RNA(ステップ8)を抽出し、ノーザンブロット(ステップ9)および転写(ステップ10)分析を行います。 - クリーンなコレクションチューブにフロースルー分画(FT)を収集します。
- 10 mLのバッファーAでカラムを3回洗います。
- バッファーEの1 mLでカラムを溶出し、2 mL マイクロチューブに溶出分 (E) を集めます。
- RNA抽出(ステップ8)まで、収集した分画をすべて氷上に保管するか、後で使用するために-20°Cで凍結します。
8. 回収された分画のRNA抽出(CE、FT、WおよびE)
- RNA抽出には各画分(FTおよびWを含む)の1mLを使用してください。
- フェノールを1巻加えます。激しく混ぜます。
注意:フェノールは揮発性と腐食性であり、注意を払い、煙のフードの下で安全に動作します。 - 遠心分離機 16,000 x g で 10°C で 10 分間
- きれいな2 mLマイクロチューブの上相を移す。
- クロロホルム/イソアミルアルコールの1ボリューム(24:1)を加え、ステップ8.3から8.4を繰り返します。
注意:ヒュームフードの下で安全に作業してください。 - 2.5量の冷エタノール100%、3M酢酸ナトリウム(NaOAc)pH5,2の1/10容量を加えます。
- -20°Cで一晩沈殿する。
注:降水は、20分の間にエタノール/ドライアイス浴で、または2時間の間に-80 °Cで行うこともできます。 - 遠心分離機 16,000 x g で 4 °C で 15 分 ペレットを乱さないよう気をつけながら、ピペットでエタノールをゆっくり取り除く。
注意:RNAペレットは必ずしも目に見えず、エタノールの存在下で緩んでいる場合があります。 - 80%の冷エタノールを500μL加えます。
- 遠心分離機 16,000 x g で 5°C で 5 分.
- エタノールをゆっくりとピペットして捨てます。真空濃縮器を使用してペレットを乾燥させ、実行モードで5分。
- ペレットを適切な体積(15~50 μL)の超純水で再懸濁します。後で使用するために-20°Cでペレットを凍結します。
- 分光光度計/フルオロメーターを使用して、RNAの量(260 nm)と品質(260/280および260/230波長比)を評価します( 材料表を参照)。製造元の指示に慎重に従ってください。
注:3〜4 μgは溶出分(E)で一般に得られる。これは主にテストされた条件に依存します。
9. ノーザンブロットによるMS2アフィニティ精製の分析36
- 10μLの超純水で5μgのCE、FT、W画分および500ngのE分画のRNAを希釈し、10μLのRNAローディングバッファーと混合します。
- 90°Cで3分間インキュベートする。
- 20 mMのグアニジウム・チオシアネートを補った1%アガロースゲルのウェルにサンプルをロードし、4°CのTBE 1xバッファーで100-150 Vでゲルを実行します。 詳細については、クーンツ (2013)37 をお読みください。
- ニトロセルロース膜上でRNAを1時間または一晩の毛細血管移動のための真空移動によって移動する。
注: 毛細血管の方法は、大きな RNA の方が効率的です。 - 紫外線架橋器を用いた膜上のUV架橋RNA(254nmで120mJ)
- RNA側を上に向けてハイブリダイゼーションボトルに膜を挿入します。
- 予熱ハイブリダイゼーション溶液の10〜20 mLを加えます。68°Cで30分インキュベート。
- 溶液を廃棄し、1μLのsRNA特異的プローブを添加したフレッシュハイブリダイゼーション溶液を10~20 mL添加します。68°Cで一晩インキュベートする。
注:DIGラベルのRNAプローブは、DIG RNAラベリングキットとメーカーの指示に従って合成されています。あるいは、ラジオラベル付きプローブを使用することもできます。 - 10~20 mLの洗浄液1(2x SSCおよび0.1%SDS)で膜を5分間20°Cで洗浄します。 1 回繰り返します。
- 洗浄液2(0.2x SSCおよび0.1%SDS)の10-20 mLで膜を68°Cで15分間洗浄します。 1 回繰り返します。
- 20°Cで少なくとも30分間、ブロッキング溶液を10〜20 mLでインキュベートする。
- 溶液を廃棄し、ポリクローナル抗ジゴキシゲニン抗体(1/1000)を添加したブロッキング溶液の10〜20mLを加え、アルカリホスファターゼに共役する。20°Cで30分インキュベート。
- 洗浄液3(マレイン酸1x、Tween200)を10~20mLで20°Cで15分間洗浄します。 1 回繰り返します。
- 検出溶液の10〜20 mLで膜をインキュベート(0.1 MトリスHClおよび0.1 M NaCl pH 9.5)20°Cで5分。
- 膜をプラスチックフィルムに付け、基板に浸します( 材料表を参照)。暗闇の中で5分をインキュベートします。
- 膜をプラスチックフィルムに密封します。膜をオートラジオグラフィーカセットに入れます。
- 専用の暗室で、膜をオートラジオグラフィーフィルムに露出します。
メモ:博覧会の時間は、数秒から数分の信号強度に依存します。 - 自動開発装置で露出したフィルムを明らかにする。
RNAシーケンシング用サンプルの調製
注: このステップは E および CE の分数から抽出された RNA に関するみ。
- 各サンプル10μLの10倍DNaseバッファとDNase I(1 U/μgの処理されたRNA)を加えます。水を加えるので、最終容積は100μLです。
- 37°Cで1時間インキュベートする。
- 前述のようにRNAを抽出して精製します(ステップ8.2~8.11)。
- RNAペレットを20μLの超純水で再懸濁します。
注:残りのDNAの存在は、PCRおよび特定のプライマー(例えば、16S遺伝子)を使用して確認することができます。 - マイクロ流体ベースの電気泳動解析システムを使用してRNAの量と品質を評価します( 材料表を参照)。
注:1μgは、一般にDNase処理後の溶出画分(E)で得られます。 - 細菌性rRNA枯渇キットでリボソームRNAを除去します。
注: 大きく豊富な RNA(すなわち、rRNA)は、アフィニティーカラムと非特異的に相互作用する傾向があります。このステップを実行するには、抽出されたRNAの500ngが必要です。 - マイクロ流体ベースの電気泳動解析システムを使用して、RNAの量と品質を再び評価します。
- cDNAライブラリ調製キットとメーカーの指示に従って、リボ枯渇したRNAの10〜20 ngでcDNAライブラリを準備します。
- シーケンシング装置を使用して取得したライブラリをシーケンスする (例えば、シングルエンド、150 bp; 材料表を参照)。
注:サンプルあたり5〜1000万読み取りは、一般的に十分です。
11. RNAseq データ分析 (図 2)
- シーケンス プラットフォームから FastQ シーケンス ファイルをダウンロードします。
- Roscoffの生物学的ステーションのギャラクシーインスタンスへのアクセス(https://galaxy.sb-roscoff.fr/)とログイン。
注: すべてのアルゴリズムは、検索バーを使用して簡単に見つけることができます。各ツールに関するユーザー ガイドが提供されています。
注意: 必要なツールのバージョンは、公開ギャラクシーサーバー38とは異なる場合があります。 - [ データの取得 ] アイコンをクリックし、[ コンピュータからファイルをアップロード]をクリックします。各 MS2 コントロールと MS2-sRNA サンプルの FastQ シーケンス ファイルをアップロードします。また、FASTA参照ゲノムファイルとGFF注釈ファイルをアップロードします。
- FastQC 読み取り品質レポート (ギャラクシー バージョン 0.69) を実行します。
注:このツールは、生のシーケンス(例えば、品質スコア、アダプタシーケンスの存在)の品質評価を提供します。 - Trimmomaticフレキシブルな読み取りトリミングツール(Galaxyバージョン0.36.6)を実行して、特にアダプタシーケンスと低品質の読み取りを削除します。ライブラリの準備に使用されるアダプター シーケンスを示します (たとえば、TruSeq 3、シングル エンド)。次のトリムモマティック演算を追加します: スライディングウィンドウ (基数 =4;平均品質=20)および最小レン(読み取りの最小長=20)。
- FastQC 読み取り品質レポート (Galaxy バージョン 0.69) を再度実行します。
- Bowtie2を実行する - マップは、参照ゲノム(ギャラクシーバージョン2.3.2.2)に対して読み取ります。履歴のゲノム参照 FASTA ファイルを使用して、読み取りを既定の設定 (非常に機密性の高いローカル) でマップします。
注: Bowtie2 ツールによって生成された BAM ファイルは、統合ゲノミクス ビューアー (IGV) を使用して視覚化できます。関連付けられた BAI ファイルも必要です。 - 必要に応じて、BAM データセットの統計をコンパイルする Run Flagstat (Galaxy Version 2.0)
- 実行 htseq-count - GFF アノテーション ファイル内の重複するフィーチャを揃えるカウント (Galaxy バージョン 0.6.1) を実行します。[交差(空でない)]モードを使用します。
- htseq-count 分析のすべての生カウントファイルを 1 つの Zip ファイルにアーカイブします。
- SARTools DESeq2 を実行してデータを比較します (Galaxy バージョン 1.6.3.0)。生のカウント ファイルとデザイン ファイルを含む Zip ファイルを指定します。指示に従って設計ファイルを生成します。
結果
代表的な結果は、S. アウレウス29の RsaC 標的の研究に由来します。RsaCは型破りな1,116 nt-long sRNAである。その5'末端は、その3'の終わり(544 nt)が構造的に独立している間、いくつかの反復領域を含み、そのmRNA標的との全ての予測された相互作用部位を含む。このsRNAの発現は、マンガン(Mn)が不足しているときに誘導され、宿主免疫応答の文脈でしばしば遭遇する。MAPS技術を用いて、RsaCと直接相互作用するいくつかのmRNAを同定し、酸化ストレス(sodA、ldh1およびsarA)および金属関連(znuBC-zurおよびsufCDSUB)応答におけるその重要な役割を明らかにした。
MS2-sRNAの構造と実験条件の検証
MAPS実験を行う前に、研究したsRNAの発現の最適条件を決定することが重要である。非ネイティブプロモーターが使用される場合、標的が存在する場合にMS2-sRNAコンストラクトを生成するのに決定的に役立ちます。また、MS2-sRNAの構造は、サイズ、安定性、発現および機能に関して慎重に検証する必要があります。MS2アプタマーは、RsaCの3'部分に対応する全長RsaC(MS2-RsaC1116)または短い形式(MS2-RsaC544)の5'末端に融合した。両方の構成体は 、S.アウレウス HG001ΔrsaC におけるクォーラムセンシング依存P3プロモーターの制御下で生体内で発現した。 rsaC 遺伝子の欠失は内因性RsaCとMS2-RsaCの間の競争を避ける。MS2タグだけで同じベクターを含む野生型株を制御として用いた。このコントロールを使用すると、MS2 タグで発生する非特異的な相互作用を減算できます。
その構造を確認し、その発現パターンを可視化するために、細菌細胞を37°CでBHI培地で2時間、4時間および6時間増殖した後に採取した。 RNA抽出後、ノーザンブロット分析はRsaC特異的DIGプローブを用いて行った(図3A)。内因性RsaC(レーン1-3)のレベルは、6時間の成長後に有意に増加し、MAPS実験のためにこの時間ポイントの選択を正当化した。重要なことに、MS2-RsaC544( レーン7-9)およびMS2-RsaC1,116( レーン10-12)のレベルは、6時間で内因性RsaCに匹敵した。したがって、それらはRsaCの内因性発現パターンを模倣すべきである。RsaCのより大きいがマイナーな形態は区別可能であり、転写の非効率的な終わりによる可能性がある。この現象は、MS2-sRNAがプラスミド21から強力なプロモーターの制御下で発現される場合にしばしば観察される。異常転写終結または分解に起因する短い形態は認められない。
SRNAの5'でMS2アプタマーを添加することも、適切な折りたたみを中断し、その機能に影響を与える可能性があります。このステップは、RsaC として高度に構造化された sRNA にとって重要です。したがって、MS2-sRNA活性は、可能な場合に内因性sRNAとテストし、比較する必要があります。以前に知られていたターゲットまたは観察可能な表現型は、それを監視するのに役立ちます。例えば、細胞内ROS蓄積に対するRsaCの影響は、MS2-RsaC544 およびMS2-RsaC1,116 の構成体29を検証するために使用された。
アフィニティー精製中に収集した分数の分析
RNAは、MS2タグ単独およびΔrsaC株を発現するWT株中のCE、FTおよびE画分から抽出し、MS2-RsaC544コンストラクトを発現した。ノーザンブロット分析を用いて、1,116nt-long内因性RsaCは溶出画率で濃縮されたが、アフィニティカラムとは特に相互作用することが判明した(図3B、レーン2-3)。MS2-RsaC1,116で同じ現象を観測しました(データは示されていません)。これは確かにその長さと複雑な二次構造によるものです。したがって、その3'部分に対応するRsaCの構造が少なく、より短い形式(544 nt)のみがMAPS実験を行うために使用された。図3Bにおいて、MS2-RsaC544は溶出画分で高く富化し、MS2-MBP融合タンパク質(レーン6)によって正常に保持されたことを示した。図3Aのように、より大きいがマイナーなMS2-RsaC544が観察された。ここで、ストリンジェンシーおよびワッシュの数は、非特異的結合を減少させるか、反対に、真の相互作用パートナーの損失を制限するように調整することができる。
MAPS分析後の推定mRNA標的の検証
生物情報学的解析の後、推定mRNA標的は、MS2-sRNAとMS2対照の間の折り畳み変化に従ってリストされ、DeSeq2を用いて得られる(図2)。例えば、MS2-RsaC544 MAPSデータ29は、S.アウレウスにおけるスーパーオキシドジスムターゼのコーディングであるsodA mRNAが主な標的であることを示唆した(最高のヒット、より高いフォールド変化)。ノーザンブロット分析は、MS2アフィニティー精製後にsodA特異的DIGプローブを用いて行い、SODAがMS2制御と比較してMS2-RsaC544と効率よく共濃縮されたことを示している(図3C)。
グローバルなトランスクリプト分析は、CE画分に体系的に行われます。MAPS データとこのトランスクリプト分析の比較は、エンリッチメント比を調整するのに役立ち、潜在的なターゲット階層を明らかにします。実際、MS2親和性精製後に高度に富化された発現性の悪いmRNAは、確かに高濃縮および高発現mRNAよりも結合親和性が高い。
MAPSによって同定されたすべての候補は、電気泳動モビリティシフトアッセイ(EMSA)またはレポーター遺伝子アッセイ(詳細についてはJagodnik et al.(2017)39を参照)などのin vitroおよび/またはin vivo実験を使用して個別に検証する必要があることに注意することが重要です。

図 1. S. アウレウスに適合した MAPS プロトコルの概略図。 プラスミドの構築からデータ解析まで。 この図の大きなバージョンを表示するには、ここをクリックしてください。
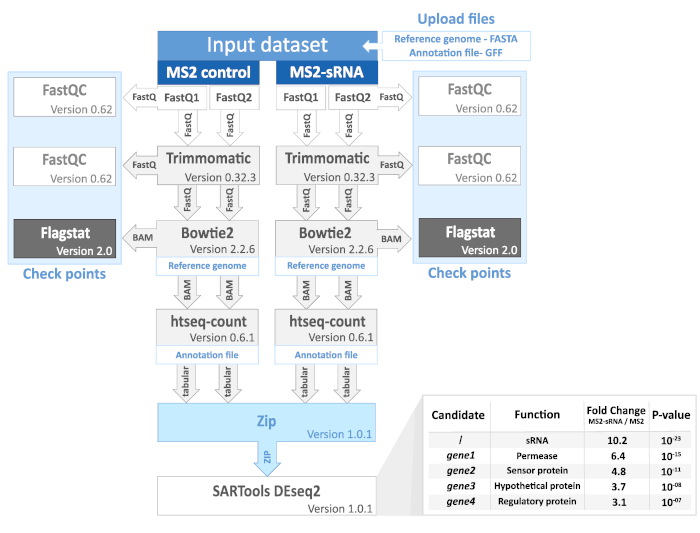
図 2.MAPS 分析ワークフローと処理されたデータ。 各ステップ、チェックポイント、ファイル形式が表されます(ステップ11も参照)。FastQフォーマットは、DNA配列と対応する品質スコアからなるテキストファイルです。BAM 形式は、整列されたシーケンスを含む圧縮ファイルです。表形式は、各ジーンのカウントを含むタブ区切りのテキスト ファイルです。結果チャートは、バイオインフォマティクス解析後に得られたデータの種類を示しています。提示された結果は架空のものであり、いかなる研究からも生じるものではありません。詳細については、基本的なチュートリアルはギャラクシープロジェクトのウェブサイト(https://galaxyproject.org/)で入手できます。 この図の大きなバージョンを表示するには、ここをクリックしてください。

図 3: 検証コントロールと MAPS コントロールを構築する内因性RsaC sRNAおよび関連MS2コンストラクトのノーザンブロット分析。WT株はpCN51-P3-MS2(対照)およびΔrsaC変異株を運ぶpCN51-P3-MS2、pCN51-P3-MS2-RsaC544またはpCN51-P3-MS2-RsaC1,116のいずれかを運ぶ。試料は、37°CでBHIで2時間、4時間および6時間の成長後に採取した。 ノーザンブロットアッセイは、RsaC特異的DIGプローブを用いて実施した。RsaC特異的DIGプローブを用いたMS2-アフィニティー精製画分のノーザンブロット分析共精製はWT株+pCN51-P3-MS2(対照)およびΔrsaC変異株+pCN51-P3-MS2-RsaC544を用いて行った。細胞は、37°CでBHIで6時間の増殖後に採取した。 粗抽出物(CE)、フロースルー(FT)、溶出(E)。C. SODA特異的DIGプローブを用いたMS2-アフィニティー精製画分(CEおよびE)のノーザンブロット分析。詳細については、 (B) を参照してください。この図の大きなバージョンを表示するには、ここをクリックしてください。
| RNA | 種類 | 参考 |
| 大腸菌 | ||
| ライブ | sRNA | ララウナ ら (2015)21 |
| ライブブ | sRNA | ララウナ ら.(2015)21 |
| 3'ETSleuZ | tRNA由来フラグメント | ララウナとマッセ (2015)26 |
| DsrA | sRNA | ララウナ ら.(2015)22 |
| hns | mRNA (5'UTR) | ララウナ ら.(2015)22 |
| シアル | sRNA | ララウナ ら.(2018)23 |
| ルプラ | sRNA | ララウナ ら.(2018)23 |
| GcvB | sRNA | ララウナ ら.(2019)24 |
| サルモネラ菌 ティフィムリウム | ||
| スラール | sRNA | シルバ ら.(2019)25 |
| 黄色 ブドウ 球菌 | ||
| RsaA | sRNA | トマシニ ら.(2017)27 |
| RsaC | sRNA | ララウナ ら.(2019)29 |
| RsaI | sRNA | ブロネスキー ら.(2019)28 |
表 1.MAPS技術は、様々な生物におけるいくつかのRNAの標的を明らかにした。
ディスカッション
グラム陽性菌の改変プロトコル
MAPSの初期プロトコルは、モデル生物大腸菌20,30におけるsRNA相互作用を研究するために開発された。ここでは、日和見ヒト病原体S.アウレウスにおけるsRNA依存的調節ネットワークの特性評価に適し、他のグラム陽性菌、病原性であろうと確実にトランスポーズ可能な改変プロトコルについて説明する。
特に注意が細胞のライシスステップに支払われました。フランスのプレスは、機械的な細胞のライシスの器械によって置き換えられた。この方法は、グラム陽性細胞を破壊し、病原性株の取り扱いに関連するリスクを制限するのに有効です。MS2親和性精製の歩留まりを改善するため、アミロース樹脂上に固定化されたMS2-MBPの量が大幅に増加している。これは、ストリンジェンシーとワッシュの数を調整する必要があります。
初期プロトコルとは異なり、MAPSは2つの生物学的複製から重複してここで行われる。統計分析を実装するワークフローが開発されました (図 2) は、得られたデータの堅牢性を明確に高めます。
MS2 コンストラクトとその式
このMAPSプロトコルは、ブドウ球菌の標的体を同定するために確立された。MS2-sRNA構築物は、低コピー数プラスミド(pCN51、20〜25コピー/細胞)および定足数センシング依存P3プロモーターの制御下で表され、主に定常期に誘発される。この発現パターンは 、S.アウレウス(すなわちRsaA、RsaIおよびRsaC)における研究されたsRNAに対応し、かなり強力なMS2-sRNA合成を保証する。しかし、他の場合には、P3プロモーターが適切でない可能性があり、sRNAが誘導されたときに細菌の自然な生理学的状態を反映していないことを排除することはできません。MS2-sRNA産生を制御するもう一つの方法は、化学的に誘導可能なプロモーター(例えば、テトラサイクリン誘導性プロモーター)を使用することです。これらのツールはMS2-sRNAコンストラクトのパルス発現を可能にしますが、この設定はRNAターゲットが同時に発現することを保証するものではありません。もう一つの欠点は、プラスミドからの発現が、高いコピー数ベクターでさらに強調された、研究されたsRNAの過剰産生につながる可能性があるということです。これは、潜在的にアルテフの実際の相互作用または関連するRNAシャペロン機能の破壊をもたらす可能性があります。最も適切な代替は、内因性sRNA遺伝子の5'末端にMS2タグを挿入することです。したがって、MS2-sRNAは染色体にコードされ、その天然プロモーターの制御下にあるであろう。これは、より良い研究sRNAの内因性発現を模倣し、研究されたsRNAの過剰産生によるバイアスを避けるべきである。いずれの場合も、重要なステップは、MS2-sRNA構築物の長さ、安定性および機能性を慎重に検証するものであり、特に内因性sRNAの産生および機能を引き起こす条件下で成長した培養物から抽出された全RNAを用いたノーザンブロットアッセイを使用することです。間違いなく、不適切/機能しないMS2-sRNA構築物の使用は、生成された結果に劇的に影響を与え、上記のコントロールの重要性を支持する可能性があります。最後に、MS2-sRNAは、常にΔsRNA バックグラウンドで産生され、mRNA標的の濃縮を最大化します。さらに、RNasesの特異的変異(例えば、欠失変異体または温度感受性変異体)を有する宿主株において実験を行い、RNasesのsRNA依存的なリクルートによるmRNA標的の分解を回避することができる。
MAPSによって特定された候補者の実験的検証はまだ必要です
考慮する必要がある1つのポイントは、MAPSが推定RNA標的のリストを提供することです。しかし、一部のRNAは、共有mRNA標的またはRNA結合タンパク質との相互作用を介して間接的に濃縮され得る。その結果、明らかにされた候補は、他の実験的アプローチによって確認されなければならない。EMSAおよびフットプリント実験は、一般的に、インビトロで推定RNA標的へのsRNAの直接結合を可視化するために使用される。次に、インビトロのトープリンティングアッセイは、sRNAがそのmRNA標的の翻訳開始に及ぼす影響を監視するのに役立ちます。最後に、ノーザンブロットおよび/または遺伝子レポーターアッセイ(lacZ または gfp)は、この実験的検証をin vivoで補完する。これらのアプローチはすべて、Jagodnikら(2017)39に記載されています。
RIL-seq および CLASH テクノロジとは異なり、MAPS は対話サイトに関する情報を直接提供しません。それにもかかわらず、マッピングされた読み取りのピークは、RNA標的の制限領域(例えば 、glyW-cysT-leuZ オペロン21の3'末端)において頻繁に観察され、ペアリング部位の同定を容易にする。あるいは、いくつかの予測ツールを使用して、IntaRNA40などの同定された標的上の推定結合部位を予測することができる。
MAPS は特定の sRNA の標的を精査します。
MAPS技術は、 大腸菌 および S. チフィムリウムなどのグラム陰性細菌におけるsRNA標的物の研究に適しているが、現在では S.アウレウスのようなグラム陽性細菌におけるこの改変プロトコルでもよい。基本的に、MAPSは、特定の時間および特定の成長条件で形成されたRNA:sRNA二重鎖のスナップショットを提供します。残念ながら、mRNA標的のリストは不完全である可能性があります。例えば、上記の予防措置を正当化する不適切な実験条件のために、同系のmRNA標的を見逃す可能性がある。
RIL-seqやCLASHなどの他の一般的に使用される方法と比較して、MAPSは特定のsRNAを使用して、相互作用するすべてのRNA標的を共精製します。ここで、sRNA:RNA相互作用は他のRBP関連デュプレックスの中で希釈されず、理論的には標的の特徴をより深く特徴付けることを可能にする。しかし、RIL-seq/CLASHおよびMAPS法は、推定sRNA標的12,41の異なるセットを明らかにし、これらの実験的アプローチが相補的であることを示唆しているようだ。この不一致は、各方法によって生成される成長条件および/またはバイアスの変動によって説明することができます。
したがって、研究の目的は、方法の選択に影響を与える必要があります。sRNA標的のグローバル分析や、RBPがsRNA媒介制御に関与する場合、RIL-seqおよびCLASH法が好ましい。どちらのアプローチも、特定のRBPに依存する規制ネットワークの概要を提供し、その結果、多種多様なSRNAおよび関連するターゲットを明らかにする。反対に、特定のsRNAの研究のために、またはRBPが知られていないか関与していない場合、MAPSは適切なオプションを表す。
開示事項
著者らは開示するものは何もない。
謝辞
この作品は、「アジェンス・ナショナル・デ・ラ・レシェルシュ」(ANR、グラントANR-16-CE11-0007-01、RIBOSTAPH、およびANR-18-CE12-002-002-04、CoNoCo、PR)によってサポートされました。また、将来のプログラムへの投資の一環としてANRが管理する国家からの資金として、LabEx NetRNA ANR-10-LABX-0036およびANR-17-EURE-0023(PRへ)の枠組みの下で出版されています。DLは、マリー・スウォトフスカ・キュリー補助金第753137-SaRNARegの下で欧州連合のHorizon 2020研究・イノベーションプログラムによって支援されました。E.マッセラボでの作業は、カナダ保健研究所(CIHR)、カナダ自然科学工学研究評議会(NSERC)、国立衛生研究所NIHチームグラントR01 GM092830-06A1からの助成金を運営することによって支援されています。
資料
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tube | Sarstedt | 72.690.001 | |
| 15 mL centrifuge tubes | Falcon | 352070 | |
| 2 mL microcentrifuge tube | Starstedt | 72.691 | |
| 2100 Bioanalyzer Instrument | Agilent | G2939BA | RNA quantity and quality |
| 250 mL culture flask | Dominique Dutscher | 2515074 | Bacterial cultures |
| 50 mL centrifuge tubes | Falcon | 352051 | Culture centrifugation |
| Absolute ethanol | VWR Chemicals | 20821.321 | RNA extraction and purification |
| Allegra X-12R Centrifuge | Beckman Coulter | Bacterial pelleting | |
| Ampicilin (amp) | Sigma-Aldrich | A9518-5G | Growth medium |
| Amylose resin | New England BioLabs | E8021S | MS2-affinity purification |
| Anti-dioxigenin AP Fab fragment | Sigma Aldrich | 11093274910 | Northern blot assays |
| Autoradiography cassette | ThermoFisher Scientific | 50-212-726 | Northern blot assays |
| BamHI | ThermoFisher Scientific | ER0051 | Plasmid construction |
| BHI (Brain Heart Infusion) Broth | Sigma-Aldrich | 53286 | Growth medium |
| Blocking reagent | Sigma Aldrich | 11096176001 | Northern blot assays |
| CDP-Star | Sigma Aldrich | 11759051001 | Northern blot assays (substrate) |
| Centrifuge 5415 R | Eppendorf | RNA extraction and purification | |
| Chloroform | Dominique Dutscher | 508320-CER | RNA extraction and purification |
| DIG-RNA labelling mix | Sigma-Aldrich | 11277073910 | Northern blot assays |
| DNase I | Roche | 4716728001 | DNase treatment |
| Erythromycin (ery) | Sigma-Aldrich | Fluka 45673 | Growth medium |
| FastPrep device | MP Biomedicals | 116004500 | Mechanical lysis |
| Guanidium Thiocyanate | Sigma-Aldrich | G9277-250G | Northern blot assays |
| Hybridization Hoven Hybrigene | Techne | FHB4DD | Northern blot assays |
| Hybridization tubes | Techne | FHB16 | Northern blot assays |
| Isoamyl alcohol | Fisher Scientific | A/6960/08 | RNA extraction and purification |
| LB (Lysogeny Broth) | Sigma-Aldrich | L3022 | Growth medium |
| Lysing Matrix B Bulk | MP Biomedicals | 6540-428 | Mechanical lysis |
| MicroPulser Electroporator | BioRad | 1652100 | Plasmid construction |
| Milli-Q water device | Millipore | Z00QSV0WW | Ultrapure water |
| NanoDrop spectrophotometer | ThermoFisher Scientific | RNA/DNA quantity and quality | |
| Nitrocellulose membrane | Dominique Dutsher | 10600002 | Northern blot assays |
| Phembact Neutre | PHEM Technologies | BAC03-5-11205 | Cleaning and decontamination |
| Phenol | Carl Roth | 38.2 | RNA extraction and purification |
| Phusion High-Fidelity DNA Polymerase | New England Biolabs | M0530 | Plasmid construction |
| pMBP-MS2 | Addgene | 65104 | MS2-MBP production |
| Poly-Prep chromatography column | BioRad | 7311550 | MS2-affinity purification |
| PstI | ThermoFisher Scientific | ER0615 | Plasmid construction |
| Qubit 3 Fluorometer | Invitrogen | 15387293 | RNA quantity |
| RNAPro Solution | MP Biomedicals | 6055050 | Mechanical lysis |
| ScriptSeq Complete Kit | Illumina | BB1224 | Preparation of cDNA librairies |
| Spectrophotometer Genesys 20 | ThermoFisher Scientific | 11972278 | Bacterial cultures |
| SpeedVac Savant vacuum device | ThermoFisher Scientific | DNA120 | RNA extraction and purification |
| Stratalinker UV Crosslinker 1800 | Stratagene | 400672 | Northern blot assays |
| T4 DNA ligase | ThermoFisher Scientific | EL0014 | Plasmid construction |
| TBE (Tris-Borate-EDTA) | Euromedex | ET020-C | Northern blot assays |
| ThermalCycler T100 | BioRad | 1861096 | Plasmid construction |
| Tween 20 | Sigma Aldrich | P9416-100ML | Northern blot assays |
| X-ray film processor | hu.q | HQ-350XT | Northern blot assays |
| X-ray films Super RX-N | FujiFilm | 4741019318 | Northern blot assays |
参考文献
- Carrier, M. C., Lalaouna, D., Masse, E. Broadening the Definition of Bacterial Small RNAs: Characteristics and Mechanisms of Action. Annual Review of Microbiology. 72, 141-161 (2018).
- Hör, J., Matera, G., Vogel, J., Gottesman, S., Storz, G. Trans-Acting Small RNAs and Their Effects on Gene Expression in Escherichia coli and Salmonella enterica. EcoSal Plus. 9 (1), (2020).
- Desgranges, E., Marzi, S., Moreau, K., Romby, P., Caldelari, I. Noncoding RNA. Microbiology Spectrum. 7 (2), (2019).
- Adams, P. P., Storz, G. Prevalence of small base-pairing RNAs derived from diverse genomic loci. Biochimica et Biophysica Acta - Gene Regulatory Mechanisms. 1863 (7), 194524 (2020).
- Pain, A., et al. An assessment of bacterial small RNA target prediction programs. RNA Biology. 12 (5), 509-513 (2015).
- Desgranges, E., Caldelari, I., Marzi, S., Lalaouna, D. Navigation through the twists and turns of RNA sequencing technologies: Application to bacterial regulatory RNAs. Biochimica et Biophysica Acta - Gene Regulatory Mechanisms. , 194506 (2020).
- Wright, P. R., et al. CopraRNA and IntaRNA: predicting small RNA targets, networks and interaction domains. Nucleic Acids Research. 42, 119-123 (2014).
- Smirnov, A., Schneider, C., Hor, J., Vogel, J. Discovery of new RNA classes and global RNA-binding proteins. Current Opinion in Microbiology. 39, 152-160 (2017).
- Saliba, A. E., Santos, S., Vogel, J. New RNA-seq approaches for the study of bacterial pathogens. Current Opinion in Microbiology. 35, 78-87 (2017).
- Melamed, S., Adams, P. P., Zhang, A., Zhang, H., Storz, G. RNA-RNA Interactomes of ProQ and Hfq Reveal Overlapping and Competing Roles. Molecular Cell. 77 (2), 411-425 (2020).
- Melamed, S., et al. Global Mapping of Small RNA-Target Interactions in Bacteria. Molecular Cell. 63 (5), 884-897 (2016).
- Iosub, I. A., et al. Hfq CLASH uncovers sRNA-target interaction networks linked to nutrient availability adaptation. Elife. 9, (2020).
- Waters, S. A., et al. Small RNA interactome of pathogenic E. revealed through crosslinking of RNase E. The EMBO Journal. 36 (3), 374-387 (2017).
- Dos Santos, R. F., Arraiano, C. M., Andrade, J. M. New molecular interactions broaden the functions of the RNA chaperone Hfq. Current Genetics. , (2019).
- Kavita, K., de Mets, F., Gottesman, S. New aspects of RNA-based regulation by Hfq and its partner sRNAs. Current Opinion in Microbiology. 42, 53-61 (2018).
- Bohn, C., Rigoulay, C., Bouloc, P. No detectable effect of RNA-binding protein Hfq absence in Staphylococcus aureus. BMC Microbiology. 7, 10 (2007).
- Jousselin, A., Metzinger, L., Felden, B. On the facultative requirement of the bacterial RNA chaperone, Hfq. Trends in Microbiology. 17 (9), 399-405 (2009).
- Olejniczak, M., Storz, G. ProQ/FinO-domain proteins: another ubiquitous family of RNA matchmakers. Molecular Microbiology. 104 (6), 905-915 (2017).
- Lalaouna, D., Desgranges, E., Caldelari, I., Marzi, S. Chapter Sixteen - MS2-Affinity Purification Coupled With RNA Sequencing Approach in the Human Pathogen Staphylococcus aureus. Methods in Enzymology. 612, 393-411 (2018).
- Lalaouna, D., Prevost, K., Eyraud, A., Masse, E. Identification of unknown RNA partners using MAPS. Methods. 117, 28-34 (2017).
- Lalaouna, D., et al. A 3' external transcribed spacer in a tRNA transcript acts as a sponge for small RNAs to prevent transcriptional noise. Molecular Cell. 58 (3), 393-405 (2015).
- Lalaouna, D., Morissette, A., Carrier, M. C., Masse, E. DsrA regulatory RNA represses both hns and rbsD mRNAs through distinct mechanisms in Escherichia coli. Molecular Microbiology. 98 (2), 357-369 (2015).
- Lalaouna, D., Prevost, K., Laliberte, G., Houe, V., Masse, E. Contrasting silencing mechanisms of the same target mRNA by two regulatory RNAs in Escherichia coli. Nucleic Acids Research. 46 (5), 2600-2612 (2018).
- Lalaouna, D., Eyraud, A., Devinck, A., Prevost, K., Masse, E. GcvB small RNA uses two distinct seed regions to regulate an extensive targetome. Molecular Microbiology. 111 (2), 473-486 (2019).
- Silva, I. J., et al. SraL sRNA interaction regulates the terminator by preventing premature transcription termination of rho mRNA. Proceedings of the National Academy of Sciences. 116 (8), 3042-3051 (2019).
- Lalaouna, D., Masse, E. Identification of sRNA interacting with a transcript of interest using MS2-affinity purification coupled with RNA sequencing (MAPS) technology. Genomics Data. 5, 136-138 (2015).
- Tomasini, A., et al. The RNA targetome of Staphylococcus aureus non-coding RNA RsaA: impact on cell surface properties and defense mechanisms. Nucleic Acids Research. 45 (11), 6746-6760 (2017).
- Bronesky, D., et al. A multifaceted small RNA modulates gene expression upon glucose limitation in Staphylococcus aureus. The EMBO Journal. 38 (6), (2019).
- Lalaouna, D., et al. RsaC sRNA modulates the oxidative stress response of Staphylococcus aureus during manganese starvation. Nucleic Acids Research. 47 (1), 9871-9887 (2019).
- Carrier, M. C., Laliberte, G., Masse, E. Identification of New Bacterial Small RNA Targets Using MS2 Affinity Purification Coupled to RNA Sequencing. Methods in Molecular Biology. 1737, 77-88 (2018).
- Garibyan, L., Avashia, N. Polymerase chain reaction. Journal of Investigative Dermatology. 133 (3), 1-4 (2013).
- Revie, D., Smith, D. W., Yee, T. W. Kinetic analysis for optimization of DNA ligation reactions. Nucleic Acids Research. 16 (21), 10301-10321 (1988).
- Seidman, C. E., Struhl, K., Sheen, J., Jessen, T. Introduction of plasmid DNA into cells. Current Protocols in Molecular Biology. , (2001).
- Sanger, F., Coulson, A. R., Barrell, B. G., Smith, A. J. H., Roe, B. A. Cloning in single-stranded bacteriophage as an aid to rapid DNA sequencing. Journal of Molecular Biology. 143 (2), 161-178 (1980).
- Grosser, M. R., Richardson, A. R. Method for Preparation and Electroporation of S. aureus and S. epidermidis. Methods in Molecular Biology. 1373, 51-57 (2016).
- Krumlauf, R. Northern blot analysis. Methods in Molecular Biology. 58, 113-128 (1996).
- Koontz, L. Agarose gel electrophoresis. Methods in Enzymology. 529, 35-45 (2013).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2016 update. Nucleic Acids Reseaerch. 44, 3-10 (2016).
- Jagodnik, J., Brosse, A., Le Lam, T. N., Chiaruttini, C., Guillier, M. Mechanistic study of base-pairing small regulatory RNAs in bacteria. Methods. 117, 67-76 (2017).
- Mann, M., Wright, P. R., Backofen, R. IntaRNA 2.0: enhanced and customizable prediction of RNA-RNA interactions. Nucleic Acids Res. 45, 435-439 (2017).
- Georg, J., et al. The power of cooperation: Experimental and computational approaches in the functional characterization of bacterial sRNAs. Molecular Microbiology. 113 (3), 603-612 (2020).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved