Method Article
Yakalama ve tıklama Kimya destekli RNA-interactome kullanarak RNA bağlanıcı proteinler tanımlaması (CARIC) strateji yakalama
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
Her iki kodlama ve kodlamayan bağlayıcı proteinler tanımlamak için tıklatın Kimya destekli RNA-interactome yakalama (CARIC) strateji uygulamak için detaylı bir protokol RNA'ların sunulur.
Özet
RNA bağlanıcı proteinler (RBPs) kapsamlı bir tanımlaması hücrelerdeki posttranscriptional düzenleyici ağ anlama anahtarıdır. RBP yakalama için yaygın olarak kullanılan bir strateji non-poly(A) RNA'ların çoğu proteinler tanımlanamayan bırakarak ökaryotik olgun mRNA üzerinde çoğunlukla oluşan Poliadenilasyon [Poli(a)] hedef RNA'ların, patlatır. Burada bir son zamanlarda bildirilen yönteminin metabolik etiketlerine göre birleştirerek poli(a) ve non-poly(A) RBPs transcriptome çapında yakalama sağlayan tıklama Kimya destekli RNA-interactome yakalama (CARIC) olarak adlandırdığı ayrıntılı yordamlar açıklar RNA'ların, in vivo UV cross-linking ve bioorthogonal etiketleme.
Giriş
İnsan genomu çeşitli mRNA'ların, rRNA'lar, tRNA da dahil olmak üzere kodlama ve kodlamayan RNA'ların (ncRNA'lar), küçük nükleer RNA'ların (snRNA), küçük genelde RNA'ların (snoRNAs) ve uzun kodlamayan RNA'ların (lncRNAs)1sentezlenir. Bu RNA'ların çoğunu RBPs giyim sahip ve Ribonükleoprotein parçacıklar (RNPs)2olarak işlev. Bu nedenle, RBPs kapsamlı bir tanımlaması çeşitli insan hastalıkları3,4,5' te karıştığı düzenleyici ağ arasında RNA'ların ve RBPs, anlamak için bir önkoşuldur.
Son birkaç yıldır insan7,8,9,10,11de dahil olmak üzere çeşitli ökaryotik sistemleri2,6' da keşfedilen RBPs büyük bir destek tanık olduk, fare12,13,14, Maya9,15,16, zebra balığı17, Drosophila melanogaster18,19 , Caenorhabditis elegans16, Arabidopsis thaliana20,21,22ve insan parazitleri23,24,25 . Bu gelişmeler tarafından Castello vd tarafından geliştirilen bir RBP yakalama stratejisi kolaylaştırdı 7 ve Baltz vd. vivo UV RNA ve onun etkileşen proteinler, poli(a) RNA'ların oligo(dT) yakalanması ve kütle spektrometresi (MS) cross-linking birleştiren 8 2012 yılında,-proteomik profil oluşturma dayalı. Ancak, bu poli(a) çoğunlukla sadece ~ %3 - %526ökaryotik transcriptome hesaba, olgun mRNA üzerinde bulunmaktadır gerçeği göz önüne alındığında bu yaygın olarak kullanılan strateji non-poly(A) RNA'ların çoğu ncRNA'lar de dahil olmak üzere, ile etkileşim RBPs esir alma yeteneğine sahip değildir ve pre-mRNA'ların.
Burada, son zamanlarda geliştirilen strateji poli(a) ve non-poly(A) RBPs27transcriptome çapında yakalanması için ayrıntılı yordamlar raporu. CARIC olarak adlandırılan, bu strateji vivo içinde UV cross-linking ve metabolik RNA'lar (tıklayın tepki olarak katılabilir bir bioorthogonal fonksiyonel grup içeren) photoactivatable ve "tıklanabilir" nükleozit analogları ile etiketleme birleştiren 4 - thiouridine (4SU) ve 5-ethynyluridine (AB). CARIC stratejisi ile ideal sonuçlar elde etmek için anahtar olan verimli metabolik etiketleme, UV cross-linking ve tıklayın tepki ve RNA bütünlük bakımından adımlardır. Tıklayın tepki katalizör olarak kullanılan Cu(I) RNA'ların parçalanma neden olabileceğinden, RNA parçalanma azaltabilir bir Cu(I) ligand esastır. Biz verimli tıklayın reaksiyonlar şiddetli RNA bozunması neden olmadan cep lysates gerçekleştirmek açıklar.
RBP yakalamak ve kimlik HeLa hücreleri içinde sadece bu protokol için açıklanan, ancak çeşitli hücre tiplerinin ve muhtemelen canlı organizmalar CARIC strateji uygulanabilir. RBP yakalama yanı sıra, bu protokolü de MS numune hazırlama ve protein tanımlama ve o kim proteomik deneyler ile aşina olmayan için yararlı olabilir miktar için aerodinamik adım adım yordamlar sağlar.
Protokol
Dikkat: uygulanabilir olduğunda kullanılan reaktifler RNase free şeklinde satın alınan veya çözünmüş RNase-serbest, çözücüler (çoğu durumda dietil pyrocarbonate (DEPC) için-tedavi su). RNA örnekleri ve Kimyasalları RNase free ele alırken, her zaman eldiven ve maske ve sık sık RNase kirlenmesini önlemek için onları değiştirmek.
1. hazırlanması, Lysate metabolik olarak etiketli ve UV çapraz bağlı hücre
-
AB ve 4SU metabolik birleşme
- Kültür HeLa hücreleri içinde Dulbecco'nın modifiye kartal Orta (% 10 fetal Sığır serum (FBS), 100 U/mL penisilin ve % 5 CO2 atmosferde 37 ° C'de 100 μg/mL streptomisin ile takıma DMEM). Deneysel bir hazırlamak için ~ 4 x 107 HeLa hücreleri (15 cm tabağı) kültür veya örnek için bir Standart MS çalıştırmak denetleyebilirsiniz.
- Kültürlü HeLa hücreleri ~ %80 izdiham ulaştığınızda, kültür orta kaldırmak ve prewarmed taze orta 15 cm çanak başına 15 mL ekleyin.
- (PBS içinde çözünmüş) 100 mM 4SU 0,5 mM için son bir konsantrasyon için fincan başına 7,5 μL ekleyip 100 mM (1 mM son bir konsantrasyon fosfat tamponlu tuz (PBS) çözünmüş) EU fincan başına 15 μL deneysel ve noUV-kontrol örnekleri. 100 mM (PBS içinde çözünmüş) EU fincan başına 15 μL 1 mM no4SU-kontrol örnekleri için son bir konsantrasyon ekleyin.
Not: 4SU fotoğraf alınabilen; Böylece, 4SU ekledikten sonra ışık koruma gereklidir. - Bulaşıkları folyo ile kapak ve 16 h. Ekle için hücreleri AB ve 4SU veya yalnızca adıma 1.1.3 deneysel, noUV- ve no4SU-kontrol örnekleri, AB'den miktarı yarısı sırasıyla kültür ve için başka bir 2 h kültür devam.
-
Vivo UV cross-linking
- Kültür orta kaldırmak için hücreleri 3 yıkama x 5 mL de PBS çanak başına ve Kaldır kalan PBS mümkün olduğu kadar ile.
Not: Kalıntıları sıvı cross-linking verimliliği önemli ölçüde azaltır. - İçin deneysel ve no4SU-kontrol örnekleri, bulaşıkları buza kaldırıldı kapaklı yerleştirin ve 365 nm UV ışığı 2 J/cm2 UV cross-linker tarafından hücrelerle ışınlatayım.
- NoUV-kontrol örnekleri, bulaşıkları buza koyun ve onları ışıktan korumak.
Not: NoUV-kontrol örnekleri için aşağıdaki adımların tümünü karanlık bir odada yapılmalıdır.
- Kültür orta kaldırmak için hücreleri 3 yıkama x 5 mL de PBS çanak başına ve Kaldır kalan PBS mümkün olduğu kadar ile.
-
Hücre lizis ve homojenizasyon
- Yemek öncesi lizis arabelleği başına 1 mL ekleyin (10 mM Tris∙HCl, pH 7.5, 50 mM LiCl, %0,02 Nonidet P-40 ve ethylenediaminetetraacetic asit (EDTA)-ücretsiz proteaz inhibitörü kokteyl) hücreleri. Kauçuk hücre lifter kullanarak hücreleri scrape ve 15 mL Tüp öncesi lizis süspansiyon toplamak.
Not: Bu adımı hücre zarı kırmak ve çözünür sitoplazmik protein ve RNA'ları bırakın. Yapmak değil tüp santrifüj kapasitesi ve süpernatant kaldırın. - İki 15 cm yemeklerin süspansiyon için 6 mL birime öncesi lizis arabelleği ekleyerek ayarlayın. Öncesi lizis süspansiyon için R-lysis arabellek (200 mM Tris∙HCl, pH 7.5, 500 mM LiCl, % 2 lityum Lauryl Sülfat [LDS]) eşit bir hacmi ekleyin.
- Lysate hücre lysate açık ve homojen olana bir şırınga ile dar bir iğneyle (27-G) birkaç kez geçen lunaparkçı. Lysate 4 ° c hafif döndürme (~ 15 dönüş/dak) 1 h için kuluçkaya.
Not: Bu son adım tam proteinlerin denaturing izin verir. Lysate güvenli bir şekilde-70 ° C'de en fazla bir ay için saklanır.
- Yemek öncesi lizis arabelleği başına 1 mL ekleyin (10 mM Tris∙HCl, pH 7.5, 50 mM LiCl, %0,02 Nonidet P-40 ve ethylenediaminetetraacetic asit (EDTA)-ücretsiz proteaz inhibitörü kokteyl) hücreleri. Kauçuk hücre lifter kullanarak hücreleri scrape ve 15 mL Tüp öncesi lizis süspansiyon toplamak.
-
Tıklayın tepki için hazırlık
- Lysate 20 birimleri seyreltme arabelleği (50 mM Tris∙HCl, pH 7.5) ekleyerek sulandırmak ve 15 mL kesirler bölmek.
Not: tuz ve deterjan yüksek konsantrasyon içeren çözümler Cu (I) verimliliğini uzlaşma olacak-katalize tıklayın tepki; Bu nedenle, arabellek lysate değiştirilmesi gerekir. - Her kesir birim 1 mL küçük olana 15 mL Ultrafiltrasyon tüp (ile molekül ağırlığı kesim, 10 kDa) kullanarak konsantre. Bir sallanan-kova rotor Ultrafiltrasyon tüp 4.000 x g 4 ° c ~ 15 dk de döndürmek için kullanın.
- Seyreltme arabellek 14 mL konsantre lysate kesir ekleyin ve 1.4.2 arasındaki adımları yineleyin. Kesirler birleştirir ve onları ultrafiltrasyon (4.000 x g ~ 15 dk 4 ° C'de) tarafından 6 mL bir birime konsantre.
Not: lysate için tıklayın tepki hazır bu yüzden tuz ve LDS şimdi çoğu kaldırılması. Lysate-70 ° C'de bir hafta kadar saklanabilir. Önemli RNA düşmesine neden, çünkü birden çok donma-çözülme çevrimleri, kaçının. Aliquot lysate küçük ölçekli karakterizasyonu gerekiyorsa.
- Lysate 20 birimleri seyreltme arabelleği (50 mM Tris∙HCl, pH 7.5) ekleyerek sulandırmak ve 15 mL kesirler bölmek.
2. RNA-interactome için örnek hazırlanması yakalama
-
Lysate preclearing
- Lysate 6 mL başına 100-μL streptavidin manyetik boncuklar ekleyin ve yavaşça doğal olarak biotinylated proteinler ortadan kaldırmak için oda sıcaklığında 30 dakika (~ 15 dönüş/dak) döndürün.
- Bir mıknatıs (~ 20 min 4 ° C'de için) ve transfer precleared için yeni bir tüp lysate kullanarak boncuk cips.
-
Performans tıklayın tepki
- Reaksiyon karışımı hazırlayın: biotin stok (100 mM azid-dimetil sülfoksit [DMSO], 100 mikron son bir konsantrasyon içinde çözünmüş biotin), 6.5 μL bakır stokunun 3.25 μL (taze yap; 1 M CuSO4 500 mikron son bir konsantrasyon, suda çözünmüş) , 65 μL ligand stok (200 mM 2 mM son bir konsantrasyon, suda çözünmüş THPTA) ve H2o 262.75 μL
Not: THPTA Tris [(1-hydroxypropyl-1H-1,2,3-triazol-4-yl) metil] Amin için duruyor. - Reaksiyon mix 6 mL için precleared lysate ekleyin ve iyice karıştırın. Sonra reaktif azaltmak 162.5 μL ekleyin (taze; olun 40 mg/mL sodyum askorbat son bir konsantrasyon, 5 mM) için lysate ve iyice karıştırın. Son hacim 6.5 mL olmalıdır.
- Reaksiyon karışımı bir orbital çalkalayıcı (800 devir/dakika) üzerinde oda sıcaklığında 2 h için kuluçkaya. 5 mM EDTA tepki karışıma ekleyin ve bu reaksiyon gidermek 5 min için kuluçkaya.
- Reaksiyon karışımı hazırlayın: biotin stok (100 mM azid-dimetil sülfoksit [DMSO], 100 mikron son bir konsantrasyon içinde çözünmüş biotin), 6.5 μL bakır stokunun 3.25 μL (taze yap; 1 M CuSO4 500 mikron son bir konsantrasyon, suda çözünmüş) , 65 μL ligand stok (200 mM 2 mM son bir konsantrasyon, suda çözünmüş THPTA) ve H2o 262.75 μL
-
Küçük ölçekli karakterizasyonu
- Boya stok (Örneğin, 100 mM azid-Cy5 DMSO içinde çözünmüş) yerine adım 2.2.1 biotin hisse senedi ile olduğu gibi tepki karışımı hazırlayın.
Not: Reaktif tutarı lysate hacmi göre ayarlanmalıdır. Genellikle, bir 20-μL aliquot lysate, karakterizasyonu bir jel floresan çözümlemesi gibi için yeterli. - Reaksiyon karıştırmak için lysate ekleyin ve oda sıcaklığında 2 h için kuluçkaya. Sonra LDS örnek arabellek (4 x) hacmi üçte birini ekleyin, 5 min için 55 ° C'de tabiatını ve örnek bir % 10 bis-Tris jel üzerinde çözmek.
Not: floresan sinyal RNA'ların üzerinde sunulan onaylamak için tıklayın tepki sonra RNase A sindirim ile denetimler içerir.
- Boya stok (Örneğin, 100 mM azid-Cy5 DMSO içinde çözünmüş) yerine adım 2.2.1 biotin hisse senedi ile olduğu gibi tepki karışımı hazırlayın.
-
Reaksiyon karışımı temizlik
- Prechilled metanol (% 100) sekiz cilt yüzeyi tepki karışıma ekleyin ve yağış için-30 ° C'de 30 dk için kuluçkaya. Yağış 50 mL konik santrifüj tüplerde gerçekleştirin.
Not: toplam hacmi 50 mL büyükse, iki 50 mL konik santrifüj tüpler içine tepki karışımı bölün. - Sulandırma arabellek hazırlamak: bir birim (%4 Sodyum Lauryl Sülfat [SDS] ve 10 mM EDTA) arabelleği A sekiz birimlerle arabelleği B (%1 Brij-97, 150 mM NaCl ve 50 mM Trietanolamin, pH 7,4) birleştirmek.
- 4000 x g 4 ° C'de 15 dakika, santrifüj kapasitesi ve süpernatant atın. ~ 1-2 mL prechilled metanol için Pelet ekleyin. Yukarı ve aşağı pipet Pelet kırmaya ve Pelet tamamen yok görünür parçalar ile askıya alınır emin olun. Tüp prechilled metanol ile doldurun. 2 x bu adımı yineleyin.
- 4000 x g 4 ° C'de 15 dakika, santrifüj kapasitesi ve süpernatant atın. Tüpler ve 5 dakika süreyle santrifüj 4.000 x g de tekrar dikkatle kalan metanol mümkün olduğunca Pelet bozmadan çizmek tekrar koy.
- 10 mL sulandırma arabellek Pelet için ekleyin. Yukarı ve aşağı Pelet çözülmeye pipet. 4 ° C'de 10 dakika için 4.000 x g , santrifüj
- Süpernatant için yeni bir tüp aktarın. 20 μL örnek kalite kontrol (bakınız Bölüm 4) için toplamak.
Not: Şimdi, örnek RNA-interactome yakalamak için hazırdır. Sulandırılan örnek-70 ° C'de bir hafta kadar saklanabilir.
- Prechilled metanol (% 100) sekiz cilt yüzeyi tepki karışıma ekleyin ve yağış için-30 ° C'de 30 dk için kuluçkaya. Yağış 50 mL konik santrifüj tüplerde gerçekleştirin.
3. RNA-interactome yakalama
-
Streptavidin-özel boncuk hazırlanması
- Streptavidin-özel Bulamaç Sulandırılan örnek 10 mL (kapatılan boncuk 800 μL) 1600 μL 15 mL konik santrifüj tüpüne götür.
- 5 dk. dikkatle için çıkarmak süpernatant kapatılan boncuk bozmadan 4000 x g de boncuk aşağı spin.
- Boncuk 10 mL 50 mM Tris∙HCl (pH 7.5) ile yıkayın. Spin aşağı boncuk (4.000 x g 5 min için) ve süpernatant kaldırın. 2 x bu adımı yineleyin.
-
Benzeşme açılan
- Temizlenmiş ve birleştirilmiş örnek--dan adım 2.4.6 (bkz. Adım 3.1) streptavidin-özel boncuk aktarın. Gecede 4 ° C'de nazik döndürme kuluçkaya
-
Çamaşır streptavidin boncuk
- Spin aşağı boncuk (4.000 x g 5 min için) ve süpernatant için yeni bir tüp aktarın. 20 μL örnek kalite kontrol için toplamak.
- Boncuk yıkama arabellek (%2 SDS) PBS, pH 7.4 içinde 10 mL ile yıkayın. Nazik döndürme (~ 12 dönüş/dak) Oda sıcaklığında 10 dakika için kuluçkaya. Spin aşağı boncuk (4.000 x g 5 min için) ve süpernatant kaldırın. 1 x tekrarlayın.
- 3.3.2 yıkama arabelleği B ile (8 M üre ve suda 250 mM NH4HCO3 ) arasındaki adımları yineleyin. 3.3.2 yıkama arabelleği C ile adımı yineleyin (PBS, pH 7,4 2,5 M NaCl). Sonra boncuk 10 mL 50 mM Tris∙HCl (pH 7.5) ile yıkayın. Spin aşağı boncuk (4.000 x g 5 min için) ve süpernatant kaldırın.
- Boncuk eşit olarak bölünmüş ve iki 1.5 mL microcentrifuge tüpler için aktarabilirsiniz.
-
Yakalanan RNPs elüsyon
- Biotin elüsyon arabellek hazırlamak: 12,5 mM biotin, 75 mM NaCl, 7.5 mM Tris∙HCl (pH 7.5), 1.5 mM EDTA, % 0.15 SDS, %0.075 sarkosyl ve % 0.02 sodyum deoxycholate.
Not: Mağaza arabellek biotin için oda sıcaklığında 4 ° C'de çökelti - İçin 400 μL yıkanmış kapatılan boncuk biotin elüsyon arabelleği 400 μL ekleyin.
- Onları bir orbital çalkalayıcı (1500 devir/dakika) Oda sıcaklığında 20 dakika için üzerinde kuluçkaya. Sonra bir ısı bloklu (1500 devir/dakika, 65 ° C) bir orbital çalkalayıcı boncuk (7800 x g için 1dk) aşağı 10 dk. Spin üzerinde kuluçkaya ve eluted RNP toplamak.
- Boncuk için taze biotin elüsyon arabelleği 400 μL ekleyin ve 3.4.3. Birleştirme iki bir 15 mL tüp içine elutes.
- Biotin elüsyon arabellek hazırlamak: 12,5 mM biotin, 75 mM NaCl, 7.5 mM Tris∙HCl (pH 7.5), 1.5 mM EDTA, % 0.15 SDS, %0.075 sarkosyl ve % 0.02 sodyum deoxycholate.
-
RNase sindirim
- Üç birim seyreltme arabelleği SDS konsantrasyonu azalmaya eluted RNP ekleyin. 0.5 mL Ultrafiltrasyon tüp kullanarak seyreltilmiş örnek konsantre (12.000 x g 4 ° c ~ 30 min için de 10 kDa; bir molekül ağırlığı kesim ile spin) ~ 40 μL için.
- 0.5 μg/μL RNase A ekleyin ve 2 h 37 ° C'de RBPs çapraz bağlı RNA'ların üzerinden serbest bırakmak için kuluçkaya. RBPs 2 μL kalite kontrol (bakınız Bölüm 4) için toplamak.
4. kalite kontrol
-
Benzeşme açılan verimliliğini kontrolünü
- 10 μL kimden adım 2.4.6 ve 10 μL adım 3.3.1 "sonra-açılan" örnek "önce-açılan" örnek al.
- Standart western blot yordamları (% 10 bis-Tris jel) kullanarak örnekleri analiz.
- Polivinilidin florid (PVDF) membran kalıntı biotin sinyalleri "sonra-açılan" örneği izlemek için streptavidin-HRP eşlenik ile leke.
Not: biotin sinyal "sonra-açılan" örnek "önce-açılan" örnek sinyal tek beşinci büyükse streptavidin-özel boncuk 3.1.1 adımda kullanılan miktarını artırın.
-
Toplam yakalama verimliliği kontrolünü
- Adım 3.5.2 ve 0,5 μL "önce-açılan" örneği yayımlanmış RBP örnekten 2 μL al (% 0,1 giriş olarak) 2.4.6 kimden adım.
- Standart gümüş boyama yordamları kullanarak örnekleri analiz.
- Jel için 20 dk Sensitizasyonu (13 mM Na2S2O3, 83 mM sodyum asetat, % 30 etanol) 30 dk ardından fiksasyon arabellek (% 40 etanol, % 10 asetik asit) ile düzeltmek.
- Yıkama jeli 3 x 5 min için su ile ve daha sonra jel 2 ile 15 mM AgNO3 çözüm 20 dk. yıkama leke x 1 dk, su ile 0,24 M Na2CO3 ve %0.012 formaldehit geliştirmek ve boyama suffici olduğunda 45 mM EDTA ile bitirmek ENT.
Not: Yakalanan RBPs gümüş boyama yoğunluğu % 0,1 giriş için benzer olmalıdır.
5. MS için örnek hazırlanması
-
Jel tripsin hazım yakalanan RBPs 28
- SDS örnek arabellek (5 x) bir dördüncü hacmi yayımlanan RBP örnekleri 3.5.2 adımından ekleyin. Örnek 10 min için 95 ° C'de denatüre.
- 1.5 mm % 10 SDS-polyacrylamide jel üzerinde RBPs çözmek.
- Gümüş, aşağıdaki standart protokolleri ile jel leke.
- Lane deneysel örnek veya denetimi örneğini yığın jel ile ve bir (~ 15 kDa) kaldırıldı RNase büyük grubun tüketim.
- Eksize lane (~ 1-1.5 mm x ~ 1-1.5 mm) küçük parçalar halinde kesin.
Not: Jel parça kısa kenarına pipet ipuçlarında tıkanma Hayır 1 mm daha kısa olması gerekir. - Microcentrifuge tüp jel parçaları aktarmak ve destaining arabellek (100 mM Na2S2O3 eşit miktarda karışımı ve 30 mM K3[Fe(CN)6]) ile destain.
- Jel adet tamamen renksiz kadar jel adet 200 mM Amonyum Bikarbonat (ABC) ile yıkayın.
- Temiz Asetonitril (ACN) 1 ml jel parçalarını kurutmak. (50 mM içinde ABC çözünmüş) 10 mM dithiothreitol 200 μL ile suyla temasa ve 45 dk 56 ° C'de kuluçkaya.
Not: Tamamen susuz jel parçalar çok sert ve opak olmalıdır. Jel adet dehidratasyon sonra hala yumuşak ACN kaldırın ve tekrar dehydrate düzgün ACN 1 mL ekleyin. - Oda sıcaklığında jel parçalara sakin. (50 mM içinde ABC çözünmüş) 58 mM iodoacetamide 200 μL ekleyin ve 45 dakika içinde belgili tanımlık karanlık oda sıcaklığında kuluçkaya.
- Su ile kısa yıkama sonra düzgün ACN 1 ml jel parçalarını kurutmak.
Not: Jel adet tamamen susuz gerekir. - Jel parçalar (50 mM içinde ABC çözünmüş) 10 ng/μL tripsin uygun miktarda suyla temasa ve 12-16 h 37 ° C'de kuluçkaya.
Not: Jel adet tamamen opak hiçbir çekirdek sayesinde rehydrated. Herhangi bir aşırı sıvı kaldırın.
-
Kararlı izotop dimetil sindirilir peptidler etiketleme 29
- 200 μL ayıklama arabelleği ekleyerek jel parçalarıyla sindirilir peptidler ayıklamak (%5 formik asit ve % 50 su ACN) ve vortexing (vasıl 1200 devir/dakika) ile 30 dk 37 ° C'de kuluçkaya.
- 5.2.1 adımı yineleyin 2 x. Bir microcentrifuge tüp içine özleri birleştirin.
- Ayıklanan peptidler vakum Santrifüjü tarafından kuru.
- 100 mM triethylammonium bikarbonat (TEAB, pH 8,5) 200 μL peptidler sulandırmak.
Dikkat: Adımları 5.2.4'ten - 5.2.6 duman başlıklı buz üzerinde yapılmalıdır. - 8 μL % 4 CH2O ve 8 μL %4 13CD2O deneysel ve kontrol örnekleri, sırasıyla ekleyin.
Not: istikrarlı izotopik etiketleme önyargı kontrol etmek için kararlı izotop takas deneysel ve kontrol diğer biyolojik olarak bağımsız Çoğaltma örnekleri. - 0,6 M NaBH3CN 8 μL ekleyin (taze olun) ve iyice karıştırın.
- Örnekleri, oda sıcaklığında vortexing ile 1 h için kuluçkaya.
- Buz üzerinde örnekleri sakin. Reaksiyon % 1 amonyak sulu çözüm 32 μL ekleyerek gidermek. O zaman, daha fazla tepki formik asitin 16 μL ekleyerek gidermek.
- Karşılık gelen denetim örneği ile deneysel örnek bir microcentrifuge tüp içine birleştirmek. Örnekleri tarafından vakum Santrifüjü kuru.
-
Dimetil etiketli peptidler ayırma
- Dur ve git çıkarma ipuçları (StageTips)30hazırlayın.
- C18 membran bir genişletilmiş uzunlukta, 10-μL ucu yerleştirin.
- Yüksek pH C18 boncuk ACN içinde ucuna kadar askıya 300 μg ekleyin.
- İpucu dik bir microcentrifuge tüp ile hangi ucunu stabilize ve alt ihbar Asansör ev yapımı bir raf yerleştirin.
- 1400 x g 2 dk. atma için ucunda akışı aracılığıyla spin.
- %80 50 μL ucuyla yıkama ACN 10 mm ABC (pH 10.0). 1 x tekrarlayın.
Not: 10 mM ABC çözüm pH % 28 amonyum hidroksit ekleyerek ayarlayın. - %50 50 μL ucuyla yıkama ACN 10 mm ABC (pH 10.0). 1 x tekrarlayın.
- 10 mm ABC (pH 10.0) 50 μL ucuyla yıkayın. 1 x tekrarlayın.
- 10 mm ABC (pH 10.0) 50 μL peptidler sulandırmak.
- Sulandırılan örnek için hazırlanan ipucu ekleyin. Akış yoluyla verimli peptid bağlama sağlamak için belgili tanımlık uç için yeniden yükleyin.
- 10 mm ABC (pH 10.0) 50 μL ucuyla yıkayın. 1 x tekrarlayın.
- % 6, % 9, % 12, % 15, % 18, % 21, % 25, % 30, % 35, %40, % 80 ve % 6, 50 μL ile 12 kesirler için kademeli peptid elute ACN 10 mm ABC (pH 10.0).
- İki kesirler eşit zaman aralığı ile birleştirmek (Kesir 1 ile 7, 8, 2 ve benzeri) altı almak için kesirler kombine.
- Örnekleri tarafından vakum Santrifüjü kuru. Kurutulmuş peptidler-30 ° C'de depolanan
- Dur ve git çıkarma ipuçları (StageTips)30hazırlayın.
6. MS ve veri analizi performansını
-
Sıvı Kromatografi-tandem kütle spektrometresi ile peptid analiz
- Kurutulmuş peptid kesirler--dan adım 15 5.3.7 μL %0,1 formik asit içeren su sulandırmak. Sulandırılan peptidler pH çözüm (pH 3 altında olmalıdır) bir pH şerit üzerinde tespit 1 μL ile kontrol edin.
- Yeniden oluşturma örnek sıvı kromatografi (LC) sütun enjekte. Çözücü (solvent A su %0,1 formik asit, çözücü B içeren %0,1 formik asit içeren ACN olduğunu) uygun bir degrade yüksek performanslı sıvı kromatografi (HPLC) uygulayın. Çözücü B tipik bir degrade aşağıdaki gibidir: %5 - %35 40 min; %35 - %70 4 dk içinde; ve düzenlenen %75 10 dk için.
- Electrospray tarafından eluted peptidler iyonize ve kütle spektrometre veri bağımlı modunda çalışır. 15 en bol iyonları seçin (çarpma ücret: 2 +, 3 + veya daha yüksek) ilk MS inceden inceye gözden geçirmek için bir tandem kütle spektrometresi (MS/MS) tarama (ayrılma, çarpışma kaynaklı CID). 500 25 maksimum süresi zamanla dinamik dışlama boyutunu ayarlamak s.
-
Protein tanımlama ve MaxQuant kullanarak miktar 31
- Protein kimlik yanlış bulma oranı (FDR) 0,01 ayarlamak ve doğruluğu ve güvenilirliği artırmak için 2'ye benzersiz peptidler sayısını ayarlayın.
- Küme en az gerekli oranı sayar (benzersiz + jilet gibi) protein miktar 2 ve olanaklı kılmak yeniden ölçmek için ve çalışmaları arasında maç işlevleri için.
-
Zenginleştirme önemi değerlendirme R/Bioconductor paket limma kullanma 32
- Denetlenen tgerçekleştirmek-limma Log2 katlı değişikliğine en az üç biyolojik çoğaltır sıfırdan karşı test etmek için uygulanan test. Veri tablosu okumak için read.table işlevini kullanın. Daha sonra lmFit ve eBayes işlevleri için uygun verileri kullanın. (Ortalama Log2 katlı değişim ve P değerleri de dahil olmak üzere) hesaplama sonuçları vermek için topTable işlevini kullanın.
- Benjamini – Hochberg yöntemiyle FDR denetlemek için P değerleri düzeltin.
- Bir FDR 0.01 proteinler önemli ölçüde deneysel örneklerinde zenginleştirilmiş bir listesini oluşturmak için geçerlidir. İki veya üç aşamalı değişim bir kesim daha ayrıntılı denetlemek için yanlış pozitif ayarla.
Sonuçlar
Kalite kontrol adımları temsilcisi sonuçları sunulmuştur. Sonuçlar 2.3.2 (Şekil 1). adımda anlatılan jel floresan analizi, 4.1.3 (Şekil 2A). adımda anlatılan western blot Analizi ve gümüş boyama analiz 4.2.2 (Şekil 2B) adımda açıklanan rakamlar içerir. Kalite kontrol adımları CARIC protokolleri optimizasyonu için kritik öneme sahiptir. Her zaman büyük ölçekli RBP tanımlama deneyleri hazırlanmasında kalite denetimleri içerir.
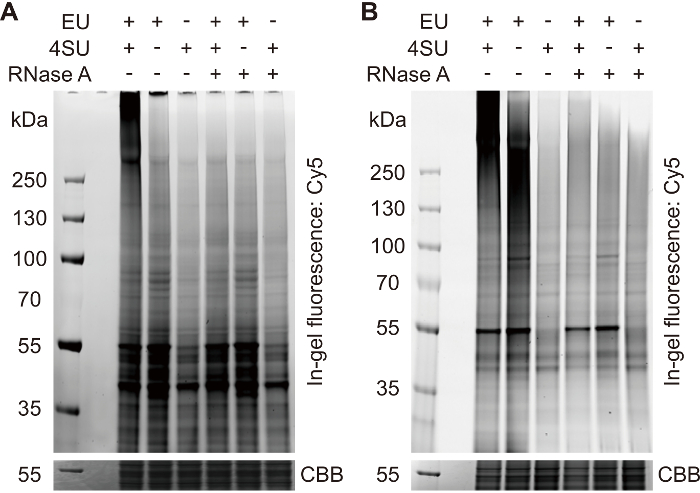
Şekil 1: jel floresans analiz 2.3.2. adımda anlatılan tıklayın etiketli örnekleri. (a) Bu paneli gösterir bir tipik olarak-jel floresans desen örnekleri tıklayın etiketli. Sadece iki kat etiketli örnek bir yüksek moleküler çapraz bağlı RNPs sinyal temsil eden kiloda (> 250 kDa), güçlü smear grubu gösterir. RNP sinyal kaldırılması için 4SU veya AB veya Özet RNase A. ile atlayın Arka plan keskin bantları daha düşük moleküler ağırlıklı, non-spesifik proteinler etiketli sinyalleri temsil eder. (B) bazı durumlarda güçlü bir lekeli grup (~ 130-250 kDa) no4SU-kontrol örneğinde görülebilir. Bu grup için çoğu zaman ısı denatürasyon sırasında bozulmuş etiketli uncross-bağlantılı RNA'lar sinyal temsil eder. Sonraki yordamlar ile müdahale değil. CBB Coomassie = parlak mavi. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
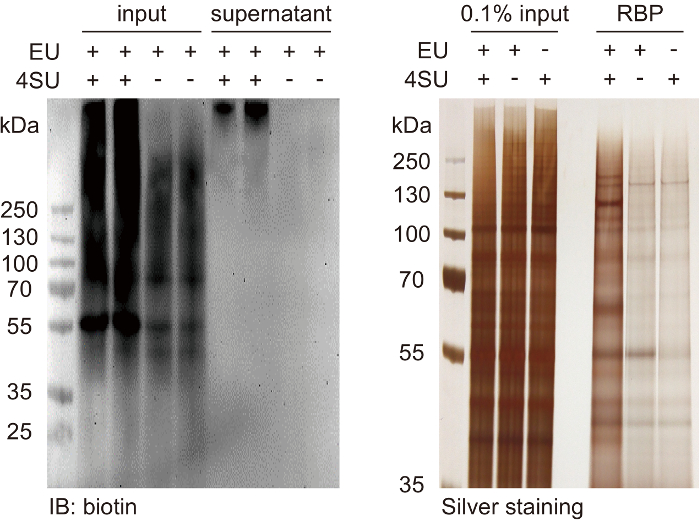
Şekil 2: kalite kontrol benzeşme açılan verimlilik ve yakalanan RBPs. Biotin bir western blot Analizi örnekleri daha önce açılan (giriş) ve örnekleri sonra açılan (süpernatant) sinyalleri (A) Bu panel gösterir. Kalan sinyalleri oranını tahmin ve 3.1.1. adımda kullanılan boncuk tutar en iyi duruma getirme. (B) Bu panel yakalanan RBPs % 0.1 giriş toplam protein karşılaştırıldığında gümüş boyama analizini gösterir. HeLa hücreleri için genel toplam yakalama verimliliği ~0.05% - giriş proteinlerin % 0,1 olduğunu. Bu değer, farklı hücre tipleri metabolik etiketleme verimliliğini farkı nedeniyle önemli ölçüde değişebilir. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.

Şekil 3: CARIC sonuçlarını temsilcisi MS. Ortalama görüntüleyen bir yanardağ Arsa Log2 katlı değiştirmek ve P değerleri quantified proteinlerin limma paket tarafından hesaplanan düzeltilmiş (A) Bu panel gösterir. > 2 Log2 katlı değişimine ve ayarlanmış bir P değeri < 0.01 proteinlerin 597 "CARIC RBPs" sınıflandırıldı. (B) Bu panel örtüşme ile önceden tanımlanan insan poli(a) RBPs7,8CARIC proteinlerin,9,10,11gösterir. CARIC RBPs diğer RBPs kodlama daha olası iken örtüşen proteinler çoğunlukla RBPs, kodlama. Bu rakam daha önce yayımlanmış çalışma izni Ulusal Bilimler Akademisi27ile olan bir yazdırılan değil. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
Tartışmalar
Adil RNA bütünlük bakımından başarılı CARIC deneyler anahtarlarına biridir. Kısmi düşme gözlendi rağmen ile uygun ligandlar Cu(I) ve dikkatli kullanım özellikleri ile RNA bozulması önemli ölçüde, azalır. AB ve deneysel örneklerinde 4SU değiştirme oranları ve % 1,18 %0,46, sırasıyla vardır (veri gösterilmez). 2.000 uzunluğu ile bozulmamış RNA'ların için nt, RNA'ların ~ %90 içeren en az bir AB ve bir 4SU. 1.000 uzunluğu ile kısmen bozulmuş RNA'ların için nt, RNA'ların ~ %70 içeren en az bir AB ve bir 4SU. Bu nedenle, şiddetli bozunması kabul edilebilir değildir, ancak kısmi bozulma RNA'ların, CARIC, verimliliğini önemli ölçüde azalmaz.
Başka bir kritik adım adım 1.4, tıklayın tepki için hazırlık olduğunu. Cu (I)-RNA'ların katalize tıklayın tepki LDS konsantrasyon için hassas. LDS yüksek yoğunlukta (> %0,1) etiketleme EU içeren RNA'ların sinyalleri ve arka plan sinyalleri proteinler (veri gösterilmez) üzerinde bir artış bir azalma sağlayacaktır.
AB ek olarak, CARIC da burda ve azido analogları adenozin33,34,35,36gibi diğer tıklanabilir nükleozit ile uyumludur. Ancak, CARIC uygulanması önemli ölçüde ilgi biyolojik bir sistem içinde doğal olmayan tıklanabilir nükleozitler metabolik verimliliği ile sınırlıdır. CARIC dışındaki koşullar kullanarak performans gösterdi bu protokol için önce bu nedenle, her zaman metabolik etiketleme verimliliği (Örneğin, yanında floresan düşsel) kontrol edin.
Son zamanlarda, sadece AB toplam RNA'ların etiketlemek için birleştirmek ve RNA ve proteinler bölünmelerini için 254-nm UV kullanır, RICK (tıklayın kimya kullanarak yeni transkripsiyonu RNA interactome yakalama), olarak adlandırılan bir strateji bildirilen37oldu. Özellikle, 254-nm UV tüm dört doğal nükleozitler yanı sıra EU etkinleştirebilirsiniz. Böylece, 254-nm UV ışınlama ücretsiz AB ve onun metabolitleri (Örneğin, EU fosfatlar) olası yanlış pozitif olarak dikkate alınması gereken karşılık gelen bağlayıcı proteinler ile çapraz.
Bir ilginç CARIC RBPs olan RNA'ların çoğunlukla sigara poliadenile çoğu bakteride tanımlamak için kullanılır. RBPs büyük ölçekli tanımlaması bakteri38posttranscriptional yönetmeliğinde moleküler temeli anlamak için çok değerli kaynakları sağlayacaktır.
Açıklamalar
Yazarlar ifşa gerek yok.
Teşekkürler
Bu eser Ulusal Doğal Bilim Vakfı, Çin hibe 91753206, 21425204 ve 21521003 ve ulusal anahtar araştırma ve geliştirme projesi 2016YFA0501500 tarafından desteklenmektedir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| HeLa | ATCC | ||
| DMEM (Dulbecco's Modified Eagle Medium) | Thermo Fisher Scientific | 11995065 | |
| FBS (Fetal Bovine Serum) | Thermo Fisher Scientific | 10099141 | |
| Penicillin & Streptomycin | Thermo Fisher Scientific | 15140122 | |
| EU (5-ethynyl uridine) | Wuhu Huaren Co. | CAS:69075-42-9 | |
| 4SU (4-thiouridine) | Sigma Aldrich | T4509 | |
| 10×PBS (Phosphate-Buffered Saline) | Thermo Fisher Scientific | AM9625 | |
| UV cross-linker | UVP | CL-1000 | Equiped with 365-nm UV lamp |
| DEPC (Diethyl pyrocarbonate) | Sigma Aldrich | D5758 | To treat water. Highly toxic! |
| Tris·HCl, pH 7.5 | Thermo Fisher Scientific | 15567027 | |
| LiCl | Sigma Aldrich | 62476 | |
| Nonidet P-40 | Biodee | 74385 | |
| EDTA-free protease inhibitor cocktail | Thermo Fisher Scientific | 88265 | One tablet for 50 mL lysis buffer. |
| LDS (Lithium dodecyl sulfate) | Sigma Aldrich | L9781 | |
| 15-mL ultrafiltration tube (10 kDa cutoff) | Millipore | UFC901024 | |
| 0.5-mL ultrafiltration tube (10 kDa cutoff) | Millipore | UFC501096 | |
| Streptavidin magnetic beads | Thermo Fisher Scientific | 88816 | |
| DMSO (Dimethyl sulfoxide) | Sigma Aldrich | 41639 | |
| Azide-biotin | Click Chemistry Tools | AZ104 | |
| Copper(II) sulfate | Sigma Aldrich | C1297 | |
| THPTA [Tris(3-hydroxypropyltriazolylmethyl)amine] | Sigma Aldrich | 762342 | |
| Sodium ascorbate | Sigma Aldrich | 11140 | |
| Azide-Cy5 | Click Chemistry Tools | AZ118 | |
| LDS sample buffer (4×) | Thermo Fisher Scientific | NP0008 | |
| 10% bis-Tris gel | Thermo Fisher Scientific | NP0301BOX | |
| EDTA | Thermo Fisher Scientific | AM9260G | |
| RNase A | Sigma Aldrich | R6513 | |
| SDS (Sodium dodecyl sulfate) | Thermo Fisher Scientific | 15525017 | |
| NaCl | Sigma Aldrich | S3014 | |
| Brij-97 [Polyoxyethylene (20) oleyl ether] | J&K | 315442 | |
| Triethanolamine | Sigma Aldrich | V900257 | |
| Streptavidin agarose | Thermo Fisher Scientific | 20353 | |
| Urea | Sigma Aldrich | U5378 | |
| Sarkosyl (N-Lauroylsarcosine sodium salt) | Sigma Aldrich | 61743 | |
| Biotin | Sigma Aldrich | B4501 | |
| Sodium deoxycholate | Sigma Aldrich | 30970 | |
| MaxQuant | Version: 1.5.5.1 |
Referanslar
- Djebali, S., et al. Landscape of transcription in human cells. Nature. 489 (7414), 101-108 (2012).
- Gerstberger, S., Hafner, M., Tuschl, T. A census of human RNA-binding proteins. Nature Reviews Genetics. 15 (12), 829-845 (2014).
- Castello, A., Fischer, B., Hentze, M. W., Preiss, T. RNA-binding proteins in Mendelian disease. Trends in Genetics. 29 (5), 318-327 (2013).
- Nussbacher, J. K., Batra, R., Lagier-Tourenne, C., Yeo, G. W. RNA-binding proteins in neurodegeneration: Seq and you shall receive. Trends in Neuroscience. 38 (4), 226-236 (2015).
- Jazurek, M., Ciesiolka, A., Starega-Roslan, J., Bilinska, K., Krzyzosiak, W. J. Identifying proteins that bind to specific RNAs - focus on simple repeat expansion diseases. Nucleic Acids Research. 44 (19), 9050-9070 (2016).
- Hentze, M. W., Castello, A., Schwarzl, T., Preiss, T. A brave new world of RNA-binding proteins. Nature Reviews Molecular Cell Biology. 19 (5), 327-341 (2018).
- Castello, A., et al. Insights into RNA biology from an atlas of mammalian mRNA-binding proteins. Cell. 149 (6), 1393-1406 (2012).
- Baltz, A. G., et al. The mRNA-bound proteome and its global occupancy profile on protein-coding transcripts. Molecular Cell. 46 (5), 674-690 (2012).
- Beckmann, B. M., et al. The RNA-binding proteomes from yeast to man harbour conserved enigmRBPs. Nature Communications. 6, 10127-10135 (2015).
- Conrad, T., et al. Serial interactome capture of the human cell nucleus. Nature Communications. 7, 11212-11222 (2016).
- Castello, A., et al. Comprehensive identification of RNA-binding domains in human cells. Molecular Cell. 63 (4), 696-710 (2016).
- Kwon, S. C., et al. The RNA-binding protein repertoire of embryonic stem cells. Nature Structural & Molecular Biology. 20 (9), 1122-1130 (2013).
- Liepelt, A., et al. Identification of RNA-binding proteins in macrophages by interactome capture. Molecular & Cellular Proteomics. 15 (8), 2699-2714 (2016).
- Liao, Y., et al. The cardiomyocyte RNA-binding proteome: Links to intermediary metabolism and heart disease. Cell Reports. 16 (5), 1456-1469 (2016).
- Mitchell, S. F., Jain, S., She, M. P., Parker, R. Global analysis of yeast mRNPs. Nature Structural & Molecular Biology. 20 (1), 127-133 (2013).
- Matia-González, A. M., Laing, E. E., Gerber, A. P. Conserved mRNA-binding proteomes in eukaryotic organisms. Nature Structural & Molecular Biology. 22 (12), 1027-1033 (2015).
- Despic, V., et al. Dynamic RNA-protein interactions underlie the zebrafish maternal-to-zygotic transition. Genome Research. 27 (7), 1184-1194 (2017).
- Wessels, H. H., et al. The mRNA-bound proteome of the early fly embryo. Genome Research. 26 (7), 1000-1009 (2016).
- Sysoev, V. O., et al. Global changes of the RNA-bound proteome during the maternal-to-zygotic transition in Drosophila. Nature Communications. 7, 12128(2016).
- Reichel, M., et al. In planta determination of the mRNA-binding proteome of Arabidopsis etiolated seedlings. Plant Cell. 28 (10), 2435-2452 (2016).
- Marondedze, C., Thomas, L., Serrano, N. L., Lilley, K. S., Gehring, C. The RNA-binding protein repertoire of Arabidopsis thaliana. Scientific Reports. 6, 29766-29778 (2016).
- Zhang, Z., et al. UV crosslinked mRNA-binding proteins captured from leaf mesophyll protoplasts. Plant Methods. 12, 42-53 (2016).
- Bunnik, E. M., et al. The mRNA-bound proteome of the human malaria parasite Plasmodium falciparum. Genome Biology. 17, 147-164 (2016).
- Lueong, S., Merce, C., Fischer, B., Hoheisel, J. D., Erben, E. D. Gene expression regulatory networks in Trypanosoma brucei: insights into the role of the mRNA-binding proteome. Molecular Microbiology. 100 (3), 457-471 (2016).
- Nandan, D., et al. Comprehensive identification of mRNA-binding proteins of Leishmania donovani by interactome capture. PLoS ONE. 12 (1), e0170068(2017).
- Jankowsky, E., Harris, M. E. Specificity and nonspecificity in RNA-protein interactions. Nature Reviews Molecular Cell Biology. 16 (9), 533-544 (2015).
- Huang, R., Han, M., Meng, L., Chen, X. Transcriptome-wide discovery of coding and noncoding RNA-binding proteins. Proceedings of the National Academy of Sciences of the United States of America. 115 (17), E3879-E3887 (2018).
- Shevchenko, A., Tomas, H., Havlis, J., Olsen, J. V., Mann, M. In-gel digestion for mass spectrometric characterization of proteins and proteomes. Nature Protocols. 1 (6), 2856-2860 (2006).
- Boersema, P. J., Raijmakers, R., Lemeer, S., Mohammed, S., Heck, A. J. R. Multiplex peptide stable isotope dimethyl labeling for quantitative proteomics. Nature Protocols. 4 (4), 484-494 (2009).
- Rappsilber, J., Mann, M., Ishihama, Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics using StageTips. Nature Protocols. 2 (8), 1896-1906 (2007).
- Cox, J., Mann, M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nature Biotechnology. 26 (12), 1367-1372 (2008).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), e47(2015).
- Grammel, M., Hang, H., Conrad, N. K. Chemical reporters for monitoring RNA synthesis and poly(A) tail dynamics. ChemBioChem. 13 (8), 1112-1115 (2012).
- Curanovic, D., et al. Global profiling of stimulus-induced polyadenylation in cells using a poly(A) trap. Nature Chemical Biology. 9 (11), 671-673 (2013).
- Zheng, Y. X., Beal, P. A. Synthesis and evaluation of an alkyne-modified ATP analog for enzymatic incorporation into RNA. Bioorganic & Medicinal Chemistry Letters. 26 (7), 1799-1802 (2016).
- Nainar, S., et al. Metabolic incorporation of azide functionality into cellular RNA. ChemBioChem. 17 (22), 2149-2152 (2016).
- Bao, X., et al. Capturing the interactome of newly transcribed RNA. Nature Methods. 15 (3), 213-220 (2018).
- Holmqvist, E., Vogel, J. RNA-binding proteins in bacteria. Nature Reviews Microbiology. , Published online (2018).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır