Method Article
Cattura e identificazione delle proteine RNA-leganti utilizzando clic chimica-assistita RNA-Interactoma (Castaldi) strategia di acquisizione
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Un protocollo dettagliato per l'applicazione di cattura (CARIC) del RNA-Interactoma di chimica-assistita clicca strategia per identificare proteine leganti alla codificazione di entrambi e non codificanti RNA è presentato.
Abstract
Una completa identificazione di RNA-binding proteins (RBPs) è la chiave per comprendere la rete di regolamentazione post-trascrizionale in cellule. Una strategia ampiamente utilizzata per la cattura RBP sfrutta la poliadenilazione [poli (a)] di RNA bersaglio, che si verifica principalmente su mRNA maturo eucariotico, lasciando la maggior parte delle proteine leganti di non-poly(A) RNA non identificato. Qui descriviamo le procedure dettagliate di un metodo recentemente segnalato chiamato click chimica-assistita RNA-Interactoma acquisire (CARIC), che consente di acquisire tutto il trascrittoma RBPs sia poli (a) e non-poly(A) combinando l'etichettatura metabolica del RNA, in vivo UV cross-linking e bioorthogonal tagging.
Introduzione
Il genoma umano è trascritto in vari tipi di RNA codificanti e non codificanti (ncRNAs), tra cui mRNA, rRNA, tRNA, piccole molecole di RNA nucleare (snRNAs), piccole molecole di RNA nucleolare (snoRNAs) e lungo non-codificazione RNAs (lncRNAs)1. La maggior parte di questi RNA possiedono abbigliamento di RBPs e funzionare come ribonucleoproteina particelle (RNP)2. Pertanto, una completa identificazione di RBPs è un prerequisito per la comprensione della rete regolamentare tra RNAs e RBPs, che è implicata in varie malattie umane3,4,5.
Ultimi anni hanno testimoniato una grande spinta di RBPs scoperto in vari sistemi eucariotici2,6, compresi umano7,8,9,10,11, del mouse12,13,14, lievito9,15,16, zebrafish17, Drosophila melanogaster18,19 , Caenorhabditis elegans16, Arabidopsis thaliana20,21,22e parassiti umani23,24,25 . Questi progressi hanno stati facilitati da una strategia di acquisizione RBP sviluppata da Castello et al. 7 e Baltz et al. 8 nel 2012, che combina in vivo UV cross-linking di RNA e di proteine interagenti, oligo (distacco) acquisizione di poli (a) RNAs e spettrometria di massa (MS)-base di analisi proteomica. Tuttavia, dato il fatto che poli (a) principalmente esiste il mRNA maturo, che rappresentano solo ~ 3% - 5% del trascrittoma eucariotiche26, questa strategia ampiamente utilizzata non è in grado di catturare RBPs interagendo con non-poly(A) RNAs, tra cui la maggior parte dei ncRNAs e pre-mRNA.
Qui, segnaliamo le procedure dettagliate di una strategia sviluppata di recente per la cattura di tutto il trascrittoma di poli (a) sia non-poly(A) RBPs27. Definito CARIC, questa strategia combina in vivo UV cross-linking ed etichettatura metabolica di RNA con fuse e "cliccabili" analoghi nucleosidici (che contengono un gruppo funzionale di bioorthogonal che possono partecipare clicca reazione), 4 - thiouridine (4SU) e 5-ethynyluridine (UE). Passaggi chiave per ottenere risultati ideali con la strategia di CARIC sono efficienti etichettatura metabolica, reazione di cross-linking e fare clic su UV e il mantenimento dell'integrità di RNA. Perché cu (i) usato come catalizzatore nella reazione di clic può causare la frammentazione degli RNA, un ligando di Cu (i) che può ridurre la frammentazione di RNA è essenziale. Descriviamo come eseguire clic efficienti reazioni in lisati cellulari senza causare grave degradazione del RNA.
Anche se catturare RBP e identificazione in cellule HeLa è descritto solo in questo protocollo, la strategia di CARIC può essere applicata a vari tipi di cellule e possibilmente agli organismi viventi. Oltre alla cattura RBP, questo protocollo fornisce anche razionalizzato le procedure dettagliate per la preparazione del campione di MS e proteina identificazione e quantificazione, che può essere utile per coloro che non hanno familiarità con gli esperimenti di proteomica.
Protocollo
Attenzione: Quando applicabile, i reattivi utilizzati devono essere acquistati sotto forma di RNAsi-libera, o disciolti in RNAsi-libera, solventi (per la maggior parte dei casi, in pirocarbonato dietilico (DEPC)-acqua trattata). Durante la manipolazione di campioni di RNA e reagenti RNAsi-libera, sempre indossare guanti e maschere e cambiarle frequentemente per evitare la contaminazione RNasi.
1. preparazione del lisato di metabolicamente etichettati e cellule reticolate UV
-
Metabolica incorporazione dell'UE e 4SU
- Cellule HeLa di cultura dell'Aquila per volta di Dulbecco Medium (DMEM) completati con 10% siero bovino fetale (FBS), 100 U/mL di penicillina e 100 μg/mL di streptomicina a 37 ° C in un'atmosfera di 5% CO2 . Coltura di ~ 4 x 10 cellule HeLa di7 (in due piatti di 15 cm) per la preparazione di uno sperimentale o campione di controllo per una che corsa standard MS.
- Quando le cellule HeLa coltivate raggiungono ~ 80% confluenza, rimuovere il terreno di coltura e aggiungere 15 mL di terreno nuovo preriscaldata per ogni piatto di 15 cm.
- Aggiungere 15 μL per ogni piatto di 100 mM EU (disciolto in tampone fosfato salino (PBS)) a una concentrazione finale di 1 mM e 7.5 μL per ogni piatto di 100mm 4SU (disciolto in PBS) ad una concentrazione finale di 0.5 mM per sperimentale e campioni di controllo noUV. Aggiungere 15 µ l per ogni piatto di 100 mM EU (disciolto in PBS) ad una concentrazione finale di 1 mM per campioni di no4SU-controllo.
Nota: 4SU è foto-activatable; Pertanto, è necessaria la protezione dalla luce dopo l'aggiunta di 4SU. - Coprire i piatti con un foglio e cultura le cellule per 16 h. Aggiungi metà dell'importo dell'UE e 4SU o solo UE dal passo 1.1.3 allo sperimentale, noUV- e campioni di no4SU-controllo, rispettivamente e continuano a coltura per un altro 2 h.
-
In vivo Reticolazione UV
- Rimuovere il terreno di coltura, lavare le cellule 3 x con 5 mL di PBS per ogni piatto e rimuovere residui PBS per quanto possibile.
Nota: È possibile che il liquido residuo ridurrà significativamente efficienza cross-linking. - Per sperimentale e campioni di no4SU-controllo, posizionare le stoviglie sul ghiaccio con il coperchio rimosso e irradiare le cellule con luce at 2 J/cm2 di un reticolante UV di UV di 365 nm.
- Per campioni di noUV-controllo, posizionare le stoviglie sul ghiaccio e proteggerli dalla luce.
Nota: Tutti i passaggi seguenti per campioni noUV-controllo dovrebbero essere eseguiti in una stanza buia.
- Rimuovere il terreno di coltura, lavare le cellule 3 x con 5 mL di PBS per ogni piatto e rimuovere residui PBS per quanto possibile.
-
Lisi cellulare e omogeneizzazione
- Aggiungere 1 mL per ogni piatto di tampone di pre-lisi (10 mM Tris∙HCl, pH 7.5, 50 mM acido LiCl, 0,02% Nonidet P-40 e acido etilendiamminotetraacetico (EDTA)-cocktail di inibitore di proteasi gratuito) alle cellule. Raschiare le celle utilizzando un sollevatore di cella di gomma e raccogliere la pre-Lisi sospensione in una provetta da 15mL.
Nota: Questo passaggio sarà rompere la membrana cellulare e rilasciare proteine citoplasmatiche solubile e RNAs. Non Centrifugare la provetta e rimuovere il surnatante. - Per la sospensione da due piatti di 15 cm, regolare il volume a 6 mL con l'aggiunta di tampone di pre lisi. Aggiungere alla sospensione pre-Lisi un uguale volume di tampone di lisi R (200 mM Tris∙HCl, pH 7.5, 500mm LiCl, 2% solfato dodecilico di litio [LDS]).
- Omogeneizzare il lisato cellulare facendolo passare attraverso una siringa con un ago sottile (27 G) diverse volte finché il lisato è chiaro e omogeneo. Incubare il lisato a 4 ° C con una delicata rotazione (~ 15 giri/min) per 1 h.
Nota: Quest'ultimo passaggio consentirà la completa denaturazione delle proteine. Il lisato possa essere memorizzato in modo sicuro a-70 ° C fino ad un mese.
- Aggiungere 1 mL per ogni piatto di tampone di pre-lisi (10 mM Tris∙HCl, pH 7.5, 50 mM acido LiCl, 0,02% Nonidet P-40 e acido etilendiamminotetraacetico (EDTA)-cocktail di inibitore di proteasi gratuito) alle cellule. Raschiare le celle utilizzando un sollevatore di cella di gomma e raccogliere la pre-Lisi sospensione in una provetta da 15mL.
-
Preparazione per la reazione di clic
- Diluire il lisato aggiungendo 20 volumi di tampone di diluizione (50mm Tris∙HCl, pH 7.5) e dividerlo in frazioni di 15 mL.
Nota: Soluzioni contenenti un'alta concentrazione di sale e detersivo compromette l'efficienza di Cu (I)-catalizzato reazione clic; così, il buffer del lisato deve essere cambiato. - Concentrare ogni frazione utilizzando un tubo di ultrafiltrazione 15 mL (con un limite di peso molecolare di 10 kDa) fino a che il volume è più piccolo di 1 mL. Utilizzare un rotore basculante per girare il tubo di ultrafiltrazione a 4.000 x g a 4 ° C per circa 15 min.
- Aggiungere 14 mL di tampone di diluizione per la frazione di lisato concentrata e ripetere il punto 1.4.2. Combinare le frazioni e concentrarli in un volume di 6 mL di ultrafiltrazione (4.000 x g a 4 ° C per circa 15 min).
Nota: la maggior parte del sale e LDS sarà ora essere rimosso, quindi lisato è pronto per la reazione di clic. Lisato può essere conservato a-70 ° C fino a una settimana. Evitare più cicli di gelo-disgelo, perché essi si tradurrà in una riduzione significativa del RNA. Aliquotare le caratterizzazioni lisate se su piccola scala sono necessari.
- Diluire il lisato aggiungendo 20 volumi di tampone di diluizione (50mm Tris∙HCl, pH 7.5) e dividerlo in frazioni di 15 mL.
2. preparazione dei campioni per RNA-Interactoma catturare
-
Preclearing del lisato
- Aggiungere 100 μL streptavidina branelli magnetici per 6 mL di lisato e ruotare delicatamente (~ 15 giri/min) per 30 min a temperatura ambiente per eliminare naturalmente proteine biotinilate.
- A pellet le perline usando un magnete (per ~ 20 min a 4 ° C) ed il trasferimento il pretrattati lisato ad un nuovo tubo.
-
Prestazioni della reazione clicca
- Preparare la miscela di reazione: 6,5 μL di stock di biotina (100mm azide-biotina disciolta in dimetilsolfossido [DMSO] ad una concentrazione finale di 100 μM), 3,25 μL di Stock in rame (fanno fresco; 1 M CuSO4 disciolto in acqua ad una concentrazione finale di 500 μM) , 65 μL di brodo ligando (200 mM THPTA disciolto in acqua ad una concentrazione finale di 2 mM) e 262.75 μL di H2O.
Nota: THPTA è acronimo di ammina di Tris [(1-hydroxypropyl-1H-1,2,3-triazol-4-yl) metil]. - Aggiungere il mix di reazione per 6ml di pretrattati lisato e mescolare bene. Quindi, aggiungere 162.5 μL di reagente di ridurre (fanno fresco; l'ascorbato di sodio 40 mg/mL ad una concentrazione finale 5 mM) MU e mescolare bene. Il volume finale deve essere 6,5 mL.
- Incubare la miscela di reazione per 2 h a temperatura ambiente su agitatore orbitale (800 giri). Aggiungere 5 mM EDTA per la miscela di reazione e incubare per 5 min placare la reazione.
- Preparare la miscela di reazione: 6,5 μL di stock di biotina (100mm azide-biotina disciolta in dimetilsolfossido [DMSO] ad una concentrazione finale di 100 μM), 3,25 μL di Stock in rame (fanno fresco; 1 M CuSO4 disciolto in acqua ad una concentrazione finale di 500 μM) , 65 μL di brodo ligando (200 mM THPTA disciolto in acqua ad una concentrazione finale di 2 mM) e 262.75 μL di H2O.
-
Caratterizzazioni su piccola scala
- Preparare la miscela di reazione come descritto al punto 2.2.1 con il brodo di biotina sostituito da Stock in tintura (per esempio, 100 mM azide-Cy5 dissolto in DMSO).
Nota: La quantità di reagente dovrebbe essere regolato secondo il volume di lisato. In genere, un'aliquota di 20 μL del lisato è abbastanza per caratterizzazioni come un'analisi di fluorescenza in gel. - Aggiungere il mix di reazione MU e incubare per 2 ore a temperatura ambiente. Quindi, aggiungere un terzo del volume del buffer del campione LDS (4x), si denaturano a 55 ° C per 5 min e risolvere il campione su un gel di bis-Tris di 10%.
Nota: Per confermare che il segnale di fluorescenza è presentato il RNAs, includono controlli con digestione di RNasi A dopo la reazione di clic.
- Preparare la miscela di reazione come descritto al punto 2.2.1 con il brodo di biotina sostituito da Stock in tintura (per esempio, 100 mM azide-Cy5 dissolto in DMSO).
-
Pulitura della miscela di reazione
- Aggiungere otto volumi di metanolo prechilled (100%) alla miscela di reazione estiguuto e Incubare 30 min a-30 ° C per precipitazione. Eseguire la precipitazione in tubi di centrifuga a fondo conico da 50 mL.
Nota: Se il volume totale è maggiore di 50 mL, dividere la miscela di reazione in due provette da centrifuga coniche da 50 mL. - Preparare il tampone di ricostituzione: combinare un volume di tampone A (solfato dodecilico di sodio 4% [SDS] e 10 mM EDTA) con otto volumi di tampone B (1% trietanolammina Brij-97, NaCl 150 mM e 50 mM, pH 7.4).
- Centrifugare a 4.000 x g per 15 min a 4 ° C e scartare il surnatante. Aggiungi ~ 1-2 mL di metanolo prechilled al pellet. Pipetta su e giù per rompere il pellet e assicurarsi che il pellet è completamente sospesa con nessun pezzi visibili. Riempire il tubo con metanolo prechilled. Ripetere questo passaggio x 2.
- Centrifugare a 4.000 x g per 15 min a 4 ° C e scartare il surnatante. Rimettere che i tubi e centrifugare nuovamente a 4.000 x g per 5 min con attenzione estrarre il metanolo residuo per quanto possibile senza disturbare il pellet.
- Aggiungere 10 mL di tampone di ricostituzione per il pellet. Pipettare su e giù per sciogliere il precipitato. Centrifuga a 4.000 x g per 10 min a 4 ° C.
- Trasferire il surnatante in una nuova provetta. Raccogliere 20 μL del campione per il controllo di qualità (Vedi sezione 4).
Nota: Ora, il campione è pronto per la cattura di RNA-Interactoma. Il campione ricostituito può essere conservato a-70 ° C fino a una settimana.
- Aggiungere otto volumi di metanolo prechilled (100%) alla miscela di reazione estiguuto e Incubare 30 min a-30 ° C per precipitazione. Eseguire la precipitazione in tubi di centrifuga a fondo conico da 50 mL.
3. RNA-Interactoma cattura
-
Preparazione dei branelli streptavidina-agarosio
- Prendere 1.600 μL di streptavidina-agarosio liquami (800 μL di perline depositati) per 10 mL di campione ricostituito in una provetta conica per centrifuga 15 mL.
- Rotazione verso il basso le perline a 4000 x g per 5 min. Rimuovere attentamente il supernatante senza disturbare le perline si stabilirono.
- Lavare le perle con 10 mL di 50 mM Tris∙HCl (pH 7.5). Rotazione verso il basso le perline (4.000 x g per 5 min) e rimuovere il surnatante. Ripetere questo passaggio x 2.
-
Affinità pulldown
- Trasferire il campione ripulito e ricostituito dal passaggio 2.4.6 ai talloni streptavidina-agarosio (Vedi punto 3.1). Incubare per una notte con delicata rotazione a 4 ° C.
-
Lavaggio dei branelli streptavidina
- Rotazione verso il basso le perline (4.000 x g per 5 min) e trasferire il surnatante in una nuova provetta. Raccogliere 20 μL del campione per il controllo qualità.
- Lavare le perle con 10 mL di tampone di lavaggio (una SDS di 2% in PBS, pH 7.4). Incubare per 10 min con delicata rotazione (~ 12 giri/min) a temperatura ambiente. Rotazione verso il basso le perline (4.000 x g per 5 min) e rimuovere il surnatante. Ripetere 1 x.
- Ripetere il passaggio 3.3.2 con tampone di lavaggio B (8m urea e 250mm NH4HCO3 disciolto in acqua). Ripetere il punto 3.3.2 con tampone di lavaggio C (2,5 M di NaCl in PBS, pH 7.4). Quindi, lavare le perle con 10 mL di 50 mM Tris∙HCl (pH 7.5). Rotazione verso il basso le perline (4.000 x g per 5 min) e rimuovere il surnatante.
- Dividere le perline in modo uniforme e trasferirli a due tubi per microcentrifuga da 1,5 mL.
-
Eluizione del RNP catturati
- Preparare il tampone di eluizione di biotina: la biotina di 12,5 mM, 75 mM NaCl, 7,5 mM Tris∙HCl (pH 7.5), 1,5 mM di EDTA, 0,15% SDS, sarkosyl 0,075% e 0,02% sodio desossicolato.
Nota: Archivio il tampone a temperatura ambiente per biotina può precipitare a 4 ° C. - A 400 μL di perline si stabilirono lavati, aggiungere 400 μL di tampone di eluizione di biotina.
- Incubare a loro su agitatore orbitale (1.500 giri/min) a temperatura ambiente per 20 min. Quindi, incubare su agitatore orbitale con un blocco di calore (1.500 giri/min, 65 ° C) per 10 min. Spin giù le perline (7.800 x g per 1 min) e raccogliere l'eluiti RNP.
- Ai talloni, aggiungere 400 μL di tampone di eluizione di biotina fresco e ripetere il punto 3.4.3. Combinare i due eluisce in una provetta 15 mL.
- Preparare il tampone di eluizione di biotina: la biotina di 12,5 mM, 75 mM NaCl, 7,5 mM Tris∙HCl (pH 7.5), 1,5 mM di EDTA, 0,15% SDS, sarkosyl 0,075% e 0,02% sodio desossicolato.
-
Digestione di RNAsi
- Aggiungere tre volumi di tampone di diluizione per il RNP eluiti per diminuire la concentrazione di SDS. Concentrare il campione diluito utilizzando un tubo di ultrafiltrazione di 0,5 mL (con un taglio di peso molecolare di 10 kDa; spin a 12.000 x g a 4 ° C per circa 30 min) a ~ 40 μL.
- Aggiungere 0,5 μg/μL RNasi A e incubare per 2 ore a 37 ° C per rilasciare RBPs da reticolato RNAs. Raccogliere 2 μL di RBPs per controllo di qualità (Vedi sezione 4).
4. controllo di qualità
-
Controllo dell'efficienza del menu a discesa di affinità
- Prendere 10 μL del campione "prima-pulldown" da passo 2.4.6 e 10 μL del campione "dopo-pulldown" dal punto 3.3.1.
- Analizzare i campioni utilizzando le procedure standard macchia occidentale (10% bis-Tris gel).
- Macchia la membrana di polivinilidene fluoruro (PVDF) con streptavidina-HRP coniugato per monitorare i segnali di biotina residuo del campione "dopo-pulldown".
Nota: Se il segnale di biotina del campione "dopo-pulldown" è maggiore di un quinto del segnale del campione "prima-pulldown", aumentare la quantità di perle di streptavidina-agarosio utilizzato nel passaggio 3.1.1.
-
Controllo dell'efficienza totale acquisizione
- Prendere 2 μL del campione RBP rilasciato da punto 3.5.2 e 0,5 μL del campione "prima-pulldown" (come 0,1% ingresso) da passo 2.4.6.
- Analizzare i campioni utilizzando procedure di macchiatura d'argento standard.
- Difficoltà il gel con un tampone di fissazione (40% di etanolo, acido acetico 10%) per 20 min seguita da sensibilizzazione (13 mM Na2S2O3, 83 mM sodio acetato, 30% etanolo) per 30 min.
- Lavare il gel 3 x con acqua per 5 minuti e, quindi, macchia con un 15 mM AgNO3 soluzione per 20 min. lavare il gel 2 x con acqua per 1 min, svilupparlo in 0.24 M Na2CO3 e 0.012% formaldeide e terminare con 45 mM EDTA, quando la colorazione è sufficiente ENT.
Nota: L'intensità di macchiatura d'argento del RBPs catturati dovrebbe essere simile a quella dell'input 0,1%.
5. preparazione dei campioni per MS
-
Digestione della tripsina del RBPs catturati in gel 28
- Aggiungere un quarto volume di tampone del campione SDS (5x) per i campioni RBP rilasciati dal punto 3.5.2. Denaturare il campione a 95 ° C per 10 min.
- Risolvere il RBPs su un gel di SDS-poliacrilammide 10% 1,5 mm.
- Macchia il gel con argento, seguenti protocolli standard.
- Asportare la corsia del campione sperimentale o controllo campione con gel d'impilamento e la band principale della RNasi A (~ 15 kDa) rimosso.
- Tagliare la corsia asportata in piccoli pezzi (~ 1-1.5 mm x ~ 1-1,5 mm).
Nota: Il bordo più corto del pezzo gel dovrebbe essere non inferiore a 1 mm per evitare intasamenti in puntali. - Trasferire i pezzi gel ad un tubo del microcentrifuge e decolorare con decolorante buffer (una miscela di volumi uguali di 100mm Na2S2O3 e 30 mM K3[Fe(CN)6]).
- Lavare i pezzi di gel con bicarbonato di ammonio 200mm (ABC) fino a quando i pezzi di gel sono totalmente incolori.
- Disidratare i pezzi di gel in 1 mL di acetonitrile ordinata (ACN). Reidratare con 200 μL di 10 mM dithiothreitol (disciolto in 50 mM ABC) e incubare a 56 ° C per 45 min.
Nota: Gel completamente disidratato pezzi devono essere molto duro e opaco. Se i pezzi di gel sono ancora morbidi dopo la disidratazione, rimuovere il ACN e aggiungere 1 mL di ACN ordinata per disidratare nuovamente. - Raffreddare i pezzi di gel a temperatura ambiente. Aggiungere 200 μL di 58mm iodoacetamide (disciolto in 50 mM ABC) e incubare a temperatura ambiente per 45 min al buio.
- Dopo un breve lavaggio con acqua, disidratare i pezzi di gel in 1 mL di ACN ordinata.
Nota: I pezzi di gel devono essere completamente disidratati. - Reidratare i pezzi di gel con la quantità appropriata di 10 tripsina ng/μL (disciolto in 50 mM ABC) e incubare a 37 ° C per 12-16 ore.
Nota: I pezzi di gel devono essere completamente reidratati con nessun core opachi. Rimuovere il liquido in eccesso.
-
Isotopo stabile dimetil etichettatura dei peptidi digeriti 29
- Estrarre i peptidi digeriti dai pezzi gel aggiungendo 200 μL di tampone di estrazione (acido formico 5% e 50% ACN in acqua) e incubare a 37 ° C per 30 min con Vortex (a 1.200 giri/min).
- Ripetere il punto 5.2.1 2x. Combinare gli estratti in una microcentrifuga.
- Asciugare i peptidi estratti mediante centrifugazione vuoto.
- Ricostituire i peptidi in 200 μL di bicarbonato di triethylammonium 100 mM (TEAB, pH 8.5).
Attenzione: La procedura 5.2.4 - 5.2.6 deve essere eseguita sul ghiaccio in una cappa aspirante. - Aggiungere 8 μL di 4% CH2O e 8 μL di 4% 13CD2O sperimentali e campioni di controllo, rispettivamente.
Nota: Per controllare il bias di etichettatura isotopica stabile, scambiare l'isotopo stabile per sperimentale e campioni di altri replica biologicamente indipendente di controllo. - Aggiungere 8 μL di 0,6 M NaBH3CN (fanno fresco) e mescolare bene.
- Incubare i campioni a temperatura ambiente per 1 h con vortex.
- Raffreddare i campioni su ghiaccio. Placare la reazione aggiungendo 32 μL di soluzione acquosa di ammoniaca 1%. Quindi, ulteriore placare la reazione aggiungendo 16 μL di acido formico.
- Combinare il campione sperimentale con il campione di controllo corrispondente in una microcentrifuga. Asciugare i campioni mediante centrifugazione vuoto.
-
Frazionamento di dimetil-labeled peptidi
- Preparare le punte di arresto-e-go-estrazione (StageTips)30.
- Inserire una membrana C18 in una punta di 10 μL di lunghezza estesa.
- Aggiungere 300 μg di perline C18 alto pH sospesa in ACN fino alla punta.
- Posizionare la punta in posizione verticale in un tubo del microcentrifuge con un rack fatta in casa che possa stabilizzare la punta e sollevare la punta sul fondale.
- Girare la punta a 1.400 x g per 2 min. scartare il flusso-attraverso.
- Lavare la punta con 50 μL di 80% ACN in 10mm ABC (pH 10.0). Ripetere 1 x.
Nota: Regolare il pH della soluzione di ABC di 10 mM con l'aggiunta di idrossido di ammonio 28%. - Lavare la punta con 50 μL di 50% ACN in 10mm ABC (pH 10.0). Ripetere 1 x.
- Lavare la punta con 50 μL di 10 mM ABC (pH 10.0). Ripetere 1 x.
- Ricostituire i peptidi in 50 μL di 10 mM ABC (pH 10.0).
- Aggiungere il campione ricostituito alla punta preparata. Ricaricare il flusso continuo fino alla punta per assicurare l'efficiente peptide associazione.
- Lavare la punta con 50 μL di 10 mM ABC (pH 10.0). Ripetere 1 x.
- Eluire il peptide graduale per 12 frazioni con 50 μL di 6%, 9%, 12%, 15%, 18%, 21%, 25%, 30%, 35%, 40%, 80% e 6% ACN in 10mm ABC (pH 10.0).
- Combinare due frazioni con un intervallo pari (frazione 1 con 7, 2 con 8, e così via) per ottenere sei combinato frazioni.
- Asciugare i campioni mediante centrifugazione vuoto. I peptidi secchi possono essere conservati a-30 ° C.
- Preparare le punte di arresto-e-go-estrazione (StageTips)30.
6. prestazioni del MS e analisi dei dati
-
Analisi di peptidi di liquida cromatografia-spettrometria di massa
- Ricostituire le frazioni secche del peptide da passo 5.3.7 in 15 μL di acqua contenente acido formico 0.1%. Controllare il pH dei peptidi ricostituiti da spotting 1 μL della soluzione su una striscia di pH (il pH deve essere inferiore a 3).
- Iniettare il campione di ricostituire la colonna cromatografia liquida (LC). Applicare una sfumatura appropriata di solvente (solvente è acqua contenente 0,1% acido formico, solvente B è ACN contenente 0,1% acido formico) in cromatografia liquida ad alte prestazioni (HPLC). Una pendenza tipica del solvente B è come segue: 5-35% in 40 min; 35-70% in 4 min; e tenuti presso il 75% per 10 min.
- Ionizzare i peptidi eluiti electrospray e operare lo spettrometro di massa in modalità dati-dipendente. Selezionare 15 ioni più abbondanti (moltiplicare carica: 2 +, 3 + o superiore) in MS iniziale esplorazione per un'analisi di spettrometria di massa (MS/MS) tandem (dissociazione indotta da collisione, CID). Impostare la dimensione di esclusione dinamico a 500 con un tempo di durata massima di 25 s.
-
Identificazione delle proteine e quantificazione mediante MaxQuant 31
- Impostare la frequenza di falsi scoperta (FDR) di identificazione delle proteine a 0.01 e impostare il numero di peptidi unici a 2 al fine di aumentare la precisione e affidabilità.
- Insieme il minimo richiesto rapporto conta (unico + rasoio) per quantificazione della proteina di 2 e di abilitare il ri-quantificare e funzioni di una partita tra le esecuzioni .
-
Valutazione di significatività di arricchimento utilizzando il R/Bioconductor pacchetto limma 32
- Eseguire un moderato t-test implementato in limma per testare il cambiamento Log2-fold contro zero da almeno tre replicati biologici. Utilizzare la funzione read.table per leggere la tabella di dati. Quindi, utilizzare le funzioni lmFit ed eBayes per i dati di montaggio. Utilizzare la funzione topTable per esportare i risultati di calcolo (compreso il cambiamento Log2-piega media e valori di P ).
- Correggere i valori di P utilizzando il metodo Benjamini – Hochberg per controllare il FDR.
- Applicare un FDR di 0,01 per generare un elenco di proteine arricchito in modo significativo nei campioni sperimentali. Impostare un taglio di due - o triplice cambiamento per controllare ulteriormente i falsi positivi.
Risultati
Vengono presentati i risultati rappresentativi dei passaggi di controllo di qualità. I risultati includono figure dell'analisi di fluorescenza in gel descritto al punto 2.3.2 (Figura 1), l'analisi di western blot descritto al punto 4.1.3 (Figura 2A) e l'analisi di macchiatura d'argento descritto al punto 4.2.2 (Figura 2B). La procedura di controllo di qualità è fondamentale per l'ottimizzazione dei protocolli CARIC. Includere sempre controlli di qualità nella preparazione di esperimenti di identificazione RBP su larga scala.
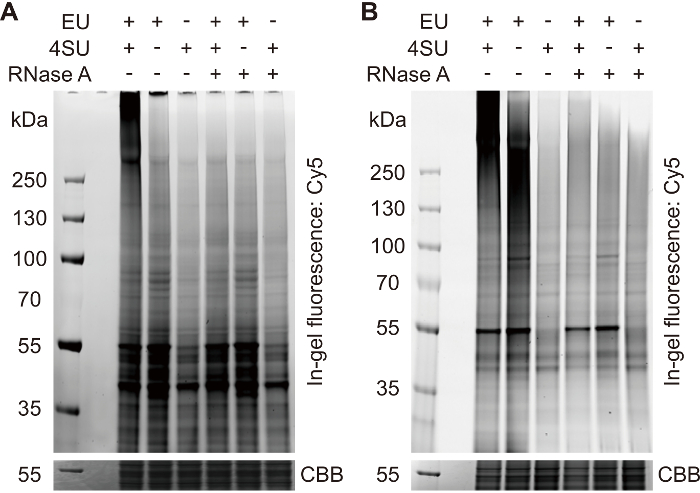
Figura 1: analisi di fluorescenza dei campioni etichettati clicca descritto al punto 2.3.2 In gel. (A) questa pannello mostra un tipico in gel fluorescenza modello di campioni con etichetta fare clic su. Solo l'esempio doppiamente etichettato Mostra una banda di striscio forte a un elevato peso molecolare (> 250 kDa), che rappresenta il segnale di reticolato RNP. Per abolire il segnale di RNP, omette 4SU o EU o digest con RNasi A. Le bande di taglienti di sfondo a un peso molecolare inferiore rappresentano i segnali di etichettato proteine aspecifiche. (B) In alcune occasioni, si può osservare una forte banda spalmata (~ 130-250 kDa) nel campione no4SU-controllo. Questo gruppo rappresenta il segnale di RNAs reticolato con etichetta, che sarà degradata durante la denaturazione al calore, per la maggior parte dei casi. Non interferirà con le procedure successive. CBB = Coomassie blu brillante. Clicca qui per visualizzare una versione più grande di questa figura.
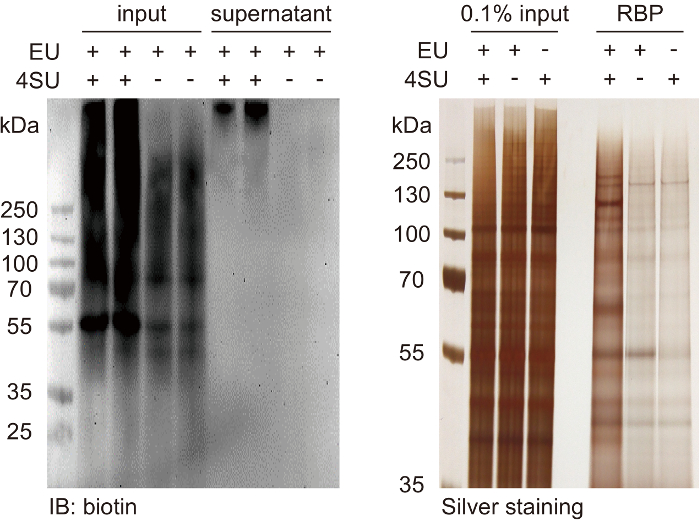
Figura 2: controllo di qualità di affinità pulldown efficienza e il catturato RBPs. (A), questo pannello mostra un'analisi di western blot di biotina segnali nei campioni prima pulldown (ingresso) e nei campioni dopo pulldown (surnatante). Stimare il rapporto dei segnali rimanenti e ottimizzare la quantità di perlina utilizzata al punto 3.1.1. (B) questo pannello mostra un'analisi di macchiatura d'argento dei catturati RBPs rispetto a 0,1% di proteine totali per ingresso. Per le cellule HeLa, l'efficienza di cattura totale generale è ~0.05% - 0.1% di proteine di ingresso. Questo valore può variare notevolmente a causa delle variazioni dell'efficienza metabolica etichettatura dei diversi tipi di cellule. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: rappresentante MS risultati di CARIC. (A), questo pannello illustra un diagramma a Vulcano visualizzati la media Log2-fold e regolate i valori di P di proteine quantificati, calcolati dal pacchetto limma. 597 di proteine con un cambiamento di Log2-fold di > 2 e un valore regolato P < 0.01 sono stati classificati come "CARIC RBPs". (B), questo pannello mostra la sovrapposizione delle proteine CARIC con poli (a) precedentemente identificato umano RBPs7,8,9,10,11. Le proteine sovrapposte sono per lo più codifica RBPs, mentre il resto della CARIC RBPs hanno maggiori probabilità di essere non-codificanti RBPs. Questa figura è una ristampa da lavoro precedentemente pubblicato con il permesso dell' Accademia nazionale delle scienze27. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Il mantenimento dell'integrità di RNA fiera è una delle chiavi del successo esperimenti CARIC. Con appropriati ligandi di Cu (i) e funzionamento attento, degradazione del RNA può essere significativamente ridotto, sebbene degradazione parziale è stata osservata. I rapporti di sostituzione dell'UE e 4SU in campioni sperimentali sono 1,18% e 0,46%, rispettivamente (dati non mostrati). Per RNA intatto con una lunghezza di 2.000 nt, ~ 90% di RNA contengono almeno un UE e uno 4SU. Per RNAs parzialmente degradato con una lunghezza di 1.000 nt, circa il 70% di RNA contengono almeno un UE e uno 4SU. Di conseguenza, parziale degradazione degli RNA non diminuisce drasticamente l'efficienza di CARIC, mentre grave degrado non è accettabile.
Un altro punto critico è passo 1.4, la preparazione per la reazione di clic. Il Cu (I)-reazione catalizzata clicca su RNAs è sensibile alla concentrazione di LDS. Un'alta concentrazione (> 0.1%) di LDS porterà ad una diminuzione di etichettatura segnali su EU-contenente RNA e un aumento dei segnali di fondo sulle proteine (dati non mostrati).
Oltre UE, CARIC è anche compatibile con altri nucleosidi cliccabile, come gli analoghi alchinil e azido di adenosina33,34,35,36. Tuttavia, l'applicazione di CARIC è significativamente limitata dall'efficienza metabolica dei nucleosidi innaturale cliccabili in un sistema biologico di interesse. Quindi, prima esecuzione CARIC utilizzando condizioni diverse da quelle ha dimostrato in questo protocollo, verificare sempre l'efficienza di etichettatura metabolico (ad es., da formazione immagine di fluorescenza).
Recentemente, una strategia simile chiamata RICK (cattura di Interactoma recentemente trascritto RNA utilizzando clic chimica), che incorpora solo UE per etichettare RNA totale e utilizza UV a 254 nm a legame incrociato di RNA e proteine, è stato segnalato37. In particolare, UV a 254 nm può attivare tutti i quattro nucleosidi naturali, come pure EU. Così, irradiazione UV 254 nm può legame incrociato libera UE e dei suoi metaboliti (ad es., fosfati EU) con corrispondenti proteine leganti, che dovrebbero essere preso in considerazione come possibili falsi positivi.
Una domanda intrigante di CARIC è identificare RBPs nei batteri cui RNAs sono per lo più non-poliadenilazione. L'identificazione su grande scala di RBPs fornirà risorse preziose per comprendere le basi molecolari di posttranscriptional regolamenti in batteri38.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Questo lavoro è supportato dalla National Natural Science Foundation di Cina sovvenzioni 91753206, 21425204 e 21521003 e di National Key Research and Development Project 2016YFA0501500.
Materiali
| Name | Company | Catalog Number | Comments |
| HeLa | ATCC | ||
| DMEM (Dulbecco's Modified Eagle Medium) | Thermo Fisher Scientific | 11995065 | |
| FBS (Fetal Bovine Serum) | Thermo Fisher Scientific | 10099141 | |
| Penicillin & Streptomycin | Thermo Fisher Scientific | 15140122 | |
| EU (5-ethynyl uridine) | Wuhu Huaren Co. | CAS:69075-42-9 | |
| 4SU (4-thiouridine) | Sigma Aldrich | T4509 | |
| 10×PBS (Phosphate-Buffered Saline) | Thermo Fisher Scientific | AM9625 | |
| UV cross-linker | UVP | CL-1000 | Equiped with 365-nm UV lamp |
| DEPC (Diethyl pyrocarbonate) | Sigma Aldrich | D5758 | To treat water. Highly toxic! |
| Tris·HCl, pH 7.5 | Thermo Fisher Scientific | 15567027 | |
| LiCl | Sigma Aldrich | 62476 | |
| Nonidet P-40 | Biodee | 74385 | |
| EDTA-free protease inhibitor cocktail | Thermo Fisher Scientific | 88265 | One tablet for 50 mL lysis buffer. |
| LDS (Lithium dodecyl sulfate) | Sigma Aldrich | L9781 | |
| 15-mL ultrafiltration tube (10 kDa cutoff) | Millipore | UFC901024 | |
| 0.5-mL ultrafiltration tube (10 kDa cutoff) | Millipore | UFC501096 | |
| Streptavidin magnetic beads | Thermo Fisher Scientific | 88816 | |
| DMSO (Dimethyl sulfoxide) | Sigma Aldrich | 41639 | |
| Azide-biotin | Click Chemistry Tools | AZ104 | |
| Copper(II) sulfate | Sigma Aldrich | C1297 | |
| THPTA [Tris(3-hydroxypropyltriazolylmethyl)amine] | Sigma Aldrich | 762342 | |
| Sodium ascorbate | Sigma Aldrich | 11140 | |
| Azide-Cy5 | Click Chemistry Tools | AZ118 | |
| LDS sample buffer (4×) | Thermo Fisher Scientific | NP0008 | |
| 10% bis-Tris gel | Thermo Fisher Scientific | NP0301BOX | |
| EDTA | Thermo Fisher Scientific | AM9260G | |
| RNase A | Sigma Aldrich | R6513 | |
| SDS (Sodium dodecyl sulfate) | Thermo Fisher Scientific | 15525017 | |
| NaCl | Sigma Aldrich | S3014 | |
| Brij-97 [Polyoxyethylene (20) oleyl ether] | J&K | 315442 | |
| Triethanolamine | Sigma Aldrich | V900257 | |
| Streptavidin agarose | Thermo Fisher Scientific | 20353 | |
| Urea | Sigma Aldrich | U5378 | |
| Sarkosyl (N-Lauroylsarcosine sodium salt) | Sigma Aldrich | 61743 | |
| Biotin | Sigma Aldrich | B4501 | |
| Sodium deoxycholate | Sigma Aldrich | 30970 | |
| MaxQuant | Version: 1.5.5.1 |
Riferimenti
- Djebali, S., et al. Landscape of transcription in human cells. Nature. 489 (7414), 101-108 (2012).
- Gerstberger, S., Hafner, M., Tuschl, T. A census of human RNA-binding proteins. Nature Reviews Genetics. 15 (12), 829-845 (2014).
- Castello, A., Fischer, B., Hentze, M. W., Preiss, T. RNA-binding proteins in Mendelian disease. Trends in Genetics. 29 (5), 318-327 (2013).
- Nussbacher, J. K., Batra, R., Lagier-Tourenne, C., Yeo, G. W. RNA-binding proteins in neurodegeneration: Seq and you shall receive. Trends in Neuroscience. 38 (4), 226-236 (2015).
- Jazurek, M., Ciesiolka, A., Starega-Roslan, J., Bilinska, K., Krzyzosiak, W. J. Identifying proteins that bind to specific RNAs - focus on simple repeat expansion diseases. Nucleic Acids Research. 44 (19), 9050-9070 (2016).
- Hentze, M. W., Castello, A., Schwarzl, T., Preiss, T. A brave new world of RNA-binding proteins. Nature Reviews Molecular Cell Biology. 19 (5), 327-341 (2018).
- Castello, A., et al. Insights into RNA biology from an atlas of mammalian mRNA-binding proteins. Cell. 149 (6), 1393-1406 (2012).
- Baltz, A. G., et al. The mRNA-bound proteome and its global occupancy profile on protein-coding transcripts. Molecular Cell. 46 (5), 674-690 (2012).
- Beckmann, B. M., et al. The RNA-binding proteomes from yeast to man harbour conserved enigmRBPs. Nature Communications. 6, 10127-10135 (2015).
- Conrad, T., et al. Serial interactome capture of the human cell nucleus. Nature Communications. 7, 11212-11222 (2016).
- Castello, A., et al. Comprehensive identification of RNA-binding domains in human cells. Molecular Cell. 63 (4), 696-710 (2016).
- Kwon, S. C., et al. The RNA-binding protein repertoire of embryonic stem cells. Nature Structural & Molecular Biology. 20 (9), 1122-1130 (2013).
- Liepelt, A., et al. Identification of RNA-binding proteins in macrophages by interactome capture. Molecular & Cellular Proteomics. 15 (8), 2699-2714 (2016).
- Liao, Y., et al. The cardiomyocyte RNA-binding proteome: Links to intermediary metabolism and heart disease. Cell Reports. 16 (5), 1456-1469 (2016).
- Mitchell, S. F., Jain, S., She, M. P., Parker, R. Global analysis of yeast mRNPs. Nature Structural & Molecular Biology. 20 (1), 127-133 (2013).
- Matia-González, A. M., Laing, E. E., Gerber, A. P. Conserved mRNA-binding proteomes in eukaryotic organisms. Nature Structural & Molecular Biology. 22 (12), 1027-1033 (2015).
- Despic, V., et al. Dynamic RNA-protein interactions underlie the zebrafish maternal-to-zygotic transition. Genome Research. 27 (7), 1184-1194 (2017).
- Wessels, H. H., et al. The mRNA-bound proteome of the early fly embryo. Genome Research. 26 (7), 1000-1009 (2016).
- Sysoev, V. O., et al. Global changes of the RNA-bound proteome during the maternal-to-zygotic transition in Drosophila. Nature Communications. 7, 12128(2016).
- Reichel, M., et al. In planta determination of the mRNA-binding proteome of Arabidopsis etiolated seedlings. Plant Cell. 28 (10), 2435-2452 (2016).
- Marondedze, C., Thomas, L., Serrano, N. L., Lilley, K. S., Gehring, C. The RNA-binding protein repertoire of Arabidopsis thaliana. Scientific Reports. 6, 29766-29778 (2016).
- Zhang, Z., et al. UV crosslinked mRNA-binding proteins captured from leaf mesophyll protoplasts. Plant Methods. 12, 42-53 (2016).
- Bunnik, E. M., et al. The mRNA-bound proteome of the human malaria parasite Plasmodium falciparum. Genome Biology. 17, 147-164 (2016).
- Lueong, S., Merce, C., Fischer, B., Hoheisel, J. D., Erben, E. D. Gene expression regulatory networks in Trypanosoma brucei: insights into the role of the mRNA-binding proteome. Molecular Microbiology. 100 (3), 457-471 (2016).
- Nandan, D., et al. Comprehensive identification of mRNA-binding proteins of Leishmania donovani by interactome capture. PLoS ONE. 12 (1), e0170068(2017).
- Jankowsky, E., Harris, M. E. Specificity and nonspecificity in RNA-protein interactions. Nature Reviews Molecular Cell Biology. 16 (9), 533-544 (2015).
- Huang, R., Han, M., Meng, L., Chen, X. Transcriptome-wide discovery of coding and noncoding RNA-binding proteins. Proceedings of the National Academy of Sciences of the United States of America. 115 (17), E3879-E3887 (2018).
- Shevchenko, A., Tomas, H., Havlis, J., Olsen, J. V., Mann, M. In-gel digestion for mass spectrometric characterization of proteins and proteomes. Nature Protocols. 1 (6), 2856-2860 (2006).
- Boersema, P. J., Raijmakers, R., Lemeer, S., Mohammed, S., Heck, A. J. R. Multiplex peptide stable isotope dimethyl labeling for quantitative proteomics. Nature Protocols. 4 (4), 484-494 (2009).
- Rappsilber, J., Mann, M., Ishihama, Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics using StageTips. Nature Protocols. 2 (8), 1896-1906 (2007).
- Cox, J., Mann, M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nature Biotechnology. 26 (12), 1367-1372 (2008).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), e47(2015).
- Grammel, M., Hang, H., Conrad, N. K. Chemical reporters for monitoring RNA synthesis and poly(A) tail dynamics. ChemBioChem. 13 (8), 1112-1115 (2012).
- Curanovic, D., et al. Global profiling of stimulus-induced polyadenylation in cells using a poly(A) trap. Nature Chemical Biology. 9 (11), 671-673 (2013).
- Zheng, Y. X., Beal, P. A. Synthesis and evaluation of an alkyne-modified ATP analog for enzymatic incorporation into RNA. Bioorganic & Medicinal Chemistry Letters. 26 (7), 1799-1802 (2016).
- Nainar, S., et al. Metabolic incorporation of azide functionality into cellular RNA. ChemBioChem. 17 (22), 2149-2152 (2016).
- Bao, X., et al. Capturing the interactome of newly transcribed RNA. Nature Methods. 15 (3), 213-220 (2018).
- Holmqvist, E., Vogel, J. RNA-binding proteins in bacteria. Nature Reviews Microbiology. , Published online (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon