Method Article
Capture et Identification des protéines de liaison à l’ARN à l’aide de Click chimie assistée par ARN-interactome Capture stratégie (CARIC)
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Un protocole détaillé pour l’application de la clic chimie assistée par RNA-interactome capture (CARIC) stratégie pour identifier les protéines liant les deux codage et non codantes RNAs est présenté.
Résumé
Une identification complète des protéines de liaison à l’ARN (pratiques commerciales restrictives) est essentielle pour comprendre le réseau de régulation post-transcriptionnelle dans les cellules. Une stratégie largement utilisée pour la capture des RBP exploite la polyadénylation [poly (a)] de l’ARN cible, qui se produit principalement sur des ARNm eucaryotes matures, laissant la plupart des protéines liant de non-poly(A) ARN non identifiés. Nous décrivons ici les procédures détaillées d’une méthode récemment appelé clic chimie assistée par RNA-interactome capture (CARIC), qui permet la capture de transcriptome à l’échelle des pratiques commerciales restrictives des poly (a) et non-poly(A) en combinant le marquage métabolique de RNAs in vivo UV réticulation et marquage de bioorthogonal.
Introduction
Le génome humain est transcrite en différents types d’ARN codantes et non codantes (ncRNA), y compris les ARNm, rRNA, ARNt, petits ARN nucléaires (snRNA), petits ARN nucléolaires (ARNpno) et longtemps non-codage RNAs (lncRNAs)1. La plupart de ces ARN possède des vêtements des pratiques commerciales restrictives et fonction ribonucléoprotéique particules (RNP)2. Par conséquent, une identification complète des pratiques commerciales restrictives est une condition sine qua non pour comprendre le réseau de régulation entre les ARN et les pratiques commerciales restrictives, qui est impliqué dans diverses maladies humaines3,4,5.
Ces dernières années ont vu un grand élan de pratiques commerciales restrictives découvert dans divers systèmes eucaryotes2,6, y compris humaine7,8,9,10,11, souris12,13,14, levure9,15,16, zebrafish17, Drosophila melanogaster18,19 , Caenorhabditis elegans16, Arabidopsis thaliana20,21,22et parasites de l’homme23,24,25 . Ces avances ont été facilitées par une stratégie de capture RBP développé par Castello et al. 7 et Baltz et al. 8 en 2012, qui combine en vivo UV réticulation des ARN et ses protéines qui interagissent, oligo (décollement) capture des poly RNAs et spectrométrie de masse (MS)-base de profilage protéomique. Toutefois, compte tenu du fait que poly (a) existe pour la plupart sur l’ARNm mature, qui représentent seulement environ 3 % - 5 % du transcriptome eucaryotes26, cette stratégie largement utilisée n’est pas capable de capturer des pratiques commerciales restrictives interagissant avec non-poly(A) ARN, dont la plupart ncRNA et pré-ARNm.
Nous rapportons ici les modalités d’une stratégie développée récemment pour la capture de l’échelle du transcriptome de pratiques commerciales restrictives à la poly (a) et non-poly(A)27. Appelé CARIC, cette stratégie combine en vivo UV réticulation et marquage métabolique d’ARN avec photoactivatable et « cliquables » analogues nucléosidiques (qui contiennent un groupe fonctionnel bioorthogonal pouvant participer à la réaction de clic), 4 - thiouridine (4SU) et 5-ethynyluridine (UE). Les étapes indispensables pour obtenir des résultats idéales avec la stratégie CARIC sont marquage métabolique efficace, réaction de réticulation et cliquez sur UV et le maintien de l’intégrité de la RNA. Cu (i) utilisé comme catalyseur dans la réaction de clic peut provoquer la fragmentation du RNAs, un ligand de Cu (i) qui peut de réduire la fragmentation de RNA est essentiel. Nous décrivons comment effectuer des réactions efficaces cliquez dans les lysats cellulaires sans causer de grave dégradation de l’ARN.
Bien que RBP capture et identification dans les cellules HeLa seulement est décrite dans le présent protocole, la CARIC stratégie peut être appliquée à différents types de cellules et éventuellement aux organismes vivants. En plus de la capture de la RBP, ce protocole prévoit également simplifiée des procédures pas à pas pour la préparation de l’échantillon MS et identification des protéines et la quantification, qui peut être utile pour ceux qui ne connaissent pas proteomic expériences.
Protocole
ATTENTION : Quand il y a lieu, les réactifs utilisés doivent être achetés sous forme de RNase-libre, ou dissous dans exempte de RNase, solvants (pour la plupart des cas, au diéthyl pyrocarbonate (DEPC)-eau traitée). Lors de la manipulation des échantillons d’ARN et de réactifs de RNase-libre, toujours porter des gants et des masques et changez-les fréquemment pour éviter toute contamination de la RNase.
1. préparation du lysat de métaboliquement marqués et UV cellules réticulées
-
Incorporation métabolique de l’UE et 4SU
- Cellules HeLa culture dans de l’aigle de la modification de Dulbecco Medium (DMEM) additionné de 10 % sérum fœtal (SVF), 100 U/mL de pénicilline et 100 μg/mL de streptomycine à 37 ° C en atmosphère 5 % CO2 . Culture ~ 4 x 107 les cellules HeLa (dans les deux plats de 15 cm) pour préparer un expérimental ou échantillon de contrôle pour une qu'exécutent standards MS.
- Quand les cellules HeLa cultivées atteignent environ 80 % de confluence, enlevez le milieu de culture et ajouter 15 mL de milieu frais préchauffée par plat de 15 cm.
- Ajouter 15 μL / plat de 100 mM EU (dissoute dans une solution saline tamponnée au phosphate (PBS)) à une concentration finale de 1 mM et 7,5 μL / plat de 100 mM 4SU (dissous dans du PBS) à une concentration finale de 0,5 mM pour expérimental et des échantillons de contrôle des noUV. Ajouter 15 μL / plat de 100 mM EU (dissous dans du PBS) à une concentration finale de 1 mM pour les échantillons de contrôle no4SU.
NOTE : 4SU est activable par photo ; ainsi, la protection contre la lumière après avoir ajouté 4SU est requise. - Couvrir les plats avec du papier et culture les cellules pendant 16 h. ajouter la moitié du montant de l’UE et la 4SU ou l’UE seulement étape 1.1.3 à l’expérimental, noUV- et no4SU-contrôle des échantillons, respectivement et continuent à cultiver pour un autre 2 h.
-
In vivo Réticulation UV
- Enlever le milieu de culture, laver les cellules 3 x 5 ml de PBS par plat et PBS résiduelle de supprimer autant que possible.
NOTE : Liquide de résidus réduira considérablement l’efficacité réticulation. - Pour expérimental et des échantillons de no4SU-contrôle, placez les plats sur la glace avec le couvercle enlevé et irradier les cellules avec la lumière à 2 J/cm2 par un réticulant UV 365 nm UV.
- Pour les échantillons de noUV-contrôle, placez les plats sur la glace et les protéger de la lumière.
Remarque : Toutes les étapes suivantes pour les échantillons de noUV-contrôle est souhaitable dans une pièce sombre.
- Enlever le milieu de culture, laver les cellules 3 x 5 ml de PBS par plat et PBS résiduelle de supprimer autant que possible.
-
Lyse des cellules et homogénéisation
- Ajouter 1 mL par plat de tampon de lyse avant (10 mM Tris∙HCl, pH 7.5, 50 mM acide LiCl, 0,02 % Nonidet P-40 et tétraacétique (EDTA)-cocktail inhibiteur de protéase libre) aux cellules. Grattez les cellules à l’aide d’un releveur de caoutchouc cellulaire et recueillir la suspension avant de la lyse dans un tube de 15mL.
Remarque : Cette étape va casser la membrane cellulaire et libèrent des protéines cytoplasmiques solubles et ARN. Ne pas Centrifuger le tube et éliminer le surnageant. - La suspension de deux plats de 15 cm, réglez le volume à 6 mL par adjonction de tampon de lyse avant. Ajouter à la suspension de lyse avant un volume équivalent de tampon de lyse-R (200 mM Tris∙HCl, pH 7.5, LiCl, 500 mM 2 % sulfate de dodécyle lithium [LDS]).
- Homogénéiser le lysat cellulaire en faisant passer dans une seringue avec une aiguille étroite (27 G) plusieurs fois jusqu'à ce que le lysat est claire et homogène. Incuber le lysat à 4 ° C, avec une légère rotation (~ 15 tr/min) pendant 1 h.
NOTE : Cette dernière étape permettra la dénaturation complète de protéines. Le lysat peut être en toute sécurité stocké à-70 ° C pendant environ un mois.
- Ajouter 1 mL par plat de tampon de lyse avant (10 mM Tris∙HCl, pH 7.5, 50 mM acide LiCl, 0,02 % Nonidet P-40 et tétraacétique (EDTA)-cocktail inhibiteur de protéase libre) aux cellules. Grattez les cellules à l’aide d’un releveur de caoutchouc cellulaire et recueillir la suspension avant de la lyse dans un tube de 15mL.
-
Préparation pour la réaction de clic
- Diluer le lysat en ajoutant 20 volumes de tampon de dilution (50 mM Tris∙HCl, pH 7,5) et diviser en fractions de 15 mL.
NOTE : Les Solutions contenant une forte concentration de sel et de détergent vont compromettre l’efficacité du Cu (I)-catalysé réaction clic ; ainsi, le tampon du lysat doit être changé. - Concentré de chaque fraction en utilisant un tube de 15 mL ultrafiltration (avec un seuil de 10 kDa de poids moléculaire) jusqu'à ce que le volume est inférieur à 1 mL. Utiliser un rotor oscillant-seau pour faire tourner le tube d’ultrafiltration à 4 000 x g à 4 ° C pendant environ 15 min.
- Ajouter 14 mL de tampon de dilution à la fraction de lysate concentrée et répétez l’étape 1.4.2. Combinez les fractions et les concentrer sur un volume de 6 mL par ultrafiltration (4 000 x g à 4 ° C pendant environ 15 min).
Remarque : La plupart des sel et LDS va maintenant être supprimé, donc le lysat est prêt pour la réaction de clic. Le lysat peut être stocké à-70 ° C pendant une semaine. Éviter de multiples cycles de gel-dégel, car ils seront traduira par l’importante dégradation de l’ARN. Aliquoter le lysat si petite échelle caractérisations sont nécessaires.
- Diluer le lysat en ajoutant 20 volumes de tampon de dilution (50 mM Tris∙HCl, pH 7,5) et diviser en fractions de 15 mL.
2. préparation des échantillons d’ARN-interactome Capture
-
Pré-approbation du lysat
- Ajouter 100 μL streptavidine des billes magnétiques pour 6 mL de lysat et faites tourner doucement (~ 15 tr/min) pendant 30 min à température ambiante pour éliminer naturellement les protéines biotinylées.
- Les perles à l’aide d’un aimant (pour environ 20 min à 4 ° C) et transférer le pré-approuvée lysat dans un nouveau tube de granule.
-
Rendement de la réaction de clic
- Préparer le mélange de la réaction : 6,5 μL de stock de biotine (100 mM azoture-biotine dissous dans le diméthylsulfoxyde [DMSO] à une concentration finale de 100 μM), 3.25 μL de stock cuivre (rendent frais ; 1 M CuSO4 dissous dans l’eau à une concentration finale de 500 μM) , 65 μL de stock ligand (THPTA dissous dans l’eau à une concentration finale de 2 mM à 200 mM) et 262.75 μL de H2O.
Remarque : THPTA est synonyme d’amine [(1-hydroxypropyl-1H-1,2,3-triazol-4-yl) méthyl] Tris. - Ajouter le mélange de la réaction à 6 mL de pré-approuvée lysat et bien mélanger. Puis, ajoutez 162,5 μl de réactif de réduire (rendent fraîche ; ascorbate de sodium 40 mg/mL à une concentration finale 5 mM) pour le lysat et bien mélanger. Le volume final devrait être de 6,5 mL.
- Incuber le mélange réactionnel pendant 2 h à température ambiante dans un agitateur orbital (800 tr/min). Ajouter 5 mM EDTA dans le mélange réactionnel et incuber pendant 5 min étancher la réaction.
- Préparer le mélange de la réaction : 6,5 μL de stock de biotine (100 mM azoture-biotine dissous dans le diméthylsulfoxyde [DMSO] à une concentration finale de 100 μM), 3.25 μL de stock cuivre (rendent frais ; 1 M CuSO4 dissous dans l’eau à une concentration finale de 500 μM) , 65 μL de stock ligand (THPTA dissous dans l’eau à une concentration finale de 2 mM à 200 mM) et 262.75 μL de H2O.
-
Caractérisations à petite échelle
- Préparer le mélange de réaction comme au point 2.2.1 avec le stock de biotine remplacé par la mère de colorant (p. ex., 100 mM azoture-Cy5 dissous dans le DMSO).
Remarque : La quantité de réactif doit être ajustée selon le volume du lysat. En règle générale, une portion de 20 μL du lysat est suffisant pour caractérisations comme une analyse de fluorescence en gel. - Ajouter le mélange de la réaction dans le lysat et il incuber pendant 2 h à température ambiante. Puis, ajoutez un tiers du volume du tampon échantillon LDS (4 x), il dénature à 55 ° C pendant 5 min et résoudre l’échantillon sur un gel de bis-Tris 10 %.
Remarque : Pour vérifier que le signal de fluorescence est présenté sur l’ARN, incluent des contrôles à la digestion de la RNase A après la réaction de clic.
- Préparer le mélange de réaction comme au point 2.2.1 avec le stock de biotine remplacé par la mère de colorant (p. ex., 100 mM azoture-Cy5 dissous dans le DMSO).
-
Nettoyage du mélange réactionnel
- Ajouter huit volumes de méthanol prérefroidie (100 %) au mélange réactionnel trempés et il incuber 30 min à-30 ° C pour la précipitation. Effectuer la précipitation dans des tubes à centrifuger conique de 50 mL.
Remarque : Si le volume total est supérieur à 50 mL, diviser le mélange réactionnel dans deux tubes à centrifuger conique de 50 mL. - Préparer le tampon de reconstitution : mélanger un volume de tampons A (dodécylsulfate de sodium 4 % [SDS] et 10 mM EDTA) avec huit volumes de tampon B (1 % des triéthanolamine Brij-97, NaCl 150 mM et 50 mM, pH 7,4).
- Centrifuger à 4 000 x g pendant 15 min à 4 ° C et éliminer le surnageant. Ajouter environ 1 à 2 mL de méthanol prérefroidie au culot. Pipette de haut en bas pour casser le culot et assurez-vous que le culot est complètement suspendu avec aucun morceaux visibles. Remplir le tube avec du méthanol prérefroidie. Répétez cette étape 2 x.
- Centrifuger à 4 000 x g pendant 15 min à 4 ° C et éliminer le surnageant. Remettre que les tubes et centrifuger à nouveau à 4 000 x g pendant 5 min Tracer soigneusement sur le méthanol résiduel autant que possible sans déranger le culot.
- Ajouter 10 mL de tampon de reconstitution au culot. Pipetter haut et bas pour dissoudre le culot. Centrifuger à 4 000 x g pendant 10 min à 4 ° C.
- Transférer le surnageant dans un nouveau tube. Recueillir 20 μl de l’échantillon pour le contrôle de la qualité (voir la section 4).
NOTE : Maintenant, l’échantillon est prêt pour la capture de RNA-interactome. L’échantillon de lait reconstitué peut être stocké à-70 ° C pendant une semaine.
- Ajouter huit volumes de méthanol prérefroidie (100 %) au mélange réactionnel trempés et il incuber 30 min à-30 ° C pour la précipitation. Effectuer la précipitation dans des tubes à centrifuger conique de 50 mL.
3. ARN-interactome Capture
-
Préparation des streptavidine-agarose perles
- Prendre 1 600 μL de streptavidine-agarose de lisier (800 μL de perles sédentarisés) pour 10 mL de l’échantillon de lait reconstitué dans un tube à centrifuger conique de 15 mL.
- Tournez en bas les perles à 4000 x g pour 5 min. Retirer délicatement le surnageant sans déranger les perles sédentarisés.
- Laver les billes avec 10 mL de 50 mM Tris∙HCl (pH 7.5). Tournez en bas les perles (4 000 x g pendant 5 min) et éliminer le surnageant. Répétez cette étape 2 x.
-
Affinité pulldown
- Transférer l’échantillon nettoyé et reconstitué à l’étape 2.4.6 aux talons streptavidine-agarose (voir étape 3.1). Incuber pendant la nuit avec rotation douce à 4 ° C.
-
Laver les billes de streptavidine
- Tournez en bas les perles (4 000 x g pendant 5 min) et transférer le surnageant dans un nouveau tube. Recueillir 20 μl de l’échantillon pour le contrôle qualité.
- Laver les billes avec 10 mL de tampon de lavage (une SDD de 2 % dans du PBS, pH 7,4). Incuber pendant 10 min avec légère rotation (~ 12 tr/min) à température ambiante. Tournez en bas les perles (4 000 x g pendant 5 min) et éliminer le surnageant. Répéter 1 x.
- Répétez l’étape 3.3.2 avec tampon de lavage B (urée 8 M et 250 mM NH4HCO3 dissous dans l’eau). Répétez l’étape 3.3.2 avec tampon de lavage C (2,5 M de NaCl dans du PBS, pH 7,4). Ensuite, laver les billes avec 10 mL de 50 mM Tris∙HCl (pH 7.5). Tournez en bas les perles (4 000 x g pendant 5 min) et éliminer le surnageant.
- Réparti sur les perles et transférez-les à deux tubes de microcentrifuge de 1,5 mL.
-
Élution de la RNP capturé
- Préparer le tampon d’élution biotine : biotine 12,5 mM, 75 mM NaCl, 7,5 mM Tris∙HCl (pH 7.5), 1,5 mM EDTA, 0,15 % SDS, sarkosyl 0,075 % et 0,02 % désoxycholate de sodium.
NOTE : Magasin de la mémoire tampon à la température ambiante, de la biotine peut précipiter à 4 ° C. - À 400 μL de perles sédentarisés lavés, ajouter 400 ml de tampon d’élution de biotine.
- Les incuber dans un agitateur orbital (1 500 tr/min) à température ambiante pendant 20 min. Puis, incuber dans un agitateur orbital avec un bloc chauffant (1 500 tr/min, 65 ° C) pendant environ 10 minutes Spin down les perles (7 800 x g pendant 1 min) et de recueillir le RNP éluée.
- Aux talons, ajouter 400 ml de tampon d’élution biotine fraîches et répétez l’étape 3.4.3. Combiner les deux élué dans un tube de 15 mL.
- Préparer le tampon d’élution biotine : biotine 12,5 mM, 75 mM NaCl, 7,5 mM Tris∙HCl (pH 7.5), 1,5 mM EDTA, 0,15 % SDS, sarkosyl 0,075 % et 0,02 % désoxycholate de sodium.
-
Digestion de la RNase
- Ajouter trois volumes de tampon de dilution à la RNP éluée pour diminuer la concentration de la SDD. Concentrer l’échantillon dilué à l’aide d’un tube de 0,5 mL ultrafiltration (avec un seuil de poids moléculaire de 10 kDa ; tournent à 12 000 x g à 4 ° C pendant environ 30 min) à ~ 40 μL.
- Ajouter 0,5 μg/μL RNase A et il incuber pendant 2 h à 37 ° C pour libérer des pratiques commerciales restrictives de RNAs réticulés. Prélever 2 μL des pratiques commerciales restrictives pour le contrôle de la qualité (voir la section 4).
4. contrôle de la qualité
-
Contrôle de l’efficacité de la conversion d’affinité
- Prendre 10 μl de l’échantillon « avant-pulldown » d’étape 2.4.6 et 10 μl de l’échantillon de le « après-pulldown » de l’étape 3.3.1.
- Analyse des échantillons à l’aide de procédures standard de la tache occidentale (gel 10 % bis-Tris).
- Colorer la membrane Polyfluorure de vinylidène (PVDF) avec conjugué streptavidine-HRP pour surveiller les signaux de biotine de résidus de l’échantillon de le « après-pulldown ».
Remarque : Si le signal de la biotine de l’échantillon de le « après-pulldown » est supérieur à un cinquième du signal de l’échantillon « avant-pulldown », augmenter la quantité de streptavidine-agarose perles utilisées à l’étape 3.1.1.
-
Contrôle de l’efficacité de la capture totale
- Prendre 2 μL de l’échantillon RBP publiée d’étape 3.5.2 et 0,5 μl de l’échantillon « avant-pulldown » (comme l’entrée de 0,1 %) d’après l’étape 2.4.6.
- Analyse des échantillons à l’aide de procédures standard de coloration à l’argent.
- Fixer le gel avec tampon de fixation (éthanol à 40 %, 10 % d’acide acétique) pendant 20 min, suivie d’une sensibilisation (13 mM Na2S2O3, acétate de sodium de 83 mM, 30 % d’éthanol) pendant 30 min.
- Laver le gel 3 x avec de l’eau pendant 5 min et, ensuite, souiller avec un 15 mM AgNO3 solution pendant 20 min. laver le gel 2 x avec de l’eau pendant 1 min, développer de 0,24 M Na2CO3 et 0,012 % de formaldéhyde et clore par 45 mM EDTA, lorsque la coloration est suffici ent.
Remarque : L’intensité de coloration à l’argent des pratiques commerciales restrictives capturés doit être similaire à celle de l’entrée de 0,1 %.
5. préparation des échantillons pour la SP
-
La digestion trypsique de pratiques commerciales restrictives capturés en gel 28
- Ajouter un quart volume de tampon de SDS (5 x) aux échantillons RBP publiées de l’étape 3.5.2. Dénaturer l’échantillon à 95 ° C pendant 10 min.
- Résoudre les pratiques commerciales restrictives sur un gel de SDS-polyacrylamide 10 % 1,5 mm.
- Souillez le gel avec de l’argent, suivant des protocoles standard.
- La voie de l’échantillon expérimental ou échantillon de contrôle avec gel de concentration et la principale bande de RNase A (~ 15 kDa) enlevé de l’accise.
- Couper la voie excisée en petits morceaux (environ 1 à 1,5 x ~ 1 à 1,5 mm).
Remarque : Le bord le plus court de la pièce de gel doit être non inférieur à 1 mm pour éviter tout colmatage en pointes de pipette. - Transférer les morceaux de gel dans un tube de microcentrifuge et décolorer avec décoloration tampon (un mélange de deux volumes égaux de 100 mM Na2S2O3 et 30 mM K3[Fe(CN)6]).
- Laver les morceaux de gel avec 200 mM de bicarbonate d’ammonium (ABC) jusqu'à ce que les morceaux de gel sont totalement incolores.
- Déshydrater les morceaux de gel dans 1 mL de pur acétonitrile (ACN). Réhydrater avec 200 μL de 10 mM le dithiothréitol (dissous dans 50 mM ABC) et incuber à 56 ° C pendant 45 min.
Remarque : Morceaux de gel complètement déshydraté doit être très dur et opaque. Si les morceaux de gel sont encore molles après déshydratation, enlever l’ACN et ajouter 1 mL d’ACN soignée pour déshydrater à nouveau. - Refroidir les morceaux de gel à la température ambiante. Ajouter 200 μl de 58 mM iodoacétamide (dissous dans 50 mM ABC) et incuber à température ambiante pendant 45 min dans le noir.
- Après un bref lavage avec de l’eau, mettre en attente les morceaux de gel dans 1 mL d’ACN soignée.
Remarque : Les morceaux de gel doivent être complètement déshydratés. - Réhydrater les morceaux de gel avec la quantité appropriée de la trypsine 10 ng/μl (dissous dans 50 mM ABC) et incuber à 37 ° C pendant 12 à 16 h.
Remarque : Les morceaux de gel doivent être complètement réhydratées avec aucun carottes opaques. Enlever tout excès de liquide.
-
Isotope stable diméthyl étiquetant des peptides digérées 29
- Extrait les morceaux de gel en ajoutant 200 μl de tampon d’extraction les peptides digérées (acide formique 5 % et 50 % ACN dans l’eau) et laisser incuber à 37 ° C pendant 30 min avec Vortex (à 1 200 tr/min).
- Répétez l’étape 5.2.1 x 2. Combiner les extraits dans un des tubes de microcentrifuge.
- Sécher les peptides extraits par centrifugation de vide.
- Reconstituer les peptides dans 200 μl de bicarbonate de triéthylammonium 100 mM (TEAB, pH 8,5).
ATTENTION : Les étapes 5.2.4 - 5.2.6 devraient être effectuées sur la glace sous une hotte. - Ajouter 8 μL de 4 % CH2O et 8 μL de 4 % 13CD2O à l’expérimental et des échantillons de contrôle, respectivement.
Remarque : Pour contrôler le parti pris de marquage isotopique stable, échanger les isotopes stables pour expérimental et contrôler des échantillons de l’autre replicate biologiquement indépendant. - Ajouter 8 μL de 0,6 M NaBH3CN (pas frais) et bien mélanger.
- Incuber les échantillons à la température ambiante pendant 1 h avec agitation.
- Refroidir les échantillons sur la glace. Étancher la réaction en ajoutant 32 μL de solution aqueuse d’ammoniaque 1 %. Puis, plus loin étancher la réaction en ajoutant 16 μL d’acide formique.
- Combiner l’échantillon expérimental avec l’échantillon de contrôle correspondant dans un des tubes de microcentrifuge. Sécher les échantillons par centrifugation de vide.
-
Fractionnement des peptides marqués au diméthyl
- Préparer les conseils arrêt-et-aller-extraction (StageTips)30.
- Insérez une membrane C18 dans une pointe de 10 μL de longueur étendu.
- Ajouter 300 μg de perles de C18 de pH élevé chez ACN a été suspendu jusqu'à la pointe.
- Placer l’embout verticalement dans un tube de microcentrifuge avec une crémaillère artisanale qui peut stabiliser la pointe et soulever la pointe au dessus du fond.
- Tourner la pointe à 1 400 g pendant 2 min. jetez l’accréditif.
- Laver l’embout avec 50 μL de 80 % ACN en 10 mM ABC (pH 10,0). Répéter 1 x.
Remarque : Ajuster le pH de la solution ABC 10 mM en ajoutant hydroxyde d’ammonium 28 %. - Laver l’embout avec 50 μL de 50 % ACN en 10 mM ABC (pH 10,0). Répéter 1 x.
- Laver l’embout avec 50 μL de 10 mM ABC (pH 10,0). Répéter 1 x.
- Reconstituer les peptides dans 50 μL de 10 mM ABC (pH 10,0).
- Ajouter l’échantillon de lait reconstitué à la pointe préparée. Recharger le cheminement jusqu'à la pointe pour assurer la liaison peptide efficace.
- Laver l’embout avec 50 μL de 10 mM ABC (pH 10,0). Répéter 1 x.
- Éluer le peptide par étapes pour 12 fractions avec 50 μL de 6 %, 9 %, 12 %, 15 %, 18 %, 21 %, 25 %, 30 %, 35 %, 40 %, 80 % et 6 % ACN en 10 mM ABC (pH 10,0).
- Combiner deux fractions avec un intervalle égal (fraction 1 à 7, 2 à 8, et ainsi de suite) pour obtenir six combinés des fractions.
- Sécher les échantillons par centrifugation de vide. Les peptides séchées peuvent être stockées à-30 ° C.
- Préparer les conseils arrêt-et-aller-extraction (StageTips)30.
6. exécution de l’analyse de données et MS
-
Analyse des peptides par spectrométrie de masse en tandem par chromatographie liquide
- Reconstituer les fractions peptidiques séchées d’étape 5.3.7 dans 15 μL d’eau contenant de l’acide formique 0,1 %. Vérifier le pH de peptides reconstitués de détachage 1 μL de la solution sur une bande de pH (pH devrait être de moins de 3 ans).
- Injecter l’échantillon de reconstituer dans la colonne de chromatographie liquide (LC). Appliquer un dégradé appropriée du solvant (solvant est l’eau contenant 0,1 % d’acide formique, solvant B est ACN contenant 0,1 % d’acide formique) en chromatographie liquide à haute performance (HPLC). Un gradient typique de solvant B se présente comme suit : 5 à 35 % en 40 min ; 35 à 70 % en 4 min ; et qui s’est tenue à 75 % pendant 10 min.
- Ionisent les peptides éluées par électrospray et exploiter le spectromètre de masse en mode dépendant des données. Choisissez 15 ions plus abondants (multichargés : 2 + 3 + et supérieur) dans la première MS scan pour une analyse de spectrométrie de masse (MS/MS) tandem (dissociation induite par collision, CID). Définir la taille de l’exclusion dynamique à 500 avec une durée maximale de 25 s.
-
Identification des protéines et la quantification à l’aide de MaxQuant 31
- Fixer le taux de fausse découverte (FDR) de l’identification des protéines à 0,01 et définissez le nombre de peptides uniques sur 2 afin d’accroître l’exactitude et la fiabilité.
- Ensemble minimal requis ratio compte (unique + rasoir) pour la quantification des protéines à 2 et permettent le ré-quantifier et fonctions correspondent entre les exécutions .
-
Évaluation de l’importance l’enrichissement à l’aide de la R/Bioconductor paquet Leduc 32
- Effectuer un modéré t-test implémenté dans Leduc pour tester la modification Log2-pli contre zéro depuis au moins trois réplicats biologiques. Utilisez la fonction read.table pour lire la table de données. Ensuite, utilisez les fonctions lmFit et eBayes pour les données de montage. Utilisez la fonction de topTable pour exporter les résultats de calcul (y compris la moyenne Log2-pli changement et les valeurs de P ).
- Corrigez les valeurs de P selon la méthode de Benjamini – Hochberg pour contrôler le FDR.
- Appliquer un FDR de 0,01 pour générer une liste de protéines considérablement enrichi dans les échantillons de laboratoire. Définir un seuil de changement de deux ou trois fois pour contrôler davantage les faux positifs.
Résultats
Les résultats représentatifs des étapes de contrôle qualité sont présentés. Les résultats incluent des chiffres de l’analyse par fluorescence en gel décrit à l’étape 2.3.2 (Figure 1), l’analyse par transfert western décrit à l’étape 4.1.3 (Figure 2 a) et l’analyse de coloration à l’argent décrite à l’étape 4.2.2 (Figure 2 b). Les mesures de contrôle de la qualité sont essentiels pour l’optimisation des protocoles CARIC. Toujours inclure des contrôles de qualité dans la préparation des expériences d’identification RBP à grande échelle.
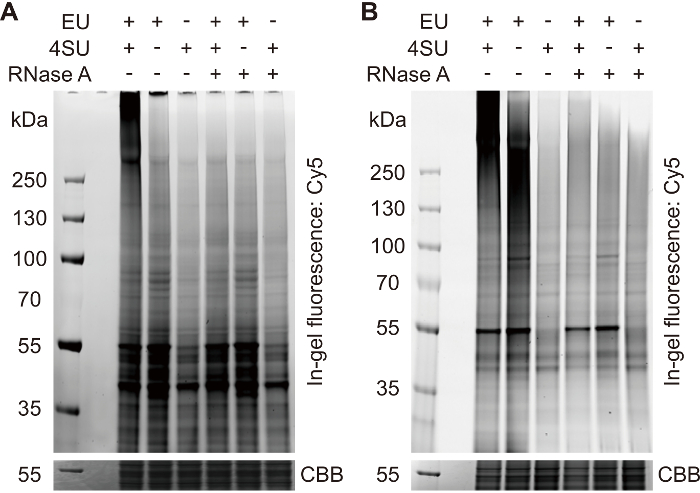
Figure 1 : analyse de la fluorescence des échantillons marqués au clic décrit à l’étape 2.3.2 en gel. (A) cette fluorescence montre un typique en gel de panneau plan des échantillons marqués au clic. Seul l’échantillon doublement marquée montre une bande forte frottis à poids moléculaire élevé (> 250 kDa), qui représente le signal du RNP réticulé. Pour supprimer le signal de RNP, omettez soit 4SU ou UE ou digérer avec de la RNase A. Les bandes de fond pointu à un poids moléculaire inférieur représentent les signaux d’étiqueté protéines non spécifiques. (B) dans certains cas, une forte bande enduite (~ 130-250 kDa) peut être observée dans l’échantillon de contrôle no4SU. Cette bande représente le signal de RNAs réticulés étiquetés, qui devrait être dégradé au cours de la dénaturation par la chaleur, pour la plupart des cas. Il n’interférera pas avec les procédures subséquentes. CBB = Coomassie brillant bleu. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
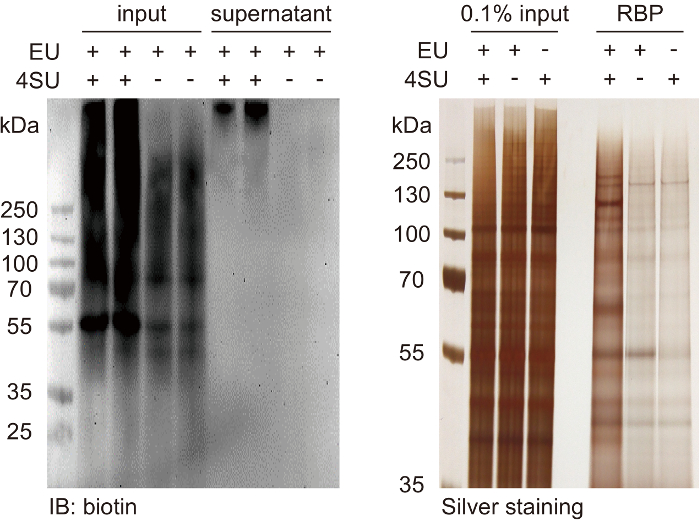
Figure 2 : contrôle de la qualité d’affinité pulldown efficacité et les pratiques commerciales restrictives capturées. (A), ce panneau présente une analyse par western blot de la biotine des signaux dans les échantillons avant pulldown (entrée) et dans les échantillons après pulldown (surnageant). Estimer le ratio des signaux restants et optimiser la quantité de perles utilisée à l’étape 3.1.1. (B) ce panneau montre une coloration à l’argent l’analyse des pratiques commerciales restrictives capturés par rapport aux protéines totales d’entrée 0,1 %. Pour les cellules HeLa, l’efficacité de captage total général est ~0.05% - 0,1 % de protéines d’entrée. Cette valeur peut varier considérablement, en raison de la variation de l’efficacité métabolique de marquage de différents types de cellules. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 3 : résultats représentant Mme de CARIC. (A), ce panneau indique une parcelle de volcan affichant la moyenne Log2-pli changer et ajusté les valeurs de P des protéines chiffrés, calculés par le paquet Leduc. 597 de protéines avec un changement de Log2-pli de > 2 et une valeur ajustée de P < 0,01 ont été classés comme « CARIC pratiques commerciales restrictives ». (B), ce panneau indique le chevauchement des protéines CARIC avec poly humain précédemment identifiés aux pratiques commerciales restrictives7,8,9,10,11. Les protéines se chevauchent codez principalement aux pratiques commerciales restrictives, alors que le reste des CARIC restrictives sont plus susceptibles d’être non codantes pratiques commerciales restrictives. Ce chiffre est un tiré à part de travaux déjà publié avec la permission de la National Academy of Sciences27. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Discussion
La préservation de l’intégrité de RNA juste est l’une des clés du succès CARIC expériences. Dégradation de l’ARN peut être significativement réduite avec les ligands appropriés de Cu (i) et opération minutieuse, bien qu’on observe une dégradation partielle. Les ratios de substitution d’UE et 4SU en échantillons expérimentaux sont 1,18 % et 0,46 %, respectivement (données non présentées). Pour ARN intact d’une longueur de 2 000 nt, environ 90 % des ARN contiennent au moins une UE et un 4SU. Pour ARN partiellement dégradés d’une longueur de 1 000 nt, environ 70 % des ARN contiennent au moins une UE et un 4SU. Donc, une dégradation partielle des ARN ne diminue pas considérablement l’efficacité de CARIC, dégradation sévère n’est pas acceptable.
Une autre étape critique est étape 1.4, la préparation de la réaction de clic. Le Cu (I)-réaction catalysée cliquez sur RNAs est sensible à la concentration de LDS. Une forte concentration (> 0,1 %) de LDS conduira à une diminution de l’étiquetage des signaux sur EU-contenant RNAs et une augmentation des signaux de fond sur les protéines (données non présentées).
En plus de l’UE, CARIC est également compatible avec les autres nucléosides cliquables, tels qu’alcynyles et azido analogues de l’adénosine33,34,35,36. Toutefois, l’application de CARIC est considérablement limitée par l’efficacité métabolique d’unnatural nucléosides cliquables dans un système biologique d’intérêt. Par conséquent, devant effectuer CARIC à l’aide de conditions autres que celles a démontré dans ce protocole, toujours vérifier l’efficacité métabolique étiquetage (p. ex., par imagerie de fluorescence).
Récemment, une stratégie similaire appelée RICK (capture de l’interactome RNA nouvellement transcrit à l’aide de la chimie de clic), qui incorpore uniquement EU pour étiqueter les ARN total et utilise UV 254 nm pour réticuler les ARN et les protéines, a été signalé37. Notamment, UV 254 nm peut activer toutes les quatre nucléosides naturels, ainsi que EU. Ainsi, l’irradiation UV 254 nm peut-être réticuler UE libre et ses métabolites (par exemple, les phosphates EU) avec des protéines de liaison correspondant, qui devraient être pris en considération comme possible de faux positifs.
Une application intéressante de CARIC est d’identifier les pratiques commerciales restrictives dans les bactéries dont RNAs sont pour la plupart non polyadénylé. L’identification à grande échelle des pratiques commerciales restrictives fournira des ressources inestimables pour comprendre les bases moléculaires des règlements post-transcriptionnelle en bactéries38.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail est soutenu par la National Natural Science Foundation de Chine subventions 91753206, 21425204 et 21521003 et par le National clé de recherche et projet de développement 2016YFA0501500.
matériels
| Name | Company | Catalog Number | Comments |
| HeLa | ATCC | ||
| DMEM (Dulbecco's Modified Eagle Medium) | Thermo Fisher Scientific | 11995065 | |
| FBS (Fetal Bovine Serum) | Thermo Fisher Scientific | 10099141 | |
| Penicillin & Streptomycin | Thermo Fisher Scientific | 15140122 | |
| EU (5-ethynyl uridine) | Wuhu Huaren Co. | CAS:69075-42-9 | |
| 4SU (4-thiouridine) | Sigma Aldrich | T4509 | |
| 10×PBS (Phosphate-Buffered Saline) | Thermo Fisher Scientific | AM9625 | |
| UV cross-linker | UVP | CL-1000 | Equiped with 365-nm UV lamp |
| DEPC (Diethyl pyrocarbonate) | Sigma Aldrich | D5758 | To treat water. Highly toxic! |
| Tris·HCl, pH 7.5 | Thermo Fisher Scientific | 15567027 | |
| LiCl | Sigma Aldrich | 62476 | |
| Nonidet P-40 | Biodee | 74385 | |
| EDTA-free protease inhibitor cocktail | Thermo Fisher Scientific | 88265 | One tablet for 50 mL lysis buffer. |
| LDS (Lithium dodecyl sulfate) | Sigma Aldrich | L9781 | |
| 15-mL ultrafiltration tube (10 kDa cutoff) | Millipore | UFC901024 | |
| 0.5-mL ultrafiltration tube (10 kDa cutoff) | Millipore | UFC501096 | |
| Streptavidin magnetic beads | Thermo Fisher Scientific | 88816 | |
| DMSO (Dimethyl sulfoxide) | Sigma Aldrich | 41639 | |
| Azide-biotin | Click Chemistry Tools | AZ104 | |
| Copper(II) sulfate | Sigma Aldrich | C1297 | |
| THPTA [Tris(3-hydroxypropyltriazolylmethyl)amine] | Sigma Aldrich | 762342 | |
| Sodium ascorbate | Sigma Aldrich | 11140 | |
| Azide-Cy5 | Click Chemistry Tools | AZ118 | |
| LDS sample buffer (4×) | Thermo Fisher Scientific | NP0008 | |
| 10% bis-Tris gel | Thermo Fisher Scientific | NP0301BOX | |
| EDTA | Thermo Fisher Scientific | AM9260G | |
| RNase A | Sigma Aldrich | R6513 | |
| SDS (Sodium dodecyl sulfate) | Thermo Fisher Scientific | 15525017 | |
| NaCl | Sigma Aldrich | S3014 | |
| Brij-97 [Polyoxyethylene (20) oleyl ether] | J&K | 315442 | |
| Triethanolamine | Sigma Aldrich | V900257 | |
| Streptavidin agarose | Thermo Fisher Scientific | 20353 | |
| Urea | Sigma Aldrich | U5378 | |
| Sarkosyl (N-Lauroylsarcosine sodium salt) | Sigma Aldrich | 61743 | |
| Biotin | Sigma Aldrich | B4501 | |
| Sodium deoxycholate | Sigma Aldrich | 30970 | |
| MaxQuant | Version: 1.5.5.1 |
Références
- Djebali, S., et al. Landscape of transcription in human cells. Nature. 489 (7414), 101-108 (2012).
- Gerstberger, S., Hafner, M., Tuschl, T. A census of human RNA-binding proteins. Nature Reviews Genetics. 15 (12), 829-845 (2014).
- Castello, A., Fischer, B., Hentze, M. W., Preiss, T. RNA-binding proteins in Mendelian disease. Trends in Genetics. 29 (5), 318-327 (2013).
- Nussbacher, J. K., Batra, R., Lagier-Tourenne, C., Yeo, G. W. RNA-binding proteins in neurodegeneration: Seq and you shall receive. Trends in Neuroscience. 38 (4), 226-236 (2015).
- Jazurek, M., Ciesiolka, A., Starega-Roslan, J., Bilinska, K., Krzyzosiak, W. J. Identifying proteins that bind to specific RNAs - focus on simple repeat expansion diseases. Nucleic Acids Research. 44 (19), 9050-9070 (2016).
- Hentze, M. W., Castello, A., Schwarzl, T., Preiss, T. A brave new world of RNA-binding proteins. Nature Reviews Molecular Cell Biology. 19 (5), 327-341 (2018).
- Castello, A., et al. Insights into RNA biology from an atlas of mammalian mRNA-binding proteins. Cell. 149 (6), 1393-1406 (2012).
- Baltz, A. G., et al. The mRNA-bound proteome and its global occupancy profile on protein-coding transcripts. Molecular Cell. 46 (5), 674-690 (2012).
- Beckmann, B. M., et al. The RNA-binding proteomes from yeast to man harbour conserved enigmRBPs. Nature Communications. 6, 10127-10135 (2015).
- Conrad, T., et al. Serial interactome capture of the human cell nucleus. Nature Communications. 7, 11212-11222 (2016).
- Castello, A., et al. Comprehensive identification of RNA-binding domains in human cells. Molecular Cell. 63 (4), 696-710 (2016).
- Kwon, S. C., et al. The RNA-binding protein repertoire of embryonic stem cells. Nature Structural & Molecular Biology. 20 (9), 1122-1130 (2013).
- Liepelt, A., et al. Identification of RNA-binding proteins in macrophages by interactome capture. Molecular & Cellular Proteomics. 15 (8), 2699-2714 (2016).
- Liao, Y., et al. The cardiomyocyte RNA-binding proteome: Links to intermediary metabolism and heart disease. Cell Reports. 16 (5), 1456-1469 (2016).
- Mitchell, S. F., Jain, S., She, M. P., Parker, R. Global analysis of yeast mRNPs. Nature Structural & Molecular Biology. 20 (1), 127-133 (2013).
- Matia-González, A. M., Laing, E. E., Gerber, A. P. Conserved mRNA-binding proteomes in eukaryotic organisms. Nature Structural & Molecular Biology. 22 (12), 1027-1033 (2015).
- Despic, V., et al. Dynamic RNA-protein interactions underlie the zebrafish maternal-to-zygotic transition. Genome Research. 27 (7), 1184-1194 (2017).
- Wessels, H. H., et al. The mRNA-bound proteome of the early fly embryo. Genome Research. 26 (7), 1000-1009 (2016).
- Sysoev, V. O., et al. Global changes of the RNA-bound proteome during the maternal-to-zygotic transition in Drosophila. Nature Communications. 7, 12128(2016).
- Reichel, M., et al. In planta determination of the mRNA-binding proteome of Arabidopsis etiolated seedlings. Plant Cell. 28 (10), 2435-2452 (2016).
- Marondedze, C., Thomas, L., Serrano, N. L., Lilley, K. S., Gehring, C. The RNA-binding protein repertoire of Arabidopsis thaliana. Scientific Reports. 6, 29766-29778 (2016).
- Zhang, Z., et al. UV crosslinked mRNA-binding proteins captured from leaf mesophyll protoplasts. Plant Methods. 12, 42-53 (2016).
- Bunnik, E. M., et al. The mRNA-bound proteome of the human malaria parasite Plasmodium falciparum. Genome Biology. 17, 147-164 (2016).
- Lueong, S., Merce, C., Fischer, B., Hoheisel, J. D., Erben, E. D. Gene expression regulatory networks in Trypanosoma brucei: insights into the role of the mRNA-binding proteome. Molecular Microbiology. 100 (3), 457-471 (2016).
- Nandan, D., et al. Comprehensive identification of mRNA-binding proteins of Leishmania donovani by interactome capture. PLoS ONE. 12 (1), e0170068(2017).
- Jankowsky, E., Harris, M. E. Specificity and nonspecificity in RNA-protein interactions. Nature Reviews Molecular Cell Biology. 16 (9), 533-544 (2015).
- Huang, R., Han, M., Meng, L., Chen, X. Transcriptome-wide discovery of coding and noncoding RNA-binding proteins. Proceedings of the National Academy of Sciences of the United States of America. 115 (17), E3879-E3887 (2018).
- Shevchenko, A., Tomas, H., Havlis, J., Olsen, J. V., Mann, M. In-gel digestion for mass spectrometric characterization of proteins and proteomes. Nature Protocols. 1 (6), 2856-2860 (2006).
- Boersema, P. J., Raijmakers, R., Lemeer, S., Mohammed, S., Heck, A. J. R. Multiplex peptide stable isotope dimethyl labeling for quantitative proteomics. Nature Protocols. 4 (4), 484-494 (2009).
- Rappsilber, J., Mann, M., Ishihama, Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics using StageTips. Nature Protocols. 2 (8), 1896-1906 (2007).
- Cox, J., Mann, M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nature Biotechnology. 26 (12), 1367-1372 (2008).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), e47(2015).
- Grammel, M., Hang, H., Conrad, N. K. Chemical reporters for monitoring RNA synthesis and poly(A) tail dynamics. ChemBioChem. 13 (8), 1112-1115 (2012).
- Curanovic, D., et al. Global profiling of stimulus-induced polyadenylation in cells using a poly(A) trap. Nature Chemical Biology. 9 (11), 671-673 (2013).
- Zheng, Y. X., Beal, P. A. Synthesis and evaluation of an alkyne-modified ATP analog for enzymatic incorporation into RNA. Bioorganic & Medicinal Chemistry Letters. 26 (7), 1799-1802 (2016).
- Nainar, S., et al. Metabolic incorporation of azide functionality into cellular RNA. ChemBioChem. 17 (22), 2149-2152 (2016).
- Bao, X., et al. Capturing the interactome of newly transcribed RNA. Nature Methods. 15 (3), 213-220 (2018).
- Holmqvist, E., Vogel, J. RNA-binding proteins in bacteria. Nature Reviews Microbiology. , Published online (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon